Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Uso de un espectrómetro de movilidad de iones cíclicos para experimentos de movilidad de iones en tándem
En este artículo
Resumen
La espectrometría de movilidad iónica (SGI) es un complemento interesante de la espectrometría de masas para la caracterización de biomoléculas, en particular porque es sensible al isomerismo. Este protocolo describe un experimento en tándem IMS (IMS/IMS), que permite el aislamiento de una molécula y la generación de los perfiles de movilidad de sus fragmentos.
Resumen
La caracterización precisa de las estructuras químicas es importante para comprender sus mecanismos biológicos subyacentes y propiedades funcionales. La espectrometría de masas (EM) es una herramienta popular, pero no siempre es suficiente para revelar completamente todas las características estructurales. Por ejemplo, aunque los carbohidratos son biológicamente relevantes, su caracterización se complica por numerosos niveles de isomerismo. La espectrometría de movilidad iónica (SGI) es un complemento interesante porque es sensible a las conformaciones iónicas y, por lo tanto, al isomerismo.
Además, los avances recientes han mejorado significativamente la técnica: la última generación de instrumentos IMS cíclicos ofrece capacidades adicionales en comparación con los instrumentos IMS lineales, como un mayor poder de resolución o la posibilidad de realizar experimentos de movilidad iónica en tándem (IMS / IMS). Durante IMS/IMS, un ion se selecciona en función de su movilidad iónica, fragmentado y reanalizado para obtener información sobre la movilidad iónica sobre sus fragmentos. Trabajos recientes mostraron que los perfiles de movilidad de los fragmentos contenidos en dichos datos IMS / IMS pueden actuar como una huella digital de un glicano en particular y pueden usarse en una estrategia de redes moleculares para organizar conjuntos de datos glucómicos de una manera estructuralmente relevante.
El objetivo de este protocolo es, por lo tanto, describir cómo generar datos IMS / IMS, desde la preparación de la muestra hasta la calibración final de la sección transversal de colisión (CCS) de la dimensión de movilidad iónica que produce espectros reproducibles. Tomando el ejemplo de un glicano representativo, este protocolo mostrará cómo construir una secuencia de control IMS / IMS en un instrumento IMS cíclico, cómo contabilizar esta secuencia de control para traducir el tiempo de llegada de IMS en tiempo de deriva (es decir, el tiempo de separación efectivo aplicado a los iones) y cómo extraer la información de movilidad relevante de los datos sin procesar. Este protocolo está diseñado para explicar claramente los puntos críticos de un experimento IMS/IMS y, por lo tanto, ayudar a los nuevos usuarios de IMS cíclico a realizar adquisiciones sencillas y reproducibles.
Introducción
La caracterización química completa de las biomoléculas es clave para comprender sus propiedades biológicas y funcionales subyacentes. Con este fin, las ciencias "ómicas" se han desarrollado en los últimos años, con el objetivo de la caracterización a gran escala de estructuras químicas en concentraciones biológicas. En proteómica y metabolómica, la EM se ha convertido en una herramienta central para desentrañar la heterogeneidad estructural que se encuentra en los medios biológicos, especialmente gracias a su sensibilidad y capacidad para proporcionar información estructural a través de la EM en tándem (EM / EM). En las estrategias de EM/EM, un ion se selecciona de acuerdo con su masa, luego se fragmenta y, finalmente, se adquieren las masas de sus fragmentos para establecer una huella digital de la molécula. Los espectros MS/MS pueden, en particular, utilizarse para hacer coincidir bases de datos espectrales1,2, o reconstruir tentativamente las estructuras madre3,4. Bajo el supuesto de que espectros similares pertenecen a compuestos similares, los datos de MS/MS también se pueden utilizar para construir redes moleculares (MNs) que conecten especies relacionadas a través de una puntuación de similitud5,6.
Sin embargo, debido a la propiedad inherente de la EM de detectar la relación masa-carga (m/z) de los iones, la técnica es ciega a una serie de características estructurales que caen dentro del rango del (estereo)isomerismo. Por ejemplo, los carbohidratos están hechos de varias subunidades de monosacáridos, muchas de las cuales son estereoisómeros o incluso epímeros (por ejemplo, Glc vs. Gal o Glc vs. Man). Estas subunidades están unidas por enlaces glicosídicos, que pueden diferir por la posición del enlace (regioisomerismo) y la configuración estérica del carbono anomérico (anomerismo). Estas características dificultan que la EM independiente distinga entre isómeros de carbohidratos7, y solo el regioisomerismo puede abordarse utilizando métodos de activación de alta energía8,9,10. Aunque la derivatización es una opción para interrumpir la equivalencia de los grupos estereoisoméricos11, requiere una extensa preparación de la muestra. Otra opción más sencilla es acoplar la EM con una dimensión analítica sensible al isomerismo, como la IMS.
Debido a que este protocolo está diseñado para usuarios que ya están familiarizados con los conceptos básicos de IMS, y debido a que las revisiones detalladas están disponibles en otros lugares12,13, solo se ofrece aquí una breve descripción de los principios de IMS. IMS es un método de separación en fase gaseosa que se basa en la interacción de iones con un gas tampón y un campo eléctrico, separando en última instancia los iones de acuerdo con sus conformaciones en fase gaseosa. Se pueden encontrar diferentes principios de IMS acoplados a MS en instrumentos comerciales: algunos operan en campos eléctricos altos y bajos alternantes (IMS asimétrico de campo, FAIMS), mientras que la mayoría opera dentro del límite de campo bajo, especialmente IMS de tubo de deriva (DTIMS, campo eléctrico linealmente decreciente), IMS de onda viajera (TWIMS, ondas de potencial simétricas) e IMS atrapado (TIMS, alto flujo de iones de captura de gas amortiguador contra campos eléctricos)13 . Los métodos de campo bajo permiten el acceso a un llamado CCS, una propiedad del par ion-gas que representa la superficie (en Å2 o nm2) del ion que interactúa con el gas tampón durante la separación. La CAC es teóricamente independiente del instrumento y, por lo tanto, es útil para generar datos que pueden reproducirse entre diferentes laboratorios14. Las separaciones de movilidad iónica pueden verse afectadas por varios parámetros y, en particular, por las fluctuaciones de la presión del gas y la temperatura del gas en la celda de movilidad. La calibración CCS es una forma de remediar esto, ya que tanto el calibrante como las especies de interés se verán afectados de manera similar13. Sin embargo, es obligatorio instalar el instrumento en una habitación con temperatura controlada y tener un sistema de control de presión de gas confiable.
Una evolución interesante de IMS es IMS/IMS, que fue introducido por primera vez en 2006 por el grupo de Clemmer como un análogo de MS/MS15,16. En IMS/IMS, un ion de interés se aísla selectivamente en función de su movilidad iónica; luego se activa (hasta una posible fragmentación), y se realiza un nuevo análisis IMS del ion o fragmentos activados. En el primer diseño instrumental, se colocaron dos células IMS en serie, separadas por un embudo iónico donde se encontraba la activación. Desde entonces, aunque se propusieron varias configuraciones IMS / IMS (para una revisión, consulte Eldrid y Thalassinos17), el primer espectrómetro de masas comercial con capacidad IMS / IMS solo estuvo disponible en 201918. Este instrumento mejoró sustancialmente el concepto inicial combinándolo con otro avance tecnológico: un diseño cíclico de la célula IMS.
La célula IMS cíclica teóricamente permite aumentar casi infinitamente la longitud de la trayectoria de deriva y, por lo tanto, el poder de resolución del instrumento19. Esto se logró por medio de una geometría de instrumento particular, donde la celda cíclica TWIMS se coloca ortogonalmente al eje óptico iónico principal. Una región de matriz multifunción en la entrada de la celda IMS permite controlar la dirección de la ruta iónica: (i) enviar iones hacia los lados para la separación IMS, (ii) hacia adelante para la detección de MS, o (iii) hacia atrás desde la celda IMS para ser almacenada en una celda de prearray. A partir de esta célula de almacenamiento previo al inicio, se pueden activar los iones y reinyectar los fragmentos en la célula IMS para la medición de la movilidad iónica, un enfoque que se ha utilizado con éxito para caracterizar estereoisómeros20. En última instancia, los datos recopilados contienen movilidad iónica e información m/z para el precursor y sus fragmentos.
En una publicación reciente que utilizó este diseño cíclico para análisis de glicanos (Ollivier et al.21), demostramos que el perfil de movilidad de los fragmentos contenidos en dichos datos IMS/IMS actúa como una huella dactilar de una biomolécula que puede ser utilizada en una estrategia de redes moleculares. La red resultante, llamada IM-MN, condujo a la organización de conjuntos de datos glucómicos de una manera estructuralmente relevante, mientras que la red construida únicamente a partir de datos de MS / MS (MS-MN) reveló poca información. Para complementar esta publicación y ayudar a los usuarios de Cyclic IMS a implementar este flujo de trabajo, este protocolo proporciona una descripción completa del protocolo utilizado para recopilar los datos. Este protocolo se centra únicamente en la generación de los datos IMS/IMS que los usuarios pueden utilizar para construir redes IM-MN (véase 21), o para cualquier otra aplicación de su elección. La construcción de IM-MN no se considerará en este documento, ya que los protocolos para redes moleculares ya están disponibles22. Se destacan los puntos cruciales que deben seguirse para generar adquisiciones IMS/IMS valiosas y reproducibles. Tomando el ejemplo de uno de los oligosacáridos estudiados por Ollivier et al. 21, se detallan los siguientes pasos: (i) preparación de la muestra, (ii) ajuste del instrumento Cyclic IMS, (iii) selección automática de picos de los datos y (iv) calibración CCS.
Protocolo
NOTA: En la Figura 1 se proporciona una descripción general del protocolo. Los parámetros utilizados para los experimentos descritos en el presente protocolo se pueden encontrar en la Tabla Suplementaria S1 y la Tabla Suplementaria S2.
1. Preparación de la solución de muestra
NOTA: El protocolo se describe utilizando un pentasacárido arabinoxilano (23-α-L-arabinofuranosil-xilotetraosa o XA2XX; ver la Tabla de Materiales) como ejemplo.
- Preparación del disolvente: 500 μM LiCl en 50:50 H2O:MeOH (vol./vol.).
- Prepare una solución madre de 100 mM de cloruro de litio (LiCl) en H2O pesando 212 mg de LiCl y agregue 50 ml de agua desionizada de alta pureza (H2O) en un tubo cónico de polipropileno de 50 ml. Agitar hasta que se disuelva por completo.
NOTA: El disolvente se dopa con una sal de litio para promover la formación de aductos [M + Li] + en la fuente de iones del espectrómetro, ya que generalmente produce espectros de fragmentación de mejor calidad en comparación con otros aductos alcalinos. Se recomienda el uso de LiCl porque previamente se ha encontrado que los ácidos orgánicos (y por lo tanto sus sales) afectan los perfiles IMS23. - En una botella de vidrio, diluya la solución madre de LiCl 200x: a 250 μL de la solución madre, agregue 24.75 mL de H2O. Agregue 25 mL de metanol (MeOH) para alcanzar una concentración final de LiCl de 500 μM en 50:50 H2O:MeOH (v/v). Sonicar durante 2 min para desgasificar el disolvente.
NOTA: MeOH presenta un peligro para la salud (H225, H301, H311, H331, H370); manipular debajo de una campana extractora usando una bata de laboratorio, guantes y protección ocular. Una proporción de 50:50 MeOH/H2O (v/v) parece ser el mejor disolvente para la ionización de oligosacáridos; sin embargo, el MeOH puede ser sustituido por acetonitrilo (ACN) si es necesario.
- Prepare una solución madre de 100 mM de cloruro de litio (LiCl) en H2O pesando 212 mg de LiCl y agregue 50 ml de agua desionizada de alta pureza (H2O) en un tubo cónico de polipropileno de 50 ml. Agitar hasta que se disuelva por completo.
- En un tubo de polipropileno de 1,5 ml, pese 1 mg del carbohidrato. Disolver con un volumen apropiado de 500 μM LiCl para alcanzar una concentración de 1 mg/ml. Diluir a una concentración final de 10 μg/mL en 50:50 MeOH/H2O + 500 μM LiCl. Conservar a 4 °C.
NOTA: La concentración de 10 μg/mL fue elegida para optimizar la señal sobre todos los iones fragmentos durante IMS/IMS-MS (esto es para un compuesto puro; aumentar la concentración cuando se trabaja en mezclas). Para la adquisición de espectros IMS/IMS de referencia, no diluya más la muestra: se espera la saturación del detector MS antes de la fragmentación, aunque el instrumento ofrece opciones para corregirla (ver paso 3.2.).
2. Ajuste del espectrómetro de masas Cíclico IMS
NOTA: Las instrucciones relacionadas con el software (ventanas, menús y comandos) se resaltan en negrita.
- Abra la consola del instrumento desde el software de control del instrumento (página de afinación de MS , consulte los detalles del software en la Tabla de materiales) y coloque el instrumento en modo de funcionamiento . Espere al menos 3 h para que los altos voltajes se estabilicen en la celda IMS.
NOTA: Para obtener la mejor reproducibilidad, los voltajes en la celda IMS deben estabilizarse por completo. Encienda los altos voltajes y deje que el instrumento se estabilice durante la noche antes de cualquier análisis cíclico de IMS. Además, la presión y la temperatura en la celda de movilidad iónica deben mantenerse lo más constantes posible. Aunque hay disponible una lectura de la presión en la pestaña Vacío , no hay ninguna lectura disponible para la temperatura. Mantenga el instrumento en un laboratorio termostatizado. El instrumento utilizado en este trabajo funciona a 1,75 mbar en un laboratorio termostatizado a 20 °C. - Configuración del instrumento IMS cíclico
NOTA: Las soluciones estándar deben infundirse utilizando el sistema de fluidos incorporado para la configuración del instrumento.- Coloque los recipientes de fluidos llenos con las normas apropiadas proporcionadas por el fabricante en el sistema de fluidos: Reservorio B ('Lockmass'): 10 pg/μL de leucina encefalina (LEU ENK) en 50:50 ACN/H2O + 0,1% de ácido fórmico; Reservorio C ('Calibrante'): MajorMix.
NOTA: En este protocolo, la solución de calibración MajorMix se utilizará para calibrar las dimensiones m/z y CCS. Por razones prácticas, se realizará una calibración CCS externa (consulte el paso 5 del protocolo); por lo tanto, también es posible utilizar una mezcla de calibrante interno para el CCS y otro calibrante para el m/z (por ejemplo, formiato de sodio o yoduro de sodio). - En la página Ajustar de la consola de Quartz, vaya a la pestaña Fluidics . Ajuste los fluidos de la muestra al reservorio C y los fluidos de referencia al reservorio B. Infundir ambas soluciones consecutivamente en la fuente de iones para verificar la señal MS.
- Realice la configuración de ADC, la configuración del detector (utilizando LEU ENK) y la calibración de masa (consulte la Tabla de materiales para la solución de calibración) desde la página Configuración del instrumento de acuerdo con las instrucciones del fabricante.
- Coloque los recipientes de fluidos llenos con las normas apropiadas proporcionadas por el fabricante en el sistema de fluidos: Reservorio B ('Lockmass'): 10 pg/μL de leucina encefalina (LEU ENK) en 50:50 ACN/H2O + 0,1% de ácido fórmico; Reservorio C ('Calibrante'): MajorMix.
- Registre una adquisición IMS de la solución de calibración con una separación de una sola pasada (utilícela para la calibración IMS externa).
NOTA: La fuente iónica y los parámetros de onda viajera (TW) (altura de onda estática y velocidad de onda) deben mantenerse constantes durante todas las adquisiciones (calibración y adquisiciones). Si el usuario no tiene conocimiento previo de los parámetros óptimos para su muestra, este paso se puede realizar después del paso 3 del protocolo (para los aductos [M + Li] + de oligosacáridos neutros, los resultados representativos utilizan una altura TW de 16 V y una velocidad TW de 350 m / s, que dan los mejores resultados).- En la pestaña Fluidics , seleccione la posición del deflector Muestra e infunda el calibrante (consulte la Tabla de materiales) en la fuente de iones (utilizando el sistema fluídico incorporado) a través de la sonda 'Muestra' a un caudal de 10 μL/min.
- Configure una secuencia IMS de una sola pasada. En la página Afinar , coloque el instrumento en modo Movilidad y abra la ventana Control de secuencia cíclica . Seleccione Modo avanzado . En la pestaña Funciones cíclicas de esta nueva ventana, seleccione Agregar paquete y, a continuación, Single/Multipass. Espere a que aparezca una secuencia de eventos de movilidad en la pestaña Secuencia de la misma ventana.
NOTA: Para activar la visualización en tiempo real, el usuario debe aplicar los parámetros del instrumento: haga clic en Sintonizar en modo TOF o Ejecutar en modo Movilidad . Antes de cambiar el instrumento entre los modos TOF y Movilidad , es necesario abortar cualquier adquisición en ejecución (incluida la visualización de la página Sintonizar ). La abundancia relativa de iones puede variar entre el modo TOF y el modo Movilidad debido a los cambios en los parámetros de transmisión de iones. - Adapte la secuencia para que todos los iones calibrantes hagan una sola pasada alrededor de la pista de carreras cíclica IMS. No cambie el tiempo de inyección o la expulsión y adquiera tiempo; sin embargo, reduzca el tiempo de separación a 1 ms (en la ficha Secuencia ). Si algunos iones de la mezcla de calibración no caben en la ventana de tiempo de llegada mostrada, cambie la sincronización del IMS con el pulsador del analizador TOF de aceleración ortogonal aumentando el número de pulsaciones por bandeja en la pestaña Configuración de ADC .
NOTA: Los tiempos en la secuencia de control sólo controlan la matriz multifunción para la apertura de iones. Mientras los iones estén comprometidos en su primer (o enésimo) paso alrededor de la pista de carreras, terminarán dicho paso incluso si la dirección del TW ha cambiado en la matriz mientras tanto. Reducir el tiempo de separación a 1 ms significa que la matriz cambiará al modo de eyección después de 1 ms. Esto asegura que los iones más rápidos no tendrán tiempo suficiente para pasar a través de la matriz y participar en una segunda pasada antes de que los iones más lentos terminen su primera pasada. Por lo tanto, todos los iones estarán sujetos al mismo número de pasadas (es decir, una pasada), que es necesario para realizar la calibración IMS. - Registre una adquisición de 2 minutos. En la ventana Control de secuencia cíclica , haga clic en Adquirir para abrir la ventana emergente Configuración de adquisición . Introduzca el nombre de archivo, la descripción y la duración de la adquisición (minutos) y haga clic en Guardar.
- Registre otros 2 minutos de adquisición de la solución de calibración en las mismas condiciones que el paso 2.3 (utilícelo para comprobar la calidad de la calibración CCS). En la ventana Control de secuencia cíclica , haga clic en Adquirir para abrir la ventana emergente Configuración de adquisición . Introduzca el nombre de archivo, la descripción y la duración de la adquisición (minutos) y haga clic en Guardar.
- Lave bien el sistema de fluidos con H2O/ACN 50:50 para evitar la cristalización del calibrante en el tubo de observación.
3. Adquisición de IMS/IMS-MS
- Usando una bomba de jeringa, infundir la muestra (dopada con litio) a 10 μg/mL a través de la sonda de muestra a un caudal de 10 μL/min.
- Cambie el instrumento al modo TOF (desde la página de sintonización de MS ) para comprobar la estabilidad de la señal. Registre una adquisición completa de EM (1 min) de la muestra, lo que será útil para verificar el patrón isotópico y la presencia de contaminantes potenciales.
NOTA: Debido a que la concentración de la muestra se elige para obtener una buena señal de iones para los fragmentos, se puede observar una saturación de TOF en este paso. La saturación de TOF se puede identificar utilizando los siguientes artefactos: (i) una resolución de EM aumentada artificialmente, (ii) un cambio en las relaciones isotópicas y (iii) una multitud de picos de baja abundancia entre isótopos. Utilice la lente DRE (dynamic range enhancement, quad/MS Profile/DRE tab de la página principal Tune) para atenuar la transmisión de iones y descartar la saturación en modo TOF (Figura 2A,B). - Coloque el instrumento en modo MSMS (pestaña Quad/MS Profile de la página principal Tune ) y seleccione la masa del ion objetivo en el campo MSMS Mass para su aislamiento en el cuadrupolo (en el ejemplo: m/z de 685.2, correspondiente a la especie iónica [M+Li]+ del pentasacárido arabinoxilano). Registre una adquisición de 1 minuto para verificar el aislamiento del precursor al procesar los datos.
NOTA: Los aductos de litio tienen un isótopo a -1 Da del pico monoisotópico, que debe eliminarse de la ventana de selección de MS / MS para que no interfiera con los pasos de procesamiento. Se puede eliminar reduciendo el rango de selección utilizando los parámetros Resolución LM y Resolución HM en la pestaña Perfil Quad/MS (Figura 2C). - Configure una secuencia IMS de "corte" para realizar una selección basada en la movilidad del isómero de interés.
- Cambie el instrumento al modo Movilidad (véase el paso 2.3.2). En la ventana Control de secuencia cíclica , en la ficha Funciones cíclicas , seleccione Agregar paquete y, a continuación, Segmentación. Espere a que aparezca una secuencia compleja de eventos de movilidad en la ficha Secuencia (Figura 3).
NOTA: Es posible visualizar cada paso del proceso IMS/IMS: haga clic en el evento Expulsar y adquirir en la pestaña Secuencia . Una vez resaltado en rojo, muévalo a la posición apropiada dentro de la secuencia usando los botones Arriba y Abajo . - Coloque el evento Expulsar y adquirir justo después del primer evento Separado (es decir, muévalo a la fila 3 en lugar de a la fila 8 de la secuencia como se muestra en la figura 3) y, a continuación, haga clic en Ejecutar. Busque los resultados de la separación inicial para que se muestren en tiempo real. Aumente la duración del primer evento Separado para una separación multipaso cambiando el valor de tiempo para este evento en la secuencia hasta que la resolución de los picos IMS sea satisfactoria. Registre una adquisición de 1 minuto como referencia.
NOTA: Tome nota del valor retraso de inicio de ADC en la ficha Configuración de ADC : será útil comprobar la calidad del aislamiento. - Haga clic en Pausa. Tenga en cuenta que se muestran los resultados de la separación inicial, aunque las modificaciones en la secuencia de control no se aplicarán hasta que el usuario vuelva a hacer clic en Ejecutar . Coloque el evento Expulsar y adquirir debajo de los eventos Expulsar, Expulsar a Pretienda y Mantener y expulsar . Ajuste la duración de los eventos para que el pico objetivo esté en la región Expulsar a Pre-Almacenar , y cualquier otro ion esté en la región Expulsar o Mantener y expulsar .
NOTA: La duración de estos tres eventos en comparación con las distribuciones de tiempo de llegada (ATD) se puede visualizar utilizando la barra codificada por colores debajo del espectro de movilidad en la pestaña Mobilogram (Figura 3). - Coloque el evento Expulsar y adquirir al final de la secuencia, debajo de los eventos Reinyect from Pre-Store y second Separate . Haga clic en Ejecutar para mostrar la población seleccionada.
NOTA: Debido a que la población seleccionada ha abandonado la celda IMS, se ha perdido toda la separación anterior y vuelve a una separación de una sola pasada (que se desea). - Compruebe la calidad del aislamiento. Para verificar que solo se ha seleccionado el pico de interés, realice la misma separación después de la reinyección que antes de la reinyección (es decir, el mismo tiempo separado ) como se muestra en la Figura 4. Registre una adquisición de 1 minuto como referencia.
NOTA: Se anima a los usuarios a comprobar la población expulsada; la ventana de tiempo Expulsar a la tienda previa debe ser el nivel de línea base (Figura 4B). Para comprobarlo, coloque el retraso de inicio de ADC en modo manual en la pestaña Configuración de ADC e introduzca el tiempo de retraso indicado en el paso 3.4.2. Registre una adquisición de 1 minuto como referencia. - En la pestaña Secuencia , en la columna situada junto a los tiempos de evento definidos por el usuario (la columna Tiempo abs , resaltada en rojo), busque los tiempos sumados de todos los eventos. Tome nota de los Abdominales de tiempo que se encuentran en la línea del evento Reinject from Pre-Store para realizar la calibración CCS.
- Cambie el instrumento al modo Movilidad (véase el paso 2.3.2). En la ventana Control de secuencia cíclica , en la ficha Funciones cíclicas , seleccione Agregar paquete y, a continuación, Segmentación. Espere a que aparezca una secuencia compleja de eventos de movilidad en la ficha Secuencia (Figura 3).
- Fragmente el pico objetivo entre las dos rondas de IMS. Cambie los voltajes del paso de reinyección para aumentar la energía cinética de los iones y fragmentarlos al colisionar con el gas de movilidad iónica.
- Establezca la duración del evento Separado que precede directamente a expulsión y adquisición en 1 ms (consulte la explicación en el paso 2.3.3).
- En la línea Reinyect from Pre-Store , marque la casilla Habilitar activación y optimice la fragmentación con el control integrado. Si el espectro es satisfactorio (por ejemplo, el pico base es un fragmento), proceda directamente al paso 3.5.4.
NOTA: Al habilitar la activación, tres voltajes en la línea se volverán grises: estos son los voltajes que el usuario necesita cambiar si se requiere la optimización manual de los voltajes (consulte el siguiente paso). Estos tres voltajes (Pre-Array Gradient, Pre-Array Bias y Array Offset) forman el gradiente utilizado para activar los iones. La energía cinética de los iones aumentará con la pendiente entre el sesgo pre-array y el desplazamiento de la matriz (ver Figura 5). Los valores predeterminados de los valores gradiente → sesgo → desvío son: sin activación 85 → 70 → 45 V; activación máxima de la función incorporada 185 → 170 → -5 V (+150 V). Después de la fragmentación, no olvide reajustar la transmisión de iones utilizando la lente DRE (disminuir la atenuación de la señal) (consulte el paso 3.2.). - Si la fragmentación no es satisfactoria con el control incorporado, desmarque la casilla Habilitar activación y proceda a optimizar manualmente los voltajes de reinyección. Aumente el voltaje de gradiente previo a la matriz (el voltaje de polarización previa a la matriz siempre debe mantenerse 15 V por debajo del gradiente de la matriz) y reduzca el voltaje de compensación de la matriz (que se puede establecer como negativo) hasta que los resultados sean satisfactorios.
NOTA: Al ajustar manualmente los voltajes de la matriz multifunción, el usuario puede cambiar de la vista 'Mobilogram' a esquemas interactivos de los voltajes aplicados en la matriz multifunción (diagrama PE) para visualizar mejor la configuración de voltaje (Figura 5A). - Registre una adquisición de 2 minutos. En la ventana emergente de adquisición, marque la opción Retener tiempo de deriva para generar un archivo que contenga solo los tiempos de llegada frente a m / z (el tiempo de adquisición utilizado para los análisis cromatográficos, el tiempo de retención, se elimina del archivo). Tenga en cuenta que este archivo está etiquetado como *_dt. CRUDO.
NOTA: Si el usuario olvida marcar la opción Retener tiempo de deriva , aún es posible extraer la dimensión IMS utilizando el software Driftscope 2.9 (File | Exportar a MassLynx | Retener el tiempo de deriva).
- Vuelva el instrumento al modo TOF en la página principal de afinación y enjuague bien el sistema con 50:50 MeOH/H2O antes de continuar con la siguiente muestra.
4. Procesamiento IMS/IMS-MS con MZmine 224
NOTA: MZmine 2 está disponible desde la URL dada en la Tabla de Materiales. Se recomienda el uso de MZmine 2.51. En el momento de la preparación de este manuscrito, las versiones posteriores no pueden abrir archivos RAW desde los instrumentos Cyclic IMS debido a un cambio en la función de importación.
- Importe los archivos sin procesar que contengan solo las dimensiones IMS y m/z (*_dt. RAW) utilizando métodos de datos Raw | Importación de datos sin procesar.
NOTA: Los archivos raw aparecerán en el lado izquierdo de la ventana principal de MZmine. No importe el original *. Archivos RAW que aún contienen la dimensión de tiempo de retención. MZmine no distingue el tiempo de retención del tiempo de llegada de IMS, y los puntos de datos de ambas dimensiones se superpondrían. - Optimice los parámetros del flujo de trabajo en un archivo representativo seleccionándolo en el cuadro Archivos de datos sin procesar lista.
- Evaluar el nivel de ruido en los datos. Haga clic con el botón derecho en el archivo en la lista Archivos de datos sin procesar , seleccione Mostrar TIC y muestre el pico base "cromatograma" (BPC). Haga doble clic en el pico más pequeño observable a simple vista para mostrar su espectro de masas. Considere que el nivel de ruido en los datos está alrededor del del segundo isótopo del pico base en este espectro, y use este mismo valor para todos los umbrales de intensidad en los siguientes pasos de procesamiento.
NOTA: Los datos se adquirieron mediante aislamiento cuadrupolo y, por lo tanto, MZmine los considera como MS / MS. A lo largo de todo el procesamiento de MZmine, asegúrese de trabajar a nivel de MS = 2. - Realizar la detección masiva utilizando métodos de datos raw | | de detección de características Detección de masas. Para los datos adquiridos en modo de perfil, utilice el algoritmo de transformación de Wavelet . Para configurar los parámetros de los algoritmos en MZmine, haga clic en el botón [...] junto al algoritmo y use la opción Mostrar vista previa para visualizar los datos mientras optimiza los parámetros.
NOTA: En esta etapa, los picos seleccionados por el algoritmo aparecerán en rojo en la ventana de vista previa. Cuando se utiliza el algoritmo de transformación wavelet en archivos RAW propietarios, MZmine a veces confunde los puntos de datos de perfil con picos centroides. El software mostrará un mensaje que indica que el usuario está ejecutando un algoritmo de perfil en espectros centroides: ignore este mensaje y haga clic en Aceptar. - Reconstruir los espectros de movilidad iónica (EIM) extraídos para cada masa de fragmento utilizando métodos de datos raw | | de detección de características Constructor de cromatogramas ADAP en la lista de masas 'masas' generada por el paso anterior. Como la entrada de tolerancia m / z en esta etapa es una tolerancia de escaneo a escaneo, asegúrese de dejarla al menos 3-4 veces más alta que la precisión general esperada.
- Como el paso anterior no tiene una opción de vista previa, verifique la calidad de la selección máxima directamente utilizando la lista de características que apareció en el panel derecho de la ventana principal de MZmine. Abra la lista Características, seleccione todas las filas, haga clic con el botón derecho y seleccione Mostrar/XIC (cuadro de diálogo). Haga clic en Todo para mostrar todos los iones en el espectro de movilidad. Inspeccione los picos seleccionados que aparecen en color para asegurarse de que no haya picos perdidos obvios.
- Desconvolucionar los EIM para dividir los m/z que contienen diferentes picos en múltiples características. Usar métodos de lista de características | | de detección de características Deconvolución cromatográfica y elija el algoritmo Wavelets (ADAP ). Optimice el algoritmo para los datos utilizando la opción Mostrar vista previa y los siguientes parámetros clave: umbral S/N, umbral de coeficiente/área y rango de wavelet RT.
NOTA: Se recomienda verificar el aspecto del espectro descontorneado. Utilice la herramienta de visualización de cromatogramas, como se describe en el paso 4.2.4. Los picos descontorneados aparecerán en color, y los picos de la misma masa deben dividirse, como se presenta en la Figura 6A. - Desisótopo los EIM descontorneados utilizando métodos de lista de características | Isótopos | Picos isotópicos mero. Utilice la precisión esperada del instrumento para el valor de tolerancia m/z y establezca la tolerancia de tiempo de llegada en 0,1 ms (que se muestra en MZmine como tolerancia de tiempo de retención 0,1 min), ya que los isótopos no se resuelven durante la separación de IMS. Compruebe la lista de características: si queda algún isótopo, aumente los valores de tolerancia.
NOTA: Aunque el desisotopado teóricamente se puede realizar en cualquier momento del procesamiento de la lista de características, es importante hacerlo al final para que los valores de carga se puedan exportar (los algoritmos utilizados para los otros pasos a veces eliminarán la información del estado de carga).
- Evaluar el nivel de ruido en los datos. Haga clic con el botón derecho en el archivo en la lista Archivos de datos sin procesar , seleccione Mostrar TIC y muestre el pico base "cromatograma" (BPC). Haga doble clic en el pico más pequeño observable a simple vista para mostrar su espectro de masas. Considere que el nivel de ruido en los datos está alrededor del del segundo isótopo del pico base en este espectro, y use este mismo valor para todos los umbrales de intensidad en los siguientes pasos de procesamiento.
- Si procesa varios espectros IMS/IMS-MS, repita el procesamiento con estos parámetros optimizados. Mantenga los mismos parámetros para todos los espectros.
- En el caso de espectros múltiples, agruparlos en una sola tabla para exportarlos; de lo contrario, vaya directamente al paso 4.5. Para agrupar los espectros, utilice métodos de lista de características | Alineación | Alineador de unión. Debido a que el objetivo no es alinear realmente los picos, use valores de tolerancia restrictivos tanto para m / z como para la hora de llegada. Dar el mismo peso a ambas dimensiones.
- Exporte la lista final de características a un archivo *.csv . Usar métodos de lista de características | | de exportación/importación Exporte a un archivo CSV y exporte los siguientes valores: Exportar fila m/z, Exportar tiempo de retención de filas (la hora real de llegada de IMS), Pico m/z y Altura máxima. Utilice una coma como separador de campos.
5. TWCCSN2 de los espectros IMS/IMS centroides
NOTA: En este protocolo, se utilizará una calibración de ajuste logarítmico25,26, que tiende a dar mejores resultados que la calibración lineal y es fácil de implementar en una hoja de cálculo o un script de procesamiento interno. Un script interno (escrito en R) está disponible en la URL dada en la Tabla de Materiales.
- Elija los valores de tiempo de llegada de referencia de la adquisición del calibrante (consulte el paso 2.3). Haga esto manualmente utilizando el software del constructor (consulte la Tabla de materiales) para verificar el aspecto de todos los picos del calibrante IMS.
- En la ventana Cromatograma , abra el *_dt. Archivo RAW correspondiente al calibrante.
- Para cada punto de calibración, genere el EIM utilizando el | de visualización Opción de masa.
- Compruebe el perfil de las EIE. Si algunos están mal definidos, ajústelos utilizando el | Opción suave (ya que los mejores resultados se obtienen típicamente con el algoritmo Savitzky-Golay, suave 2 veces en 3 contenedores). Informe de los valores del ápice en una hoja de cálculo.
NOTA: Debido a que los puntos de referencia generalmente se adquieren utilizando dispositivos DTIMS de baja resolución, algunas distribuciones multimodales pueden aparecer en Cyclic IMS dependiendo de los calibrantes. Elimine cualquier pico que presente dicha distribución de la lista de calibración.
- Calcule los parámetros de ajuste logarítmico a partir de los calibrantes.
- Para todos los puntos de calibración, calcule lo siguiente.
- Calcule el tiempo de deriva usando Eq (1):
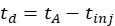 (1)
(1)
con td el tiempo de deriva, tA el tiempo de llegada medido, y tinj el tiempo de inyección en la célula IMS (todo en ms).
NOTA: Para moléculas pequeñas, como fragmentos de oligosacáridos, la variación del tiempo muerto (tiempo de vuelo entre la salida de la célula IMS y el detector) entre diferentes masas está dentro del rango de error de la calibración CCS y puede ignorarse. - Calcule la masa neutra de los iones usando Eq (2):
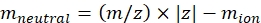 (2)
(2)
con z el estado de carga del ion, y mion la masa del contra-ion (en Da). Utiliza masas exactas para evitar introducir incertidumbre. Si hay una pérdida de átomos en lugar de un contra-ion, use valores de iones negativos (por ejemplo, para [M-H]-mneutral = (m/z) * |z| - (- 1.007276) = (m/z) * |z| + 1.007276). - Calcule el parámetro CCS usando Eq (3):
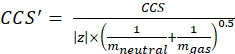 (3)
(3)
con CCS el valor de referencia del tubo de deriva DTCCSN2 (en nm2), y mgas la masa del gas de deriva (en Da; ej. para el nitrógeno: mgas = 28,01 Da). - Calcule el parámetro td' usando Eq (4):
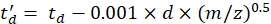 (4)
(4)
con d el retardo de inicio del detector utilizado experimentalmente para corregir el tiempo muerto (típicamente ~ 1.5 ms). - Calcule el logaritmo de los parámetros anteriores:
ln (CCS') y ln (t'd)
- Calcule el tiempo de deriva usando Eq (1):
- Realizar una regresión lineal para determinar el coeficiente R2 y los parámetros x e y del ajuste logarítmico (con x la pendiente y ln(y) la intersección) usando Eq (5):
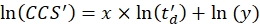 (5)
(5)
NOTA: El usuario puede trazar los valores ln(CCS') vs ln(td') para comprobar visualmente los resultados de la calibración, aunque esto es opcional.
- Para todos los puntos de calibración, calcule lo siguiente.
- Aplique la calibración a los datos experimentales para calibrar los picos seleccionados por MZmine para cada espectro IMS/IMS exportado al archivo *.csv. Para cada punto, calcule lo siguiente.
- Calcule el tiempo de deriva usando Eq (6):
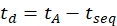 (6)
(6)
con tseq el tiempo anterior a la separación final de IMS (el valor 'Time Abs' indicado en el paso 3.4.6).
NOTA: Si calibra varios espectros IMS/IMS adquiridos con diferentes secuencias, compruebe cuidadosamente los valores tseq. - Calcule la masa neutra de los iones usando Eq (7):
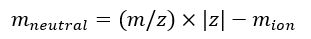 (7)
(7) - Calcule los parámetros td' y td'' utilizando Eq (8) y Eq (9):
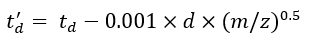 (8)
(8)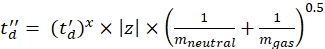 (9)
(9) - Calcule los valores finales calibrados de CCS (TWCCSN2 en nm2) mediante Eq (10):
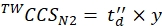 (10)
(10)
NOTA: Aunque paso 5.2.2. da ln(y) como la intersección, y debe usarse para obtener el valor final de CCS. No olvides aplicar una función exponencial.
- Calcule el tiempo de deriva usando Eq (6):
- Compruebe la exactitud de la calibración aplicando la calibración a la segunda adquisición de la solución de calibración adquirida en el paso 2.4.
NOTA: La calibración debe dar resultados con un error de ~1-2%.
Resultados
Se eligió un pentasacárido arabinoxilano, XA2XX, como ejemplo para ilustrar este protocolo. Este compuesto está disponible comercialmente, pero solo como mezcla con otro pentasacárido arabinoxilano, XA3XX (XA3XX puro también está disponible comercialmente). Las estructuras de XA2XX y XA3XX se dan en la Figura Suplementaria S1. Como la proporción de XA2XX y XA3XX en la mezcla comercial es de ~50:50, se preparó una soluci?...
Discusión
El SELECT SERIES Cyclic IMS es una poderosa herramienta que permite seleccionar una población iónica definida, de una movilidad m/z e iónica dada, sin necesidad de separación cromatográfica aguas arriba. El instrumento ofrece la posibilidad de generar un mapa de fragmentación bidimensional de esta población iónica, del que se pueden extraer los espectros MS/MS e IMS/IMS. Sin embargo, el usuario debe tener en cuenta varios puntos críticos que requieren atención durante el proceso experimental.
Divulgaciones
Los autores no tienen ningún conflicto de intereses que revelar.
Agradecimientos
S.O. agradece a la Agencia Nacional de Investigación de Francia la financiación de su doctorado (beca ANR-18-CE29-0006).
Materiales
| Name | Company | Catalog Number | Comments |
| 33-α-L- plus 23-α-L-Arabinofuranosyl-xylotetraose (XA3XX/XA2XX) mixture | Megazyme Ltd., Wicklow, Ireland | O-XAXXMIX | XA2XX + XA3XX mixture |
| 33-α-L-Arabinofuranosyl-xylotetraose (XA3XX) | Megazyme Ltd., Wicklow, Ireland | O-XA3XX | Pure XA3XX standard |
| Eppendorf Safe-Lock Tubes, 1.5 mL, Eppendorf Quality, colorless, 1,000 tubes | Eppendorf, Hamburg, Germany | 0030120086 | Used to prepare the carbohydrate stock solution and dilution |
| FALCON 50 mL Polypropylene Conical Tube 30 x 115 mm | Corning Science México S.A. de C.V., Reynosa, Tamaulipas, Mexico | 352070 | Used to prepare the aqueous stock solution of 100 mM LiCl |
| Lithium Chloride (ACS reagent, ≥99 %) | Sigma-Aldrich Inc., Saint Quentin Fallavier, France | 310468 | Used to dope the sample with lithium |
| Major Mix IMS/Tof Calibration Kit | Waters Corp., Wilmslow, UK | 186008113 | Calibration solution for MS and IMS |
| MassLynx 4.2 SCN1016 Release 6 (Waters Embedded Analyser Platform for Cyclic IMS 2.9.1 Release 9) | Waters Corp., Wilmslow, UK | 721022377 | Cyclic IMS vendor software for instrument control and data processing |
| Methanol for HPLC PLUS Gradient grade | Carlo-Erba Reagents, Val de Reuil, France | 412383 | High-purity solvent |
| MS Leucine Enkephaline Kit | Waters Corp., Wilmslow, UK | 700002456 | Reference compound used for tuning of the mass spectrometer |
| SCHOTT DURAN 100 mL borosilicate glass bottle | VWR INTERNATIONAL, Radnor, Pennsylvania, US | 218012458 | Used to prepare the solution of 500 µM LiCl in 50:50 MeOH/Water |
| SELECT SERIES Cyclic IMS | Waters Corp., Wilmslow, UK | 186009432 | Ion mobility-mass spectrometer equipped with a cylic IMS cell |
| Website: http://mzmine.github.io/ | MZmine Development Team | - | Link to download the MZmine software |
| Website: https://github.com/siollivier/IM-MN | INRAE, UR BIA, BIBS Facility, Nantes, France | - | Link to an in-house R script containing a CCS calibration function |
Referencias
- Allard, P. -. M., et al. Integration of molecular networking and in-silico MS/MS fragmentation for natural products dereplication. Analytical Chemistry. 88 (6), 3317-3323 (2016).
- Wang, M., et al. Mass spectrometry searches using MASST. Nature Biotechnology. 38 (1), 23-26 (2020).
- David, M., Fertin, G., Rogniaux, H., Tessier, D. SpecOMS: a full open modification search method performing all-to-all spectra comparisons within minutes. Journal of Proteome Research. 16 (8), 3030-3038 (2017).
- Dührkop, K., et al. SIRIUS 4: a rapid tool for turning tandem mass spectra into metabolite structure information. Nature Methods. 16 (4), 299-302 (2019).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Nothias, L. -. F., et al. Feature-based molecular networking in the GNPS analysis environment. Nature Methods. 17 (9), 905-908 (2020).
- Gray, C. J., et al. Advancing solutions to the Carbohydrate Sequencing Challenge. Journal of the American Chemical Society. 141 (37), 14463-14479 (2019).
- Ropartz, D., et al. Online coupling of high-resolution chromatography with extreme UV photon activation tandem mass spectrometry: Application to the structural investigation of complex glycans by dissociative photoionization. Analytica Chimica Acta. 933, 1-9 (2016).
- Wolff, J. J., et al. Negative electron transfer dissociation of glycosaminoglycans. Analytical Chemistry. 82 (9), 3460-3466 (2010).
- Ropartz, D., et al. Charge transfer dissociation of complex oligosaccharides: comparison with collision-induced dissociation and extreme ultraviolet dissociative photoionization. Journal of the American Society for Mass Spectrometry. 27 (10), 1614-1619 (2016).
- Morelle, W., et al. Fragmentation characteristics of permethylated oligosaccharides using a matrix-assisted laser desorption/ionization two-stage time-of-flight (TOF/TOF) tandem mass spectrometer. Rapid Communications in Mass Spectrometry. 18 (22), 2637-2649 (2004).
- Gabelica, V., Marklund, E. Fundamentals of ion mobility spectrometry. Current Opinion in Chemical Biology. 42, 51-59 (2018).
- Gabelica, V., et al. Recommendations for reporting ion mobility mass spectrometry measurements. Mass Spectrometry Reviews. 38 (3), 291-320 (2019).
- Hernandez-Mesa, M., et al. Interlaboratory and interplatform study of steroids collision cross section by traveling wave ion mobility spectrometry. Analytical Chemistry. 92 (7), 5013-5022 (2020).
- Koeniger, S. L., et al. An IMS-IMS analogue of MS-MS. Analytical Chemistry. 78 (12), 4161-4174 (2006).
- Merenbloom, S. I., Koeniger, S. L., Valentine, S. J., Plasencia, M. D., Clemmer, D. E. IMS−IMS and IMS−IMS−IMS/MS for separating peptide and protein fragment ions. Analytical Chemistry. 78 (8), 2802-2809 (2006).
- Eldrid, C., Thalassinos, K. Developments in tandem ion mobility mass spectrometry. Biochemical Society Transactions. 48 (6), 2457-2466 (2020).
- Giles, K., et al. A cyclic ion mobility-mass spectrometry system. Analytical Chemistry. 91 (13), 8564-8573 (2019).
- Merenbloom, S. I., Glaskin, R. S., Henson, Z. B., Clemmer, D. E. High-resolution ion cyclotron mobility spectrometry. Analytical Chemistry. 81 (4), 1482-1487 (2009).
- Ollivier, S., et al. Anomeric retention of carbohydrates in multistage cyclic ion mobility (IMSn): de novo structural elucidation of enzymatically produced mannosides. Analytical Chemistry. 93 (15), 6254-6261 (2021).
- Ollivier, S., Fanuel, M., Rogniaux, H., Ropartz, D. Molecular networking of high-resolution tandem ion mobility spectra: a structurally relevant way of organizing data in glycomics. Analytical Chemistry. 93 (31), 10871-10878 (2021).
- Aron, A. T., et al. Reproducible molecular networking of untargeted mass spectrometry data using GNPS. Nature Protocols. 15 (6), 1954-1991 (2020).
- McKenna, K. R., Li, L., Krishnamurthy, R., Liotta, C. L., Fernández, F. M. Organic acid shift reagents for the discrimination of carbohydrate isobars by ion mobility-mass spectrometry. The Analyst. 145 (24), 8008-8015 (2021).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11, 395 (2010).
- Ruotolo, B. T., Benesch, J. L. P., Sandercock, A. M., Hyung, S. -. J., Robinson, C. V. Ion mobility-mass spectrometry analysis of large protein complexes. Nature Protocols. 3 (7), 1139-1152 (2008).
- Bush, M. F., Hall, Z., Giles, K., Hoyes, J., Robinson, C. V., Ruotolo, B. T. Collision cross sections of proteins and their complexes: a calibration framework and database for gas-phase structural biology. Analytical Chemistry. 82 (22), 9557-9565 (2010).
- Ropartz, D., et al. Structure determination of large isomeric oligosaccharides of natural origin through multipass and multistage cyclic traveling-wave ion mobility mass spectrometry. Analytical Chemistry. 91 (18), 12030-12037 (2019).
- Tolmachev, A. V., et al. Characterization of ion dynamics in structures for lossless ion manipulations. Analytical Chemistry. 86 (18), 9162-9168 (2014).
- Arndt, J. R., et al. High-resolution ion-mobility-enabled peptide mapping for high-throughput critical quality attribute monitoring. Journal of the American Society for Mass Spectrometry. 32 (8), 2019-2032 (2021).
- Le Fèvre, A., Dugourd, P., Chirot, F. Exploring conformational landscapes using trap and release tandem ion mobility spectrometry. Analytical Chemistry. 93 (9), 4183-4190 (2021).
- Ohshimo, K., He, X., Ito, R., Misaizu, F. Conformer separation of dibenzo-crown-ether complexes with Na+ and K+ ions studied by cryogenic ion mobility-mass spectrometry. The Journal of Physical Chemistry A. 125 (17), 3718-3725 (2021).
- Purves, R. W., Barnett, D. A., Ells, B., Guevremont, R. Gas-phase conformers of the [M + 2H]2+ ion of bradykinin investigated by combining high-field asymmetric waveform ion mobility spectrometry, hydrogen/deuterium exchange, and energy-loss measurements. Rapid Communications in Mass Spectrometry. 15 (16), 1453-1456 (2001).
- Ujma, J., et al. Cyclic ion mobility mass spectrometry distinguishes anomers and open-ring forms of pentasaccharides. Journal of the American Society for Mass Spectrometry. 30 (6), 1028-1037 (2019).
- Warnke, S., Faleh, A. B., Scutelnic, V., Rizzo, T. R. Separation and identification of glycan anomers using ultrahigh-resolution ion-mobility spectrometry and cryogenic ion spectroscopy. Journal of The American Society for Mass Spectrometry. 30 (11), 2204-2211 (2019).
- Williamson, D. L., Bergman, A. E., Nagy, G. Investigating the structure of α/β carbohydrate linkage isomers as a function of group I metal adduction and degree of polymerization as revealed by cyclic ion mobility separations. Journal of the American Society for Mass Spectrometry. 32 (10), 2573-2582 (2021).
- Myers, O. D., Sumner, S. J., Li, S., Barnes, S., Du, X. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: new algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks. Analytical Chemistry. 89 (17), 8696-8703 (2017).
- Marchand, A., Livet, S., Rosu, F., Gabelica, V. Drift tube ion mobility: how to reconstruct collision cross section distributions from arrival time distributions. Analytical Chemistry. 89 (23), 12674-12681 (2017).
- Davis, D. M., et al. Analysis of ion mobility spectra for mixed vapors using Gaussian deconvolution. Analytica Chimica Acta. 289 (3), 263-272 (1994).
- Polasky, D. A., Dixit, S. M., Fantin, S. M., Ruotolo, B. T. CIUSuite 2: next-generation software for the analysis of gas-phase protein unfolding data. Analytical Chemistry. 91 (4), 3147-3155 (2019).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Gelb, A. S., Jarratt, R. E., Huang, Y., Dodds, E. D. A study of calibrant selection in measurement of carbohydrate and peptide ion-neutral collision cross sections by traveling wave ion mobility spectrometry. Analytical Chemistry. 86 (22), 11396-11402 (2014).
- Richardson, K., Langridge, D., Dixit, S. M., Ruotolo, B. T. An improved calibration approach for traveling wave ion mobility spectrometry: robust, high-precision collision cross sections. Analytical Chemistry. 93 (7), 3542-3550 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados