È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Dimostrazione dell'allineamento delle sequenze per prevedere lo strumento di suscettibilità tra le specie per una rapida valutazione della conservazione delle proteine
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per utilizzare l'ultima versione dello strumento SeqAPASS (Sequence Alignment to Predict Across Species Suceptibility) dell'Agenzia per la protezione ambientale degli Stati Uniti. Questo protocollo dimostra l'applicazione dello strumento online per analizzare rapidamente la conservazione delle proteine e fornire previsioni personalizzabili e facilmente interpretabili della suscettibilità chimica tra le specie.
Abstract
Lo strumento SeqAPASS (Sequence Alignment to Predict Across Species Suceptibility) dell'Agenzia per la protezione dell'ambiente degli Stati Uniti è un'applicazione di screening online veloce e disponibile gratuitamente che consente ai ricercatori e alle autorità di regolamentazione di estrapolare informazioni sulla tossicità tra le specie. Per i bersagli biologici in sistemi modello come cellule umane, topi, ratti e zebrafish, sono disponibili dati di tossicità per una varietà di sostanze chimiche. Attraverso la valutazione della conservazione del bersaglio proteico, questo strumento può essere utilizzato per estrapolare i dati generati da tali sistemi modello a migliaia di altre specie prive di dati sulla tossicità, fornendo previsioni di suscettibilità chimica intrinseca relativa. Le ultime versioni dello strumento (versioni 2.0-6.1) hanno incorporato nuove funzionalità che consentono la rapida sintesi, interpretazione e utilizzo dei dati per la pubblicazione oltre a grafici di qualità di presentazione.
Tra queste funzionalità ci sono visualizzazioni di dati personalizzabili e un rapporto di riepilogo completo progettato per riassumere i dati SeqAPASS per facilitare l'interpretazione. Questo documento descrive il protocollo per guidare gli utenti attraverso l'invio di processi, la navigazione nei vari livelli di confronto delle sequenze proteiche e l'interpretazione e la visualizzazione dei dati risultanti. Vengono evidenziate le nuove funzionalità di SeqAPASS v2.0-6.0. Inoltre, vengono descritti due casi d'uso incentrati sulla conservazione della transtiretina e della proteina del recettore oppioide utilizzando questo strumento. Infine, i punti di forza e i limiti di SeqAPASS sono discussi per definire il dominio di applicabilità per lo strumento ed evidenziare diverse applicazioni per l'estrapolazione cross-specie.
Introduzione
Tradizionalmente, il campo della tossicologia si è basato fortemente sull'uso della sperimentazione animale intera per fornire i dati necessari per le valutazioni della sicurezza chimica. Tali metodi sono in genere costosi e richiedono molte risorse. Tuttavia, a causa del gran numero di sostanze chimiche attualmente utilizzate e del rapido ritmo con cui vengono sviluppate nuove sostanze chimiche, a livello globale vi è una riconosciuta necessità di metodi più efficienti di screening chimico 1,2. Questa necessità e il conseguente cambiamento di paradigma dalla sperimentazione animale ha portato allo sviluppo di molti nuovi metodi di approccio, tra cui saggi di screening ad alto rendimento, trascrittomica ad alto rendimento, sequenziamento di prossima generazione e modellazione computazionale, che sono promettenti strategie di sperimentazione alternative 3,4.
Valutare la sicurezza chimica attraverso la diversità delle specie potenzialmente influenzate dalle esposizioni chimiche è stata una sfida duratura, non solo con i test di tossicità tradizionali, ma anche con nuovi metodi di approccio. I progressi nella tossicologia comparativa e predittiva hanno fornito quadri per comprendere la sensibilità relativa delle diverse specie e i progressi tecnologici nei metodi computazionali continuano ad aumentare l'applicabilità di questi metodi. Nell'ultimo decennio sono state discusse diverse strategie che sfruttano i database di sequenze geniche e proteiche esistenti, insieme alla conoscenza di specifici bersagli molecolari chimici, per supportare approcci predittivi per l'estrapolazione tra specie e migliorare le valutazioni di sicurezza chimica oltre i tipici organismi modello 5,6,7,8.
Per far progredire la scienza in azione, basarsi su questi studi fondamentali in tossicologia predittiva, dare priorità agli sforzi di test chimici e supportare il processo decisionale, è stato creato lo strumento SeqAPASS (Sequence Alignment to Predict Across Species Suceptibility) dell'Agenzia per la protezione ambientale degli Stati Uniti. Questo strumento è un'applicazione pubblica e liberamente disponibile basata sul web che utilizza archivi pubblici di informazioni sulla sequenza proteica in costante espansione per prevedere la suscettibilità chimica attraverso la diversità delle specie9. Sulla base del principio che la suscettibilità intrinseca relativa di una specie a una particolare sostanza chimica può essere determinata valutando la conservazione dei bersagli proteici noti di quella sostanza chimica, questo strumento confronta rapidamente le sequenze di amminoacidi proteici di una specie con sensibilità nota a tutte le specie con dati di sequenza proteica esistenti. Questa valutazione è completata attraverso tre livelli di analisi, tra cui (1) sequenza aminoacidica primaria, (2) dominio funzionale e (3) confronti critici dei residui di amminoacidi, ognuno dei quali richiede una conoscenza più approfondita dell'interazione chimica-proteina e fornisce una maggiore risoluzione tassonomica nella previsione della suscettibilità. Uno dei principali punti di forza di SeqAPASS è che gli utenti possono personalizzare e perfezionare la loro valutazione aggiungendo ulteriori linee di evidenza verso la conservazione del target in base alla quantità di informazioni disponibili per quanto riguarda l'interazione chimica-proteina o proteina-proteina di interesse.
La prima versione è stata rilasciata nel 2016, che ha permesso agli utenti di valutare sequenze di amminoacidi primari e domini funzionali in modo semplificato per prevedere la suscettibilità chimica e conteneva capacità minime di visualizzazione dei dati (Tabella 1). Le differenze individuali di amminoacidi hanno dimostrato di essere importanti determinanti delle differenze tra specie nelle interazioni chimico-proteiche, che possono influenzare la suscettibilità chimica delle specie10,11,12. Pertanto, sono state sviluppate versioni successive per considerare gli amminoacidi critici che sono importanti per l'interazione chimica diretta13. In risposta al feedback delle parti interessate e degli utenti, questo strumento è stato sottoposto a rilasci annuali con nuove funzionalità aggiuntive progettate per soddisfare le esigenze sia dei ricercatori che delle comunità normative per affrontare le sfide nell'estrapolazione tra specie (Tabella 1). Il lancio della versione 5.0 di SeqAPASS nel 2020 ha portato avanti funzionalità incentrate sull'utente che incorporano opzioni di visualizzazione e sintesi dei dati, collegamenti esterni, tabelle di riepilogo e opzioni di report e funzionalità grafiche. Nel complesso, i nuovi attributi e le nuove capacità di questa versione hanno migliorato la sintesi dei dati, l'interoperabilità tra database esterni e la facilità di interpretazione dei dati per le previsioni di suscettibilità tra specie.
Protocollo
1. Per iniziare
NOTA: il protocollo qui presentato è incentrato sull'utilità dello strumento e sulle funzionalità chiave. Descrizioni dettagliate di metodi, caratteristiche e componenti sono disponibili sul sito Web in una Guida utente completa (Tabella 1).
Tabella 1: Evoluzione dello strumento SeqAPASS. Un elenco di funzionalità e aggiornamenti aggiunti allo strumento SeqAPASS dalla sua distribuzione iniziale. Abbreviazioni: SeqAPASS = allineamento della sequenza per prevedere la suscettibilità delle specie; ECOTOX = ECOTOXicology knowledge base. Clicca qui per scaricare questa tabella.
- Vai a https://seqapass.epa.gov/seqapass utilizzando Chrome. Selezionare Login per utilizzare un account esistente o seguire le istruzioni per creare un account SeqAPASS, che consentirà agli utenti di eseguire, archiviare, accedere e personalizzare i lavori completati.
- Prima di condurre un'analisi, identificare una proteina di interesse e una specie mirata o sensibile esaminando la letteratura esistente o i dati preesistenti (Figura 1). Poiché SeqAPASS contiene collegamenti a risorse esterne per aiutare a identificare la proteina della query, fare clic sui pulsanti a discesa sotto Identificare un target proteico per accedere alle risorse pertinenti.
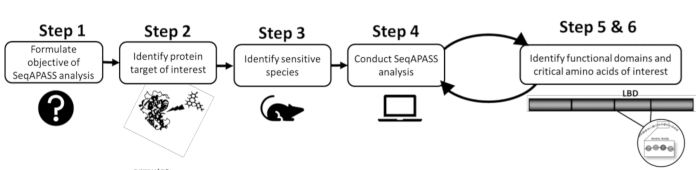
Figura 1: Formulazione del problema SeqAPASS: diagramma schematico delle informazioni preliminari necessarie per un'analisi di successo. Abbreviazioni: SeqAPASS = allineamento della sequenza per prevedere la suscettibilità delle specie; LBD = dominio legante il ligando. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Interoperabilità di SeqAPASS tra database. Diagramma schematico di strumenti esterni, database e risorse integrati in SeqAPASS. Abbreviazioni: SeqAPASS = allineamento della sequenza per prevedere la suscettibilità delle specie; AOP = via degli esiti avversi; NCBI = Centro nazionale per l'informazione biotecnologica; ECOTOX = ECOTOXicology knowledge base. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 2: Collegamenti, risorse e strumenti integrati nello strumento SeqAPASS. Un elenco delle varie fonti di dati, collegamenti e risorse sfruttate nello strumento SeqAPASS. Abbreviazione: SeqAPASS = allineamento della sequenza per prevedere la suscettibilità delle specie. Clicca qui per scaricare questa tabella.
2. Sviluppo ed esecuzione di una query SeqAPASS: Level 1
NOTA: In un'analisi di livello 1, l'intera sequenza di amminoacidi primari di una proteina di query viene confrontata con le sequenze di amminoacidi primari di tutte le specie con informazioni di sequenza disponibili. Questo strumento utilizza algoritmi per estrarre, raccogliere e compilare dati disponibili pubblicamente per allineare e confrontare rapidamente le sequenze di amminoacidi tra le specie. Il backend memorizza le informazioni dai database del National Center for Biotechnology Information (NCBI) e utilizza strategicamente le versioni standalone del Protein Basic Local Alignment Search Tool (BLASTp)54 e del Constraint-based Multiple Alignment Tool (COBALT)55.
- In Confronta sequenze di amminoacidi primari fare clic su Per specie o Per accesso. Utilizzare la selezione Per specie per digitare o selezionare da un elenco di specie per scegliere il target proteico di interesse.
- Inviare direttamente le accessioni proteiche (ad esempio, l'ID proteina NCBI) inserendo l'adesione o le adesioni nella casella di testo Per adesione .
- Selezionare Esegui richiesta per inviare la query. Una volta inviato, attendi che venga visualizzata una notifica nell'angolo in alto a destra della finestra del browser che indica un invio riuscito.
- Selezionare la scheda SeqAPASS Run Status nella parte superiore della pagina per visualizzare un elenco di tutte le esecuzioni di SeqAPASS condotte con quell'account utente e controllare la percentuale di completamento.
- Fare clic su Aggiorna dati mentre è selezionato il pulsante di opzione appropriato per verificare lo stato delle esecuzioni di livello 2 e livello 3.
- Seleziona la scheda Visualizza report SeqAPASS nella parte superiore della pagina per accedere a un elenco di tutti i report completati con quell'account.
- Nella scheda Visualizza report SeqAPASS , selezionare la proteina di query di interesse. Fare clic su Richiedi report selezionato per aprire la pagina Informazioni sulle proteine query di livello 1 e visualizzare i risultati, le opzioni di personalizzazione dei dati, le visualizzazioni e i report di riepilogo.
- Per impostazione predefinita, selezionare Visualizza report per visualizzare i dati nel Web browser. In alternativa, selezionare Salva report per scaricare i dati non elaborati come file .zip.
NOTA: il tempo necessario per un'analisi di livello 1 varia (in media 23 minuti per la versione 5.1) a seconda della domanda globale degli utenti in quel momento, del numero di processi inviati alla coda e della quantità di informazioni sulle proteine esistenti per un processo inviato. Se un bersaglio proteico è stato precedentemente completato, i dati saranno disponibili in pochi secondi dopo l'invio.
3. Sviluppo ed esecuzione di una query SeqAPASS: Level 2
NOTA: Poiché l'intera sequenza proteica non è direttamente coinvolta in un'interazione chimica, un'analisi di livello 2 confronta solo la sequenza amminoacidica del dominio funzionale per fare previsioni di suscettibilità a ranghi tassonomici inferiori (ad esempio, classe, ordine, famiglia).
- Dalla pagina Informazioni sulle proteine della query di livello 1, fare clic sul segno più + accanto all'intestazione di livello 2 per popolare il menu Query di livello 2 .
- Identificare i domini appropriati nella proteina di interesse (proteina di query).
- Se un dominio non è stato identificato, fare clic sul collegamento integrato al database dei domini conservati (CDD) NCBI (Tabella 1), che può aiutare nell'identificazione della selezione del dominio appropriato.
NOTA: in genere, solo i domini di hit specifici vengono selezionati come query nel livello 2.
- Se un dominio non è stato identificato, fare clic sul collegamento integrato al database dei domini conservati (CDD) NCBI (Tabella 1), che può aiutare nell'identificazione della selezione del dominio appropriato.
- Fare clic sulla casella Seleziona dominio per popolare automaticamente un elenco di domini funzionali per la proteina della query.
- Selezionare le accessioni al dominio dall'elenco a discesa e avviare la query di livello 2 facendo clic sul pulsante Richiedi esecuzione dominio . Una volta inviato, attendi che venga visualizzata una notifica che indica che l'invio è stato eseguito correttamente.
- Fare clic su Aggiorna esecuzioni di livello 2 e 3 per popolare i dati di livello 2, che saranno disponibili entro pochi secondi dall'invio.
- In Visualizza dati di livello 2, selezionare le accessioni al dominio completate dall'elenco a discesa e fare clic sul pulsante Visualizza dati di livello 2 per aprire i risultati in una nuova pagina.
4. Accesso e comprensione dei dati: SeqAPASS Livello 1 e Livello 2
- Scorri fino alla fine della pagina Query Protein Information per visualizzare un report dei risultati: per impostazione predefinita viene fornito un report primario con analisi di livello 1 e 2. Selezionare il pulsante di opzione Report completo per visualizzare un report più dettagliato che fornisce tutti i riscontri della sequenza e le metriche di allineamento. Fare clic sull'accesso/ID/nome appropriato in entrambi i report per accedere alle informazioni trasparenti sull'allineamento delle proteine e sulla tassonomia nel database NCBI.
- Scorrere fino al lato destro della tabella dei risultati per visualizzare la colonna ECOTOX . Fare clic sui collegamenti alla knowledge base di ECOTOXicology (ECOTOX) per raccogliere rapidamente i dati di tossicità corrispondenti per le specie con previsioni di suscettibilità.
NOTA: ECOTOX è una knowledge base completa e pubblicamente disponibile che fornisce singoli dati sulla tossicità chimica per piante acquatiche e terrestri e fauna selvatica. SeqAPASS v6.0 include un widget ECOTOX per connettersi più rapidamente con i dati ECOTOX rilevanti per sostanza chimica e specie di interesse. - Fare clic su Scarica tabella per salvare la tabella come file di foglio di calcolo. Fare clic sul pulsante Visualizza report di riepilogo per visualizzare e scaricare una tabella di report di riepilogo che presenta i dati ordinati per gruppo tassonomico.
NOTA: le tabelle di riepilogo dei dati sono disponibili sia per i report primari che per quelli completi e forniscono una panoramica delle previsioni per una determinata destinazione.
5. Manipolazione delle impostazioni dei dati: SeqAPASS Livello 1 e Livello 2
NOTA: In entrambe le analisi di livello 1 e livello 2, si presume che maggiore è la somiglianza proteica, maggiore è la probabilità che una sostanza chimica interagisca con la proteina in modo simile alla specie / proteina interrogata, rendendole suscettibili ai potenziali impatti delle sostanze chimiche con questo bersaglio molecolare. A causa della somiglianza di questi dati, i passaggi per comprendere i dati di livello 1 e 2 sono delineati insieme in un unico protocollo.
- Fare riferimento ai sottomenu nella parte superiore di Query Protein Information per accedere e manipolare le impostazioni del report e utilizzare le impostazioni predefinite per tutte le opzioni di report per la maggior parte delle analisi. Se esiste una giustificazione scientifica per modificare l'impostazione predefinita, attenersi alla seguente procedura facoltativa:
- (FACOLTATIVO) Fare clic sul segno più + accanto a Soglia di suscettibilità per visualizzare e regolare le impostazioni di interruzione della suscettibilità in una nuova scheda. Selezionare un nuovo valore limite da un elenco a discesa o immettere un valore limite definito dall'utente.
- (FACOLTATIVO) Modificare il numero nel campo E-Value (il numero di diversi allineamenti previsti per caso) se si desidera qualcosa di diverso da quello predefinito.
NOTA: Qualsiasi proteina con un valore E maggiore del numero nella casella verrà eliminata dal rapporto primario. - (FACOLTATIVO) Utilizzare l'opzione Ordina per gruppo tassonomico per scegliere il livello della gerarchia tassonomica da visualizzare nella colonna Gruppo tassonomico filtrato della tabella dei risultati.
NOTA: la modifica della gerarchia tassonomica altererà anche la previsione della suscettibilità basata sulle specie di ciascun gruppo filtrato che si trovano al di sopra del limite. - (FACOLTATIVO) Modificare il campo Common Domain (quanti domini comuni una proteina deve condividere con la proteina di query da includere nei risultati) se si desidera qualcosa di diverso da quello predefinito.
Nota : poiché l'impostazione predefinita è 1, qualsiasi sequenza che non condivide almeno un dominio comune con la proteina di query verrà esclusa. - (FACOLTATIVO) Selezionare No in Specie Read Across per ottenere le previsioni di suscettibilità di Y solo se la percentuale di somiglianza è maggiore o uguale al cutoff o se il hit viene identificato come candidato ortologo.
NOTA: per impostazione predefinita questa impostazione è Sì, il che significa che verrà riportata una previsione di suscettibilità di Y per tutti i candidati ortologi, tutte le specie elencate sopra il cutoff di suscettibilità e tutte le specie al di sotto del cutoff dello stesso gruppo tassonomico con una o più specie al di sopra del cutoff.
- Fare clic sul pulsante Scarica impostazioni report correnti per scaricare un file che acquisisce le impostazioni correnti applicate.
NOTA: il livello di valutazione specifico (1, 2 o 3) selezionato determinerà le impostazioni presentate nel report.
6. Visualizzazione dei dati: SeqAPASS Livello 1 e Livello 2
- Fare clic sul segno più + accanto a Visualizzazione e fare clic sul pulsante Visualizza dati per aprire una scheda separata che visualizza le informazioni definite dall'utente e l'opzione per selezionare un grafico interattivo dei risultati.
- Fare clic su Boxplot per aprire i controlli boxplot e plot interattivi e consentire l'aggiornamento attivo della visualizzazione boxplot per riflettere le modifiche apportate alla tabella dati e fornire grafica di qualità di pubblicazione e presentazione.
NOTA: il boxplot predefinito visualizza i gruppi di specie sull'asse x e la somiglianza percentuale sull'asse y. I boxplot mostrano il cutoff di suscettibilità (linea tratteggiata), la somiglianza percentuale tra le specie rispetto alle specie di query e i valori medi e mediani per ciascun gruppo tassonomico insieme al 25 ° e 75 ° percentile e all'intervallo interquartile. A seconda dell'obiettivo dell'analisi e delle esigenze dell'utente, molte caratteristiche del boxplot possono essere modificate attraverso i seguenti passaggi opzionali.- (FACOLTATIVO) Per personalizzare i gruppi tassonomici visualizzati, fare riferimento alla casella Gruppi tassonomici nella sezione Controlli . Rimuovi i gruppi scorrendo i nomi e selezionando x o utilizzando il menu a discesa Gruppi tassonomici .
- (FACOLTATIVO) Per aggiungere una legenda che indichi una specie di interesse o specifici gruppi predefiniti (ad esempio, specie in via di estinzione o minacciate), passa il mouse sopra il nome di un gruppo tassonomico sull'asse x per attivare una finestra pop-up che elenca le prime tre specie ordinate in base alla più alta percentuale di somiglianza. Passa il mouse sopra le specie nella legenda per generare una finestra popup con le informazioni sulle specie corrispondenti. Fare clic sulla casella relativa a un gruppo tassonomico specifico per generare una tabella riassuntiva scaricabile che elenca specie e previsioni.
- Fare clic su Scarica Boxplot per scegliere un tipo di file, personalizzare la risoluzione di larghezza/altezza e salvare la visualizzazione.
7. Sviluppo ed esecuzione di un'analisi SeqAPASS: Level 3
NOTA: Un'analisi di livello 3 valuta i residui di amminoacidi identificati dall'utente all'interno della proteina di query e confronta rapidamente la conservazione di questi residui tra le specie. Si presume che le specie in cui questi residui sono conservati abbiano maggiori probabilità di interagire con una sostanza chimica in modo simile alla specie/proteina modello. Poiché il livello 3 si concentra sui singoli amminoacidi, un'analisi può essere eseguita solo quando è disponibile una conoscenza dettagliata dei residui di amminoacidi critici per l'interazione chimica-proteina o proteina-proteina.
- Fare clic sul segno più + accanto all'intestazione di livello 3 nella pagina Informazioni sulle proteine della query di livello 1 per popolare il menu Query di livello 3.
- Fare clic sul segno più + accanto a Esplora riferimenti per aprire lo strumento Esplora riferimenti, che genera una stringa booleana predefinita per interrogare la letteratura disponibile e aiuta gli utenti a identificare la letteratura appropriata per supportare l'identificazione di amminoacidi critici da utilizzare nella valutazione di livello 3 (Tabella 2 e Figura 2).
- (FACOLTATIVO) Una volta che la query protein si popola automaticamente, utilizzare la funzione Add Protein Name per aggiungere altre proteine.
- Fai clic sul link Genera Google Scholar per aprire un popup contenente una stringa di ricerca generata automaticamente che include termini di ricerca pertinenti.
- Fai clic su Cerca in Google Scholar per eseguire query nel database della letteratura utilizzando la stringa di ricerca.
- In alternativa, fare clic su Copia negli Appunti e personalizzare la stringa di ricerca aggiungendo o rimuovendo termini utilizzando le funzioni di Esplora riferimenti.
8. Identificare i residui critici di aminoacidi utilizzando la letteratura identificata
- Selezionare la sequenza di modelli a cui verranno allineate le specie selezionate dall'utente nel menu Query di livello 3.
NOTA: Questa sequenza modello viene comunemente scelta in base alla letteratura per la quale sono stati identificati gli amminoacidi critici e può essere la stessa specie o una specie diversa da quelle interrogate nel livello 1 e nel livello 2.- (FACOLTATIVO) Utilizzare la casella Confronti aggiuntivi per confrontare eventuali accessioni/sequenze non visualizzate nelle tabelle Report primario/completo.
- Immettere un nome definito dall'utente per l'esecuzione di livello 3 nella casella di testo Immettere il nome dell'esecuzione di livello 3 per identificare l'esecuzione di livello 3 completata. Scegli un nome univoco per ogni valutazione.
- Selezionare il gruppo tassonomico di interesse nel campo Scegli gruppo tassonomico. Selezionare un gruppo tassonomico per filtrare automaticamente la tabella in base a tale gruppo tassonomico.
- Nella tabella dei risultati, fare clic manualmente sulla casella di controllo accanto a qualsiasi specie da allineare alla sequenza del modello.
NOTA: per garantire un allineamento appropriato, è necessario confrontare un gruppo tassonomico alla volta con il modello. Selezionare solo proteine annotate in modo simile per le specie di interesse. Quando si selezionano le sequenze per il confronto, è importante prestare attenzione a determinate sequenze (ad esempio, ipotetiche, BASSA QUALITÀ o parziali). A meno che non vi sia una giustificazione trasparente per l'inclusione, è meglio escludere queste sequenze in quanto potrebbero distorcere le previsioni a causa di informazioni di sequenza incomplete o inappropriate. - Ripetere i passaggi per allineare tutti i gruppi tassonomici di interesse.
- Fare clic su Aggiorna esecuzioni di livello 2 e 3 una volta che tutte le specie sono state allineate per popolare il menu Seleziona nome esecuzione livello 3 con i processi di livello 3 completati e ottenere immediatamente i dati da un allineamento di livello 3.
- Fare clic su Combina dati di livello 3 per combinare allineamenti da più gruppi tassonomici.
- In alternativa, per visualizzare un singolo report, selezionare il nome definito dall'utente in Scegli query da visualizzare e fare clic su Visualizza dati di livello 3.
- Selezionare il modello di livello 3 da utilizzare come base per il confronto dei residui di amminoacidi nel menu Combina report di livello 3 e fare clic su Avanti.
- In Processi di livello 3, selezionare i processi completati per il confronto e fare clic su Avanti. Utilizzare la funzione Processi di livello 3 dell'ordine per riordinare i gruppi tassonomici, se lo si desidera. Fare clic su Visualizza dati di livello 3 per creare una pagina di report di livello 3 con i gruppi tassonomici combinati allineati.
- Selezionare le posizioni degli amminoacidi identificate in precedenza per la specie modello digitando le posizioni degli amminoacidi, separate da virgole, nella casella Immetti posizioni dei residui di amminoacidi e quindi selezionando Copia nell'elenco dei residui . Selezionare direttamente i residui nella sequenza del modello dalla casella shuttle.
- Fare clic su Aggiorna report per aggiornare la pagina e visualizzare le previsioni di suscettibilità di livello 3.
NOTA: Il livello 3 utilizza un semplice insieme di regole derivate da descrittori di base delle proprietà funzionali della catena laterale (ad esempio, alifatiche, aromatiche) e delle dimensioni molecolari (differenze di peso molecolare >30 g / mol) per determinare se le differenze nelle posizioni chiave possono influenzare le interazioni proteiche13.
9. Visualizzazione dei dati SeqAPASS di livello 3
NOTA: come nei livelli precedenti, sono disponibili i report primari e completi. Oltre ai dati identici ai dati di livello 1 e 2, il rapporto primario visualizza le posizioni degli amminoacidi, le abbreviazioni e una suscettibilità sì/no (Y/N) simile alla previsione del modello. Allo stesso modo, il rapporto completo contiene informazioni sulla classificazione delle catene laterali degli aminoacidi e sul peso molecolare.
- Nella pagina Report livello 3 scorrere fino in fondo per visualizzare un report dei risultati. Fare clic su Scarica tabella nella parte inferiore del report per salvare la tabella .
- Fare clic su Visualizza report di riepilogo di livello 3 per visualizzare e scaricare una tabella di report di riepilogo che presenta i dati ordinati per gruppo tassonomico. Fare clic sul segno più + accanto a Visualizzazione nella pagina Report di livello 3 per aprire una scheda del browser separata che visualizza le informazioni definite dall'utente e l'opzione per visualizzare i risultati sotto forma di una mappa termica interattiva.
- Fare clic su Mappa termica nella pagina Informazioni di visualizzazione per aprire l'elemento grafico e i controlli interattivi e consentire l'aggiornamento attivo della visualizzazione della mappa di calore per riflettere le modifiche apportate alla tabella dati. Eseguire i seguenti passaggi facoltativi per personalizzare la mappa di calore.
- (FACOLTATIVO) Selezionare Opzioni report per passare da un report semplice, che visualizza la posizione dell'amminoacido, l'abbreviazione di una lettera e la somiglianza tra amminoacidi, o un report completo, che visualizza informazioni dettagliate su ogni amminoacido selezionato.
- (FACOLTATIVO) Selezionare Opzioni report per modificare la modalità di visualizzazione delle specie, in base al nome comune o al nome scientifico.
NOTA: all'interno del rapporto semplice, gli amminoacidi sono classificati come Corrispondenza totale (blu scuro), Corrispondenza parziale (azzurro, sostituzioni che soddisfano un solo criterio) o Non corrispondenza (giallo, sostituzioni che non soddisfano nessuno dei due criteri) all'amminoacido del modello. Il report completo visualizza i confronti come Corrispondenza totale (blu scuro) o Non corrispondenza (giallo). - (FACOLTATIVO) Selezionare Selezioni facoltative per evidenziare informazioni utili quali candidati ortologi, specie minacciate, specie minacciate o organismi modello comuni.
- (FACOLTATIVO) Seleziona Impostazioni mappa termica per selezionare ulteriori opzioni di personalizzazione, tra cui l'aggiunta o la rimozione di colonne, legende e testo.
- Fare clic su Scarica Boxplot per scegliere un tipo di file e salvare la visualizzazione.
10. Interpretazione dei risultati di SeqAPASS: linee di prova per la conservazione delle proteine
NOTA: per facilitare l'interpretazione, questo strumento include un rapporto di riepilogo delle decisioni (DS Report) progettato per integrare i dati tra i livelli. Il DS Report contiene i risultati (cioè tabelle di dati e/o visualizzazioni) selezionati dall'utente e consente la rapida valutazione delle previsioni di suscettibilità su più livelli per più specie contemporaneamente.
- Fare clic su Push Level # to DS Report nelle pagine dei risultati o della visualizzazione dei dati e attendere che i dati vengano "spinti" e che la scheda DS Report diventi attiva.
NOTA: se i risultati o le modifiche non sono stati inviati al report DS, il report Push Level # to DS rimarrà attivo fino a quando non sarà selezionato. Se un'impostazione è stata modificata, verrà visualizzato il testo Fare clic per eseguire il push di nuove modifiche finché non verranno inviate le modifiche al report. Le visualizzazioni possono essere inviate al DS Report in qualsiasi momento durante la valutazione. - Selezionare la scheda DS Report in qualsiasi momento per accedere alla pagina DS .
NOTA: Per tutte le specie allineate nel livello 1, la tabella del rapporto riassuntivo della decisione finale contiene i dati importanti e le previsioni di suscettibilità per ciascuna analisi. Se una specie nella tabella DS non è stata inclusa nel rapporto di livello 3 ma è stata trovata in lavori di livello 1 e/o livello 2, la cella nella tabella riceverà una designazione non applicabile (NA) per la previsione di suscettibilità di livello 3.
Risultati
Per dimostrare l'applicazione dello strumento SeqAPASS ed evidenziare nuove funzionalità, vengono descritti due casi di studio che rappresentano casi in cui la conservazione delle proteine prevede che ci siano differenze nella suscettibilità chimica tra le specie (transtiretina umana) e che non ci siano differenze (μ recettore oppioide [MOR]). Il primo di questi esempi affronta la sequenza proteica / confronti strutturali per prevedere il dominio di applicabilità per le vie di esito avverso (AOP, vedi Tabella...
Discussione
C'è un riconoscimento diffuso che non è possibile testare empiricamente abbastanza specie per catturare la diversità genomica, fenotipica, fisiologica e comportamentale degli organismi viventi che possono essere esposti a sostanze chimiche di interesse tossicologico. L'obiettivo di SeqAPASS è massimizzare l'uso di sequenze proteiche esistenti e in continua espansione e dati strutturali per aiutare e informare l'estrapolazione dei dati / conoscenze sulla tossicità chimica dagli organismi testati a centinaia o migliai...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori ringraziano il Dr. Daniel L. Villeneuve (U.S. EPA, Center for Computational Toxicology and Exposure) e il Dr. Jon A. Doering (Department of Environmental Sciences, Louisiana State University) per aver fornito commenti su una precedente bozza del manoscritto. Questo lavoro è stato sostenuto dalla US Environmental Protection Agency. Le opinioni espresse in questo documento sono quelle degli autori e non riflettono necessariamente le opinioni o le politiche dell'Agenzia per la protezione ambientale degli Stati Uniti, né la menzione di nomi commerciali o prodotti commerciali indica l'approvazione da parte del governo federale.
Materiali
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Riferimenti
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001 (2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982 (2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506 (2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A., Richardson, S. J., Cody, V. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. , 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529 (2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency Available from: https://www.epa.gov/npdes/npdes-resources (2018)
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053 (2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636 (2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740 (2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595 (2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021)
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service Available from: https://ecos.fws.gov/ecp/ (2021)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon