Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Bir Hücre Tipine Özel Ölçekte Arabidopsis thaliana Kök Gelişimini Araştırmak Için Ribozom Affinity Arınma (TRAP) Çevirisi
Bu Makalede
Özet
Ribozom yakınlık arınması (TRAP) çevirisi, organ ve dokuların en az işlenmesi yle gelişim programlarını inceleme olanağı sunar. Protokol, yeşil floresan protein (GFP) etiketli ribozomal alt ünite ile hedeflenen hücrelerden yüksek kaliteli RNA verir. QRT-PCR veya RNA-seq gibi alt akış analiz araçları doku ve hücre tipine özgü ifade profillerini ortaya çıkarır.
Özet
Bu makalede, ribozom afinite arınma (TRAP) yöntemi ve ardışık optimize edilmiş düşük girişli kitaplık hazırlama yöntemi ile farklı Arabidopsis thaliana kök hücre tiplerinden translatom verileri elde etmek için uygulamalı talimatlar veriyoruz.
Başlangıç malzemesi olarak, yeterli organizatörlerin kullanımı ile gfp etiketli ribozomal protein RPL18'i hücre tipine özgü bir şekilde ifade eden bitki hatlarını kullanırız. Önce immünpurifikasyon ve RNA ekstraksiyon, doku, doku bütünlüğünü korur ve aynı anda yüksek zamansal çözünürlük ile zaman serisi çalışmaların yürütülmesine olanak sağlar dondurulmuş snap. Özellikle, hücre duvarı yapıları bozulmadan kalır, bu da farklı hücre popülasyonlarını izole etmek için protoplasting dokuya dayanan floresan-aktive hücre tasnisi temelli yaklaşımlar gibi alternatif prosedürlerde büyük bir dezavantajdır. Ayrıca, yüksek kaliteli RNA elde edilmesine olanak sağlayan lazer yakalama mikrodiseksiyon tabanlı teknikler gibi doku fiksasyonu gerekli değildir.
Ancak, hücrelerin alt popülasyonlarından örnekleme ve sadece polizom ilişkili RNA izole ciddi RNA verimleri sınırlar. Bu nedenle, RNA-seq tarafından başarılı veri toplama için yeterince hassas kütüphane hazırlama yöntemleri uygulamak gereklidir.
Trap, hücre duvarıyla ilgili ve mekanik sinyal yolları içerdiğinden, birçok gelişimsel süreç hücre duvarı yla ilgili olduğu için bitki araştırmaları için ideal bir araç tır. Organizatörlerin belirli hücre popülasyonlarını hedeflemek için kullanımı, organ ve tek hücre düzeyi arasındaki boşluğu köprülemektedir ve bu da çok az çözünürlük veya çok yüksek maliyetlerden muzdariptir. Burada, lateral kök oluşumunda hücre-hücre iletişimini incelemek için TRAP uyguluyoruz.
Giriş
Yeni nesil sıralama tekniklerinin giderek artan uygulamasıyla, gelişim biyolojisinde uzamsal çözünürlük artırılabilir. Çağdaş çalışmalar, tek hücreli seviye,1,2,3,,4değilse, dokuların özel hücre tiplerine kadar incelenmesini amaçlamaktadır. Bu amaçla, son elli yıl içinde farklı yöntemlerbir bolluk geliştirilmiştir (Şekil 1Abakınız )5,6,,7,8,9,10,11,12,13,14,15.
Bitki bilimindeki pek çok araç, hayvan araştırmalarında öncülük eden tekniklerin adaptasyonları olmuştur. Burada ayrıntılı olarak sunduğumuz yöntem için durum böyle değildir. 2005 yılında, protein çevirisi güçlü bir arka plan ile donatılmış, Bailey-Serres Lab sonraki yakınlık saflaştırma16için ribozomal proteinleri mühendisi yola çıktı. Böylece, bir sakaroz gradyan ile ultracentrifugation dayalı ve 1960'lardan beri ribozomçevirme değerlendirmek için kullanılan zaman alıcı ve emek yoğun polizom profilleme, önlemek olabilir17,18. Yöntem o zamandan beri çevirisel ribozom afinite arınma (TRAP)16olarak anılacaktır. Bitkilerde başarılı translatome çalışmalardan sonra, Heiman ve ark. hayvanlar için TRAP adapte19 ve diğerleri maya20, Drosophila21, Xenopus22 ve zebra balığı23,24.
Model sisteminin genetik modifikasyonu TRAP için bir ön koşul olmasına rağmen, hangi türler genetik dönüşüm için uygun uygulama sınırlar, bir aynı anda özel ilgi ve bozulmamış doku / organ25 izole etmek için son derece zor hücrelerin alt kümeleri hedef bu itiraz koşum olabilir (örneğin, bir fare beyin veya enfekte bitki dokusunda mantar hyphae yüksek dallı dendritik hücreler). Bitkilerde, tüm hücreler hidrostatik iskelet26temelini oluşturan hücre duvarları ile yerinde tutulur. Bu matris bir bitki hücresi serbest etmek için, bilim adamları ya fiziksel lazer yakalama mikrodizeksiyon (LCM)27 veya hücre duvarları28enzimatik sindirim gerçekleştirilen yoluyla çevresindeki doku hücre kesip var . İkinci hücreler arasında, sözde protoplastlar, ilgi popülasyonu floresan etiketli ve floresan aktif hücre sıralama (FACS)7ile ayrılabilir. LCM genellikle sabit ve balmumu gömülü bir örnek gerektirir, hangi sonuçta onun RNA kalitesini bozar29. FACS tabanlı yöntemler yüksek kaliteli RNA verim, ancak protoplasting kendisi süreci gen ekspresyonu farklılıkları tanıttı30 ve modifiye ve kalın ikincil hücre duvarları ile dokuların tedavi etmek için kötü üne sahiptir. Ayrıca, bitkilerdeki birçok gelişimsel sürecin mekanik olarak iletilen sinyallere dayandığı varsayılır ve bu nedenle hücre duvarının bütünlüğü büyük önem taşımaktadır31. Çekirdek seviyesinde hareket ederek hücre izolasyonundan maksatlamak için kısayol kullanan iki yöntem, floresan tarafından aktive edilmiş nükleer sıralama (FANS) ve belirli hücre tiplerinde etiketlenmiş çekirdeklerin izolasyonudur (BOZULMASIN). TRAP olduğu gibi, onlar çekirdekleri işaretlemek için hücre tipine özgü organizatörleri kullanmak, daha sonra sıralama yoluyla zenginleştirilmiş olsun veya aşağı çekmek, sırasıyla8,15. Tüm bu yaklaşımlar için önemli bir sorun bir doku hücrelerinin alt kümelerinden yeterli RNA malzeme elde etmektir. TRAP hücresel RNA'ların sadece bir kısmını ele geçirirken, numune toplama önemli bir darboğazdır. Bu nedenle, düşük giriş tutarlarından yüksek kaliteli veri üretmek için özellikle hassas kitaplık hazırlama protokolleri gereklidir.
Kuruluşundan bu yana, TRAP ya DNA mikrodizileri ile birlikte kullanılmıştır ya da, sıralama maliyetleri son yıllarda önemli ölçüde düştü gibi, RNA-seq10,32,33. Sablok ve ark.34'teincelenmiş çok sayıda araştırma sorusu zaten açıklığa kavuşturulmuştur. Belirli hücre türlerini hedeflemek için farklı organizatörleri birleştirerek teknik çok yönlü olduğu için önümüzdeki yıllarda daha fazla rapor takip edeceğine eminiz. Sonunda, Bu indüklenebilir bir şekilde bile yapılacak, ve birçok biyotik ve abiyotik stres faktörlerine bitkinin reaksiyonu probing ile kombine edilebilir. Ayrıca, istikrarlı transgenik hatları mevcut değildir, kıllı kök ekspresyon sistemleri de başarıyla domates ve medicago35,36TRAP gerçekleştirmek için kullanılmıştır.
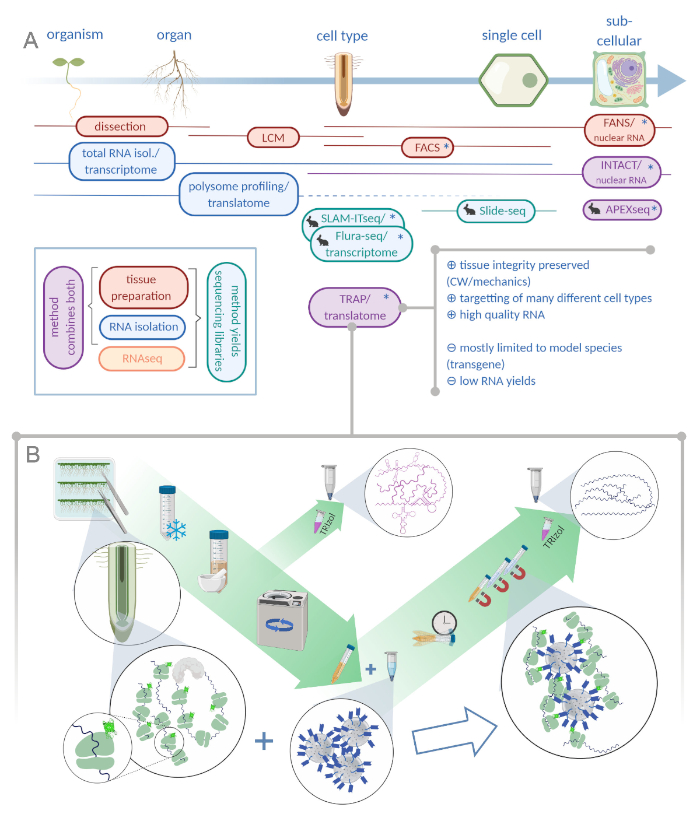
Şekil 1: Ribozom afinite arınma (TRAP) çevirisi "omiks" analiz portföyünü tamamlar. A. Analitik hassasiyet düzeylerini artırmak, aşağı tek hücreli hatta hücre altı çözünürlüğü yöntemleri veya bunların kombinasyonları bir bolluk ile elde edilebilir. Şema bitki ve hayvan alanında şu anda mevcut araçlar alabilen bir genel bakış sağlar. Hücresel çözünürlükte doku toplama lcm veya FACS gibi protokoller ile elde edilebilir, daha sonra standart transkripsime veya polizom profilleme / translatome analizi ile birleştiğinde. TRAP ve INTACT, epitop etiketlemeye dayalı olarak hem doku yakalama hem de RNA izolasyonu entegre ederler. Ancak, INTACT sadece hücre çekirdeklerini örnekler ve bu nedenle, transkripsiyon analizi özel bir durum oluşturur. Küçük bir tavşan simgesi hayvan alanında yeni geliştirilen yöntemleri işaretler: SLAM-ITseq ve Flura-seq izin veren enzim ifade hücrelerde değiştirilmiş uracil üsleri ile yeni doğan RNA metabolik targetting güveniyor iken, Slide-seq hücresel aralığında konumsal bilgi sağlayan DNA barkodları ile kaplı cam slayt kullanır. APEX-seq'da belirli hücre altı bölmelerde RNA örneklemesi için yakınlık etiketleme yaklaşımı izlenir. Özellikle, artan çözünürlük genellikle transgenik malzeme (yıldız) üretimi gerektirir ve bu nedenle bu yöntemler ağırlıklı olarak model türler için kullanılır. TRAP özellikle hücre duvarı (CW) veya mekanik sinyalizasyon ve CW matrisserbest bırakmak zor hücre türleri içeren bitki bilimi çalışmaları için uygundur. B. TRAP prosedürünün ayrıntılı ıslak laboratuvar adımları: Farklı hücre tiplerinde (örn. kök endodermisi) GFP etiketli ribozomal proteini ifade eden fideler Petri kaplarında yedi gün boyunca yetiştirilir ve kök malzemesi yapışma donma ile hasat edilir. Toplam RNA kontrol numunesi, santrifüj yoluyla enkaz peletleme önce homojenize ham ekstresi toplanır. Manyetik anti-GFP boncukimmünenfazla yapmak için temizlenmiş ekstrese eklenir. Kuluçka ve üç yıkama adımından sonra polizomla ilişkili RNA (TRAP/polizom RNA) doğrudan fenol-kloroform ekstraksiyonu ile elde edilir. LCM: lazer yakalama mikrodiseksiyon, FACS / FANLAR: floresan aktif hücre / nükleer sıralama, APEX-seq: mühendislik askorbate peroksidaz dayalı yöntem, INTACT: belirli hücre tiplerinde etiketli çekirdeklerin izolasyonu, SLAM-ITseq: dokuda RNA metabolik sıralama için thiol(SH) bağlı alkylation, Flura-seq: fluorourasil etiketli RNA sıralama (Biorender.com ile oluşturulmuş) Bu rakam daha büyük bir sürümünü görmek için lütfen buraya tıklayın.
Bu makalenin amacı, TRAP yönteminin ayrıntılı bir açıklamasını sağlamak, kritik adımları vurgulamak ve olası bir kitaplık hazırlama yöntemi için rehberlik sağlamaktır.
Genel bir TRAP deneyi temelde aşağıdaki adımlardan oluşacaktır (ayrıca bkz. Şekil 1B):(1) Ribozom etiketleme yapısının klonlanması, transgenik hat üretimi ve seçimi, tohumların büyümesi ve şişirilmesi, sterilizasyon ve kaplama, stres uygulaması/tedavisi (isteğe bağlı) ve doku hasadı; (2) doku homojenizasyonu ve ham ekstresi temizlenmesi, boncuk yıkama ve immünpurifikasyon ve yıkama adımları da dahil olmak üzere immünsafifikasyon; (3) RNA çıkarma ve kalite değerlendirmesi; ve (4) kütüphane hazırlama.
Arabidopsis kökü bir model bitki olarak piyasaya beri bitki geliştirme çalışma için bir model sistemi olmuştur37,38. Burada TRAP uygulaması bitki yanal kök gelişimi bağlamında sergilenir. Bitkilerde, tüm kök sisteminin birikimi bu programın yürütülmesine dayanır ve bu nedenle organizmanın hayatta kalması için çok önemlidir39. Arabidopsis'telateral kökler ksilem damarlarının yanında bulunan pericycle dokusundan kaynaklanır ve bu nedenle ksilem direk pericycle (XPP; bkz. Şekil 2C)40olarak adlandırılır. Kökün derinliklerinde bulunan bazı XPP hücreleri, kurucu hücre kimliği kazanır ve yerel bir hormonal tetikleyici üzerine, anticlinal41'işişirerek ve bölerek çoğalmaya başlar. Ancak, sert bir hücre duvarı matris varlığı nedeniyle, bu süreç çevre dokular üzerinde mekanik stres uygular. Özellikle, örten endodermis etkilenir, yanal kök büyüme ekseni şekilde olduğu gibi42,43,44. Gerçekten de, yeni oluşan primordium örten endodermis hücre yoluyla büyümek zorunda kalacak(Şekil 2C2) korteks ve epidermis hücreleri sadece sonunda ortaya çıkarmak için primordium için kenara itilirise 45,46. Laboratuarımızda yapılan son çalışmalar, endodermisin peridöngüsündeki çoğalmaya aktif olarak katkıda bulundugunu göstermiştir. Endodermal hormonal sinyalhedefli engelleme XPP hücrelerinde bile ilk bölümü inhibe etmek için yeterlidir47. Bu nedenle, pericycle-endodermis iletişim Arabidopsislateral kök gelişimi için çok erken bir kontrol noktası oluşturmaktadır. Ancak bu çapraz konuşmanın nasıl yapıldığı bilinmemektedir. Bu gizemi çözmek için XPP ve endodermal hücreleri hedeflemek için TRAP-seq yaklaşımını seçtik. Lateral kök programında hücreler için zenginleştirmek için, biz ekseksen bir auxin analog uygulayarak hormonal tetikleyici taklit (1-naftaneasetik asit, NAA)48, hangi aynı zamanda zamansal lateral kök oluşumunun ilk aşamasıçözmek için izin.
Protokol
1. Transgenin klonlanması, transgenik hat üretimi ve seçimi
- Uygun giriş vektöründe tercih edilen organizatörü klonlayın. PDONRP1-P249'darekombinasyon bazlı klonlama yöntemi(Malzeme Tablosu)kullanın ve pDONRP4-P1r'deki organizatörleri (tercih edilen afinitasyon etiketi veya floresan proteinle) yeniden birleştirin.
- Transgenik tohumların doğrudan seçilmesini kolaylaştırmak için RPL18 içeren giriş vektörü ile iki parçarekombinasyon reaksiyonunda, fast-red selection kaset50 ile uygun hedef vektöründe organizatör içeren giriş vektörü ile birleştirin.
- Yeniden birleştirilmiş vektörü sıralama yaparak doğrulayın ve uygun, yetkin agrobacteria'a dönüştürün. Çiçek daldırma Arabidopsis bitkiler ve 3-4 hafta hasat ve seçim T1 tohumları51sonra .
- İyi ifade eden çizgileri tanımlamak ve birden çok bağımsız satırda bildirilen organizatör etkinliğine göre ifade desenlerini doğrulamak için mikroskopiyi kullanın. Tek bir T-DNA ekleme ile temsili ifade deseni gösteren satırları seçin. Bu susturma en aza indirmek için yardımcı olabilir ve genetik haçlar için avantajlı olacaktır.
- Marker geni için homozigot olan T3 yavrularını seçin.
2. Yayılma ve sterilizasyon
- Hücre tipine özgü TRAP, RNA'yı kök başına sınırlı sayıda hedef hücreden yalıtır. Gerekli başlangıç malzemesini oluşturmak için homozigot çizgileri çoğaltın. Bu amaçla, mantar büyüme kontrolü özel bir odak ile standart büyüme koşulları kullanın.
NOT: Tek ekleme hatları elde edilemiyorsa, T-DNA kaynaklı transjenerasyonel susturma önlemek için birkaç nesil boyunca büyük popülasyonlarda toplu gruplar büyür. - Bir tur klor gazı ve bir tur %70 EtOH ile büyük miktarlarda Arabidopsis tohumunu sterilize edin.
- Tohumları 12 cm x 12 cm kare Petri tabaklarına (0,3 mL'den az tohum/tabak) eşit olarak yayın ve kurutucu veya diğer uygun bir kap içine istifleyin. Tohumların gaziçin erişilebilir olması gerektiğinden küme veya yığın oluşumundan kaçının. Rapor edildiği gibi çamaşır suyu ve HCl hacimleri ile gaz sterilizasyonunu bir gecedegerçekleştirin 52: 100 mL çamaşır suyu (%13) 60 L desiccator içinde eksi HCl 6 mL ile. Steril bir kapta tohumları toplamadan önce en az 1 saat defumigate.
DİkKAT: %37 HCl son derece aşındırıcıdır ve dikkatli kullanım gerektirir. Klor gazı zehirlidir, duman kaputu kullanın. - Tabak başına 0,1 mL kuru, gazla sterilize edilmiş tohumları alın ve oda sıcaklığında sterilizasyon çözeltisi (%70 EtOH, %0,01 Ara) ile karıştırın. 20 dk kuluçka, etOH decant ve steril H2O ile 3-4 kez tohumları yıkayın.
- ıslatılmış tohumları 50 mL tüplere aktarın ve steril %0,1 agar ile seyrelterek plaka başına 1 mL imbibed tohum bulamacı (0,1 mL tohum/1 mL bulamaç) elde edin.
NOT: Transgen entegrasyon olayları nedeniyle, bitki hatları farklı sterilizasyon tekniklerine duyarlı olabilir; özellikle EtOH kuluçka süresinin kritik olduğu bulunmuştur. Elimizde, çift sterilizasyon adımları deneyler sırasında mantar kontaminasyonunu önlemek için gerekli ydi. Bu, tek bir zaman noktasının kirlenmesi tüm deneyi engellediği için zaman serileri yaparken özellikle önemlidir. Yerel büyüme koşullarına bağlı olarak çift sterilizasyona her zaman ihtiyaç duyulmayabilir.
- Tohumları 12 cm x 12 cm kare Petri tabaklarına (0,3 mL'den az tohum/tabak) eşit olarak yayın ve kurutucu veya diğer uygun bir kap içine istifleyin. Tohumların gaziçin erişilebilir olması gerektiğinden küme veya yığın oluşumundan kaçının. Rapor edildiği gibi çamaşır suyu ve HCl hacimleri ile gaz sterilizasyonunu bir gecedegerçekleştirin 52: 100 mL çamaşır suyu (%13) 60 L desiccator içinde eksi HCl 6 mL ile. Steril bir kapta tohumları toplamadan önce en az 1 saat defumigate.
3. Kaplama
- Bu adımları önceden hazırlayın. 1/2 MS plakaları dökün (pH 5.8) deney için gerekli miktarlarda %1 agar ile (numune/zaman noktası başına 20-30). Ucuçapı jiletle yaklaşık 3-4 mm'ye kadar büyütmek için 1 mL pipet ucu kesin. İpuçlarını otoklavla. Kare Petri çanak kapakları ile plaka başına tohum üç satır kaplama için bir şablon tutucu oluşturun. Steril bir çalışma ortamı sağlamak ve işlenecek plakaları etiketlemek için laminar akış başlığı hazırlayın.
NOT: Aynı anda çok sayıda plaka işlenirse, renkli etiketler etiketlemeyi hızlandırabilir. - Boş agar tabaklarını şablon tutucuya yerleştirin ve 1 mL'lik imbibed tohumu üç satırüzerine eşit olarak dağıtın. İşlenmiş tabakları tohumlar kuruyana kadar (yani agar yüzeyine yapışın) laminar akışa yığınlar halinde yerleştirin. Agar da kuruyacağı için tabakları daha uzun süre bırakmayın.
- Tohumlar yeterince kuruduktan sonra kapakları kapatın ve her tabağı mikropore bantla kapatın. Tohumları karanlıkta 4 °C'de iki gün boyunca tesniye edin ve daha sonra bir büyüme odasına yerleştirin.
4. Doku tedavisi (isteğe bağlı)
NOT: Bu protokolde Arabidopsis köklerinin sentetik auxin varyantı NAA ile eksojen tedavisini ana hatlar. Eldeki deneysel soruya bağlı olarak, bu bölümün ayarlanması veya tamamen atlanabilmesi gerekir.
- 1,5 - 2 cm yüksekliğinde ve 10 cm uzunluğunda kağıt mendil şeritler hazırlayın. Uzatılmış kuluçka süreleri, dokunun kullanılmadan önce otoklavlatılmasını gerektirir.
- Hormon tedavisi geçmesi gereken tüm plakalar mikropore bant çıkarın. 10 mM NAA'nın seyreltilmesi 1 mL 'lik (DMSO'da çözünmüş) 1 L sıvı, otoklavlı 1/2 MS çözeltisi (pH 5.8) ve doku kağıdını çözeltide ıslatın (10 μM NAA).
- Köklerin her satırı üzerine kağıt mendil bir şerit uygulamak için cımbız kullanın. Hava kabarcıklarını çıkarmak için parmaklarınızı hafifçe kullanın. Plakadan fazla sıvıyı boşaltın, kapağı kapatın ve plakayı zamanla etiketleyin. Uzun kuluçka süreleri için, plakaları büyüme odasına geri yerleştirin.
5. Hasat
- Her biyolojik çoğaltma/zaman noktası/işleme için plakaları alın. Farklı doku örnekleri için temiz bir Dewar kabıve etiket tüplerinde (15 veya 50 mL) sıvı nitrojen toplayın. Bir Strafor tutucu hazırlayın.
DİkKAT: Sıvı nitrojen işleme prosedürlerine (havalandırma, donma, potansiyel olarak patlayan tüpler) aşina olun. - Plakayı açın ve kökleri agar yüzeyinden ayırmamaya dikkat edin. Cerrahi bir bıçakla, tek, kararlı bir inme de ateş-kök-kavşak boyunca satır başına bir kez kesilir. Keskinliği garanti etmek için bıçakları numuneler arasında temizleyin ve sık sık değiştirin.
- Cımbızla, her satırın kökleri boyunca kaydırarak üç demet halinde toplayın. Kökleri kapmak ve dondurma snap sıvı nitrojen dolu bir 50 mL tüp içine boşaltın.
NOT: Bir sonraki adımda öğütülmesi zor olduğundan kökleri yoğun yapılara (toplar gibi) monte etmeye çalışmayın. - Bir numune oluşturan tüm plakalarla (kuluçka süreleri sırasına göre) devam edin ve fazla sıvı nitrojen dökün. Köklerin dökülmesini önlemek için tüp kapağını kullanın. Sonra kapağını kapatın ve Dewar gemide tüm tüpleri toplamak. Kök dokusunu -80 °C'de saklayın.
6. İmmünopurifikasyon
NOT: Bu adım yüksek kaliteli TRAP/polizom RNA elde etmeyi amaçlamaktadır. Bu nedenle, kesinlikle RNA işleme için iyi uygulama tavsiye izleyin. Steril bir tezgah içinde bu bölümdeki tüm adımları gerçekleştirin ve bir RNase kaldırma çözeltisi(Malzeme Tablosu)ile tüm ekipman ve labware temizleyin. Eldiven giyin ve numune, buz veya temizlenmemiş diğer kaynaklarla kirlendiğinde hemen değiştirin. Bu çok önemli bir yönü olduğundan, atık bertaraf tavsiyesi ile birlikte ekipman yeniden bir bölüm dahildir.
- Arabellek hazırlığı
- Tablo 1 ve otoklav (A) veya filtre sterilize (1) göre stok çözümleri hazırlamak. Aksi belirtilmedikçe, çözücü RNase içermeyen sudur.
- Tablo 1'de belirtildiği gibi dithiothreitol (DTT), fenilmetilsülfonil florür (PMSF), sikloheximid (CHX) ve kloramfenikol (CAM) çözücülerini tablo 1'de belirtildiği gibi eritin ve aliquot dithiothreitol (DTT), feniltilsülfonil florür (PMSF), siklohekmid (CAM) ve -20 °C'de saklayın. Diğer tüm stoklar oda sıcaklığında kalabilir.
- Her ekstraksiyondan önce zaman alan tampon karıştırmayı önlemek için , yıkama tamponu (WB) için 1-4 ve poliom bazlı ekstraksiyon tamponu (PEB) için 1-6 malzemeyle stokları önceden karıştırın. Böylece, sadece su ve dondurulmuş maddeler (7-10) çıkarma gününde ekleyin. Önceden karıştırılmış stokları ve RNase içermeyen suyu 4 °C'de saklayın.
NOT: GFP ile nanobody etkileşimi yüksek DTT konsantrasyonlarına duyarlı olduğundan, DTT konsantrasyonu Zanetti ve ark. 2005'ten bildirilen konsantrasyonun 1/5'idir.
| Malzemeler | Stok konsantrasyonu | 50 mL WB* için mL hacim ekleme | 50 mL PEB için mL hacim ekleyin* | ||
| 1 | Tris, pH 9 | A | 2 M | 5 | 5 |
| 2 | Kartal | A | 2 M | 5 | 5 |
| 3 | EGTA | A | 0,5 M | 2.5 | 2.5 |
| 4 | MgCl2 | A | 1 M | 1.75 | 1.75 |
| 5 | Pte | A | %20 (v/v) | 0 | 2.5 |
| 6 | deterjan karışımı | A | 0 | 2.5 | |
| Tween 20 | %20 (v/v) | ||||
| Triton-X 100 | %20 (v/v) | ||||
| Brij-35 | %20 (w/v) | ||||
| Igepal | %20 (v/v) | ||||
| 7 | Dtt | ₳ | 0,5 M | 0.1 | 0.1 |
| 8 | PMSF | ₳ | 0.1 M (İzopropanol) | 0.5 | 0.5 |
| 9 | Siklohekmid | ₳ | 25 mg/mL (EtOH) | 0.1 | 0.1 |
| 10 | Kloramfenikol | ₳ | 50 mg/mL (EtOH) | 0.05 | 0.05 |
Tablo 1: Arabellek kompozisyonu ve karıştırma tavsiyesi. Verilen miktarlarda karıştırılan stok konsantrasyonları olan malzemeler 50 mL WB veya PEB verir. Tris: tris-(hidroksimetil)-aminometan, EGTA: etilen glikol-bis(β-aminoetil eter)-N,N,N,N,N,N,N'-tetra-asetik asit, PTE: Polioksietilen-(10)-tridesetil eter, A: otoklav, filtre-sterilize; *RNase içermeyen su ile 50 mL'ye kadar doldurun.
- Doku homojenizasyonu/taşlama
- Santrifüjü soğutun ve homogenizers ve santrifüj tüpleri buz üzerine yerleştirin. DTT, PMSF, CHX ve CAM'in eritme aliquot'larını çöz. Günün gereksinimlerine göre 50 mL tüplerde (numunelerin#) stok çözeltilerinden PEB ve WB'yi karıştırın ve buz üzerinde soğutun.
NOT: PMSF'nin sudaki yarı ömrü sadece 30 dk olduğundan, kullanımdan kısa bir süre önce PMSF ekleyin. - Bir Dewar kabında bol miktarda sıvı nitrojen hazırlayın ve -80 °C depolamadan doku örnekleri alın. Soğuk havan toplarından kaynaklanan yanıkları önlemek için standart laboratuvar eldivenlerinin altına pamuklu eldivenler takın. Onlar taşlama izin verecek kadar soğuk olana kadar harçlar ve pestles içine sıvı nitrojen dökün. Harçları ayırt etmek için bir sistem tasarlamak önerilir (etiketveya belirli bir sırada tutmak).
- Bir harç içine boş doku örneği ve tüm malzeme beyaz bir toz olana kadar dikkatle eziyet. Gerekirse, doku dondurulmuş tutmak veya daha iyi taşlama kolaylaştırmak için sıvı nitrojen ekleyin.
- Numuneye 5 mL PEB ekleyin ve tampon donmadan önce tozla hızlıca karıştırın. Bu örnek çözülürken (zaman zaman karışım) başka bir numuneyi işler.
- Karışım aktarılır aktarılabildiği anda bulamacı cam bir homojene boşaltın ve buzda tutun. 2 mL peb ile harcı ve havanelidurur ve homogenizer numuneye ekleyin.
NOT: RNA bozulmasına izin verdiği için tamamen sıvı bir numuneden kaçının. - Ekstre homojen olana kadar bulamacı elle öğütün. En az 4-5 dalma öneririz.
NOT: Bulamacın daha fazla çözülmesi için biraz ek bekleme süresi gerekebilir. Homogenizers kullanımı bazı çalışkanlık gerektirir. Kaba kuvvet uygulamayın ve emme kuvvetlerine karşı dikkatli olun. Dikkate alınmazsa, bu homogenizer dökülmesi, kirlenme veya imha yol açacaktır. - Ham kök ekstresini 50 mL'lik bir santrifüj tüpüne dökün (buzda tutun).
NOT: Genellikle transferden önce birkaç örnek öğütülmüş olabilir. Taşlama, aktarma ve homojenleştirme paralel işleme gereklidir. Hızlı çalışmaya çalışın ama acele etmeyin; sakin ol. Homojenize örnekleri her zaman buzüzerinde saklayın.
- Santrifüjü soğutun ve homogenizers ve santrifüj tüpleri buz üzerine yerleştirin. DTT, PMSF, CHX ve CAM'in eritme aliquot'larını çöz. Günün gereksinimlerine göre 50 mL tüplerde (numunelerin#) stok çözeltilerinden PEB ve WB'yi karıştırın ve buz üzerinde soğutun.
- Toplam RNA örnek toplama
- Her ham numunenin 200 μL aliquot'larını temiz bir mikrosantrifüj tüpüne aktarın (önceden buz üzerinde etiketlenmiş ve soğutulmuş).
- 7.1 ve 7.2 noktalarındaTRAP örnekleri için ayrıntılı olarak rna ekstraksiyonu ile devam edin. Örnekler santrifüjde temizlerken bu adımları yapın.
- DNA kontaminasyonunu ortadan kaldırmak ve reaksiyonu ticari bir kit(Malzeme Tablosu)kullanarak temizlemek için resuspended total RNA ile dNase tedavisi gerçekleştirin.
NOT: Toplam RNA ekstraksiyonları genellikle yüksek konsantrasyonlarda verim verir ve numunelerin önemli ölçüde seyreltilmesi gerekir. Hassas Qubit protokolü ile seyreltme sonrası konsantrasyonun ölçülmesini öneririz.
- Ham özü temizleme
- 6.2.7'den örnekleriçeren buz kovasını alın ve 16.000 x g ve 4 °C'de 15 dk santrifüj edin.
NOT: Santrifüjdengelemek için, buna göre örnekleri eşleştirin. Bunun tamamen mümkün olmaması durumunda, PEB ekleyerek bir örneği ayarlayın. - Supernatant'ı taze bir santrifüj tüpüne dökün (önceden buzda soğutulmuş) ve santrifüjü tekrarlayın (16.000 x g ve 4 °C'de 15 dk). Bu aktarım, santrifüjün yanında hızlı bir şekilde gerçekleştirilebilir.
- Ham ekstresi temizlerken, adım 6.6 için GFP-boncukyıkama başlatın.
NOT: Shaker üzerinde sallanan için bu buz kovası tutun ama kontamine olabilir gibi steril tezgah içine geri yerleştirin.
- 6.2.7'den örnekleriçeren buz kovasını alın ve 16.000 x g ve 4 °C'de 15 dk santrifüj edin.
- Boncuk yıkama
- Aliquot manyetik GFP-boncuklar (#samples x 60 μL, Malzeme Tablosu)1,5 mL tüp içine. Manyetik standa yerleştirin. Boncuklar toplandıktan sonra, supernatant çıkarın.
- 1 mL soğuk WB ekleyin, boncukları yeniden askıya alın ve tekrar toplayın. Yıkama tamponu atın ve 1 mL WB ile bir kez daha tekrarlayın.
- Sonuç olarak, WB'deki boncukları adım 6.5.1'de kullanılan ilk hacmine yeniden askıya alın.
- İmmünopurifikasyon (IP)
- Santrifüjden hemen sonra, temizlenmiş supernatant'ı etiketli 15 mL tüplere dökün ve numune başına 60 μL yıkanmış boncuk ekleyin.
- Tüm örnekleri yatay olarak buz kovasına yerleştirin ve çalkalayıcıya koyun. GFP etiketli polizomları boncuklara bağlamak için karışımın 2 saat kuluçkaya yatmasını sağlar.
- 15 mL tüpler (buz üzerinde) için manyetik stand üzerinde boncuk toplamak ve kalan PEB PMSF ekleyin. Supernatant atın. Boncuklara yaklaşık 5 mL PEB dökün ve yatırarak tekrar askıya alın. Bölüm 6.6.2'deki gibi aynı kurulumda 15 dk boyunca numuneleri çalkalayın.
- WB ile yıkıntıları toplam 3 yıkama (1 x PEB, 2 x WB) tekrarlayın. Her arabellek değişiminden önce PMSF ekleyin.
- Boncukları 1 mL WB'de toplayın ve 1,5 mL'lik bir tüpe aktarın. Son olarak, manyetik standı üzerinde boncuk bir kez daha toplamak ve tüm sıvı kaldırın. Tüpü kapatın ve tüm numuneler işlenene kadar buzda tutun.
- RNA çıkarma için bir duman başlık için örnekleri taşıyın.
- Atık ların bertaraf edilmesi ve laboratuvar malzemelerinin yenilenmesi.
- İyi bir laboratuvar uygulamasına göre yapılırsa (bkz. bölüm 2.2.1), sterilizasyon işlemi sulu bir NaCl çözeltisi verir. Klor gazı, yanı sıra artık HCl ve çamaşır suyu, duman kaputunda defumigate bırakın.
- PEB ve WB bertaraf: CHX yüksek pH'da ayrıştırken, tüm sıvıları toplayın ve pH>9'a getirin. Sıvı atıkları halojenli kimyasal atıklara atın. Tüm katı maddeler (dokular, serolojik pipetler, eldivenler, vb.) kimyasal atık olarak atılmalıdır.
- Fenol içeren sıvıları ayrı ayrı toplayın, ayrıca fenol lemine olmuş maddeleri (uçlar, tüpler ve eldivenler) toplayın.
- El yıkama harçları, pestles ve homogenizers (sünger ve fırça) sabun ve iyice durulayın. Daha sonra malzemeyi bir gecede >220 °C'de pişirin. Ya tedaviden önce folyo ya da ısı yalıtımlı, kapalı bir konteyner içine yerleştirin.
- Fırça temiz santrifüj tüpler deterjan ve sonra diethylpyrocarbonate (DEPC)-duman kaputunda tedavi. Bu amaçla, deiyonize suya sıvı DEPC ekleyin (1 mL DEPC ila 1 L H2O) ve sallayarak karıştırın. Santrifüj tüplerini dökülen DEPC-su yakalar otoklavable tepsi üzerine yerleştirin. Tüpler içine süspansiyon dökün ve 3 saat veya gece bırakın. DEPC sonraki otoklavlama işleminde ayrışır.
DİkKAT: DEPC son derece zehirlidir.
7. RNA çıkarma ve QC
- RNA çıkarma
- 4 °C'ye kadar masa üstü santrifüjsoso.
- Her numuneye 1 mL asit-guanidinium-fenol bazlı reaktif(Malzeme Tablosu)ekleyin, boncukları veya toplam RNA bulamacını yeniden askıya almak için ters çevirin ve 5 dakika boyunca buz üzerinde kuluçkaya yatırın. Girdap etmeyin!
- 200 μL kloroform ekleyin ve buz üzerinde 3 dakika kuluçka. Sonra iyice örnekleri girdap.
- Faz ayrımına yardımcı olmak için, santrifüj maksimum. hızı 10-15 dk, 4 °C.
- Etiket 1.5 mL düşük tutma tüpleri(Malzeme Tablosu)ve aliquot 650 μL isopropanol her içine.
- Üst sulu fazı (650°L) dikkatlice alın ve izopropanol ile hazırlanan tüplere aktarın. Pembe organik faz dokunmadan kaçının.
- RNA'yı geceleme -20 °C'de çökeltin.
NOT: Numunelerin izopropanol'de -20 °C veya -80 °C'de saklanıp sadece gerektiğinde suda suda çözünmesi tavsiye edilir. Sulu RNA haftalarca/aylarca depolandığında -80 °C'de bile bozulur.
- RNA yağış
- 4 °C'ye kadar masa üstü santrifüjsoso.
- Taze %80 EtOH'u RNase içermeyen su ile hazırlayın ve -20 °C'de (-80 °C'de 5 dk) soğuması işlemi hızlandırmaya yardımcı olur.
- Numuneleri maksimum hızda (yaklaşık 13.000 x g)30 dk için santrifüj edin ve süpernatantı atın. Pelet görünür olmayacak, bu yüzden dikkatli bir şekilde pipet sanki orada. 1 mL soğuk %80 EtOH ekleyin ve tüpü bir veya iki kez ters çevirin.
- Maksimum hızda 30 dakika tekrar santrifüj ve iki yıkama toplam yıkama tekrarlayın.
- 2 dakika boyunca aşağı doğru döndürün ve 10 μL uçlu tüm artık EtOH'u çıkarın. Peleti oda sıcaklığında 3-5 dakika (daha fazla değil) kurumaya bırakın ve 20 μL RNase içermeyen suda tekrar bekletin.
- Numuneleri buzda tutun ve mümkün olan en kısa sürede kalite kontrol yapın. Örnekleri -80 °C'de saklamaya devam edin. Donma-çözülme döngülerinden kaçının.
- Üreticinin tavsiyelerine göre özel ekipman(Malzeme Tablosu)kullanılarak kalite kontrol.
8. Kütüphane hazırlama
- SMARTer v4 Ultra Düşük Girişli RNA Kiti ile cDNA sentezi ve amplifikasyon
- Her numunenin seyreltilmesini 4,75 μL hacimde 1,5 ng TRAP-RNA veya toplam RNA'ya sahip olarak hesaplayın. Tüm reaksiyonları PCR-tüplerde gerçekleştirin ve numuneleri taze RNase içermeyen su ile seyreltin.
- Üreticinin önerilerine göre tüm adımları 1/2 reaksiyon hacimleri ile gerçekleştirin. CDNA'yı 12-13 PCR çevrimiyle yükseltin.
- PCR'yi 0,5 μL 10x lysis tampon ve 25 μL SPRI boncuk(Malzeme Tablosu)ekleyerek temizleyin. Birçok örnek işlenirse lysis tampon ve boncuklar önceden karıştırılabilir. Boncuklar borulama önce eşit dağılmış olduğundan emin olun.
- Protokolü tam tepkihacimlerinde (17 μL elüsyon arabelleği) uygulayın. Boncukları 3 dakikadan fazla kurutmayın. Aşırı kurutulmuş numuneler uzun kuluçka süreleri ile potansiyel olarak kurtarılabilir.
- Qubit HS DNA kiti ile örnek konsantrasyonlarını ölçün.
NOT: SMARTer v4 kiti 200 pg'ye kadar girişi tolere edebilir. Qubit değerlerinin 16 çevrim PCR ile belirlenemeyen (250 pg, algılama sınırının altında) olduğu durumlarda kütüphaneler elde ettik. Ancak, sınırlı giriş malzemesi de daha az karmaşık kitaplıklar verebilir.
- Nextera XT DNA Kütüphane Hazırlama Kiti ile parçalanma ve adaptör ligasyon PCR
- Bir PCR tüpünde 200 pg/μl ve pipet 1,25 l konsantrasyon elde etmek için cDNA'yı RNase içermeyen su ile seyreltin.
- Reaksiyon hacimleri ile üreticiye göre tüm adımları gerçekleştirin. Bir sıralama havuzuna ait numuneler için cDNA'yı 12 PCR çevrimi ve uyumlu adaptörlerle yükseltin. Illumina'nın Indeks Kitleri A ve D ile 384'e kadar örnek çokkatlı olabilir.
- PCR temizleme için 12,5 μL resuspension tampon ve 22,5 μL SPRI boncuk (0,9x oranı) ekleyin. Numuneyi 22 μL elüsyon tamponu ile verin.
NOT: QC ve havuzlama sıralama şirketi(Malzeme Tablosu)tarafından gerçekleştirildi ve bu nedenle boncuk bazlı normalleştirme gerekmedi. Enzimomatik parçalanma reaksiyonu (tagmentation) her enzim sadece bir kez kestiği için malzeme girişine karşı çok hassastır. Bu nedenle, konsantrasyon tavsiyesini aşmayın.
Sonuçlar
Kalite değerlendirmesi için, yukarıda belirtilen prosedür birkaç ara adımda incelenmelidir: planta'da ifade deseni doğrulaması, izole edilmiş polizomal RNA'nın ve son kütüphanelerin kalite kontrolü. qRT-PCR bilinen marker genleri kullanarak, ek olarak, tedavi durumuna yanıtı onaylamak veya deneysel koşullar ince ayar yapılabilir.
GFP sinyal dağılımının konfokal analizi
Hem endodermal hem...
Tartışmalar
RPL18 yerelleştirme deseni doğrulaması
Herhangi bir TRAP deneyinden elde edilen verilerin yanlış yorumlanmasını önlemek için çok önemli olan, etiketli ribozomal alt birimin uygun ifade desenidir. Bu nedenle, RPL18 bir epitop etiketi olarak GFP dahil çok zarif istenilen ifade deseni doğrulama ve ardışık, aynı dokudan polizom fraksiyonu çekme sağlar. Uygun organizatör desenleri sağlamak için daha invaziv yaklaşımlar Jiao ve Mayerowitz 2010, GUS boyama gerektirir ve Tian ve ark 20...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Zürih Genetik Çeşitlilik Merkezi'nden Jean-Claude Walser'a bu projenin erken evresinde önemli uzman tavsiyeleri için teşekkür ederiz. Vermeer laboratuvarındaki çalışmalar, JEMV'ye verilen İsviçre Ulusal Bilim Vakfı'ndan (SNSF) SNF Profesörlük hibesi (PP00P3_157524) ve R'EQUIP ekipman hibesi (316030_164086) tarafından desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120x120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only - SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC | |
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT | |
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA | |
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG | |
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
Referanslar
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır