需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
翻译核糖体亲和纯化(TRAP)以研究细胞类型特异性尺度下阿拉伯生物基塔利亚纳根发育
摘要
翻译核糖体亲和纯化(TRAP)提供了以最少的器官和组织处理来解剖发育方案的可能性。该协议从绿色荧光蛋白(GFP)标记的核糖体亚组细胞中产生高质量的RNA。下游分析工具(如 qRT-PCR 或 RNA-seq)可显示组织和细胞类型特定的表达特征。
摘要
在本文中,我们给出了通过翻译核糖体亲和纯化(TRAP)方法和连续优化的低输入库制备从不同阿拉伯生物基系thaliana根细胞类型获取的翻译数据的实践说明。
作为起始材料,我们使用植物生产线,使用适当的促进剂,以细胞类型特定的方式表达GFP标记核糖蛋白RPL18。在免疫纯化和RNA提取之前,组织被卡扣冷冻,从而保持组织的完整性,同时允许以高时间分辨率执行时间序列研究。值得注意的是,细胞壁结构保持不变,这是替代程序的一个主要缺点,例如荧光激活细胞排序方法,依靠组织原面分离不同的细胞群。此外,无需像激光捕获微解剖技术那样组织固定,从而获得高质量的RNA。
然而,从细胞亚群的取样,仅分离多体相关RNA,严重限制了RNA的产生。因此,有必要应用足够灵敏的库制备方法,以便RNA-seq成功采集数据。
TRAP 为植物研究提供了理想的工具,因为许多开发过程都涉及与细胞壁相关和机械信号通路。利用促进剂瞄准特定细胞群,正在缩小器官和单细胞水平之间的差距,而单细胞水平又因分辨率小或成本非常高而受到影响。在这里,我们应用TRAP来研究侧根形成的细胞-细胞通信。
引言
在下一代测序技术日益应用的推动下,空间分辨率在发育生物学中可以增强。当代研究旨在解剖组织到专门的细胞类型,如果不是单细胞水平11,2,3,4。2,3,4为此,在过去五十年中,已设计了大量不同的方法(见图1A)5、6、7、8、9、10、11、12、13、14、15。,14,,7,8,9,10,11,12,135,6 Figure 1A15
植物科学中的许多工具都是对动物研究首创的技术的适应。我们在此处详细介绍的方法不是这样。2005年,凭借蛋白质翻译的强势背景,贝利-塞雷斯实验室开始设计核糖蛋白,用于后续的亲和力纯化16。因此,它们可以避免耗时和劳动密集型的多体分析,这是基于超中心与蔗糖梯度,并用于评估翻译核糖体自20世纪60年代17,18。17,该方法自即被称为转化核糖体亲和纯化(TRAP)16。在成功的植物翻译研究后,Heiman19等人将其应用扩展到酵母20、果蝇21、异种鱼23、24。,2422
虽然模型系统的遗传改造是TRAP的先决条件,它将其应用限制在适应遗传转化的物种,人们可以同时利用这种异议,以针对特别感兴趣的细胞子集,否则极难从完整的组织/器官25(例如,在小鼠大脑中高度分支的树突状细胞或受感染的植物组织中的真菌催眠)。在植物中,所有细胞都通过细胞壁固定到位,形成静水骨架26的基础。为了将植物细胞从这个基质中释放出来,科学家要么通过激光捕获微切(LCM)27从周围组织中物理地切27出细胞,要么对细胞壁进行酶消化28。在后一个细胞中,所谓的正代,感兴趣的种群被荧光标记,可以通过荧光激活细胞分拣(FACS)7分离。7LCM通常需要将样品固定并嵌入蜡中,这最终会降低其RNA29的质量。基于FACS的方法产生高质量的RNA,但提供过程本身引入了基因表达的差异30和具有修饰和厚的继生细胞壁组织是出了名的难以治疗。此外,植物中的许多发育过程被认为是依靠机械传输的信号,因此细胞壁的完整性是最重要的31。两种方法,使用快捷方式,通过在核水平上操作来规避细胞分离,是荧光激活核分拣(FANS)和特定细胞类型(INTACT)标记的核分离。与TRAP一样,它们使用细胞类型特定的促进器来标记核,然后通过排序或拉下获得丰富,分别为,8,15。所有这些方法的一个主要挑战是从组织中的细胞子集获得足够的RNA材料。由于 TRAP 只捕获蜂窝 RNA 的一小部分,因此样本采集是一个相当大的瓶颈。因此,需要特别敏感的库准备协议来生成低输入量的高质量数据。
自建立以来,TRAP 要么与 DNA 微阵列结合使用,要么随着近年来测序成本的显著下降,RNA-seq 10、32、33 。10,32,33在Sablok等人34日回顾了许多研究问题。我们相信,在未来几年中,将会有更多的报告,因为结合不同的启动子来靶向特定的细胞类型时,该技术非常通用。最终,即使以可诱导的方式完成,也可以结合探测植物对许多生物和非生物应激因素的反应。此外,在没有稳定的转基因线的情况下,毛茸茸根表达系统也已成功用于在番茄和药质35,36,36中执行TRAP。
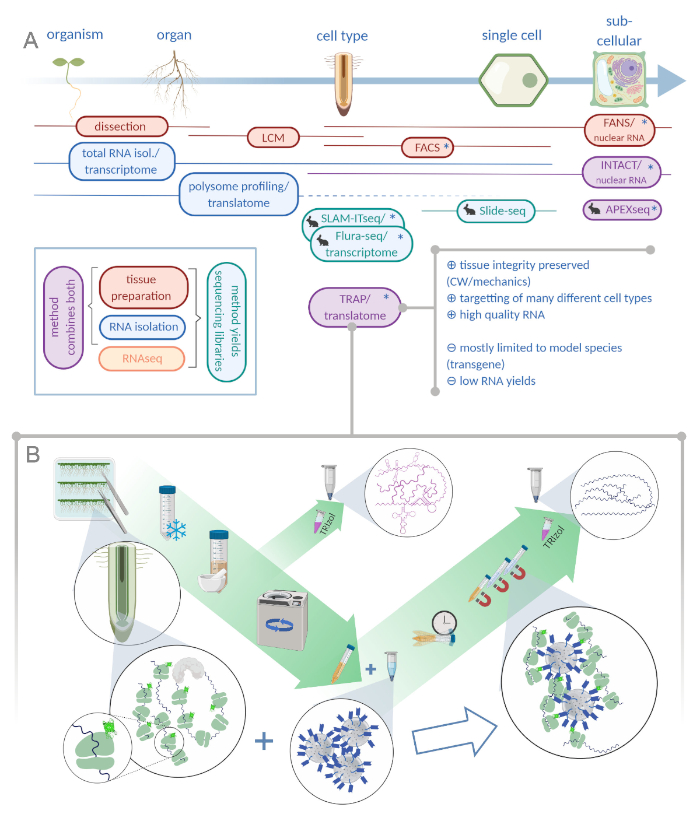
图1:翻译核糖体亲和力纯化(TRAP)补充了"经济学"分析组合。A.通过大量方法或组合,可以提高分析精度,甚至单细胞甚至亚细胞分辨率。该计划概述了目前在植物和动物领域可用的工具。细胞分辨率下的组织收集可以通过LCM或FACS等协议实现,然后与标准转录组或多体分析/翻译分析耦合。TRAP 和 INTACT 将组织捕获和 RNA 分离结合在一起,因为它们基于表上标记。然而,INTACT只样本细胞核,因此,构成转录分析的特殊情况。一个小兔子图标标记了动物领域新开发的方法:虽然SLAM-ITseq和Flura-seq依赖于在表达放任性酶的细胞中具有改性尿素碱的新生RNA的代谢靶向,Slide-seq使用带有DNA条形码的涂层玻璃幻灯片,在细胞范围内提供位置信息。APEX-seq 中遵循接近标记方法,在特定亚细胞隔间中对 RNA 进行采样。值得注意的是,分辨率提高通常需要生成转基因材料(星号),因此这些方法主要用于模型物种。TRAP 特别适用于涉及细胞壁 (CW) 或机械信号的植物科学研究,以及难以从其 CW 基质中释放的细胞物种。B. TRAP程序的详细湿实验室步骤:在不同细胞类型(如根内皮)中表达GFP标记核糖蛋白的幼苗在培养皿上生长7天,通过卡扣冷冻收获根料。在通过离心对碎片进行颗粒化之前,从均质原油提取物中采集总RNA控制样本。磁性抗GFP珠被添加到清除的提取物中,以执行免疫沉淀。孵化和三洗步骤后,通过酚-氯仿提取直接获得多体相关RNA(TRAP/多体RNA)。LCM:激光捕获微切解剖,FACS/FANS:荧光激活细胞/核分拣, APEX-seq:基于工程抗坏血剂过氧化物酶的方法,INTACT:特定细胞类型中标记的核分离,SLAM-ITseq:用于组织RNA代谢测序的THiol(SH)相关烷基化,Flura-seq:氟酸酯标记RNA测序(用Biorender.com创建)请点击这里查看这个数字的较大版本。
本文的目的是提供 TRAP 方法的详细说明,以突出显示关键步骤,并为可能的库准备方法提供指导。
通用 TRAP 实验将基本包括以下步骤(另见图1B):(1)植物材料的准备,包括核糖体标记结构的克隆、转基因生产线的生产和选择、种子的生长和扩充、灭菌和电镀以及应力应用/处理(可选)和组织收获;(二)免疫纯化,包括组织均质化、清纯粗提取物、珠洗、免疫净化、洗涤步骤;(3) RNA提取和质量评估;(4) 图书馆准备。
阿拉伯植物根自引进示范植物37、38,38以来,一直是研究植物发展的示范系统。在这里,TRAP的应用在植物横向根开发的背景下被展示出来。在植物中,整个根系的建立依赖于这个程序的执行,因此对生物体39的生存非常重要。在Arabidopsis中,侧根起源于位于赛莱默血管旁边的围轮组织,因此被称为木质杆围轮(XPP;参见图2C)40。40一些XPP细胞,位于根的深处,获得一个创始人细胞的身份,并在局部荷尔蒙触发下,开始通过肿胀和分裂41开始增殖。然而,由于存在刚性细胞壁基质,这一过程对周围的组织施加机械应力。特别是,覆盖内皮受影响,因为它是在横向根生长轴42,43,4443,44的方式。42事实上,新形成的原体将不得不通过覆盖的内皮细胞(图2C2)生长,而皮层和表皮细胞只是被推到一边,使原体最终出现45,46。,46我们实验室最近的工作表明,内皮病正在积极推动适应围周期的增殖。靶点阻塞内皮激素信号足以抑制甚至第一分裂在XPP细胞47。因此,围周期-内皮斯通信是阿拉伯细胞横向根系发展的一个早期检查点。然而,不知道这个相声是如何进行的。为了解开这个谜团,我们选择了TRAP-seq方法来瞄准XPP和内皮细胞。为了丰富横向根程序中的细胞,我们通过外生应用辅助辛酸(1-naph甲酸,NAA)48来模拟荷尔蒙触发,同时允许暂时解决侧根形成48的初始阶段。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 转基因克隆、转基因生产线生产和选择
- 在适当的入口矢量中克隆所选择的启动子。使用基于重组的克隆方法(材料表),并使用pDONRP1-P249中基于重组的克隆重新组合pDONRP4-P1r.克隆RPL18(具有亲和力标记或荧光蛋白)。
- 使用 FAST-RED 选择盒50将包含RPL18的入口矢量与包含启动子的入口矢量在两个片段重组反应中合并到适当的目标矢量中,以方便直接选择转基因种子。
- 通过测序验证重组的病媒,并将其转化为合适、称职的农细菌。花浸阿拉伯植物和3-4周收获后,选择T1种子51。
- 使用显微镜识别表达良好的行,并根据多个独立行中报告的启动器活动验证表达模式。选择显示具有代表性的表达模式的线,并插入单个 T-DNA。这可能有助于减少沉默,并将有利于遗传交叉。
- 选择标记基因的同源基因T3后代。
2. 传播和灭菌
- 细胞类型特异性TRAP从每个根的有限数量的目标细胞中分离RNA。要生成所需的起始材料,应传播同源线。为此,使用标准生长条件,特别注重真菌生长控制。
注:如果无法获得单插入线,则在几代大种群中成批生长,以避免T-DNA引起的跨代沉默。 - 用一轮氯气和一轮70%的EtOH对大量阿拉伯化种子进行消毒。
- 将种子均匀地铺在 12 厘米 x 12 厘米方形培养皿(小于 0.3 mL 种子/板)上,并将其放入干燥器或其他合适的容器中。避免团状或堆形成,因为种子需要接触气体。在漂白剂和 HCl 体积下进行气体灭菌,报告为52: 100 mL 漂白剂 (13%)60 L 干燥器中具有 6 mL 的 Conc. HCl。在无菌容器中收集种子之前,请至少1小时进行脱泡。
注意:37% HCl 具有高度腐蚀性,需要小心处理。氯气是有毒的,使用烟气罩。 - 每盘取0.1 mL干式气体灭菌种子,并在室温下与灭菌溶液(70%EtOH,0.01% Tween)混合。孵育20分钟,切碎EtOH,用无菌H2O洗涤种子3-4次。
- 将浸泡的种子转移到50 mL管中,用无菌0.1%的龙gar稀释,每盘获得1 mL浸渍种子浆料(0.1 mL种子/1 mL浆)。
注:由于转基因整合事件,植物系可能容易受到不同的灭菌技术的影响;特别是EtOH孵育时间被发现是至关重要的。在我们手中,双灭菌步骤是必要的,以避免真菌污染在实验期间。当执行时间序列时,这一点尤其重要,因为单个时间点的污染会妨碍整个实验。很可能,根据当地的生长条件,并不总是需要双重灭菌。
- 将种子均匀地铺在 12 厘米 x 12 厘米方形培养皿(小于 0.3 mL 种子/板)上,并将其放入干燥器或其他合适的容器中。避免团状或堆形成,因为种子需要接触气体。在漂白剂和 HCl 体积下进行气体灭菌,报告为52: 100 mL 漂白剂 (13%)60 L 干燥器中具有 6 mL 的 Conc. HCl。在无菌容器中收集种子之前,请至少1小时进行脱泡。
3. 电镀
- 提前准备这些步骤。在实验所需的量中浇注 1⁄2 MS 板 (pH 5.8), 加盖为 1% agar(每个样品/时点 20-30 个)。切割 1 mL 移液器尖端,用剃刀刀片将尖端直径放大至约 3-4 mm。高压提示。创建模板支架,用于用方形培养皿盖为每个板电镀三排种子。准备层状流量罩,提供无菌工作环境,并标记要处理的板。
注:如果同时处理许多板,彩色标签可以加快标签。 - 将空的 agar 板放入模板支架中,将 1 mL 浸埋种子均匀地分布到三行上。将加工的板放入层状流中,直到种子干燥(即粘在加面上)。不要让盘子变长,因为加盘也会干涸。
- 一旦种子足够干燥,关闭盖子,用微孔胶带密封每个板。在黑暗中将种子分层两天,在4°C下,然后把它们放入生长室。
4. 组织治疗(可选)
注:在此协议中,我们概述了合成辅助基因中NAA对阿拉伯蛋白酶根的外源处理。根据手头的实验问题,需要调整或完全省略此部分。
- 准备1.5 - 2厘米高、10厘米长的纸巾条。延长的孵育时间要求组织在使用前进行解剖。
- 从所有必须接受激素治疗的板中取出微孔胶带。在 1 L 液体中稀释 1 mL 10 mM NAA(溶解在 DMSO 中),对 1⁄2 MS 溶液(pH 5.8)进行自切除,并将纸巾浸泡在溶液中 (10 μM NAA)。
- 使用钳子将一条纸巾涂在每排根上。轻轻地用手指去除气泡。从盘子中倒出多余的液体,关闭盖子,用时间标记盘子。对于延长的孵育时间,将板放回生长室。
5. 收获
- 检索每个生物复制/时间点/处理的板。收集液氮在干净的Dewar容器和标签管(15或50 mL)为不同的组织样本。准备发泡胶支架。
注意:熟悉液氮处理程序(曝气、冻伤、潜在爆炸管)。 - 打开板,用钳子取出纸巾,小心不要从加面上分离根部。使用手术刀片,在单次确定的行程中沿射根结每行切割一次。清洁样品之间的刀片并频繁更换,以确保锐度。
- 使用钳子,沿每行的根部轻扫以收集它们三个捆绑包。抓住根部,将其倒入一个装满液氮的 50 mL 管中,以卡死。
注:不要试图将根部组装成致密的结构(如球),因为它们在下一步中很难磨碎。 - 继续所有构成一个样品的板(按孵育时间顺序),并倒出多余的液氮。使用管盖防止根部溢出。然后关闭盖子,收集德瓦尔容器中的所有管。将根组织储存在-80°C。
6. 免疫净化
注:此步骤旨在获得高质量的 TRAP/多体RNA。因此,严格遵循RNA处理的良好做法建议。在无菌工作台上执行本节中的所有步骤,并使用 RNase 去除溶液(材料表)清洁所有设备和实验室软件。当样品、冰块或其他未清洁的源受到污染时,戴上手套并立即更换。由于这是一个非常重要的方面,因此包括关于设备再利用的一节以及废物处理建议。
- 缓冲准备
- 根据表 1和高压灭菌器 (A) 准备库存解决方案或过滤器消毒 (*)。除非另有说明,否则溶剂为无无Nase水。
- 如表1所示,在各自的溶剂中溶解和分值二硫磷醇(DTT)、苯乙二硫芬基氟化物(PMSF)、环己烷(CHX)和氯霉素(CAM),并将其储存在-20°C。所有其他股票可以保持在室温。
- 预混合库存 - 与成分 1-4 用于洗涤缓冲液 (WB) 和 1-6 用于多体萃取缓冲液 (PEB) ) - 以避免每次萃取前耗时的缓冲混合。因此,仅在提取当天加入水和冷冻成分(7-10)。将预混合库存和无 RNase 水保持在 4 °C。
注:DTT浓度是Zanetti等人2005年报告的浓度的1/5,因为与GFP的nann相互作用对高DTT浓度敏感。
| 成分 | 库存浓度 | 为 50 mL 的 WB* 添加 mL 的体积 | 50 mL PEB 的体积以 mL 添加* | ||
| 1 | 三重奏,pH 9 | A | 2 米 | 5 | 5 |
| 2 | 氯化钾 | A | 2 米 | 5 | 5 |
| 3 | EGTA | A | 0.5 M | 2.5 | 2.5 |
| 4 | 毫克Cl2 | A | 1 米 | 1.75 | 1.75 |
| 5 | Pte | A | 20%(v/v) | 0 | 2.5 |
| 6 | 洗涤剂混合物 | A | 0 | 2.5 | |
| 补间 20 | 20%(v/v) | ||||
| 特里顿-X 100 | 20%(v/v) | ||||
| 布里杰-35 | 20% (v/v) | ||||
| 伊格帕尔 | 20%(v/v) | ||||
| 7 | DTT | ₳ | 0.5 M | 0.1 | 0.1 |
| 8 | PMSF | ₳ | 0.1 M (异丙醇) | 0.5 | 0.5 |
| 9 | 环氧西米德 | ₳ | 25毫克/米升(埃特霍) | 0.1 | 0.1 |
| 10 | 氯霉素 | ₳ | 50毫克/米升(EtOH) | 0.05 | 0.05 |
表1:缓冲组成和混合建议。给定库存浓度混合在给定量中的成分可产生 50 mL 的 WB 或 PEB。三酯:三酯-(羟基甲基)-氨基甲烷,EGTA:乙二醇-乙二醇(β-氨基乙醚)-N、N、N'-四乙酸、PTE:聚乙烯-(10)-三丁基乙醚,A:高压灭菌器,*:过滤器-灭菌;•使用无RNase水填充高达50 mL。
- 组织均质化/研磨
- 冷却离心机,将同质器和离心管放在冰上。解冻 DTT、PMSF、CHX 和 CAM 的等号。根据当天的要求(样品*)将50 mL管中的库存溶液中的PEB和WB混合,并在冰上冷却。
注:仅在使用前不久添加 PMSF,因为 PMSF 在水中的半衰程仅为 30 分钟。 - 在 Dewar 容器中准备大量液氮,并从 -80 °C 储存中检索组织样本。在标准实验室手套下方佩戴棉手套,以防止冷砂浆灼伤。将液氮倒入砂浆和虫子中,直到其足够冷,以便进行研磨。建议设计一个系统来区分迫击炮(标签或保持一定顺序)。
- 空组织样品进入砂浆并仔细研磨,直到所有材料都是白色粉末。如果需要,加入液氮以保持组织冻结或促进更好的研磨。
- 在样品中加入 5 mL PEB,并在缓冲液冻结前快速与粉末混合。当此示例解冻(不时混合)处理另一个样本时。
- 一旦混合物可以转移,将浆料倒入玻璃均质器中,并保持在冰上。再加2 mL PEB,冲洗砂浆并缠住,并将其添加到均质剂中的样品中。
注:避免完全液体样品,因为这允许RNA降解。 - 手动研磨浆料,直到提取物均匀。我们建议至少 4 到 5 次跳水。
注:可能需要一些额外的等待时间,以便泥浆进一步解冻。处理均质器需要一些勤奋。不要使用蛮力和小心吸力。如果不加考虑,这将导致均匀器的溢出、污染或破坏。 - 将粗根提取物倒入 50 mL 离心管中(保持冰)。
注:通常几个样品可以在转移前接地。需要平行处理磨削、转移和均质化。尽量快点工作,但不要匆忙;保持冷静。始终将均质样品保存在冰上。
- 冷却离心机,将同质器和离心管放在冰上。解冻 DTT、PMSF、CHX 和 CAM 的等号。根据当天的要求(样品*)将50 mL管中的库存溶液中的PEB和WB混合,并在冰上冷却。
- RNA 样本收集总量
- 将每个粗样品的 200 μL 等分转移到干净的微离心管(事先在冰上标记和冷却)。
- 继续RNA提取,如第7.1点和7.2点中TRAP样品的详细详细内容。当样品在离心机中清除时,执行这些步骤。
- 使用重新悬浮的总RNA进行DNase处理,以消除DNA污染,并使用商业试剂盒(材料表)清理反应。
注:总RNA萃取通常产生高浓度,样品需要大大稀释。我们建议通过敏感的Qubit协议测量稀释后的浓度。
- 清除原油提取物
- 将装有 6.2.7 样品的冰桶放在 16,000 x g和 4 °C 下,将其离心 15 分钟。
注:要平衡离心机,应相应地对样品进行配对。如果不能完全可能,请通过添加 PEB 调整一个样本。 - 将上清液倒入新鲜的离心管(事先在冰上冷却)和重复离心(15 分钟,16,000 x g和 4 °C)。此转移可在离心机旁边快速执行。
- 当原油萃取物清除时,开始清洗步骤 6.6 的 GFP-珠。
注:保留这个冰桶在摇床上摇动,但不要放回无菌的长凳上,因为它可能被污染。
- 将装有 6.2.7 样品的冰桶放在 16,000 x g和 4 °C 下,将其离心 15 分钟。
- 珠洗
- 阿利quot磁性 GFP-珠(#samples x 60 μL,材料表)成 1.5 mL 管。放在磁性支架上。收集珠子后,取出上清液。
- 加入 1 mL 的冷 WB,重新悬挂珠子并再次收集它们。丢弃洗涤缓冲液,用 1 mL 的 WB 重复一次。
- 最终,将 WB 中的珠子重新挂起到步骤 6.5.1 中使用的初始体积。
- 免疫纯化(IP)
- 离心后,立即将清除的上清液倒入标有15 mL的管中,并每样品加入60μL的洗珠。
- 将所有样品水平放入冰桶中,并将其放在摇床上。让混合物孵育2小时,以便将 GFP 标记的多体与珠子结合。
- 收集磁架上的珠子,用于 15 mL 管(冰上),并将 PMSF 添加到剩余的 PEB 中。丢弃上清剂。将大约 5 mL 的 PEB 倒入珠子,然后倾斜重新悬挂它们。与第 6.6.2 节中相同的设置中摇动样品 15 分钟。
- 使用 WB 重复洗场,总共 3 次洗(1 x PEB,2 个 WB)。在每次缓冲区交换之前,添加 PMSF。
- 将珠子收集在 1 mL 的 WB 中,并将其转移到 1.5 mL 管中。最后,在磁架上再收集一次珠子,并去除所有液体。关闭管子并保持冰,直到处理所有样品。
- 将样品运送到烟雾罩进行RNA提取。
- 废物处理和实验室用品的修复。
- 如果按照良好的实验室实践执行(参见第 2.2.1 节),灭菌程序会产生水性 NaCl 溶液。将氯气以及残留的 HCl 和漂白剂留在烟雾罩中。
- PEB 和 WB 处置:当 CHX 在高 pH 时分解,收集所有液体并带到 pH>9。处理卤化化学废物中的液体废物。所有固体(组织、血清学移液器、手套等)应作为化学废物处理。
- 分别收集含苯酚的液体以及酚类污染物质(提示、管和手套)。
- 用肥皂洗手砂浆、虫子和均质器(海绵和刷子),彻底冲洗。随后,在 >220 °C 下烘烤材料过夜。在处理前用锡箔包裹,或放入防热、覆盖的容器中。
- 用洗涤剂刷干净离心管,然后在烟罩中处理二乙丙酸酯(DEPC)。为此,将液体 DEPC 添加到去离子水中(1 mL DEPC 到 1 L 的 H2O),并通过摇动混合。将离心机管放在可分离的托盘上,以捕获溢出的 DEPC 水。将悬架倒入管中,离开 3 小时或过夜。DEPC 在后续的高压灭菌过程中分解。
注意:DEPC 是剧毒的。
7. RNA 提取和质量控制
- RNA 提取
- 将桌面离心机冷却至 4 °C。
- 在每个样品中加入1mL的酸碱-酚基试剂(材料表),倒置以重新悬浮珠子或总RNA浆,并在冰上孵育5分钟。不要旋涡!
- 加入200μL的氯仿,在冰上孵育3分钟。然后彻底涡旋样品。
- 为了帮助相分离,最大离心机。速度为10-15分钟,4°C。
- 标签 1.5 mL 低保留管 (材料表) 和等分 650 μL 异丙醇到每个。
- 小心地取上水相(约650 μL),并转移到制备的管与异丙醇。避免接触粉红色有机相。
- 在-20°C下一夜之间沉淀RNA。
注:建议将样品储存在异丙醇中,温度为-20°C或-80°C,并在需要时仅在水中溶解。水性RNA即使在-80°C下,在储存数周/数月时也会降解。
- RNA沉淀
- 将桌面离心机冷却至 4 °C。
- 使用无RNase水制备新鲜的 80% EtOH,并在 -20°C 下冷却(在 -80 °C 下 5 分钟有助于加快工艺速度)。
- 以最大速度离心样品(约13,000 x gg),30分钟,并丢弃上清液。颗粒将不可见,如此仔细移液,就好像它在那里。加入 1 mL 的冷 80% EtOH 并反转管一两次。
- 以最大速度再次离心30分钟,并重复洗涤,共洗涤两次。
- 向下旋转 2 分钟,用 10 μL 尖端去除所有残留的 EtOH。在室温下,将颗粒干燥 3-5 分钟(不要更多),并在 20 μL 无 Nase 水中重新悬浮。
- 将样品放在冰上,并尽快进行质量控制。继续将样品储存在-80°C。避免冻结-解冻循环。
- 根据制造商的建议,使用专用设备(材料表)进行质量控制。
8. 图书馆准备
- cDNA合成和放大与SMARTer v4超低输入RNA套件
- 计算每个样品的稀释,以4.75μL的体积计算1.5纳克的TRAP-RNA或总RNA。 在PCR管中执行所有反应,用无RNase水的新鲜等值点稀释样品。
- 根据制造商的建议,使用 1/2 反应体积执行所有步骤。使用 12-13 PCR 周期放大 cDNA。
- 通过添加 0.5 μL 的 10x 集塞缓冲液和 25 μL 的 SPRI 磁珠(材料表),清理 PCR。如果处理许多样品的液化缓冲液和珠子可以预混合。确保珠子在移液前均匀分散。
- 以全反应体积(17 μL的洗脱缓冲液)继续处理协议。不要让珠子干燥超过3分钟。过度干燥的样品可以通过延长的潜伏时间来挽救。
- 使用Qubit HS DNA试剂盒测量样品浓度。
注: SMARTer v4 套件可以容忍到 200 pg 输入。在无法确定库比特值(低于 250 pg,检测限制)的情况下,我们确实获得了库,该值具有 16 个周期 PCR。但是,有限的输入材料也可能产生不太复杂的库。
- 使用 Nextera XT DNA 库制备套件进行碎片和适配器连接 PCR
- 用无RNase水稀释cDNA,获得PCR管中的浓度为200皮克/μl,移液器浓度为1.25μL。
- 根据制造商执行所有步骤,其反应量为 1/4。使用 12 个 PCR 周期和兼容适配器放大 cDNA,用于属于一个测序池的样本。使用 Illumina 的索引套件 A 和 D,可以多路复用多达 384 个样本。
- 对于 PCR 清理添加 12.5 μL 的重新悬浮缓冲液和 22.5 μL 的 SPRI 磁珠(0.9 倍比)。用22μL的洗脱缓冲液对样品进行脱脂。
注:QC和池由测序公司(材料表)执行,因此不需要基于珠子的规范化。酶分片反应(标记)对材料输入非常敏感,因为每个酶只切割一次。因此,不要超过浓度建议。
Access restricted. Please log in or start a trial to view this content.
结果
在质量评估方面,应探讨上述过程的几个中间步骤:植物中的表达模式验证、分离的多体层RNA的质量控制以及最终库。此外,还可以使用已知标记基因进行qRT-PCR,以确认对治疗条件的反应或微调实验条件。
GFP信号分布的共聚焦分析
为了检查内皮和XPP表达模式,我们通过共聚焦显微镜分析了pELTP:::GFP-RPL18和<...
Access restricted. Please log in or start a trial to view this content.
讨论
验证 RPL18 本地化模式
避免任何 TRAP 实验的数据被误解的关键,是标记的核糖体子单位的正确表达模式。因此,将 GFP 作为表位标记与 RPL18 的上部标签非常优雅地允许验证所需的表达模式,并连续从同一组织中下拉多体分数。更多侵入性的方法,以确保适当的促进者模式遵循焦和梅耶罗维茨2010年,这需要GUS染色和在Tian等人2019年依靠免疫染色与抗FLAG抗体58,59。
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
我们要感谢苏黎世遗传多样性中心的让-克洛德·沃尔瑟在该项目的早期阶段提供重要的专家建议。Vermeer 实验室的工作得到了瑞士国家科学基金会 (SNSF) 授予 JEMV 的 SNF 教授授予 (PP00P3_157524) 和 R'EQUIP 设备赠款 (316030_164086) 的支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120x120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only - SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC | |
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT | |
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA | |
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG | |
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
参考文献
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673(2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. Kaufmann, K., Mueller-Roeber, B. , Springer. New York, NY. 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. Nielsen, H. , Humana Press. Totowa, NJ. 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276(2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436(2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956(2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354(2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443(2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71(1993).
- Ristova, D., Barbez, E. Root Development. , Springer. New York, NY. (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587(2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3(2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985(2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419(2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141(2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366(2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696(2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801(2007).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。