Method Article
قكات: "كتابة سيميوتوماتيد الكمية" من القاتل-الخلية "جينات مستقبلات" الغلوبولين المناعي مثل
In This Article
Summary
الكمية القاتل خلية الغلوبولين المناعي مثل مستقبلات (كيريباتي) كتابة شبه الآلي (قكات) أسلوب بسيط والفائق، وفعالة من حيث التكلفة لنسخ رقم نوع قير الجينات لتطبيقها في الرابطة للدراسات السكانية والمرض.
Abstract
خلية القاتل الغلوبولين المناعي مثل مستقبلات (كيرس) مجموعة من المستقبلات المناعية المثبطة وتفعيل، القاتل الطبيعية (ناغورني كاراباخ) وخلايا T، مرمزة بواسطة كتلة متعددة الأشكال من الجينات على الكروموسوم 19. على يغاندس أفضل تتسم هي جزيئات مستضد (هلا) الكريات البيض البشرية التي تم ترميزها ضمن محور histocompatibility الرئيسية المعقدة (MHC) على الكروموسوم 6. وهناك أدلة كبيرة على أنها تلعب دوراً هاما في الحصانة والاستنساخ وزرع الأعضاء، مما يجعل من أهمية حاسمة لأن التقنيات التي يمكن بدقة التركيب الوراثي لهم. بيد عالية-تسلسل التماثل، كذلك الاليلي وتباين عدد النسخ، تجعل من الصعب على تصميم الأساليب التي يمكن بدقة وكفاءة النمط الوراثي جميع قير الجينات. الأساليب التقليدية عادة ما تكون محدودة في دقة البيانات المتحصل عليها والإنتاجية والفعالية من حيث التكلفة والوقت اللازم لإعداد وتشغيل التجارب. يصف لنا طريقة تسمى الكمية قير شبه الآلي كتابة (قكات)، وهو أسلوب سلسلة من ردود فعل الفائق متعدد بوليميراز في الوقت الحقيقي التي يمكن تحديد أرقام نسخ الجينات لجميع الجينات في موضع كيريباتي . قكات هو أسلوب الفائق بسيطة التي يمكن أن توفر عالية الاستبانة قير نسخ عدد البيانات، التي يمكن أن تستخدم كذلك لاستنتاج الاختلافات في haplotypes هيكلياً متعددة الأشكال التي تشتمل عليها. يمكن أن يكون هذا العدد و haplotype نسخ البيانات مفيداً للدراسات المتعلقة بالجمعيات المرض على نطاق واسع، وعلم الوراثة السكانية، فضلا عن التحقيقات في التعبير والتفاعلات الوظيفية بين قير و هلا.
Introduction
في البشر، ومستقبلات الغلوبولين المناعي مثل القاتل(قير) يتم تعيين موضع على الذراع الطويلة كروموسوم 19 داخل مجمع مستقبلات الكريات البيض (LRC). هذا المكان هو حوالي 150 كيلو بايت في طول ويتضمن 15 قير الجينات مرتبة رئيس-إلى-الذيل. المكاني قير المعروفة حاليا هي KIR2DL1، KIR2DL2/KIR2DL3، KIR2DL4، KIR2DL5A، KIR2DL5B، KIR2DS1-5، KIR3DL1/KIR3DS1، KIR3DL2-3، وهما بسيودوجينيس، KIR2DP1 و KIR3DP1. ترميز الجينات قير لثنائي الأبعاد (2D) ومستقبلات المجال مثل الغلوبولين المناعي (3D) ثلاثي الأبعاد مع القصيرة (S؛ وتفعيل) أو طويلة (L؛ والمثبطة) ذيول هيولى، التي يعرب عنها الخلايا القاتلة الطبيعية (ناغورني كاراباخ) ومجموعات فرعية من T الخلايا. تباين عدد نسخ معروضة ضمن أشكال محور قير haplotypes المتنوعة مع الجينات متغيرة المحتوى1. عدم الاليلي جزئ مثلى (نهر)، يسر بترتيب إغلاق الرأس إلى الذيل الجينات وتسلسل عالية التماثل، هو الآلية المقترحة لتكون مسؤولة عن تقلب هابلوتيبيك. وأبلغ ما يزيد على 100 من haplotypes مختلفة في السكان في جميع أنحاء العالم1،2،،من34. جميع هذه haplotypes يمكن أن تقسم إلى فئتين رئيسيتين: haplotypes A و B. هابلوتيبي A يحتوي على جينات قير 7: KIR3DL3، KIR2DL1، KIR2DL3، KIR2DL4، KIR3DL1، و KIR3DL2، هما المثبطة قير الجينات، و تفعيل قير جين KIR2DS4. ومع ذلك، حتى إلى 70% من الأصل الأوروبي الأفراد الذين متماثل قير haplotype A حصرا تحمل شكلاً غير وظيفية 'حذف'5، KIR2DS46. جميع مجموعات الجينات قير أخرى تشكل haplotypes المجموعة باء، بما في ذلك واحد على الأقل من جينات محددة قير KIR2DS1، KIR2DS2، KIR2DS3، KIR2DS5، KIR3DS1، KIR2DL2، و KIR2DL5، وعادة ما تشمل اثنين أو أكثر من الجينات قير تفعيل.
هلا الفئة الأولى جزيئات قد حددت يغاندس لبعض المستقبلات المثبطة (KIR2DL1، KIR2DL2، KIR2DL3، و KIR3DL1)، تفعيل مستقبلات (KIR2DS1، KIR2DS2، KIR2DS4، KIR2DS5، و KIR3DS1)، و KIR2DL4، وهو فريد كيريباتي التي تحتوي على ذيول طويلة هيولى مثل مستقبلات قير المثبطة الأخرى ولكن له أيضا بقايا إيجابيا يحمل قرب المجال خارج الخلية التي مشترك ميزة أخرى تفعيل مستقبلات كيريباتي . مزيج المتغيرات داخل كيريباتي الجينات والجينات هلا يؤثر على مستقبلات يجند تفاعل تلك الأشكال المحتملة ناغورني كاراباخ خلية الاستجابة على المستوى الفردي7،8. وأوضحت الأدلة المستمدة من الدراسات الوراثية الرابطة أن كيريباتي يلعب دوراً في المقاومة الفيروسية (.على سبيل المثال،9 من فيروس نقص المناعة البشرية (الإيدز) والتهاب الكبد الوبائي فيروس [سي]10)، نجاح زرع 11، خطر الإصابة باضطرابات الحمل والتناسل بنجاح12،13، الحماية ضد الانتكاس بعد متمكنة من الخلايا الجذعية المكونة للدم (هسكت) زرع14،15، 16، وخطر السرطان17.
بمزيج من التماثل عالية-تسلسل والاليلي والتحديات يعرض التنوع هابلوتيبيك في مهمة التنميط قير الجينات بدقة. وتشمل الأساليب التقليدية لكتابة قير الجينات الخاصة بتسلسل التمهيدي (SSP) بوليميريز سلسلة من ردود الفعل (PCR)18،،من1920، اليغنوكليوتيد الخاصة بتسلسل التحقيق بكر (SSOP)21، و مصفوفة الليزر ساعد الامتزاز التأين-وقت الرحلة الكتلي (TOF استخدام MS)22. المآخذ على هذه التقنيات أنها توفر فقط جزئية من التبصر في النمط الوراثي للفرد في حين يجري أيضا شاقة لأداء. مؤخرا طبق تسلسل الجيل القادم (خ ع) اكتب موضع كيريباتي على وجه التحديد. بينما يكون هذا الأسلوب قوية جداً، فإنه يمكن أن تكون مكلفة لتشغيل، وأنها تستغرق وقتاً طويلاً لإجراء تدقيق البيانات والتحليل المتعمق.
قكات أسلوب PCR كمي الفائق. بينما الأساليب التقليدية شاقة وتستغرق وقتاً طويلاً، هذا الأسلوب يجعل من الممكن لتشغيل ما يقرب من 000 1 عينة الحمض النووي (جدنا) الجينوم في خمسة أيام ويعطي النمط الوراثي كيريباتي ، فضلا عن عدد نسخ الجينات. قكات يتكون من عشرة ردود فعل متعدد، يستهدف كل منها اثنين قير المكاني ونسخ الجينات مرجع واحد عدد نسخة ثابتة في الجينوم (STAT6) تستخدم للتحديد الكمي النسبي للجينات قير رقم23. هذا التحليل قد استخدمت بنجاح في الدراسات التي تنطوي على لوحات كبيرة من السكان والأفواج المرض عن الأمراض المعدية مثل فيروس التهاب الكبد جيم، شروط المناعة الذاتية مثل داء السكري من النوع 1، واضطرابات الحمل مثل بريكلامبسيا، فضلا عن توفير الوراثية التي تدعم بدراسات تهدف إلى فهم الخلية ناغورني كاراباخ وظيفة4،1،24،،من2526.
Protocol
1-إعداد وطلاء من الحمض النووي
- دقة قياس تركيز جدنا باستخدام أداة سبيكتروفوتوميتريك أو فلوروميتريك.
- تمييع الحمض النووي إلى 4 نانوغرام/ميليلتر في لوحة 96 بئر عميق. وتشمل مراقبة جدنا عينة واحدة على الأقل مع عدد نسخ معروفة وعنصر تحكم غير قالب واحد.
- الطرد المركزي لوحات 96-جيدا في 450 س ز 2 دقيقة.
- باستخدام أداة التعامل مع سائل والاستغناء عن كل عينة في البيلوروسية على لوحات qPCR 384-جيدا حيث يكون لكل بئر 10 نانوغرام من الحمض النووي (2.5 ميليلتر في البئر). إعداد لوحات 384-جيدا على الأقل عشرة، واحدة لكل رد فعل قكات.
- إذا كان هو يجري الاستغناء عن جدنا من أكثر من 96-جيدا لوحة، أداء يغسل وحدة التخزين بالكامل مع التبييض 2% والماء عالي النقاوة لتنظيف إبر السائل التعامل مع النظام بين كل لوحة 96-جيدا من عينات جدنا.
- أيردري الحمض النووي التي تفرخ لوحات 384-جيدا في منطقة نظيف في درجة حرارة الغرفة لمالا يقل عن 24 ساعة.
2-إعداد أجهزة الإشعال والمسابير
ملاحظة: قكات يتكون من عشرة ردود الفعل المتعددة. ويتضمن رد فعل كل ثلاثة أزواج التمهيدي وثلاثة تحقيقات المسمى fluorescence تضخيم على وجه التحديد هما كيريباتي الجينات والجينات مرجعاً واحداً. تم تعديل المسابير التي تم نشرها في جيانغ et al.27 حيث الآن المسماة النوكليوتيد مع الأصباغ أتو نظراً لأنها توفر فوتوستابيليتي محسنة وعمر طويل إشارة. تركيبات التمهيدي قبل الكوتيد متوفرة تجارياً (انظر الجدول للمواد).
- إعداد مجموعات التمهيدي لكل رد فعل وفقا لتخفيف الوارد في الجدول 1.
- إعداد مجموعات التحقيق لكل رد فعل وفقا الجدول 1. اختبار كل مسبار الفردية قبل صنع التركيبة.
3-إعداد مزيج ماجستير
ملاحظة وحدات التخزين المذكورة أدناه لأداء واحد قكات الرد على مجموعة من لوحات 10 × 384-جيدا.
- ضمان أن العينات جدنا مطلي على لوحات 384-بئر جافة تماما. إجراء جميع خطوات على الجليد وإبقاء الكواشف تغطيتها من التعرض للضوء بقدر الإمكان نظراً لتحقيقات المسمى fluorescence الحساسة صور والحرارية.
- تذويب qPCR المخزن المؤقت، والتمهيدي، ومختبرين التحقيق في 4 درجات مئوية.
- على الجليد، إعداد مزيج رئيسي للوحات 10 × 384-جيدا بإضافة 18.86 مل الماء عالي النقاوة، 20 مل من المخزن المؤقت قبكر و 1,000 ميليلتر من تركيبة التمهيدي بريبريباريد ميليلتر 180 من تركيبة مسبار بريبريباريد (الجدول 2).
- توزيع مزيج الرئيسي بالتساوي عبر صفيحة جيدا عمق 96 استخدام ماصة متعددة القنوات، بيبيتينج ميليلتر 415 في كل بئر. إبقاء هذه اللوحة في مربع جليد تغطي من الضوء.
- باستخدام أداة التعامل مع سائل، الاستغناء عن 9.5 ميليلتر من مزيج الرئيسي في كل من لوحة 384-جيدا مع جدنا المجفف جيدا. ختم اللوحة بإحباط ثم ضعه مباشرة في 4 درجات مئوية. كرر هذه العملية من أجل لوحات المتبقية، وضمان أن إبر السائل التعامل مع نظام يتم غسلها بالماء بين كل لوحة.
- الطرد المركزي لوحات 384-جيدا في ز 450 x لمدة 3 دقائق واحتضانها لهم عند 4 درجة مئوية بين عشية وضحاها، أو بين 6-12 ح ريسوسبيند في الحمض النووي، وتبديد أي فقاعات الهواء.
4-قبكر الإنزيم
- وعقب الحضانة بين عشية وضحاها، الطرد المركزي في 450 x ز لمدة 3 دقيقة لتبديد أي فقاعات الهواء المتبقية.
- لأغراض التشغيل الآلي للمكاتب، ربط الجهاز قبكر (مثلاً، لايتسيكلير 480) إلى معالج ميكروسكوبية (انظر الجدول للمواد). برنامج معالج الميكروسكوبية لوضع اللوحات في الجهاز قبكر من رصيف تبريد تخزين محمياً من الضوء.
ملاحظة فحوصات ينبغي، من الناحية النظرية، العمل على الأجهزة الأخرى qPCR مع إعدادات الألياف الضوئية المتوافقة. - استخدام الدراجات الشروط التالية: 95 درجة مئوية لمدة 5 دقائق تليها دورات 40 من 95 درجة مئوية عن 15 ثانية و 66 درجة مئوية لمدة 50 ثانية، مع جمع البيانات في 66 ° C.
- بمجرد اكتمال التشغيل، يكون الروبوت جمع اللوحة من آلة قبكر ووضعه في قفص الاتهام المرتجع.
5-بعد انتهاء تشغيل تحليل
- بعد التضخيم، حساب القيم دورة (Cq) القياس الكمي باستخدام الأسلوب الثاني أقصى مشتقة أو أسلوب النقاط تناسب مع البرنامج الخاص بالجهاز قبكر (انظر الجدول للمواد)، اتباع الخطوات التالية.
- فتح برنامج قبكر، وفي علامة التبويب المستكشف ، فتح ملف تجربة رد فعل المحفوظة للوحة واحدة.
-
لتحليل استخدام أسلوب الحد الأقصى المشتقة الثانية، حدد علامة التبويب تحليل، وإنشاء تحليل جديد لاستخدام أسلوب "ماكس مشتق Quant/Second إس".
- في إطار تحليل جديد لإنشاء ، حدد نوع التحليل: أسلوب Abs Quant/Second مشتق ماكس، مجموعة فرعية: جميع عينات، البرنامج: التضخيم، واسم: Rx-DFO (حيث x هو رقم رد الفعل).
- حدد عامل تصفية مشط واختر فيك/عرافة/Yellow555 (533-580). وهذا ما يضمن أن يتم تحديد البيانات التي يتم جمعها ل STAT6 .
- حدد تعويض اللون ل VIC/HEX/Yellow555(533-580). انقر فوق حساب. كرر هذه العملية للاتحاد الماليزي (465-510) و Cy5/Cy5.5(618-660). انقر فوق حفظ ملف.
-
للتحليل استخدام أسلوب النقاط تناسب، حدد "نقاط" ضليع في الرياضيات/تناسب القيمة المطلقة في علامة التبويب تحليل.
- في إطار تحليل جديد لإنشاء ، حدد نوع التحليل: أسلوب "النقاط" ضليع في الرياضيات/تناسب القيمة المطلقة، مجموعة فرعية: جميع العينات، والبرنامج: التضخيم، والاسم: DFO ركسف (حيث x هو رقم رد الفعل).
- حدد عوامل التصفية الصحيحة وتعويضات اللون ل STAT6 وكل من الجينات كيريباتي (الاتحاد الماليزي/Cy5). في علامة التبويب نويسيباند ، تعيين الفرقة الضوضاء استبعاد ضجيج الخلفية.
- في علامة التبويب تحليل ، تعيين النقاط التي تناسب إلى 3 وحدد إظهار تناسب النقاط. انقر فوق حساب. انقر فوق حفظ ملف.
6-تصدير النتائج
- في برنامج قبكر، فتح المستكشف تحديد التبويب تصدير مجموعة النتائج.
- افتح المجلد الذي يتم حفظ ملفات التجربة ونقل الملفات في الجزء الجانب الأيمن من الإطار. انقر فوق التالي. حدد الاسم وموقع ملف التصدير.
- حدد نوع تحليل أسلوب "ماكس مشتق Quant/Second إس" أو إس ضليع في الرياضيات/تناسب النقاط. انقر فوق التالي. تحقق من أن اسم الملف والمجلد التصدير، ونوع التحليل صحيحان ثم انقر فوق التالي لبدء عملية التصدير.
- انتظر حتى حالة تصدير على ما يرام. الشاشة سوف ينتقل تلقائياً إلى الخطوة التالية. تحقق من أن كافة الملفات المحددة قد تم تصديرها بنجاح حيث أن فشل عدد الملفات = 0. انقر فوق تم.
- استخدام البرامج النصية split_file.pl و roche2sds.pl لتقسيم لوحات المصدرة إلى ردود الفعل الفردية لكل لوحة.
ملاحظة يتم توفير البرامج النصية على طلب/GitHub.
7-نسخ عدد العمليات الحسابية
- فتح برنامج تحليل عدد النسخ (مثلاً، كوبيكالير). حدد استيراد ملف نتائج PCR الوقت الحقيقي وتحميل الملفات النصية التي تم إنشاؤها بواسطة roche2sds.pl.
- حدد تحليل وإجراء التحليل بتحديد أما معايرة العينة مع عدد النسخ المعروفة أو بواسطة تحديد عدد النسخ الأكثر شيوعاً. انظر الجدول 5 لعدد الجينات قير عادة ما يلاحظ في السكان الأصل الأوروبي النسخة الأكثر شيوعاً.
8-يتحقق من نوعية البيانات
- استخدام برنامج نصي للبحث والتطوير KIR_CNVdata_analysis_for_Excel_ver020215. ص لضم البيانات عدد نسخ من جميع لوحات في جدول بيانات.
ملاحظة يتم توفير البرامج النصية على طلب/GitHub. - تدقيق البيانات الخام في برنامج تحليل عدد نسخ للعينات التي لا تتوافق مع اختلال الصلة المعروفة (LD) للجينات كيريباتي (الجدول 6).
النتائج
ويمكن إجراء تحليل رقم نسخة عن طريق تصدير الملفات إلى برنامج تحليل عدد نسخة، التي تنص على عدد النسخ المتوقعة والمقدرة استناداً إلى أسلوب ΔΔCq.
يمكن التنبؤ بعدد نسخ أما استناداً إلى عدد النسخ المعروفة لعينات من الحمض النووي التحكم على اللوحة، أو عن طريق إدخال عدد نسخ الجينات الأكثر شيوعاً (الجدول 5). ويبين الشكل 1 نتائج لوحة لرد فعل يستهدف KIR2DL4 و KIR3DS1، فضلا عن الجين مرجع STAT6. رقم KIR2DL4، جين إطار في موضع كيريباتي ، النسخة الأكثر شيوعاً نسختين، بينما رقم KIR3DS1، تفعيل جينات، النسخة الأكثر شيوعاً نسخة واحدة. وتظهر النتائج في الشكل المؤامرات التضخيم بكر يحتفل به في البرنامج قبكر ونسخة عدد البيانات التي تم إنشاؤها من البيانات qPCR. كما هو موضح، التحليل قادراً على التمييز بين 0، 1، 2، 3، والجينات قير 4 نسخ الأرقام. كما يتيح البرنامج تحليل عدد نسخ عرض توزيع عدد نسخ عبر اللوحة كمخطط دائري أو رسم بياني شريطي. الفعالية للتنبؤ بعدد نسخ أقل بالنسبة للعينات مع ارتفاع عدد نسخ.
جودة جميع المواد المستخدمة في ردود الفعل، جدنا، المخزن المؤقت، والإشعال، والمجسات، يمكن أن تؤثر على دقة النتائج التي تم الحصول عليها. الخلافات في النتائج غير الأكثر احتمالاً أن تكون بسبب التباين في تركيز الحمض النووي عبر صفيحة. نقاء جدنا المستخرجة، التي يمكن أن تقاس باستخدام 260/280 و 260/نسب 230، يمكن أيضا أن يكون لها تأثير على النوعية. ومن المستصوب بنسبة 260/280 نسبة 1.8-2 و 260/230 2-2-2. يمكن أن يؤدي تركيزات مجموعة متفاوتة من الحمض النووي عبر لوحة تقلب عالية في دورة عتبة (جر) بين العينات والخلافات في نطاق عدد نسخ المقدرة. وتظهر النتائج في الشكل 2 التأثير يمكن أن يكون التباين بين قيمt C عبر صفيحة على الدقة في التنبؤ بعدد النسخ. الخط الأحمر يشير إلى نطاق عدد نسخ المقدرة عينة، ومن الناحية المثالية، ينبغي أن يكون أقرب عدد صحيح قدر الإمكان.
يمكن تصدير البيانات عدد نسخة، وحللت مرة واحدة، كملف جدول بيانات في تنسيق 96-جيدا. قمنا باستخدام برنامج نصي R (متوفرة عند الطلب) الجمع بين البيانات رقم نسخة من كل 10 من الألواح التي يتم تشغيلها كمجموعة في جدول واحد. البيانات المنشورة حول كيرس من السكان الأصل الأوروبي معظمها يتيح التنبؤ بالقواعد LD موجودة بين الجينات المختلفة في مجمع قير 1. وتستخدم هذه التنبؤات لإجراء فحوص المصب في نسخ عدد النتائج المتحصل عليها (الجدول 6). قد تحتوي على العينات التي لا تتوافق مع LD المتوقع بين الجينات تعدد الأشكال غير عادية أو الاختلافات الهيكلية هابلوتيبيك. مخطط انسيابي تصف البروتوكول يرد في الشكل 3.
تم تطوير أداة تسمى قير Haplotype المعرف (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) لتيسير الإسناد haplotypes من مجموعة البيانات. احتساب يعمل على أساس قائمة haplotypes مرجع الملاحظة في سكان أصل الأوروبي1. ومع ذلك، الأداة تسمح أيضا لمجموعة مخصصة من haplotypes مرجع لاستخدامها بدلاً من ذلك. يتم إنشاء ثلاثة ملفات منفصلة؛ أول ملف يسرد كافة تركيبات haplotype عينة والملف الثاني يوفر قائمة المشذبة تركيبات haplotypes التي لها ترددات مجتمعة أعلى، ويسرد الملف الثالث العينات التي لا يمكن تعيين haplotypes. ويمكن استخدام عدم إحالة haplotypes كمؤشر لرواية haplotypes.
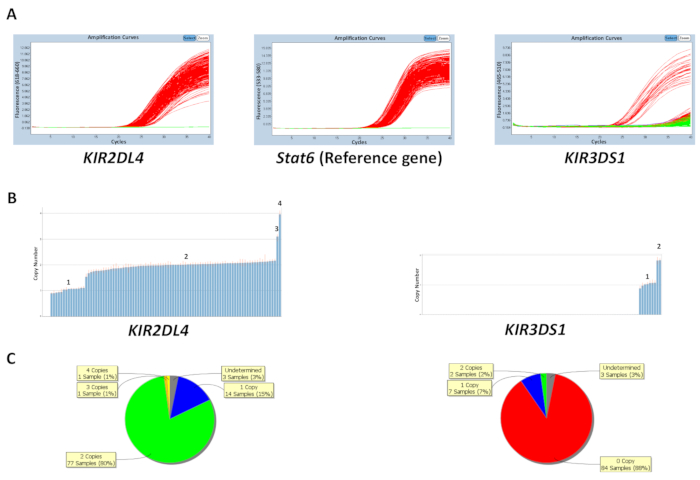
رقم 1: نتائج تمثيلية للوحة للرد رقم 5- (أ) هذا التضخيم يظهر الفريق المؤامرات. (ب) هذا الفريق يظهر نسخ عدد قطع الأراضي. (ج) هذا الفريق يوضح توزيع عدد النسخ. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
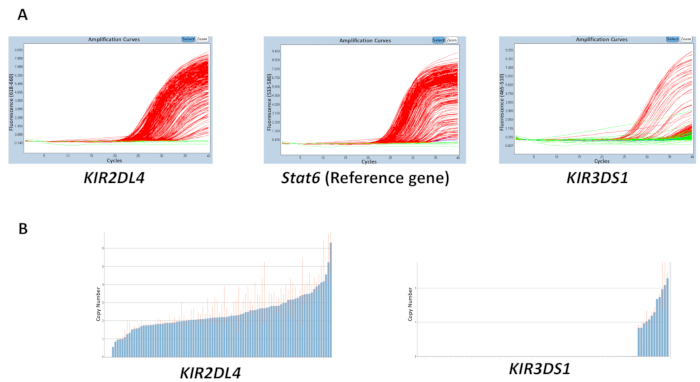
رقم 2: نتائج تمثيلية لصفيحة مع تركيز الحمض النووي متغير لرد فعل رقم 5- (أ) هذا التضخيم يظهر الفريق المؤامرات. (ب) هذا الفريق يظهر نسخ عدد قطع الأراضي. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
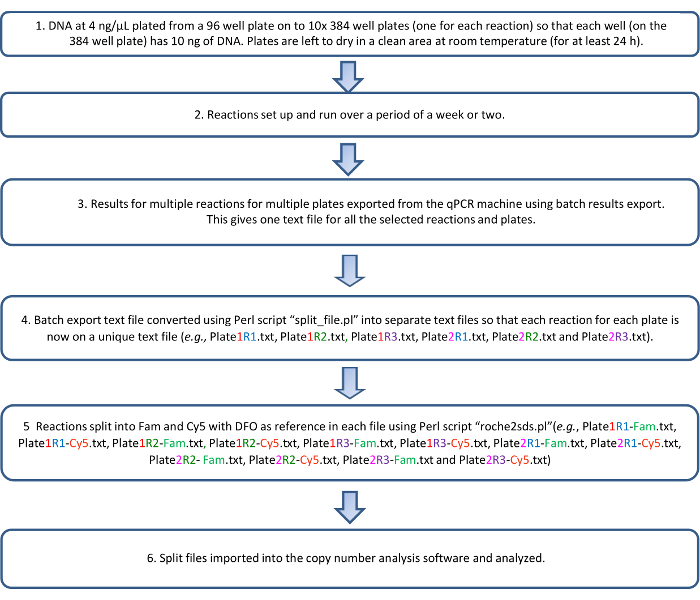
الشكل 3: مخطط انسيابي لبروتوكول قكات. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
| الفحص | الجينات | الإشعال إلى الأمام | تركيز (nM) | عكس أجهزة الإشعال | تركيز (nM) | تحقيقات | تركيز (nM) |
| 1 لا | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2 دل 2 | 2DL2F4 | 400 | C3R2 | 600 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| دل 2 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 3 لا | دل 3 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 4 | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 5 لا | 3DS1 | B2F | 250 | B1R | 250 | P4b | 150 |
| 2DL 4 | C1F | 200 | C1R | 200 | P5b-2 دل 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 6 | 2DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| دل 2 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 8 | 2DS3 | B5F | 250 | B5R | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | D5R | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| رقم 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 10 لا | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
الجدول 1: تركيبة وتركيز أجهزة الإشعال والمجسات المستخدمة في كل رد فعل قكات 27 .
| رد الفعل | مختبرين التمهيدي (ميليلتر) | التحقيق مختبرين (ميليلتر) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | المياه | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2 دل 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | المياه | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| دل 2 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| ملاحظة: تحتاج إلى 20 ميليلتر أقل المياه في MasterMix | |||||||||||
| R3 | دل 3 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | المياه | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | المياه | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | المياه | STAT6F | STAT6R | P4B | P5B-2 ل 4 | PSTAT6 |
| 2DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | المياه | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | المياه | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| دل 2 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | المياه | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | المياه | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | المياه | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
الجدول 2: وحدات التخزين (ميليلتر) من 100 ميكرومتر التمهيدي/مسبار الأسهم الحلول لجعل التمهيدي والتحقيق مختبرين تركيبة.
| الاسم | الاتجاه | تعديل 5´ | تعديل 3´ | تسلسل (5 '→3') | طول | الخرائط المواضيعية | GC % | إكسون | موقف |
| P4a | معنى | الاتحاد الماليزي | بك-1 | تكاتككتجك آتجتتغت كاجاتجتكا | 27 | 60 | 44.4 | 4 | 425-451 |
| P4b | أنتيسينسي | الاتحاد الماليزي | بك-1 | آكاجاكك جتاجكاتكت جتاجتككك T | 28 | 62 | 50 | 4 | 576-603 |
| P5b | معنى | ATTO647N | بك-2 | آكاتككا جككجاكت تككتكتج | 25 | 60 | 52 | 5 | 828-852 |
| P5b-2 دل 4 | معنى | ATTO647N | بهق-2 | آكاتككا جككجاكت تكككتكتج | 25 | 61 | 56 | 5 | 828-852 |
| P9 | معنى | ATTO647N | بك-2 | كككتكتكا جاجكككا أجاكاكك | 24 | 60 | 62.5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | بك-2 | كتجاتككت ككاتجاجكا تجكاجكت | 26 | 62 | 50 |
الجدول 3: قائمة المسابر المستخدمة في قكات 1، 27-تم تعديل الأصباغ الفلورية تستخدم في 5 ' نهاية تحقيقات اليغو P5b، P5b-2 دل 4، P9، و PSTAT6 بالأصباغ أتو.
| الجينات | كبسولة تفجير | الاتجاه | تسلسل (5´-3´) | طول | الخرائط المواضيعية | GC % | إكسون | موقف | Amplicon (bp) | وقد غاب الآليلات | ||
| 3DL2e4 | A1F | إلى الأمام | جككككتجكتجا أتكاج | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 2 * 008، * 021، * 027، * 038. | ||
| F15 | عكس | كتجكاجاكاج جكاتكا | 19 | 53 | 52.6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | إلى الأمام | جتككككتجتجا آتكاجا | 19 | 49 | 52.6 | 4 | 398-416 | 112 | لا شيء | ||
| A5R | عكس | جتجاجكجكاا جتجتكا | 18 | 52 | 55.6 | 492-509 | لا شيء | |||||
| 2DS2 | A2F | إلى الأمام | جتكجككتجتجا آتكاجا | 19 | 49 | 52.6 | 4 | 398-416 | 111 | لا شيء | ||
| A6R | عكس | تجاجتجكااج تجتككتات | 21 | 51 | 42.9 | 488-508 | لا شيء | |||||
| دل 3 3 | A8Fa | إلى الأمام | جتجااتكججا جاجاكج | 18 | 50 | 55.6 | 4 | 406-423 | 139 | لا شيء | ||
| A8Fb | إلى الأمام | جتجااتكاج أجاجاكج | 19 | 50 | 52.6 | 405-423 | 3DL 3 * 054، دل 3 3 * 00905. | |||||
| A8R | عكس | أجتجاككتجج آكككج | 18 | 51 | 61.1 | 526-543 | لا شيء | |||||
| 3DL1e4 | M1) | إلى الأمام | كاتكجتكككات جاتجكت | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DL 1 * 00505، دل 3 1 * 006، 3DL 1 * 054، دل 3 1 * 086، دل 3 1 * 089 | ||
| B1R | عكس | ججاجكتجاكا كتجاتاج | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | إلى الأمام | كاتكجتككات جاتجكج | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DS1 * 047؛ وقد التقط دل 3 1 * 054. | ||
| B1R | عكس | ججاجكتجاكا كتجاتاج | 20 | 52 | 55 | 614-633 | لا شيء | |||||
| 2DL 1 | B3F | إلى الأمام | تكتككاتكاجت كجكاتجاك | 20 | 52 | 50 | 4 | 544-563 | 96 | 2DL 1 * 020، دل 2 1 * 028 | ||
| B3R | عكس | جتكاكتججاجك تجاكاك | 18 | 50 | 61.1 | 622-639 | 2DL 1 * 023، دل 2 1 * 029، دل 2 1 * 030 | |||||
| 2DS1 | B4F | إلى الأمام | تكتككاتكاجتك جكاتجا | 19 | 51 | 47.4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | عكس | جتكاكتججاج كتجاك | 17 | 49 | 64.7 | 624-640 | لا شيء | |||||
| 2DS3 | B5F | إلى الأمام | كتككاتكجتكج كاتجاج | 18 | 53 | 61.1 | 4 | 546-563 | 96 | لا شيء | ||
| B5R | عكس | ججتكاكتججا جكتجا | 18 | 51 | 61.1 | 624-641 | لا شيء | |||||
| 2DS5 | B6F2 | إلى الأمام | أجاجاجججاكج تتاكك | 19 | 50 | 52.6 | 4 | 475-493 | 173 | لا شيء | ||
| B6R3 | عكس | تككاجاججتكا كتججك | 18 | 53 | 66.7 | 630-647 | 2DS5 * 003 | |||||
| 2DL 4 | C1F | إلى الأمام | جكاجتجكككاجك أتكات | 18 | 52 | 55.6 | 5 | 808-825 | 83 | لا شيء | ||
| C1R | عكس | ككجاجكاتكتج تاجتكت | 19 | 52 | 52.6 | 872-890 | 2DL 4 * 018، 2 دل 4 * 019 | |||||
| 2 دل 2 | 2DL2F4 | إلى الأمام | جاجتجاجكك كاتجات | 19 | 52 | 57.9 | 5 | 778-796 | 151 | 2 دل 2 * 009؛ ز 782 تغيرت إلى ألف. | ||
| C3R2 | عكس | تكجاجتتجاكك أكتكجتات | 20 | 51 | 45 | 909-928 | لا شيء | |||||
| 2DS4 | C5F | إلى الأمام | تكككتجكاجتجك جكاجك | 17 | 57 | 70.6 | 5 | 803-819 | 120 | لا شيء | ||
| C5R | عكس | تجاككاكتكجت أججاجك | 19 | 52 | 57.9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | إلى الأمام | ككتجتككتجكا جكتككات | 19 | 54 | 57.9 | 5 | 750-768 | 203 | لا شيء | ||
| 2DS4R2 | عكس | تجاكجااكا جكاجتجا | 20 | 53 | 50 | 933-952 | لا شيء | |||||
| 2DS4FL | 2DS4FL | إلى الأمام | ككجاجكتككتا تجاكاتج | 19 | 53 | 57.9 | 5 | 744-762 | 209 | لا شيء | ||
| 2DS4R2 | عكس | تجاكجااكا جكاجتجا | 20 | 53 | 50 | 933-952 | لا شيء | |||||
| دل 2 3 | D1F | إلى الأمام | أجاكككتكاجا جتجا | 17 | 48 | 58.8 | 9 | 1180-1196 | 156 | لا شيء | ||
| D1R | عكس | كاجاجاكاكت تجاتكا | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010 ودل 2 3 * 01801 ودل 2 3 * 017 دل 2 3 * 01802 | |||||
| دل 2 5 | D2F | إلى الأمام | كاكتجكجتتتك أكاكاجاك | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 و 2DL5B * 020 | ||
| D2R | عكس | جكاجاجاكا تجاتكت | 19 | 49 | 47.4 | 1315-1333 | لا شيء | |||||
| 2DP1 | D3F | إلى الأمام | ككتكاجاجتج أكاتاكجت | 20 | 53 | 55 | 9 | 1184-1203 | 121 | لا شيء | ||
| D3R | عكس | تجاجتككج تجتاكاكت | 20 | 50 | 45 | 1285-1304 | لا شيء | |||||
| 3DL1e9 | D4F | إلى الأمام | كاكاجتجاتك أكتجكجت | 19 | 52 | 52.6 | 9 | 1203-1221 | 93 | 3DL 1 * 061، دل 3 1 * 068 | ||
| D4R2 | عكس | ككجتجتاكاجا تجتاتكتجتا | 23 | 53 | 43.5 | 1273-1295 | 3DL 1 * 05901، دل 3 1 * 05902، دل 3 1 * 060، دل 3 1 * 061، دل 3 1 * 064، دل 3 1 * 065، دل 3 1 * 094N، دل 3 1 * 098 | |||||
| 3DL2e9 | D4F | إلى الأمام | كاكاجتجاتك أكتجكجت | 19 | 52 | 52.6 | 9 | 1203-1221 | 156 | لا شيء | ||
| D5R | عكس | جاككتجاكتجتج جتجكتكج | 19 | 54 | 63.2 | 1340-1358 | لا شيء | |||||
| STAT6 | STAT6F | إلى الأمام | ككاجاتجككتاك كاتجتجك | 20 | 54 | 60 | 129 | |||||
| STAT6R | عكس | ككاتكتجكاكاج أككاكتكك | 20 | 54 | 60 | |||||||
الجدول 4: تسلسل الإشعال المستخدمة في قكات 1، 27-
| كيريباتي الجينات | دل 3 3 | 2DS2 | 2 دل 2 | دل 2 3 | 2DP1 | 2DL 1 | 3DP1 | 2DL 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | دل 2 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 المجموع | 2DS4 فلوريدا | 2DS4 ديل | 3DL 2 ex4 | 3DL 2 EX9 | |
| عدد النسخ الأكثر شيوعاً | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
الجدول 5: رقم النسخة الأكثر شيوعاً كيريباتي الجينات في عينات المنشأ الأوروبي-
| الربط اختلال قواعد قكات استناداً إلى سكان أوروبا | نسخ الاختيار رقم | |||||||
| 1 | KIR3DL3، KIR3DP1، KIR2DL4 ، و KIR3DL2 ، هي إطار الجينات موجودة على كلا haplotypes. | KIR3DL3، KIR3DP1، KIR2DL4 ، و KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 و KIR2DL2 في دينار مع بعضها البعض | 2DS2=2 دل 2 | ||||||
| 3 | KIR2DL2 و KIR2DL3 من الآليلات لنفس الجين | 2 دل 2+دل 2 3= 2 | ||||||
| 4 | KIR2DP1 و KIR2DL1 في دينار مع بعضها البعض | 2DP1=دل 2 1 | ||||||
| 5 | إكسون 4 KIR3DL1 و KIR3DL2 يتساوى إكسون 9 من KIR3DL1 و KIR3DL2 على التوالي. | 3DL1ex4=3DL1ex9 و 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 و KIR3DS1 من الآليلات | 3 DL 1+3DS1= 2 | ||||||
| 7 | LD مع KIR2DL5 KIR2DS3 و KIR2DS5 | 2DS3+2DS5=دل 2 5 | ||||||
| 8 | KIR3DS1 و KIR2DS1 بدينار | 3DS1=2DS1 | ||||||
| 9 | وجود أتال KIR2DS1 و KIR2DS4Tالتبادلية في هابلوتيبي | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL و KIR2DS4del هي المتغيرات من KIR2DS4TOTAL | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
الجدول 6: اختلال الربط بين كيريباتي يمكن استخدام الجينات في السكان الأصل الأوروبي للتحقق من عدد نسخ البيانات 1،27-
Discussion
وصفت لنا طريقة الفائق رواية شبه الآلي، تسمى قكات، مما يسهل نسخ كتابة عدد الجينات قير . الأسلوب هو تحسين عبر الطرق التقليدية مثل موفر الخدمات المشتركة-بكر، وهي منخفضة الإنتاجية ويمكن إلا أن يشير إلى وجود أو عدم وجود هذه الجينات عالية متعددة الأشكال.
تعتمد على عدة عوامل، بما في ذلك نوعية وتركيز-التوحيد عينات جدنا ونوعية المواد الكاشفة دقة البيانات عدد النسخ التي تم الحصول عليها. جودة ودقة عينات جدنا عبر صفيحة تكتسي أهمية بالغة نظراً للاختلافات في التركيز عبر اللوحة يمكن أن ينتج خطأ في حساب عدد النسخ. البيانات من الأفواج من أجزاء أخرى من العالم حيث تم التحقق من صحة فحوصات باستخدام مجموعات عينة الأصل الأوروبي، تتطلب عمليات تفتيش أكثر دقة. هذا لضمان أن حالات التسرب اليل أو الربط غير محددة التمهيدي/التحقيق لا تفسر خطأ كتباين عدد النسخ.
بينما صممت فحوصات والأمثل لتشغيل الفائق، يمكن تعديلها لتشغيل نماذج أقل. مقياس الثقة في برنامج تحليل عدد نسخ يتأثر عند تحليل العينات أقل، ولكن هذا يمكن أن تتحسن إذا تدرج عينات الحمض النووي الجينوم التحكم بعدد نسخ جينات قير معروفة على اللوحة و replicates عينة إضافية وشملت.
للمختبرات دون الروبوتات السائل/لوحة معالجة، يمكن أن يكون الاستغناء عن مزيج الرئيسي استخدام الماصات متعدد القنوات ولوحات يمكن تحميلها يدوياً في الصك qPCR.
وكان الهدف الرئيسي وراء تطوير قكات لإنشاء بسيطة والفائق والدقة والفعالية من حيث التكلفة أسلوب للنمط الوراثي كيرس للمرض الرابطة للدراسات. وقد تحقق ذلك بنجاح منذ قكات وقد استخدمت في التحقيق في دور كيريباتي في العديد من الدراسات رابطة الأمراض الكبيرة، بما في ذلك مجموعة من الأمراض المعدية، والظروف الذاتية، و اضطرابات الحمل4، 24 , 25 , 26.
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
تلقي المشروع تمويلاً من مجلس البحوث الطبية (MRC) ومجلس البحوث الأوروبي (المنسق) ضمن أفق 2020 البحث والابتكار في البرنامج الاتحاد الأوروبي في (منحة الاتفاق رقم 695551) كامبردج "المعهد الوطني للصحة" (NIH) الطبية مركز البحوث والمعاهد الوطنية للصحة أبحاث الدم وزرع وحدة البحوث (NIHR بترو) في التبرع بالأعضاء وزرعها في جامعة كامبردج وفي شراكة مع دائرة الصحة الوطنية الدم و "زرع الأعضاء" (نسبت). الآراء التي أعرب عنها هي آراء المؤلفين وليس بالضرورة من دائرة الصحة الوطنية، في NIHR، ووزارة الصحة، أو نسبت.
Materials
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
References
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved