Method Article
qKAT: quantitativa coeficiente digitação de assassino-célula Genes de receptores de imunoglobulina-como
Neste Artigo
Resumo
Pilha de assassino quantitativa imunoglobulina-como receptor (KIR) semi-automático digitação (qKAT) é um método simples, alto rendimento e baixo custo para copiar números genes KIR de tipo para sua aplicação em estudos de associação de população e a doença.
Resumo
Receptores de imunoglobulina-like da pilha de assassino (KIRs) são um conjunto de receptores imunes inibitórios e ativando, assassinas naturais (NK) e células T, codificadas por um cluster polimórfico de genes no cromossomo 19. Seus ligantes mais bem caracterizadas são as moléculas de antigénios (HLA) de leucócito humano que são codificadas dentro o locus de (MHC) complexo principal de histocompatibilidade no cromossomo 6. Não há evidências substanciais de que desempenham um papel significativo na imunidade, reprodução e transplante de órgãos, tornando-se crucial ter técnicas que podem com precisão genótipo-los. No entanto, sequência de alta homologia, também como alélica e variação do número de cópia, dificultam ao genótipo e, eficientemente, métodos de projeto que podem com precisão todos os genes KIR . Os métodos tradicionais são geralmente limitados a resolução dos dados obtidos, produtividade, rentabilidade e o tempo necessário para configurar e executar os experimentos. Nós descrevemos um método chamado quantitativo KIR digitação semi-automática (qKAT), que é um método de reação em cadeia de polimerase em tempo real multiplex do elevado-throughput que pode determinar o número de cópias do gene para todos os genes no locus do KIR . qKAT é um método simples de alto rendimento que pode fornecer dados de alta resolução KIR cópia números, que podem ser mais usados para inferir as variações nos haplótipos estruturalmente polimórficas que eles englobam. Dados de número e haplótipo esta cópia podem ser benéficos para estudos sobre associações de doença em grande escala, genética de populações, bem como investigações sobre a expressão e interações funcionais entre KIR e HLA.
Introdução
Em humanos, os receptores de imunoglobulina-como assassino(KIR) locus é mapeado no braço longo do cromossomo 19 dentro o leucócitos do receptor complexo (LRC). Esse locus é cerca de 150 kb de comprimento e inclui 15 KIR genes arranjados cabeça a cauda. Os loci KIR que é atualmente conhecidos são KIR2DL1, KIR2DL2/KIR2DL3 KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1-5, KIR3DL1/KIR3DS1, KIR3DL2-3, e dois pseudogenes, KIR2DP1 e KIR3DP1. Os genes KIR codificam para bidimensional (2D) e tridimensionais (3D) domínio de imunoglobulina-como receptores com short (S; ativando) ou longa (L; inibitório) cauda citoplasmática, que é expressos por células assassinas naturais (NK) e subconjuntos de T células. Variação de número de cópia exibiu dentro das formas de locus KIR diversos haplótipos com gene variável conteúdo1. Recombinação homóloga não-alélica (NAHR), facilitada por um arranjo de gene de cabeça-de-cauda estreita e sequência de alta homologia, é o mecanismo proposto para ser responsável para a variabilidade de haplotypic. Mais de 100 diferentes haplótipos foram relatados em populações em todo o mundo1,2,3,4. Todos estes haplótipos podiam ser divididos em dois grandes grupos: A e B haplótipos. O haplótipo A contém 7 genes KIR : KIR3DL3, KIR2DL1, KIR2DL3, KIR2DL4, KIR3DL1e KIR3DL2, que são inibitórios genes KIR e a activação KIR gene KIR2DS4. No entanto, até 70% de indivíduos de origem europeia que são homozigotos para KIR haplótipo A exclusivamente levar para uma forma de "exclusão" não-funcional de KIR2DS45,6. Todas as outras combinações de genes KIR formam grupo B haplótipos, incluindo pelo menos um dos genes KIR específicos KIR2DS1, KIR2DS2, KIR2DS3, KIR2DS5, KIR3DS1, KIR2DL2, e KIR2DL5e tipicamente incluem dois ou mais genes KIR ativando.
Moléculas de HLA classe I foram identificados como ligantes para determinados receptores inibitórios (KIR2DL1, KIR2DL2, KIR2DL3e KIR3DL1), ativando os receptores (KIR2DS1, KIR2DS2, KIR2DS4, KIR2DS5e KIR3DS1), e para KIR2DL4, que é um exclusivo KIR que contém um longo citoplasmático rabos como outros receptores KIR inibitórios, mas também tem um resíduo de carga positiva perto o domínio extracelular, que é um comum característica de outros receptores KIR ativando. A combinação de variantes dentro dos genes KIR e os genes HLA influencia a interação do ligante do receptor que formas potencial NK célula capacidade de resposta a nível individual7,8. Evidências de estudos genéticos Associação indicou que KIR desempenha um papel na resistência viral (ex.., vírus de imunodeficiência humana [HIV]9 e hepatite C vírus [HCV]10), o sucesso do transplante de 11, o risco de gravidez transtornos e sucesso reprodutivo12,13, a proteção contra a recaída após células-tronco hematopoéticas alogênico transplante (TCTH)14,15, 16e o risco de cancros17.
A combinação de sequência de alta homologia e alélica e haplotypic diversidade apresenta desafios na tarefa de genes KIR com precisão genotipagem. Métodos convencionais para digitar genes KIR incluem a primeira demão de sequência-específica (SSP) do polymerase reação em cadeia (PCR)18,19,20, sequência específica oligonucleotide sonda PCR (SSOP)21, e laser assistida matriz desorção ionização-tempo de voo de espectrometria de massa (MALDI-TOF MS)22. As desvantagens dessas técnicas são que eles só fornecem visão parcial sobre o genótipo de um indivíduo enquanto também ser trabalhoso executar. Recentemente, sequenciamento de próxima geração (NGS) tem sido aplicado para digitar o locus KIR especificamente. Enquanto este método é muito poderoso, pode ser caro executar, e é demorado para realizar verificações de dados e análise em profundidade.
qKAT é um método PCR quantitativo elevado-throughput. Enquanto os métodos convencionais são laborioso e demorado, este método torna possível executar quase 1.000 amostras de DNA (gDNA) genômicas em cinco dias e dá o genótipo KIR , bem como o número de cópia do gene. qKAT é composto por dez reações multiplex, cada um dos quais metas dois loci KIR e um gene de referência de um número fixo de cópia no genoma (STAT6) utilizado para a quantificação relativa do gene KIR copiar número23. Este ensaio tem sido usado com sucesso em estudos envolvendo painéis de grande população e doença coortes sobre doenças infecciosas como HCV, doenças auto-imunes, como diabetes tipo 1 e transtornos da gravidez como pré-eclâmpsia, bem como fornecendo uma genética na base de estudos que visa compreender o NK celular função1,4,24,25,26.
Protocolo
1. preparação e chapeamento do DNA
- Quantificar com precisão a concentração de gDNA usando um instrumento espectrofotométrico ou fluorométrica.
- Dilua o DNA a 4 ng / µ l em uma placa de 96 poços profundos-bem. Incluem pelo menos uma amostra de gDNA de controle com um número de cópia conhecida e um modelo de controle.
- Centrifugar as placas de 96 poços a 450 x g por 2 min.
- Usando um instrumento de manipulação de líquidos, dispensar cada amostra em quadriplicado nas chapas de qPCR 384-bem, para que cada poço tem 10 ng de DNA (2,5 µ l/poço). Prepare pelo menos dez chapas de 384-bem, uma para cada reação qKAT.
- Se gDNA fornecimento de mais de uma placa de 96 poços, realize uma lavagem de volume completo com 2% de lixívia e água ultrapura para limpar as agulhas do líquido sistema entre cada placa de 96 poços de gDNA amostras de manipulação.
- Secar o DNA por incubar as placas de 384-bem em uma área limpa, em temperatura ambiente pelo menos 24 h.
2. preparação das sondas e Primers
Nota: qKAT é composto por dez reações multiplex. Cada reação inclui três pares da primeira demão e três sondas de fluorescência-etiquetada que amplificam especificamente dois genes KIR e referência de um gene. As sondas que foram publicadas no Jiang et al.27 foram modificadas para que os oligonucleotides agora são rotulados com corantes ATTO desde que eles oferecem maior fotoestabilidade e vidas sinal longo. Cartilha pré-aliquotadas combinações são comercialmente disponíveis (ver Tabela de materiais).
- Prepare as combinações de cartilha para cada reação conforme as diluições indicado na tabela 1.
- Prepare sonda combinações para cada reação, conforme tabela 1. Teste cada sonda individual antes de fazer a combinação.
3. preparação do Master Mix
Nota Os volumes mencionados abaixo são para a realização de uma reação de qKAT em um conjunto de placas de 10 x 384-bem.
- Certifique-se de que as amostras de gDNA chapeadas com as placas do 384-bem estão completamente secas. Realizar todos os passos no gelo e manter os reagentes cobertos da exposição à luz tanto quanto possível, desde que as sondas de fluorescência-etiquetada são foto - e termo-sensível.
- Degele o buffer de qPCR, primer e alíquotas de sonda a 4 ° C.
- No gelo, prepare uma mistura de mestre para placas de 10 x 384-bem adicionando 18,86 mL de água ultrapura, 20 mL de tampão de qPCR, 1.000 µ l de combinação preprepared cartilha e 180 µ l de combinação de sonda preprepared (tabela 2).
- Distribua uniformemente a mistura de mestre através de uma placa de 96 de profundidade bem usando uma pipeta multicanal, pipetagem 415 µ l em cada poço. Manter esta placa em uma caixa de gelo coberta de luz.
- Usando um instrumento de manipulação de líquidos, dispense 9,5 µ l do mix mestre em cada poço da placa 384-bem com gDNA seca. Selar o prato com uma folha e coloque-o imediatamente a 4 ° C. Repita este processo para as restantes placas, garantindo que as agulhas do líquido sistema de manipulação são lavadas com água entre cada prato.
- Centrifugar as placas de 384 poços a 450 x g por 3 min e incube-os a 4 ° C durante a noite ou entre 6-12 h para Ressuspender o DNA e para dissipar quaisquer bolhas de ar.
4. ensaio de qPCR
- Após a incubação durante a noite, centrifugar a 450 x g durante 3 min dissipar quaisquer bolhas de ar restantes.
- Para efeitos de automação, ligue a máquina de qPCR (por exemplo, LightCycler 480) a um manipulador de microplaca (ver Tabela de materiais). Programa manipulador de microplacas para colocar as placas na máquina qPCR de uma doca de armazenamento refrigerado protegido da luz.
Nota os ensaios devem, em teoria, trabalhar em outras máquinas de qPCR com configurações de ópticas compatíveis. - Use as seguintes condições de ciclismo: 95 ° C por 5 min, seguido de 40 ciclos de 95 ° C por 15 s e 66 ° C para 50 s, com coleta de dados a 66 ° C.
- Uma vez concluída a execução, tem o robô recolher a placa da máquina qPCR e colocá-lo no banco do réus de descarte.
5. após a execução da análise
- Após a amplificação, calcular os valores de ciclo (Cq) de quantificação usando o segundo método de máximo derivativo ou o método de pontos de ajuste com o software da máquina qPCR (ver Tabela de materiais), seguindo os passos abaixo.
- Abra o software de qPCR e, na aba do navegador , abra o arquivo de experimento de reação salvos para um prato.
-
Para a análise usando o segundo método de máximo derivado, selecione a guia de análise e criar uma nova análise, usando o método Abs Quant/Second derivado Max.
- Na janela criar nova análise , seleccione o tipo de análise: o método Abs Quant/Second derivado Max, subconjunto: todas as amostras, programa: amplificação, nome: Rx-DFO (onde x é o número de reação).
- Selecione Filtro Comb e escolha VIC/HEX/Yellow555 (533-580). Isso garante que os dados coletados para STAT6 estão selecionados.
- Selecione cor compensação para VIC/HEX/Yellow555(533-580). Clique em calcular. Repita isto para Fam (465-510) e Cy5/Cy5.5(618-660). Clique em salvar arquivo.
-
Para a análise usando o método de pontos de ajuste, selecione Abs Quant/Fit pontos na guia de análise.
- Na janela criar nova análise , seleccione o tipo de análise: o método Abs Quant/Fit pontos, subconjunto: todas as amostras, programa: amplificação, nome: RxF-DFO (onde x é o número de reação).
- Selecione os filtros corretos e compensações de cor para cada um dos genes KIR (Fam/Cy5) e o STAT6 . Na guia Noiseband , defina a banda ruído para eliminar o ruído de fundo.
- Na guia análise , definir os pontos de ajuste para 3 e selecione que mostrar pontos de ajuste. Clique em calcular. Clique em salvar arquivo.
6. exportação dos resultados
- No software qPCR, abra o navegador Tab. Selecione Exportar de lote de resultados.
- Abra a pasta na qual os arquivos do experimento são salvos em transferir os arquivos para a seção do lado direito da janela. Clique em seguinte. Selecione o nome e o local do arquivo de exportação.
- Selecione o tipo de análise o método Abs Quant/Second derivado Max ou Abs Quant/Fit pontos. Clique em seguinte. Verifique se o nome do arquivo, a pasta de exportação e o tipo de análise estão correto e clique em próximo para iniciar o processo de exportação.
- Espere até que o Status de exportação é Okey. A tela automaticamente se moverá para o próximo passo. Verifique que todos os arquivos selecionados foram exportados com sucesso para que o número de arquivos falhou = 0. Clique em concluído.
- Use scripts split_file.pl e roche2sds.pl para dividir as placas exportadas em reações individuais para cada placa.
Nota os scripts são fornecidos na solicitação/GitHub.
7. Copie o número de cálculos
- Abra o software de análise número da cópia (por exemplo, CopyCaller). Selecione Importar arquivo de resultados PCR em tempo real e carregar arquivos de texto criados pelo roche2sds.pl.
- Selecione analisar e realizar a análise ou selecionando amostra Calibrador com número de cópia conhecida ou selecionando o número mais frequente de cópia. Ver tabela 5 para o número mais frequente de cópia de genes KIR tipicamente observada em populações de origem europeia.
8. qualidade dos dados verifica
- Use script de R KIR_CNVdata_analysis_for_Excel_ver020215. R para combinar dados de número de cópia de todas as placas em uma planilha.
Nota os scripts são fornecidos na solicitação/GitHub. - Verifique novamente os dados brutos sobre o software de análise de número de cópia para amostras que não estão em conformidade com o desequilíbrio de ligação conhecida (LD) para genes KIR (quadro 6).
Resultados
Análise do número de cópia pode efectuar exportando os arquivos para o software de análise número de cópia, que fornece o número de cópia previsto e estimado baseado no método ΔΔCq.
O número de cópia pode ser previsto com base no número de amostras de DNA de controle na placa ou inserindo o número de cópia de gene mais frequente (tabela 5) cópia conhecida. A Figura 1 mostra os resultados de uma placa para uma reação que tem como alvo KIR2DL4 e KIR3DS1, bem como o gene de referência STAT6. O número de cópia mais frequente para KIR2DL4, um gene de quadro no locus KIR , é duas cópias, Considerando que o número de cópia mais frequente para KIR3DS1, um gene de activação, é uma cópia. Os resultados na figura mostram as parcelas de amplificação do PCR observadas sobre o software de qPCR e os dados de número de cópia gerados a partir dos dados de qPCR. Como mostrado, o ensaio é capaz de distinguir entre 0, 1, 2, 3 e 4 número de cópias do gene de KIR . O software de análise de número de cópia também permite uma visualização da distribuição do número de cópia através da placa como um gráfico de pizza ou um gráfico de barras. A eficácia da previsão de número de cópia é inferior para amostras com um número maior de cópia.
A qualidade de todos os materiais usados nas reações gDNA, tampão, primers e sondas, pode afetar a precisão dos resultados obtidos. No entanto, a discordância nos resultados é mais provável ser causado devido a variação na concentração de DNA através de uma placa. A pureza do gDNA extraído, o que pode ser medido usando a 260/280 e 260/230 rácios, também pode ter um efeito sobre a qualidade. Um rácio de 260/280 cuja relação 1,8-2 e 260/230 2-2.2 são desejáveis. Um concentrações de intervalo irregular de DNA através de uma placa podem levar a uma alta variabilidade no ciclo de limiar (Ct) entre amostras e discordância no intervalo do número estimado de cópia. Os resultados na Figura 2 mostram o efeito que a disparidade entre os valores det C através de uma placa pode ter sobre a exactidão na predição do número de cópia. A linha vermelha indica o intervalo do número estimado de cópia para uma amostra e, idealmente, deveria ser mais próximo de um número inteiro possível.
Os dados de número de cópia, uma vez analisados, podem ser exportados como um arquivo de planilha em um formato de 96 poços. Usamos um script de R (disponível quando solicitado) para combinar os dados de número de cópia de todos os 10 placas que são executados como um conjunto em uma planilha. Os dados publicados sobre KIRs de populações de origem europeia na maior parte permite a previsão de regras de LD que existem entre os vários genes no complexo KIR 1. Estas previsões são usados para conduzir verificações a jusante na cópia números resultados obtidos (tabela 6). Amostras que não estão em conformidade com o previsto LD entre os genes podem conter incomum polimorfismo ou variações estruturais de haplotypic. Um fluxograma descrevendo o protocolo é mostrado na Figura 3.
Uma ferramenta chamada KIR haplótipo identificador (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) foi desenvolvida para facilitar a imputação de haplótipos do conjunto de dados. A imputação funciona com base numa lista de haplótipos de referência observadas em uma população de origem europeia1. No entanto, a ferramenta também permite um conjunto personalizado de haplótipos de referência a ser usado em vez disso. Três arquivos separados são gerados; o primeiro arquivo lista todas as combinações de haplótipo para uma amostra, o segundo arquivo fornece uma lista aparada as combinações de haplótipos que têm as mais altas frequências combinadas, e o terceiro arquivo listas de exemplos que não podem ser atribuídos haplótipos. Non-atribuição de haplótipos poderia ser usado como um indicador de haplótipos de romance.
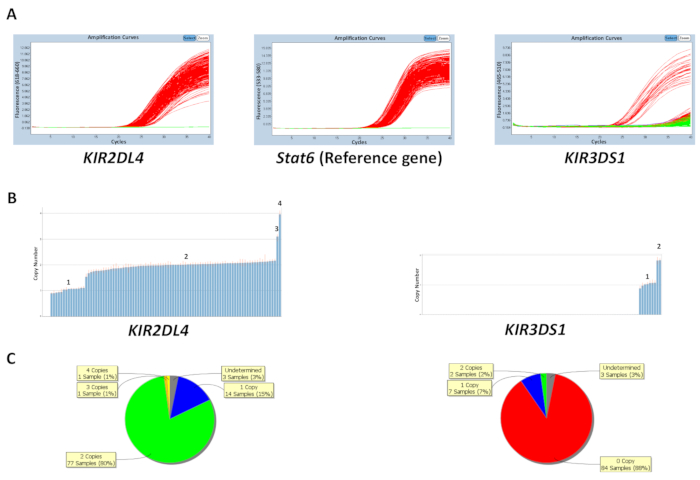
Figura 1: número de resultados representativos de uma placa para a reação 5. (A), este painel mostra amplificação parcelas. (B), este painel mostra copiar números parcelas. (C), este painel mostra a distribuição do número de cópia. Clique aqui para ver uma versão maior desta figura.
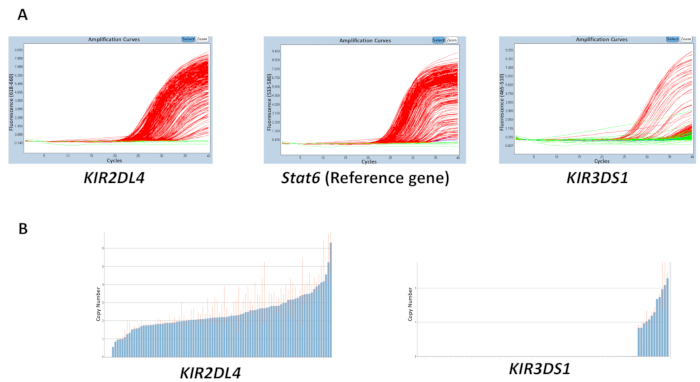
Figura 2: número de resultados representativos de uma placa com uma concentração variável de DNA pela reação 5. (A), este painel mostra amplificação parcelas. (B), este painel mostra copiar números parcelas. Clique aqui para ver uma versão maior desta figura.
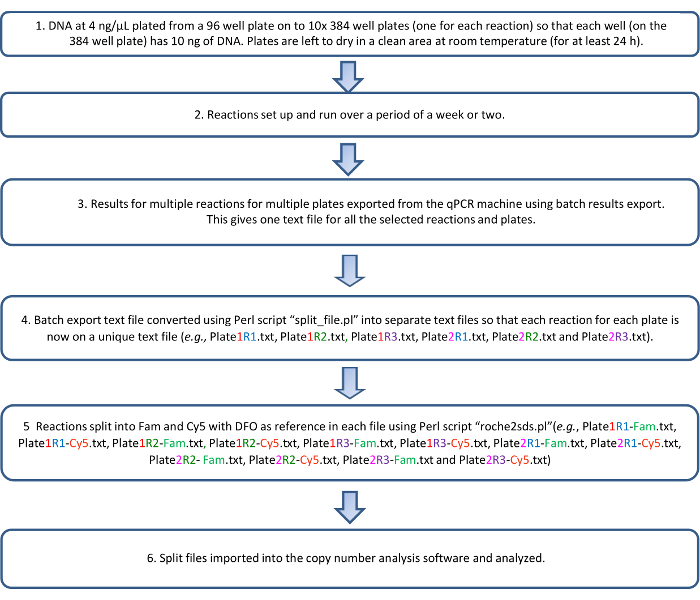
Figura 3: fluxograma do protocolo qKAT. Clique aqui para ver uma versão maior desta figura.
| Ensaio | Genes | Primers para a frente | Concentração (nM) | Primers reversos | Concentração (nM) | Sondas | Concentração (nM) |
| N º 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2DL 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 3 | 3DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 4 | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 5 | 3DS1 | B2F | 250 | B1R | 250 | P4b | 150 |
| 2DL 4 | C1F | 200 | C1R | 200 | P5B - 2DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 6 | 2DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| 2DL 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 8 | 2DS3 | B5F | 250 | E55 | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | C55 | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N º 10 | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
Tabela 1: combinação e concentração de primers e sondas utilizadas em cada reação de qKAT 27 .
| Reação | Primeira demão alíquotas (µ l) | Alíquotas de sonda (µ l) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | ÁGUA | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | ÁGUA | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| Nota: preciso de 20 µ l de menos água no MasterMix | |||||||||||
| R3 | 3DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | ÁGUA | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | ÁGUA | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | ÁGUA | STAT6F | STAT6R | P4B | P5B - 2L 4 | PSTAT6 |
| 2DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | ÁGUA | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | ÁGUA | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | E55 | D4F | C55 | ÁGUA | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | ÁGUA | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | ÁGUA | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
Tabela 2: Volumes (µ l) da cartilha de 100 µM/ponta de estocam soluções para fazer a primeira demão e sonda alíquotas de combinação.
| Nome | Direção | modificação de 5´ | modificação de 3´ | Sequência (5' →3') | Comprimento | TM | % DE GC | Exão | Posição |
| P4a | Sentido | FAM | BHQ-1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44,4 | 4 | 425-451 |
| P4b | Antisentido | FAM | BHQ-1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5B | Sentido | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828-852 |
| P5B - 2DL 4 | Sentido | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828-852 |
| P9 | Sentido | ATTO647N | BHQ-2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62,5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ-2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
Tabela 3: lista das sondas utilizadas em qKAT 1, 27. os corantes fluorescentes usados na extremidade 5' das sondas oligo P5b, P5b - 2 DL 4, P9 e PSTAT6 foram modificados para corantes ATTO.
| Gene | Primeiras demão | Direção | Sequência (5´-3´) | Comprimento | TM | % DE GC | Exão | Posição | Amplicon (bp) | Alelos podem ser desperdiçados. | ||
| 3DL2e4 | A1F | Para a frente | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 008 * 2, * 021, * 027, * 038. | ||
| A1R | Inverter | CTGCAAGGACAG GCATCAA | 19 | 53 | 52,6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | Para a frente | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 112 | Nenhum | ||
| A5R | Inverter | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55.6 | 492-509 | Nenhum | |||||
| 2DS2 | A2F | Para a frente | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 111 | Nenhum | ||
| A6R | Inverter | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42,9 | 488-508 | Nenhum | |||||
| 3DL 3 | A8Fa | Para a frente | GTGAAATCGGGA GAGACG | 18 | 50 | 55.6 | 4 | 406-423 | 139 | Nenhum | ||
| A8Fb | Para a frente | GGTGAAATCAGG AGAGACG | 19 | 50 | 52,6 | 405-423 | 3DL 3 * 054, 3DL 3 * 00905. | |||||
| A8R | Inverter | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | Nenhum | |||||
| 3DL1e4 | B1F | Para a frente | CATCGGTCCCAT GATGCT | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DL 1 * 00505, 3DL 1 * 006, 3DL 1 * 054, 3DL 1 * 086, 3DL 1 * 089 | ||
| B1R | Inverter | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | Para a frente | CATCGGTTCCAT GATGCG | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DS1 * 047; pode pegar 3DL 1 * 054. | ||
| B1R | Inverter | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | Nenhum | |||||
| 2DL 1 | B3F | Para a frente | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | 2DL 1 * 020, 2DL 1 * 028 | ||
| B3R | Inverter | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | 2DL 1 * 023, 2DL 1 * 029, 2DL 1 * 030 | |||||
| 2DS1 | B4F | Para a frente | TCTCCATCAGTC GCATGAA | 19 | 51 | 47,4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | Inverter | GGTCACTGGGAG CTGAC | 17 | 49 | 64,7 | 624-640 | Nenhum | |||||
| 2DS3 | B5F | Para a frente | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546-563 | 96 | Nenhum | ||
| E55 | Inverter | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | Nenhum | |||||
| 2DS5 | B6F2 | Para a frente | AGAGAGGGGACG TTTAACC | 19 | 50 | 52,6 | 4 | 475-493 | 173 | Nenhum | ||
| B6R3 | Inverter | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66,7 | 630-647 | 2DS5 * 003 | |||||
| 2DL 4 | C1F | Para a frente | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55.6 | 5 | 808-825 | 83 | Nenhum | ||
| C1R | Inverter | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52,6 | 872-890 | 2DL 4 * 018, 2DL 4 * 019 | |||||
| 2DL 2 | 2DL2F4 | Para a frente | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57.9 | 5 | 778-796 | 151 | 2DL 2 * 009; 782G mudado para r. | ||
| C3R2 | Inverter | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | Nenhum | |||||
| 2DS4 | C5F | Para a frente | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | Nenhum | ||
| C5R | Inverter | TTGACCACTCGT AGGGAGC | 19 | 52 | 57.9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | Para a frente | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57.9 | 5 | 750-768 | 203 | Nenhum | ||
| 2DS4R2 | Inverter | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Nenhum | |||||
| 2DS4FL | 2DS4FL | Para a frente | CCGGAGCTCCTA TGACATG | 19 | 53 | 57.9 | 5 | 744-762 | 209 | Nenhum | ||
| 2DS4R2 | Inverter | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Nenhum | |||||
| 2DL 3 | D1F | Para a frente | AGACCCTCAGGA GGTGA | 17 | 48 | 58,8 | 9 | 1180-1196 | 156 | Nenhum | ||
| D1R | Inverter | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010, 2DL 3 * 017, 2DL 3 * 01801 e 2DL 3 * 01802 | |||||
| 2DL 5 | D2F | Para a frente | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 e 2DL5B * 020 | ||
| D2R | Inverter | GGCAGGAGACAA TGATCTT | 19 | 49 | 47,4 | 1315-1333 | Nenhum | |||||
| 2DP1 | D3F | Para a frente | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | Nenhum | ||
| D3R | Inverter | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | Nenhum | |||||
| 3DL1e9 | D4F | Para a frente | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 93 | 3DL 1 * 061, 3DL 1 * 068 | ||
| D4R2 | Inverter | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43,5 | 1273-1295 | 3DL 1 * 05901, 3DL 1 * 05902, 3DL 1 * 060, 3DL 1 * 061, 3DL 1 * 064, 3DL 1 * 065, 3DL 1 * 094N, 3DL 1 * 098 | |||||
| 3DL2e9 | D4F | Para a frente | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 156 | Nenhum | ||
| D5R | Inverter | GACCTGACTGTG GTGCTCG | 19 | 54 | 63,2 | 1340-1358 | Nenhum | |||||
| STAT6 | STAT6F | Para a frente | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129. | |||||
| STAT6R | Inverter | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
Tabela 4: sequências dos primers utilizados em qKAT 1, 27.
| KIR gene | 3DL 3 | 2DS2 | 2DL 2 | 2DL 3 | 2DP1 | 2DL 1 | 3DP1 | 2DL 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | 2DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 Total | 2DS4 FL | 2DS4 DEL | 3DL 2 EX4 | 3DL 2 EX9 | |
| Número de cópia mais frequente | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
Tabela 5: Número de cópia mais frequente para KIR genes comumente observados em amostras de origem europeia.
| Regras de desequilíbrio de ligação para qKAT com base em populações europeias | Verificação do número de cópia | |||||||
| 1 | KIR3DL3, KIR3DP1, KIR2DL4 e KIR3DL2 são quadro genes presentes em ambos os haplótipos. | KIR3DL3, KIR3DP1, KIR2DL4 e KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 e KIR2DL2 são em LD uns com os outros | 2DS2=2 DL 2 | ||||||
| 3 | KIR2DL2 e KIR2DL3 são alelos de um mesmo gene | 2 DL de 2+2 DL 3= 2 | ||||||
| 4 | KIR2DP1 e KIR2DL1 são em LD uns com os outros | 2DP1=2 DL 1 | ||||||
| 5 | Exon 4 de KIR3DL1 e KIR3DL2 é igual a exon 9 de KIR3DL1 e KIR3DL2 respectivamente. | 3DL1ex4=3DL1ex9 e 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 e KIR3DS1 são alelos | 3 DL 1+3DS1= 2 | ||||||
| 7 | KIR2DS3 e KIR2DS5 estão em LD com KIR2DL5 | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1 e KIR2DS1 estão em LD | 3DS1=2DS1 | ||||||
| 9 | Presença de KIR2DS1 e KIR2DS4Tvirgula é mutuamente exclusiva em um haplótipo | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL e KIR2DS4del são variantes de KIR2DS4TOTAL | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
Tabela 6: desequilíbrio de ligação entre KIR os genes comumente observados em populações de origem Europeia podem ser usados para verificar os dados de número de cópia 1,27.
Discussão
Nós descrevemos um método de alta produtividade semi-automatizadas romance, chamado qKAT, que facilita a digitação de número cópia dos genes KIR . O método é uma melhoria sobre os métodos convencionais como SSP-PCR, que são de baixo rendimento e só podem indicar a presença ou ausência destes genes altamente polimórficos.
A precisão dos dados cópia números obtidos é dependente de vários fatores, incluindo a qualidade e uniformidade-concentração de amostras gDNA e a qualidade dos reagentes. A qualidade e a precisão das amostras gDNA através de uma placa são extremamente importantes, uma vez que as variações na concentração do outro lado da placa podem resultar em erros no cálculo do número de cópia. Desde os ensaios foram validados utilizando conjuntos de exemplo de origem europeia, dados de coortes de outras partes do mundo exigem verificações mais minuciosa. Isto é para garantir que instâncias do abandono do alelo ou ligação não-específica da primeira demão/sonda não são mal interpretadas como variação de número de cópia.
Enquanto os ensaios foram projetados e otimizados para rodar como alta produtividade, eles podem ser modificados para executar amostras menos. A métrica de confiança no software de análise do número cópia é afectada quando analisando amostras de menos, mas isso pode ser melhorado se amostras de DNA genômicas controle com um número de cópia de gene conhecido do KIR estão incluídas na placa e repetições de exemplo adicionais são incluído.
Para os laboratórios sem líquido/placa-manipulação de robôs, mistura de mestre pode ser dispensada usando pipetas multicanais e chapas podem ser carregadas manualmente dentro do instrumento qPCR.
O principal objetivo por trás do desenvolvimento de qKAT foi criar um simples, alta produtividade, alta resolução e econômica método genótipo KIRs para doença estudos de associação. Isto foi conseguido com sucesso desde que qKAT tem sido empregado em investigar o papel do KIR em vários estudos de associação grande doença, incluindo uma variedade de doenças infecciosas, doenças auto-imunes e distúrbios de gravidez4, 24 , 25 , 26.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
O projeto recebido financiamento do Conselho de pesquisa médica (MRC), o Conselho Europeu de investigação (CEI) sob o programa União Europeia do Horizonte 2020 pesquisa e inovação (grant acordo n. º 695551) e a National Institute of Health (NIH) Cambridge Biomedical Research Centre e sangue de pesquisa de NIH e Transplant Research Unit (NIHR BTRU) na transplantação e na Universidade de Cambridge e em parceria com o sangue de NHS e transplante (NHSBT). As opiniões expressadas são as dos autores e não necessariamente aqueles de NHS, o NIHR, o departamento de saúde ou o NHSBT.
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
Referências
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados