Method Article
qKAT : quantitatif semi-Automated typage des cellules tueuses-gènes de récepteurs immunoglobuline-like
Dans cet article
Résumé
Quantitative des cellules tueuses récepteur immunoglobuline-like (KIR) saisie semi-automatique (qKAT) est une méthode simple, haut-débit et rentable pour copier le numéros gènes de type KIR pour leur application dans les études d’association de la population et de la maladie.
Résumé
Récepteurs de cellules tueuses immunoglobuline-like (RIC) sont un ensemble de récepteurs immunitaires inhibitrices et activation, tueuses naturelles (NK) et de lymphocytes T, codées par un cluster polymorphe des gènes sur le chromosome 19. Leurs ligands mieux caractérisés sont les molécules d’antigen (HLA) de leucocytes humains qui sont codés dans le locus (MHC) complexe majeur d’histocompatibilité sur le chromosome 6. Il y a des preuves substantielles qu’ils jouent un rôle important dans l’immunité, la reproduction et la transplantation, rend crucial d’avoir des techniques qui peuvent précisément génotype eux. Cependant, haute-homologie, aussi bien qu’allélique et variation numéro de copie, rendent difficile de génotype et efficacement les méthodes de conception qui peuvent avec précision tous les gènes KIR . Les méthodes traditionnelles sont généralement limitées dans la résolution des données obtenues, débit, la rentabilité et le temps nécessaire à la mise en place et d’exploiter les expériences. Les auteurs décrivent une méthode appelée quantitative KIR saisie semi-automatique (qKAT), qui est une méthode de PCR multiplex Real-Time polymerase haut débit qui permet de déterminer le nombre de copies du gène pour tous les gènes dans le locus KIR . qKAT est une méthode simple de haut débit qui peut fournir des données à haute résolution KIR copie numéros, qui permet plus d’en déduire les variations dans les haplotypes structurellement polymorphes qui englobent les. Ces données de nombre et haplotype de copie peuvent être bénéfiques pour les études sur les associations de maladies à grande échelle, la génétique des populations, ainsi que les enquêtes sur l’expression et les interactions fonctionnelles entre KIR et HLA.
Introduction
Chez l’homme, le tueur récepteur immunoglobuline-like(KIR) locus est localisé sur le bras long du chromosome 19 dans le complexe de récepteur de leucocyte (LRC). Ce locus est d’environ 150 Ko de long et comprend 15 KIR gènes disposées tête à queue. Les locus KIR qui sont actuellement connues sont KIR2DL1, KIR2DL2/KIR2DL3, KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1-5, KIR3DL1/KIR3DS1, KIR3DL2-3, et deux pseudogènes, KIR2DP1 et KIR3DP1. KIR gènes codent pour deux dimensions (2D) et tridimensionnels (3D) domaine d’immunoglobuline-like récepteurs avec short (S ; activation) ou long (L ; inhibitrice) pastilles cytoplasmique, qui sont exprimés par les cellules tueuses naturelles (NK) et de sous-ensembles de T cellules. Copie variation numéro exposé dans les formes de locus KIR différents haplotypes avec gène variable contenu1. Recombinaison homologue non-alléliques (NAHR), facilitée par un arrangement de gène de tête-à-queue étroite et haute-homologie, est le mécanisme proposé d’être responsable pour la variabilité haplotypique. Plus de 100 différents haplotypes ont été signalés dans les populations dans le monde1,2,3,4. Tous ces haplotypes pourrait être divisées en deux grands groupes : les haplotypes A et B. L’haplotype A contient 7 gènes KIR : KIR3DL3, KIR2DL1, KIR2DL3, KIR2DL4, KIR3DL1et KIR3DL2, qui sont des gènes inhibiteurs de KIR et l’activation KIR gène KIR2DS4. Cependant, jusqu'à 70 % de personnes d’origine européenne qui sont homozygotes pour KIR haplotype A exclusivement effectuer une forme non fonctionnelle « suppression » de KIR2DS45,6. Toutes les autres combinaisons de gènes KIR forment d’haplotypes du groupe B, dont au moins un des gènes spécifiques KIR KIR2DS1, KIR2DS2, KIR2DS3, KIR2DS5, KIR3DS1, KIR2DL2, et KIR2DL5et comprennent généralement deux ou plusieurs gènes KIR activant.
Molécules HLA classe I ont été identifiés comme les ligands pour certains récepteurs inhibiteurs (KIR2DL1, KIR2DL2, KIR2DL3et KIR3DL1), activant les récepteurs (KIR2DS1, KIR2DS2, KIR2DS4, KIR2DS5et KIR3DS1), et pour KIR2DL4, qui est un unique KIR qui contient un long cytoplasmique queues comme autres récepteurs inhibiteurs de KIR, mais dispose également d’un résidu chargé positivement près le domaine extracellulaire qui est un commun fonction d’autres récepteurs KIR activant. La combinaison des variantes dans les gènes KIR et les gènes HLA influence interaction ligand du récepteur cette réactivité de cellules NK potentielle de formes au niveau individuel7,8. Preuves provenant d’études de liaison génétique a indiqué que le KIR joue un rôle dans la résistance virale (par exemple., le virus d’immunodéficience humaine [VIH]9 et l’hépatite C [VHC]10), le succès de la greffe 11, le risque de troubles de la grossesse et le succès reproducteur12,13, la protection contre les rechutes après allogreffe de cellules souches hématopoïétiques (CSH) la transplantation14,15, 16et le risque de cancers17.
La combinaison d’homologie de séquence haute et alléliques et haplotypiques diversité présente défis dans la tâche de façon précise les gènes KIR génotypage. Les méthodes conventionnelles de type KIR gènes comprennent amorce séquence-spécifique (SSP) polymerase chain reaction (PCR)18,19,20, séquence spécifique oligonucléotide sonde PCR (SSOP)21, et laser assistée matrice desorption ionisation et l’heure de vol de spectrométrie de masse (MALDI-TOF MS)22. Les inconvénients de ces techniques sont qu’ils fournissent seulement partiel aperçu le génotype d’un individu tout en étant aussi laborieux à effectuer. Récemment le séquençage de prochaine génération (NGS) a été appliqué pour taper le locus KIR spécifiquement. Alors que cette méthode est très puissante, il peut être coûteux à exécuter, et c’est beaucoup de temps mener des contrôles approfondis de données et d’analyse.
qKAT est une méthode PCR quantitative de haut débit. Alors que les méthodes conventionnelles sont laborieux et fastidieux, cette méthode permet d’exécuter presque 1 000 échantillons d’ADN (génomique) génomiques en cinq jours et donne le génotype KIR , ainsi que le nombre de copies du gène. qKAT se compose de dix réactions multiplexes, dont chacun vise deux loci KIR et un gène de référence d’un nombre de copie fixée dans le génome (STAT6) utilisé pour la quantification relative du gène KIR copier le numéro23. Ce test a été utilisé avec succès dans les études portant sur les panneaux de la grande population et cohortes de la maladie sur les maladies infectieuses comme le VHC, des maladies auto-immunes comme le diabète de type 1 et des troubles de la grossesse comme la pré-éclampsie, ainsi que fournir une génétique base à des études visant à comprendre les NK cell function1,4,24,25,26.
Protocole
1. préparation et électrodéposition d’ADN
- Quantifier précisément la concentration de génomique à l’aide d’un instrument spectrophotométrique ou fluorimétrique.
- Diluer l’ADN à 4 ng/µL sur une plaque de 96 puits puits profonds. Inclure au moins un échantillon d’Adng de contrôle avec un nombre de copies connues et un contrôle sans modèle.
- Centrifuger les plaques 96 puits à 450 x g pendant 2 min.
- À l’aide d’un instrument de manipulation de liquides, répartir chaque échantillon en quatre exemplaires sur plaques 384 puits qPCR afin que chaque cupule a 10 ng d’ADN (2,5 µL/puits). Préparer au moins dix plaques 384 puits, un pour chaque réaction de qKAT.
- Si Adng est actuellement distribué depuis plus d’une plaque à 96 puits, effectuer un lavage de volume complet avec 2 % eau de Javel et eau ultrapure pour nettoyer les aiguilles du liquide système entre chaque plaque à 96 puits d’échantillons Adng de manutention.
- Sécher à l’air l’ADN en incubant les plaques 384 puits dans un endroit propre à température ambiante pendant au moins 24 h.
2. préparation des amorces et des sondes
Remarque : qKAT se compose de dix réactions multiplexes. Chaque réaction comprend trois paires d’amorces et de trois sondes marquées fluorescence qui amplifient spécifiquement deux gènes KIR et gène une seule référence. Les sondes qui ont été publiés dans Jiang et al.,27 ont été modifiés afin que les oligonucléotides sont maintenant marqués avec ATTO colorants car ils offrent photostabilité améliorée et longues durées de vie. Pré-aliquotés d’amorces sont disponibles dans le commerce (voir Table des matières).
- Préparer des paires d’amorces pour chaque réaction selon les dilutions indiquées au tableau 1.
- Préparer les combinaisons de sonde pour chaque réaction conformément au tableau 1. Chaque sonde individuel avant de faire la combinaison de contrôle.
3. préparation du Mix Master
NOTE Les volumes mentionnés ci-dessous sont pour effectuer une réaction de qKAT sur un ensemble de plaques de 10 x 384 puits.
- S’assurer que les échantillons d’Adng plaqués sur les plaques 384 puits soient complètement secs. Procéder à toutes les étapes sur la glace et garder les réactifs couverts contre l’exposition à la lumière autant que possible, puisque les sondes marquées fluorescence sont photosensibles et thermo.
- Décongeler le tampon de qPCR, apprêt et sonde aliquotes à 4 ° C.
- Sur la glace, préparer un mélange maître pour plaques de 10 x 384 puits en ajoutant 18,86 mL d’eau ultrapure, 20 mL de tampon de qPCR et 1 000 µL de combinaison d’amorces des 180 µL de combinaison des sonde (tableau 2).
- Répartir le mélange maître à travers une plaque de profondeur 96 puits à l’aide d’une pipette multicanaux, pipetage 415 µL à chaque puits. Garder cette plaque dans une glacière couverte de la lumière.
- À l’aide d’un instrument de manipulation de liquides, distribuer 9,5 µL du mélange maître dans chaque puits de la plaque 384 puits avec Adng séchée. Sceller la plaque avec une feuille et le placer immédiatement à 4 ° C. Répétez ce processus pour les plaques restantes, veiller à ce que les aiguilles du liquide système de manutention sont lavés avec de l’eau entre chaque plaque.
- Centrifuger les plaques 384 puits à 450 x g pendant 3 min et les incuber à 4 ° C durant la nuit ou entre 6 à 12 h pour remettre en suspension l’ADN et à dissiper les bulles d’air.
4. qPCR Assay
- Après l’incubation durant la nuit, centrifuger à 450 g pendant 3 min dissiper les bulles d’air restantes.
- Aux fins de l’automatisation, brancher la machine de qPCR (p. ex., LightCycler 480) à un gestionnaire de la microplaque (voir Table des matières). Programme gestionnaire microplaque afin de placer les plaques dans la machine de qPCR partir d’un quai de stockage refroidi abri de la lumière.
NOTE les essais devraient, en théorie, travailler sur d’autres machines de qPCR avec paramètres optiques compatibles. - Utiliser les conditions suivantes de cyclisme : 95 ° C pendant 5 min, suivi de 40 cycles de 95 ° C pour 15 s et 66 ° C pendant 50 s, avec la collecte de données à 66 ° C.
- Une fois que la série est terminée, avoir le robot recueillir la plaque de la machine de qPCR et placez-le dans le dock de défausse.
5. après l’exécution de l’analyse
- Après amplification, calculer les valeurs de cycle (Cq) quantification à l’aide de la méthode maximale dérivée seconde ou la méthode des Points de lissage avec le logiciel de la machine de qPCR (voir Table des matières), suivant les étapes ci-dessous.
- Ouvrez le logiciel de qPCR et, dans l’onglet navigateur , ouvrez le fichier d’expérience de réaction enregistrée pour une plaque.
-
Pour l’analyse en utilisant la méthode maximale dérivée seconde, sélectionnez l’onglet analyse et de créer une nouvelle analyse à l’aide de la méthode Abs Quant/Second dérivé du Max.
- Dans la fenêtre Créer nouvelle analyse , sélectionnez le type d’analyse : méthode Abs Quant/Second dérivé du Max, sous-ensemble : tous les échantillons, programme : Amplification, nom : Rx-MPO (où x est le nombre de réaction).
- Sélectionnez Filtre peigne et choisissez VIC/HEX/Yellow555 (533-580). Cela garantit que les données recueillies pour STAT6 sont sélectionnées.
- Sélectionnez couleur Compensation pour VIC/HEX/Yellow555(533-580). Cliquez sur calculer. Répétez cette opération pour Fam (465-510) et Cy5/Cy5.5(618-660). Cliquez sur enregistrer le fichier.
-
Pour l’analyse à l’aide de la méthode d’ajustement Points, sélectionnez Abs Quant/Fit Points dans l’onglet analyse.
- Dans la fenêtre Créer nouvelle analyse , sélectionnez le type d’analyse : méthode Abs Quant/Fit Points, sous-ensemble : tous les échantillons, programme : Amplification, nom : RxF-MPO (où x est le nombre de réaction).
- Sélectionnez les filtres corrects et les compensations de couleur pour STAT6 et chacun des gènes KIR (Fam/Cy5). Dans l’onglet Noiseband , sélectionner la bande de bruit d’exclure le bruit de fond.
- Dans l’onglet analyse , la valeur des points de lissage 3 et sélectionnez que show points de lissage. Cliquez sur calculer. Cliquez sur enregistrer le fichier.
6. l’exportation des résultats
- Dans le logiciel de qPCR, ouvrez le navigateur onglet sélectionnez Résultats Batch Export.
- Ouvrez le dossier dans lequel les fichiers de l’expérience sont enregistrés et transférer les fichiers dans la section droite de la fenêtre. Cliquez sur suivant. Sélectionnez le nom et l’emplacement du fichier d’exportation.
- Sélectionnez le type d’analyse méthode Abs Quant/Second dérivé du Max ou Abs Quant/Fit Points. Cliquez sur suivant. Vérifiez que le nom du fichier, le dossier d’exportation et le type d’analyse sont correct et cliquez sur suivant pour démarrer le processus d’exportation.
- Attendez que l' État d’exportation est Ok. L’écran passera automatiquement à l’étape suivante. Vérifiez que tous les fichiers sélectionnés ont été exportés avec succès pour que le nombre de fichiers a échoué = 0. Cliquez sur terminé.
- Utiliser les scripts split_file.pl et roche2sds.pl pour couper les plaques exportés en réactions individuelles pour chaque plaque.
NOTE les scripts sont fournis sur demande/GitHub.
7. copiez le numéro de calculs
- Ouvrez le logiciel d’analyse numéro copie (par exemple, CopyCaller). Sélectionnez importer fichier de résultats de PCR en temps réel et charger des fichiers texte créés par roche2sds.pl.
- Sélectionnez analyser et procéder à l’analyse de chaque sélection échantillon étalon avec nombre de copie connue ou en sélectionnant le numéro de copie plus fréquent. Voir tableau 5 pour le nombre de copies plus fréquent des gènes KIR généralement observées dans les populations d’origine européenne.
8. qualité des données vérifie
- Utilisez le script R KIR_CNVdata_analysis_for_Excel_ver020215. R de combiner des données de numéros de copie de toutes les plaques dans un tableur.
NOTE les scripts sont fournis sur demande/GitHub. - Revérifier les données brutes sur le logiciel d’analyse numéro de copie pour les échantillons qui ne respectent pas le déséquilibre de liaison connue (LD) pour KIR gènes (tableau 6).
Résultats
Analyse numéro copie peut être effectuée en exportant les fichiers dans le logiciel d’analyse numéro de copie, qui fournit le nombre de copies prédites et estimée selon la méthode de ΔΔCq.
On peut prédire le nombre de copies que soit basée sur le nombre de copie connue contrôle d’échantillons d’ADN sur la plaque ou en entrant le nombre de copies de gènes plus fréquent (tableau 5). La figure 1 montre les résultats d’une plaque pour une réaction qui cible KIR2DL4 et KIR3DS1, ainsi que le gène de référence STAT6. Le nombre de copies plus fréquent pour KIR2DL4, un gène de cadre dans le locus KIR , est deux copies, alors que le nombre de copies plus fréquent pour KIR3DS1, un gène activant, est une copie. Les résultats de la figure montrent les parcelles d’amplification PCR observées sur le logiciel de qPCR et les données de numéro de copie générées à partir des données de qPCR. Comme indiqué, le test est capable de distinguer entre 0, 1, 2, 3 et 4 nombre de copies de gènes KIR . Le logiciel d’analyse numéro de copie permet aussi une visualisation de la distribution du nombre de copies à travers la plaque comme un graphique en secteurs ou un graphique à barres. L’efficacité de la prédiction de nombre de copie est plus faible pour les échantillons ayant un plus grand nombre de copie.
La qualité de tous les matériaux utilisés dans les réactions Adng, tampon, amorces et sondes, peut affecter la précision des résultats obtenus. Toutefois, une discordance dans les résultats est plus susceptible d’être causé par une variation de la concentration de l’ADN sur une plaque. La pureté de l’Adng extrait, qui peut être mesurée à l’aide de la 260/280 et 260/230 rapports, peut aussi avoir un effet sur la qualité. Un ratio 260/280 du ratio 1,8-2 et 260/230 du 2-2. 2 sont souhaitables. Une concentration de portée inégale de l’ADN à travers une plaque peut conduire à une forte variabilité du cycle seuil (Ct) entre les échantillons et une discordance dans la gamme du nombre estimé de copies. Les résultats de la Figure 2 montrent l’effet de que la disparité entre les valeurs det C sur une plaque peut avoir sur l’exactitude dans la prédiction du nombre de copies. La ligne rouge indique la fourchette du nombre estimé de copies pour un échantillon et, idéalement, devrait être aussi proche d’un entier que possible.
Les données de numéro de copie, une fois analysées, peuvent être exportées dans un fichier de feuille de calcul dans un format de 96 puits. Nous avons utilisé un script R (disponible sur demande) pour combiner les données de numéro de copie de toutes les 10 plaques qui fonctionnent comme un ensemble dans un tableur. Les données publiées sur KIRs provenant pour la plupart des populations d’origine européenne permettent la prédiction des règles LD qui existent entre différents gènes dans le complexe de KIR 1. Ces prédictions sont utilisées pour effectuer des vérifications en aval sur l’exemplaire numéro résultats obtenus (tableau 6). Les échantillons qui ne sont pas conforment à la LD prévu entre les gènes pourraient contenir polymorphisme inhabituelle ou haplotypique variations structurales. Un organigramme décrivant le protocole est illustré à la Figure 3.
Un outil appelé identificateur de Haplotype KIR (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) a été développé afin de faciliter l’imputation des haplotypes de l’ensemble de données. L’imputation fonctionne sur la base d’une liste de référence haplotypes observés dans une population d’origine européenne1. Toutefois, l’outil permet également pour un jeu personnalisé des haplotypes de référence à utiliser à la place. Trois dossiers distincts sont produisent ; le premier fichier répertorie toutes les combinaisons de haplotype pour obtenir un exemple, le deuxième fichier fournit une liste ajustée des combinaisons haplotypes disposant des plus hautes fréquences combinées et le troisième fichier répertorie les exemples qu’il ne peut pas être assignés des haplotypes. Non autorisation de cession des haplotypes pourrait servir d’indicateur des haplotypes roman.
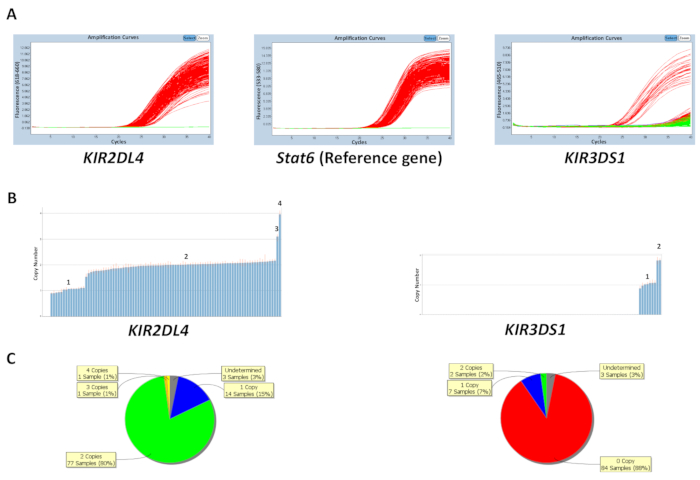
Figure 1 : résultats représentatifs d’une plaque pour la réaction numéro 5. (A), ce panneau affiche amplification parcelles. (B), ce panneau affiche copie nombre emplacements. (C), ce panneau montre la distribution de nombre de copie. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
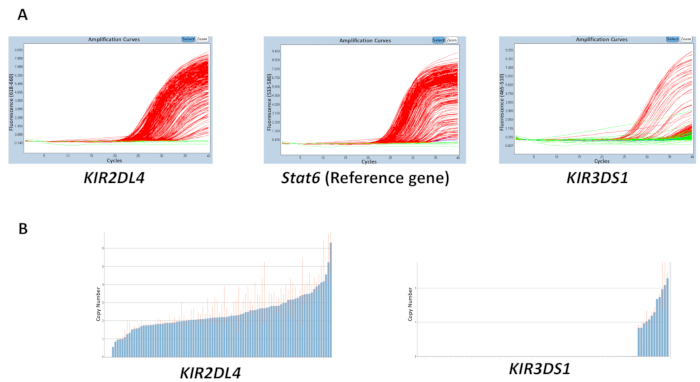
Figure 2 : résultats représentatifs d’une plaque avec une concentration d’ADN variable pour la réaction numéro 5. (A), ce panneau affiche amplification parcelles. (B), ce panneau affiche copie nombre emplacements. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
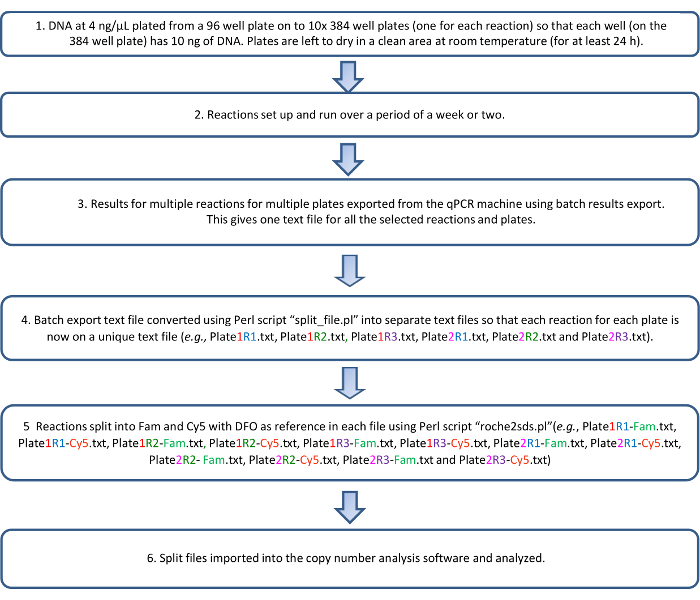
Figure 3 : diagramme de flux du protocole qKAT. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Test | Gènes | Amorces avant | Concentration (nM) | Inverser les amorces | Concentration (nM) | Sondes | Concentration (nM) |
| N ° 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2DL 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 3 | 3DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 4 | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 5 | 3DS1 | B2F | 250 | B1R | 250 | P4b | 150 |
| 2DL 4 | C1F | 200 | C1R | 200 | P5B - 2DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 6 | 2DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| 2DL 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 8 | 2DS3 | B5F | 250 | B5R | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | D5R | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| N° 10 | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
Tableau 1 : combinaison et concentration des amorces et des sondes utilisées dans chaque réaction de qKAT 27 .
| Réaction | Apprêt aliquotes (µL) | Sonde aliquotes (µL) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | EAU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | EAU | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| Remarque : besoin 20 µL moins d’eau dans le MasterMix | |||||||||||
| R3 | 3DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | EAU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | EAU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | EAU | STAT6F | STAT6R | P4B | P5B - 2L 4 | PSTAT6 |
| 2DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | EAU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | EAU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | EAU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | EAU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | EAU | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
Tableau 2 : Volumes (µL) de 100 µM amorce/sonde des solutions mères de faire primer et sonder les aliquotes de combinaison.
| Nom | Direction | modification de 5´ | modification de 3´ | Séquence (5' →3') | Longueur | TM | % GC | Exon | Position |
| P4a | Sens | FAM | BHQ-1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44,4 | 4 | 425-451 |
| P4b | Antisens | FAM | BHQ-1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5B | Sens | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828-852 |
| P5B - 2DL 4 | Sens | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828-852 |
| P9 | Sens | ATTO647N | BHQ-2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62,5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ-2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
Tableau 3 : liste des sondes utilisées en qKAT 1, 27. les colorants fluorescents utilisés à l’extrémité 5' des sondes oligo P5b, P5b - 2 DL 4, P9 et PSTAT6 ont été modifiés aux colorants ATTO.
| Gène | Amorces | Direction | Séquence (5´-3´) | Longueur | TM | % GC | Exon | Position | Amplicon (bp) | Allèles peuvent être manqués | ||
| 3DL2e4 | A1F | Vers l’avant | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 2 * 008, * 021, * 027, * 038. | ||
| A1R | Marche arrière | CTGCAAGGACAG GCATCAA | 19 | 53 | 52,6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | Vers l’avant | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 112 | Aucun | ||
| A5R | Marche arrière | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55,6 | 492-509 | Aucun | |||||
| 2DS2 | A2F | Vers l’avant | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 111 | Aucun | ||
| A6R | Marche arrière | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42,9 | 488-508 | Aucun | |||||
| 3DL 3 | A8Fa | Vers l’avant | GTGAAATCGGGA GAGACG | 18 | 50 | 55,6 | 4 | 406-423 | 139 | Aucun | ||
| A8Fb | Vers l’avant | GGTGAAATCAGG AGAGACG | 19 | 50 | 52,6 | 405-423 | 3DL 3 * 054, 3DL 3 * 00905. | |||||
| A8R | Marche arrière | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | Aucun | |||||
| 3DL1e4 | B1F | Vers l’avant | CATCGGTCCCAT GATGCT | 18 | 51 | 55,6 | 4 | 549-566 | 85 | 3DL 1 * 00505, 3DL 1 * 006, 3DL 1 * 054, 3DL 1 * 086, 3DL 1 * 089 | ||
| B1R | Marche arrière | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | Vers l’avant | CATCGGTTCCAT GATGCG | 18 | 51 | 55,6 | 4 | 549-566 | 85 | 3DS1 * 047 ; peuvent se procurer 3DL 1 * 054. | ||
| B1R | Marche arrière | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | Aucun | |||||
| 2DL 1 | B3F | Vers l’avant | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | 2DL 1 * 020, 2DL 1 * 028 | ||
| B3R | Marche arrière | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | 2DL 1 * 023, 2DL 1 * 029, 2DL 1 * 030 | |||||
| 2DS1 | B4F | Vers l’avant | TCTCCATCAGTC GCATGAA | 19 | 51 | 47,4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | Marche arrière | GGTCACTGGGAG CTGAC | 17 | 49 | 64,7 | 624-640 | Aucun | |||||
| 2DS3 | B5F | Vers l’avant | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546-563 | 96 | Aucun | ||
| B5R | Marche arrière | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | Aucun | |||||
| 2DS5 | B6F2 | Vers l’avant | AGAGAGGGGACG TTTAACC | 19 | 50 | 52,6 | 4 | 475-493 | 173 | Aucun | ||
| B6R3 | Marche arrière | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66,7 | 630-647 | 2DS5 * 003 | |||||
| 2DL 4 | C1F | Vers l’avant | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55,6 | 5 | 808-825 | 83 | Aucun | ||
| C1R | Marche arrière | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52,6 | 872-890 | 2DL 4 * 018, 2DL 4 * 019 | |||||
| 2DL 2 | 2DL2F4 | Vers l’avant | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57,9 | 5 | 778-796 | 151 | 2DL 2 * 009 ; 782G changé à A. | ||
| C3R2 | Marche arrière | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | Aucun | |||||
| 2DS4 | C5F | Vers l’avant | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | Aucun | ||
| C5R | Marche arrière | TTGACCACTCGT AGGGAGC | 19 | 52 | 57,9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | Vers l’avant | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57,9 | 5 | 750-768 | 203 | Aucun | ||
| 2DS4R2 | Marche arrière | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Aucun | |||||
| 2DS4FL | 2DS4FL | Vers l’avant | CCGGAGCTCCTA TGACATG | 19 | 53 | 57,9 | 5 | 744-762 | 209 | Aucun | ||
| 2DS4R2 | Marche arrière | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Aucun | |||||
| 2DL 3 | D1F | Vers l’avant | AGACCCTCAGGA GGTGA | 17 | 48 | 58,8 | 9 | 1180-1196 | 156 | Aucun | ||
| D1R | Marche arrière | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010, 2DL 3 * 017, 2DL 3 * 01801 et 2DL 3 * 01802 | |||||
| 2DL 5 | D2F | Vers l’avant | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 et 2DL5B * 020 | ||
| D2R | Marche arrière | GGCAGGAGACAA TGATCTT | 19 | 49 | 47,4 | 1315-1333 | Aucun | |||||
| 2DP1 | D3F | Vers l’avant | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | Aucun | ||
| D3R | Marche arrière | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | Aucun | |||||
| 3DL1e9 | D4F | Vers l’avant | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 93 | 3DL 1 * 061, 3DL 1 * 068 | ||
| D4R2 | Marche arrière | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43,5 | 1273-1295 | 3DL 1 * 05901, 3DL 1 * 05902, 3DL 1 * 060, 3DL 1 * 061, 3DL 1 * 064, 3DL 1 * 065, 3DL 1 * 094N, 3DL 1 * 098 | |||||
| 3DL2e9 | D4F | Vers l’avant | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 156 | Aucun | ||
| D5R | Marche arrière | GACCTGACTGTG GTGCTCG | 19 | 54 | 63,2 | 1340-1358 | Aucun | |||||
| STAT6 | STAT6F | Vers l’avant | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129 | |||||
| STAT6R | Marche arrière | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
Tableau 4 : les séquences des amorces utilisées dans qKAT 1, 27.
| KIR gène | 3DL 3 | 2DS2 | 2DL 2 | 2DL 3 | 2DP1 | 2DL 1 | 3DP1 | 2DL 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | 2DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 Total | 2DS4 FL | 2DS4 DEL | 3DL 2 EX4 | 3DL 2 EX9 | |
| Nombre de copies plus fréquent | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
Tableau 5 : Nombre de copies plus fréquent pour KIR gènes couramment observés dans les échantillons d’origine européenne.
| Règles de déséquilibre d’assemblage pour qKAT issu des populations européennes | Vérification de numéro de copie | |||||||
| 1 | KIR3DL3, KIR3DP1, KIR2DL4 et KIR3DL2 sont des gènes de cadre présents sur les deux haplotypes. | KIR3DL3, KIR3DP1, KIR2DL4 et KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 et KIR2DL2 sont en LD entre eux | 2DS2=2 DL 2 | ||||||
| 3 | KIR2DL2 et KIR2DL3 sont des allèles du même gène | 2 DL 2+2 DL 3= 2 | ||||||
| 4 | KIR2DP1 et KIR2DL1 sont en LD entre eux | 2DP1=2 DL 1 | ||||||
| 5 | Exon 4 du KIR3DL1 et KIR3DL2 est égal à exon 9 de KIR3DL1 et KIR3DL2 respectivement. | 3DL1ex4=3DL1ex9 et 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 et KIR3DS1 sont des allèles | 3 DL 1+3DS1= 2 | ||||||
| 7 | KIR2DS3 et KIR2DS5 sont en LD avec KIR2DL5 | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1 et KIR2DS1 sont en LD | 3DS1=2DS1 | ||||||
| 9 | Présence d’otal KIR2DS1 et KIR2DS4Test mutuellement exclusive sur un haplotype | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL et KIR2DS4del sont des variantes du KIR2DS4TOTAL | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
Tableau 6 : déséquilibre de liaison entre KIR gènes couramment observés dans les populations d’origine européenne peuvent être utilisés pour vérifier les données de numéros de copie 1,27.
Discussion
Nous avons décrit une nouvelle méthode semi-automatisée haut-débit, appelée qKAT, ce qui facilite la copie numéro typage des gènes KIR . La méthode est une amélioration par rapport aux méthodes conventionnelles comme fournisseur de services partagés-PCR, qui sont de faible débit et peut seulement indiquer la présence ou l’absence de ces gènes très polymorphes.
L’exactitude des données numéros copie obtenues dépend de multiples facteurs, y compris la qualité et la concentration-homogénéité des échantillons Adng et la qualité des réactifs. La qualité et la précision des échantillons Adng à travers une plaque sont extrêmement importantes car les variations de concentration à travers la plaque peuvent entraîner des erreurs dans le calcul du nombre de copie. Étant donné que les tests ont été validés à l’aide des ensembles d’échantillons origine européenne, données de cohortes provenant d’autres parties du monde exigent des vérifications plus approfondies. Il s’agit de faire en sorte que les instances de décrochage de l’allèle ou liaison non-spécifique apprêt/sonde ne sont pas interprété à tort comme variation numéro de copie.
Alors que les tests ont été conçus et optimisés pour exécuter en tant que haut-débit, ils peuvent être modifiés pour exécuter moins d’échantillons. La métrique de confiance dans le logiciel d’analyse numéro de copie est affectée lors de l’analyse des échantillons de moins, mais cela peut être améliorée si les échantillons d’ADN génomiques contrôle avec un nombre connu de la copie du gène KIR figurent sur la plaque et répétitions d’échantillonnage supplémentaires sont inclus.
Pour les laboratoires sans liquide/plaque-manutention robots, mélange principal peut être dispensée à l’aide de pipettes multicanaux et plaques peuvent être chargés manuellement dans l’instrument de qPCR.
L’objectif principal derrière le développement de qKAT devait créer une méthode simple, de haut débit, haute résolution et rentable au génotype KIRs pour maladie études d’association. Ceci a été réalisé avec succès puisque qKAT a été employé dans l’enquête sur le rôle de KIR dans plusieurs études d’association de grandes maladies, y compris une gamme de maladies infectieuses, maladies auto-immunes et grossesse troubles4, 24 , 25 , 26.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le projet a reçu des fonds du Medical Research Council (MRC), le Conseil européen de la recherche (CER) en vertu du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (subvention, contrat n° 695551) et le National Institute of Health (NIH) de Cambridge Biomedical Research Centre et NIH recherche sang et Transplant Research Unit (NIHR BTRU) dans le don et la Transplantation de l’Université de Cambridge et en partenariat avec le sang de la NHS et transplantation (NHSBT). Les opinions exprimées sont celles des auteurs et pas nécessairement celles de la NHS, le NIHR, le ministère de la santé ou le NHSBT.
matériels
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
Références
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon