Method Article
qKAT: количественные Semi-automated типирование убийца клетки генов иммуноглобулинов подобных рецепторов
В этой статье
Резюме
Количественные клеток-киллеров иммуноглобулина подобный рецептор (KIR) полуавтоматические ввод (qKAT) является простой, высокой пропускной способности и экономически метод, чтобы скопировать номер типа Кир генов для их применения в ассоциации исследований населения и болезней.
Аннотация
Иммуноглобулин подобные рецепторы клеток-киллеров (Кирс) представляют собой набор тормозящее и активация иммунных рецепторов, на природные убийца (НК) и Т-клеток, кодируемых кластер полиморфных генов на хромосоме 19. Их наиболее характерны лигандов являются человеческого лейкоцитарного антигена (HLA) молекулы, которые кодируются в комплекс (MHC) локуса гистосовместимости на хромосоме 6. Есть существенные доказательства того, что они играют значительную роль в иммунитет, воспроизводства и трансплантации, что делает его крайне важно иметь методы, которые можно точно генотип их. Однако, высокий последовательности гомологии, а также аллельных и копии номер изменения, затрудняют методов проектирования, которые можно точно и эффективно генотип все гены Кир . Традиционные методы обычно ограничены в резолюции полученных данных, пропускная способность, эффективности затрат и времени для настройки и выполнения экспериментов. Мы описываем метод, называемый количественных Кир полуавтоматические ввода (qKAT), который является высок объём Мультиплекс в реальном времени полимеразной цепной реакции метод, который можно определить номера копии гена для всех генов в локусе Кир . qKAT представляет собой простой метод высокой пропускной способности, которая может обеспечить высоким разрешением Кир копирования числовые данные, которые могут в дальнейшем использоваться для выведения вариации в структурно полиморфных гаплотипов, что охватить их. Эта копия номер и гаплотип данные могут быть полезными для исследований на крупномасштабных болезни ассоциаций, популяционной генетики, а также расследований на выражение и функционального взаимодействия между Кир и HLA.
Введение
В организме человека, убийца рецептора иммуноглобулина как(Кир) локус сопоставляется на длинном плече хромосомы 19 в рамках комплекс рецептор лейкоцитов (LRC). Этот локус в длину около 150 КБ и включает в себя 15 Кир гены расположены головы до хвоста. Кир локусов, которые известны в настоящее время являются KIR2DL1, KIR2DL2/KIR2DL3, KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1-5, KIR3DL1/KIR3DS1, KIR3DL2-3, и два pseudogenes, KIR2DP1 и KIR3DP1. Кир гены кодировать для двумерного (2D) и трехмерных (3D) домен иммуноглобулина подобных рецепторов с короткими (S; активации) или длинным (L; тормозящее) цитоплазматических хвосты, которые выражаются в естественных киллеров (НК) и подмножество T клетки. Номер варианта копирования выставлены в Кир локуса формы различных гаплотипов с переменной ген содержание1. Non аллельные гомологичная рекомбинация (НАХР), способствовало тесное голова хвост гена договоренности и высокой последовательности гомологии, является механизм, предложенный отвечает за haplotypic изменчивости. Более 100 различных гаплотипов были зарегистрированы в населения во всем мире1,2,3,4. Все эти гаплотипов можно разделить на две основные группы: A и B гаплотипов. Гаплотип A содержит 7 Кир генов: KIR3DL3, KIR2DL1, KIR2DL3, KIR2DL4, KIR3DL1и KIR3DL2, которые ингибирующее Кир генов и активации Кир гена KIR2DS4. Однако до 70% лиц европейского происхождения, которые гомозиготных для Кир гаплотип А исключительно несут нефункциональные «удаление» форма KIR2DS45,6. Все другие комбинации генов Кир формируют группы B гаплотипов, включая по крайней мере один из конкретных генов Кир , KIR2DS1, KIR2DS2, KIR2DS3, KIR2DS5, KIR3DS1, KIR2DL2, и KIR2DL5и обычно включают в себя два или более Активация Кир генов.
HLA класса I молекулы были названы лигандов для определенных рецепторов ингибирующих (KIR2DL1, KIR2DL2, KIR2DL3и KIR3DL1), активируя рецепторы (KIR2DS1, KIR2DS2, KIR2DS4, KIR2DS5и KIR3DS1), и для KIR2DL4, который является уникальный Кир , который содержит долго цитоплазматических хвосты, как других рецепторов ингибирующих Кир, но также имеет положительно заряженных остатков вблизи внеклеточного домена, которая является общей особенность других активирующий рецепторы Кир . Сочетание вариантов внутри Кир генов и HLA генов влияет взаимодействия лигандов рецепторов потенциал клеток отзывчивость НК что фигуры на индивидуальный уровень7,-8. Доказательства из исследований генетических ассоциации указал, что Кир играет определенную роль в вирусной устойчивости (например,., вирус иммунодефицита человека [ВИЧ]9 и гепатит C вирус [гепатита с]10), успех трансплантации 11, риск беременности расстройств и репродуктивный успех12,13, защиты от рецидивов после аллогенной гемопоэтических стволовых клеток трансплантации (ТГСК)14,15, 16и риск рака17.
Сочетание высокой последовательности гомологии и аллельные и haplotypic разнообразие представляет проблемы в задаче точно генотипирования Кир генов. Обычные методы типа Кир генов включают последовательности специфического праймера (SSP) полимеразной цепной реакции (ПЦР)18,19,20, специфичные для последовательности олигонуклеотида зонд PCR (SSOP)21, и Матрица помощь лазерной десорбции ионизации время полета масс-спектрометрия (MALDI-TOF MS)22. Недостатки этих методов являются, что они только обеспечивают частичное понимание генотип человека, а также трудоемкий для выполнения. Недавно секвенирование нового поколения (НГС) был применен к типу Кир Локус специально. Хотя этот метод является очень мощным, он может быть дорогим для запуска, и это требует много времени для проведения углубленного анализа и данных проверок.
qKAT является методом высок объём количественного PCR. В то время как обычные методы трудоемким и длительным, этот метод позволяет запускать почти 1000 образцов геномной ДНК (геномная ДНК) в течение пяти дней и дает Кир генотипа, а также номер копии гена. qKAT состоит из десяти мультиплекс реакций, каждая из которых ориентирована на две Кир локусов и один ген ссылки Фиксированная копия числа в геноме (STAT6) используется для относительной квантификация гена Кир скопировать номер23. Этот assay успешно используется в исследованиях с участием большого числа панелей и когорты болезни на инфекционные заболевания, как гепатит с, аутоиммунные заболевания, как диабета типа 1 и беременность расстройств, таких как преэклампсия, а также предоставление генетических основу для исследований, направленных на понимание NK клеток функция1,4,24,25,26.
протокол
1. Подготовка и обшивка из ДНК
- Точно подсчитать концентрация геномная ДНК с помощью спектрофотометрические или флуориметрический инструмента.
- Разбавляют ДНК до 4 нг/мкл на плиту 96-луночных глубокой скважины. Включать, по крайней мере один образец управления геномная ДНК с числом известных копии и один элемент управления-шаблон.
- Центрифуга 96-луночных пластины на 450 x g на 2 мин.
- С помощью инструмента обработки жидких, обойтись каждый образец в составленном на 384-ну ПЦР плиты так, что каждый хорошо имеет 10 нг ДНК (2,5 мкл/хорошо). Подготовка по меньшей мере десять 384-ну пластины, один для каждой qKAT реакции.
- Если геномная ДНК отказаться от более чем одной 96-луночных пластины, выполните полный объем промыть 2% отбеливателя и ультрачистая вода для очистки иглы жидкости системы между каждой пластины 96-луночных геномная ДНК образцов обработки.
- Просушите ДНК, инкубации и 384-ну пластины в чистой зоне при комнатной температуре в течение 24 часов.
2. Подготовка грунтовки и зонды
Примечание: qKAT состоит из десяти мультиплекс реакций. Каждая реакция включает в себя три пары праймера и три флуоресцировани обозначенного зондами, которые специально усиливают два Кир генов и ссылку на один ген. Датчики, которые были опубликованы в Цзян et al.27 были изменены таким образом, чтобы олигонуклеотиды теперь помечены Атто красителями, так как они предлагают улучшенный светостойкость и длинный сигнал продолжительностей жизни. Pre-aliquoted грунтовка комбинации являются коммерчески доступных (см. Таблицу материалы).
- Подготовьте грунт комбинации для каждой реакции согласно разведений, приведенные в таблице 1.
- Подготовьте зонд комбинации для каждой реакции согласно таблице 1. Каждый индивидуальный пробник до принятия комбинации.
3. Подготовка Мастер микс
Примечание Томов, упомянутых ниже, для выполнения одного qKAT реакция на наборе 10 x 384-ну пластин.
- Убедитесь, что геномная ДНК образцов покрытием на тарелках 384-ну абсолютно сухие. Провести все шаги на льду и держать реагентов, распространяется от воздействия света как можно больше, поскольку флуоресцировани обозначенного зонды чувствительны к фото - и термо.
- Размораживание буфер ПЦР, грунтовка и зонд аликвоты при 4 ° C.
- На льду Подготовьте мастер смесь для 10 x 384-ну пластин, добавив 18,86 мл ультрачистая вода, 20 мл ПЦР буфер, 1000 мкл вкладываемое грунтовка комбинации и 180 мкл вкладываемое зонд комбинации (Таблица 2).
- Равномерно распределяет Мастер микс по 96-глубокие хорошо пластину, используя многоканальные пипетки, закупорить 415 мкл в каждой скважине. Держите эту пластину в поле льда распространяется от света.
- С помощью инструмента обработки жидких, лунки 9,5 мкл мастер смесь в каждой скважине 384-ну пластины с сушеной геномная ДНК. Печать пластину с фольгой и сразу же поместить его на 4 ° C. Повторите этот процесс для оставшихся пластины, обеспечивая, что иглы жидкости системы обработки промывают водой между каждой плиты.
- Центрифуга 384-ну пластины на 450 x g 3 мин и инкубировать их в 4 ° C на ночь или между 6-12 ч до Ресуспензируйте ДНК и рассеивать любые воздушные пузыри.
4. ПЦР анализа
- После ночи инкубации центрифуги на 450 x g 3 мин рассеивать любые оставшиеся пузырьков воздуха.
- Для целей автоматизации, подключите машину ПЦР (например, LightCycler 480) Гонав обработчик (см. Таблицу материалы). Программа обработчик Гонав размещать пластины в машину ПЦР с охлаждением хранения док, который защищен от света.
Примечание анализов должны в теории, работать на других машинах ПЦР с совместимых оптических параметров. - Используйте следующие условия Велоспорт: 95 ° C за 5 мин, после чего 40 циклов 95 ° c 15 s и 66 ° C 50 сек, сбора данных на 66 ° C.
- После завершения выполнения, у робота собрать пластину от машины ПЦР и поместите его в док отменить.
5. После выполнения анализа
- После усиления, вычислить количественная оценка цикла (ов) с помощью второй производной максимальная метода или метода определяющих точек с помощью программного обеспечения машины ПЦР (см. Таблицу материалы), следуя инструкциям ниже.
- Откройте программное обеспечение ПЦР, на вкладке " Навигатор ", файл и открыть сохраненный реакции эксперимент для одной пластины.
-
Для анализа с использованием метода второй производной максимальная выберите вкладку анализ и создать новый анализ с помощью метода Abs Quant/Second производная Макс.
- В окне Создание нового анализа , выберите тип анализа: метод Abs Quant/Second производная Макс, подмножества: все образцы, программа: усиление, имя: Rx-ДФО (где x является номером реакции).
- Выберите Фильтр расческу и выберите Вик/HEX/Yellow555 (533-580). Это гарантирует, что данные, собранные для STAT6 установлен.
- Выберите Цвет компенсации для VIC/HEX/Yellow555(533-580). Нажмите кнопку вычислить. Повторите это действие для Fam (465-510) и Cy5/Cy5.5(618-660). Нажмите кнопку сохранить файл.
-
Для анализа с использованием метода определяющих точек выберите Abs Квант/Fit точки на вкладке анализ.
- В окне Создание нового анализа , выберите тип анализа: метод Abs Квант/определяющих точек, подмножества: все образцы, программа: усилители, имя: RxF-ДФО (где x является номером реакции).
- Выберите правильные фильтры и цвет компенсации для STAT6 и каждый из генов Кир (Fam/Cy5). В Noiseband на вкладке установите шума группу, чтобы исключить фоновый шум.
- На вкладке анализ равным 3 определяющих точек и выберите Показать определяющих точек. Нажмите кнопку вычислить. Нажмите кнопку сохранить файл.
6. Экспорт результатов
- В программном обеспечении ПЦР, откройте Навигатор tab. Выберите Экспорт пакетов результаты.
- Откройте папку, в которой эксперимент файлы сохраняются и передачи файлов в правой части окна. Нажмите кнопку Далее. Выберите имя и расположение файла экспорта.
- Выберите тип анализа, метода Abs Quant/Second производная Макс или Abs Квант/определяющие точки. Нажмите кнопку Далее. Проверьте, что имя файла, папку экспорта и тип анализа являются правильными и нажмите кнопку следующий чтобы начать процесс экспорта.
- Подождите, пока Состояние экспорта ОК. Экран автоматически перейдет к следующему шагу. Проверьте, что все выбранные файлы были успешно экспортированы тем, что количество файлов не = 0. Нажмите кнопку Готово.
- Использовать скрипты split_file.pl и roche2sds.pl разделить экспортируемый пластины на индивидуальных реакций для каждой пластины.
Примечание сценарии предоставляются по запросу/GitHub.
7. Скопируйте число вычислений
- Открываете копию номером анализ программного обеспечения (например, CopyCaller). Выберите файл результатов в реальном времени PCR импорта и загрузить текстовые файлы, созданные roche2sds.pl.
- Выберите анализировать и проводить анализ, либо выбрав образец калибратор с числом известных копии или выбрав наиболее частого копирования номер. Смотрите в таблице 5 для наиболее частых копии количество генов Кир , обычно наблюдается в населения европейского происхождения.
8. качество данных проверяет
- Используйте сценарий R KIR_CNVdata_analysis_for_Excel_ver020215. R объединить копирования данных из всех плит в электронную таблицу.
Примечание сценарии предоставляются по запросу/GitHub. - Проверьте исходные данные на копию номером анализ программного обеспечения для образцов, которые не соответствуют известных связей неравновесия (LD) для Кир генов (Таблица 6).
Результаты
Анализ копировать номера может осуществляться путем экспорта файлов для копирования номер анализ программного обеспечения, которое предоставляет номер предсказал и оценкам копирования, основанный на методе ΔΔCq.
Номер копии может быть предсказано, либо на основе известных копии количество контрольных образцов ДНК на плите или путем ввода наиболее часто номер копии гена (Таблица 5). Рисунок 1 показывает результаты пластины для реакции, предназначенный для KIR2DL4 и KIR3DS1, а также ссылка гена STAT6. Наиболее частые копии номер для KIR2DL4, рамки гена в Кир локус, — две копии, тогда как наиболее часто копии номер для KIR3DS1, активируя гена, одна копия. Результаты на рисунке показывают участки амплификации PCR, наблюдается на ПЦР программного обеспечения и копии данных создается из данных ПЦР. Как показано, assay возможность различать 0, 1, 2, 3 и 4 номеров копии гена Кир . Копия числовой анализ программного обеспечения также позволяет Просмотр распределения числа копирования через пластину как круговую диаграмму или гистограмму. Эффективность копирования номер предсказание ниже для образцов с более высоким номером копии.
Качество всех материалов, используемых в реакции, геномная ДНК, буфер, грунтовки и зонды, может повлиять на точность полученных результатов. Однако скорее всего, вызвана из-за различия в концентрации ДНК через тарелку несоответствие в результатах. Чистота извлеченные геномная ДНК, которая может быть измерена с помощью 260/280 и 260/230 коэффициенты, также может иметь влияние на качество. 260/280 отношение коэффициента 1,8-2 и 260/230 2-2,2 желательны. Неравномерным диапазон ДНК концентрации через пластины может привести к высокой изменчивости в круговороте порога (tC) между образцов и отсутствие согласованности в диапазоне сметных копия числа. Результаты на рисунке 2 показывают эффект, что может иметь расхождение между значениямиt C через тарелку на точность прогнозирования числа копирования. Красная линия указывает диапазон оценкам копии номер для образца и, в идеале, должно быть как можно ближе к целое как можно скорее.
Копирование данных, после проанализированы могут быть экспортированы в файл электронной таблицы в формате 96-луночных. Мы использовали сценарий R (доступно по запросу) для объединения копирование данных всех 10 пластин, которые запускаются как набор в одну таблицу. Опубликованные данные о Кирс основном европейского происхождения населения позволяет предсказания LD правил, которые существуют между различными генов в Кир комплекс1. Эти прогнозы используются для проверки ниже по течению на копии число результатов (Таблица 6). Образцы, которые не соответствуют прогнозируемым LD между гены могут содержать необычных полиморфизм или haplotypic структурных изменений. Блок-схемы, описывающие протокол показан на рисунке 3.
Инструмент называется Кир гаплотип идентификатор (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) была разработана для облегчения вменение гаплотипов из набора данных. Вменением работает на основе перечня справочных гаплотипов наблюдается в населения европейского происхождения1. Однако инструмент также позволяет для набора справочных гаплотипов использоваться вместо. Создаются три отдельных файлов; Первый файл содержит список всех комбинаций гаплотип для образца, второй файл предоставляет список обрезается гаплотипов комбинации, которые имеют высокие комбинированные частоты, и третий файл список образцов, которые не могут быть назначены гаплотипов. Non назначение гаплотипов могут использоваться в качестве показателя Роман гаплотипов.
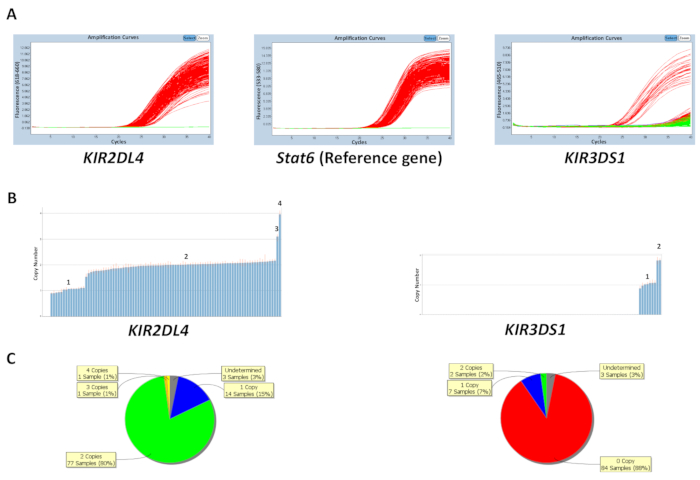
Рисунок 1: представитель результаты пластины для реакции номер 5. (A) это усиление шоу группа участков. (B) Эта панель показывает скопировать несколько участков. (C) Эта группа показывает номер распределения копий. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
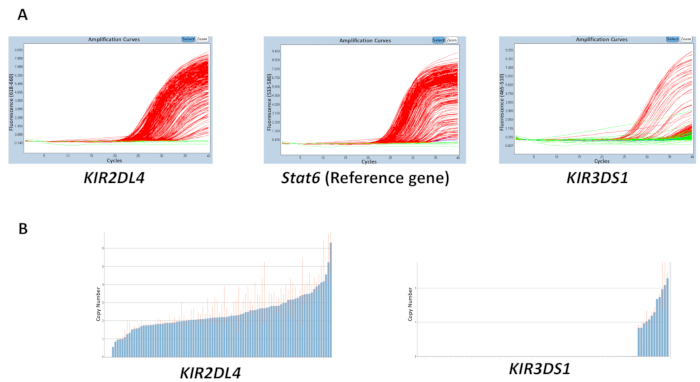
Рисунок 2: представитель результаты плиты с переменной концентрации ДНК для реакции номер 5. (A) это усиление шоу группа участков. (B) Эта панель показывает скопировать несколько участков. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
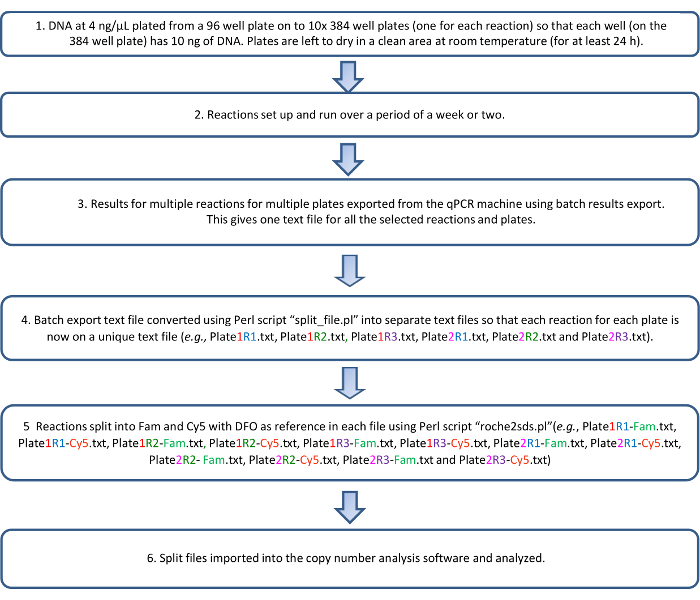
Рисунок 3: блок-схемы протокола qKAT. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
| Проба | Гены | Форвард праймеры | Концентрация (Нм) | Обратный праймеры | Концентрация (Нм) | Зонды | Концентрация (Нм) |
| No 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2DL 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 3 | 3DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 4 | 3DL1e4 | B1F | 250 | B1R | 125 | Expal P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 5 | 3DS1 | B2F | 250 | B1R | 250 | Expal P4b | 150 |
| 2 ДЛ 4 | C1F | 200 | C1R | 200 | P5b - 2DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 6 | 2DL 1 | B3F | 500 | B3R | 125 | Expal P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 7 | 2DS1 | B4F | 500 | B4R | 250 | Expal P4b | 150 |
| 2DL 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 8 | 2DS3 | B5F | 250 | B5R | 250 | Expal P4b | 150 |
| 3DL2e9 | D4F | 250 | D5R | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 10 | 2DS5 | B6F2 | 200 | B6R3 | 200 | Expal P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
Таблица 1: сочетание и концентрации грунтовки и датчики, используемые в каждой реакции qKAT 27 .
| Реакция | Грунтовка аликвоты (мкл) | Зонд аликвоты (мкл) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | ВОДЫ | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | ВОДЫ | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| Примечание: требуется 20 мкл меньше воды в MasterMix | |||||||||||
| R3 | 3DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | ВОДЫ | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P5B - 2L 4 | PSTAT6 |
| 2 ДЛ 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P9 | PSTAT6 |
| 2DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | ВОДЫ | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | ВОДЫ | STAT6F | STAT6R | EXPAL P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
Таблица 2: Тома (мкл) 100 мкм праймер/зонд складе решения сделать грунт и зонд аликвоты комбинации.
| Имя | Направление | модификация 5´ | модификация 3´ | Последовательность (5' →3') | Длина | TM | GC % | Эксон | Позиция |
| P4a | Чувство | FAM | BHQ-1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44,4 | 4 | 425-451 |
| Expal P4b | Antisense | FAM | BHQ-1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5b | Чувство | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828-852 |
| P5b - 2DL 4 | Чувство | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828-852 |
| P9 | Чувство | ATTO647N | BHQ-2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62,5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ-2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
Таблица 3: список датчиков, используемых в qKAT 1, 27. люминесцентные красители, используемые в конце 5' oligo зонды P5b, P5b - 2 DL 4, P9 и PSTAT6 были изменены для Атто красители.
| Джин | Праймеры | Направление | Последовательность (5´-3´) | Длина | TM | GC % | Эксон | Позиция | Ампликон (bp) | Может быть упущена аллели | ||
| 3DL2e4 | A1F | Вперед | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 2 * 008, * 021, * 027, * 038. | ||
| A1R | Реверс | CTGCAAGGACAG GCATCAA | 19 | 53 | 52,6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | Вперед | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 112 | Нет | ||
| A5R | Реверс | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55,6 | 492-509 | Нет | |||||
| 2DS2 | A2F | Вперед | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52,6 | 4 | 398-416 | 111 | Нет | ||
| A6R | Реверс | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42,9 | 488-508 | Нет | |||||
| 3DL 3 | A8Fa | Вперед | GTGAAATCGGGA GAGACG | 18 | 50 | 55,6 | 4 | 406-423 | 139 | Нет | ||
| A8Fb | Вперед | GGTGAAATCAGG AGAGACG | 19 | 50 | 52,6 | 405-423 | 3DL 3 * 054, 3DL 3 * 00905. | |||||
| A8R | Реверс | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | Нет | |||||
| 3DL1e4 | B1F | Вперед | CATCGGTCCCAT GATGCT | 18 | 51 | 55,6 | 4 | 549-566 | 85 | 3DL 1 * 00505, 3DL 1 * 006, 3DL 1 * 054, 3DL 1 * 086, 3DL 1 * 089 | ||
| B1R | Реверс | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | Вперед | CATCGGTTCCAT GATGCG | 18 | 51 | 55,6 | 4 | 549-566 | 85 | 3DS1 * 047; могут забрать 3DL 1 * 054. | ||
| B1R | Реверс | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | Нет | |||||
| 2DL 1 | B3F | Вперед | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | 2DL 1 * 020, 2DL 1 * 028 | ||
| B3R | Реверс | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | 2DL 1 * 023, 2DL 1 * 029, 2DL 1 * 030 | |||||
| 2DS1 | B4F | Вперед | TCTCCATCAGTC GCATGAA | 19 | 51 | 47,4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | Реверс | GGTCACTGGGAG CTGAC | 17 | 49 | 64,7 | 624-640 | Нет | |||||
| 2DS3 | B5F | Вперед | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546-563 | 96 | Нет | ||
| B5R | Реверс | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | Нет | |||||
| 2DS5 | B6F2 | Вперед | AGAGAGGGGACG TTTAACC | 19 | 50 | 52,6 | 4 | 475-493 | 173 | Нет | ||
| B6R3 | Реверс | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66,7 | 630-647 | 2DS5 * 003 | |||||
| 2 ДЛ 4 | C1F | Вперед | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55,6 | 5 | 808-825 | 83 | Нет | ||
| C1R | Реверс | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52,6 | 872-890 | 2 ДЛ 4 * 018, 2DL 4 * 019 | |||||
| 2DL 2 | 2DL2F4 | Вперед | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57,9 | 5 | 778-796 | 151 | 2DL 2 * 009; 782G изменено на а. | ||
| C3R2 | Реверс | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | Нет | |||||
| 2DS4 | C5F | Вперед | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | Нет | ||
| C5R | Реверс | TTGACCACTCGT AGGGAGC | 19 | 52 | 57,9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | Вперед | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57,9 | 5 | 750-768 | 203 | Нет | ||
| 2DS4R2 | Реверс | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Нет | |||||
| 2DS4FL | 2DS4FL | Вперед | CCGGAGCTCCTA TGACATG | 19 | 53 | 57,9 | 5 | 744-762 | 209 | Нет | ||
| 2DS4R2 | Реверс | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Нет | |||||
| 2DL 3 | D1F | Вперед | AGACCCTCAGGA GGTGA | 17 | 48 | 58,8 | 9 | 1180-1196 | 156 | Нет | ||
| D1R | Реверс | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010, 2DL 3 * 017, 2DL 3 * 01801 и 2DL 3 * 01802 | |||||
| 2DL 5 | D2F | Вперед | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 и 2DL5B * 020 | ||
| D2R | Реверс | GGCAGGAGACAA TGATCTT | 19 | 49 | 47,4 | 1315-1333 | Нет | |||||
| 2DP1 | D3F | Вперед | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | Нет | ||
| D3R | Реверс | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | Нет | |||||
| 3DL1e9 | D4F | Вперед | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 93 | 3DL 1 * 061, 3DL 1 * 068 | ||
| D4R2 | Реверс | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43,5 | 1273-1295 | 3DL 1 * 05901, 3DL 1 * 05902, 3DL 1 * 060, 3DL 1 * 061, 3DL 1 * 064, 3DL 1 * 065, 3DL 1 * 094N, 3DL 1 * 098 | |||||
| 3DL2e9 | D4F | Вперед | CACAGTTGGATC ACTGCGT | 19 | 52 | 52,6 | 9 | 1203-1221 | 156 | Нет | ||
| D5R | Реверс | GACCTGACTGTG GTGCTCG | 19 | 54 | 63,2 | 1340-1358 | Нет | |||||
| STAT6 | STAT6F | Вперед | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129 | |||||
| STAT6R | Реверс | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
Таблица 4: последовательности праймеров, используемые в qKAT 1, 27.
| Кир Джин | 3DL 3 | 2DS2 | 2DL 2 | 2DL 3 | 2DP1 | 2DL 1 | 3DP1 | 2 ДЛ 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | 2DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 Итого | 2DS4 FL | 2DS4 DEL | 3DL 2 EX4 | 3DL 2 EX9 | |
| Наиболее частое копирование номер | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
Таблица 5: Наиболее частое копирование номер для Кир генов, обычно наблюдается в образцах европейского происхождения.
| Связь неравновесия правила для qKAT, основанные на европейских популяций | Скопируйте номер проверки | |||||||
| 1 | KIR3DL3, KIR3DP1, KIR2DL4 и KIR3DL2 являются рамки гены присутствуют на обеих гаплотипов. | KIR3DL3, KIR3DP1, KIR2DL4 и KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 и KIR2DL2 находятся в LD друг с другом | 2DS2=2 DL 2 | ||||||
| 3 | KIR2DL2 и KIR2DL3 являются аллели гена же | 2 DL 2+2 DL 3= 2 | ||||||
| 4 | KIR2DP1 и KIR2DL1 находятся в LD друг с другом | 2DP1=2 DL 1 | ||||||
| 5 | Эксон 4 KIR3DL1 и KIR3DL2 равен Эксон 9 KIR3DL1 и KIR3DL2 соответственно. | 3DL1ex4=3DL1ex9 и 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 и KIR3DS1 являются аллели | 3 DL 1+3DS1= 2 | ||||||
| 7 | KIR2DS3 и KIR2DS5 находятся в LD с KIR2DL5 | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1 и KIR2DS1 находятся в LD | 3DS1=2DS1 | ||||||
| 9 | Наличие KIR2DS1 и KIR2DS4Total является взаимоисключающим на гаплотипа | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL и KIR2DS4del являются варианты KIR2DS4TOTAL | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
Таблица 6: взаимосвязь равновесия между Кир гены, обычно наблюдается у населения европейского происхождения может использоваться для проверки копии данных 1,-27.
Обсуждение
Мы описали Роман полуавтоматические высок объём метод, называемый qKAT, который облегчает копирование номер ввода Кир генов. Этот метод является улучшением по сравнению традиционными методами как SSP-PCR, низкой пропускной способностью и может только указать наличие или отсутствие этих весьма полиморфных генов.
Точность копирования число полученных данных зависит от нескольких факторов, включая качество и концентрации единообразие геномная ДНК образцов и качество реагентов. Качество и точность геномная ДНК образцов через тарелку чрезвычайно важны для различия в концентрации через пластины может привести к ошибкам при расчете числа копирования. После анализов были проверены с использованием комплектов образцов европейского происхождения, данные из когорты из других частей мира требуют более тщательной проверки. Это необходимо для того, что экземпляры аллеля отсева или неспецифических праймер/зонд привязки не истолкованы как копирование количество вариантов.
В то время как анализы были разработаны и оптимизированы для работы в качестве высокой пропускной способности, они могут быть изменены для запуска меньшее количество образцов. Метрику уверенность в копии номер анализ программного обеспечения пострадавших при анализе образцов меньше, но это можно улучшить, если управления геномной ДНК образцов с известный номер копии гена Кир включены на плите и реплицирует дополнительные примеры включены.
Для лабораторий без жидкости/плита обработки роботы мастер смесь можно обойтись с помощью многоканальных пипеток и пластины могут загружаться вручную в ПЦР инструмент.
Основная цель за развитие qKAT было создание простой, высокой пропускной способности, с высоким разрешением и экономически метод генотип Кирс болезни ассоциации исследований. Это была успешно достигнута, поскольку qKAT был нанят в расследовании роли Кир в нескольких крупных болезни ассоциации исследований, включая широкий спектр инфекционных заболеваний, аутоиммунные условий и беременность расстройств4, 24 , 25 , 26.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Проект получил финансирование от Совета медицинских исследований (СМИ), Совет европейских исследований (ERC) под исследовательской и инновационной программы Европейского союза по Horizon 2020 (Грант соглашение № 695551) и национального института здравоохранения (НИЗ) Кембридж Биомедицинских научно-исследовательский центр и низ исследования крови и трансплантации исследовательское подразделение (NIHR BTRU) в донорства и трансплантации в Кембриджском университете и в партнерстве с NHS крови и трансплантации (NHSBT). Мнения, выраженные являются те из авторов и не обязательно ГСЗ, NIHR, Департамент здравоохранения, или NHSBT.
Материалы
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
Ссылки
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены