Method Article
qkat: 杀手细胞免疫球蛋白样受体基因的定量半自动分型
摘要
定量杀伤细胞免疫球蛋白样受体 (kir) 半自动分型 (qkat) 是一种简单、高通量、经济高效的方法, 用于复制数字型kir基因, 用于人群和疾病关联研究。
摘要
杀手细胞免疫球蛋白样受体 (kir) 是一组抑制和激活免疫受体, 对自然杀伤细胞 (nk) 和 t 细胞, 编码的多态性基因群在染色体19。他们最典型的配体是人类白细胞抗原 (hla) 分子, 这些分子被编码在6号染色体上的主要组织相容性复合体 (mhc) 位点内。有大量证据表明, 它们在免疫、生殖和移植中发挥着重要作用, 因此拥有能够准确对它们进行基因型的技术至关重要。然而, 高序列同源性, 以及等位基因和拷贝数的变化, 使得很难设计出能够准确和有效地基因型所有kir基因的方法。传统方法在获得的数据的分辨率、吞吐量、成本效益以及设置和运行实验所需的时间方面通常受到限制。我们描述了一种称为定量kir半自动分型 (qkat) 的方法, 这是一种高通量多重实时聚合酶链反应方法, 可以确定kir位点中所有基因的基因复制数。qkat 是一种简单的高吞吐量方法, 可以提供高分辨率的 kir拷贝数数据, 可进一步用于推断包含这些数据的结构多态型单倍型中的变化。这个拷贝数和单倍型数据可以有利于研究大规模疾病关联、群体遗传学, 以及调查 kir和hla之间的表达和功能相互作用。
引言
在人类中, 杀手免疫球蛋白样受体 (kir) 位点被映射在白细胞受体复合体 (lrc) 内19号染色体的长臂上。这个位点的长度约为 150 kb, 包括从尾部排列的 15个kb基因。目前已知的kir位点是kr2dl1, kir2dl2/kir2dl3, KIR2DL1,kir2dl5a, KIR2DL1, kir3dl1/kir3ds1 , KIR3DL1, 和两个伪基因, kir2dp1和kir3dp1。kir基因编码二维 (2d) 和三维 (3d) 免疫球蛋白样域受体, 具有短 (s; 激活) 或长 (l; 抑制) 细胞质尾, 由自然杀伤细胞和 t 亚群表达细胞。在 kir位点中表现出的拷贝数变异形成了基因含量可变的不同单倍型。非等位基因同源重组 (nahr), 由紧密的头尾基因排列和高序列同源性促成, 是提出的负责单倍型变异的机制。据报道, 全世界1、2、3、4的人群中有100多个不同的单倍型。所有这些单倍型可分为两大组: a 和 b 单倍型。a 单倍型包含 7个kir基因: kir3dl3、 kir2dl1、 KIR2DL1、 kr2dl4、KIR3DL3和KIR3DL3, 它们是抑制kir 基因和激活 kir 的基因基因 kir2ds4。然而, 多达70% 的欧洲起源的个体谁是纯合为 kir 单倍型 a 完全携带一个非功能的 "删除" 形式的kir2ds45,6。所有其他kir基因组合形成 b 组单倍型, 包括至少一个特定的kir基因kir2ds1, kir2ds2, kir2ds3, kr2ds5, kir3ds1, kir2dl2, 和kir2dl5, 通常包括两个或多个激活kir基因。
hla i 类分子已被确定为某些抑制受体 (kir2dl1、 KIR2DL1、 kr2dl3和kir3dl1) 的配体, 激活受体 (kr2ds1、 KIR2DL1、KIR2DL1、 kir2ds5和kl3ds1), 以及kir2dl4,这是一个独特的kir , 包含一个长的细胞质尾巴, 像其他抑制 kir 受体, 但也有一个带正电的残留附近的细胞外域, 这是一个常见的其他激活kir受体的特征。kir 基因和 hla 基因中的变异组合影响受体配体相互作用, 在个体7,8水平上塑造潜在的 nk 细胞反应能力。基因关联研究的证据表明, kir在病毒抗性 (例如人体免疫缺陷病毒 [hiv] 9 和丙型肝炎病毒 [hcv]10 ) 中发挥着重要作用, 移植的成功11、妊娠期疾病的风险和生殖成功12,13, 防止异基因造血干细胞移植 (hsct)后复发14, 15, 1 6日, 癌症风险17日。
高序列同源性以及等位基因和单倍型多样性的结合对准确基因分型kir基因的任务提出了挑战。kir 基因类型的传统方法包括序列特异性引物 (ssp) 聚合酶链反应 (pcr)18、19、20、序列特异性寡核苷酸探针 (ssop) pcr21, 以及矩阵辅助激光解吸电离-飞行质谱法 (aldi-tof ms)的时间22。这些技术的缺点是, 他们只提供部分洞察个人的基因型, 同时也是费力的执行。最近, 下一代测序 (ngs) 被特别应用于kir位点的类型。虽然此方法非常强大, 但运行成本可能很高, 并且进行深入分析和数据检查非常耗时。
qkat 是一种高通量定量 pcr 方法。虽然传统的方法是费力和耗时的, 这种方法可以运行近 1, 000个基因组 dna (gdna) 样本在五天内, 并给出kir基因型, 以及基因复制的数字。qkat 由10个多重反应组成, 每个反应的目标是两个kir位点和一个用于kir基因副本编号23的基因组 (stat6) 中固定拷贝数的参考基因.该检测方法已成功地应用于涉及大量人口面板和疾病队列的研究, 涉及传染性疾病, 如 hcv、1型糖尿病等自身免疫性疾病和先兆子宫炎等妊娠疾病, 并提供了遗传研究的基础, 旨在了解 nk 细胞功能1,4,24,25,26。
研究方案
1. dna 的制备和电镀
- 使用分光光度法或荧光仪器准确量化 gdna 浓度。
- 在96井深井板上稀释 dna 至4μμl。包括至少一个具有已知副本编号和一个非模板控件的控制 gdna 样本。
- 以 450 x g 离心96孔板 2分钟.
- 使用液体处理仪器, 将每个样品中的四聚一式贴在384井 qpcr 板上, 使每口井都有10纳克的 dna (2.5μl·井)。准备至少 10个384个井板, 每个 qkat 反应一个。
- 如果从一个96孔以上的板中提取 gdna, 请使用2% 的漂白剂和超纯水进行全体积清洗, 以清洁每个96孔板之间的液体处理系统的针头。
- 在室温下将384个井板在干净的地方孵育至少 24小时, 从而将 dna 风干。
2. 底漆和探针的制备
注: qkat 由10个多重反应组成。每个反应包括三个引物对和三个荧光标记探针, 专门放大两个kir基因和一个参考基因。对在江泽民等人的第27号文件中公布的探针进行了修改, 使寡核苷酸现在被标记为 atto 染料, 因为它们提供了更好的光稳定性和较长的信号寿命。预报价底漆组合可在市场上获得 (见材料表)。
- 根据表 1中给出的稀释量, 为每个反应准备引物组合。
- 根据表1为每个反应准备探头组合。在进行组合之前, 请先测试每个探头。
3. 主混料的制备
注:下面提到的卷用于在一组 10倍384孔板上执行一个 qkat 反应。
- 确保在384个油井板上镀金的 gdna 样本完全干燥。在冰上执行所有步骤, 并尽可能防止试剂暴露在光线下, 因为荧光标记的探头是光和热敏感的。
- 在4°c 下对 qpcr 缓冲液、底漆和探针进行除霜。
- 在冰上, 通过添加18.86 毫升的超纯水、20毫升的 qpcr 缓冲液、1, 000μl 的预熟引物组合和180μl 的预制备探针组合, 制备 10x 384 孔板的主混合物 (表 2)。
- 使用多通道移液器将主混料均匀地分布在96深的井板上, 将415μl 移液入每口井。把这个盘子放在一个被光线遮挡的冰盒里。
- 使用液体处理仪器, 将主混合物的9.5μl 分配到384个井板的每口井中, 并将其干燥。用铝箔密封盘子, 并立即将其放置在4°c。对剩余的板材重复此过程, 确保液体处理系统的针头在每个板材之间用水清洗。
- 以 450 x 克离心384孔板, 以 450 x 克离心 3分钟, 并在4°c 下隔夜或6-12小时之间孵育, 以重新悬浮 dna 并驱散任何气泡。
4. qpcr 检测
- 经过隔夜孵化后, 离心机以 450 x g的速度进行3分钟的工作, 以驱散任何剩余的气泡。
- 为了实现自动化, 请将 qpcr 机器 (例如, light cycler 480) 连接到微板处理程序 (请参见材料表)。对微板处理程序进行编程, 使其从可防止光线照射的冷却存储基座放入 qpcr 机器。
注意 从理论上讲, 该检测应在具有兼容光学设置的其他 qpcr 机器上工作。 - 使用以下循环条件: 95°c 5分钟, 然后40个周期 95°c 15秒, 66°c, 数据收集在66°c。
- 一旦运行完成, 让机器人从 qpcr 机器收集板, 并将其放置在丢弃的码头。
5. 运行后分析
- 扩增后, 按照以下步骤, 使用 qpcr 机器软件的二阶导数最大值法或 "拟合点" 方法 (参见材料表) 计算定量周期 (cq) 值。
- 打开 qpcr 软件, 然后在 "导航"选项卡中, 打开一个板的保存反应实验文件。
-
对于使用二阶导数最大值方法进行分析, 请选择 "分析" 选项卡, 然后使用 abs quant/第二个导数最大值方法创建新的分析。
- 在 "创建新的分析" 窗口中, 选择分析类型: abs quant/j3vert倒数 max 方法, 子集:所有示例, 程序: 放大, 名称: Rx-DFO (其中x是反应编号).
- 选择滤光片, 然后选择vicx\ hex/yl5055 (533-580)。这可确保选择为 stat6收集的数据。
- 选择VIC/HEX/Yellow555(533-580)的颜色补偿.单击 "计算"。对 fam (465-510) 和 Cy5/Cy5.5(618-660 重复此操作)。单击 "保存文件"。
-
对于使用 "拟合点" 方法进行分析, 请在 "分析" 选项卡中选择 "abs quant/fit 点"。
- 在 "创建新的分析" 窗口中, 选择分析类型: abs quant-fitt 方法, 子集:所有示例, 程序: 放大, 名称: RxF-DFO (其中x是反应编号)。
- 为stat6和每个kir基因 (famcy5) 选择正确的过滤器和颜色补偿。在 " noiseband " 选项卡中, 设置噪声带以排除背景噪声。
- 在 "分析" 选项卡中, 将适合点设置为 3 , 然后选择 "显示适合点"。单击 "计算"。单击 "保存文件"。
6. 结果的导出
- 在 qpcr 软件中, 打开"导航器" 选项卡. 选择"结果批量导出"。
- 打开保存实验文件的文件夹, 并将文件传输到窗口的右侧部分。单击"下一步"。选择导出文件的名称和位置。
- 选择分析类型abs quant\ 第二个导数最大方法或abs quant\ fit 点。单击"下一步"。检查文件的名称、导出文件夹和分析类型是否正确, 然后单击"下一步"开始导出过程。
- 等待, 直到导出状态确定.屏幕将自动移动到下一步。检查所有选定的文件是否已成功导出, 以便失败的文件数 = 0。单击"完成"。
- 使用脚本拆分 _ file. pl 和 roche2sds. pl 将导出的板拆分为每个板块的单独反应。
注意 脚本在请求/github 上提供。
7. 复制编号计算
- 打开拷贝数分析软件 (例如, 复制来电显示器)。选择"导入实时 pcr 结果文件"并加载由roche2sds. pl 创建的文本文件。
- 选择"分析" 并进行分析, 方法是选择具有已知复制编号的校准器样品, 或者选择最常见的复制编号.有关通常在欧洲起源人群中观察到的最常见的 kir基因拷贝数, 请参见表 5 。
8. 数据质量检查
- KIR_CNVdata_analysis_for_Excel_ver020215 使用 r 脚本。r将所有印版中的复制数字数据合并到电子表格中。
注意 脚本在请求/github 上提供。 - 对不符合kir基因已知链接不平衡 (ld) 的样品的拷贝数分析软件上的原始数据进行重新检查 (表 6)。
结果
复制编号分析可以通过将文件导出到拷贝数分析软件来实现, 该软件提供了基于 cq 方法的预测和估计的拷贝数。
复制编号可以根据板上已知的对照 dna 样本复制数量或通过输入最频繁的基因复制编号来预测 (表 5)。图 1显示了针对kir2dl4和kir3ds1的反应板的结果, 以及参考基因stat6.kir2dl4 是 kir位点中的一个框架基因, 最常见的拷贝数是两个拷贝, 而kir3ds1(一种激活基因) 最常见的拷贝数是一个拷贝。图中的结果显示了在 qpcr 软件上观察到的 pcr 扩增图以及从 qpcr 数据生成的拷贝数数据。如图所示, 该检测能够区分0、1、2、3和 4 kir基因拷贝数。复制编号分析软件还可以将复制编号在印版上的分布情况视为饼图或条形图。对于复制编号较高的样本, 复制编号预测的效果较低。
反应中使用的所有材料、gdna、缓冲液、引物和探针的质量都会影响所获得结果的准确性。然而, 结果的不一致最可能是由于一个板块上 dna 浓度的变化造成的。提取的 gdna 的纯度, 可以用26/280 和26-230 的比例进行测量, 也会对质量产生影响。需要 26/2 2602 比和26°230比率2-2.2。一个板块上 dna 浓度的不均匀范围可能导致样品之间阈值周期 (ct) 的高度变异性, 并在估计的副本数量范围内出现不和谐。图 2中的结果显示了板之间的 ct 值之间的差异对副本编号预测精度的影响。红线指示样本的估计副本编号的范围, 理想情况下, 应尽可能接近整数。
复制编号数据一旦分析, 就可以以96孔格式导出为电子表格文件。我们使用 r 脚本 (可根据要求提供) 将作为一个集运行的所有10个板块的复制编号数据合并到一个电子表格中。关于主要来自欧洲种群的kir 的已发布数据使人们能够预测kir 复合物 1中各种基因之间存在的 ld 规则。这些预测用于对获得的副本编号结果进行下游检查 (表 6)。不符合预测的 ld 的基因之间的样本可能包含异常多态性或单倍型结构变化。图 3显示了描述协议的流程图。
开发了一种名为kir单倍型标识符 (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) 的工具, 以促进对数据集中的单倍型进行归因。根据在欧洲裔人口1中观察到的参考单倍型清单,这些估算起作用。但是, 该工具还允许使用自定义的引用单倍型集。生成三个单独的文件;第一个文件列出了样本的所有单倍型组合, 第二个文件提供了组合频率最高的单倍型组合的裁剪列表, 第三个文件列出了不能分配单倍型的样本。单倍型的非分配可以作为新单倍型的指标。
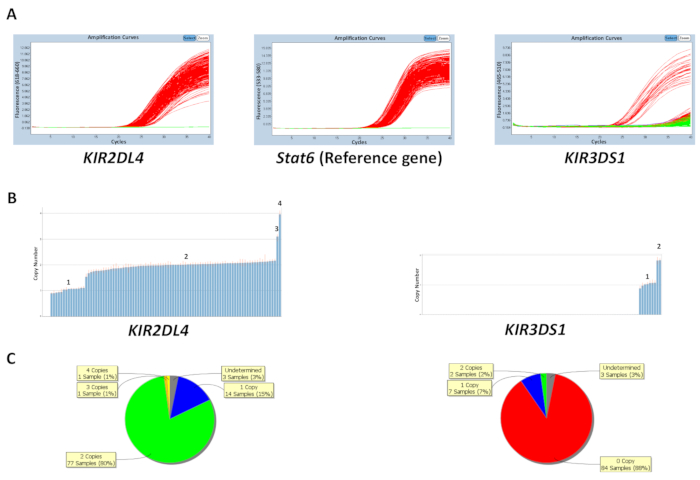
图 1: 5号反应板的代表性结果.(a) 此面板显示放大图。(b) 此面板显示复制编号图。(c) 此面板显示副本编号分布。请点击这里查看此图的较大版本.
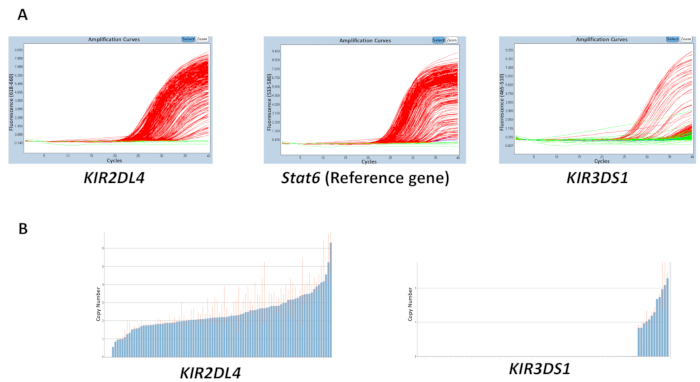
图 2: 反应数5具有可变 dna 浓度的板的代表性结果.(a) 此面板显示放大图。(b) 此面板显示复制编号图。请点击这里查看此图的较大版本.
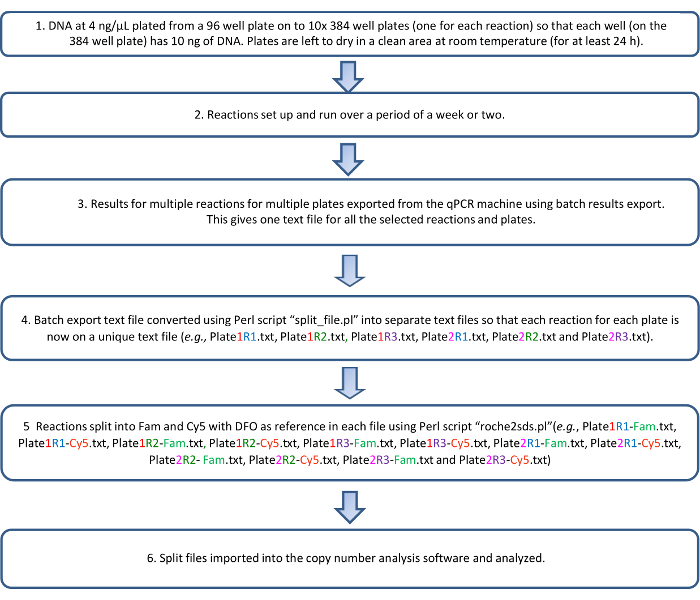
图 3: qkat 协议的流程图.请点击这里查看此图的较大版本.
| 测定 | 基因 | 前进底漆 | 浓度 (nm) | 反向底漆 | 浓度 (nm) | 探针 | 浓度 (nm) |
| 1号 | 3dp1 | a4f | 250人 | a5r | 250人 | p4a | 150人 |
| 2dl2 | 2dl2f4 | 400人 | c3r2 | 600元 | p5b | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 2号 | 2ds2 | a4f | 400人 | a6r | 400人 | p4a | 200人 |
| 2dl3 | d1f | 400人 | d1r | 400人 | p9 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 3号 | 3dl3 | a8 楼 | 500元 | a8r | 500元 | p4a | 150人 |
| 2ds4del | 2ds4del | 250人 | 2ds4r2 | 250人 | p5b | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 4号 | 3dl1e4 | b1f | 250人 | b1r | 125 | p4b | 150人 |
| 3dl1e9 | d4f | 250人 | d4r2 | 500元 | p9 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 5号 | 3ds1 | b2f | 250人 | b1r | 250人 | p4b | 150人 |
| 2dl4 | c1f | 200人 | c1r | 200人 | p5b-2 dl4 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 第6期 | 2dl1 | b3f | 500元 | b3r | 125 | p4b | 150人 |
| 2dp1 | d3f | 250人 | d3r | 500元 | p9 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 7号 | 2ds1 | b4f | 500元 | b4r | 250人 | p4b | 150人 |
| 2dl5 | d2f | 500元 | d2r | 500元 | p9 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 8号 | 2ds3 | b5f | 250人 | b5r | 250人 | p4b | 150人 |
| 3dl2e9 | d4f | 250人 | d5r | 125 | p9 | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 9号 | 3dl2e4 | a1f | 200人 | a1r | 200人 | p4a | 150人 |
| 2ds4fl | 2ds4fl | 250人 | 2ds4r2 | 500元 | p5b | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 | |
| 10号 | 2ds5 | b6f2 | 200人 | b6r3 | 200人 | p4b | 150人 |
| 2ds4 | c5f | 250人 | c5r | 250人 | p5b | 150人 | |
| 第6-stat6 | 状态6楼 | 200人 | stat6r | 200人 | pstat6 | 150人 |
表 1: 在每个 qkat 反应中使用的引物和探针的组合和浓度27.
| 反应 | 底漆液体 (μl) | 探针 (μl) | |||||||||
| r1 | 3dp1 | a4f | a5r | 2dl2f4 | c3r2 | 水 | 状态6楼 | stat6r | p4a | p5b | pstat6 |
| 2dl2 | 100元 | 100元 | 160 | 240 | 200人 | 80 | 80 | 60 | 60 | 60 | |
| R 2 | 2ds2 | a2f | a6r | d1f | d1r | 水 | 状态6楼 | stat6r | p4a | p9 | pstat6 |
| 2dl3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| 注: 只需使用20μl 的水 | |||||||||||
| r3 | 3dl3 | a8f a8fb | a8r | 2ds4delf | 2ds4r2 | 水 | 状态6楼 | stat6r | p4a | p5b | pstat6 |
| 2DS4DEL | 100 100 | 200人 | 100元 | 100元 | 200人 | 80 | 80 | 60 | 60 | 60 | |
| r4 | 3dl1e4 | b1f | b1r | d4f | d4r2 | 水 | 状态6楼 | stat6r | p4b | p9 | pstat6 |
| 3dl1e9 | 100元 | 50 | 100元 | 200人 | 350 | 80 | 80 | 60 | 60 | 60 | |
| r5 | 3ds1 | b2f | b1r | c1f | c1r | 水 | 状态6楼 | stat6r | p4b | p5b-2l4 | pstat6 |
| 2dl4 | 100元 | 100元 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| r6 | 2dl1 | b3f | b3r | d3f | d3r | 水 | 状态6楼 | stat6r | p4b | p9 | pstat6 |
| 2dp1 | 200人 | 50 | 100元 | 200人 | 250人 | 80 | 80 | 60 | 60 | 60 | |
| r7 | 2ds1 | b4f | b4r | d2f | d2r | 水 | 状态6楼 | stat6r | p4b | p9 | pstat6 |
| 2dl5 | 200人 | 100元 | 200人 | 200人 | 100元 | 80 | 80 | 60 | 60 | 60 | |
| r8 | 2ds3 | b5f | b5r | d4f | d5r | 水 | 状态6楼 | stat6r | p4b | p9 | pstat6 |
| 3dl2e9 | 100元 | 100元 | 100元 | 50 | 450元 | 80 | 80 | 60 | 60 | 60 | |
| r9 | 3dl2e4 | a1f | a1r | 2ds4wtf | 2ds4r2 | 水 | 状态6楼 | stat6r | p4a | p5b | pstat6 |
| 2ds4wt | 80 | 80 | 100元 | 200人 | 340 | 80 | 80 | 60 | 60 | 60 | |
| r10 | 2ds5 | b6f2 | b6r3 | c5f | c5r | 水 | 状态6楼 | stat6r | p4b | p5b | pstat6 |
| 2DS4TOTAL | 80 | 80 | 100元 | 100元 | 440 | 80 | 80 | 60 | 60 | 60 | |
表 2: 100μm 引物/探针库存解决方案的体积 (μl), 用于制造底漆和探针组合等价物。
| 名字 | 方向 | 5 ' 改装 | 3 ' 改装 | 序列 (5 ' →3 ') | 长度 | Tm | gc% | 外 显 子 | 位置 |
| p4a | 感悟 | Fam | bhq-1 | tcatcctgc aatttggt 卡加特加卡 | 27 | 60 | 44。4 | 4个 | 425-451 |
| p4b | 反 义 | Fam | bhq-1 | acacaaacc gtamatct ggggccc t | 28 | 62 | 50 | 4个 | 576-603 |
| p5b | 感悟 | atto647n | bhq-2 | aacattcca ggccagact ttcctctg | 25 | 60 | 52 | 5 | 828-852 |
| p5b-2 dl4 | 感悟 | atto647n | bhq-2 | aacattcca ggccagact tccctg | 25 | 61 | 56 | 5 | 828-852 |
| p9 | 感悟 | atto647n | bhq-2 | ccctctcta gggccca agacacc | 24 | 60 | 62。5 | 9 | 124-1269 |
| pstat6 | atto550 | bhq-2 | ctgattcct ccatgagca tgcagct | 26 | 62 | 50 |
表 3: qkat 中使用的探头列表 1、 27. 在寡核苷酸探针 5 ' 端使用的荧光染料 (p5b)、p5b-2dl4、p9 和 pstat6 被修改为 atto 染料。
| 基因 | 引 | 方向 | 序列 (5 克-3 ') | 长度 | Tm | gc% | 外 显 子 | 位置 | 放大器 (bp) | 可能会漏掉等位基因 | ||
| 3dl2e4 | a1f | 向前 | gcctgctgaa atcagg | 18 | 52 | 61。1 | 4个 | 399-416 | 179 | 3DL2*008, * 021, * 021, * 021。 | ||
| a1r | 反向 | ctgcaggagag gccatcaa | 19 | 53 | 52。6 | 559-577 | 3DL2*048 | |||||
| 3dp1 | a4f | 向前 | gtcctgggga aatcaga | 19 | 49 | 52。6 | 4个 | 398-416 | 112 | 没有 | ||
| a5r | 反向 | ggaggggcaaa gtgca | 18 | 52 | 55。6 | 492-509 | 没有 | |||||
| 2ds2 | a2f | 向前 | gtgcctggggga aatcaga | 19 | 49 | 52。6 | 4个 | 398-416 | 111 | 没有 | ||
| a6r | 反向 | tgagggcaaag tgttcctat | 21 | 51 | 42。9 | 488-508 | 没有 | |||||
| 3dl3 | a8fa | 向前 | gggaatcgga gagacg | 18 | 50 | 55。6 | 4个 | 406-423 | 139 | 没有 | ||
| a8fb | 向前 | gggaaatacagg agagacg | 19 | 50 | 52。6 | 405-423 | 3DL3*054, 3DL3*00905。 | |||||
| a8r | 反向 | agtgaccgg aaccg | 18 | 51 | 61。1 | 526-543 | 没有 | |||||
| 3dl1e4 | b1f | 向前 | catcggcccat 关贸总协定 | 18 | 51 | 55。6 | 4个 | 549-566 | 85 | 3DL1*00505、3DL1*006、3DL1*054、3DL1*086、3DL1*089 | ||
| b1r | 反向 | ggagct ggacaa ctgatagg | 20 | 52 | 55 | 614-633 | 3DL1*00502 | |||||
| 3ds1 | b2f | 向前 | catcgttccat gatgcg | 18 | 51 | 55。6 | 4个 | 549-566 | 85 | 3DS1*047;可能会拿起3DL1*054。 | ||
| b1r | 反向 | ggagct ggacaa ctgatagg | 20 | 52 | 55 | 614-633 | 没有 | |||||
| 2dl1 | b3f | 向前 | ttccacatcagt cgcatgac | 20 | 52 | 50 | 4个 | 544-563 | 96 | 2DL1*020, 2DL1*028 | ||
| b3r | 反向 | gtcacggagc tgacac | 18 | 50 | 61。1 | 622-639 | 2DL1*023、2DL1*029、2DL1*030 | |||||
| 2ds1 | b4f | 向前 | tcccatacagtc gcatgaa | 19 | 51 | 47。4 | 4个 | 545-563 | 96 | 2DS1*001 | ||
| b4r | 反向 | gggcactgggag ctgac | 17 | 49 | 64。7 | 624-640 | 没有 | |||||
| 2ds3 | b5f | 向前 | ctccatcgggcg catgag | 18 | 53 | 61。1 | 4个 | 546-563 | 96 | 没有 | ||
| b5r | 反向 | gggcactggga gctgaa | 18 | 51 | 61。1 | 624-641 | 没有 | |||||
| 2ds5 | b6f2 | 向前 | agaggggagg tttaacc | 19 | 50 | 52。6 | 4个 | 475-493 | 173 | 没有 | ||
| b6r3 | 反向 | tccaggggtca ctgggc | 18 | 53 | 66。7 | 630-647 | 2DS5*003 | |||||
| 2dl4 | c1f | 向前 | gcaggccacgc atcaat | 18 | 52 | 55。6 | 5 | 808-825 | 83 | 没有 | ||
| c1r | 反向 | ccgagcatcg taggtct | 19 | 52 | 52。6 | 872-890 | 2DL4*018, 2DL4*019 | |||||
| 2dl2 | 2dl2f4 | 向前 | gggggggcc catgaat | 19 | 52 | 57。9 | 5 | 778-796 | 151 | 2DL2*009;782g 改为 a。 | ||
| c3r2 | 反向 | tcgagtttgacc actcgtat | 20 | 51 | 45 | 909-928 | 没有 | |||||
| 2ds4 | c5f | 向前 | tcccgcagggc gcagc | 17 | 57 | 70。6 | 5 | 803-819 | 1120 | 没有 | ||
| c5r | 反向 | ttgaccctcgt aggagc | 19 | 52 | 57。9 | 904-922 | 2DS4*013 | |||||
| 2ds4del | 2ds4del | 向前 | ccttccttgca gccccat | 19 | 54 | 57。9 | 5 | 77.0-768 | 203 | 没有 | ||
| 2ds4r2 | 反向 | tgaggaaacaa gcaggga | 20 | 53 | 50 | 93-952 | 没有 | |||||
| 2ds4fl | 2ds4fl | 向前 | ccggccccta tgacatg | 19 | 53 | 57。9 | 5 | 744-762 | 209 | 没有 | ||
| 2ds4r2 | 反向 | tgaggaaacaa gcaggga | 20 | 53 | 50 | 93-952 | 没有 | |||||
| 2dl3 | d1f | 向前 | agcctgcagga ggtga | 17 | 48 | 58。8 | 9 | 1140-1196 | 156 | 没有 | ||
| d1r | 反向 | cagagacacact ttggatca | 20 | 50 | 45 | 1316-1335 | 2DL3*010、2DL3*017、2DL3*01801 和2DL3*01802 | |||||
| 2dl5 | d2f | 向前 | cacgctttttc 中国民航 | 20 | 52 | 50 | 9 | 1214-1233 | 1120 | 2DL5B*011 和2DL5B*020 | ||
| d2r | 反向 | ggaggagacaa tgatctt | 19 | 49 | 47。4 | 1315-1333 | 没有 | |||||
| 2dp1 | d3f | 向前 | cctcagggg acatacgt | 20 | 53 | 55 | 9 | 1184-1203 | 121 | 没有 | ||
| d3r | 反向 | ttggagttcg tgtatact | 20 | 50 | 45 | 1285-1304 | 没有 | |||||
| 3dl1e9 | d4f | 向前 | cacattggatc actgcgt | 19 | 52 | 52。6 | 9 | 1203-1221 | 93 | 3DL1*061, 3DL1*068 | ||
| d4r2 | 反向 | ccggtacaaga tgttttgta | 23 | 53 | 43。5 | 1273-1295 | 3DL1*05901、3DL1*05902、3DL1*060、3DL1*061、3DL1*064、3DL1*065、3DL1*094N 3DL1*098 | |||||
| 3dl2e9 | d4f | 向前 | cacattggatc actgcgt | 19 | 52 | 52。6 | 9 | 1203-1221 | 156 | 没有 | ||
| d5r | 反向 | gaccactgg ggctcg | 19 | 54 | 63。2 | 1340-1358 | 没有 | |||||
| 第6-stat6 | 状态6楼 | 向前 | ccagaggcctac catgggc | 20 | 54 | 60 | 1130 | |||||
| stat6r | 反向 | ccatcgcaag acctcc | 20 | 54 | 60 | |||||||
表 4: qkat 中使用的引物序列 1、 27岁
| kir基因 | 3dl3 | 2ds2 | 2dl2 | 2dl3 | 2dp1 | 2dl1 | 3dp1 | 2dl4 | 3dl1 ex9 | 3dl1 ex9 | 3ds1 | 2dl5 | 2ds3 | 2ds5 | 2ds1 | 2ds4 总 | 2ds4 佛罗里达州 | 2ds4 戴尔 | 3dl2 ex4 | 3dl2 ex9 | |
| 最常见的副本编号 | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
表 5:最常见的复制编号kir在欧洲来源的样本中常见的基因.
| 基于欧洲人口的 qkat 连锁不平衡规则 | 复制编号检查 | |||||||
| 1 | kir3dl3、KIR3DL3, kr2dl4和KIR3DL3是存在于两种单倍型上的框架基因。 | kir3dl3, kir3d1 1, kir2dl4 和 KIR3DL3 = 2 | ||||||
| 2 | kir2ds2和kir2dl2彼此在 ld 中 | 2ds2=2dl2 | ||||||
| 3个 | kir2dl2和KIR2DL2是同一基因的等位基因 | 2dl2+2DL2= 2 | ||||||
| 4个 | kir2dp1和kir2dl1彼此处于 ld 中 | 2dp1=2dl1 | ||||||
| 5 | kir3dl1和KIR3DL1的 exon 4 分别等于kir3dl1和KIR3DL1的 exon 9。 | 3dl1ex4=3DL1ex4和3DL1ex4=3DL1ex4 | ||||||
| 6 | kir3dl1和KIR3DL1均为等位基因 | 3dl1+3DL1= 2 | ||||||
| 7。 | kir2ds3和kir2ds5与kr2dl5 一起在 ld 中 | 2ds3+2ds5=2dl5 | ||||||
| 8 | kir3ds1和kir2ds1在 ld | 3ds1=2ds1 | ||||||
| 9 | kir2ds1和kir2ds4t的存在在单倍型上是相互排斥的 | 2ds1+2ds4total= 2 | ||||||
| 10 | kr2ds4fl和kr2ds4del 是 kr2ds4total的变种 | 2ds4fl+2ds4del =2ds4tal | ||||||
表 6: 相互之间的联系不平衡kir在欧洲来源人群中常见的基因可用于检查拷贝数数据 1,27.
讨论
我们描述了一种新的半自动高通量方法, 称为 qkat, 它有利于kir基因的拷贝数分型.与 ssp-pcr 等传统方法相比, 该方法是一种改进, 后者是低吞吐量的, 只能表明这些高度多态性基因的存在或不存在。
所获得的拷贝数数据的准确性取决于多种因素, 包括 gdna 样品的质量和浓度均匀性以及试剂的质量。板上 gdna 样本的质量和准确性极其重要, 因为板上浓度的变化会导致复制编号的计算错误。由于检测是使用欧洲来源的样本集进行验证的, 来自世界其他地区的队列数据需要进行更彻底的检查。这是为了确保等位基因丢失或非特异性引物探针绑定的情况不会被误解为拷贝数的变化。
虽然检测的设计和优化是为了以高通量的方式运行, 但可以对其进行修改, 以运行较少的示例。在分析较少的样本时, 复制数量分析软件中的置信度指标会受到影响, 但如果在平板上包含具有已知kir基因复制号的控制基因组 dna 样本, 并且在平板上包含额外的样本复制, 则可以改进这种情况。包括。
对于没有液体/板处理机器人的实验室, 可以使用多通道移液器进行主混料, 也可以手动将板材加载到 qpcr 仪器中。
开发 qkat 的主要目的是创建一种简单、高吞吐量、高分辨率和具有成本效益的方法, 用于疾病关联研究的基因型 kir 。这是成功地实现的, 因为 qkat 已被用于调查kir的作用, 在几个大型疾病协会研究, 包括一系列传染病, 自身免疫性条件, 和妊娠疾病4,24,25,26岁
披露声明
作者没有什么可透露的。
致谢
该项目得到了医学研究理事会、欧洲联盟 "地平线 2020" 研究和创新方案 (第669 5551 号赠款协议) 下的欧洲研究理事会和剑桥国家卫生研究所的资助。与国家医疗服务体系血液和移植系统 (nhsbt) 合作, 在剑桥大学器官捐献和移植方面设立生物医学研究中心和 nih 血液和移植研究小组 (nihr btru)。所表达的观点是作者的观点, 不一定是国家医疗服务体系、国家人权研究所、卫生部或 nhsbt 的观点。
材料
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
参考文献
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。