Method Article
qKAT: nicel Semi-automated yazmaktan katil hücreli immünglobulin benzeri reseptör gen
Bu Makalede
Özet
Nicel katil hücre immünglobulin benzeri reseptör (kır) yarı otomatik (qKAT) yazarak sayı türü kır genler kendi uygulama için nüfus ve hastalığı Derneği çalışmalarda kopyalamak için basit, yüksek performans ve düşük maliyetli bir yöntemdir.
Özet
Katil hücre immünglobulin benzeri reseptörler (KIRs) doğal öldürücü (NK) ve T hücreleri, kromozom 19 genlerin Polimorfik bir kümeye tarafından kodlanmış üzerinde inhibitör ve aktive bağışıklık reseptörleri kümesidir. Onların en çok karakterize ligandlar MHC kompleksi (MHC) odağı Kromozom 6 Tarih içinde kodlanmış insan lökosit antijeni (HLA) moleküllerdir. Önemli kanıt onlar bağışıklık, üreme ve nakli, doğru bir şekilde genotip olabilir teknikleri çok önemli hale önemli bir rol oynamak onları. Ancak, yüksek-sıra homoloji, de allelic ve kopya numarası varyasyon, zor yapmak doğru olabilir tasarım yöntemleri ve verimli bir şekilde genotip tüm KIR genler. Geleneksel yöntemler genellikle çözünürlük elde edilen veri, performans, maliyet-etkililik ve ayarlama ve deneyler çalıştırmak için geçen süre sınırlıdır. Biz nicel kır yarı otomatik (qKAT), tipik olarak adlandırılan bir yöntem tarif tüm KIR locus genlerinde Gen kopya numaraları belirlemek yüksek üretilen iş çok katmanlı gerçek zamanlı Polimeraz zincir reaksiyonu yöntemi olduğu. qKAT daha da onları kapsayacak yapısal olarak çok biçimli haplotypes değişimler anlaması için kullanılan yüksek çözünürlüklü kır sayı veri kopyalama, sağlayan basit bir yüksek üretilen iş yöntemidir. Bu kopya sayısı ve haplogrubundan veri büyük ölçekli hastalığı dernekler, Populasyon genetiği gibi ifade ve KIR ve HLAarasındaki işlevsel etkileşimler üzerine araştırmalar çalışmaları için yararlı olabilir.
Giriş
İnsanlarda, katil immünglobulin benzeri reseptör(kır) odağı kromozom 19 lökosit reseptör kompleksi (LRC) içinde uzun kolundaki eşleştirilir. Bu locus yaklaşık 150 kb uzunluğunda ve 15 kır düzenlenmiş genler baş-için-kuyruk içerir. Şu anda bilinir kır loci vardır KIR2DL1, KIR2DL2/KIR2DL3, KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1-5, KIR3DL1/KIR3DS1, KIR3DL2-3, ve iki psödogenler, KIR2DP1 ve KIR3DP1. Kır genler iki boyutlu (2D) kodlamak ve üç boyutlu (3D) immünglobulin benzeri etki alanı reseptörleri ile kısa (S; aktive) veya uzun (L; inhibitör) doğal öldürücü (NK) hücreleri ve T alt kümeleri tarafından ifade edilir sitoplazmik kuyruk hücreleri. Kopya numarası varyasyon kır locus formları içinde değişken gen içerik1çeşitli haplotypes sergiledi. Bir yakın kafa kuyruk gen düzenlemesi ve yüksek-sıra homoloji, Sigara allelic homolog rekombinasyon (NAHR), haplotypic değişkenliği için sorumlu olmak için önerilen mekanizmasıdır. 100'den fazla farklı haplotypes nüfus dünya çapında1,2,3,4' te bildirilmiştir. Bu haplotypes iki ana gruba ayrılmıştır olabilir: A ve B haplotypes. A haplogrubundan 7 kır gen içermektedir: KIR3DL3, KIR2DL1, KIR2DL3, KIR2DL4, KIR3DL1ve KIR3DL2, inhibitör kır genler ve aktive kır gen KIR2DS4. Ancak, yukarı sadece KIR haplogrubundan A için homozigoz Avrupa kökenli bireylerin % 70 KIR2DS45,6işlevsel olmayan "silme" şeklinde taşımak. Tüm diğer KIR gen kombinasyonları oluşturmak dahil en az bir özel KIR genlerin KIR2DS1, B grubu haplotypes, KIR2DS2, KIR2DS3, KIR2DS5, KIR3DS1, KIR2DL2, ve KIR2DL5ve genel olarak iki veya daha fazla aktive kır genler içerir.
HLA sınıf ı molekülleri ligandlar için belirli inhibitör reseptörler (KIR2DL1, KIR2DL2, KIR2DL3ve KIR3DL1), aktive reseptörleri (KIR2DS1, KIR2DS2, KIR2DS4, olarak tespit edilmiştir KIR2DS5ve KIR3DS1), ve KIR2DL4için hangi diğer inhibitör kır reseptörleri gibi uzun sitoplazmik kuyruk içerir ancak Ayrıca bir ortak olan ekstrasellüler etki alanı pozitif yüklü bir kalıntı sahip benzersiz bir kır diğer aktive kır reseptörleri özelliğidir. Kasanın içinde KIR genler ve HLA genleri versiyonlarının Reseptör ligand etkileşim o şekiller potansiyel NK hücre yanıt bireysel düzeyde7,8etkiler. Genetik Derneği çalışmaları kanıt belirtilen kır viral direnç (e.g., insan immün yetmezlik virüsü [HIV]9 ve hepatit C virüsü [HCV]10), bir rol oynamaktadır nakli başarısı 11, gebelik bozuklukları ve üreme başarı12,13, allojeneik hematopoietik kök hücre nakli (HKHT)14,15, sonra nüks karşı koruma risk 16ve17Kanser riski.
Kombinasyon yüksek sıra homoloji ve allelic ve haplotypic çeşitlilik sunar sorunları genlerin doğru Genotipleme KIR görevde. Kır genler yazmanız için geleneksel yöntemler arasında fingerprinting astar (SSP) Polimeraz zincir reaksiyonu (PCR)18,19,20, fingerprinting Oligonükleotid (SSOP) PCR21, yoklama ve matris yardımlı lazer desorption iyonlaşma-saat uçuş kütle spektrometresi (MALDI-TOF MS)22. Bu teknikler sakıncaları onların sadece kısmi bir bireyin genotipi içgörü iken de gerçekleştirmek zahmetli olma sağlamak vardır. Son zamanlarda yeni nesil sıralama (NGS) KIR locus özellikle yazın uygulandı. Bu yöntem çok güçlü olsa da, çalıştırmak pahalı olabilir ve derinlemesine analiz ve veri denetimleri yapmak zaman alır.
qKAT bir yüksek-den geçerek kantitatif PCR yöntemidir. Geleneksel yöntemlerle zahmetli ve zaman alıcı olmakla birlikte, bu yöntem, beş gün içinde yaklaşık 1000 genomik DNA (gDNA) örnekleri çalıştırmak olanak verir ve KIR genotip Gen kopya numarası verir. qKAT her biri iki KIR loci hedefleyen on multiplex reaksiyonları, oluşur ve bir referans gen kır gen göreli miktar için kullanılan genom (STAT6) bir sabit kopya sayısının sayı23kopyalayın. Bu tahlil başarıyla büyük nüfus panellere ve hastalığı kohort HCV, tip 1 diyabet gibi otoimmün koşulları gibi bulaşıcı hastalıklar ve preeklampsi gibi gebelik bozuklukları ile ilgili olarak bir genetik çalışmalarda kullanılan NK hücre işlevi1,4,24,25,26anlama amaçlı çalışmalar için destek.
Protokol
1. hazırlık ve DNA dışında kaplama
- Doğru bir spektrofotometrik veya Fluorometrik enstrüman kullanarak gDNA konsantrasyon ölçmek.
- DNA 4 ng/µL 96-şey derin-şey tabakta oranında seyreltin. En az bir denetim gDNA örnek bir bilinen kopya numarası ile ve bir şablon sigara denetimi içerir.
- 96-şey plakaları 450 x g 2 min için de santrifüj kapasitesi.
- Sıvı işleme aracı kullanarak dağıtmak quadruplicate 384-de qPCR plakaları üzerine her örnekte böylece her şey 10 ng DNA'ın (2.5 µL/de). En az on 384-şey levha, her qKAT reaksiyon için hazırlayın.
- GDNA birden fazla 96-şey plaka reçete, tam birim yıkama % 2 çamaşır suyu ve işleme sistemi her 96-şey plaka gDNA örneklerin arasında sıvı iğnelerden temizlemek için ultrasaf su ile gerçekleştirin.
- DNA, oda sıcaklığında en az 24 saat için temiz bir alan 384-şey levha kuluçka tarafından kuruması.
2. Astar ve sondalar hazırlanması
Not: qKAT on multiplex reaksiyonları oluşur. Her reaksiyon üç astar çift ve özellikle iki KIR genler ve bir referans gen yükseltmek üç Floresans etiketli sondalar içerir. Geliştirilmiş photostability ve uzun sinyal yaşam süreleri sundukları beri oligonucleotides şimdi ATTO boyalar ile etiketlenir böylece Jiang vd.27 ' de Yayınlandı sondalar modifiye edilmiştir. Pre-bölünmemeli astar piyasada bulunan (bakınız Tablo reçetesi) oluşur.
- Astar bileşimleri her reaksiyon başı olarak Tablo 1' de verilen dilutions için hazır olun.
- Sonda kombinasyonları her reaksiyon başı olarak Tablo 1için hazır olun. Kasanın yapmadan önce tek tek her sonda sınayın.
3. Master Mix hazırlanması
Not Aşağıda belirtilen birimleri bir qKAT reaksiyon 10 x 384-şey kaplamalar kümesi üzerinde gerçekleştirmek için vardır.
- 384-şey Tabaklarda kaplama gDNA örnekleri tamamen kuru olduğundan emin olun. Buz üstünde tüm adımları yapmak ve floresan etiketli probları fotoğraf ve ısı duyarlı olduğundan mümkün olduğu kadar gün ışığına maruz kaplı reaktifler tutun.
- QPCR arabellek, astar ve sonda aliquots 4 ° C'de defrost
- Buz üzerinde 10 x 384-şey plakalar için 18.86 mL ultrasaf su, 20 mL qPCR arabellek, preprepared astar kombinasyonu 1000 µL ve preprepared sonda birleşimi (Tablo 2) 180 µL ekleyerek ana bir karışım hazır olun.
- Ana karışımı eşit bir 96-derin iyi plaka 415 µL her kuyuya pipetting bir çok kanallı pipet kullanarak dağıtmak. Bu plaka bir buz kutusuna gelen ışık kapalı tutun.
- Sıvı işleme enstrüman kullanarak, kurutulmuş gDNA ile 384-şey plaka her kuyuya ana karışımı 9.5 µL dağıtmak. Plaka bir folyo ile kapatın ve hemen 4 ° C'de yer Sistem işleme sıvı iğnelerden her plaka arasında su ile yıkanır sağlanması kalan plakaları için bu işlemi yineleyin.
- 450 x g 3 dk de 384-şey plakaları santrifüj kapasitesi ve gecede 4 ° C'de veya arasında 6-12 h DNA resuspend için ve herhangi bir hava kabarcıkları dağılımı için kuluçkaya.
4. qPCR tahlil
- Gecede kuluçka vasıl 450 x g için kalan herhangi bir hava kabarcıkları dağılımı 3 dk santrifüj kapasitesi.
- Otomasyon amacıyla, qPCR makine (örneğin, LightCycler 480) Mikroplaka işleyicisine bağlanmak ( Tablo malzemelerigörmek). Program Mikroplaka işleyicinin ışıktan korunan bir soğutulmuş depolama dock qPCR makineden tabak yerleştirin.
Not deneyleri teorik olarak, diğer qPCR makine ile uyumlu optik ayarları üzerinde çalışması gerektiğini. - Aşağıdaki bisiklet koşullarını kullanın: 95 ° C 5 min için takip 95 ° C 40 döngüsü tarafından 15 s ve 50 66 ° C için veri toplama 66 ° C'de ile s
- Çalışma tamamlandıktan sonra robot plaka qPCR makineden toplamak ve atma havuzda yer var.
5. çalıştırma sonrası analizi
- Amplifikasyon sonra ikinci türev maksimum yöntemi veya uyum Puan yöntemi qPCR makine yazılım programıyla miktar döngüsü (Cq) değerleri hesaplamak (bkz. Tablo malzemeler), aşağıdaki adımları izleyerek.
- QPCR yazılımını açın ve Navigator sekmede bir tabak kaydedilmiş tepki deneme dosyasını açın.
-
İkinci türev maksimum yöntemiyle analizi için analiz sekmesini seçin ve yeni bir analiz Abs Quant/Second türev Max yöntemini kullanarak oluşturun.
- Oluştur yeni analiz penceresinde analiz türünü seçin: Abs Quant/Second türev Max yöntemi, alt: tüm örnekleri, programı: amplifikasyon, adı: Rx-DFO ( x nerede tepki numarası).
- Filtre tarak ve VIC/HEX/Yellow555 (533-580)seçin. Bu STAT6 için toplanan veriler seçilir sağlar.
- Renk tazminat VIC/HEX/Yellow555(533-580)için seçin. Hesaplar' ı tıklatın. Bu Fam (465-510) ve Cy5/Cy5.5(618-660) için yineleyin. Dosya Kaydet' i tıklatın.
-
Uyum Puan yöntemiyle analizi için Abs Quant/Fit noktaları analiz sekmesinde seçin.
- Oluştur yeni analiz penceresinde analiz türünü seçin: Abs Quant/uyum Puan yöntemi, alt: tüm örnekleri, programı: amplifikasyon, adı: RxF-DFO ( x nerede tepki numarası).
- Doğru filtreleri ve renk tazminatlar için STAT6 ve her kır genlerin (Fam/Cy5) seçin. Noiseband sekmesinde, arka plan gürültü çıkarmak için gürültü grubu ayarlayın.
- Analiz sekmesi uygun Puan 3 ' e ayarlayın ve gösteri Puan Sığdır' ı. Hesaplar' ı tıklatın. Dosya Kaydet' i tıklatın.
6. ihracat sonuçları
- QPCR yazılımında Navigator açın sekmesini seçin Toplu iş sonuçlarını aktarın.
- Deney dosyaları kaydedilir ve pencerenin sağ tarafında bölümüne transfer dosyaları içeren klasörü açın. İleri' yi tıklatın. Adı ve dışa aktarma dosyasının konumunu seçin.
- Analiz türü Abs Quant/Second türev Max yöntemi veya Abs Quant/Fit noktalarıseçin. İleri' yi tıklatın. Dosya, ihracat klasör ve analiz türünün adını doğru olduğundan ve verme işlemini başlatmak için İleri ' yi tıklatın emin olun.
- Verme durumu Okolana kadar bekleyin. Ekran otomatik olarak bir sonraki adıma hareket edecek. Seçilen tüm dosyalar başarıyla verildiğini kontrol böylece dosya sayısı başarısız oldu = 0. Bitti' yi tıklatın.
- Komut dosyaları split_file.pl ve roche2sds.pl verilen plakaları her plaka için bireysel tepkiler içine bölmek için kullanın.
Not komut dosyalarını istek/GitHub sağlanır.
7. kopya sayı hesaplamaları
- Kopya numara analiz yazılımı (örneğin, CopyCaller) açın. Alma gerçek zamanlı PCR sonuçları dosyasını seçin ve roche2sds.pltarafından oluşturulan metin dosyaları yükleyin.
- Analiz seçin ve her iki seçme Kalibratör örnek bilinen kopya numarası ile veya en sık kopya numarasıseçerek analiz yapmak. Bkz. Tablo 5 kır genler genellikle Avrupa kökenli nüfus içinde gözlenen en sık kopya sayısı için.
8. veri kalite denetimleri
- KIR_CNVdata_analysis_for_Excel_ver020215 komut dosyası kullan R. R bir elektronik tabloya tüm plakaları kopya sayı veri birleştirmek için.
Not komut dosyalarını istek/GitHub sağlanır. - Kır genler (Tablo 6) için bilinen bağlantı dengesizliği (LD) için uygun olmayan örnekleri için kopya numara analiz yazılımı ham veri yeniden denetleyin.
Sonuçlar
Kopya numara analizi ΔΔCq yöntemine göre tahmini ve tahmini kopya numarası sağlayan kopya numara analiz yazılım, dosyaları dışa aktararak yürütülen olabilir.
Kopya sayısı da kontrol DNA örnekleri plaka üzerinde veya en sık Gen kopya sayısı (Tablo 5) giren tarafından bilinen kopya sayısına dayalı tahmin edilebilir. Şekil 1 gösterir KIR2DL4 ve KIR3DS1gibi referans gen hedefleyen bir tepki için bir tabak sonuçlarını STAT6. En sık kopya KIR2DL4, kır odağı bir çerçeve gen numarasıdır iki kopya, KIR3DS1, aktive bir gen için en sık kopya numarası bir kopya ise. Rakam sonuçlarında qPCR yazılım ve qPCR verilerinden oluşturulan kopya sayı veri üzerinde gözlenen PCR güçlendirme araziler göster. Görüldüğü gibi tahlil 0, 1, 2, 3 ve 4 kır Gen kopya numaraları arasında ayrım yapabiliyor. Kopya numara analiz yazılımı da bir kopya sayısı dağılımı bir pasta grafik veya çubuk grafik olarak plaka arasında sağlar. Kopya sayı tahmin etkinlik örnekleri ile daha yüksek bir kopya sayı için düşüktür.
Reaksiyonlar, gDNA, tampon, astar ve sondalar, kullanılan malzemelerin kalitesi elde edilen sonuçları doğruluğunu etkileyebilir. Ancak, sonuçlarında uyumsuzluk nedeniyle DNA toplama bir plaka arasında varyasyon nedeni büyük olasılıkla. 260/280 ve 260 kullanarak ölçülen ayıklanan gDNA saflığı/230 oranları, ayrıca kalitesi üzerinde bir etkisi olabilir. 2-2,2 1,8-2 ve 260/230 a oranı 260/280 oranında arzu vardır. Bir plaka üzerinde bir DNA düzensiz aralığı konsantrasyonları yüksek bir değişkenlik örnekleri ve uyumsuzluk tahmini kopya numarası aralığında arasındaki eşik döngüsü (Ct) yol açabilir. Sonuçlar Şekil 2 Ct değerleri arasında bir plaka arasında eşitsizlik kopya numarası tahminindeki doğruluğu üzerinde olabilir etkisi gösterir. Kırmızı çizgi bir örnek için tahmini kopya numarası aralığını gösterir ve ideal olarak, bir tamsayı olarak mümkün olduğunca yakın olmalıdır.
Bir kez analiz, sayı veri kopyalama, 96-şey biçiminde elektronik tablo dosyası olarak dışa aktarılabilir. Biz bir R komut dosyası (istek üzerine temin edilebilir) bir elektronik tabloya bir küme olarak çalıştırılan tüm 10 plakaları kopya sayı veri birleştirmek için kullanılır. Yayınlanan verilerden çoğunlukla Avrupa kökenli nüfus KIRs hakkında tahmin kır karmaşık1çeşitli genler arasındaki mevcut LD kuralları sağlar. Bu tahminler (Tablo 6) elde edilen kopya numara sonuçları karşıdan akış denetimi yapmak için kullanılır. Genler arasında tahmin edilen LD için uygun olmayan örnekleri olağandışı çok biçimlilik veya haplotypic yapısal farklılıklar içeriyor olabilir. Protokol açıklayan bir akış çizelgesi şekil 3' te gösterilmiştir.
Kır haplogrubundan tanımlayıcı (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) olarak adlandırılan bir araç zina ile haplotypes veri kümesinden kolaylaştırmak için geliştirilmiştir. Zina bir Avrupa kökenli nüfus1' gözlenen başvuru haplotypes listesini temelinde çalışır. Ancak, bu araç da başvuru haplotypes özel bir kümesi için onun yerine kullanılacak sağlar. Üç ayrı dosyaları oluşturulur; ilk dosyanın bir örneği için tüm haplogrubundan birleşimleri listelenir, ikinci dosya kesilmiş en yüksek kombine frekansları var haplotypes birleşimlerinin listesini sağlar ve üçüncü dosyası, haplotypes atanamaz örnekleri listeler. Sigara-atama haplotypes, roman haplotypes bir göstergesi olarak kullanılabilir.
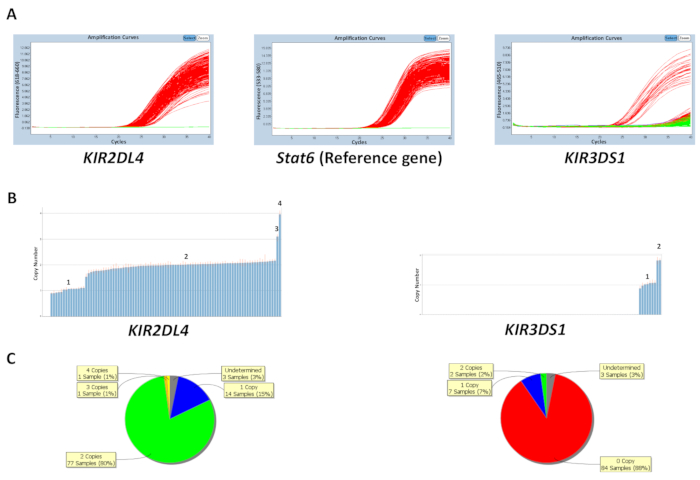
Şekil 1: tepki için bir tabak temsilcisi sonuçları 5 numara. (A) bu paneli gösterir amplifikasyon araziler. (B) bu panel programları numara araziler kopyalar. (C) bu paneli kopya numarası dağıtım gösterir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
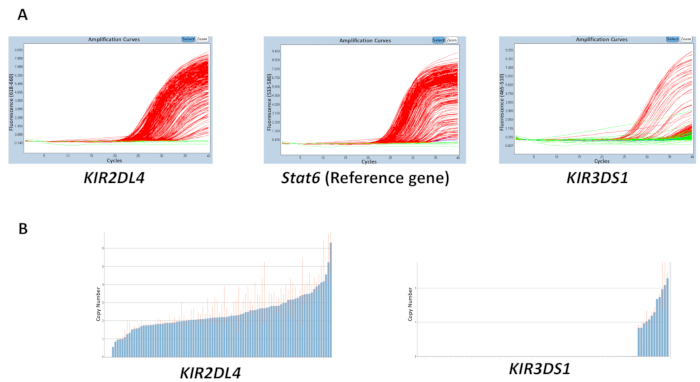
Resim 2: bir tabak tepki için değişken bir DNA konsantrasyon ile temsilcisi sonuçları 5 numara. (A) bu paneli gösterir amplifikasyon araziler. (B) bu panel programları numara araziler kopyalar. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
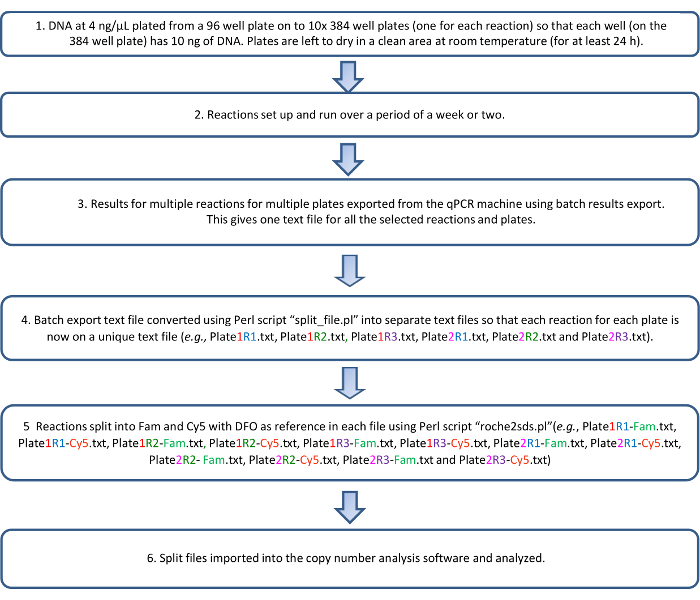
Şekil 3: qKAT iletişim kuralının akış şeması. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| Tahlil | Genler | İleri astar | Konsantrasyonu (nM) | Ters astar | Konsantrasyonu (nM) | Probları | Konsantrasyonu (nM) |
| No 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2DL 3 | D1F | 400 | 1ÖK | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 3 | 3DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 4 | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 5 | 3DS1 | B2F | 250 | B1R | 250 | P4b | 150 |
| 2DL 4 | C1F | 200 | C1R | 200 | P5B - 2DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 6 | 2DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| 2DL 5 | D2F | 500 | 2ÖK | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 8 | 2DS3 | B5F | 250 | B5R | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | D5R | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 10 | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5B | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
Tablo 1: kombinasyon ve astar ve her qKAT tepki olarak kullanılan problar konsantrasyon 27 .
| Reaksiyon | Astar Aliquots (µL) | Sonda Aliquots (µL) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | SU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | 1ÖK | SU | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| Not: daha az MasterMix suda 20 µL gerekir | |||||||||||
| R3 | 3DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | SU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | SU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | SU | STAT6F | STAT6R | P4B | P5B - 2L 4 | PSTAT6 |
| 2DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | SU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | 2ÖK | SU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | SU | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | SU | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | SU | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
Tablo 2: astar yapmak ve birlikte aliquots soruşturma birimleri (µL) 100 µM astar/sonda hisse senedi çözümleri.
| Adı | Yön | 5´ değişiklik | 3´ değişiklik | Sıra (5' →3') | Uzunluğu | TM | GC % | EXoN | Pozisyon |
| P4a | Anlamda | FAM | BHQ-1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44.4 | 4 | 425-451 |
| P4b | Antisens | FAM | BHQ-1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5B | Anlamda | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828-852 |
| P5B - 2DL 4 | Anlamda | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828-852 |
| P9 | Anlamda | ATTO647N | BHQ-2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62,5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ-2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
Tablo 3: qKAT içinde kullanılan problar listesi 1, 27. 5' sonunda oligo probları P5b, P5b - 2 DL 4, P9 ve PSTAT6 kullanılan floresan boyalar ATTO boyalar için modifiye edilmiştir.
| Gen | Astar | Yön | Sıra (5´-3´) | Uzunluğu | TM | GC % | EXoN | Pozisyon | Amplicon (bp) | Gen cevapsız | ||
| 3DL2e4 | A1F | İleri | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 2 * 008, * 021, * 027, * 038. | ||
| A1R | Ters | CTGCAAGGACAG GCATCAA | 19 | 53 | 52.6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | İleri | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398-416 | 112 | Hiçbiri | ||
| A5R | Ters | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55.6 | 492-509 | Hiçbiri | |||||
| 2DS2 | A2F | İleri | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398-416 | 111 | Hiçbiri | ||
| A6R | Ters | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42,9 | 488-508 | Hiçbiri | |||||
| 3DL 3 | A8Fa | İleri | GTGAAATCGGGA GAGACG | 18 | 50 | 55.6 | 4 | 406-423 | 139 | Hiçbiri | ||
| A8Fb | İleri | GGTGAAATCAGG AGAGACG | 19 | 50 | 52.6 | 405-423 | 3DL 3 * 054, 3DL 3 * 00905. | |||||
| A8R | Ters | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | Hiçbiri | |||||
| 3DL1e4 | B1F | İleri | CATCGGTCCCAT GATGCT | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DL 1 * 00505, 3DL 1 * 006, 3DL 1 * 054, 3DL 1 * 086, 3DL 1 * 089 | ||
| B1R | Ters | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | İleri | CATCGGTTCCAT GATGCG | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DS1 * 047; 3 DL 1 * 054 kadar teslim alabilirsiniz. | ||
| B1R | Ters | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | Hiçbiri | |||||
| 2DL 1 | B3F | İleri | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | 2DL 1 * 020, 2DL 1 * 028 | ||
| B3R | Ters | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | 2DL 1 * 023, 2DL 1 * 029, 2DL 1 * 030 | |||||
| 2DS1 | B4F | İleri | TCTCCATCAGTC GCATGAA | 19 | 51 | 47.4 | 4 | 545 563 | 96 | 2DS1 * 001 | ||
| B4R | Ters | GGTCACTGGGAG CTGAC | 17 | 49 | 64.7 | 624-640 | Hiçbiri | |||||
| 2DS3 | B5F | İleri | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546-563 | 96 | Hiçbiri | ||
| B5R | Ters | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | Hiçbiri | |||||
| 2DS5 | B6F2 | İleri | AGAGAGGGGACG TTTAACC | 19 | 50 | 52.6 | 4 | 475-493 | 173 | Hiçbiri | ||
| B6R3 | Ters | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66,7 | 630-647 | 2DS5 * 003 | |||||
| 2DL 4 | C1F | İleri | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55.6 | 5 | 808-825 | 83 | Hiçbiri | ||
| C1R | Ters | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52.6 | 872-890 | 2DL 4 * 018, 2DL 4 * 019 | |||||
| 2DL 2 | 2DL2F4 | İleri | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57,9 | 5 | 778-796 | 151 | 2DL 2 * 009; 782G a ana bilgisayarına değişti | ||
| C3R2 | Ters | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | Hiçbiri | |||||
| 2DS4 | C5F | İleri | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | Hiçbiri | ||
| C5R | Ters | TTGACCACTCGT AGGGAGC | 19 | 52 | 57,9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | İleri | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57,9 | 5 | 750-768 | 203 | Hiçbiri | ||
| 2DS4R2 | Ters | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Hiçbiri | |||||
| 2DS4FL | 2DS4FL | İleri | CCGGAGCTCCTA TGACATG | 19 | 53 | 57,9 | 5 | 744-762 | 209 | Hiçbiri | ||
| 2DS4R2 | Ters | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | Hiçbiri | |||||
| 2DL 3 | D1F | İleri | AGACCCTCAGGA GGTGA | 17 | 48 | 58.8 | 9 | 1180-1196 | 156 | Hiçbiri | ||
| 1ÖK | Ters | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010, 2DL 3 * 017, 2DL 3 * 01801 ve 2DL 3 * 01802 | |||||
| 2DL 5 | D2F | İleri | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 42U | 2DL5B * 011 ve 2DL5B * 020 | ||
| 2ÖK | Ters | GGCAGGAGACAA TGATCTT | 19 | 49 | 47.4 | 1315-1333 | Hiçbiri | |||||
| 2DP1 | D3F | İleri | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | Hiçbiri | ||
| D3R | Ters | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | Hiçbiri | |||||
| 3DL1e9 | D4F | İleri | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 93 | 3DL 1 * 061, 3DL 1 * 068 | ||
| D4R2 | Ters | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43.5 | 1273-1295 | 3DL 1 * 05901, 3DL 1 * 05902, 3DL 1 * 060, 3DL 1 * 061, 3DL 1 * 064, 3DL 1 * 065, 3DL 1 * 094N, 3DL 1 * 098 | |||||
| 3DL2e9 | D4F | İleri | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 156 | Hiçbiri | ||
| D5R | Ters | GACCTGACTGTG GTGCTCG | 19 | 54 | 63,2 | 1340-1358 | Hiçbiri | |||||
| STAT6 | STAT6F | İleri | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129 | |||||
| STAT6R | Ters | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
Tablo 4: qKAT içinde kullanılan astar dizisi 1, 27.
| Kır Gen | 3DL 3 | 2DS2 | 2DL 2 | 2DL 3 | 2DP1 | 2DL 1 | 3DP1 | 2DL 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | 2DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 Toplam | 2DS4 FL | 2DS4 DEL | 3DL 2 EX4 | 3DL 2 EX9 | |
| En sık kopya numarası | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
Tablo 5: En sık kopya numarası Kır yaygın olarak gözlenen Avrupa kökenli örneklerinde genler.
| Avrupa nüfus tabanlı qKAT için bağlantı dengesizlik kuralları | Kopya numarası onay | |||||||
| 1 | KIR3DL3, KIR3DP1, KIR2DL4 ve KIR3DL2 framework genler üzerinde her iki haplotypes mevcut bulunmaktadır. | KIR3DL3, KIR3DP1, KIR2DL4 ve KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 ve KIR2DL2 LD içinde birbirleriyle olan | 2DS22 DL 2 = | ||||||
| 3 | KIR2DL2 ve KIR2DL3 aynı gen allelleri vardır | 2 DL 2+2 DL 3= 2 | ||||||
| 4 | KIR2DP1 ve KIR2DL1 LD içinde birbirleriyle olan | 2DP12 DL 1 = | ||||||
| 5 | EXoN 4 KIR3DL1 ve KIR3DL2 sırasıyla exon 9 KIR3DL1 ve KIR3DL2 eşittir. | 3DL1ex43DL1ex9 = ve 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 ve KIR3DS1 gen vardır | 3 DL 1+3DS1= 2 | ||||||
| 7 | LD KIR2DL5 ile KIR2DS3 ve KIR2DS5 bulunmaktadır | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1 ve KIR2DS1 LD içinde vardır | 3DS12DS1 = | ||||||
| 9 | KIR2DS1 ve KIR2DS4Total varlığı bir haplogrubundan birlikte kullanılamaz | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL ve KIR2DS4del KIR2DS4TOTAL türevleri vardır | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
Tablo 6: arasında bağlantı dengesizlik Kır Avrupa kökenli nüfus yaygın olarak gözlenen genlerin kopya sayı veri kontrol etmek için kullanılan 1,27.
Tartışmalar
Biz kopya kır genler. numara yazarak kolaylaştırır qKAT olarak adlandırılan bir roman yarı otomatik yüksek üretilen iş yöntemi, açıklanan Düşük işlem hacmi ve yalnızca son derece Polimorfik bu genlerin olup belirtebilirsiniz geleneksel yöntemler üzerinde SSP-PCR gibi bir gelişme yöntemidir.
Elde edilen kopya sayı verilerin doğruluğunu kalite ve konsantrasyon-homojenlik reaktifleri kalite ve gDNA örnekleri de dahil olmak üzere birden fazla etkene bağlıdır. Konsantrasyon plaka arasında farklılığı kopya numarası hesaplama hataları neden olabilir beri kalitesini ve doğruluğunu gDNA örnekler bir plaka üzerindeki son derece önemlidir. Deneyleri Avrupa kökenli örnek kümesi'ni kullanma doğrulanmış beri tabur dünyanın diğer bölgelerinden gelen verileri daha kapsamlı denetimler gerektirir. Bu formda çıkarma veya non-spesifik astar/sonda bağlama örneklerini kopya numarası varyasyon yanlış yorumlandığını değil sağlamaktır.
Deneyleri tasarlanmış ve üretilen yüksek-iş çalıştırmak için en iyi duruma getirilmiş olsa da, daha az numunelerinin çalışılabilmesi için değiştirilebilir. Güven metrik kopya numara analiz yazılımı olarak daha az örnekleri analiz ederken etkilenir ama bu kontrol genomik DNA örnekleri ile bilinen bir kır Gen kopya numarası plaka üzerinde bulunan ve ek örnek çoğaltır geliştirilebilir dahil.
Laboratuarlar sıvı/plaka-taşıma robotlar olmadan için ana mix çok kanallı Pipetler kullanarak reçete ve plakaları el ile qPCR araç yüklenebilir.
QKAT gelişimi arkasındaki temel amacı bir basit, yüksek performans, yüksek çözünürlüklü ve maliyet-etkin yöntem genotip KIRs hastalığı için dernek çalışmaları yaratmaktı. QKAT kır rolde bulaşıcı hastalıklar, otoimmün koşullar ve gebelik bozuklukları4, bir dizi de dahil olmak üzere birçok büyük hastalığı Derneği araştırma soruşturma istihdam beri bu başarıyla sağlandı 24 , 25 , 26.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Tıbbi Araştırma Konseyi (MRC), Avrupa Birliği'nin ufuk 2020 araştırma ve yenilik programı (hibe sözleşmesi No 695551) altında Avrupa Araştırma Konseyi (ERC) ve Ulusal Sağlık Enstitüsü (NIH) Cambridge fon alınan proje Biyomedikal Araştırma Merkezi ve NIH araştırma kan ve nakli araştırma birimi (NIHR BTRU) Organ bağışı ve nakli Cambridge Üniversitesi ve USS kan ve nakli (NHSBT) ile işbirliği içinde. Görüşler yazarlar ve ille bu NHS, NIHR, Sağlık Bakanlığı veya NHSBT vardır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
Referanslar
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır