Method Article
qKAT: キラー細胞免疫グロブリン様受容体遺伝子の定量的半自動化された入力
要約
定量のキラー細胞免疫グロブリン様受容体 (KIR) 半自動入力 (qKAT) は、人口と病気の関連研究での応用数型KIRの遺伝子をコピーする簡単な高スループット、およびコスト効率の高い方法です。
要約
キラー細胞免疫グロブリン様受容体 (KIRs)、ナチュラル キラー (NK) と 19 番染色体上の遺伝子の多形クラスターによって符号化される T 細胞の抑制、活性化の免疫受容体のセットです。最も特徴付けられるリガンドは染色体 6 の主要な組織適合性の複雑な (MHC) 軌跡内にエンコードされている人間の白血球の抗原 (HLA) 分子。免疫・再生・移植、遺伝子型を正確にできる技術を持っている重要なことで重要な役割を果たしているという相当な証拠があるそれら。ただし、高配列相同性と同様、対立およびコピー数変化を難しく正確にすることができます設計手法と効率的に遺伝子型すべてKIRの遺伝子。従来の方法は、得られたデータの解像度、スループット、費用対効果、およびセットアップと実験の実行に要する時間通常制限されます。定量的なキール半自動入力 (qKAT) と呼ばれる手法について述べるキール軌跡のすべての遺伝子の遺伝子のコピー数を決定することができます高速多重リアルタイムのポリメラーゼ連鎖反応法であります。qKAT は、データを提供できる高解像度キールコピー数、それらを包含構造多型のハプロタイプの変化を推論するさらに使用できる単純な高スループット方法です。このコピーの数とハプロタイプのデータを式とキールとHLAとの間の機能的相互作用に関する調査と同様に大規模病協会、集団遺伝学の研究のために有益にすることができます。
概要
人間、キラー免疫グロブリン様受容体の(キール)、白血球受容体複合体 (LRC) 内 19 番染色体の長腕に遺伝子座が割り当てられています。この軌跡は、長さ約 150 kb で 15 KIR遺伝子配置頭にテールが含まれています。現在知られているKIRの遺伝子座がKIR2DL1、 KIR2DL2/KIR2DL3、 KIR2DL4、 KIR2DL5A、 KIR2DL5B KIR2DS1 5、 KIR3DL1/KIR3DS1 KIR3DL2-32 つの的偽遺伝子、 KIR2DP1 、 KIR3DP1。KIR遺伝子は二次元 (2 D) のエンコードと短い (S; 活性化) と 3次元 (3 D) 免疫グロブリン様ドメイン受容体または長い (L; 抑制) 細胞質尾、ナチュラル キラー (NK) 細胞と T のサブセットで表されるセルです。コピー数変異キール軌跡フォーム内で変数遺伝子コンテンツ1多様なハプロタイプを展示しました。近くに頭-尾の遺伝子配列と高い相同によって促進される非対立遺伝子相同組換え (NAHR) は、haplotypic 変動を担当する提案メカニズムです。100 以上の異なるハプロタイプは、人口世界中1,2,3,4で報告されています。すべてのこれらのハプロタイプは、2 つの主要なグループに分けることができる: A と B のハプロタイプ。A ハプロタイプには 7 KIRの遺伝子が含まれています: KIR3DL3、 KIR2DL1、 KIR2DL3、 KIR2DL4、 KIR3DL1、 KIR3DL2、抑制のKIR遺伝子と活性化キール遺伝子KIR2DS4。ただしをキールのホモは、ヨーロッパ起源の人の 70% にハプロタイプは専ら運ぶKIR2DS45,6の非機能的な「削除」フォーム。すべての他のKIRの遺伝子の組み合わせを形成グループ B ハプロタイプ、特定のKIR遺伝子KIR2DS1の少なくとも 1 つを含むKIR2DS2、 KIR2DS3、 KIR2DS5、 KIR3DS1、 KIR2DL2、KIR2DL5と通常 2 つ以上の活性化KIR遺伝子が含まれます。
HLA のクラス I の分子特定抑制性受容体 (KIR2DL1、 KIR2DL2、 KIR2DL3とKIR3DL1) 活性化受容体 (KIR2DS1、 KIR2DS2、 KIR2DS4の配位子として識別されています。KIR2DS5、およびKIR3DS1)、 KIR2DL4は他の抑制のキールの受容器のような長い細胞質尾が含まれていますが、共通である細胞外ドメインに近い正荷電残基がありますユニークなキールとその他の活性化のキールの受容体の機能。KIR 遺伝子と HLA 遺伝子の変異の組み合わせは、その図形潜在的な NK 細胞応答性個々 レベル7、8時を受容体リガンド相互作用に影響します。遺伝的関連研究からの証拠はキールが (例えば.、ひと免疫不全ウイルス [HIV]9 C 型肝炎ウイルス [c 型肝炎]10) ウイルスの抵抗の役割を果たしていることを示しているの移植の成功11、妊娠障害と繁殖成功12,13、同種造血幹細胞移植 (造血幹細胞移植)14,15後、再発に対する保護のリスク 16、および癌の17のリスク。
高配列相同性の対立遺伝子の組み合わせと haplotypic の多様性は、正確なジェノタイピングKIRの遺伝子の作業の課題を提示。KIR遺伝子を入力する従来の方法は、シーケンス特定のプライマー (SSP) ポリメラーゼの連鎖反応 (PCR)18,19、20、シーケンス固有オリゴヌクレオチド プローブ (SSOP) PCR21とマトリックス支援レーザ脱イオン化-時間の飛行質量分析法 (MALDI-TOF MS)22。これらの手法の欠点は、一方、実行する骨の折れるだけ個人の遺伝子型に部分的な洞察力を提供すること。最近特にキール軌跡を入力する次世代シーケンサー (NGS) が適用されています。このメソッドは非常に強力なそれは、実行する高価なことができます、詳細な解析やデータのチェックを行う時間がかかる。
qKAT は、高スループットの定量的 PCR 法です。従来の方法は手間と時間のかかる、このメソッドは 5 日間で約 1,000 のゲノム DNA (gDNA) のサンプルを実行することが可能になり、 KIR遺伝子と同様の遺伝子コピー数を与えます。qKAT から成っているそれぞれの目標 2 つのキール座位 10 の多重反応と、 KIRの遺伝子の相対的な定量化のため使用されるゲノム (STAT6) の固定コピー数の 1 つの参照の遺伝子コピー数23。この試金は遺伝的提供し同様、人口パネルや 1 型糖尿病のような自己免疫疾患、c 型肝炎などの感染症、妊娠中毒症などの妊娠障害疾患コホート研究で正常に使用されていますNK ・細胞機能1,4,24,25,26を理解することを目的とした研究を支えます。
プロトコル
1. 準備と DNA からめっき
- 吸光光度または蛍光器具を使用して gDNA 濃度を正確に測定します。
- DNA を深井戸 96 ウェル プレートの 4 ng/μ L に希釈します。知られているコピー数と少なくとも 1 つの gDNA サンプルおよび 1 つの非テンプレート コントロールが含まれます。
- 2 分の 450 x gで 96 ウェルのプレートを遠心します。
- 各ウェルに 10、qPCR 384 ウェル プレートに 4 連の各サンプルを調剤リキッドハンド リング装置を用いて DNA の ng (2.5 μ L/ウェル)。少なくとも 10 の 384 ウェル プレートを各 qKAT の反作用のための 1 つを準備します。
- GDNA は 1 つ以上の 96 ウェル プレートから調剤されて場合は、2% の漂白剤と純水 gDNA サンプルの各 96 ウェル プレート間のシステムの処理液の針をきれいにフル ボリューム洗浄を実行します。
- 少なくとも 24 時間室温で清潔で 384 ウェル プレートの孵化によって DNA を風乾します。
2. プライマーおよびプローブの準備
注: qKAT は 10 の多重反応で構成されています。各反応には、プライマーの 3 つのペアと 2 つのKIR遺伝子と 1 つの参照の遺伝子を増幅する具体的には 3 つの蛍光標識プローブが含まれています。江ら27に掲載されたプローブは、オリゴヌクレオチドが今付けられて ATTO 色素改善光安定性と長い信号の有効期間を提供するので、変更されました。前検体プライマーの組み合わせは、市販 (材料の表を参照してください)。
- 表1 希釈に従って各反作用のためのプライマー組み合わせを準備します。
- 表 1に従って各反作用のためのプローブの組み合わせを準備します。組み合わせを行う前に各それぞれのプローブをテストします。
3. マスター ミックスの準備
メモ下記のボリュームは、10 x 384 ウェル プレートのセットに qKAT 反応の 1 つを実行するため。
- 384 ウェル プレートにメッキ gDNA サンプルが完全に乾燥を確認します。氷の上のすべての手順を行い、蛍光標識プローブが写真と熱敏感なので可能な限り、光への暴露から覆われて試薬を維持します。
- QPCR バッファー、プライマーおよびプローブ 4 ° C で因数を解凍します。
- 氷の上 18.86 mL 純水 20 mL、qPCR バッファー、作り置きのプライマー組み合わせの 1,000 μ L 作り置きプローブの組み合わせ (表 2) の 180 μ L を追加することによって 10 x 384 ウェル プレート用マスター ミックスを調製します。
- 415 μ L を分注を各ウェルに、マルチ チャンネル ピペットを使用して深い 96 well プレートにマスター ミックスを均等に分散します。このプレートを光から覆われているアイス ボックスにしてください。
- 液体処理機器を使用して、乾燥された gDNA を 384 ウェル プレートの各ウェルにマスター ミックスの 9.5 μ L を調剤します。箔押しの入ったプレートをシール、すぐに 4 ° C で液体処理システムの針が各プレートの間に水で洗っていることを確認、残りの板は、このプロセスを繰り返します。
- 3 分の 450 x g で 384 ウェル プレートを遠心し、4 ° c 夜間、または 6-12 h に DNA を再懸濁します、空気の泡を放散する間、それらを孵化させなさい。
4. qPCR 測定
- 一晩インキュベート後残り空気の泡を放散する 3 分の 450 × gで遠心分離機します。
- 自動化のために、qPCR マシン (例えば、LightCycler 480) をマイクロ プレート ハンドラーに接続 (材料の表を参照してください)。プログラムは、光から保護冷却貯蔵ドックから qPCR マシンにプレートを配置するマイクロ プレート ハンドラー。
注 法、理論的で動作するはず他の qPCR のマシンに互換性のある光設定。 - 次のサイクリング条件を使用: 95 ° C、5 分が 95 ° C の 40 のサイクルが続く 15 s の 66 ° C、50 秒、66 ° C でデータ収集を
- 実行が完了したら、一度、qPCR マシンからプレートを収集し、破棄ドックのロボットを持っています。
5. 実行後の分析
- 増幅後 qPCR マシンのソフトウェアで、2 番目の派生最大メソッドまたはフィット点メソッドを使用して定量化サイクル (Cq) 値を計算 (材料の表を参照)、以下の手順します。
- QPCR ソフトウェアを開き、1 つのプレートの保存された反応実験ファイルナビゲーターのタブで開きます。
-
2 番目の派生最大法を用いた解析、[分析] タブを選択し、Abs Quant/Second 誘導体 Max メソッドを使用して新しい分析を作成します。
- 作成新しい解析] ウィンドウで、[解析タイプを選択: Abs Quant/Second 誘導体 Max メソッドサブセット:すべてのサンプルプログラム:増幅名前: Rx DFO ( xは反応数)。
- フィルター櫛とヴィック/六角/Yellow555 (533-580)を選択します。これにより、 STAT6で収集されたデータが選択されています。
- VIC/HEX/Yellow555(533-580)のカラー補正を選択します。計算をクリックします。ファム (465-510) と Cy5/Cy5.5(618-660) のため、この手順を繰り返します。ファイルを保存をクリックします。
-
フィット点法を用いた解析、解析タブで Abs クオンツ/フィット点を選択します。
- 作成新しい解析] ウィンドウで、[解析タイプを選択: Abs クオンツ/フィット点法サブセット:すべてのサンプルプログラム:増幅名前: RxF DFO ( xは反応数)。
- 適切なフィルターとSTAT6と各KIR遺伝子 (ファム/cy5 の組合せ) のカラー補正を選択します。[ Noiseband ] タブでバック グラウンド ノイズを除外するノイズ ・ バンドを設定します。
- [分析] タブで、フィット点を3に設定し、ショーのフィット点を選択します。計算をクリックします。ファイルを保存をクリックします。
6. 結果のエクスポート
- QPCR ソフトウェアでナビゲーターを開きます結果バッチ書き出し」タブを選択します。
- 実験ファイルが保存、ウィンドウの右側にあるセクションに、ファイルを転送するフォルダーを開きます。[次へ] をクリックします。名前とエクスポート ファイルの場所を選択します。
- Abs Quant/Second 誘導体 Max メソッドまたはAbs クオンツ/フィット点の解析タイプを選択します。[次へ] をクリックします。ファイル、エクスポート フォルダー、および解析のタイプの名前が正しいことと、次のエクスポート プロセスを開始するをクリックを確認します。
- エクスポートのステータス]が[ok]になるまで待ちます。画面は、次のステップに自動的に移動します。選択したすべてのファイルが正常にエクスポートされましたを確認して失敗したファイルの数 = 0。完了をクリックします。
- 各プレートの個々 の反作用にエクスポートされた板を分割するのにスクリプト split_file.pl と roche2sds.pl を使用します。
注 スクリプトが要求/GitHub 上で提供されます。
7. コピー数の計算
- コピー数解析ソフトウェア (例えば、CopyCaller) を開きます。リアルタイム PCR の結果ファイルのインポートを選択し、 roche2sds.plで作成したテキスト ファイルをロードします。
- 分析を選択し、いずれか選択する知られているコピー数と校正サンプルまたは最も頻繁にコピーの番号を選択して、分析を実施します。通常ヨーロッパ起源の集団で観察されたKIR遺伝子の最も頻繁なコピー数の表 5を参照してください。
8. データの品質をチェックします。
- R スクリプトKIR_CNVdata_analysis_for_Excel_ver020215 を使用します。Rすべての板からコピー数データをスプレッドシートに結合します。
注 スクリプトが要求/GitHub 上で提供されます。 - KIR遺伝子 (表 6) 知られている連鎖不平衡 (LD) に準拠していないサンプルのコピー数解析ソフトウェア上の raw データを再確認します。
結果
コピー数解析は ΔΔCq 法に基づく予測と推定されるコピー数を示しますコピー数解析ソフトウェアにファイルをエクスポートすることによって実施することができます。
コピー数は、コントロール DNA サンプル プレートまたは最も頻繁に遺伝子コピー数 (表 5) を入力することで知られているコピー数に基づいて予測できます。図 1に示しますKIR2DL4 KIR3DS1、参照の遺伝子を対象とする反応のプレートの結果STAT6 。KIR3DS1、活性化遺伝子の最も頻繁なコピー数は 1 つのコピーに対し、 KIR2DL4、 キール軌跡のフレームワーク遺伝子の最も頻繁なコピー数は 2 つのコピーです。図の結果は、qPCR ソフトウェアと qPCR データから生成されたコピー数データで観測された PCR の拡大のプロットを表示します。示すように、アッセイは 0、1、2、3、および 4 のKIR遺伝子のコピー数を区別することができます。コピー数解析ソフトウェアが円グラフまたは棒グラフとして板全体コピー数の分布を表示できます。コピー数予測の有効性が低い高コピー数とサンプルです。
反応、gDNA、バッファー、プライマーおよびプローブで使用されるすべての材料の品質は、得られる結果の精度に影響を与えます。ただし、結果の不一致はプレート間で DNA の濃度の変動に起因する可能性が高いです。260/280 と 260 を使用して測定することができます抽出された gDNA の純度/230 比が品質に及ぼす影響を持つことができます。2 2.2 1.8 2、260/230 比の 260/280 の比率が望ましいです。プレート間で不均一な範囲の DNA 濃度高い可変性しきい値サイクル (Ct) サンプルと推定されるコピー数の範囲における不一致の可能性があります。図 2の結果は、効果を示すプレート間で Ct値間の格差はコピー数の予測の精度を持つことができます。赤い線はサンプル推定コピー数の範囲を示し、理想的には、可能な限り整数に近いする必要があります。
数値データをコピーするには、一度、分析は、96 ウェル フォーマットでスプレッドシート ファイルとしてエクスポートできます。R スクリプト (要リクエスト) を使って 1 つのワークシートにセットとして実行されるすべての 10 皿のコピー番号データを結合です。ほとんどヨーロッパ起源集団からKIRsについて公開されたデータキール複雑な1に様々 な遺伝子との間に存在する LD ルールの予測ができます。これらの予測を使用して、コピー数結果 (表 6) に下流のチェックを実施します。異常な多態性や haplotypic の構造変化、遺伝子間予測の LD に準拠していないサンプルが含まれます。プロトコルを記述するフローチャートを図 3に示します。
キールのハプロタイプの識別子 (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) と呼ばれるツールは、データ セットからハプロタイプの補完を容易にするために開発されました。帰属は、ヨーロッパ起源の人口1に見られた参照ハプロタイプのリストに基づいて動作します。ただし、代わりに使用する、ツールは参照ハプロタイプのカスタム セットにもできます。3 つのファイルが生成されます。最初のファイルは、サンプルのすべてのハプロタイプの組み合わせを一覧表示、2 番目のファイルを提供しています最高の結合された周波数を持つハプロタイプの組み合わせのトリム一覧、3 番目のファイルはハプロタイプを割り当てることができないサンプルを示します。ハプロタイプの非割り当ては新規ハプロタイプのインジケーターとして使用できます。
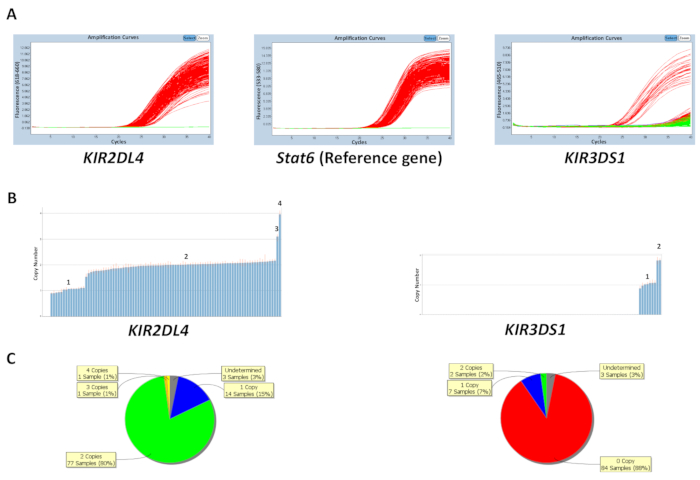
図 1: 反応プレートの代表の結果数 5 。(A) このパネル ショー増幅プロット。(B) このパネルを示していますコピー数をプロットします。(C) このパネルは、コピー数の分布を示しています。この図の拡大版を表示するのにはここをクリックしてください。
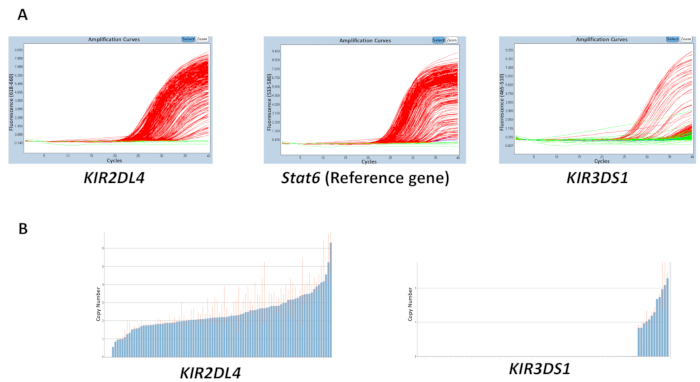
図 2: 反応変数 DNA 濃度を有する平板の代表の結果数 5 。(A) このパネル ショー増幅プロット。(B) このパネルを示していますコピー数をプロットします。この図の拡大版を表示するのにはここをクリックしてください。
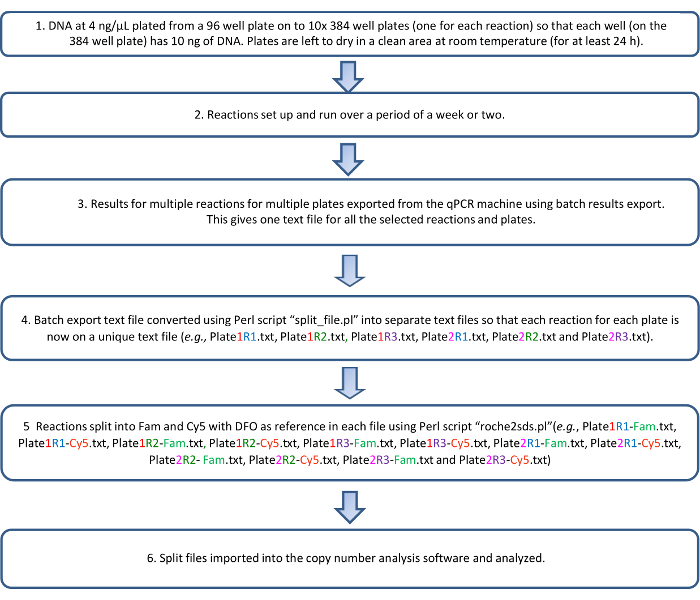
図 3: qKAT プロトコルのフローチャート。この図の拡大版を表示するのにはここをクリックしてください。
| アッセイ | 遺伝子 | 前方のプライマー | 濃度 (nM) | 逆のプライマー | 濃度 (nM) | プローブ | 濃度 (nM) |
| いいえ 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2 DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| なし 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2 DL 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 3 | 3 DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 4 | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 5 | 3DS1 | 地下 2 階 | 250 | B1R | 250 | P4b | 150 |
| 2 DL 4 | C1F | 200 | C1R | 200 | P5b-2 DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 6 | 2 DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| 2 DL 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 8 | 2DS3 | B5F | 250 | B5R | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | D5R | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| いいえ 10 | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
表 1: 組み合わせとプライマーおよびプローブの各 qKAT の反作用で使用される濃度27.
| 反応 | プライマー因数 (μ L) | プローブ因数 (μ L) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | 水 | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2 DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | 水 | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2 DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| 注: 必要が少ない、マスター ミックスで水 20 μ L | |||||||||||
| R3 | 3 DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | 水 | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | 水 | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | 地下 2 階 | B1R | C1F | C1R | 水 | STAT6F | STAT6R | P4B | P5B-2 L 4 | PSTAT6 |
| 2 DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2 DL 1 | B3F | B3R | D3F | D3R | 水 | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | 水 | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2 DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | 水 | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | 水 | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | 水 | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
表 2: ボリューム (μ L) 100 μ M プライマー ・ プローブ溶液のプライマーし、プローブの組み合わせ因数。
| 名 | 方向 | 5´ 変更 | 3´ 変更 | シーケンス (5' →3') | 長さ | Tm | GC % | エクソン | 位置 |
| P4a | 感覚 | ファム | BHQ 1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44.4 | 4 | 425 451 |
| P4b | アンチセンス | ファム | BHQ 1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5b | 感覚 | ATTO647N | BHQ 2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828-852 |
| P5b-2 DL 4 | 感覚 | ATTO647N | BHQ 2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828-852 |
| P9 | 感覚 | ATTO647N | BHQ 2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62.5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ 2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
テーブル 3: qKAT で使用されているプローブのリスト 1, 27. ATTO 色素にオリゴ プローブ P5b、P5b-2 DL 4、P9、および PSTAT6 の 5' 端に使用される蛍光染料が変更されました。
| 遺伝子 | プライマー | 方向 | シーケンス (5´-3´) | 長さ | Tm | GC % | エクソン | 位置 | 私アンプリコン (bp) | 対立遺伝子が失われる可能性が | ||
| 3DL2e4 | A1F | フォワード | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3 DL 2 * 008 * 021 * 027、* 038。 | ||
| A1R | 逆 | CTGCAAGGACAG GCATCAA | 19 | 53 | 52.6 | 559-577 | 3 DL 2 * 048 | |||||
| 3DP1 | A4F | フォワード | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398 416 | 112 | どれも | ||
| A5R | 逆 | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55.6 | 492-509 | どれも | |||||
| 2DS2 | A2F | フォワード | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398 416 | 111 | どれも | ||
| A6R | 逆 | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42.9 | 488-508 | どれも | |||||
| 3 DL 3 | A8Fa | フォワード | GTGAAATCGGGA GAGACG | 18 | 50 | 55.6 | 4 | 406-423 | 139 | どれも | ||
| A8Fb | フォワード | GGTGAAATCAGG AGAGACG | 19 | 50 | 52.6 | 405 423 | 3 DL 3 * 054 3 DL 3 * 00905。 | |||||
| A8R | 逆 | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | どれも | |||||
| 3DL1e4 | B1F | フォワード | CATCGGTCCCAT GATGCT | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3 DL 3 DL 1 * 006 3 DL 1 * 054 3 DL 3 DL 1 * 089 1 * 086 1 * 00505 | ||
| B1R | 逆 | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3 DL 1 * 00502 | |||||
| 3DS1 | 地下 2 階 | フォワード | CATCGGTTCCAT GATGCG | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DS1 * 047;3 DL 1 * 054 を拾うことがあります。 | ||
| B1R | 逆 | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | どれも | |||||
| 2 DL 1 | B3F | フォワード | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | 2 DL 2 DL 1 * 028 1 * 020 | ||
| B3R | 逆 | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | 2 DL 2 DL 1 * 030 2 DL 1 * 029 1 * 023 | |||||
| 2DS1 | B4F | フォワード | TCTCCATCAGTC GCATGAA | 19 | 51 | 47.4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | 逆 | GGTCACTGGGAG CTGAC | 17 | 49 | 64.7 | 624-640 | どれも | |||||
| 2DS3 | B5F | フォワード | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546 563 | 96 | どれも | ||
| B5R | 逆 | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | どれも | |||||
| 2DS5 | B6F2 | フォワード | AGAGAGGGGACG TTTAACC | 19 | 50 | 52.6 | 4 | 475-493 | 173 | どれも | ||
| B6R3 | 逆 | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66.7 | 630-647 | 2DS5 * 003 | |||||
| 2 DL 4 | C1F | フォワード | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55.6 | 5 | 808 825 | 83 | どれも | ||
| C1R | 逆 | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52.6 | 872-890 | 2 DL 2 DL 4 * 019 4 * 018 | |||||
| 2 DL 2 | 2DL2F4 | フォワード | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57.9 | 5 | 778-796 | 151 | 2 DL 2 * 009;782 G を A に変更 | ||
| C3R2 | 逆 | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | どれも | |||||
| 2DS4 | C5F | フォワード | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | どれも | ||
| C5R | 逆 | TTGACCACTCGT AGGGAGC | 19 | 52 | 57.9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | フォワード | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57.9 | 5 | 750-768 | 203 | どれも | ||
| 2DS4R2 | 逆 | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | どれも | |||||
| 2DS4FL | 2DS4FL | フォワード | CCGGAGCTCCTA TGACATG | 19 | 53 | 57.9 | 5 | 744-762 | 209 | どれも | ||
| 2DS4R2 | 逆 | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | どれも | |||||
| 2 DL 3 | D1F | フォワード | AGACCCTCAGGA GGTGA | 17 | 48 | 58.8 | 9 | 1180-1196 | 156 | どれも | ||
| D1R | 逆 | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 3 * 01802 2 DL 2 DL 3 * 01801、2 DL 3 * 017 2 DL 3 * 010 | |||||
| 2 DL 5 | D2F | フォワード | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 と 2DL5B * 020 | ||
| D2R | 逆 | GGCAGGAGACAA TGATCTT | 19 | 49 | 47.4 | 1315-1333 | どれも | |||||
| 2DP1 | D3F | フォワード | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | どれも | ||
| D3R | 逆 | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | どれも | |||||
| 3DL1e9 | D4F | フォワード | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 93 | 3 DL 3 DL 1 * 068 1 * 061 | ||
| D4R2 | 逆 | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43.5 | 1273 1295 | 3 DL 3 DL 1 * 05902 3 DL 1 * 060 3 DL 3 DL 1 * 065, 3 DL 1 * 094N 3 DL 1 * 098 3 DL 1 * 064 1 * 061 1 * 05901 | |||||
| 3DL2e9 | D4F | フォワード | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 156 | どれも | ||
| D5R | 逆 | GACCTGACTGTG GTGCTCG | 19 | 54 | 63.2 | 1340-1358 | どれも | |||||
| STAT6 | STAT6F | フォワード | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129 | |||||
| STAT6R | 逆 | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
表 4: qKAT で使用されるプライマーのシーケンス 1, 27 。
| キール遺伝子 | 3 DL 3 | 2DS2 | 2 DL 2 | 2 DL 3 | 2DP1 | 2 DL 1 | 3DP1 | 2 DL 4 | 3 DL 1 EX9 | 3 DL 1 EX9 | 3DS1 | 2 DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 合計 | 2DS4 フロリダ | 2DS4 デル | 3 DL 2 ex4 | 3 DL 2 EX9 | |
| 最も頻繁なコピー数 | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
表 5:ための最も頻繁なコピー数キールヨーロッパ起源のサンプルで一般的に観察される遺伝子。
| 不均衡のリンケージの規則 qKAT のヨーロッパの人口に基づく | コピーの番号の確認 | |||||||
| 1 | KIR3DL3、KIR3DP1、KIR2DL4 、 KIR3DL2は、フレームワーク遺伝子両方のハプロタイプの存在です。 | KIR3DL3、KIR3DP1、KIR2DL4 、 KIR3DL2 = 2 | ||||||
| 2 | お互いに LD、 KIR2DS2 、 KIR2DL2 | 2DS2=2 DL 2 | ||||||
| 3 | KIR2DL2とKIR2DL3が同じ遺伝子の対立遺伝子です。 | 2 DL 2+2 DL 3= 2 | ||||||
| 4 | お互いに LD、 KIR2DP1とKIR2DL1 | 2DP1=2 DL 1 | ||||||
| 5 | KIR3DL1とKIR3DL2の exon 4 それぞれエクソン 9 KIR3DL1とKIR3DL2のと同じです。 | 3DL1ex4=3DL1ex9 、 3DL2ex4=3DL2ex9 | ||||||
| 6 | 対立遺伝子は、 KIR3DS1 KIR3DL1 | 3 DL 1+3DS1= 2 | ||||||
| 7 | KIR2DS3とKIR2DS5は、 KIR2DL5と LD | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1とKIR2DS1は、LD | 3DS1=2DS1 | ||||||
| 9 | KIR2DS1とKIR2DS4Tの計の存在は相互に排他的なハプロタイプ | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FLとKIR2DS4delは、 KIR2DS4TOTALの亜種 | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
表 6: 間の連鎖不平衡キール一般的にヨーロッパ起源の人口で観察される遺伝子コピー数データを確認する使用できます。 1,27 。
ディスカッション
KIR遺伝子.のコピー数の入力を容易にする qKAT と呼ばれる新規半自動化されたハイスループット法について述べるメソッドは、低スループットおよびこれらの非常に多型の遺伝子の有無を示すことができますのみ SSP PCR のような従来の方法と比べて改善。
得られた数値データをコピーの精度が品質と gDNA サンプルおよび試薬の品質の均一性の濃度を含む複数の要因に依存しています。品質とプレート間で gDNA サンプルの精度は、プレート間の濃度の変化は、コピー数の計算でエラーが発生するので非常に重要です。以来、法は、ヨーロッパ起源のサンプル セットを使用して検証された、世界の他の部分からのコホートからデータはより徹底的にチェックを必要です。これは、対立遺伝子ドロップ アウトまたは非特異的プライマー ・ プローブ バインディングのインスタンスが誤ってコピー数変異として解釈されないことを確保するためです。
法は、設計され、高スループットとして実行するために最適化された、間は、以下のサンプルを実行する変更できます。少ないサンプルを分析する際、コピー数解析ソフトウェアで信頼の指標が影響を受けるが、プレートに知られているKIR遺伝子コピー数と制御ゲノム DNA のサンプルが含まれ、その他のサンプルの複製がある場合、これは向上させることが含まれています。
液体/プレート-ハンドリング ロボットなし所マルチ チャンネル ピペットの使用のマスターの組合せを分配することができます、プレートは qPCR 測定器に手動で読み込むことができます。
QKAT の開発の後ろの主な目的は、メソッドを作成する単純な高スループット、高解像度、およびコスト効率の高い遺伝子KIRs病協会研究。QKAT は、感染症、自己免疫疾患、妊娠障害4,の範囲を含むいくつかの大きい疾患関連研究におけるキールの役割の調査に採用されているので、これは正常に達成されました。24,25,26。
開示事項
著者が明らかに何もありません。
謝辞
医療研究評議会 (MRC)、欧州連合のホライゾン 2020年研究と技術革新プログラム (グラント契約号 695551) の下で欧州研究会議 (ERC)、国立衛生研究所 (NIH) ケンブリッジからの資金援助を受けたプロジェクト生物医学の研究センターと NIH 研究血液・移植研究ユニット (NIHR BTRU) 臓器提供とケンブリッジ大学の移植および NHS の血および移植 (NHSBT) とのパートナーシップ。見解では、これらの者とは必ずしもこれらの NHS、NIHR、保健省や、NHSBT。
資料
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
参考文献
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved