Method Article
qKAT: Semi-automated כמותיים הקלדה של הרוצח-תא גנים קולטני דמוי נוגדן
In This Article
Summary
תא הרג כמותיים קולטן דמוי אימונוגלובולינים (קיר) חצי אוטומטיות הקלדה (qKAT) היא שיטה פשוטה, תפוקה גבוהה וחסכונית להעתיק מספר גנים "קיר" סוג של היישום שלהם במחקרים האגודה האוכלוסייה ומחלות.
Abstract
תא הרג בנוגדנים דמוי קולטנים (KIRs) הם קבוצה של רצפטורים מעכבות, הפעלת המערכת החיסונית, על הרוצח הטבעיים (NK) ותאי T, מקודד על ידי מקבץ רב שלבית של גנים על כרומוזום 19. ליגנדים מאופיין ביותר שלהם הם מולקולות אנטיגן (הלע) לויקוציטים אנושיים מקודדים בתוך histocompatibility הגדולות מורכבות (MHC) לוקוס על כרומוזום 6. יש עדויות משמעותיות כי הם לשחק תפקיד משמעותי חסינות, רבייה, ההשתלה, שהופך אותו מכריע יש טכניקות יכולים במדויק גנוטיפ אותם. עם זאת, הומולוגיה גבוהה-רצף, כמו גם כמו allelic, וריאציה מס העתק, להקשות על שיטות עיצוב יכול בצורה מדויקת ויעילה גנוטיפ כל הגנים "קיר" . שיטות מסורתיות מוגבלים בדרך כלל את הרזולוציה של נתונים שהושגו, תפוקה, משתלמת, הזמן שנדרשו עבור התקנה והפעלה הניסויים. אנו מתארים שיטה הנקראת כמותיות וקיר חצי אוטומטיות הקלדה (qKAT), אשר היא שיטה תגובת שרשרת פולימראז בזמן אמת מולטיפלקס תפוקה גבוהה שיכול לקבוע את מספרי עותק ג'ין עבור כל הגנים במיקומה "קיר" . qKAT היא שיטה פשוטה תפוקה גבוהה שיכול לספק ברזולוציה גבוהה וקיר עותק מספר נתונים, אשר יכול לשמש עוד יותר להסיק הווריאציות haplotypes רב-צורתיות מבנית, שמעבירים אותם. מספר זה העתק נתונים haplotype יכול להועיל במחקרים על עמותות המחלה בקנה מידה גדול, גנטיקה של אוכלוסיות, כמו גם חקירות על הביטוי ואינטראקציות פונקציונלי בין קיר הלע.
Introduction
בבני אדם, הקולטן בנוגדנים כמו רוצח("קיר") לוקוס ממופה על הזרוע הארוכה של כרומוזום 19 בתוך הקולטן ליקוציט מורכבים (LRC). לוקוס זה בסביבות 150 kb אורך וכוללת 15 וקיר הגנים מסודרים ראש זנב. לוקוסים וקיר הידועות כיום הם KIR2DL1, KIR2DL2/KIR2DL3, KIR2DL4, KIR2DL5A, KIR2DL5B, KIR2DS1-5, KIR3DL1/KIR3DS1, KIR3DL2-3, שני pseudogenes, KIR2DP1 ו- KIR3DP1. הגנים וקיר קידוד עבור דו מימדי (2D) ואת רצפטורים תלת מימדי (3D) בנוגדנים כמו תחום עם קצר (S; הפעלת) או ארוכים (L; מעכבות) cytoplasmic זנבות, הבאים לידי ביטוי על ידי תאים הרוצח הטבעיים (NK) קבוצות משנה של T תאים. וריאציה מספר עותק הציג בתוך הצורות לוקוס וקיר haplotypes מגוונת עם תוכן משתנה ג'ין1. Non-allelic רקומבינציה הומולוגית (לבנון), בהנחייתם של סידור הגן ראש זנב קרוב והומולוגיה גבוהה-רצף, הוא המנגנון המוצע להיות אחראי על ההשתנות haplotypic. מעל 100 haplotypes שונות דווחו אוכלוסיות ברחבי העולם1,2,3,4. כל haplotypes האלה יכול להיות מחולק לשתי קבוצות עיקריות: A ו- B haplotypes. A haplotype מכיל גנים קיר 7: KIR3DL3, KIR2DL1, KIR2DL3, KIR2DL4, KIR3DL1, KIR3DL2, ואשר הינם גנים וקיר מעכבות, את הפעלת "קיר" ג'ין KIR2DS4. עם זאת, למעלה עד 70% של אנשים ממוצא אירופי homozygous על קיר haplotype A באופן בלעדי לשאת צורה פונקציונלי "מחיקת" של5, KIR2DS46. כל השילובים גנים אחרים וקיר בצורת haplotypes קבוצה B, כולל לפחות באחד מהגנים קיר מסוים KIR2DS1, KIR2DS2, KIR2DS3, KIR2DS5, KIR3DS1, KIR2DL2, KIR2DL5, וכוללים בדרך כלל שניים או יותר גנים "קיר" הפעלת.
הלע מחלקה אני מולקולות זוהו על ליגנדים עבור מסוימים רצפטורים מעכבים (KIR2DL1, KIR2DL2, KIR2DL3, KIR3DL1), הפעלת קולטנים (KIR2DS1, KIR2DS2, KIR2DS4, KIR2DS5, ו KIR3DS1), KIR2DL4, אשר הוא ייחודי קיר מכיל של זנב ארוך cytoplasmic כמו אחרים קולטנים וקיר מעכבות, אך גם יש משקע הטעון חיובית ליד המחשבים חוץ-תאית אשר נפוצה תכונה של אחרים הפעלת קולטני "קיר" . השילוב של משתנים בתוך הגנים "קיר", הגנים הלע משפיע על האינטראקציה ליגנד לקולטן הזה צורות אפשריות NK תא תגובתיות-ברמת הפרט7,8. עדויות ממחקרים שיוך גנטי ציין כי וקיר משחק תפקיד ההתנגדות ויראלי (למשל., וירוס הכשל החיסוני האנושי [HIV]9 ו הפטיטיס C וירוס [בזכות]10), ההצלחה של השתלת 11, הסיכון של הפרעות הריון12,הצלחה הרבייה13, הגנה מפני נסיגה לאחר14,(HSCT) השתלת תא גזע allogeneic hematopoietic15, 16, את הסיכון של סרטן17.
השילוב של high-רצף הומולוגיה, allelic ואתגרים haplotypic גיוון מתנות במשימה של מדויק genotyping וקיר גנים. שיטות קונבנציונליות כדי להקליד "קיר" הגנים כוללים רצף ספציפי פריימר (SSP) פולימראז תגובת שרשרת (PCR)18,19,20, רצף ספציפי oligonucleotide בדיקה PCR (SSOP)21, ו לייזר בסיוע מטריקס desorption יינון-שעת הטיסה ספקטרומטר מסה (MALDI-TOF MS)22. החסרונות של טכניקות אלה הן רק הם מספקים תובנה חלקית גנוטיפ של הפרט אך גם מפרך כדי לבצע. לאחרונה הוחל הדור הבא רצפי (הגדרות) כדי להקליד את לוקוס וקיר במיוחד. בעוד ששיטה זו היא רבת עוצמה, זה יכול להיות יקר כדי להפעיל, וזה זמן רב כדי לערוך בדיקות מעמיקה בניתוח ונתונים.
qKAT היא שיטת ה-PCR כמותי תפוקה גבוהה. בעוד שיטות קונבנציונליות מפרך וצורך, שיטה זו מאפשרת להפעיל כמעט 1,000 דגימות דנ א (gDNA) גנומית בחמישה ימים ונותן של גנוטיפ "קיר" , כמו גם את מספר עותק גנטי. qKAT מורכב עשר תגובות מולטיפלקס, שכל אחד מהם מטרות שני לוקוסים וקיר והעתק אחד הגנים הפניה של מספר עותק קבוע של הגנום (STAT6) משמש כימות היחסי של הגן וקיר מספר23. Assay הזה שימש בהצלחה בלימודי מעורבים לוחות אוכלוסייה גדולה לבין המחלה המפחידה על מחלות זיהומיות כגון בזכות, מחלות אוטואימוניות כמו בסוכרת מסוג 1, הריון הפרעות כגון רעלת, וכן מתן גנטי לגורם המרכזי עליו מושתתת על מחקרים להבנת NK תא פונקציה1,4,24,25,26.
Protocol
1. ציפוי מתוך ה-DNA והכנה
- לכמת במדויק את הריכוז gDNA שימוש בכלי spectrophotometric או fluorometric.
- לדלל את הדנ א כדי ng/µL 4 על צלחת עמוקה 96-טוב-טוב. כוללים אחד לפחות שליטה מדגם gDNA עם מספר עותק הידוע ושליטה שאינם מתבנית אחת.
- Centrifuge הלוחות 96-ובכן ב x 450 גרם למשך 2 דקות.
- שימוש בכלי טיפול בנוזלים, לוותר על כל מדגם ב ארבע פעמים על גבי לוחות qPCR 384-ובכן כך בכל טוב יש 10 ng של DNA (2.5 µL טוב). להכין לפחות 10 צלחות 384-. ובכן, אחד עבור כל תגובה qKAT.
- אם הוא להיות בגליל gDNA באמצעות גלופה 96-ובכן אחד או יותר, לבצע שטיפה מלא בנפח 2% אקונומיקה ומים הנדסה גנטית כדי לנקות את המחטים של הנוזל טיפול מערכת בין כל צלחת 96-ובכן דוגמאות gDNA.
- מילה נהדרת ה-DNA על ידי המקננת הלוחות 384-ובכן באזור נקי בטמפרטורת החדר במשך לפחות 24 שעות.
2. הכנת תחל וזונדים
הערה: qKAT מורכב תגובות מולטיפלקס עשר. כל התגובה כוללת שלושה זוגות פריימר וזונדים שלושה התווית על-ידי קרינה פלואורסצנטית המדגישים במיוחד שני גנים וקיר וג'ין הפניה אחת. הגששים שפורסמו בשנת ג'יאנג ואח27 שונו כך oligonucleotides מסומנות כעת עם צבעי אטו מאז הם מציעים פוטוסטביליטי משופרת ואורך זמן שידור. שילובים פריימר Pre-aliquoted הינם זמינים מסחרית (ראו טבלה של חומרים).
- הכנת תחל השילובים עבור כל תגובה לפי דילולים נתון בטבלה 1.
- להכין שילובים בדיקה עבור כל תגובה לפי טבלה 1. בדיקת כל בדיקה בודדים לפני קבלת השילוב.
3. הכנת המיקס מאסטר
הערה אמצעי האחסון המוזכרים להלן הם עבור ביצוע התגובה qKAT אחד על קבוצה של 10 x 384-ובכן צלחות.
- להבטיח כי הדגימות gDNA מצופה על לוחות 384-ובכן יבש לחלוטין. לנהל את כל השלבים על הקרח ולשמור את ריאגנטים מכוסה מחשיפה לאור ככל האפשר מאחר התווית על-ידי קרינה פלואורסצנטית הגששים צילום התרמו-והרגיש.
- מפשירים את מאגר qPCR, פריימר ולאחר בדיקה aliquots ב 4 º C.
- על קרח, להכין תערובת בסיס 10 x 384-ובכן הצלחות על-ידי הוספת 18.86 מ ל מים הנדסה גנטית, 20 מ"ל של מאגר qPCR, µL 1,000 שילוב פריימר preprepared µL 180 שילוב בדיקה preprepared (טבלה 2).
- פזר את תערובת הבסיס באופן שווה על פני צלחת 96-עמוק היטב באמצעות פיפטה של רב ערוצית, pipetting 415 µL לבאר כל. לשמור על הצלחת הזו בתיבת קרח מכוסה אור.
- שימוש בכלי טיפול בנוזלים, לוותר על µL 9.5 של המיקס מאסטר לבאר כל צלחת 384-טוב עם gDNA מיובשים. לאטום את הצלחת בנייר ולמקם אותו מיד ב 4 º C. חזור על תהליך זה עבור לוחות הנותרים, המבטיח כי המחטים של הנוזל טיפול מערכת נשטפים במים בין כל צלחת.
- Centrifuge הלוחות 384-ובכן ב 450 x g למשך 3 דקות, דגירה אותם ב 4 מעלות צלזיוס למשך הלילה או בין 6-12 שעות כדי resuspend את ה-DNA וכדי להפיג כל בועות האוויר.
4. qPCR Assay
- בעקבות הדגירה לילה, צנטריפוגה ב x 450 גרם במשך 3 דקות לפרוק את כל בועות האוויר הנותרים.
- למטרות של אוטומציה, לחבר את המכונה qPCR (למשל, LightCycler 480) מלווה microplate (ראה טבלה של חומרים). תוכנית המטפל microplate כדי למקם את הצלחות לתוך המכונה qPCR מן מזח אחסון מקורר זה מוגן מפני אור.
הערה מבחני צריך, בתיאוריה, לעבוד על מכונות אחרות qPCR עם הגדרות אופטיים תואם. - השתמש אופניים התנאים הבאים: 95 מעלות צלזיוס למשך 5 דקות ואחריו 40 מחזורי 95 ° C עבור 15 s ו- C ° 66 תמורת 50 s, עם איסוף נתונים ב 66 º C.
- לאחר סיום ההפעלה, יש את הרובוט לאסוף את הצלחת מהמכונה qPCR ולמקם אותו לרציף להשליך.
5. פוסט-לרוץ לניתוח
- לאחר הגברה, לחשב את הערכים כימות מחזור (סי) באמצעות השיטה השניה המרבי נגזרות או השיטה מתאימה נקודות עם התוכנה של המכונה qPCR (ראה טבלה של חומרים), ביצוע השלבים הבאים.
- פתח את תוכנת qPCR, בכרטיסייה ' ניווט ', פתח את הקובץ ניסוי התגובה שנשמרו עבור לוח אחד.
-
על הניתוח באמצעות השיטה השניה המרבי נגזרות, בחר בכרטיסיה ניתוח, וליצור ניתוח חדש בשיטת Abs Quant/Second נגזרת מקס.
- בחלון ' יצירת ניתוח חדש ', בחר סוג ניתוח: Abs Quant/Second נגזרת מקס שיטה, משנה: כל דגימות, תוכנית: הגברה, שם: Rx-DFO (כאשר x הוא מספר התגובה).
- בחר מסנן מסרק ובחר הקס/ויק/Yellow555 (533-580). פעולה זו מבטיחה כי הנתונים שנאספו עבור STAT6 נבחרה.
- בחר צבע פיצוי עבור VIC/HEX/Yellow555(533-580). לחץ על חשב. חזור על הפעולה עבור המשפחה (465-510) ו- Cy5/Cy5.5(618-660). לחץ על שמור קובץ.
-
לניתוח בשיטת נקודות מתאים, בחר נקודות חשבים/Fit Abs בכרטיסיה ניתוח.
- בחלון ' יצירת ניתוח חדש ', בחר סוג ניתוח: נקודות חשבים/Fit Abs שיטה, משנה: כל דגימות, תוכנית: הגברה, שם: RxF-DFO (כאשר x הוא מספר התגובה).
- בחר את המסננים הנכונה ואת צבע פיצויים עבור STAT6 וכל אחת של גנים "קיר" (Fam/Cy5). בכרטיסייה ' Noiseband ', הגדר את להקת רעש כדי לא לכלול את רעשי הרקע.
- בכרטיסייה ' ניתוח ', הגדר את הנקודות מתאים ל- 3 ובחר שהצג מתאים נקודות. לחץ על חשב. לחץ על שמור קובץ.
6. ייצוא של תוצאות
- בהתוכנה qPCR, פתח את הנווט בכרטיסיית לבחור לייצא את התוצאות אצווה.
- פתח את התיקייה שבה הקבצים ניסוי נשמרות, להעביר את הקבצים לתוך המקטע מצד ימין של החלון. לחץ על הבא. בחר את השם והמיקום של קובץ הייצוא.
- בחר סוג ניתוח Abs Quant/Second נגזרת מקס שיטה או Abs חשבים/Fit נקודות. לחץ על הבא. בדוק השם של הקובץ, התיקיה לייצא את סוג ניתוח הן נכונות, לחץ על הבא כדי להתחיל את תהליך הייצוא.
- המתן עד מצב ייצוא הוא בסדר. המסך יעבור אוטומטית לשלב הבא. . תבדוק את כל הקבצים שנבחרו יוצאו בהצלחה כך מספר הקבצים נכשל = 0. לחץ על לעשות.
- להשתמש בקבצי script split_file.pl roche2sds.pl כדי לפצל את הצלחות מיוצא תגובות בודדות עבור כל צלחת.
הערה קבצי ה-script מסופקים על פי בקשה/GitHub.
7. העתק מספר חישובים
- פתח את מספר ניתוח התוכנה להעתיק (לדוגמה, CopyCaller). בחר ייבוא קובץ תוצאות ה-PCR בזמן אמת , לטעון קבצי טקסט שנוצר על-ידי roche2sds.pl.
- בחרו נתח , לערוך את הניתוח על-ידי גם בחירת כיל לדוגמה עם עותק הידוע מספר או על-ידי בחירת מספר עותק בתדירות הגבוהה ביותר. ראה טבלה 5 עבור המספר עותק בתדירות הגבוהה ביותר של גנים "קיר" בדרך כלל נצפתה באוכלוסיות ממוצא אירופי.
8. איכות הנתונים בודק
- להשתמש בקובץ script R KIR_CNVdata_analysis_for_Excel_ver020215. R לשלב העתק נתונים מספר הצלחות לתוך גיליון אלקטרוני.
הערה קבצי ה-script מסופקים על פי בקשה/GitHub. - בדוק מחדש את הנתונים הגולמיים על התוכנה ניתוח מספר עותק עבור דגימות לא תואמות הידוע תאחיזה לא שוויונית (LD) עבור קיר גנים (טבלה 6).
תוצאות
ניתוח מספר עותק יכול להתבצע על-ידי ייצוא הקבצים לתוכנה עותק מספר ניתוח, אשר מספק מספר להעתיק החזוי המשוערת בהתבסס על שיטת ΔΔCq.
המספר עותק ניתן לחזות שגם בהתבסס על עותק הידוע מספר דגימות די אן איי שליטה בצלחת או על-ידי הזנת המספר עותק גנטי בתדירות הגבוהה ביותר (טבלה 5). איור 1 מציג את התוצאות של צלחת לתגובה שמכוונת KIR2DL4 , KIR3DS1, כמו גם את הגן הפניה STAT6. המספר עותק בתדירות הגבוהה ביותר עבור KIR2DL4, גן מסגרת במיקומה "קיר" , היא שני עותקים, בעוד מספר עותק בתדירות הגבוהה ביותר של KIR3DS1, הפעלת הגן, אינו עותק אחד. התוצאות באיור מציגות החלקות הגברה PCR שנצפו על התוכנה qPCR, הנתונים מספר עותק שנוצר מהנתונים qPCR. כפי שמוצג, וזמינותו הוא היכולת להבחין בין 0, 1, 2, 3 ו- 4 מספרים עותק של ג'ין "קיר" . ניתוח מספר עותק התוכנה גם מאפשר צפייה של ההתפלגות של מספר עותק על-פני לוח תרשים עוגה או תרשים עמודות. היעילות של התחזית מספר העותק הוא נמוך יותר עבור דגימות עם העתק מספר גבוה יותר.
האיכות של כל החומרים המשמשים את התגובות gDNA, מאגר, תחל, רגשים, יכול להשפיע על הדיוק של התוצאות המתקבלות. הכתבים בתוצאות זאת, סביר להניח להיגרם עקב וריאציה הריכוז של ה-DNA על פני צלחת. טוהר gDNA חילוץ, אשר ניתן למדוד באמצעות של 260/280 ו- 260/230 יחסי, גם יכולה להיות השפעה על האיכות. יחס 260/280 של 1.8-2 ו 260/230 יחס של 2-2.2 הם רצויים. ריכוזים טווח לא אחידה של הדנ א על פני צלחת יכול להוביל השתנות גבוהה במחזור הסף (סיטי) בין הכתבים בטווח של המספר המשוער עותק ודוגמאות. התוצאות באיור 2 מציגות האפקט שהפער בין ערכיt C על פני צלחת יכולה להיות על הדיוק בניבוי של המספר עותק. הקו האדום מציין את טווח המספר המשוער עותק עבור מדגם ו, באופן אידיאלי, צריך להיות קרוב שלם ככל האפשר.
ניתן לייצא את עותק מספר נתונים, פעם ניתח, בתור קובץ הגיליון האלקטרוני בתבנית 96-ובכן. השתמשנו קובץ script R (זמין על פי בקשה) כדי לשלב את הנתונים מספר עותק של כל לוחות 10 שמופעלות כערכה לתוך גיליון אלקטרוני אחד. נתונים שפורסמו על KIRs של אוכלוסיות ממוצא אירופאי בעיקר מאפשרת נבואתו של LD הכללים הקיימים בין גנים שונים מורכבים "קיר" 1. תחזיות אלה משמשים לערוך בדיקות במורד הזרם על עותק מספר התוצאות המתקבלות (טבלה 6). דוגמאות לא תואמות תעודת הזהות החזוי בין הגנים עשוי להכיל פולימורפיזם יוצא דופן או וריאציות מבניות haplotypic. תרשים זרימה המתאר את הפרוטוקול מוצג באיור3.
כלי שנקרא "קיר" Haplotype המזהה (http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/) פותחה כדי להקל את מעומק של haplotypes של ערכת הנתונים. מעומק עובד על בסיס רשימת הפניה haplotypes הנהוגות האוכלוסייה האירופית-מקור1. עם זאת, הכלי מאפשרת גם עבור ערכה מותאמת אישית של הפניה haplotypes כדי להשתמש במקום זאת. שלושה קבצים נפרדים נוצרים; הקובץ הראשון מפרטת את כל השילובים haplotype עבור מדגם, הקובץ השני מספק רשימת הצירופים haplotypes שיש משולב התדרים הגבוהים החתוך, ומפרטת הקובץ השלישי כדוגמאות אין אפשרות להקצות haplotypes. Non-הקצאה של haplotypes יכול לשמש כמחוון של haplotypes רומן.
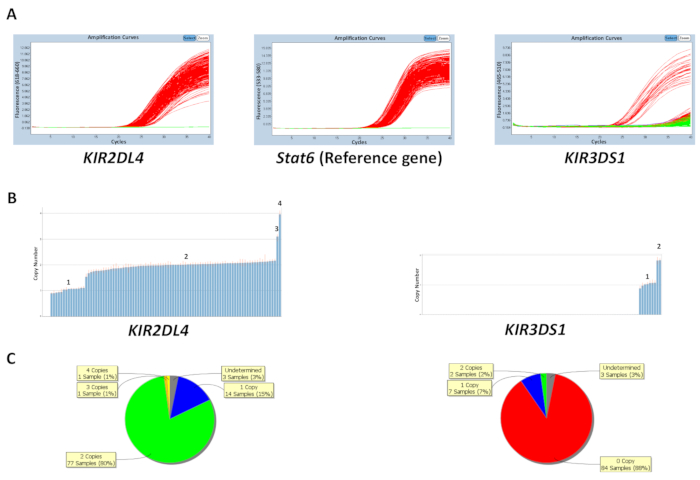
איור 1: התוצאות נציג של צלחת על תגובה מספר 5- (א) זה הגברה מראה החלונית חלקות. (B) לוח זה מראה להעתיק מספר חלקות. (ג) לוח זה מציג את התפלגות מספר עותק. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
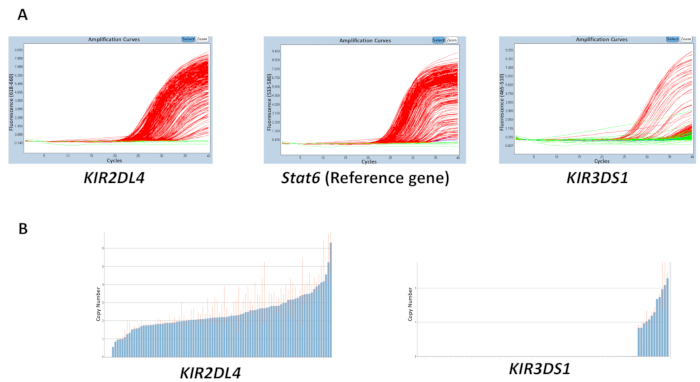
איור 2: התוצאות נציג של צלחת עם ריכוז ה-DNA משתנה עבור התגובה מספר 5- (א) זה הגברה מראה החלונית חלקות. (B) לוח זה מראה להעתיק מספר חלקות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
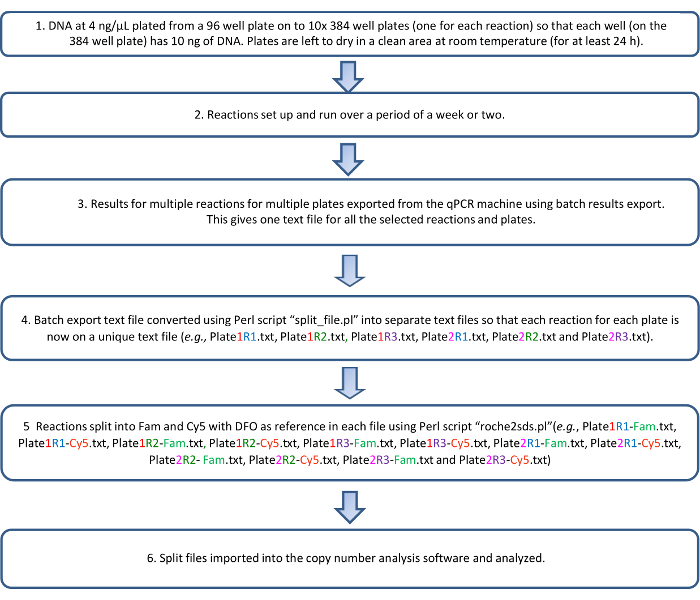
איור 3: תרשים זרימה של פרוטוקול qKAT. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| Assay | גנים | תחל קדימה | ריכוז (אן אם) | תחל הפוכה | ריכוז (אן אם) | הגששים | ריכוז (אן אם) |
| מס ' 1 | 3DP1 | A4F | 250 | A5R | 250 | P4a | 150 |
| 2DL 2 | 2DL2F4 | 400 | C3R2 | 600 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 2 | 2DS2 | A4F | 400 | A6R | 400 | P4a | 200 |
| 2DL 3 | D1F | 400 | D1R | 400 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| מס ' 3 | 3DL 3 | A8F | 500 | A8R | 500 | P4a | 150 |
| 2DS4Del | 2DS4Del | 250 | 2DS4R2 | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 4. לא | 3DL1e4 | B1F | 250 | B1R | 125 | P4b | 150 |
| 3DL1e9 | D4F | 250 | D4R2 | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 5 | 3DS1 | B2F | 250 | B1R | 250 | P4b | 150 |
| 2DL 4 | C1F | 200 | C1R | 200 | P5b - 2DL 4 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 6. לא | 2DL 1 | B3F | 500 | B3R | 125 | P4b | 150 |
| 2DP1 | D3F | 250 | D3R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| . מספר 7 | 2DS1 | B4F | 500 | B4R | 250 | P4b | 150 |
| 2DL 5 | D2F | 500 | D2R | 500 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 8 | 2DS3 | B5F | 250 | B5R | 250 | P4b | 150 |
| 3DL2e9 | D4F | 250 | G51 | 125 | P9 | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| No 9 | 3DL2e4 | A1F | 200 | A1R | 200 | P4a | 150 |
| 2DS4FL | 2DS4FL | 250 | 2DS4R2 | 500 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 | |
| 10. לא | 2DS5 | B6F2 | 200 | B6R3 | 200 | P4b | 150 |
| 2DS4 | C5F | 250 | C5R | 250 | P5b | 150 | |
| STAT6 | STAT6F | 200 | STAT6R | 200 | PSTAT6 | 150 |
טבלה 1: שילוב עם ריכוז תחל וזונדים בשימוש כל תגובה qKAT 27 .
| התגובה | פריימר Aliquots (µL) | בדיקה Aliquots (µL) | |||||||||
| R1 | 3DP1 | A4F | A5R | 2DL2F4 | C3R2 | מים | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DL 2 | 100 | 100 | 160 | 240 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R2 | 2DS2 | A2F | A6R | D1F | D1R | מים | STAT6F | STAT6R | P4A | P9 | PSTAT6 |
| 2DL 3 | 160 | 160 | 160 | 160 | 160 | 80 | 80 | 80 | 60 | 60 | |
| שים לב: עליך µL 20 פחות מים MasterMix | |||||||||||
| R3 | 3DL 3 | A8F A8FB | A8R | 2DS4DELF | 2DS4R2 | מים | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4DEL | 100 100 | 200 | 100 | 100 | 200 | 80 | 80 | 60 | 60 | 60 | |
| R4 | 3DL1E4 | B1F | B1R | D4F | D4R2 | מים | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL1E9 | 100 | 50 | 100 | 200 | 350 | 80 | 80 | 60 | 60 | 60 | |
| R5 | 3DS1 | B2F | B1R | C1F | C1R | מים | STAT6F | STAT6R | P4B | P5B - 2L 4 | PSTAT6 |
| 2DL 4 | 100 | 100 | 80 | 80 | 440 | 80 | 80 | 60 | 60 | 60 | |
| R6 | 2DL 1 | B3F | B3R | D3F | D3R | מים | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DP1 | 200 | 50 | 100 | 200 | 250 | 80 | 80 | 60 | 60 | 60 | |
| R7 | 2DS1 | B4F | B4R | D2F | D2R | מים | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 2DL 5 | 200 | 100 | 200 | 200 | 100 | 80 | 80 | 60 | 60 | 60 | |
| R8 | 2DS3 | B5F | B5R | D4F | D5R | מים | STAT6F | STAT6R | P4B | P9 | PSTAT6 |
| 3DL2E9 | 100 | 100 | 100 | 50 | 450 | 80 | 80 | 60 | 60 | 60 | |
| R9 | 3DL2E4 | A1F | A1R | 2DS4WTF | 2DS4R2 | מים | STAT6F | STAT6R | P4A | P5B | PSTAT6 |
| 2DS4WT | 80 | 80 | 100 | 200 | 340 | 80 | 80 | 60 | 60 | 60 | |
| R10 | 2DS5 | B6F2 | B6R3 | C5F | C5R | מים | STAT6F | STAT6R | P4B | P5B | PSTAT6 |
| 2DS4TOTAL | 80 | 80 | 100 | 100 | 440 | 80 | 80 | 60 | 60 | 60 | |
טבלה 2: כרכים (µL) של 100 מיקרומטר פריימר/בדיקה במלאי פתרונות פריימר ולבצע בדיקה בשילוב aliquots.
| שם | כיוון | שינוי 5´ | שינוי 3´ | רצף (5' →3') | אורך | Tm | GC % | אקסון | מיקום |
| P4a | הגיוני | FAM | BHQ-1 | TCATCCTGC AATGTTGGT CAGATGTCA | 27 | 60 | 44.4 | 4 | 425-451 |
| P4b | Antisense | FAM | BHQ-1 | AACAGAACC GTAGCATCT GTAGGTCCC T | 28 | 62 | 50 | 4 | 576-603 |
| P5b | הגיוני | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TTCCTCTG | 25 | 60 | 52 | 5 | 828 – 852 |
| P5b - 2DL 4 | הגיוני | ATTO647N | BHQ-2 | AACATTCCA GGCCGACT TCCCTCTG | 25 | 61 | 56 | 5 | 828 – 852 |
| P9 | הגיוני | ATTO647N | BHQ-2 | CCCTTCTCA GAGGCCCA AGACACC | 24 | 60 | 62.5 | 9 | 1246-1269 |
| PSTAT6 | ATTO550 | BHQ-2 | CTGATTCCT CCATGAGCA TGCAGCTT | 26 | 62 | 50 |
טבלה 3: רשימה של הגששים בשימוש qKAT 1, 27. צבעי פלורסנט השתמשו בקצה 5' של הגששים oligo P5b, P5b - 2 DL 4, P9, PSTAT6 שונו כדי צבעי אטו.
| ג'ין | תחל | כיוון | רצף (5´-3´) | אורך | Tm | GC % | אקסון | מיקום | אמפליקון (bp) | אללים עלול לפספס | ||
| 3DL2e4 | A1F | קדימה | GCCCCTGCTGAA ATCAGG | 18 | 52 | 61.1 | 4 | 399-416 | 179 | 3DL 2 * 008, * 021, * 027, * 038. | ||
| A1R | הפוך | CTGCAAGGACAG GCATCAA | 19 | 53 | 52.6 | 559-577 | 3DL 2 * 048 | |||||
| 3DP1 | A4F | קדימה | GTCCCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398-416 | 112 | אף אחד | ||
| A5R | הפוך | GTGAGGCGCAAA GTGTCA | 18 | 52 | 55.6 | 492-509 | אף אחד | |||||
| 2DS2 | A2F | קדימה | GTCGCCTGGTGA AATCAGA | 19 | 49 | 52.6 | 4 | 398-416 | 111 | אף אחד | ||
| A6R | הפוך | TGAGGTGCAAAG TGTCCTTAT | 21 | 51 | 42.9 | 488-508 | אף אחד | |||||
| 3DL 3 | A8Fa | קדימה | GTGAAATCGGGA GAGACG | 18 | 50 | 55.6 | 4 | 406-423 | 139 | אף אחד | ||
| A8Fb | קדימה | GGTGAAATCAGG AGAGACG | 19 | 50 | 52.6 | 405-423 | 3DL 3 * 054, 3DL 3 * 00905. | |||||
| A8R | הפוך | AGTTGACCTGGG AACCCG | 18 | 51 | 61.1 | 526-543 | אף אחד | |||||
| 3DL1e4 | B1F | קדימה | CATCGGTCCCAT GATGCT | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DL 1 * 00505, DL 3 1 * 006, DL 3 1 * 054, DL 3 1 * 086, DL 3 1 * 089 | ||
| B1R | הפוך | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | 3DL 1 * 00502 | |||||
| 3DS1 | B2F | קדימה | CATCGGTTCCAT GATGCG | 18 | 51 | 55.6 | 4 | 549-566 | 85 | 3DS1 * 047; ייתכן לאסוף 3DL 1 * 054. | ||
| B1R | הפוך | GGGAGCTGACAA CTGATAGG | 20 | 52 | 55 | 614-633 | אף אחד | |||||
| 2DL 1 | B3F | קדימה | TTCTCCATCAGT CGCATGAC | 20 | 52 | 50 | 4 | 544-563 | 96 | DL 2 1 * 020, DL 2 1 * 028 | ||
| B3R | הפוך | GTCACTGGGAGC TGACAC | 18 | 50 | 61.1 | 622-639 | DL 2 1 * 023, DL 2 1 * 029, DL 2 1 * 030 | |||||
| 2DS1 | B4F | קדימה | TCTCCATCAGTC GCATGAA | 19 | 51 | 47.4 | 4 | 545-563 | 96 | 2DS1 * 001 | ||
| B4R | הפוך | GGTCACTGGGAG CTGAC | 17 | 49 | 64.7 | 624-640 | אף אחד | |||||
| 2DS3 | B5F | קדימה | CTCCATCGGTCG CATGAG | 18 | 53 | 61.1 | 4 | 546-563 | 96 | אף אחד | ||
| B5R | הפוך | GGGTCACTGGGA GCTGAA | 18 | 51 | 61.1 | 624-641 | אף אחד | |||||
| 2DS5 | B6F2 | קדימה | AGAGAGGGGACG TTTAACC | 19 | 50 | 52.6 | 4 | 475-493 | 173 | אף אחד | ||
| B6R3 | הפוך | TCCAGAGGGTCA CTGGGC | 18 | 53 | 66.7 | 630-647 | 2DS5 * 003 | |||||
| 2DL 4 | C1F | קדימה | GCAGTGCCCAGC ATCAAT | 18 | 52 | 55.6 | 5 | 808-825 | 83 | אף אחד | ||
| C1R | הפוך | CCGAAGCATCTG TAGGTCT | 19 | 52 | 52.6 | 872-890 | 2DL 4 * 018, DL 2 4 * 019 | |||||
| 2DL 2 | 2DL2F4 | קדימה | GAGGTGGAGGCC CATGAAT | 19 | 52 | 57.9 | 5 | 778-796 | 151 | 2DL 2 * 009; 782G לשנות לא | ||
| C3R2 | הפוך | TCGAGTTTGACC ACTCGTAT | 20 | 51 | 45 | 909-928 | אף אחד | |||||
| 2DS4 | C5F | קדימה | TCCCTGCAGTGC GCAGC | 17 | 57 | 70.6 | 5 | 803-819 | 120 | אף אחד | ||
| C5R | הפוך | TTGACCACTCGT AGGGAGC | 19 | 52 | 57.9 | 904-922 | 2DS4 * 013 | |||||
| 2DS4Del | 2DS4Del | קדימה | CCTTGTCCTGCA GCTCCAT | 19 | 54 | 57.9 | 5 | 750-768 | 203 | אף אחד | ||
| 2DS4R2 | הפוך | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | אף אחד | |||||
| 2DS4FL | 2DS4FL | קדימה | CCGGAGCTCCTA TGACATG | 19 | 53 | 57.9 | 5 | 744-762 | 209 | אף אחד | ||
| 2DS4R2 | הפוך | TGACGGAAACAA GCAGTGGA | 20 | 53 | 50 | 933-952 | אף אחד | |||||
| 2DL 3 | D1F | קדימה | AGACCCTCAGGA GGTGA | 17 | 48 | 58.8 | 9 | 1180-1196 | 156 | אף אחד | ||
| D1R | הפוך | CAGGAGACAACT TTGGATCA | 20 | 50 | 45 | 1316-1335 | 2DL 3 * 010, DL 2 3 * 017, DL 2 3 * 01801, DL 2 3 * 01802 | |||||
| 2DL 5 | D2F | קדימה | CACTGCGTTTTC ACACAGAC | 20 | 52 | 50 | 9 | 1214-1233 | 120 | 2DL5B * 011 ו- 2DL5B * 020 | ||
| D2R | הפוך | GGCAGGAGACAA TGATCTT | 19 | 49 | 47.4 | 1315-1333 | אף אחד | |||||
| 2DP1 | D3F | קדימה | CCTCAGGAGGTG ACATACGT | 20 | 53 | 55 | 9 | 1184-1203 | 121 | אף אחד | ||
| D3R | הפוך | TTGGAAGTTCCG TGTACACT | 20 | 50 | 45 | 1285-1304 | אף אחד | |||||
| 3DL1e9 | D4F | קדימה | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 93 | 3DL 1 * 061, DL 3 1 * 068 | ||
| D4R2 | הפוך | CCGTGTACAAGA TGGTATCTGTA | 23 | 53 | 43.5 | 1273-1295 | 3DL 1 * 05901, DL 3 1 * 05902, DL 3 1 * 060, DL 3 1 * 061, DL 3 1 * 064, DL 3 1 * 065, DL 3 1 * 094N, DL 3 1 * 098 | |||||
| 3DL2e9 | D4F | קדימה | CACAGTTGGATC ACTGCGT | 19 | 52 | 52.6 | 9 | 1203-1221 | 156 | אף אחד | ||
| D5R | הפוך | GACCTGACTGTG GTGCTCG | 19 | 54 | 63.2 | 1340-1358 | אף אחד | |||||
| STAT6 | STAT6F | קדימה | CCAGATGCCTAC CATGGTGC | 20 | 54 | 60 | 129 | |||||
| STAT6R | הפוך | CCATCTGCACAG ACCACTCC | 20 | 54 | 60 | |||||||
בטבלה 4: רצף צבעי יסוד בשימוש qKAT 1, 27-
| "קיר" ג'ין | 3DL 3 | 2DS2 | 2DL 2 | 2DL 3 | 2DP1 | 2DL 1 | 3DP1 | 2DL 4 | 3DL 1 EX9 | 3DL 1 EX9 | 3DS1 | 2DL 5 | 2DS3 | 2DS5 | 2DS1 | 2DS4 סה | 2DS4 פל | 2DS4 דל | 3DL 2 ex4 | 3DL 2 EX9 | |
| מספר עותק בתדירות הגבוהה ביותר | 2 | 1 | 1 | 2 | 2 | 2 | 2 | 2 | 2 | 2 | 1 | 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 | 2 | |
טבלה 5: העתק בתדירות הגבוהה ביותר למספר "קיר" גנים שנצפתה בדרך כלל נקודת המוצא האירופי דגימות.
| תאחיזה לא שוויונית לכללי qKAT מבוסס על אוכלוסיות האירופית | סימון מספר עותק | |||||||
| 1 | KIR3DL3, KIR3DP1, KIR2DL4 ו KIR3DL2 הם מסגרת הגנים המצויים על שתי haplotypes. | KIR3DL3, KIR3DP1, KIR2DL4 , KIR3DL2 = 2 | ||||||
| 2 | KIR2DS2 , KIR2DL2 הינם LD אחד עם השני | 2DS2=2 DL 2 | ||||||
| 3 | KIR2DL2 ו KIR2DL3 הם אללים של אותו גן | 2 DL 2+2 DL 3= 2 | ||||||
| 4 | KIR2DP1 , KIR2DL1 הינם LD אחד עם השני | 2DP1=2 DL 1 | ||||||
| 5 | אקסון 4 של KIR3DL1 ו- KIR3DL2 שווה אקסון 9 של KIR3DL1 ו- KIR3DL2 בהתאמה. | 3DL1ex4=3DL1ex9 , 3DL2ex4=3DL2ex9 | ||||||
| 6 | KIR3DL1 ו KIR3DS1 הם אללים | 3 DL 1+3DS1= 2 | ||||||
| 7 | KIR2DS3 , KIR2DS5 הינם LD עם KIR2DL5 | 2DS3+2DS5=2 DL 5 | ||||||
| 8 | KIR3DS1 , KIR2DS1 הינם LD | 3DS1=2DS1 | ||||||
| 9 | הנוכחות של otal KIR2DS1 , KIR2DS4Tהיא שבחירה-haplotype | 2DS1+2DS4TOTAL= 2 | ||||||
| 10 | KIR2DS4FL ו KIR2DS4del הם וריאציות של KIR2DS4TOTAL | 2DS4FL+2DS4DEL=2DS4TOTAL | ||||||
טבלה 6: תאחיזה לא שוויונית בין "קיר" גנים נצפתה נפוץ בקרב אוכלוסיות ממוצא אירופאי יכול לשמש כדי לבדוק את הנתונים מספר עותק 1,27-
Discussion
אנו המתואר הרומן חצי אוטומטיות תפוקה גבוהה בשיטה, הנקראת qKAT, אשר מקלה על הקלדת מספר עותק של "קיר" גנים. השיטה היא שיפור על פני שיטות קונבנציונליות כמו SSP-PCR, תפוקה נמוכה והם יכולים רק להצביע על נוכחות או היעדרות של גנים אלה מאוד רב שלבית.
הדיוק של הנתונים מספר עותק שהושג תלויה גורמים רבים, כולל את האיכות ואת ריכוז-אחידות של הדגימות gDNA והאיכות של ריאגנטים. האיכות והדיוק של הדגימות gDNA על פני צלחת חשובים ביותר מאז וריאציות בריכוז על פני הצלחת עלולה לגרום לשגיאות בחישוב של המספר עותק. מאז מבחני היו מאומת באמצעות ערכות המדגם האירופי-מקור, נתונים גדודים משאר חלקי העולם דורשות בדיקות יסודית יותר. זו היא להבטיח כי מקרים של נשירת אלל או שאינם ספציפיים פריימר/בדיקה איגוד הם לא טעו לחשוב כמו העתק מספר וריאציות.
בעוד מבחני היו מעוצבות וממוטבות לרוץ כמו תפוקה גבוהה, ניתן לשנותן כדי להפעיל פחות דגימות. מדד ביטחון בתוכנת ניתוח מספר עותק מושפע בעת ניתוח דגימות פחות, אבל זה יכול להשתפר אם בקרת דגימות די אן איי גנומית עם מספר ידוע של עותק הגן וקיר כלולים בצלחת נוספים דוגמת משכפל כלל.
עבור מעבדות ללא נוזל/צלחת-טיפול רובוטים, מיקס מאסטר יכול ובשמפו באמצעות פיפטות רב ערוצית, צלחות ניתן לטעון ידנית לתוך הכלי qPCR.
המטרה העיקרית מאחורי התפתחות qKAT הייתה ליצור פשוטה, תפוקה גבוהה, ברזולוציה גבוהה, וחסכונית שיטה גנוטיפ KIRs למחלה האגודה מחקרים. דבר זה הושג בהצלחה מאז qKAT ננקטה חוקרים את התפקיד של "קיר" במספר מחקרים האגודה מחלה גדולה, כולל מגוון של מחלות זיהומיות, מחלות אוטואימוניות הריון הפרעות4, 24 , 25 , 26.
Disclosures
המחברים אין לחשוף.
Acknowledgements
הפרויקט קיבל מימון של המועצה למחקר רפואי (MRC), אירופה מחקר המועצה (ERC) תחת אופק 2020 מחקר וחדשנות התכנית של האיחוד האירופי (גרנט הסכם מס 695551) וקיימברידג המכון הלאומי לבריאות (NIH) ביו המרכז לחקר דם מחקר NIH והשתלת יחידת המחקר (NIHR BTRU) תרומת איברים והשתלות -מנדרינית, בשיתוף עם דם NHS השתלה (NHSBT). הדיעות הם אלה היוצרים ואלה לא בהכרח של בקופת חולים, את NIHR, מחלקת הבריאות או את NHSBT.
Materials
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| Oligonucleotides | Sigma | Custom order | SEQUENCES: Listed in Table 4 |
| Probes labelled with ATTO dyes | Sigma | Custom order | SEQUENCES: Listed in Table 3 |
| SensiFAST Probe No-ROX Kit | Bioline | BIO-86020 | − |
| MilliQ water | − | − | − |
| Name | Company | Catalog Number | Comments |
| EQUIPMENT | |||
| Centrifuge with a swinging bucket rotor | Eppendorf(or equivalent) | Eppendorf 5810R or equivalent system | |
| NanoDrop | Thermo Scientific | ND-2000 | |
| OR | |||
| QuBit Fluorometer | Life Technologies | Q33216 | |
| Matrix Hydra | Thermo Scientific | 109611 | |
| LightCycler 480 II Instrument 384-well | Roche | 05015243001 | |
| Twister II Microplate Handler with MéCour Thermal Plate Stacker (MéCour) | Caliper Life Sciences | 204135 | |
| Vortex mixer | Biosan | BS-010201-AAA | |
| Single-channel pipettes (volume range: 0.5–10 µL, 2–20 µL, 20–200 µL, 200–1,000 µL; 1-10 mL) | Gilson(or equivalent) | F144801, F123600, F123615, F123602, F161201 | |
| RNase- and DNase-free pipette tips filtered (10 µL, 20 µL, 200 µL, 1,000 µL, 10 mL) | Starlab (or equivalent) | S1111-3810, S1120-1810, S1120-8810, S1111-6810, I1054-0001 | |
| StarTub PS Reagent Reservoir, 55 mL | STARLAB | E2310-1010 | |
| 50 mL Centrifuge Tube | STARLAB | E1450-0200 | |
| 96-well deep well plate | Fisher Scientific | 12194162 | |
| LC480 384 Multi-well plates | Roche | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche | 04729757001 | |
| Name | Company | Catalog Number | Comments |
| SOFTWARE | |||
| Roche LightCycler 480 Software v1.5 | |||
| Applied Biosystems CopyCaller Software v2.1 | https://www.thermofisher.com/uk/en/home/technical-resources/software-downloads/copycaller-software.html | ||
| KIR haplotype identifier | http://www.bioinformatics.cimr.cam.ac.uk/haplotypes/ |
References
- Jiang, W., et al. Copy number variation leads to considerable diversity for B but not A haplotypes of the human KIR genes encoding NK cell receptors. Genome Research. 22, 1845-1854 (2012).
- Nemat-Gorgani, N., et al. Different Selected Mechanisms Attenuated the Inhibitory Interaction of KIR2DL1 with C2 + HLA-C in Two Indigenous Human Populations in Southern Africa. The Journal of Immunology. 200, 2640-2655 (2018).
- Norman, P. J., et al. Co-evolution of human leukocyte antigen (HLA) class I ligands with killer-cell immunoglobulin-like receptors (KIR) in a genetically diverse population of sub-Saharan Africans. PLoS Genetics. 9, e1003938 (2013).
- Nakimuli, A., et al. Killer cell immunoglobulin-like receptor (KIR) genes and their HLA-C ligands in a Ugandan population. Immunogenetics. 65, 765-775 (2013).
- Bontadini, A., et al. Distribution of killer cell immunoglobin-like receptors genes in the Italian Caucasian population. Journal of Translational Medicine. 4, 1-9 (2006).
- Graef, T., et al. KIR2DS4 is a product of gene conversion with KIR3DL2 that introduced specificity for HLA-A*11 while diminishing avidity for HLA-C. The Journal of Experimental Medicine. 206, 2557-2572 (2009).
- Béziat, V., Hilton, H. G., Norman, P. J., Traherne, J. A. Deciphering the killer-cell immunoglobulin-like receptor system at super-resolution for natural killer and T-cell biology. Immunology. 150, 248-264 (2017).
- Blokhuis, J. H., et al. KIR2DS5 allotypes that recognize the C2 epitope of HLA-C are common among Africans and absent from Europeans. Immunity, Inflammation and Disease. 5, 461-468 (2017).
- Martin, M. P., et al. Epistatic interaction between KIR3DS1 and HLA-B delays the progression to AIDS. Nature Genetics. 31, 429-434 (2002).
- Khakoo, S. I., et al. HLA and NK cell inhibitory receptor genes in resolving hepatitis C virus infection. Science. 305, 872-874 (2004).
- van Bergen, J., et al. KIR-ligand mismatches are associated with reduced long-term graft survival in HLA-compatible kidney transplantation. American Journal of Transplantation. 11, 1959-1964 (2011).
- Hiby, S. E., et al. Association of maternal killer - cell immunoglobulin-like receptors and parental HLA - C genotypes with recurrent miscarriage. Human Reproduction. 23, 972-976 (2008).
- Nakimuli, A., et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. Proceedings of the National Academy of Sciences. 112, 845-850 (2015).
- van Bergen, J., et al. HLA reduces killer cell Ig-like receptor expression level and frequency in a humanized mouse model. The Journal of Immunology. 190, 2880-2885 (2013).
- Bachanova, V., et al. Donor KIR B Genotype Improves Progression-Free Survival of Non-Hodgkin Lymphoma Patients Receiving Unrelated Donor Transplantation. Biology of Blood and Marrow Transplantation. 22, 1602-1607 (2016).
- Cooley, S., et al. Donor selection for natural killer cell receptor genes leads to superior survival after unrelated transplantation for acute myelogenous leukemia. Blood. 116, 2411-2419 (2010).
- Barani, S., Khademi, B., Ashouri, E., Ghaderi, A. KIR2DS1, 2DS5, 3DS1 and KIR2DL5 are associated with the risk of head and neck squamous cell carcinoma in Iranians. Human Immunology. 79, 218-223 (2018).
- Vilches, C., Castaño, J., Gómez-Lozano, N., Estefanía, E. Facilitation of KIR genotyping by a PCR-SSP method that amplifies short DNA fragments. Tissue Antigens. 70, 415-422 (2007).
- Ashouri, E., Ghaderi, A., Reed, E. F., Rajalingam, R. A novel duplex SSP-PCR typing method for KIR gene profiling. Tissue Antigens. 74, 62-67 (2009).
- Martin, M. P., Carrington, M. KIR locus polymorphisms: genotyping and disease association analysis. Methods in Molecular Biology. , 49-64 (2008).
- Crum, K. A., Logue, S. E., Curran, M. D., Middleton, D. Development of a PCR-SSOP approach capable of defining the natural killer cell inhibitory receptor (KIR) gene sequence repertoires. Tissue Antigens. 56, 313-326 (2000).
- Houtchens, K. A., et al. High-throughput killer cell immunoglobulin-like receptor genotyping by MALDI-TOF mass spectrometry with discovery of novel alleles. Immunogenetics. 59, 525-537 (2007).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods. 25, 402-408 (2001).
- Traherne, J. A., et al. KIR haplotypes are associated with late-onset type 1 diabetes in European-American families. Genes and Immunity. 17, 8-12 (2016).
- Hydes, T. J., et al. The interaction of genetic determinants in the outcome of HCV infection: Evidence for discrete immunological pathways. Tissue Antigens. 86, 267-275 (2015).
- Dunphy, S. E., et al. 2DL1, 2DL2 and 2DL3 all contribute to KIR phenotype variability on human NK cells. Genes and Immunity. 16, 301-310 (2015).
- Jiang, W., et al. qKAT: A high-throughput qPCR method for KIR gene copy number and haplotype determination. Genome Medicine. 8, 1-11 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved