Method Article
استخدام خط أنابيب MALDI TOF-MS IDBac المفتوح المصدر لتحليل البروتين الميكروبي وبيانات المستقلب المتخصصة
In This Article
Summary
IDBac هو خط أنابيب المعلوماتية الحيوية القائمة على الطيف الكتلي المفتوح المصدر الذي يدمج البيانات من كل من البروتين السليم وأطياف المستقلب المتخصصة، التي تم جمعها على مواد الخلايا كشط من المستعمرات البكتيرية. يسمح خط الأنابيب للباحثين بتنظيم المئات إلى الآلاف من المستعمرات البكتيرية بسرعة في مجموعات تصنيفية مفترضة، وتمييزها بشكل أكبر على أساس إنتاج المستقلب المتخصص.
Abstract
من أجل تصور العلاقة بين الفيلوجيني البكتيري وإنتاج المستقلب المتخصص للمستعمرات البكتيرية التي تنمو على أجار المغذيات، قمنا بتطوير IDBac - وهو امتصاص ليزر بمساعدة مصفوفة منخفضة التكلفة وعالية الإنتاجية بمساعدة المصفوفة/التأين خط أنابيب المعلوماتية الحيوية (MALDI-TOF MS) في وقت الطيران. تم تصميم برنامج IDBac لغير الخبراء، وهو متاح بحرية، وقادر على تحليل عدد قليل إلى الآلاف من المستعمرات البكتيرية. هنا، نقدم إجراءات لإعداد المستعمرات البكتيرية لتحليل MALDI-TOF MS، وتشغيل أداة MS، ومعالجة البيانات والتصور في IDBac. على وجه الخصوص، نحن نوجه المستخدمين كيفية تجميع البكتيريا في dendrograms على أساس بصمات البروتين MS وإنشاء شبكات جمعية المستقلب بشكل تفاعلي (MANs) من بيانات المستقلب المتخصصة.
Introduction
ومن العوائق الرئيسية أمام الباحثين الذين يدرسون الوظيفة البكتيرية القدرة على تقييم الهوية التصنيفية للكائنات الدقيقة وقدرتها على إنتاج الأيض المتخصص بسرعة وفي وقت واحد. وقد حال ذلك دون إحراز تقدم كبير في فهم العلاقة بين الفيلوجيني البكتيري وإنتاج المستقلب المتخصص في غالبية البكتيريا المعزولة عن البيئة. على الرغم من أن الأساليب المستندة إلىمرض التصلب العصبي المتعدد التي تستخدم بصمات البروتين لتجميع وتحديد البكتيريا هي وصف جيد 1،2،3،4، وقد أجريت هذه الدراسات عموما على مجموعات صغيرة من العزلات، بطريقة خاصة بالأنواع. والأهم من ذلك أن المعلومات المتعلقة بإنتاج المستقلب المتخصص، وهو محرك رئيسي للوظيفة الميكروبية في البيئة، ظلت غير مدمجة في هذه الدراسات. وقد قدم سيلفا وآخرون5 مؤخراً تاريخاً شاملاً يفصّل الاستخدام الناقص لـ MALDI-TOF MS لتحليل الأيض المتخصص ونقص البرمجيات اللازمة للتخفيف من الاختناقات الحالية في مجال المعلوماتية الأحيائية. من أجل معالجة أوجه القصور هذه، أنشأنا IDBac، خط أنابيب المعلوماتية الحيوية التي تدمج كل من وسائط الخطية والعاكسة من MALDI-TOF MS6. وهذا يسمح للمستخدمين لتصور بسرعة وتمييز العزلات البكتيرية على أساس كل من البروتين والمتخصصة metabolite MS بصمات الأصابع، على التوالي.
IDBac هو فعال من حيث التكلفة، والإنتاجية العالية، ومصممة للمستخدم العادي. وهي متاحة بحرية (chasemc.github.io/IDBac)، وتتطلب فقط الوصول إلى مطياف كتلة MALDI-TOF (سيكون وضع الانعكاس مطلوبا لتحليل المستقلب المتخصص). يعتمد إعداد العينات على طريقة "النقل المباشر الموسع" البسيطة7 و8 ويتم جمع البيانات مع عمليات الاستحواذ الخطية والانعكاسية المتتالية على بقعة واحدة من أهداف MALDI. مع IDBac، فمن الممكن تحليل إنتاج فيلوجيني المفترضة والمستقلب المتخصصة من مئات المستعمرات في أقل من أربع ساعات، بما في ذلك إعداد العينة، والحصول على البيانات، والتصور البيانات. وهذا يمثل ميزة كبيرة من الوقت والتكلفة على الأساليب التقليدية لتحديد البكتيريا (مثل تسلسل الجينات)، وتحليل الناتج الأيضي (قياس الطيف الكتلي للكروماتوغرافيا السائلة [LCMS] والأساليب الكروماتوغرافية المماثلة).
باستخدام البيانات التي تم الحصول عليها في تحليل الوضع الخطي، يستخدم IDBac التجميع الهرمي لتمثيل الصلة بين أطياف البروتين. وبما أن الأطياف تمثل في الغالب بروتينات ريبوسومال المأينة، فإنها توفر تمثيلاً للتنوع الجيني الفيوجيني الموجود في عينة. وبالإضافة إلى ذلك، يتضمن IDBac بيانات وضع reflectron لعرض بصمات الأصابع المستقلب المتخصصة كشبكات جمعية المستقلب (MANs). MANs هي شبكات ثنائية تسمح بالتصور السهل لإنتاج المستقلب المشترك والفريد بين العزلات البكتيرية. منصة IDBac يسمح للباحثين لتحليل كل من البروتين وبيانات المستقلب المتخصصة جنبا إلى جنب ولكن أيضا بشكل فردي إذا تم الحصول على نوع واحد فقط من البيانات. الأهم من ذلك، IDBac يعالج البيانات الخام من أدوات بروكر وشيامن، فضلا عن TXT، علامة التبويب، csv، mzXML، وmzML. يؤدي ذلك إلى إزالة الحاجة إلى التحويل اليدوي وتنسيق مجموعات البيانات، ويقلل بشكل كبير من خطر خطأ المستخدم أو سوء التعامل مع بيانات MS.
Protocol
1 - إعداد مصفوفة مالدي
- إعداد 10 ملغ / مل MALDI الصف، و / أو تبلور α-سيانو-4-هيدروكسيسيناميك حمض (CHCA) في المذيبات MS-الصف: 50٪ أسيتوتريل (ACN)، 47.5٪ المياه (H2O)، 2.5٪ حمض ثلاثي الفلوروسيتيك (TFA). مثال: 100 ميكرولتر الحل = 50 ميكرولتر ACN + 47.5 ميكرولتر H2O + 2.5 ميكرولتر TFA + 1 ملغ CHCA
- إعداد ما لا يقل عن 1 ميكرولتر من محلول المصفوفة لكل بقعة لوحة MALDI ودوامة أو sonicate حتى في الحل (حوالي 5 دقائق sonication أو لا المواد الصلبة مرئية).
تحذير: TFA هو حمض قوي التي ينبغي التعامل معها في غطاء الدخان الكيميائي في حين يرتدي معدات الوقاية الشخصية المناسبة، كما أنه يمكن أن تضر الجلد والعينين، والخطوط الجوية مع الاتصال أو الاستنشاق.
ملاحظة: CHCA هو استرطابي وحساس للضوء وينبغي تخزينها في قارورة العنبر في المجفف. هناك العديد من خيارات مصفوفة MALDI المتاحة. CHCA هو الأكثر شيوعا لتوصيف البروتين من البكتيريا، ولكن يعمل أيضا لتحليل المستقلب المتخصصة. يعتمد اختيار المصفوفة على احتياجات المستخدم/التجربة الفردية.
- إعداد ما لا يقل عن 1 ميكرولتر من محلول المصفوفة لكل بقعة لوحة MALDI ودوامة أو sonicate حتى في الحل (حوالي 5 دقائق sonication أو لا المواد الصلبة مرئية).
2- إعداد لوحات الهدف التابعة لـ MALDI
ملاحظة: انظر ساوروآخرون 7، للحصول على مزيد من التفاصيل.
- شطف لوحة MALDI مع الميثانول (HPLC الصف أو أعلى) ومسح الجافة مع مناديل ورقية لينة. لا تستخدم فرش جلخ عند تنظيف لوحات الهدف، وهذا يمكن أن تضر بشكل دائم سطح اللوحة المستهدفة.
- تعيين البروتين والبقع كاليبرانت المستقلب المتخصصة. تنظيم بقع المعايرة بالتساوي عبر مجموعة العينات، لحساب المخالفات MALDI لوحة والانجراف الصك مع مرور الوقت. (أ) تعيين عدد مناسب من الوسائط/البقع الفارغة بالمصفوفة للدراسة؛ هذه البقع سوف تحتوي على الوسائط والمصفوفة فقط، أو مصفوفة فقط.
- باستخدام مسواك معقم، نقل جزء صغير من مستعمرة بكتيرية إلى المكان المناسب على لوحة MALDI. نشر المستعمرة البكتيرية بالتساوي على الفور. يجب أن تظهر البقعة مسطحة قدر الإمكان.
ملاحظة: سيكون من الأسهل لتسطيح المستعمرات البكتيرية التي هي أكثر المخاطية / غير متبلور. للحصول على مستعمرات أكثر صلابة / الصلبة، وتجنب تركمجموعات مرئية من كتلة الخلية على بقعة MALDI (الشكل 1).
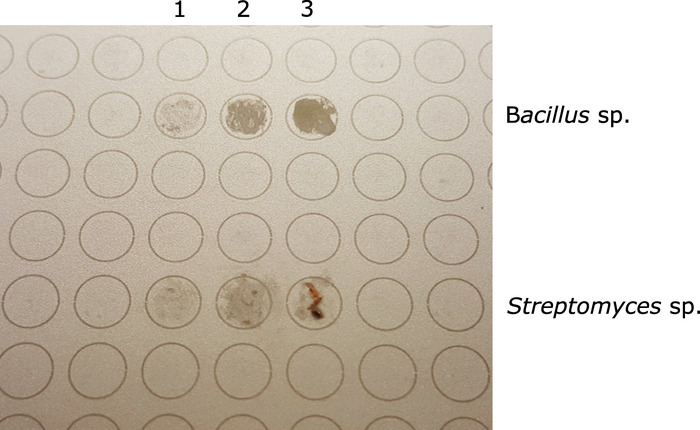
الشكل 1: لوحة MALDI المستهدفة تظهر اثنين من عزلات مختلفة قبل إضافة حمض الفورميك ومصفوفة MALDI (أعلى 3 بقع - عصية sp.; أسفل 3 البقع - العقدية sp.). وبالنسبة لكليهما، يمثل العمود 3 عينة زائدة؛ يمثل العمود 2 الكمية المناسبة من العينة؛ يمثل العمود 1 عينة غير كافية لتحليل MALDI. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- قم بإعداد مصفوفة/تحكم في الوسائط باستخدام مسواك معقم لنقل الحد الأدنى من الأجار/الوسائط إلى البقعة (البقعة) المناسبة على لوحة MALDI.
- تراكب 1 μL من 70٪ درجة قياس الطيف الكتلي حمض الفورميك على كل بقعة عينة، بما في ذلك بقع التحكم في المصفوفة. السماح للحمض بتجفيف الهواء تماما في غطاء الدخان الكيميائي (حوالي 5 دقائق).
تحذير: حمض الفورميك هو مادة كيميائية كاوية وينبغي التعامل معها في أغطية الدخان الكيميائي. يمكن أن تلحق الضرر بالخطوط الجوية إذا استنشق. - إضافة 1 μL من حل مصفوفة MALDI المعدة لكل بقعة عينة، وكذلك إلى مصفوفة / وسائل الإعلام البقع التحكم. السماح حل مصفوفة لتجف الهواء تماما (حوالي 5 دقائق).
ملاحظة: من الممكن تخزين اللوحة في المجفف، في الظلام، حتى يمكن تحليلها على مطياف كتلة MALDI-TOF. قد تختلف أوقات التخزين المسموح بها وفقًا لاستقرار العينة. - أضف 0.5-1.0 ميكرولتر إلى بقع المعايرة المخصصة، متبوعاً بمحلول مصفوفة MALDI 1 ميكرولتر. ماصة الحل الناتج صعودا وهبوطا لخلط. السماح لجميع البقع لتجف تماما قبل إدخالفي مطياف كتلة MALDI-TOF.
ملاحظة: يجب إضافة البروتين وكاليباتالمستقلت المتخصصة في غضون 30 دقيقة من تحليل MALDI، وكلاهما عرضة للتدهور.
3 - الحصول على البيانات
ملاحظة: يتم سرد المعلمات العامة للحصول على البيانات في الجدول 1.
| المعلمه | البروتين | المستقلب المتخصص |
| بداية جماعية (دا) | عام 1920 | 60 |
| نهاية الشامل (دا) | 21000 | 2700 |
| انحراف جماعي (دا) | عام 1900 | 50 |
| لقطات | 500 | ألف شخص |
| التردد (هرتز) | عام 2000 | عام 2000 |
| حجم الليزر | كبيره | المتوسطه |
| MaxStdDev (جزء في المليون) | 300 | 30 |
الجدول 1
- بعد البروتوكولات الخاصة بالأداة المستخدمة، قم بإعداد كل من البروتين ومعايرة المستقلب المتخصصة.
- اختبار عدد قليل من البقع المستهدفة منفصلة لتحديد قوة الليزر الأمثل واكتساب كاشف لاستخدامها عند الحصول على الأطياف (وهذا سوف تختلف يوما بعد يوم وبواسطة أداة).
ملاحظة: الشكل 2ألف والشكل 3ألف يبينان الأطياف المثلى، في حين أن الشكل 2D والشكل 3D مثالان على الأطياف ذات النوعية السيئة.
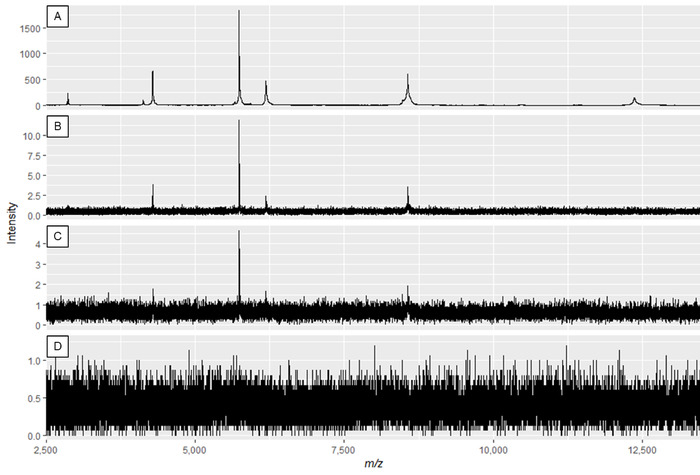
الشكل 2: مثال على أطياف البروتين التي تعرض تأثير تعديل قوة الليزر وكسب كاشف. نوعية الأطياف هي الأفضل في اللوحة A، وينخفض حتى جودة الأطياف غير كافية في لوحات C و D. في حين أن الطيف في اللوحة B قد يؤدي إلى قمم قابلة للاستخدام، تعرض اللوحة A البيانات المثلى. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
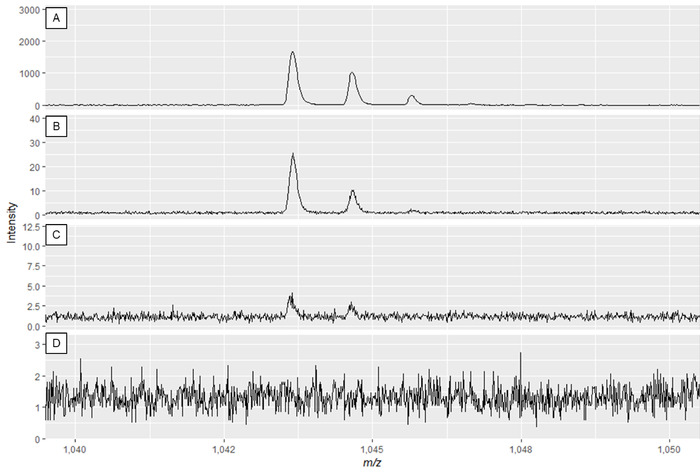
الشكل 3: مثال على أطياف المستقلب المتخصصة التي تعرض تأثير تعديل قوة الليزر وكسب كاشف. نوعية الأطياف هي الأفضل في اللوحة A وتنخفض حتى جودة الأطياف غير كافية في لوحات C و D. في حين أن الطيف في اللوحة B قد يؤدي إلى قمم قابلة للاستخدام، تعرض اللوحة A البيانات المثلى. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- الحصول على الأطياف، وتوفير أطياف البروتين في مجلد واحد وأطياف المستقلب المتخصصة في مجلد منفصل ثان.
4. تنظيف لوحة الهدف MALDI (تكييفها منساور وآخرون 7)
- إزالة لوحة الهدف MALDI من حاملها وشطف مع الأسيتون.
- اغسل هُنا بصابون سائل غير كاشط لإزالة البروتينات النزرة والدهون، ومناديل ورقية ناعمة/فرشاة أسنان ناعمة.
- شطف بالماء منزوع المتأين لمدة 2 دقيقة تقريبا لإزالة الصابون تماما.
- Sonicate لوحة الهدف في الماء (HPLC الصف أو أعلى) لمدة 5 دقائق.
- شطف لوحة الهدف بالماء (HPLC الصف أو أعلى).
- شطف لوحة الهدف مع الميثانول (HPLC الصف أو أعلى).
5. تثبيت برنامج IDBac
- قم بتنزيل برنامج IDBac.
ملاحظة: النسخ الاحتياطية الدائمة التي تم إصدارها متوفرة أيضًا للتنزيل (راجع جدول المواد). - انقر نقراً مزدوجاً فوق "Install_IDBac.exe" الذي تم تنزيله لبدء المثبت واتبع الإرشادات التي تظهر على الشاشة.
6. بدءا من البيانات الخام
ملاحظة: يتم تضمين التفسيرات التفصيلية والتعليمات لكل خطوة معالجة البيانات داخل IDBac، ولكن يتم وصف التحليلات الرئيسية والمدخلات التفاعلية أدناه.
- انقر نقراً مزدوجاً فوق اختصار سطح المكتب IDBac لتشغيل IDBac. سيتم فتح IDBac في علامة التبويب مقدمة بشكل افتراضي.
- استخدم الزر التحقق من وجود تحديثات للتأكد من استخدام أحدث إصدار من IDBac (يتطلب الوصول إلى الإنترنت). في حالة توفر إصدار أحدث، سيقوم IDBac بتنزيل التحديث وتثبيته تلقائيًا، وبعد ذلك سيطلب IDBac إعادة تشغيله.
- انقر على علامة التبويب البدء بالبيانات الأولية واختر من القائمة نوع البيانات التي سيتم استخدامها مع IDBac; متابعة باتباع الإرشادات داخل التطبيق.
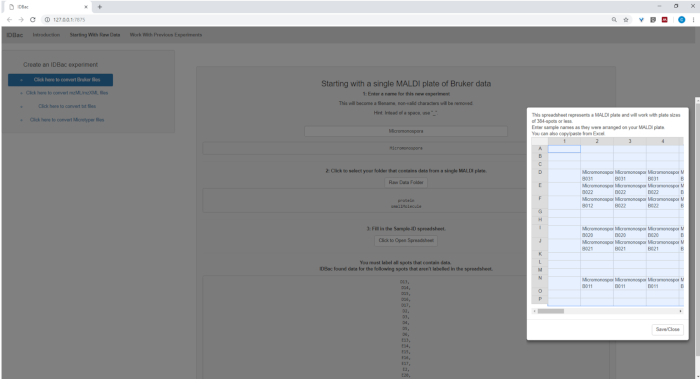
- عند إعداد تحويل ملفات البيانات ومعالجتها، أدخل اسمًا وصفيًا للتجربة عند المطالبة (راجع الشكل4). وسيتم عرض التجارب في وقت لاحق أبجديا، لذلك استراتيجية مفيدة هي البدء في أسماء التجارب مع سمة المجموعة (على سبيل المثال، "عصية التجارب_التجربة-1"؛ "عصية-تجارب_تجربة-2").
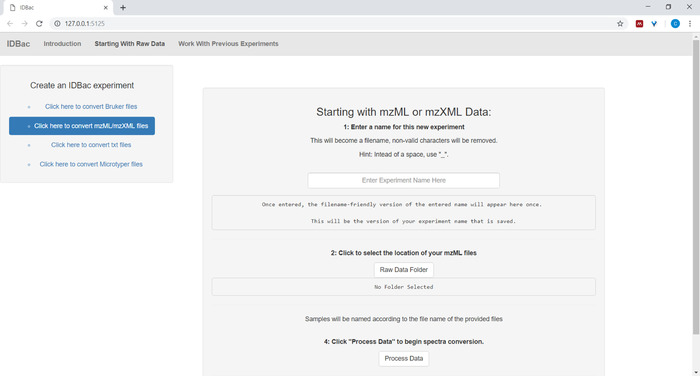
الشكل 4: تحويل بيانات IDBac وخطوة المعالجة المسبقة. يقوم IDBac بتحويل الأطياف الخام إلى تنسيق mzML المفتوح ويخزن mzML وقوائم الذروة وعينة المعلومات في قاعدة بيانات لكل تجربة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
7. العمل مع التجارب السابقة
- بعد تحويل الملفات ومعالجتها مع IDBac، أو في أي وقت يرغب المرء في إعادة تحليل تجربة، انتقل إلى صفحة العمل مع التجارب السابقة وحدد تجربة للعمل مع ( الشكل5).
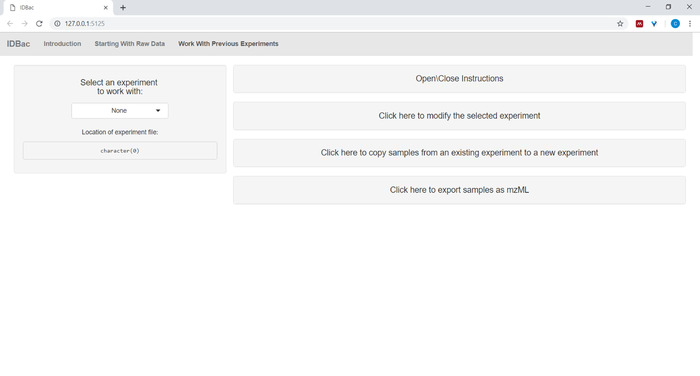
الشكل 5: صفحة "العمل مع التجارب السابقة". استخدم صفحة "العمل مع التجارب السابقة" في IDBac لتحديد تجربة لتحليلها أو تعديلها. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- (اختياري) إضافة معلومات حول العينات باستخدام القائمة اضغط هنا لتعديل التجربة المحددة. إدخال المعلومات في جدول البيانات المعبأة تلقائيًا واضغط على حفظ (الشكل6). يسمح هذا الخيار للمستخدم بتلوين بيانات التعليمات البرمجية أثناء التحليلات.
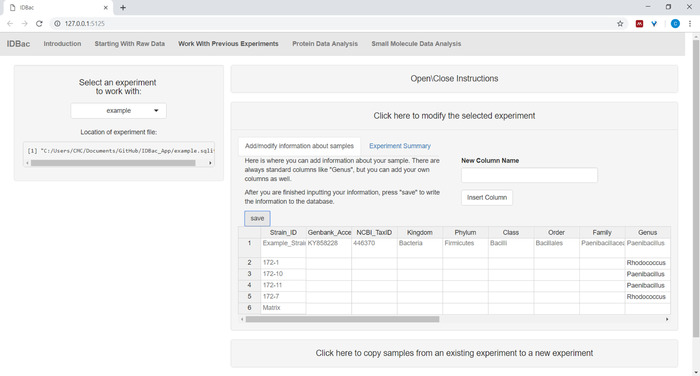
الشكل 6: معلومات عينة الإدخال. في صفحة "العمل مع التجارب السابقة" يمكن للمستخدمين إدخال معلومات حول عينات مثل الهوية التصنيفية، موقع التحصيل، وظروف العزل، وما إلى ذلك، يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
- (اختياري) نقل كافة العينات، أو مجموعة فرعية من العينات إلى تجربة جديدة أو تجربة أخرى بالنقر فوق نقل العينات من التجارب السابقة إلى تجارب جديدة/أخرى واتباع التعليمات المتوفرة (الشكل7).
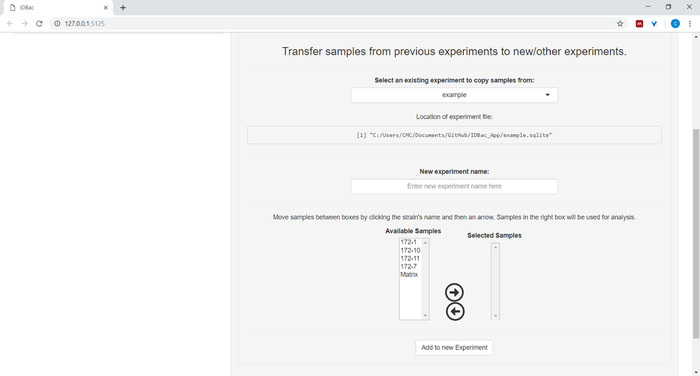
الشكل 7: نقل البيانات. تحتوي صفحة "العمل مع التجارب السابقة" على خيار نقل البيانات بين التجارب الحالية والتجارب الجديدة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- عندما تكون جاهزة لبدء التحليل، تأكد من تحديد التجربة للعمل معها. حدد إما تحليل بيانات البروتين أو تحليل بيانات جزيء صغير.
8. إعداد تحليل بيانات البروتين وإنشاء قطع أرض متطابقة
- إذا تم تحليل بيانات البروتين، انتقل أولاً إلى صفحة تحليل بيانات البروتين. اختر إعدادات انتقاء الذروة وتقييم أطياف البروتين من العيناتعبر قطع المرايا المعروضة (الشكل 8).
ملاحظة: في المؤامرات المرآة، ذروة حمراء يدل على وجود تلك الذروة فقط في الطيف العلوي، في حين تمثل القمم الزرقاء تلك التي تحدث في كل من الأطياف.
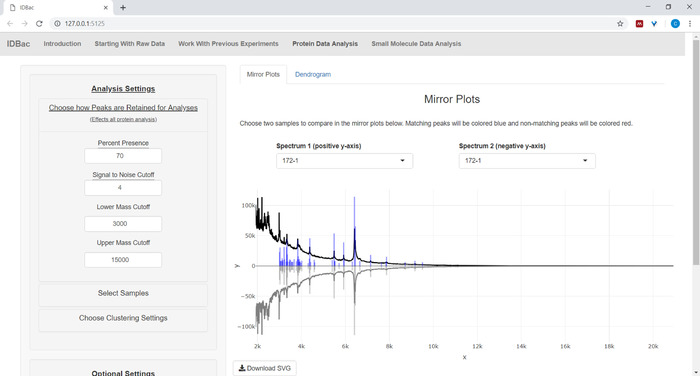
الشكل 8: اختيار كيفية الاحتفاظ بالقمم للتحليل. بعد اختيار تجربة لتحليلها، فإن زيارة صفحة "تحليل بيانات البروتين" ثم فتح القائمة "اختيار كيفية الاحتفاظ بالقمم للتحليل" يسمح للمستخدمين باختيار إعدادات مثل نسبة الإشارة إلى الضوضاء للاحتفاظ بالقمم. سيتم تحديث رسم المرآة المعروضة (أو dendrogram) تلقائيًا ليعكس الإعدادات المختارة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- تعديل النسبة المئوية للتكرارات التي يجب أن تكون فيها الذروة موجودة من أجل إدراجها في التحليلات (على سبيل المثال، إذا تم تعيين العتبة إلى 70٪ وتحدث ذروة في ما لا يقل عن 7 من أصل 10 تكرارات، سيتم تضمينها).
- باستخدام المؤامرات مرآة كتوجيه البصرية، وضبط إشارة إلى انقطاع الضوضاء التي تحتفظ القمم الأكثر "حقيقية" وأقل ضوضاء، مشيرا إلى أن أكثر تكرار وأعلى "نسبة وجود الذروة" سوف تسمح اختيار إشارة أقل إلى انقطاع الضوضاء.
- حدد الفواصل الدنيا والعليا لـ m/z، مع إملاء نطاق القيم الكتلية داخل كل طيف لاستخدامها في مزيد من التحليلات من قبل IDBac.
9- تجميع العينات باستخدام بيانات البروتين
- ضمن صفحة تحليل بيانات البروتين، حدد علامة التبويب Dendrogram. يسمح هذا بتجميع العينات في مخطط الأسنان وفقًا لتدابير المسافة التي يختارها المستخدم وخوارزميات التجميع.
- انقر فوق تحديد نماذج في القائمة واتبع الإرشادات لتحديد عينات لتضمينها في التحليلات. سيتم عرض العينات التي تحتوي على أطياف البروتين فقط داخل مربع العينات المتوفرة (الشكل9).
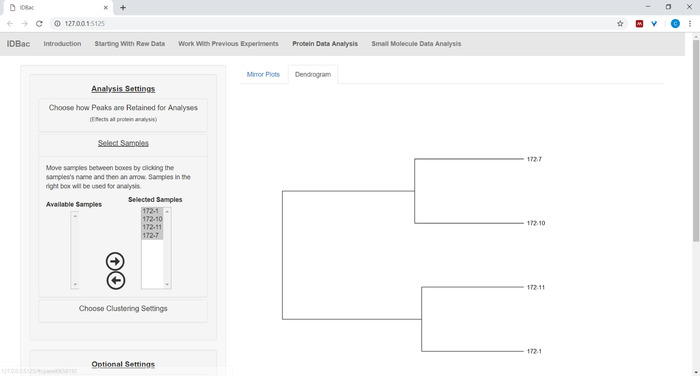
الشكل 9: حدد عينات من التجربة المختارة لإدراجها في مخطط الدندروغرام المعروض. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- استخدم القيم الافتراضية أو، ضمن اختيار إعداداتالتجميع، حدد خوارزميات المسافة والتجميع المطلوبة ليتم تطبيقها على إنشاء مخطط الطباعة.
- حدد التواجد/الغياب كإدخال. بدلاً من ذلك، إذا كنت واثقًا من ذروة ارتفاع العينات (على سبيل المثال، بعد إجراء دراسة لتقييم تقلب شدة الذروة)، فحدد الكثافات كمدخلات.
ملاحظة: في وقت النشر، يوفر IDBac المرونة في إعدادات التجميع، بالاعتماد على المستخدمين لاختيار المجموعات المناسبة. إذا لم تكن غير مألوفة مع هذه الخيارات، ويقترح لإقران إما A: "جيب التمام" المسافة، و "متوسط (UPGMA)" تجميع; أو B: "الإقليدين" المسافة، و "Ward.D2" تجميع. - لعرض قيم التمهيد على مخطط الأسنان، أدخل رقمًا بين 2 و1000 ضمن Bootstraps.
- عند الإبلاغ عن النتائج، انسخ النص ضمن فقرة الاقتراحات الخاصة بتحليل البروتين. يوفر هذا الإعدادات المعرفة من قبل المستخدم التي تم إنشاؤها مخطط الأسنان المحدد.
10. تخصيص dendrogram البروتين
- لبدء تخصيص مخطط الأسنان، افتح قائمة ضبط مخطط الأسنان (الشكل10).
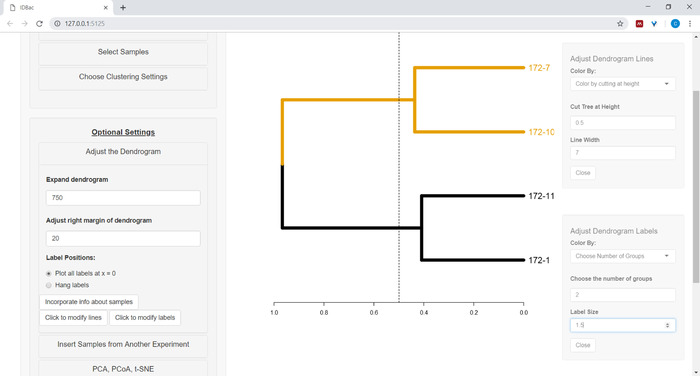
الشكل 10: ضبط مخطط الأسنان. IDBac يوفر بعض الخيارات لتعديل كيف تبدو dendrogram، ويمكن العثور على هذه داخل القائمة "ضبط Dendrogram". وهذا يشمل فروع التلوين والتسميات بواسطة k-means، أو عن طريق "قطع" الدندروغرام في ارتفاع مقدم من المستخدم. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- لتلوين خطوط و/أو تسميات الدندروغرام حدد الزر المناسب: انقر لتعديل الخطوط أو انقر لتعديل التسميات وحدد الخيارات المطلوبة.
- لرسم معلومات من جدول البيانات بجوار مخطط الأسنان (راجع الخطوة 7.2)، حدد الزر تضمين معلومات حول العينات. سيؤدي ذلك إلى فتح لوحة حيث تقوم فئة (عمود في جدول البيانات) بالملء الذاتي استنادًا إلى القيم المدخلة (الشكل11).
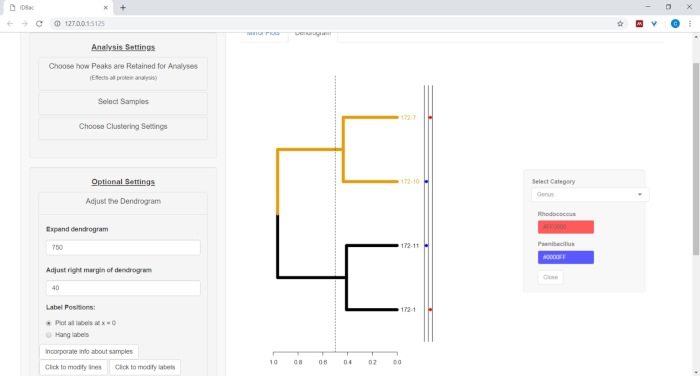
الشكل 11: إدراج معلومات عن العينات. ضمن القائمة "ضبط Dendrogram" هو الخيار "تضمين معلومات حول العينات". سيؤدي تحديد هذا إلى السماح بتخطيط معلومات حول العينات بجوار مخطط الأسنان. عينة المعلومات هو الإدخال داخل صفحة "العمل مع التجارب السابقة". الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
11. إدراج عينات من تجربة منفصلة في مخطط الأسنان
- لإدراج نماذج من تجربة أخرى، حدد زر القائمة إدراج عينات من تجربة أخرى. اتبع الإرشادات في اللوحة التي تم افتتاحها حديثًا (الشكل12).
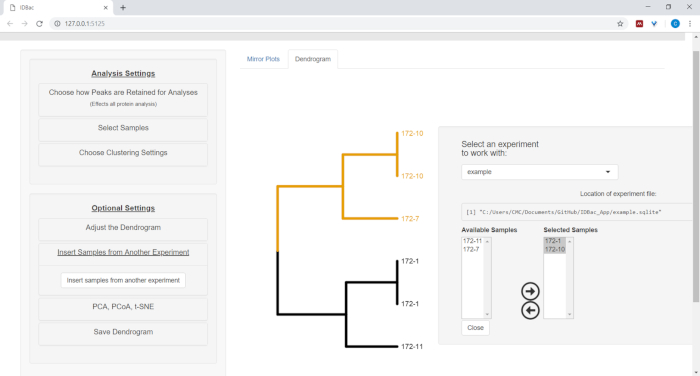
الشكل 12: إدراج عينات من قائمة تجربة أخرى. في بعض الأحيان يكون من المفيد مقارنة عينات من تجربة أخرى. استخدم القائمة "إدراج نماذج من تجربة أخرى" لاختيار عينات لتضمينها ضمن مخطط الدندروغرام المعروض حالياً. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
12. تحليل بيانات المستقلب المتخصصة وشبكات رابطة المستقلب (MANs)
- انتقل إلى صفحة شبكة جمعية المستقلب (جزيء صغير). تسمح هذه الصفحة بتصور البيانات حسب تحليل المكونات المبدئية (PCA) وMANs، التي تستخدم الشبكات الثنائية لعرض ارتباط قيم جزيء صغير m/z مع العينات.
- إذا تم إنشاء مخطط الأسنان البروتين (القسم 9)، سيتم عرضه على هذه الصفحة أيضا. انقر واسحب على مخطط الأسنان لتسليط الضوء على عينات مختارة من الفائدة لتحليلها. إذا لم يتم تمييز أي عينات أو لم يتم إنشاء مخطط دندروغرام البروتين، سوف يظهر رجل من مجموعة فرعية عشوائية أو جميع العينات، مع الاحترام (الشكل 13).
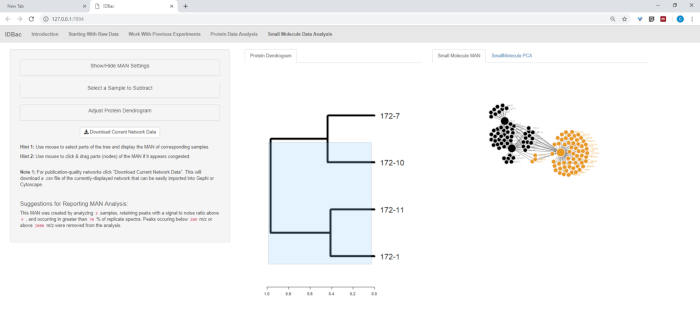
الشكل 13: صفحة تحليل بيانات الجزيئات الصغيرة. إذا تم إنشاء مخطط دندروغرام من أطياف البروتين، سيتم عرضه ضمن صفحة "تحليل بيانات الجزيئات الصغيرة". ستعرض هذه الصفحة أيضًا شبكات Metabolite Associate (MANs) وتحليل المكونات الرئيسية (PCA) لبيانات الجزيئات الصغيرة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
- لطرح مصفوفة/وسائط فارغة في MAN، افتح القائمة حدد نموذج ًا إلى طرح واختر النموذج المناسب لاستخدامه كفراغ.
- فتح القائمة إظهار / إخفاء إعدادات MAN لتحديد القيم المطلوبة للنسبة المئوية من وجود الذروة في تكرار، إشارة إلى الضوضاء، وقطع الكتلة العليا والسفلى، كما تم ذلك لأطياف البروتين في القسم 9. استخدم قطع مرآة الجزيء الصغيرة لتوجيه اختيار هذه الإعدادات.
- حدد "تنزيل بيانات الشبكة الحالية" لحفظ بيانات MAN المعروضة حالياً. يمكن استخدام هذه البيانات في برامج تحليل الشبكة غير IDBac.
- للإبلاغ عن النتائج، انسخ النص ضمن الفقرة "اقتراحات لتحليل تقرير الرجل". يوفر هذا الإعدادات المعرفة من قبل المستخدم المستخدمة لإنشاء MAN تم إنشاؤه.
13. مشاركة البيانات
- يتم حفظ كل IDBac "تجربة" كقاعدة بيانات SQLite واحدة. وهو يحتوي على الأطياف الخام mzML المحولة، والقمم المكتشفة، وجميع معلومات إدخال المستخدم حول العينات. لذلك، لمشاركة تجربة IDBac ببساطة مشاركة ملف SQlite الذي له نفس اسم التجربة (يتم عرض موقع الملف في صفحة العمل مع التجارب السابقة).
النتائج
قمنا بتحليل ست سلالات من Micromonospora chokoriensis وسلالات اثنين من عصية subtilis, التي كانت تتميز سابقا6, باستخدام البيانات المتاحة في DOI: 10.5281/zenodo.2574096. باتباع الإرشادات في علامة التبويب البدء بالبيانات الأولية، اخترنا الخيار انقر هنا لتحويل ملفات Bruker واتبعنا التعليمات التي يوفرها IDBac لكل مجموعة بيانات (الشكل 14).
بعد الانتهاء من التحويل الآلي والمعالجة المسبقة/خطوات الذروة، شرعنا في إنشاء تجربة IDBac مشتركة جديدة عن طريق نقل عينات من التجربتين إلى تجربة واحدة تحتوي على كل من عصية و عينات ميكرومونوسوبورا (الشكل 15). وشمل التحليل الناتج مقارنة أطياف البروتين باستخدام قطع المرايا، كما هو موضح في الشكل 16،الذي كان مفيداً في تقييم نوعية الأطياف وتعديل إعدادات انتقاء الذروة. يعرض الشكل 17 لقطة شاشة لنتائج تجميع البروتين مع تحديد الإعدادات الافتراضية. تم تلوين مخطط الأسنان عن طريق ضبط العتبة على المؤامرة (يظهر كخط منقط). وتجدر الإشارة إلى الفصل الواضح بين الأجناس، حيث يعزل كل من M. chokoriensis وB. subtilis التجميع بشكل منفصل.
الشكل 18والشكل 19والشكل 20 يسلط الضوء على القدرة على توليد الشبكات الانشؤية للمناطق التي يختارها المستخدم عن طريق النقر والسحب عبر مخطط الدندروغرام البروتيني. مع هذا كنا قادرين على إنشاء بسرعة MANs لمقارنة سلالات Subtilis B. فقط (الشكل 18)، فقط سلالات M. chokoriensis (الشكل 19) ، وجميع السلالات في وقت واحد (الشكل 20). وتتمثل الوظيفة الرئيسية لهذه الشبكات في تزويد الباحثين بنظرة عامة واسعة على درجة التداخل المتخصص بين البكتيريا. مع هذه البيانات في متناول اليد، والباحثين لديهم الآن القدرة على اتخاذ قرارات مستنيرة من كمية صغيرة فقط من المواد كشط من مستعمرة بكتيرية.
الشكل 14: معالجة الأطياف. تم تحويل تحميل Bruker autoFlex الأطياف ومعالجتها باستخدام IDBac. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
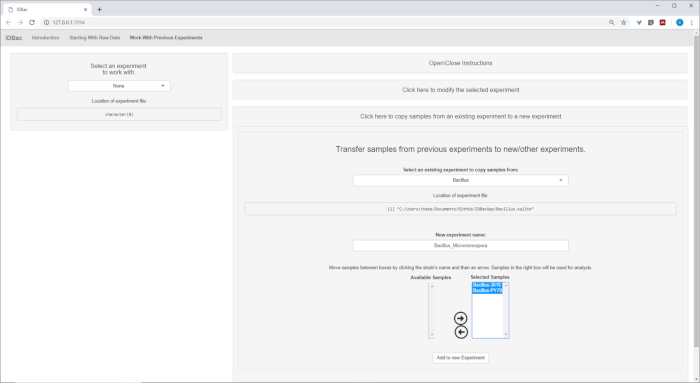
الشكل 15: تجربة IDBac المشتركة. ونظرا ً لأن الأطياف الميكرومونوسبورة وعصية جمعت على لوحات مستهدفة مختلفة من MALDI، فقد تم دمج التجربتين في وقت لاحق في تجربة واحدة - "Bacillus_Micromonsopora". وقد تم ذلك ضمن علامة التبويب "العمل مع التجارب السابقة"، باتباع الاتجاهات ضمن القائمة "نقل العينات من التجارب السابقة إلى تجارب جديدة/أخرى". الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
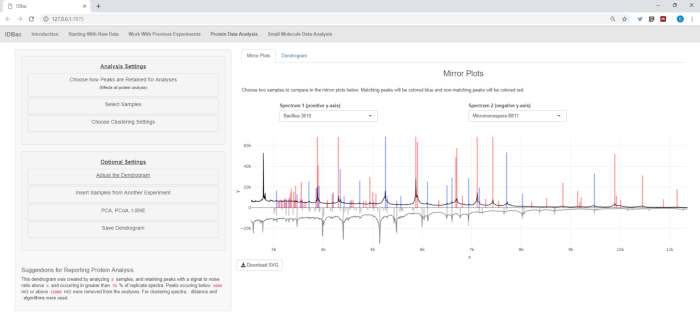
الشكل 16: المقارنة. تمت مقارنة Micromonspora وعصية الأطياف باستخدام المؤامرات مرآة داخل صفحة "تحليل بيانات البروتين". في نهاية المطاف، تم اختيار إعدادات الذروة الافتراضية. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
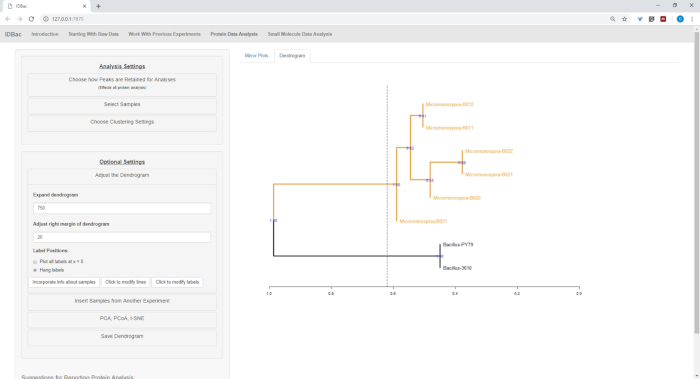
الشكل 17: التجميع الهرمي. التجميع الهرمي، باستخدام الإعدادات الافتراضية، مجمعة بشكل صحيح عصية وعزل Micromonospora. تم تلوين مخطط الأسنان من خلال "قطع" مخطط الأسنان على ارتفاع تعسفي (عرض كخط متقطع) و 100 bootstraps تستخدم لإظهار الثقة في المتفرعة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
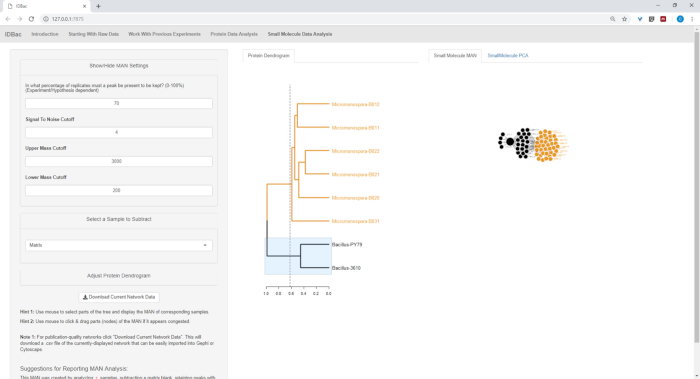
الشكل 18: أظهر الإنسان الذي تم إنشاؤه عن طريق اختيار سلالات عصية sp. من مخطط الأسنان البروتيني إنتاجًا تفاضليًا من الأيضالمتخصص. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
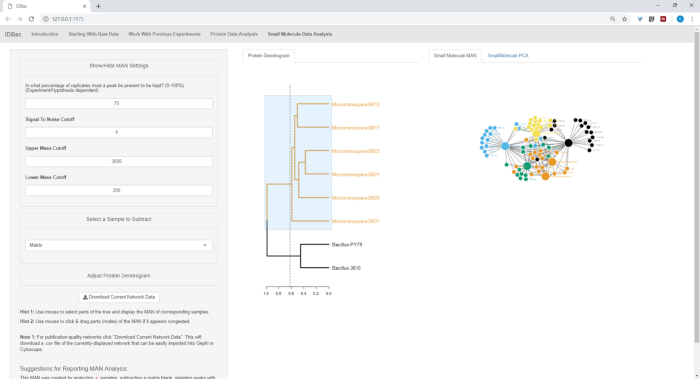
الشكل 19: أظهر الإنسان الذي تم إنشاؤه عن طريق اختيار سلالات Micromonospora sp. الست من مخطط الأسنان البروتيني إنتاجًا تفاضليًا من الأيض المتخصص. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
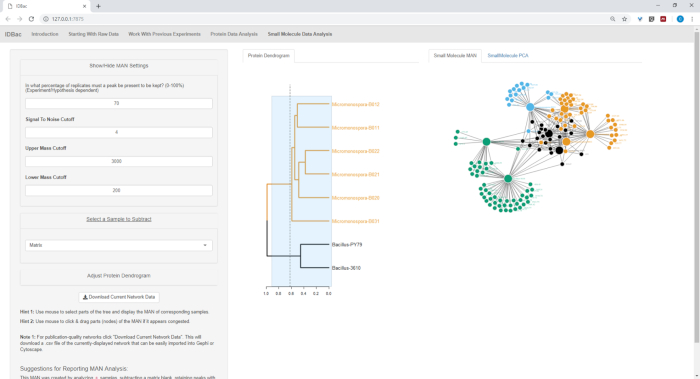
الشكل 20: MAN of Bacillus sp. and Micromonospora sp. s. السلالات التي تظهر إنتاج تفاضلي من الأيض المتخصصة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
بروتوكول IDBac تفاصيل البروتين البكتيري والمستقلب المتخصصة الحصول على البيانات وتحليل ما يصل إلى 384 عزلات بكتيرية في 4 ح من قبل باحث واحد. مع IDBac ليست هناك حاجة لاستخراج الحمض النووي من العزلات البكتيرية أو توليد مقتطفات المستقلب المتخصصة من مرق التخمير السائل وتحليلها باستخدام أساليب كروماتوغرافية. بدلا من ذلك، يتم جمع البروتين وبيانات المستقلب المتخصصة ببساطة عن طريق نشر المواد من المستعمرات البكتيرية مباشرة على لوحة الهدف MALDI. وهذا يقلل إلى حد كبير من الوقت والتكلفة المرتبطة بالتقنيات البديلة مثل تسلسل الجينات 16S rRNA وLCMS9.
من المهم إضافة مصفوفة فارغة وبقع معايرة إلى لوحة MALDI، ونحن نوصي باستخدام عدد مناسب من النسخ المتماثلة لضمان استنساخ والثقة الإحصائية. وسوف تكون أعداد النسخ المتماثلة تعتمد على التجربة. على سبيل المثال، إذا كان المستخدم ينوي التمييز بين آلاف المستعمرات ومجموعة من لوحات التنوع البيئي، فقد يكون من الضروري إجراء نسخ متماثل أقل (يقوم مختبرنا بجمع ثلاثة نسخ تقنية في كل مستعمرة). بدلا من ذلك، إذا كان المستخدم يرغب في إنشاء قاعدة بيانات مخصصة من سلالات من taxa البكتيرية محددة لتحديد بسرعة تصنيفات الأنواع الفرعية من عزلات غير معروفة، ثم أكثر تكرارات مناسبة (مختبرنا يجمع ثمانية نسخ بيولوجية لكل سلالة).
IDBac هو أداة للتثواب بسرعة عالية ذات الصلة العزلات البكتيرية على أساس المعلومات التصنيفية المفترضة وإنتاج المستقلب المتخصصة. يمكن أن تكمل أو تكون بمثابة مقدمة لطرق متعامدة مثل التحليلات الوراثية المتعمقة، والدراسات التي تنطوي على إنتاج المستقلب والوظيفة، أو توصيف بنية المستقلب المتخصصة عن طريق التحليل الطيفي للرنين المغناطيسي النووي و / أو LC-MS/MS.
إنتاج المستقلب المتخصصة (IDBac MANs) هو عرضة للغاية لظروف النمو البكتيري، وخاصة باستخدام وسائل الإعلام المختلفة، وهو الحد المحتمل للطريقة. ومع ذلك يمكن استغلال هذه الصفات من قبل المستخدم، كما IDBac يمكن أن تولد بسهولة MANs تبين الاختلافات في إنتاج المستقلب المتخصصة في ظل مجموعة متنوعة من ظروف النمو. من المهم أن نلاحظ أنه في حين أن بصمات المستقلب المتخصصة قد تختلف حسب حالة النمو، فقد أظهرنا في السابق أن بصمات البروتين لا تزال مستقرة نسبيا عبر هذه المتغيرات (انظر Clark et al.6). عند التعامل مع لوحات التنوع البيئي، نوصي بتنقية العزلات البكتيرية قبل التحليل من أجل تقليل المساهمات المحتملة من الحديث البكتيري المجاور.
وأخيرا، فإن عدم وجود قاعدة بيانات عامة يمكن البحث فيها من بصمات البروتين MS هو قصور كبير في استخدام هذه الطريقة لتصنيف البكتيريا البيئية غير معروف. أنشأنا IDBac مع هذا في الاعتبار، وشملت التحويل الآلي للبيانات إلى شكل مفتوح المصدر مقبول من قبل المجتمع (mzML)10،11،12 وتصميم البرنامج للسماح بالبحث، وتقاسم، وإنشاء قواعد بيانات مخصصة. ونحن بصدد إنشاء قاعدة بيانات عامة كبيرة (>000 10 سلالات تتميز بالكامل)، مما سيسمح بتصنيف بعض العزلات إلى مستوى الأنواع، بما في ذلك وصلات بأرقام الانضمام إلى GenBank عند توافرها.
IDBac هو مفتوح المصدر والتعليمات البرمجية متاحة لأي شخص لتخصيص تحليل البيانات واحتياجات التصور. نوصي المستخدمين باستشارة مجموعة واسعة من المؤلفات (ساور وآخرون7وسيلفا وآخرون5)للمساعدة في دعم وتصميم أهدافهم التجريبية. نستضيف منتدى للمناقشة في: https://groups.google.com/forum/#!forum/idbac ووسيلة للإبلاغ عن القضايا مع البرنامج في: https://github.com/chasemc/IDBacApp/issues.
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
وقد دعم هذا العمل المعهد الوطني للعلوم الطبية العامة منحة R01 GM125943، المنحة الجغرافية الوطنية CP-044R-17؛ منحة صندوق البحوث الآيسلندي ة 152336-051؛ وجامعة إلينوي في صناديق بدء التشغيل شيكاغو. كما نشكر المساهمين التالين: الدكتورة أماندا بولمان على مساعدتها في معايير اكتساب بروتين MALDI-TOF MS. الدكتور تيري مور والدكتور أتول جاين لبلورة ألفا-سيانو-4-هيدروكسيسيناميك حمض مصفوفة (CHCA).
Materials
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
References
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved