Method Article
微生物タンパク質および特殊代謝産物データの分析にオープンソースのMALDI TOF-MS IDBacパイプラインを使用
要約
IDBacは、細菌コロニーから掻き取られた細胞材料に収集された、無傷のタンパク質と特殊な代謝スペクトルの両方からのデータを統合するオープンソースの質量分析ベースのバイオインフォマティクスパイプラインです。このパイプラインにより、研究者は数百から数千の細菌コロニーを種置分類群に迅速に組織し、特殊な代謝産物生産に基づいてそれらをさらに区別することができます。
要約
栄養寒天で増殖する細菌コロニーの細菌系統と特殊な代謝産物産生との関係を可視化するために、低コストで高スループットのマトリックス支援レーザー脱着/イオン化を実現するIDBacを開発しました。飛行時間質量分析(MALDI-TOF MS)バイオインフォマティクスパイプライン。IDBacソフトウェアは、非専門家のために設計されており、自由に利用可能であり、細菌のコロニーの数〜数千を分析することができます。ここでは、MALDI-TOF MS解析、MS計測器の操作、IDBacにおけるデータ処理と可視化のための細菌コロニーの調製手順を紹介する。特に、タンパク質MS指紋に基づいて細菌をデンドログラムにクラスター化する方法をユーザーに指示し、特殊な代謝産物データから代謝物アソシエーションネットワーク(MAN)をインタラクティブに作成する方法を指示します。
概要
細菌機能を研究する研究者の主な障壁は、微生物の分類的アイデンティティと特殊な代謝産物を生産する能力を迅速かつ同時に評価する能力です。これは、細菌の系統と環境から分離された細菌の大部分における特殊な代謝産物産生との関係を理解する上で重要な進歩を防いだ。タンパク質フィンガープリントを用いて細菌を同定するMSベースの方法は、1、2、3、4についてよく説明されているが、これらの研究は一般的に、単離物の小群で行われている。種固有の方法で。重要なのは、環境における微生物機能の主要な推進要因である特殊代謝産物産生に関する情報は、これらの研究に組み込まれないままである。Silva et al.5は最近、MALDI-TOF MSの使用不足を詳細に説明し、専門の代謝産物を分析し、現在のバイオインフォマティクスのボトルネックを解消するためのソフトウェアの不足を詳述した包括的な歴史を提供した。これらの欠点に対処するために、MALDI-TOF MS6の線形モードとリフレクトロン モードの両方を統合するバイオインフォマティクス パイプラインである IDBac を作成しました。これにより、ユーザーはタンパク質と特殊な代謝産物MS指紋の両方に基づいて細菌単離物を迅速に視覚化し、区別することができます。
IDBac は、コスト効率が高く、スループットが高く、レイ ユーザー向けに設計されています。それは自由に利用できる(chasemc.github.io/IDBac)、MALDI-TOF質量分析計へのアクセスだけを要求する(リフレクターモードは、特殊な代謝産物分析のために必要とされる)。サンプル調製は、単純な「拡張直接転写」方法7、8に依存し、データは単一のMALDIターゲットスポット上で連続した線形および反射ロンの獲得で収集される。IDBacを使用すると、サンプル調製、データ取得、データビジュアライゼーションなど、数百のコロニーの植物学および特殊な代謝産物の生産を4時間以内に分析することが可能です。これは、細菌(遺伝子シーケンシングなど)を同定し、代謝出力(液体クロマトグラフィー質量分析法[LCMS]および同様のクロマトグラフィー法)を分析する従来の方法に比べて、かなりの時間とコストの優位性を提示します。
線形モード解析で得られたデータを使用して、IDBacはタンパク質スペクトルの関連性を表すために階層クラスタリングを採用しています。スペクトルは主にイオン化リボソームタンパク質を表すので、サンプル中に存在する系統的多様性の表現を提供する。さらに、IDBacは、代謝物アソシエーションネットワーク(MAN)として特殊な代謝物指紋を表示するためにリフレクターモードデータを組み込んでいます。MANは、細菌単離物間の共有およびユニークな代謝産物産生を容易に視覚化することを可能にするバイパルティネットワークです。IDBacプラットフォームにより、研究者はタンパク質と特殊な代謝産物データの両方を並行して分析できますが、1つのデータタイプのみが取得された場合は個別に分析できます。重要なのは、IDBac は Bruker および Xiamen インストゥルメントの生データ、および txt、タブ、csv、mzXML、および mzML から生データを処理することです。これにより、データ・セットの手動変換と書式設定が不要になり、ユーザー・エラーや MS データの誤った処理のリスクが大幅に軽減されます。
プロトコル
1. マルディマトリックスの調製
- MSグレード溶媒中の10mg/mL MALDIグレード、および/または再結晶化α-シアノ-4-ヒドロキシシナミン酸(CHCA):50%アセトニトリル(ACN)、47.5%水(H2O)、2.5%トリフルオロ酢酸(TFA)を再結晶化する。例: 100 μL 溶液 = 50 μL ACN + 47.5 μL H2O + 2.5 μL TFA + 1 mg CHCA
- MALDIプレートスポットあたりのマトリックス溶液の少なくとも1 μLを調べ、溶液中まで渦または超音波処理(約5分の超音波処理または可視固体なし)を調べます。
注意:TFAは、適切な個人用保護具を装着しながら化学ヒュームフードで取り扱うべき強酸であり、接触や吸入で皮膚、目、気道に損傷を与える可能性があります。
注:CHCAは吸湿性と光感受性であり、デシケータ内の琥珀色のバイアルに格納する必要があります。多くの MALDI マトリックス オプションを使用できます。CHCAは、細菌のタンパク質プロファイリングのために最も一般的ですが、また、専門的な代謝産物分析のために働きます。マトリックスの選択は、個々のユーザー/実験のニーズによって異なります。
- MALDIプレートスポットあたりのマトリックス溶液の少なくとも1 μLを調べ、溶液中まで渦または超音波処理(約5分の超音波処理または可視固体なし)を調べます。
2. MALDIターゲットプレートの調製
注:詳細については、ザウアーら7を参照してください。
- MALDIプレートをメタノール(HPLCグレード以上)ですすり、柔らかい紙拭きで拭き取ります。ターゲットプレートの表面が永久に損傷する可能性がありますので、ターゲットプレートをクリーニングする際には研磨ブラシを使用しないでください。
- タンパク質と特殊な代謝産物のキャリブラント スポットを割り当てます。MALDIプレートの不規則性と計器のドリフトを考慮して、サンプル母集団全体でキャリブレーションスポットを均等に整理します。スタディに適切な数のメディア/マトリックスブランクスポットを割り当てます。これらのスポットには、メディアとマトリックスのみが含まれるか、マトリックスのみが含まれます。
- 無菌のつまようじを使用して、細菌のコロニーの小さな部分をMALDIプレート上の適切な場所に移します。細菌のコロニーをその場に均等に広げる。スポットは可能な限り平坦に表示されます。
注:より粘膜/非晶質である細菌コロニーを平坦化することが容易になります。より堅い/固体コロニーのために、MALDIスポットに細胞質量の目に見えるクラスターを残さないようにしてください(図1)。
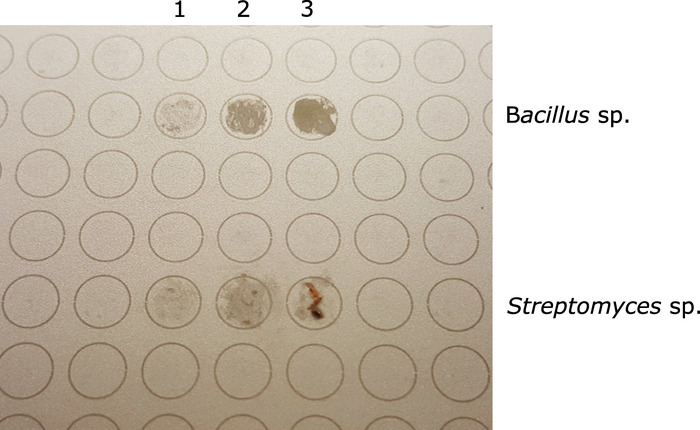
図1:ギ酸およびMALDIマトリックスを添加する前に2つの異なる単離物を示すMALDI標的プレート(上3箇所 -バチルスsp.;下3スポット-連鎖膿菌sp.)。どちらの場合も、列3は過剰なサンプルを表します。列2は、適切な量のサンプルを表します。列1は、MALDI 分析のサンプルが不十分です。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- 無菌のつまようじを使用して、MALDIプレート上の適切なスポットに最小限の量の寒天/メディアを転送することにより、マトリックス/メディアコントロールを準備します。
- マトリックス制御スポットを含む各サンプルスポットに70%質量分析グレードの1 μLをオーバーレイします。化学ヒュームフード(約5分)で酸を完全に乾燥させます。
注意:ギ酸は腐食性の化学物質であり、化学ヒュームフードで処理する必要があります。吸入すると気道にダメージを与える可能性があります。 - 各サンプルスポットおよびマトリックス/メディア制御スポットに、調製されたMALDIマトリックス溶液の1 μLを追加します。マトリックス溶液を完全に乾燥させます(約5分)。
注:MALDI-TOF質量分析計で分析できるまで、プレートを暗闇の中でデシケータに保存することができます。許容貯蔵時間はサンプルの安定性によって変わる。 - 割り当てられたキャリブレーションスポットに0.5~1.0 μLのキャリブラントを追加し、続いて1 μL MALDIマトリックス溶液を追加します。得られた溶液を上下にピペットして混ぜます。MALDI-TOF質量分析計に導入する前に、すべてのスポットが完全に乾燥するようにします。
注:タンパク質と特殊な代謝物キャリブラントは、両方とも分解の影響を受けやすいので、MALDI分析の30分以内に添加する必要があります。
3. データ取得
注: データ取得の一般的なパラメータは、表 1に示されています。
| パラメーター | タンパク質 | 特殊代謝産物 |
| マススタート (Da) | 1920年 | 60歳 |
| マスエンド (Da) | 21000年 | 2700年 |
| 質量偏向 (Da) | 1900年 | 50歳 |
| ショット | 500名 | 1000年 |
| 周波数(Hz) | 2000年 | 2000年 |
| レーザーサイズ | 大きな | 媒体 |
| マックスストドデブ (ppm) | 300人 | 30歳 |
表 1.
- 使用する器具に固有のプロトコルに従って、タンパク質と特殊な代謝物のキャリブレーションの両方を設定します。
- スペクトルを取得する際に使用する最適なレーザーパワーと検出器のゲインを決定するために、いくつかの別々のターゲットスポットをテストします(これは日々、計測器によって異なります)。
注:図2Aと図3Aは最適なスペクトルを示し、図2Dと図3Dは低品質スペクトルの例です。
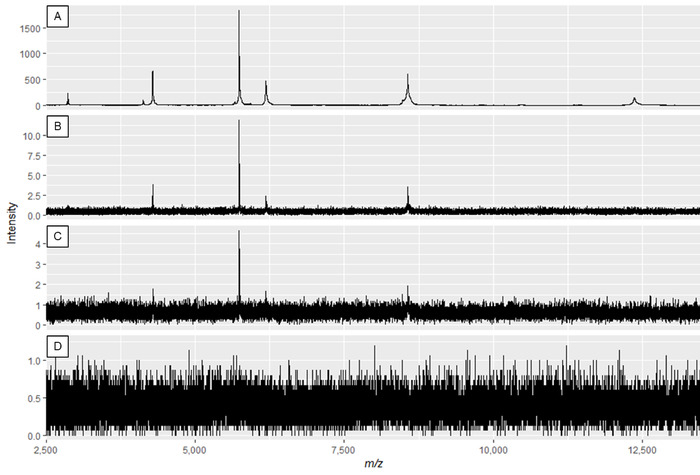
図 2:レーザーパワーおよび検出器ゲインを修飾する効果を示すタンパク質スペクトルの例。スペクトル品質はパネルAに最適であり、パネルCとDのスペクトル品質が不十分になるまで減少します。パネルBのスペクトルは使用可能なピークをもたらすかもしれませんが、パネルAは最適なデータを表示します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
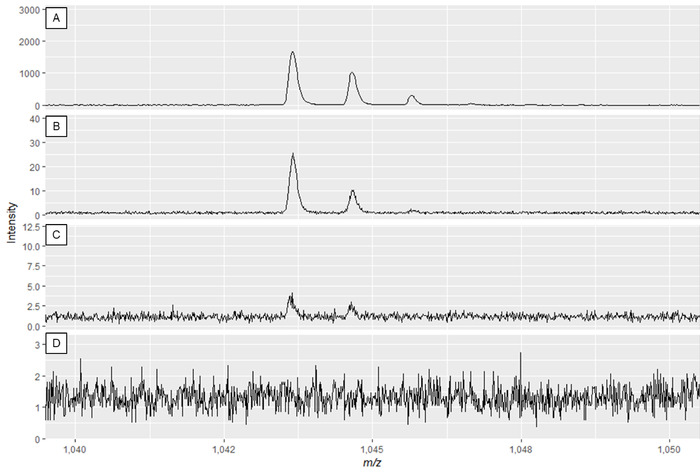
図 3:レーザーパワーおよび検出器ゲインを修飾する効果を示す特殊な代謝スペクトルを例とする。スペクトル品質はパネルAに最適であり、パネルCとDのスペクトル品質が不十分になるまで減少します。パネルBのスペクトルは使用可能なピークをもたらすかもしれませんが、パネルAは最適なデータを表示します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- スペクトルを取得し、タンパク質スペクトルを1つのフォルダに保存し、特殊な代謝スペクトルを2番目の別個のフォルダに保存します。
4. MALDIターゲットプレートのクリーニング(ザウアーら7から適応)
- MALDIターゲットプレートをホルダーから取り出し、アセトンですすいでください。
- 無研至の液体石鹸で洗い、微量タンパク質や脂質を除去し、柔らかい紙拭き取り/柔らかい毛の歯ブラシを取り除きます。
- 脱イオン水で約2分間すすいで、石鹸を完全に取り除きます。
- 対象プレートを水中(HPLCグレード以上)で5分間超音波処理します。
- 対象プレートを水ですすいでください(HPLCグレード以上)。
- ターゲットプレートをメタノール(HPLCグレード以上)ですすください。
5. IDBacソフトウェアのインストール
- IDBac ソフトウェアをダウンロードします。
注: 永続的なバージョン管理されたバックアップもダウンロードできます (資料の表を参照)。 - ダウンロードした "Install_IDBac.exe" をダブルクリックしてインストーラを起動し、画面の指示に従います。
6. 生データから始める
注: 各データ処理ステップの詳細な説明と手順は IDBac 内に埋め込まれていますが、主な分析と対話式入力については以下に説明します。
- IDBac デスクトップ ショートカットをダブルクリックして IDBac を起動します。IDBac はデフォルトで[はじめに]タブで開きます。
- [更新の確認]ボタンを使用して、最新バージョンの IDBac が使用されていることを確認します (インターネットアクセスが必要です)。新しいバージョンが利用可能な場合、IDBac は更新プログラムを自動的にダウンロードしてインストールし、その後 IDBac を再起動するように要求します。
- [生データで開始]タブをクリックし、IDBac で使用するデータの種類をメニューから選択します。アプリ内の指示に従って続行します。
- データ ファイルの変換と処理を設定する場合は、プロンプトが表示された実験の説明名を入力します (図 4を参照)。実験は後でアルファベット順に表示されますので、グループ属性(例えば「バチルス-trials_experiment-1」)を使って実験名を開始するのが役立つ戦略です。"バチルス試験_実験-2")。
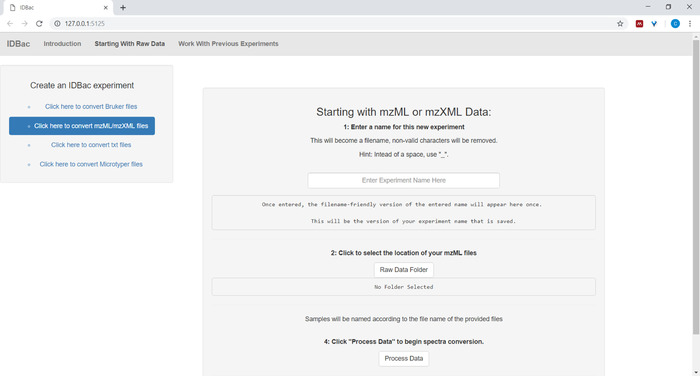
図 4: IDBac データ変換および前処理ステップ。IDBac は、未加工スペクトルを開いている mzML 形式に変換し、各実験のデータベースに mzML、ピーク リスト、およびサンプル情報を格納します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
7. 以前の実験を行う
- ファイルを変換して IDBac で処理した後、または実験を再分析する場合はいつでも、[前の実験を使用して作業] ページに移動し、使用する実験を選択します (図5)。
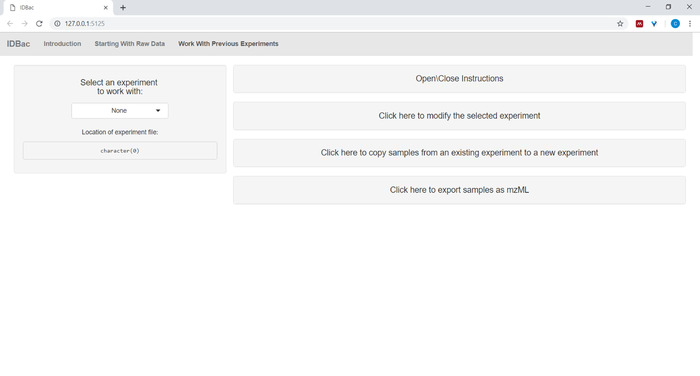
図 5: 「以前の実験を行う」ページ。IDBac の「以前の実験を使用する」ページを使用して、分析または変更する実験を選択します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- (オプション)メニューを使用してサンプルに関する情報を追加する 選択した実験を変更するには、ここをクリックします。自動入力されたスプレッドシートに情報を入力し、[保存] (図 6)を押します。 このオプションを使用すると、解析中にデータを色分けできます。
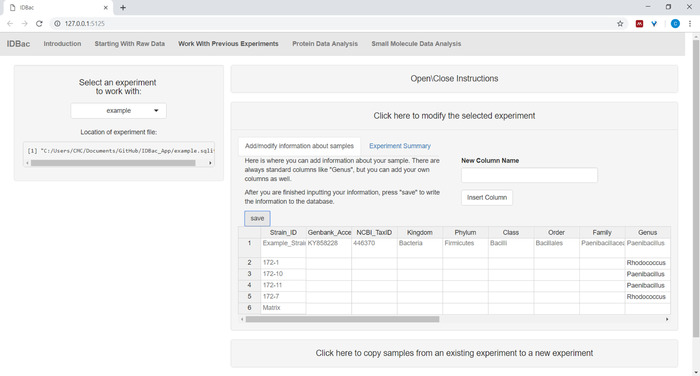
図 6: サンプル情報を入力します。「以前の実験で作業」ページ内では、分類のアイデンティティ、収集場所、隔離条件などのサンプルに関する情報を入力できます。
- (オプション)以前の実験から新しい/他の実験にサンプルを転送し、提供された指示に従ってサンプルの全部または別の実験にサンプルのサブセットを転送します(図7)。
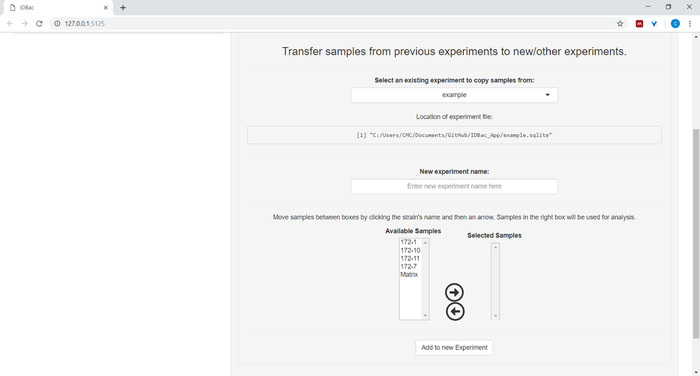
図 7: データの転送。[以前の実験を使用して作業する] ページには、既存の実験と新しい実験の間でデータを転送するオプションが含まれています。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- 解析を開始する準備ができたら、使用する実験が選択されていることを確認します。タンパク質データ分析または低分子データ分析のいずれかを選択します。
8. タンパク質データ解析の設定とミラープロットの作成
- タンパク質データを分析する場合は、まず[タンパク質データ分析]ページに移動します。ピークピッキング設定を選択し、表示されたミラープロットを介してサンプルのタンパク質スペクトルを評価します(図8)。
注: ミラー プロットでは、赤いピークはトップ スペクトルでのみそのピークの存在を示し、青いピークは両方のスペクトルで発生するピークを表します。
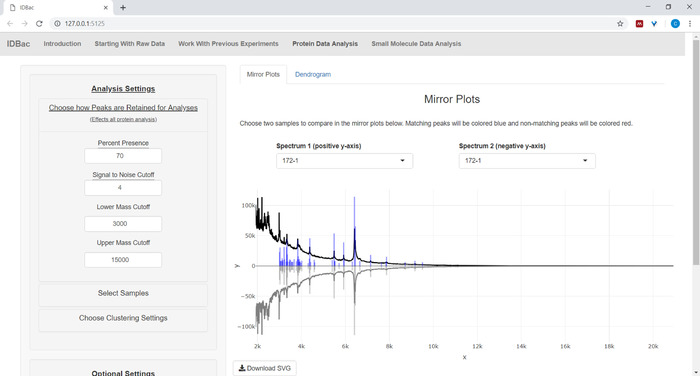
図 8: 解析のためにピークを保持する方法を選択します。分析する実験を選択した後、「タンパク質データ分析」ページにアクセスし、「分析のためにピークを保持する方法を選択する」メニューを開くと、ピークを保持するための信号対雑音比などの設定を選択できます。表示されたミラープロット(またはデンドログラム)は、選択した設定を反映するように自動的に更新されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- 分析にピークを含める必要がある反復のパーセンテージを調整します (たとえば、しきい値が 70% に設定され、10 回の反復のうち少なくとも 7 回でピークが発生した場合は、そのピークが含まれます)。
- ミラープロットを視覚的なガイダンスとして使用して、最も「本物の」ピークと最小ノイズを保持するノイズカットオフに信号を調整し、より多くの反復と高い「パーセンテージピークプレゼンス」値が高いほど、ノイズカットオフに対する低い信号の選択が可能であることを指摘します。
- 下側のm/zカットオフを指定し、IDBac によるさらなる分析に使用する各スペクトル内の質量値の範囲をディクテーションします。
9. タンパク質データを用いてサンプルをクラスタリングする
- [タンパク質データ分析]ページで、[デンドログラム]タブを選択します。これにより、ユーザーが選択した距離メジャーとクラスタリング アルゴリズムに従って、サンプルをデンドログラムにグループ化できます。
- メニューの[サンプルを選択]をクリックし、指示に従って分析に含めるサンプルを選択します。タンパク質スペクトルを含むサンプルのみが[利用可能なサンプル]ボックス(図9)に表示されます。
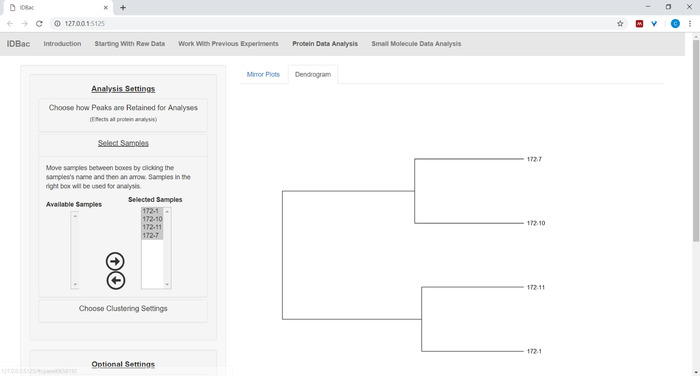
図 9: 選択した実験からサンプルを選択し、表示されたデンドログラム内に含めます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- 既定値を使用するか、[クラスタリング設定を選択]で、デンドログラムの生成に適用する必要な距離アルゴリズムとクラスタリングアルゴリズムを選択します。
- 入力として[プレゼンス/不在]を選択します。あるいは、サンプルのピーク高さに自信がある場合(例えば、ピーク強度の変動性を評価するスタディを行った後)、入力として強度を選択します。
注: 公開時点では、IDBac はクラスタリングの設定を柔軟に行い、ユーザーが適切な組み合わせを選択するように依存します。これらのオプションに慣れていない場合は、A:"cosine"距離と"平均(UPGMA)"クラスタリングのいずれかをペアリングすることをお勧めします。または B: "ユークリッド" 距離、および "Ward.D2" クラスタリング。 - デデンドログラムにブートストラップ値を表示するには、ブートストラップの下に 2 から 1000 の間の数値を入力します。
- 結果を報告する場合は、タンパク質分析の提案段落内にテキストをコピーします。これにより、特定のデンドログラムを生成したユーザー定義の設定が提供されます。
10. タンパク質デンドログラムのカスタマイズ
- デンドログラムのカスタマイズを開始するには、[デンドログラムの調整]メニューを開きます (図 10)。
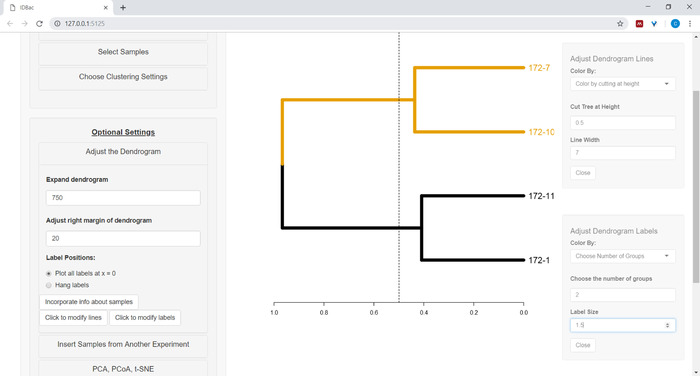
図 10:デンドログラムを調整します。IDBacは、デンドログラムがどのように見えるかを変更するためのいくつかのオプションを提供し、これらはメニュー「デンドログラムを調整」内で見つけることができます。これには、k 平均による分岐とラベルの色付け、またはユーザーが提供する高さでデンドログラムを「切断」する色付けが含まれます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- デンドログラムの線やラベルに色を付けるには、適切なボタンを選択します: [クリック] をクリックして行を変更するか、[クリックしてラベルを変更して、目的のオプションを選択します。
- デンドログラムの横にあるスプレッドシートから情報をプロットするには(手順 7.2 を参照)、[サンプルに関する情報を組み込む]ボタンを選択します。これにより、入力された値に基づいてカテゴリ (スプレッドシート内の列) が自己入力されるパネルが開きます (図 11)。
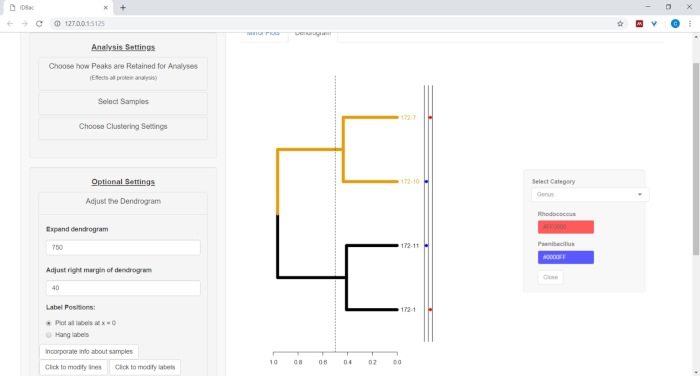
図 11: サンプルに関する情報を組み込む。「デンドログラムを調整する」メニューには、「サンプルに関する情報を組み込む」オプションがあります。これを選択すると、デンドログラムの横にサンプルに関する情報をプロットできます。サンプル情報は、「以前の実験を使用して作業」ページ内で入力されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
11. 別の実験のサンプルをデンドログラムに挿入する
- 別の実験からサンプルを挿入するには、メニュー ボタンを選択して、別の実験からサンプルを挿入します。新しく開いたパネルの指示に従ってください (図 12)。
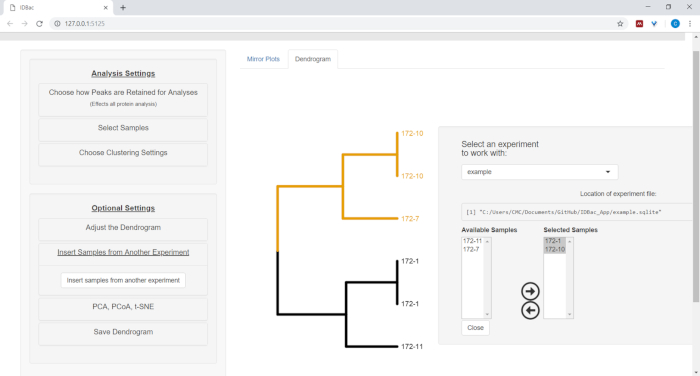
図 12: 別の実験メニューからサンプルを挿入します。別の実験のサンプルを比較すると便利な場合があります。「別の実験からサンプルを挿入」メニューを使用して、現在表示されているデンドログラム内に含めるサンプルを選択します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
12. 特殊代謝産物データと代謝関連ネットワークの解析(MAN)
- 代謝産物協会ネットワーク(低分子)のページに進みます。このページでは、原理成分分析(PCA)とMANによるデータビジュアライゼーションを可能にし、バイパルティネットワークを使用して、小分子m/z値とサンプルの相関関係を表示します。
- タンパク質デンドログラムが作成された場合(セクション9)、このページにも表示されます。デンドログラム上でクリックアンドドラッグして、分析対象の選択したサンプルをハイライト表示します。サンプルが強調表示されていない場合、またはタンパク質デンドログラムが作成されなかった場合は、ランダムサブセットまたはすべてのサンプルの MAN が丁寧に表示されます (図 13)。
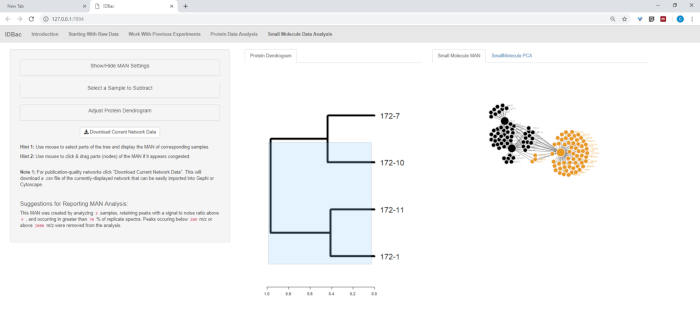
図13:小分子データ分析」ページ。デンドログラムがタンパク質スペクトルから作成された場合、「低分子データ分析」ページに表示されます。このページでは、低分子データの代謝アソシテネットワーク(MAN)と原理成分分析(PCA)も表示されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- MAN で行列/メディアブランクを減算するには、メニューを開き、減算するサンプルを選択し、ブランクとして使用する適切なサンプルを選択します。
- セクション9のタンパク質スペクトルで行われたように、メニューを開いて、レプリケート、ノイズへの信号、上下の質量カットオフのピークプレゼンスのパーセンテージの目的の値を選択します。これらの設定の選択を導くために、小分子ミラープロットを使用します。
- 現在表示されている MAN のデータを保存するには、「現在のネットワーク データをダウンロード」を選択します。これらのデータは、IDBac 以外のネットワーク解析ソフトウェアで使用できます。
- 結果を報告するには、MAN 分析の提案の段落内にテキストをコピーします。これにより、作成された MAN の生成に使用されるユーザー定義の設定が提供されます。
13. データの共有
- 各 IDBac "実験" は、単一の SQLite データベースとして保存されます。これには、変換された mzML 生スペクトル、検出されたピーク、およびサンプルに関するすべてのユーザー入力情報が含まれています。したがって、IDBac 実験を共有するには、実験と同じ名前の SQlite ファイルを共有するだけです (ファイルの場所は、前の実験の操作ページに表示されます)。
結果
DOIで入手可能なデータを用いて、以前に特徴づけられた6株のマイクロモノスポラチョコリエンシスと2株のバチルス・サチリスを分析した: 10.5281/zenodo.2574096.[生データを使用して開始] タブの指示に従って、[ここをクリック] を選択して Bruker ファイルを変換し、各データセットに対して IDBac が提供する手順に従います (図 14)。
自動変換と前処理/ピークピーク時のステップが完了した後、我々は、2つの実験からサンプルをバチルスと両方を含む単一の実験に移すことによって、新しい組み合わせIDBac実験を作成し、新しい複合IDBac実験を作成しました。マイクロモノソポラサンプル(図15)。結果として得られた分析では、図16に描かれたミラープロットを用いたタンパク質スペクトルの比較が含まれており、これはスペクトル品質の評価とピークピッキング設定の調整に役立ちました。図 17は、既定の設定が選択されたタンパク質 クラスタリング結果のスクリーンショットを表示します。デンドログラムは、プロットのしきい値を調整することによって色分けされました(点線で表示されます)。注目すべきは、M.チョコリエンシスとB.サブティリスの両方が別々にクラスタリングを分離して、属間の明確な分離である。
図 18、図 19、および図 20は、タンパク質デンドログラムをクリックしてドラッグして、ユーザーが選択した領域の MAN を生成する機能を強調表示します。これにより、B.サブティリス株(図18)、M.チョコリエンシス株(図19)、およびすべての株を同時に比較するMANを迅速に作成することができました(図20)。これらのネットワークの主な機能は、細菌間の特殊な代謝産物の重複の程度の広範な概要を研究者に提供することです。これらのデータを手にして、研究者は細菌のコロニーから削り取られた少量の材料から情報に基づいた意思決定を行う能力を持つようになりました。
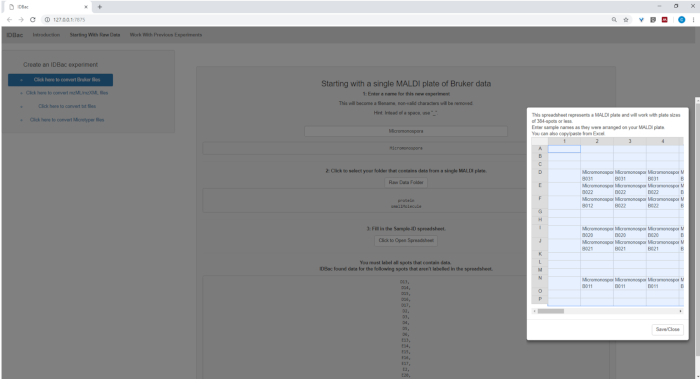
図14:スペクトル処理。ダウンロードしたBruker autoFlexスペクトルは、IDBacを使用して変換および処理されました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
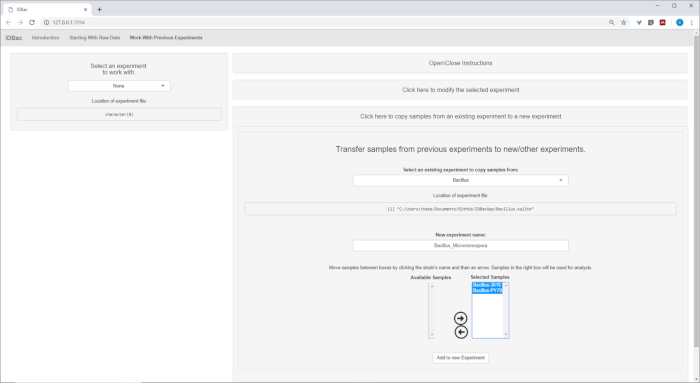
図15:組み合わせたIDBac実験。マイクロモノスポラとバチルススペクトルは異なるMALDIターゲットプレート上で収集されたため、2つの実験は、その後、単一の実験に組み合わされました-「バチルス_マイクロモンソポラ」。これは「以前の実験で作業する」タブで行われ、メニュー「以前の実験から新しい/他の実験にサンプルを転送する」の指示に従って行われました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
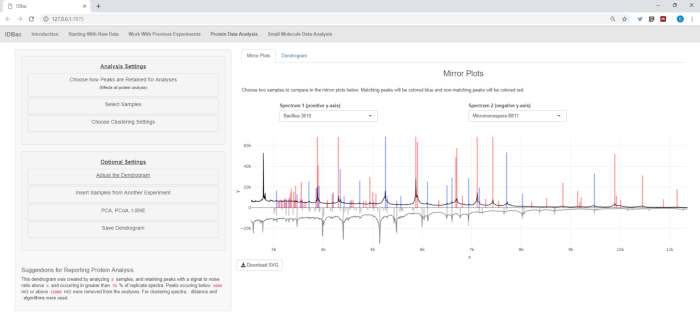
図 16: 比較。マイクロモンスポラとバチルススペクトルを「タンパク質データ分析」ページ内のミラープロットを用いて比較した。最終的に、デフォルトのピーク設定が選択されました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
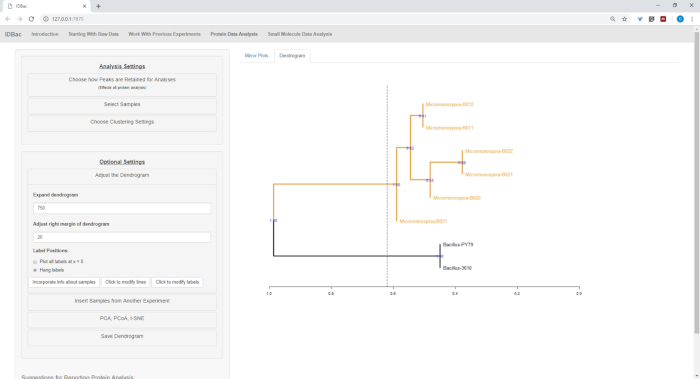
図 17: 階層クラスタリング。階層クラスタリングは、デフォルト設定を使用して、正しくグループ化されたBacillusとMicromonospora分離。デンドログラムは、任意の高さでデンドログラムを「切断」し(破線として表示)、分岐に自信を示すために使用される100のブートストラップによって着色されました。この図のより大きなバージョンを表示するには、ここをクリックしてください。
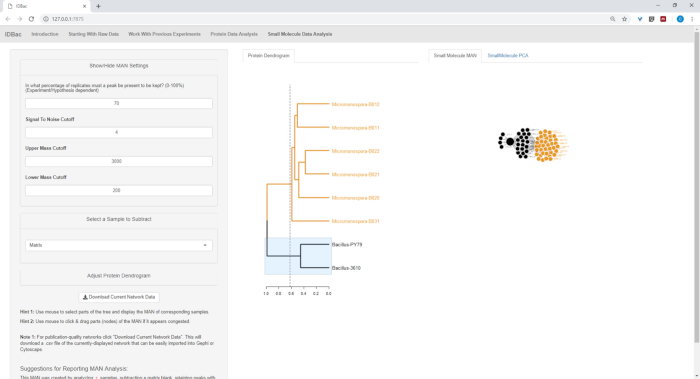
図18:タンパク質デンドログラムからバチルスsp.株を選択して作成されたMANは、特殊代謝産物の微分産生を示した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
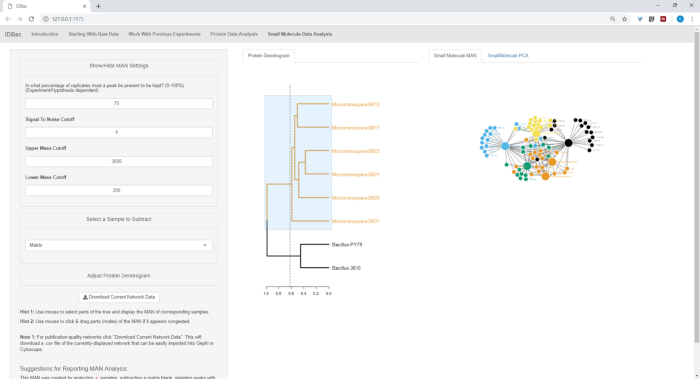
図19:タンパク質デンドログラムから6つのマイクロモノスポラsp.株を選択して作成されたMANは、特殊な代謝産物の微分産生を示した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
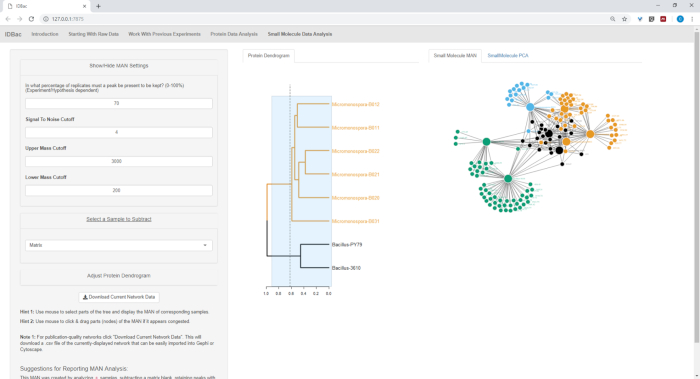
図20:特殊代謝産物の微分産生を示すバチルス属およびマイクロモノスポラ属株のMAN。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
IDBacプロトコルは、細菌タンパク質と特殊な代謝産物データの取得と分析を1人の研究者によって4時間で最大384の細菌単離物の詳細に説明します。IDBacを使用すると、細菌分離からDNAを抽出したり、液体発酵ブロスから特殊な代謝物抽出物を生成し、クロマトグラフィー法を使用してそれらを分析する必要はありません。代わりに、タンパク質と特殊な代謝産物データは、細菌コロニーからMALDIターゲットプレートに直接材料を広げることによって収集されます。これにより、16S rRNA遺伝子シーケンシングやLCMS 9などの代替技術に関連する時間とコストが大幅に削減されます。
MALDIプレートにマトリックスブランクとキャリブレーションスポットを追加することが重要であり、再現性と統計的な信頼性を確保するために適切な数の反復を使用することをお勧めします。反復数は実験に依存します。たとえば、ユーザーが何千ものコロニーを環境ダイバーシティプレートのコレクションから区別する場合、必要な複製数が少なくなる可能性があります(当研究室では、コロニーごとに 3 つの技術的な反復を収集します)。あるいは、ユーザーが特定の細菌タキサから株のカスタムデータベースを作成して未知の単離物の亜種分類を迅速に決定したい場合、より多くの複製が適切です(私たちの研究室は、1回あたり8つの生物学的反復を収集します)ひずみ)。
IDBacは、栄養分類情報と特殊な代謝産物生産に基づいて、関連性の高い細菌単離物を迅速に分離するためのツールです。これは、詳細な遺伝子解析、代謝物の産生と機能を含む研究、核磁気共鳴分光法による特殊な代謝物構造の特性解析などの直交法の前駆体として機能することができます。LC-MS/MS
特殊な代謝産物産生(IDBac MAN)は、細菌の増殖条件、特に異なる培地を使用して、この方法の潜在的な制限である可能性があります。しかし、IDBacは様々な成長条件下で特殊な代謝産物産生の違いを示すMANを容易に生成することができるので、これらの特性は、ユーザーによって利用される可能性があります。特殊な代謝産物の指紋は成長条件によって異なる可能性がありますが、タンパク質フィンガープリントはこれらの変数にわたって比較的安定していることを以前に示しました(Clark et al.6を参照)。環境ダイバーシティプレートを扱う場合は、近隣の細菌クロストークからの寄与を減らすために、分析前に細菌単離物を精製することをお勧めします。
最後に、タンパク質MSフィンガープリントの検索可能なパブリックデータベースの欠如は、未知の環境細菌を分類するためにこの方法を使用する上で大きな欠点です。これを念頭に置いてIDBacを作成し、コミュニティが受け入れるオープンソース形式(mzML)10、11、12へのデータの自動変換を含み、検索、共有、および作成を可能にするソフトウェアを設計しました。カスタム データベース。私たちは、大規模なパブリックデータベース(>10,000完全に特徴付けられた株)を作成中で、利用可能な場合はGenBankの加盟番号へのリンクを含む種レベルへの一部の分離物の分類を可能にします。
IDBac はオープン ソースであり、データ分析と視覚化のニーズをカスタマイズできるユーザーがコードを使用できます。我々は、ユーザーが彼らの実験目標をサポートし、設計するのを助けるために文献の広範なボディを参照することをお勧めします (Sauer et al.7, Silva et al.5)私たちは、ディスカッションのためのフォーラムを主催しています: https://groups.google.com/forum/#!forum/idbacとソフトウェアに関する問題を報告する手段: https://github.com/chasemc/IDBacApp/issues.
開示事項
著者は何も開示していない。
謝辞
この研究は、国立一般医学研究所グラントR01 GM125943、ナショナルジオグラフィックグラントCP-044R-17によってサポートされました。アイスランド研究基金助成金 152336-051;そして、シカゴのスタートアップファンドでイリノイ大学。また、以下の貢献者に感謝します: アマンダ・ブルマン博士は、MALDI-TOF MSタンパク質取得パラメータの支援を受けました。テリー・ムーア博士とアトゥル・ジャイン博士は、α-シアノ-4-ヒドロキシシナミン酸マトリックス(CHCA)を再結晶化した。
資料
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
参考文献
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved