Method Article
Mikrobiyal protein ve özel metabolit verilerinin analizi için açık kaynak MALDı TOF-MS ıdbac Pipeline kullanma
Bu Makalede
Özet
IDBac, bakteriyel kolonilerden kazınmış hücre malzemesinde toplanan, hem bozulmamış protein hem de özel metabolit spektrumlu verileri birleştiren açık kaynaklı Kütle Spektrometrisi bazlı Biyoinformatik boru hattıdır. Boru hattı araştırmacılar hızlı bir şekilde on binlerce bakteriyel koloniler Putatif taksonom gruplara düzenlemek için izin verir ve daha fazla özel metaboliti üretimi dayalı ayırt.
Özet
Bakteri Phylogeny ve besin agar üzerinde büyüyen bakteriyel kolonilerin özel metaboliti üretimi arasındaki ilişkiyi görselleştirmek için, biz IDBac geliştirilen-düşük maliyetli ve yüksek verim Matris Destekli Lazer Desorpsiyon/İyonizasyon Uçuş süresi kitle spektrometresi (MALDı-TOF MS) Biyoinformatik boru hattı. Idbac yazılımı olmayan uzmanlar için tasarlanmıştır, serbestçe kullanılabilir ve bakteriyel kolonilerin birkaç binlerce analiz yeteneğine sahiptir. Burada, MALDı-TOF MS analizi, MS enstrüman operasyonu ve ıdbac 'de veri işleme ve görselleştirme için bakteriyel kolonilerin hazırlanması için prosedürler sunuyoruz. Özellikle, biz nasıl dendrograms içine protein MS parmak izleri dayalı ve interaktif metabolit Derneği Networks (MANs) özel metabolit verilerden oluşturmak bakteri küme kullanıcılara talimat.
Giriş
Bakteriyel fonksiyon Etüdü araştırmacılar için büyük bir engel hızlı ve eşzamanlı bir mikroorganizma ve özel metabolitleri üretmek için kapasitesini taksononomik kimliğini değerlendirmek yeteneğidir. Bu, ortamdan izole edilen bakterilerin çoğunda bakteriyel Phylogeny ve özel metabolit üretimi arasındaki ilişkiyi anlamada önemli gelişmeler önledi. Protein parmak izlerini grup olarak tanımlamak ve bakterileri belirlemek için kullanılan MS tabanlı yöntemler1,2,3,4, bu çalışmalar genellikle küçük izolatlar gruplarında gerçekleştirilmesine karşın, bir türe özgü şekilde. Önemlisi, özel metabolit üretimi hakkında bilgi, çevresel mikrobiyal fonksiyonun büyük bir sürücü, bu çalışmalarda dahil edilmemiş kaldı. Silva ve ark.5 son zamanlarda özel metabolitleri ve yazılım eksikliği mevcut Biyoinformatik darboğazları rahatlatmak için analiz etmek MALDI-TOF MS altkullanımı ayrıntılarıyla kapsamlı bir tarih sağladı. Bu eksiklikleri gidermek amacıyla, MALDı-TOF MS6' nın hem doğrusal hem de reflectron modlarını birleştiren bir Biyoinformatik boru hattı olan ıdbac 'i oluşturduk. Bu kullanıcıların hızla görselleştirmek ve bakteri izolatlar hem protein ve özel metaboliti MS parmak izleri, sırasıyla dayalı ayırt sağlar.
Idbac uygun maliyetli, yüksek verimlilik ve Kullanıcı yatıyordu için tasarlanmıştır. Serbestçe kullanılabilir (chasemc.github.io/IDBac), ve sadece bir MALDı-TOF Kütle spektrometresi erişim gerektirir (reflectron modu özel metabolit analizi için gerekli olacaktır). Numune hazırlama basit "genişletilmiş doğrudan transfer" yöntemine dayanır7,8 ve veri tek bir Maldi-hedef noktada ardışık doğrusal ve reflectron alımları ile toplanır. Idbac ile, örnek hazırlama, veri edinme ve veri görselleştirme dahil olmak üzere dört saat içinde kolonilerin yüzlerce sözde Phylogeny ve özel metaboliti üretimini analiz etmek mümkündür. Bu, bakteri tanımlama (gen sıralaması gibi) ve metabolik çıktıyı analiz etme (sıvı kromatografi-kütle spektrometresi [LCMS] ve benzer kromatografik yöntemler) ile ilgili geleneksel yöntemlerle önemli bir zaman ve maliyet avantajı sunar.
Doğrusal mod analizinde elde edilen verileri kullanarak, ıdbac protein spektrumunun relatedness temsil etmek için hiyerarşik kümeleme kullanır. Spektrum çoğunlukla iyonize ribozomal proteinleri temsil ettiğinden, bir örnekteki fitogenetik çeşitliliğin bir gösterimini sağlar. Buna ek olarak, idbac metaboliti Derneği Networks (MANs) olarak özel metabolit parmak izleri görüntülemek için reflectron modu verileri içerir. MANs, bakteriyel izolatlar arasında ortak ve benzersiz metabolit üretiminin kolay görselleştirilmesini sağlayan iki taraflı ağlardan biridir. IDBac platformu, araştırmacılar hem protein hem de özel metabolit veri tandem aynı zamanda tek bir veri türü elde edilir, aynı şekilde analiz etmek için izin verir. Önemlisi, ıdbac Bruker ve Xiamen enstrümanların yanı sıra txt, sekme, CSV, mzXML ve mzML ham verileri işler. Bu, el ile dönüştürme ve veri kümeleri biçimlendirme gereksinimini ortadan kaldırır ve önemli ölçüde Kullanıcı hatası veya MS veri yanlış işleme riskini azaltır.
Protokol
1. MALDı matrisinin hazırlanması
- MS dereceli çözücülerde 10 mg/mL MALDı-Grade ve/veya rerystalize α-Cyano-4-hydroxycinnamic Acid (CHCA) hazırlayın: 50% Asetonitril (ACN), 47,5% su (H2O), 2,5% trifluoasetik ASIT (TFA). Örnek: 100 μL çözeltisi = 50 μL ACN + 47,5 μL H2O + 2,5 ΜL TFA + 1 mg chca
- MALDı plaka spot ve Vortex başına en az 1 μL matris çözeltisi hazırlayın veya çözüme kadar sonikat (yaklaşık 5 dk sonication veya görünür katılar).
DIKKAT: TFA, cilt, gözler ve hava yollarının temas veya inhalasyon ile zarar görmesini sağlayan, uygun kişisel koruyucu ekipman takarken kimyasal duman kaputu içinde ele alınması gereken güçlü bir asittir.
Not: CHCA higroskopik ve ışığa duyarlıdır ve bir kurutucu içinde Amber şişeleri içinde depolanmalıdır. Birçok MALDı matris seçeneği mevcuttur. CHCA bakterilerin protein profil için en yaygındır, aynı zamanda özel metabolit analizi için çalışır. Matris seçimi, bireysel kullanıcı/deneme gereksinimlerine bağlıdır.
- MALDı plaka spot ve Vortex başına en az 1 μL matris çözeltisi hazırlayın veya çözüme kadar sonikat (yaklaşık 5 dk sonication veya görünür katılar).
2. MALDı hedef plakalarının hazırlanması
Not: daha fazla ayrıntı için Sauer ve al.7' ye bakın.
- MALDı plakasını metanol (HPLC-Grade veya üzeri) ile durulayın ve yumuşak kağıt mendilleri ile kurutun. Bu hedef plakanın yüzeyine kalıcı olarak zarar verebilecek şekilde, hedef plakaları temizlerken aşındırıcı fırçalar kullanmayın.
- Protein ve özel metabolit kalibreli lekeler atayın. Zaman içinde MALDI-Plate-düzensizlikler ve enstrüman kayması için hesap, örnek nüfus arasında eşit kalibrasyon noktalarını düzenleyin. Çalışma için uygun sayıda ortam/matris boş nokta atayın; Bu noktalar yalnızca medya ve matris veya yalnızca matris içerecektir.
- Steril bir kürdan kullanarak, bir bakteriyel koloninin küçük bir kısmını MALDı plakasında uygun noktaya aktarın. Bakteri kolonisini yere eşit olarak yayın. Spot mümkün olduğunca düz görünmelidir.
Not: daha fazla mukoid/amorf olan bakteriyel kolonileri yassılaştırmak daha kolay olacaktır. Daha sert/katı koloniler için, MALDı spot üzerinde hücre kütlesi görünür kümeleri bırakarak kaçının (Şekil 1).
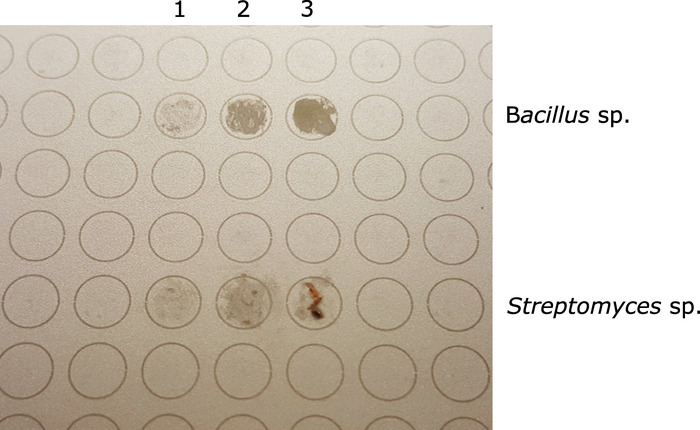
Şekil 1: formülik asit ve MALDı matrisini (ilk 3 nokta- Bacillus Sp.; alt 3 Spotlar- Streptomyces Sp.) eklemeden önce ıkı farklı yalıtılmış gösteren Maldi-Target plakası . Her ikisi için de sütun 3 aşırı örnek temsil eder; sütun 2 örnek uygun miktarını temsil eder; sütun 1 maldı çözümlemesi için yetersiz örneği temsil eder. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Az miktarda agar/medyayı MALDı plakasında uygun noktaya (ler) aktarmak için steril bir kürdan kullanarak bir matris/medya kontrolü hazırlayın.
- Matris kontrol noktaları da dahil olmak üzere her örnek noktaya 1 μL 70% Kütle Spektrometrisi sınıf formik asit bindirme. Asitlere kimyasal duman kaputu içinde tamamen kurumasına izin verin (yaklaşık 5 dakika).
DIKKAT: formik asit, kostik bir kimyasaldır ve kimyasal duman davlumbazında ele alınması gerekir. Eğer solunabilir hava yolları zarar verebilir. - Her örnek noktanın yanı sıra matris/medya kontrol noktalarına hazırlanan MALDı matris çözümünü 1 μL ekleyin. Matris çözümünün tamamen kurumasına izin verin (yaklaşık 5 dakika).
Not: bir MALDı-TOF Kütle spektrometresi üzerinde analiz edilebilir kadar, karanlıkta, bir kurutucu plaka saklamak mümkündür. İzin verilen depolama süreleri örnek kararlılığa bağlı olarak farklılık gösterebilir. - Atanan kalibrasyon noktaların ardından 1 μL MALDı matris çözeltisi ile 0.5 – 1.0 μL kalibre ekleyin. Elde edilen solüsyonu karıştırmak için yukarı ve aşağı pipet. MALDı-TOF kütle spektrometresine giriş yapmadan önce tüm noktalardan tamamen kurumaya izin verin.
Not: protein ve özel metabolit kalibraları, her iki bozulma duyarlı olduğu gibi, MALDı Analizi 30 dk içinde eklenmesi gerekir.
3. veri edinme
Not: veri edinme için genel parametreler Tablo 1' de listelenir.
| Parametre | Protein | Uzmanlaşmış metaboliti |
| Kütle başlangıcı (da) | 1920 | 60 |
| Kütle sonu (da) | 21000 | 2700 |
| Kütle sapması (da) | 1900 | 50 |
| Çekim | 500 | 1000 |
| Frekans (Hz) | 2000 | 2000 |
| Lazer boyutu | Büyük | Orta |
| MaxStdDev (ppm) | 300 | 30 |
Tablo 1.
- Kullanılan enstrümana özgü protokollerin ardından hem protein hem de özel metabolit kalibrasyonları ayarlayın.
- Spektrumları alırken kullanılacak optimum lazer gücünü ve dedektör kazanımı belirlemek için birkaç ayrı hedef spot testi yapın (Bu, günlük ve enstrüman ile değişir).
Not: şekil 2A ve şekil 3A optimum spektrumları gösterirken Şekil 2D ve şekil 3D düşük kaliteli spektrumların örnekleridir.
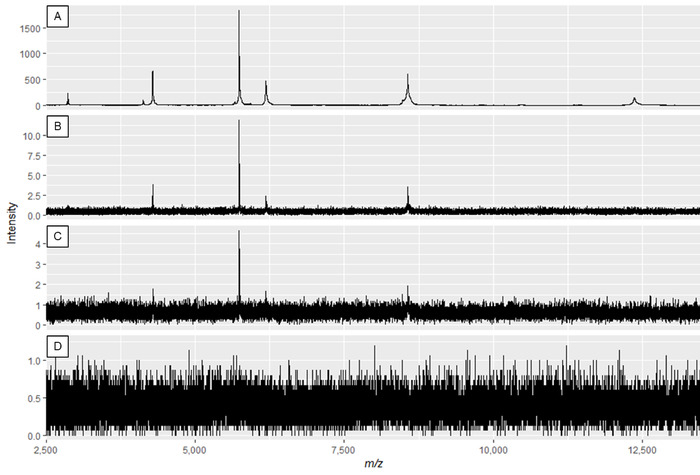
Şekil 2: Lazer güç ve dedektör kazanç değiştirme etkisini gösteren örnek protein spektrumları. Spectra kalite panel Aen iyisidir, ve paneller C ve Dyetersiz spektrum kalitesine kadar azalır. Panel B 'de spektrumunda kullanışlı doruklara neden olabilir, panel A en iyi verileri görüntüler. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
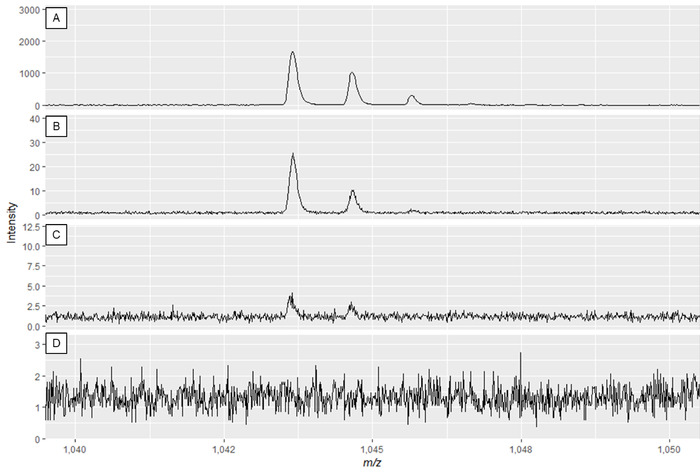
Şekil 3: Örnek özel metabolit Spectra lazer güç ve dedektör kazanç değiştirme etkisini gösteren. Spectra kalite panelin en iyisidir A ve paneller C ve Dyetersiz spektrum kalitesi kadar azalır. Panel B 'de spektrumunda kullanışlı doruklara neden olabilir, panel A en iyi verileri görüntüler. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Spektrum kazanın, bir klasöre protein spektrumları tasarrufu ve ikinci, ayrı bir klasöre özel metaboliti Spectra.
4. MALDı hedef plakasını Temizleme (Sauer ve al.7' den uyarlanmış)
- MALDı hedef plakasını kendi tutucusuna çıkarın ve aseton ile durulayın.
- İzleme proteinlerini ve lipidlerini ve yumuşak kağıt mendilleri/yumuşak kıl diş fırçasını kaldırmak için aşındırıcı olmayan bir sıvı sabun ile yıkayın.
- Sabun tamamen kaldırmak için yaklaşık 2 dakika için de-iyonize su ile durulayın.
- Hedef plakayı su içinde (HPLC Grade veya üzeri) 5 dakika boyunca sonikat.
- Hedef plakayı su ile durulayın (HPLC sınıfı veya üzeri).
- Hedef plakayı metanol (HPLC sınıfı veya üzeri) ile durulayın.
5. ıdbac yazılımını yükleme
- Idbac yazılımını indirin.
Not: kalıcı, sürümlü yedeklemeler de indirilebilir (bkz. malzeme tablosu). - Yükleyici başlatmak için indirilen "Install_IDBac. exe" çift tıklatın ve ekrandaki yönergeleri izleyin.
6. RAW verilerle başlayarak
Not: her veri işleme adımının ayrıntılı açıklamaları ve talimatları ıdbac içinde gömülür, ancak ana analizler ve interaktif girişler aşağıda açıklanmıştır.
- Idbac başlatmak için ıdbac masaüstü kısayolunu çift tıklatın. Idbac varsayılan olarak giriş sekmesinde açılacaktır.
- Idbac 'in en güncel sürümünün kullanıldığından emin olmak için Güncelleştirmeleri denetle düğmesini kullanın (internet erişimi gerektirir). Daha yeni bir sürüm varsa, ıdbac otomatik olarak karşıdan yükleyip yükledikten sonra ıdbac yeniden başlatılması isteğinde olacaktır.
- Ham veri sekmesi ile başlayarak tıklatın ve menüden ıdbac ile kullanılacak veri türünü seçin; Uygulama içi yönergeleri izleyerek devam edin.
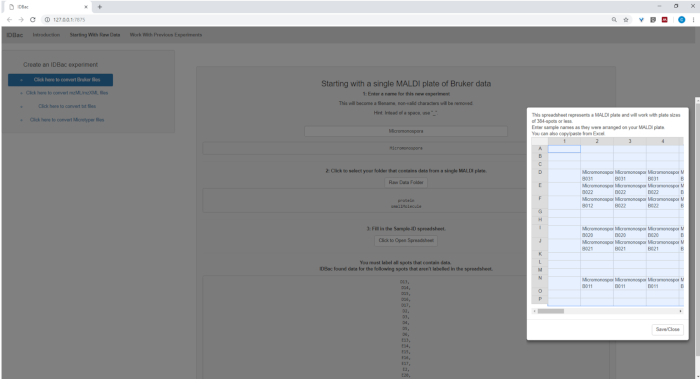
- Veri dosyalarının dönüşümünü ve işlenmesini ayarlarken, istendiğinde deneme için açıklayıcı bir ad girin (bkz. Şekil 4). Denemeler daha sonra alfabetik olarak görüntülenecektir, böylece yararlı bir strateji, deneme adlarını grup özniteliğiyle başlatacaktır (örn., "Bacillus-trials_experiment-1"; "Bacillus-trials_experiment-2").
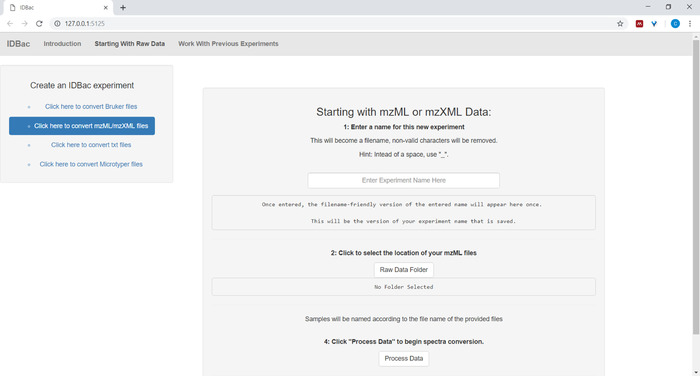
Şekil 4: IDBac veri dönüştürme ve ön işlem adım. Idbac açık mzML formatında ham Spectra dönüştürür ve her deneme için bir veritabanında mzML, tepe listeleri ve örnek bilgileri depolar. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
7. önceki deneylerle çalışın
- Dosyaları dönüştürdükten ve onları IDBac ile işlerken veya bir deneyle yeniden analiz etmek istediğinde, önceki deneyler sayfasıyla çalışmaya gidin ve çalışmak Için bir deneme seçin (Şekil 5).
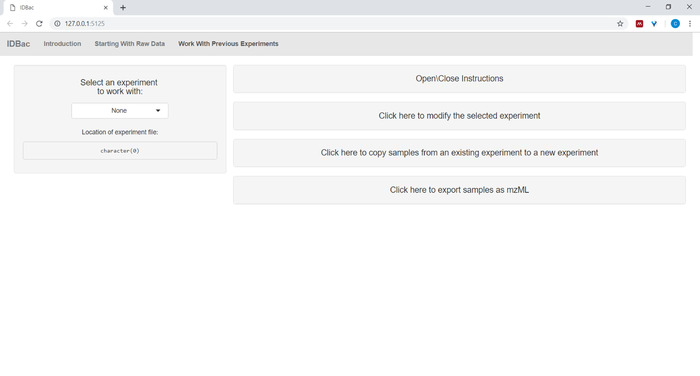
Şekil 5: "önceki deneylerle çalışma" sayfası. Çözümlemek veya değiştirmek için bir deneme seçmek için ıdbac 's "önceki denemeleri ile çalışma" sayfasını kullanın. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Isteğe bağlı Menü kullanarak örnekler hakkında bilgi ekleme Seçili deneyi değiştirmek için burayı tıklatın. Otomatik doldurulan elektronik tabloya bilgi girin ve Kaydet tuşuna basın (Şekil 6). Bu seçenek, kullanıcının analizler sırasında renk kodu verilerine izin verir.
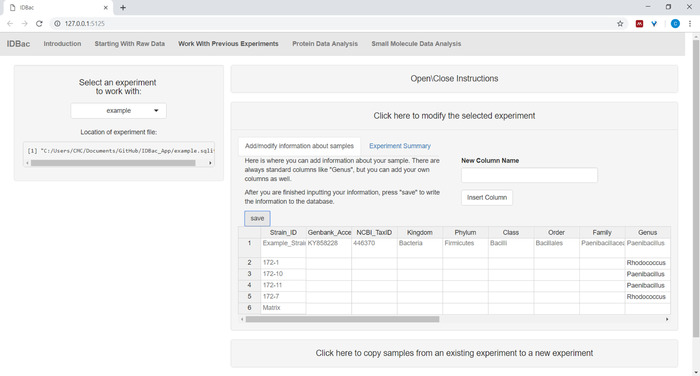
Şekil 6: örnek bilgileri girin. "Önceki deneylerle çalışma" sayfasında kullanıcılar, taksononomik kimlik, toplama konumu, yalıtım koşulları vb. gibi örnekler hakkında bilgi girebilmek Için lütfen buraya tıklayarak bu figürün daha büyük bir versiyonunu görüntüleyebilirsiniz .
- Isteğe bağlı Önceki deneylerden gelen örnekleri yeni/diğer deneylere aktar 'ı tıklatarak ve sağlanan yönergeleri Izleyerek (Şekil 7), tüm veya numunelerin bir alt kümesini yeni veya başka bir denemeye aktarın.
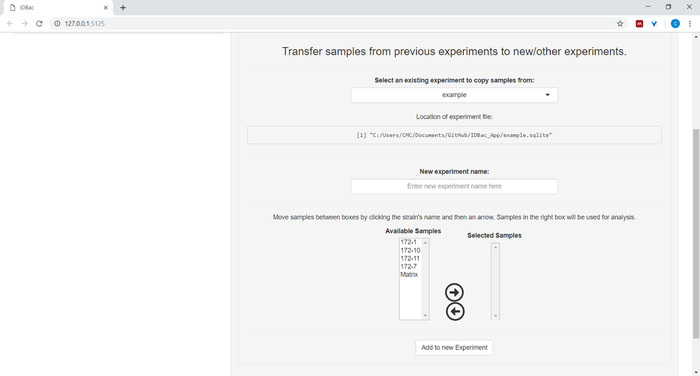
Şekil 7: verileri aktarın. "Önceki denemeleri ile çalışma" sayfası, varolan denemeler ve yeni deneyler arasında veri aktarımı seçeneği içerir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Analize başlamaya hazır olduğunda, çalışacağı denemenin seçildiğinden emin olun. Protein veri analizi veya küçük molekül veri analizi'ni seçin.
8. protein veri analizi kurma ve ayna çizimleri oluşturma
- Protein verilerini analiz ederseniz, önce protein veri analizi sayfasına gidin. Üst düzey çekme ayarlarını seçin ve görüntülenen ayna çizimleri aracılığıyla numunelerin protein spektrumları değerlendir (Şekil 8).
Not: ayna çizimlerinde, kırmızı bir tepe sadece üst spektrumunda o zirve varlığını belirtir, mavi zirveler her iki spektrum içinde meydana gelen temsil ederken.
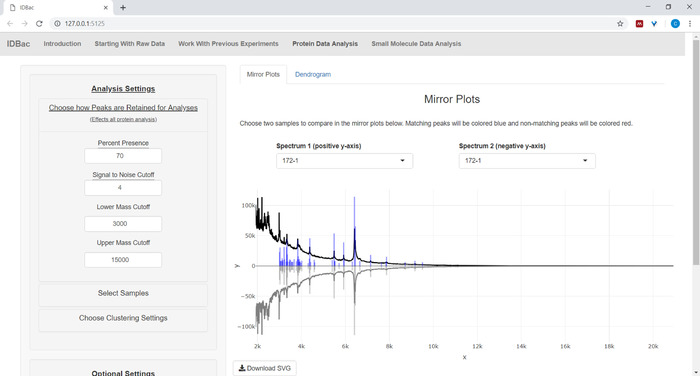
Şekil 8: doruklarına analiz için nasıl korunur seçin. Analiz etmek için bir deneme seçtikten sonra, "protein veri analizi" sayfasını ziyaret edin ve daha sonra "doruklarda analiz için nasıl tutulur seçin" menüsünü açarak kullanıcıların zirveleri korumak için sinyal-gürültü oranı gibi ayarları seçmesine izin verir. Görüntülenen yansıtma grafiği (veya dendrogram) otomatik olarak seçilen ayarları yansıtacak şekilde güncellenir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Analizlere dahil edilmesi için bir zirvede mevcut olması gereken çoğaltır yüzdesini ayarlayın (örneğin, eşik% 70 olarak ayarlanmışsa ve en az 7 ' si 10 çoğaltır içinde bir tepe meydana gelirse, dahil edilecektir).
- Görsel rehberlik olarak ayna çizimleri kullanarak, daha fazla çoğaltır ve daha yüksek bir "yüzde zirve varlığı" değeri gürültü kesme için daha düşük bir sinyal seçimi izin verir belirterek, en "hakiki" doruklarına ve az gürültü koruyan gürültü kesme sinyali ayarlayın.
- Idbac tarafından daha fazla analizlerde kullanılacak her spektrumdaki kütle değerlerinin aralığını dikte ederek, alt ve üst m/z cutoffs belirtin.
9. protein verilerini kullanarak kümeleme örnekleri
- İçinde protein veri analizi sayfa, seçin dendrogram sekmesi. Bu örnekleri bir dendrogram Kullanıcı tarafından seçilen uzaklık önlemleri ve kümeleme algoritmaları göre gruplandırma sağlar.
- Menüde örnekler Seç 'e tıklayın ve analizlere dahil edilen örnekleri seçmek için yönergeleri izleyin. Yalnızca protein spektrumları içeren örnekler mevcut numune kutusu içinde görüntülenir (Şekil 9).
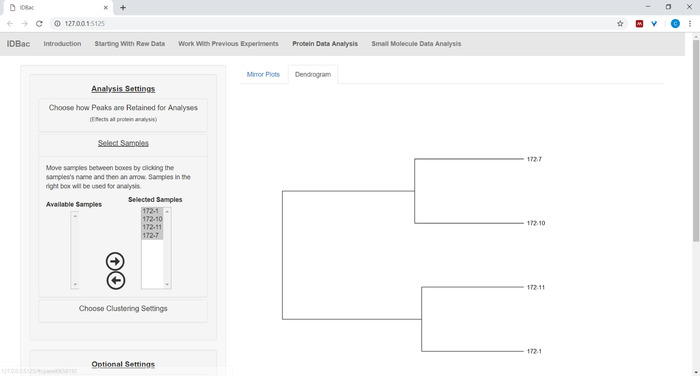
Şekil 9: görüntülenen dendrogram içinde dahil etmek için seçilen deneyden örnekleri seçin. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Varsayılan değerleri kullanın veya altında kümeleme ayarları seçin, istenen uzaklık ve kümeleme algoritmaları dendrogram nesil uygulanacak seçin.
- Giriş olarak varlığı/devamsızlık seçeneğini belirleyin. Alternatif olarak, numunelerin tepe yükseklikleri hakkında emin iseniz (örn., en yüksek yoğunluğun değişkenliğini değerlendirmek için bir çalışma yaptıktan sonra), giriş olarak yoğunluklar 'ı seçin.
Not: yayın sırasında ıdbac uygun kombinasyonları seçmek için kullanıcılara güveniyor, kümeleme için ayarları esneklik sağlar. Bu seçenekler ile aşina değilseniz, ya da eşleştirmek için önerilen A: "kosinüs" uzaklığı ve "ortalama (UPGMA)" kümeleme; veya B: "Euclidean" uzaklık ve "Ward. D2" kümeleme. - Dendrogram üzerindeki Bootstrap değerlerini görüntülemek için, bootasklarıaltında 2 ile 1000 arasında bir sayı girin.
- Sonuçları raporlarken, Raporlama protein analizi paragrafı Için öneriler içindeki metni kopyalayın. Bu, belirli dendrogram oluşturulan kullanıcı tanımlı ayarları sağlar.
10. protein dendrogram özelleştirme
- Dendrogram 'ı özelleştirmeye başlamak için dendrogram menüsünü ayarlayın (Şekil 10).
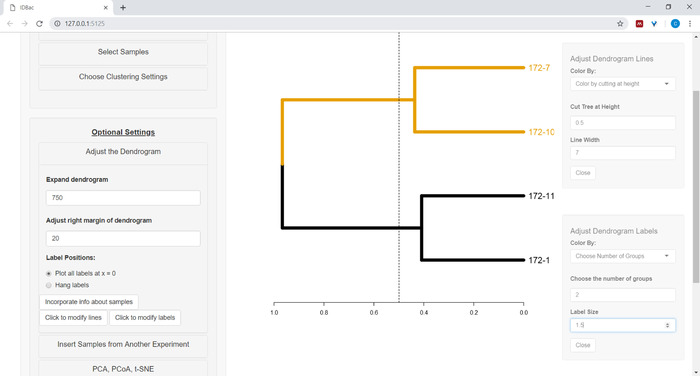
Şekil 10: Dendrogram ayarlayın. IDBac dendrogram nasıl göründüğünü değiştirmek için birkaç seçenek sağlar, bu menü içinde bulunabilir "Dendrogram ayarlayın". Bu, k-araçlarla dalları ve etiketleri renklendirme veya bir kullanıcı tarafından sağlanan yükseklikte dendrogram "kesme" içerir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- Dendrogram 'ın çizgilerini ve/veya etiketlerini renklendirmek için uygun düğmeyi seçin: satırları değiştirmek veya etiketleri değiştirmek için tıklayın ve istediğiniz seçenekleri belirleyin.
- Dendrogram yanındaki elektronik tabloya bilgi çizmek için (bkz: adım 7,2), düğme örnekleri hakkında bilgi birleştirmekseçin. Bu, girilen değerlere göre bir kategorinin (elektronik tablodaki sütunun) kendi kendine dolduracağı bir panel açacaktır (Şekil 11).
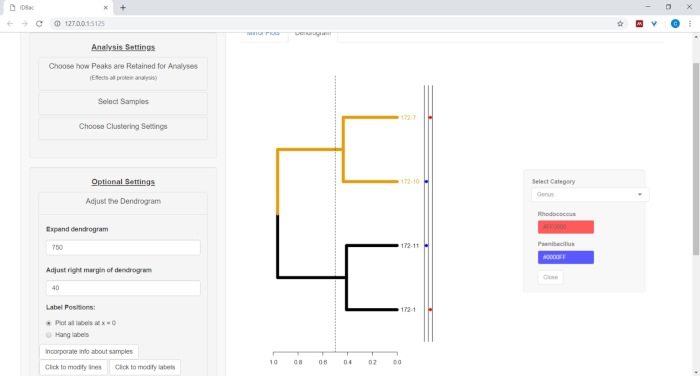
Şekil 11: örnekler hakkında bilgi ekleyin. "Dendrogram ayarla" menüsü içinde "örnekleri hakkında bilgi birleştirmek" seçeneğidir. Bu seçme, dendrogram yanında örnekleri hakkında komplo bilgileri izin verecektir. Örnek bilgiler "önceki denemeleri ile çalışma" sayfasında girdi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
11. ayrı bir deney dendrogram içine örnekleri ekleyin
- Başka bir deneyden örnek eklemek için, başka bir deneyden örnekler eklemekiçin menü düğmesini seçin. Yeni açılan paneldeki yönergeleri izleyin (Şekil 12).
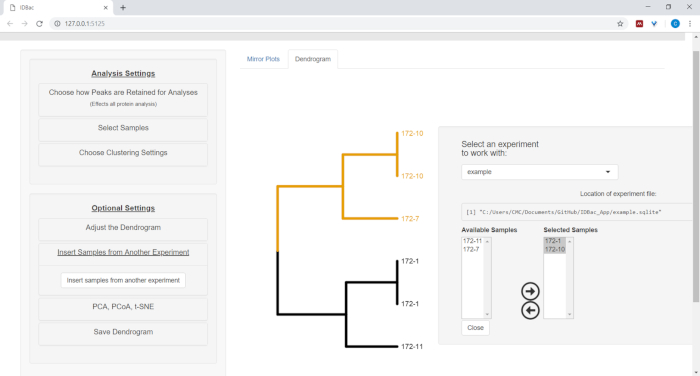
Şekil 12: başka bir deney menüsünden örnekler ekleyin. Bazen başka bir denemenin örneklerini karşılaştırmak yararlıdır. Şu anda görüntülenen dendrogram içinde dahil etmek için örnekleri seçmek için "başka bir deneyden örnekler Ekle" menüsünü kullanın. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
12. özel metabolit veri ve metabolit dernek ağları (MANs) analiz
- Metabolit dernek ağı (küçük-molekül) sayfasına geçin. Bu sayfa, küçük molekül m/z değerlerinin örneklerle korelasyonunu görüntülemek için iki taraflı ağlar kullanan ilke bileşenleri analizi (PCA) ve MANs tarafından veri görselleştirme sağlar.
- Eğer bir protein dendrogram (Bölüm 9) oluşturuldu, bu da bu sayfada görüntülenir. Tıklayın ve-sürükle dendrogram üzerinde analiz edilecek ilgi seçme örneklerini vurgulamak için. Hiçbir numune vurgulanmışsa veya hiçbir protein dendrogram oluşturulmuşsa, rasgele bir alt veya tüm numunelerin bir MAN 'ı saygıyla görünür (Şekil 13).
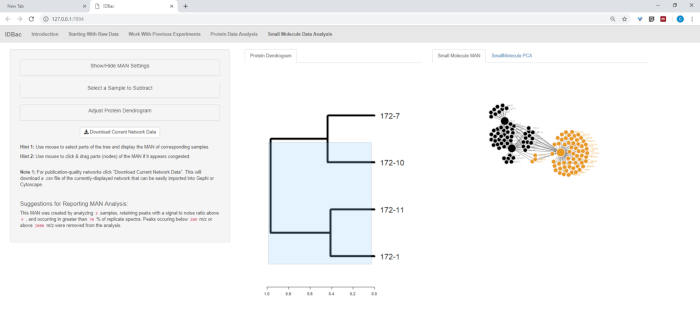
Şekil 13: küçük molekül veri analizi "sayfası. Eğer bir dendrogram protein spektrumunda oluşturulmuşsa, "küçük molekül veri analizi" sayfası içinde görünecektir. Bu sayfa ayrıca küçük molekül verileri için metabolit Ilişkilendirir ağları (MANs) ve Ilke bileşenleri analizi (PCA) görüntüler. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
- MAN 'de boş bir matris/medyayı çıkarmak için, menüyü açın, çıkarmak için bir örnek seçin ve boş olarak kullanmak üzere uygun örneği seçin.
- Bölüm 9 ' da protein spektrumları için yapıldığı gibi, çoğaltır, gürültü sinyali ve üst ve alt kitle kesitlerinde zirve varlığının yüzdesi için istenilen değerleri seçmek üzere Man ayarlarını göster/gizle menüsünü açın. Bu ayarların seçimini yönlendirmek için küçük molekül aynası grafiklerini kullanın.
- Şu anda görüntülenen MAN verilerini kaydetmek için "geçerli ağ verilerini Indir" seçeneğini belirleyin. Bu veriler, ıdbac dışındaki ağ çözümleme yazılımında kullanılabilir.
- Raporlama sonuçları için, Man Analizi paragrafını bildirdiği Için öneriler içindeki metni kopyalayın. Bu, oluşturulan MAN oluşturmak için kullanılan Kullanıcı tanımlı ayarları sağlar.
13. veri paylaşma
- Her ıdbac "deneme" tek bir SQLite veritabanı olarak kaydedilir. Dönüştürülmüş mzML ham spektrumları, algılanan zirveleri ve örneklerle ilgili tüm Kullanıcı giriş bilgilerini içerir. Bu nedenle, bir ıdbac deneyi paylaşmak için yalnızca denemede aynı ada sahip SQlite dosyasını paylaşabilirsiniz (dosya konumu önceki denemeleri Ile çalışma sayfasında görüntülenir).
Sonuçlar
Biz Micromonospora chokoriensis altı suşları ve Bacillus subtilis, daha önce6, veri DOI: 10.5281/zenodo. 2574096 kullanılabilir kullanarak karakterize iki suzlar analiz etti. Ham veri sekmesi Ile başlayarak talimatların ardından, Bruker dosyalarını dönüştürmek için burayı tıklatın seçeneğini seçtiniz ve her veri kümesi için ıdbac tarafından sağlanan yönergeleri izledi (Şekil 14).
Otomatik dönüşüm ve ön işlem/Peak-peaking adımları tamamlandıktan sonra, iki denemeden gelen örnekleri hem Bacillus hem de içeren tek bir denemede aktararak yeni bir kombine idbac deneyi oluşturmaya devam ettik Micromonosopora örnekleri (Şekil 15). Sonuç analizi, spektrum kalitesini değerlendirmek ve tepe çekme ayarlarını ayarlamak için yararlı olan Şekil 16' da resimde gösterildiği gibi, ayna grafiklerini kullanarak protein Spektrumları karşılaştırarak yer alıyor. Şekil 17 seçilen varsayılan ayarlarla protein kümeleme sonuçlarının bir ekran görüntüsünü görüntüler. Dendrogram arsa üzerinde eşik ayarlayarak renklidir (noktalı bir çizgi olarak görünür). Not, her iki M. chokoriensis ve B. subtilis ayrı olarak kümeleme yalıtır ile, Genera arasındaki net ayrım.
Şekil 18, Şekil 19ve Şekil 20 , protein dendrogram arasında tıklayarak ve sürükleyerek Kullanıcı tarafından seçilen bölgelerin MANs oluşturmak için yeteneğini vurgulayın. Bu biz hızlı bir şekilde sadece B. subtilis suşları karşılaştırmak için MANs oluşturmak başardık (Şekil 18), sadece M. chokoriensis suşları (Şekil 19), ve aynı anda tüm suşları (Şekil 20). Bu ağlar birincil işlevi, bakteriler arasında özel metabolit çakışmasını derecesi geniş bir genel bakış ile araştırmacılar sağlamaktır. Bu veriler el ile, araştırmacılar şimdi bir bakteriyel koloniden kazınmış malzeme sadece küçük bir miktar bilinçli kararlar almak için kapasitesine sahip.
Şekil 14: Spectra işleme. İndirilen Bruker autoFlex Spectra ıdbac kullanılarak dönüştürülmüş ve işlenir. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
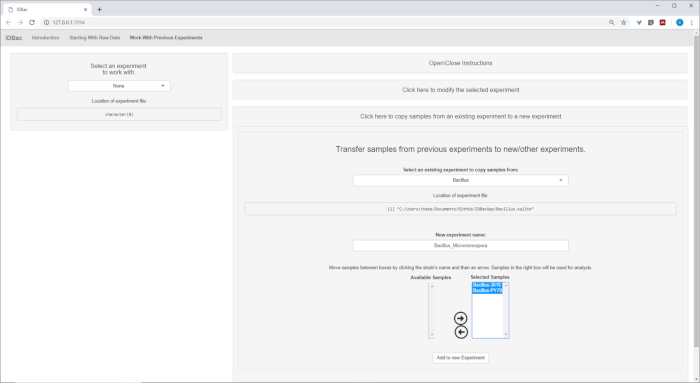
Şekil 15: kombine ıdbac deneyi. Micromonospora ve Bacillus Spectra farklı Maldi hedef plakaları üzerinde toplanan çünkü, iki deney daha sonra tek bir deney Içine birleştirilir-"Bacillus_Micromonsopora". Bu "önceki deneylerle çalışma" sekmesinde, "önceki deneylerden örnekleri yeni/diğer deneylere aktar" menüsündeki yönergeleri izleyerek yapıldı. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
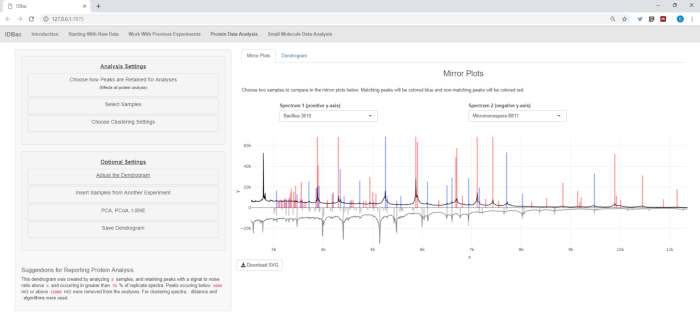
Şekil 16: karşılaştırma. Micromonspora ve Bacillus Spectra, "protein veri analizi" sayfasındaki ayna grafiklerini kullanarak karşılaştırıldı. Sonuçta, varsayılan tepe ayarları seçildi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
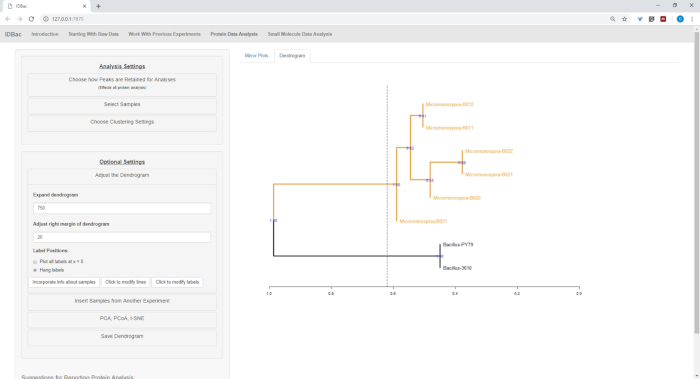
Şekil 17: hiyerarşik kümeleme. Hiyerarşik kümeleme, varsayılan ayarları kullanarak, doğru gruplandırılmış Bacillus ve Micromonospora yalıtır. Dendrogram (bir kesikli çizgi olarak görüntülenir) bir keyfi yükseklikte dendrogram "kesme" ile renkli ve 100 dallanma güven göstermek için kullanılan Boot,. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
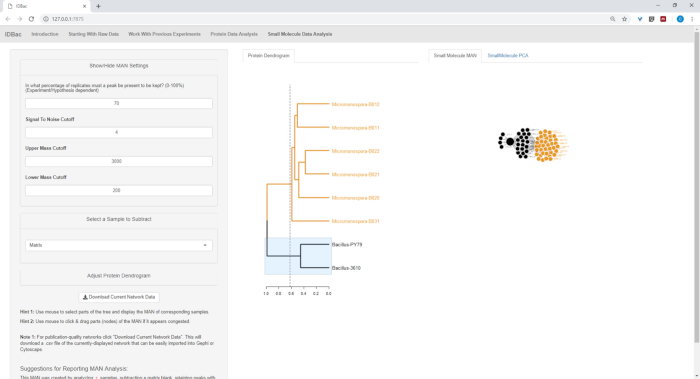
Şekil 18: Man protein dendrogram gelen Bacillus Sp. suşları seçerek oluşturulan özel metabolitlerin diferansiyel üretimi gösterdi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
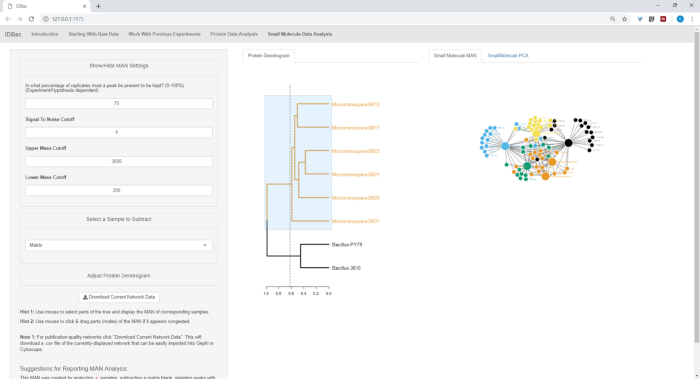
Şekil 19: Man protein dendrogram altı Micromonospora Sp. suşları seçerek oluşturulan özel metabolitlerin diferansiyel üretimi gösterdi. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
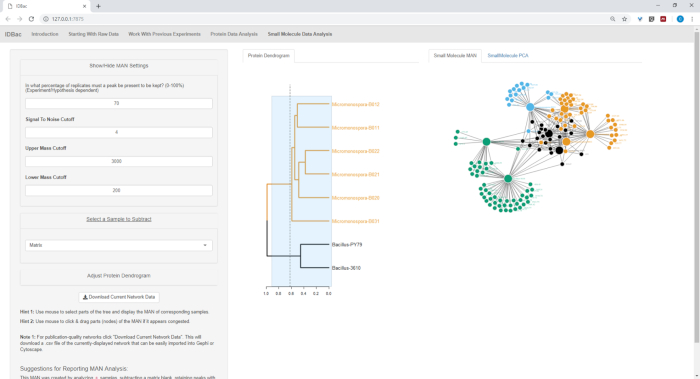
Şekil 20: Bacillus Sp. ve micromonospora Sp. suşların özel metabolitlerin diferansiyel üretimini gösteren Man . Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.
Tartışmalar
Idbac protokol detayları bakteriyel protein ve özel metabolit veri edinme ve analiz kadar 384 bakteriyel izolatlar 4 h tek bir araştırmacı tarafından. Idbac ile bakteriyel izolatlar DNA ayıklamak veya sıvı fermantasyon et suyu özel metaboliti özler oluşturmak ve kromatografik yöntemleri kullanarak analiz etmek için gerek yoktur. Bunun yerine, protein ve özel metabolit verileri sadece bir MALDı hedef plaka üzerine doğrudan bakteriyel kolonilerden malzeme yayma tarafından toplanır. Bu, 16S rRNA gen sıralaması ve LCMS9gibi alternatif tekniklerle ilişkili zaman ve maliyeti büyük ölçüde azaltır.
MALDı plakasına bir matris boş ve kalibrasyon noktaları eklemek önemlidir ve yeniden üretilebilirliği ve istatistiksel güveni sağlamak için uygun sayıda çoğaltır kullanmanızı öneririz. Çoğaltır sayıları deneme bağımlı olacaktır. Örneğin, bir Kullanıcı çevresel çeşitlilik plakaları koleksiyonundan binlerce kolonileri ayırt etmek niyetinde ise, daha az çoğaltır gerekli olabilir (laboratuvarımız koloni başına üç teknik çoğaltır toplar). Alternatif olarak, bir Kullanıcı belirli bakteriyel takson 'dan gelen suşların özel bir veritabanı oluşturmak istiyorsa, bilinmeyen izolatların alt türler sınıflandırmalarını hızla belirlemek için daha fazla çoğaltır uygundur (laboratuvarımız her bir sekiz biyolojik çoğaltır toplar strain).
IDBac hızlı bir şekilde son derece ilgili bakteriyel izolatlar Putatif taksonom bilgi ve özel metabolit üretimi dayalı ayırt etmek için bir araçtır. Derinlemesine genetik analizler, metabolit üretimi ve fonksiyonuyla ilgili çalışmalar veya nükleer manyetik rezonans spektroskopisi ve/veya özel metabolit yapısının karakterize edilmesi gibi ortogonal yöntemlerle öncü olarak hizmet verebilir LC-MS/MS.
Özel metabolit üretimi (ıdbac MANs), özellikle yöntemin potansiyel bir sınırlama olan farklı medya kullanarak, bakteriyel büyüme koşullarına son derece duyarlıdır. Ancak bu özellikleri Kullanıcı tarafından istismar edilebilir, ıdbac kolayca büyüme koşulları çeşitli altında özel metaboliti üretiminde farklılıkları gösteren MANs üretebilir gibi. Özel metaboliti parmak izi büyüme durumuna göre değişebilir iken, daha önce protein parmak izleri bu değişkenler arasında nispeten kararlı kaldığını göstermiştir dikkat etmek önemlidir (bkz. Clark ve al.6). Çevresel çeşitlilik plakaları ile uğraşırken, komşu bakteriyel çapraz konuşma olası katkıları azaltmak için analiz öncesinde bakteriyel izolatlar arındırmak öneririz.
Son olarak, protein MS parmak izleri aranabilir bir genel veritabanı eksikliği bilinmeyen çevresel bakterileri sınıflandırmak için bu yöntemin kullanımı büyük bir eksiklik olduğunu. Biz bu akılda ıdbac oluşturuldu ve bir topluluk kabul açık kaynak formatı (mzml)10,11,12 içine veri otomatik dönüşüm dahil ve arama, paylaşım ve oluşturulması izin vermek için yazılım tasarlanmış özel veritabanları. Biz büyük bir kamu veritabanı oluşturma sürecinde (> 10000 tam olarak karakterize suşları), hangi türler için bazı izolatlar sınıflandırılması için izin verecektir düzeyi, GenBank üyelik numaraları kullanılabilir olduğunda bağlantılar da dahil olmak üzere.
Idbac açık kaynak ve kod herkes kendi veri analizi ve görselleştirme ihtiyaçlarını özelleştirmek için kullanılabilir. Kullanıcıların deneysel hedeflerini desteklemesine ve tasarlamalarına yardımcı olmak için geniş bir literatür gövdesine (Sauer vd.7, Silva ve al.5) danışmanızı öneririz. Biz tartışma için bir forum barındırıyoruz: https://groups.Google.com/forum/#!forum/idbac ve yazılım ile ilgili sorunları bildirmek için bir araç: https://github.com/chasemc/IDBacApp/issues.
Açıklamalar
Yazarların ifşa etmesi gereken hiçbir şey yok.
Teşekkürler
Bu çalışma ulusal genel Tıp Bilimleri Enstitüsü Grant R01 GM125943, National Geographic Grant CP-044R-17 tarafından destekleniyordu; İzlanda araştırma fonu Grant 152336-051; ve University of Illinois Chicago başlangıç fonları. Ayrıca, aşağıdaki katılımcılar teşekkür: Dr Amanda bulman MALDı-TOF MS protein edinme parametreleri ile yardım için; Dr. Terry Moore ve Dr. Atul Jain rerystallizing Alfa-Cyano-4-hidroxycinnamic asit matris (CHCA) için.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
Referanslar
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır