Method Article
Uso de la tubería de código abierto MALDI TOF-MS IDBac para el análisis de datos de proteínas microbianas y metabolitos especializados
En este artículo
Resumen
IDBac es una tubería bioinformática basada en espectrometría de masas de código abierto que integra datos de espectros de proteínas intactos y metabolitos especializados, recogidos en material celular raspado de colonias bacterianas. La tubería permite a los investigadores organizar rápidamente cientos a miles de colonias bacterianas en grupos taxonómicos putativos, y diferenciarlos aún más en función de la producción especializada de metabolitos.
Resumen
Con el fin de visualizar la relación entre la filogenia bacteriana y la producción especializada de metabolitos de colonias bacterianas que crecen en agar nutritivo, desarrollamos IDBac, una desorción/ionización asistida por matriz de bajo costo y alto rendimiento bioinformática de espectrometría de masas de tiempo de vuelo (MALDI-TOF MS). El software IDBac está diseñado para no expertos, está disponible libremente y es capaz de analizar algunas o miles de colonias bacterianas. Aquí, presentamos procedimientos para la preparación de colonias bacterianas para el análisis de MS MALDI-TOF, operación de instrumentos MS y procesamiento y visualización de datos en IDBac. En particular, instruimos a los usuarios cómo agrupar bacterias en dendrogramas basados en huellas dactilares de LA proteína MS y crear interactivamente redes de asociación de metabolitos (MAN) a partir de datos de metabolitos especializados.
Introducción
Una barrera importante para los investigadores que estudian la función bacteriana es la capacidad de evaluar rápida y simultáneamente la identidad taxonómica de un microorganismo y su capacidad para producir metabolitos especializados. Esto ha evitado avances significativos en la comprensión de la relación entre la filogenia bacteriana y la producción especializada de metabolitos en la mayoría de las bacterias aisladas del medio ambiente. Aunque los métodos basados en MS que utilizan huellas dactilares de proteínas para agrupar e identificar bacterias están bien descritos1,2,3,4, estos estudios se han realizado generalmente en pequeños grupos de aislados, de una manera específica de cada especie. Es importante destacar que la información sobre la producción especializada de metabolitos, un importante impulsor de la función microbiana en el medio ambiente, ha permanecido sin incorporarse en estos estudios. 5 recientemente proporcionaron una historia completa que detalla el uso bajo de MALDI-TOF MS para analizar metabolitos especializados y la escasez de software para aliviar los cuellos de botella bioinformáticos actuales. Para abordar estas deficiencias, creamos IDBac, una canalización bioinformática que integra los modos linealy reflectron de MALDI-TOF MS 6. Esto permite a los usuarios visualizar y diferenciar rápidamente los aislados bacterianos basados en las huellas dactilares de la BIÓ de proteínas y metabolitos especializados, respectivamente.
IDBac es rentable, de alto rendimiento y está diseñado para el usuario laico. Está disponible libremente (chasemc.github.io/IDBac), y sólo requiere acceso a un espectrómetro de masas MALDI-TOF (se requerirá el modo de reflejo para el análisis especializado de metabolitos). La preparación de la muestra se basa en el método simple de "transferencia directa extendida"7,8 y los datos se recopilan con adquisiciones lineales y de reflectron consecutivas en un único punto de destino maldiva. Con IDBac, es posible analizar la filogenia putativa y la producción especializada de metabolitos de cientos de colonias en menos de cuatro horas, incluyendo la preparación de muestras, la adquisición de datos y la visualización de datos. Esto presenta una ventaja significativa de tiempo y costo sobre los métodos tradicionales de identificación de bacterias (como la secuenciación de genes) y el análisis de la producción metabólica (cromatografía líquida-espectrometría de masas [LCMS] y métodos cromatográficos similares).
Utilizando datos obtenidos en el análisis en modo lineal, IDBac emplea clustering jerárquico para representar la relación de los espectros de proteínas. Dado que los espectros representan principalmente proteínas ribosomales ionizadas, proporcionan una representación de la diversidad filogenética presente en una muestra. Además, IDBac incorpora datos de modo reflectron para mostrar huellas dactilares de metabolitos especializados como redes de asociación de metabolitos (MÁN). Las MAN son redes bipartitas que permiten una fácil visualización de la producción de metabolitos compartidos y únicos entre aislados bacterianos. La plataforma IDBac permite a los investigadores analizar los datos de proteínas y metabolitos especializados en tándem, pero también individualmente si solo se adquiere un tipo de datos. Es importante destacar que IDBac procesa datos sin procesar de instrumentos Bruker y Xiamen, así como txt, tab, csv, mzXML y mzML. Esto elimina la necesidad de conversión manual y formato de conjuntos de datos, y reduce significativamente el riesgo de error del usuario o mal manejo de los datos de MS.
Protocolo
1. Preparación de la matriz MALDI
- Preparar 10 mg/ml de MALDI-grado, y/o recristalizado -ciano-4-ácido hidroxicinnamico (CHCA) en disolventes de grado MS: 50% acetonitrilo (ACN), 47,5% agua (H2O), 2,5% ácido trifluoroacético (TFA). Ejemplo: Solución de 100 l a 50 a/Ml aCN + 47,5 l H2O + 2,5 l de TFA + 1 mg de CHCA
- Preparar al menos 1 l de solución de matriz por punto de placa MALDI y vórtice o sonicado hasta que esté en solución (aproximadamente 5 min de sonicación o sin sólidos visibles).
ADVERTENCIA: El AFC es un ácido fuerte que debe manipularse en una campana de humos químicos mientras lleva el equipo de protección personal adecuado, ya que puede dañar la piel, los ojos y las vías respiratorias con contacto o inhalación.
NOTA: CHCA es higroscópico y sensible a la luz y debe almacenarse en viales de ámbar en un desecador. Hay muchas opciones de matriz MALDI disponibles. CHCA es más común para el perfilado de proteínas de bacterias, pero también funciona para el análisis de metabolitos especializados. La selección de matrices depende de las necesidades individuales del usuario/experimento.
- Preparar al menos 1 l de solución de matriz por punto de placa MALDI y vórtice o sonicado hasta que esté en solución (aproximadamente 5 min de sonicación o sin sólidos visibles).
2. Preparación de placas diana MALDI
NOTA: Para más detalles, véase Sauer et al.
- Enjuague la placa MALDI con metanol (grado HPLC o superior) y séquela con toallitas de papel blandas. No utilice cepillos abrasivos al limpiar las placas de destino, ya que esto puede dañar permanentemente la superficie de la placa de destino.
- Asigne puntos de calibrador de proteínas y metabolitos especializados. Organice los puntos de calibración uniformemente en toda la población de muestras, para tener en cuenta las irregularidades de placas MALDI y la deriva del instrumento a lo largo del tiempo. Asigne un número apropiado de puntos de medios/matriz en blanco para el estudio; estos puntos contendrán sólo medios y matriz, o sólo matriz.
- Usando un palillo estéril, transfiera una pequeña porción de una colonia bacteriana al lugar apropiado en la placa MALDI. Esparce la colonia bacteriana uniformemente sobre el lugar. El punto debe aparecer lo más plano posible.
NOTA: Será más fácil aplanar colonias bacterianas que son más mucoides/amorfas. Para colonias más rígidas/sólidas, evite dejar racimos visibles de masa celular en el punto MALDI (Figura1).
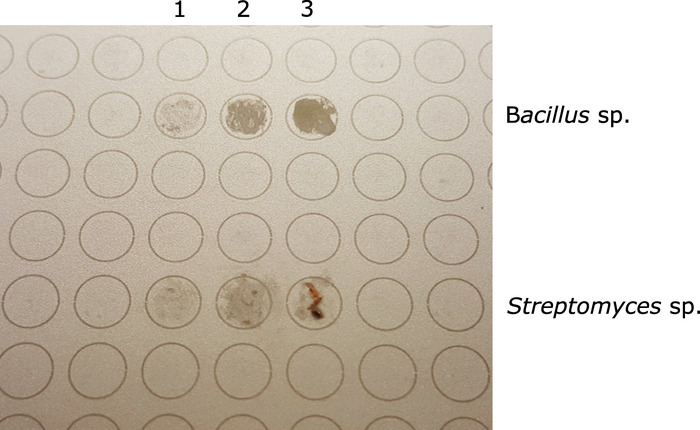
Figura 1: Placa objetivo MALDI que muestra dos aislados diferentes antes de añadir ácido fórmico y matriz MALDI (top 3 puntos - Bacillus sp.; inferior 3 puntos - Streptomyces sp.). Para ambos, la columna 3 representa el exceso de muestra; la columna 2 representa la cantidad adecuada de muestra; la columna 1 representa una muestra insuficiente para el análisis maldi(. Haga clic aquí para ver una versión más grande de esta figura.
- Prepare un control de matriz/medios utilizando un palillo estéril para transferir una cantidad mínima de agar/medios a los puntos apropiados de la placa MALDI.
- Superponer 1 l de ácido fórmico de grado de espectrometría de masas del 70% en cada punto de muestra, incluidos los puntos de control de matriz. Deje que el ácido se seque completamente en una campana de humo químico (aproximadamente 5 min).
ADVERTENCIA: El ácido fórmico es un producto químico cáustico y debe manipularse en campanas de humos químicos. Puede dañar las vías respiratorias si se inhala. - Agregue 1 l de la solución de matriz MALDI preparada a cada punto de muestra, así como a los puntos de control de matriz/medios. Deje que la solución de matriz se seque completamente al aire (aproximadamente 5 min).
NOTA: Es posible almacenar la placa en un desecador, en la oscuridad, hasta que pueda ser analizada en un espectrómetro de masas MALDI-TOF. Los tiempos de almacenamiento permitidos pueden variar dependiendo de la estabilidad de la muestra. - Añadir un calibrante de 0,5–1,0 l a los puntos de calibración asignados, seguido de una solución de matriz MALDI de 1 l. Pipetear la solución resultante hacia arriba y hacia abajo para mezclar. Deje que todas las manchas se sequen completamente antes de introducirlas en el espectrómetro de masas MALDI-TOF.
NOTA: Los calibradores de proteínas y metabolitos especializados deben añadirse dentro de los 30 minutos del análisis maldi,, ya que ambos son susceptibles a la degradación.
3. Adquisición de datos
NOTA: Los parámetros generales para la adquisición de datos se enumeran en el Cuadro 1.
| Parámetro | Proteína | Metabolito especializado |
| Inicio masivo (Da) | 1920 | 60 |
| Fin de masa (Da) | 21000 | 2700 |
| Desviación en masa (Da) | 1900 | 50 |
| Tiros | 500 | 1000 |
| Frecuencia (Hz) | 2000 | 2000 |
| Tamaño del láser | Grande | Medio |
| MaxStdDev (ppm) | 300 | 30 |
Tabla 1.
- Siguiendo los protocolos específicos del instrumento utilizado, configure calibraciones de proteínas y metabolitos especializados.
- Pruebe algunos puntos de destino separados para determinar la potencia láser óptima y la ganancia del detector para usar al adquirir espectros (esto variará día a día y por instrumento).
NOTA: La Figura 2A y la Figura 3A muestran espectros óptimos, mientras que la Figura 2D y la Figura 3D son ejemplos de espectros de mala calidad.
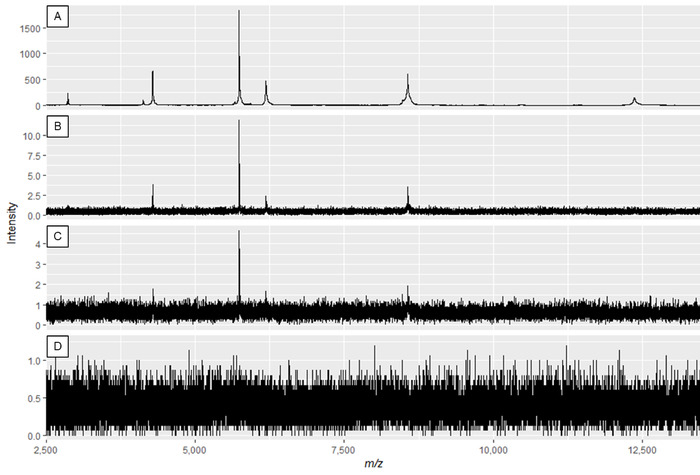
Figura 2: Ejemplo de espectros de proteínas que muestran el efecto de modificar la potencia del láser y la ganancia del detector. La calidad de los espectros es mejor en el panel A, y disminuye hasta que la calidad de los espectros es insuficiente en los paneles C y D. Mientras que el espectro en el panel B puede dar lugar a picos útiles, el panel A muestra datos óptimos. Haga clic aquí para ver una versión más grande de esta figura.
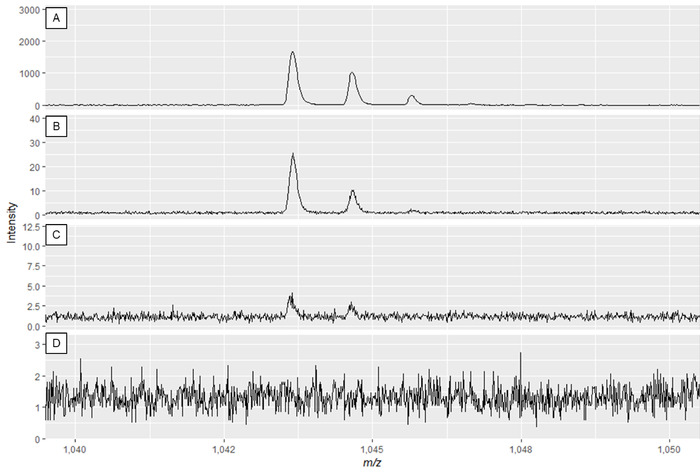
Figura 3: Ejemplo de espectros de metabolitos especializados que muestran el efecto de modificar la potencia del láser y la ganancia del detector. La calidad de los espectros es mejor en el panel A y disminuye hasta que la calidad de los espectros es insuficiente en los paneles C y D. Mientras que el espectro en el panel B puede dar lugar a picos útiles, el panel A muestra datos óptimos. Haga clic aquí para ver una versión más grande de esta figura.
- Adquiera espectros, ahorrando espectros de proteínas en una carpeta y espectros de metabolitos especializados en una segunda carpeta separada.
4. Limpieza de la placa de destino MALDI (adaptada de Sauer et al.7)
- Retire la placa de destino MALDI de su soporte y enjuague con acetona.
- Lávese con un jabón líquido no abrasivo para eliminar las proteínas traza y los lípidos, y las toallitas de papel suave/cepillo de dientes de cerdas suaves.
- Enjuagar con agua desionizada durante aproximadamente 2 minutos para eliminar completamente el jabón.
- Sonicar la placa objetivo en agua (grado HPLC o superior) durante 5 min.
- Enjuague la placa de destino con agua (grado HPLC o superior).
- Enjuague la placa diana con metanol (grado HPLC o superior).
5. Instalación del software IDBac
- Descargue el software IDBac.
NOTA: Las copias de seguridad permanentes y versionadas también están disponibles para su descarga (consulte la Tabla de materiales). - Haga doble clic en el programa descargado "Install_IDBac.exe" para iniciar el instalador y siga las instrucciones en pantalla.
6. Comenzando con los datos sin procesar
NOTA: Las explicaciones detalladas y las instrucciones de cada paso de procesamiento de datos se incrustan en IDBac, sin embargo, los principales análisis y entradas interactivas se describen a continuación.
- Haga doble clic en el acceso directo del escritorio IDBac para iniciar IDBac. IDBac se abrirá en la pestaña Introducción de forma predeterminada.
- Utilice el botón Buscar actualizaciones para asegurarse de que se está utilizando la versión más reciente de IDBac (requiere acceso a Internet). Si hay una versión más reciente disponible, IDBac descargará e instalará automáticamente la actualización, después de lo cual IDBac solicitará que se reinicie.
- Haga clic en la pestaña Comenzar con datos sin procesar y elija en el menú el tipo de datos que se utilizarán con IDBac; seguir las instrucciones de la aplicación.
- Al configurar la conversión y el procesamiento de archivos de datos, introduzca un nombre descriptivo para el experimento en el que se le solicite (consulte la figura 4). Los experimentos se mostrarán más adelante alfabéticamente, por lo que una estrategia útil es comenzar los nombres de experimentos con un atributo de grupo (por ejemplo, "bacillus-trials_experiment-1"; "bacillus-trials_experiment-2").
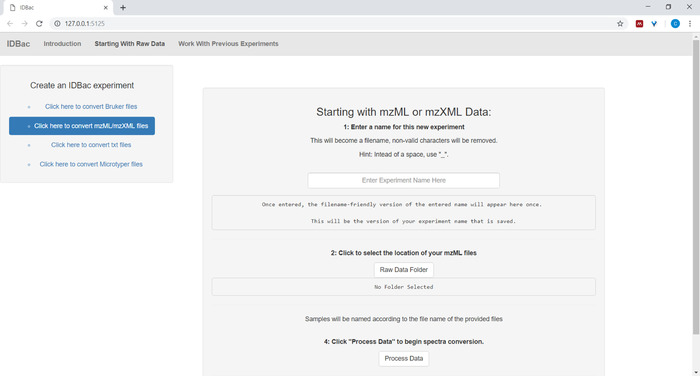
Figura 4: Paso de conversión y preprocesamiento de datos IDBac. IDBac convierte los espectros sin procesar en el formato mzML abierto y almacena mzML, listas de picos e información de muestra en una base de datos para cada experimento. Haga clic aquí para ver una versión más grande de esta figura.
7. Trabajar con experimentos anteriores
- Después de convertir archivos y procesarlos con IDBac, o en cualquier momento que desee volver a analizar un experimento, vaya a la página Trabajar con experimentos anteriores y Seleccione un experimento con el que trabajar ( Figura5).
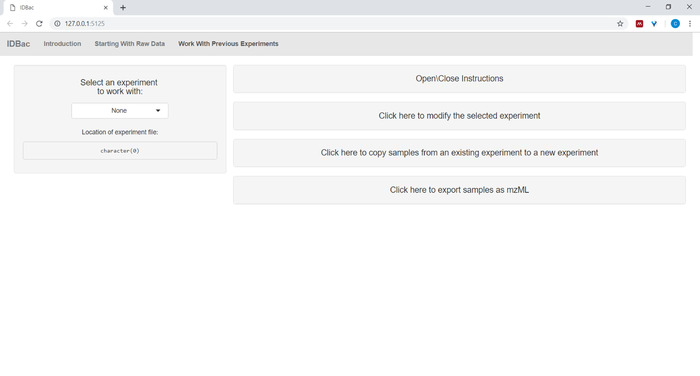
Figura 5: Página "Trabajar con experimentos anteriores". Utilice la página "Trabajar con experimentos anteriores" de IDBac para seleccionar un experimento para analizar o modificar. Haga clic aquí para ver una versión más grande de esta figura.
- (Opcional) Añadir información sobre ejemplos mediante el menú Haga clic aquí para modificar el experimentoseleccionado. Introduzca información en la hoja de cálculo rellenada automáticamente y pulse Guardar (Figura 6). Esta opción permite al usuario codificar datos por colordurante durante los análisis.
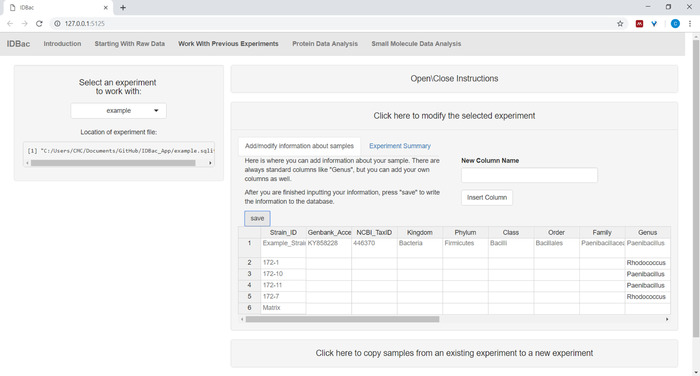
Figura 6: Información de muestra de entrada. Dentro de la página "Trabajar con experimentos anteriores" los usuarios pueden introducir información sobre muestras como identidad taxonómica, ubicación de la colección, condiciones de aislamiento, etc. Haga clic aquí para ver una versión más grande de esta figura.
- (Opcional) Transfiera todos o un subconjunto de muestras a un experimento nuevo u otro haciendo clic en Transferir muestras de experimentos anteriores a experimentos nuevos o otros y siguiendo las instrucciones proporcionadas (Figura7).
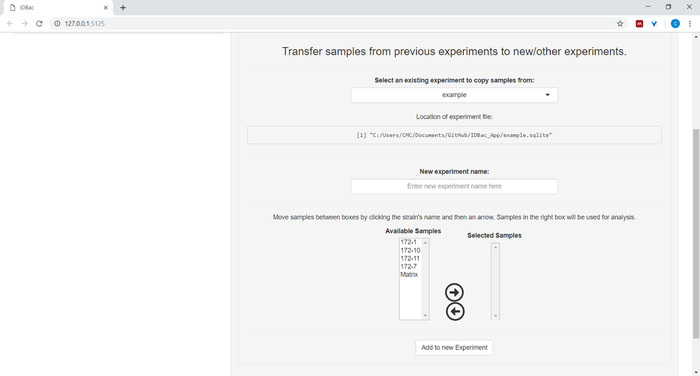
Figura 7: Transferir datos. La página "Trabajar con experimentos anteriores" contiene la opción de transferir datos entre experimentos existentes y a nuevos experimentos. Haga clic aquí para ver una versión más grande de esta figura.
- Cuando esté listo para comenzar el análisis, asegúrese de que el experimento con el que trabajar está seleccionado. Seleccione Análisis de datos de proteínas o Análisis de datos de moléculas pequeñas.
8. Configuración del análisis de datos proteicos y creación de gráficas espejo
- Si analiza los datos de proteínas, vaya primero a la página Análisis de datos de proteínas. Elija la configuración de selección de picos y evalúe los espectros de proteínas de las muestras a través de las gráficas de espejo mostradas (Figura8).
NOTA: En las gráficas de espejo, un pico rojo significa la presencia de ese pico sólo en el espectro superior, mientras que los picos azules representan aquellos que ocurren en ambos espectros.
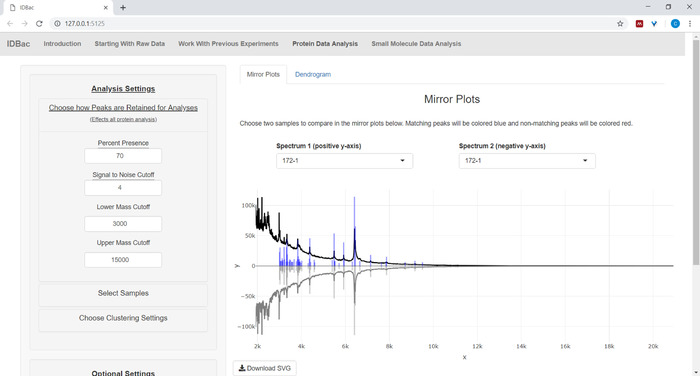
Figura 8: Elija cómo se conservan los picos para el análisis. Después de seleccionar un experimento para analizar, visitar la página "Análisis de datos de proteínas" y, posteriormente, abrir el menú "Elegir cómo se retienen los picos para el análisis" permite a los usuarios elegir ajustes como la relación señal-ruido para retener picos. El trazado de espejo mostrado (o dendrograma) se actualizará automáticamente para reflejar los ajustes elegidos. Haga clic aquí para ver una versión más grande de esta figura.
- Ajustar el porcentaje de réplicas en las que debe estar presente un pico para que se incluya para los análisis (por ejemplo, si el umbral se establece en 70% y un pico se produce en al menos 7 de cada 10 réplicas, se incluirá).
- Usando las gráficas de espejo como guía visual, ajuste la señal al corte de ruido que retenga los picos más "auténticos" y el menor ruido, señalando que más réplicas y un valor más alto de "presencia máxima porcentual" permitirán seleccionar un corte de señal más bajo al ruido.
- Especifique los cortes m/z inferior y superior, dictando el rango de valores de masa dentro de cada espectro que se utilizará en análisis posteriores por IDBac.
9. Agrupación de muestras utilizando datos proteicos
- En la página Análisis de datos de proteínas, seleccione la pestaña Dendrogram. Esto permite agrupar muestras en un dendrograma de acuerdo con las medidas de distancia seleccionadas por el usuario y los algoritmos de agrupación en clústeres.
- Haga clic en Seleccionar muestras en el menú y siga las instrucciones para seleccionar las muestras que desea incluir en los análisis. Solo las muestras que contengan espectros de proteínas se mostrarán en el cuadro Muestras disponibles (Figura9).
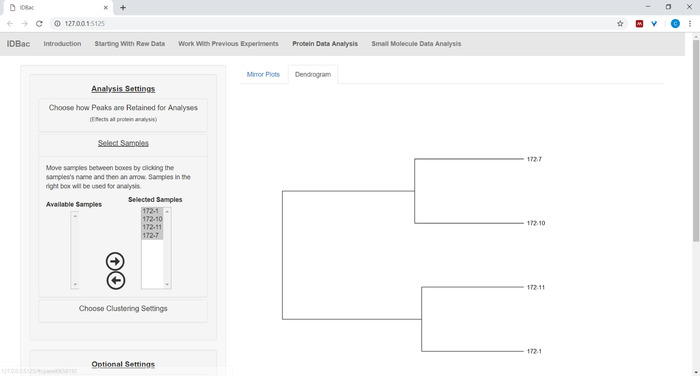
Figura 9: Seleccione muestras del experimento elegido para incluirlas en el dendrograma mostrado. Haga clic aquí para ver una versión más grande de esta figura.
- Utilice los valores predeterminados o, en Elegir configuración de agrupaciónen clústeres , seleccione los algoritmos de distancia y agrupación en clústeres deseados que se aplicarán a la generación del dendrograma.
- Seleccione Presencia/Ausencia como entrada. Alternativamente, si confía en las alturas máximas de las muestras (por ejemplo, después de realizar un estudio para evaluar la variabilidad de la intensidad máxima), seleccione Intensidades como entrada.
NOTA: En el momento de la publicación, IDBac proporciona flexibilidad en la configuración de la agrupación en clústeres, confiando en que los usuarios elijan las combinaciones adecuadas. Si no está familiarizado con estas opciones, se sugiere emparejar A: distancia "coseno" y clustering "promedio (UPGMA)"; o B: distancia "euclidiana" y agrupación "Ward.D2". - Para mostrar los valores de arranque en el dendrograma, escriba un número entre 2 y 1000 en Bootstraps.
- Al informar de los resultados, copie el texto en el párrafo Sugerencias para informar del análisis de proteínas. Esto proporciona la configuración definida por el usuario que generó el dendrograma específico.
10. Personalización del dendrograma proteico
- Para comenzar a personalizar el dendrograma, abra el menú Ajustar el dendrograma (Figura10).
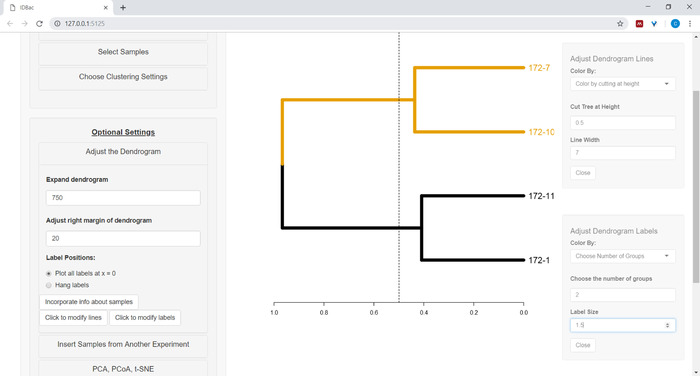
Figura 10: Ajuste el dendrograma. IDBac proporciona algunas opciones para modificar el aspecto del dendrograma, estas se pueden encontrar en el menú "Ajustar el dendrograma". Esto incluye colorear ramas y etiquetas por k-means, o "cortar" el dendrograma a una altura proporcionada por el usuario. Haga clic aquí para ver una versión más grande de esta figura.
- Para colorear las líneas y/o etiquetas del dendrograma, seleccione el botón correspondiente: Haga clic para modificar las líneas o haga clic para modificar las etiquetas y seleccione las opciones deseadas.
- Para trazar información de la hoja de cálculo junto al dendrograma (consulte el paso 7.2), seleccione el botón Incorporar información sobre muestras. Esto abrirá un panel donde una categoría (columna en la hoja de cálculo) se rellenará automáticamente en función de los valores introducidos (Figura 11).
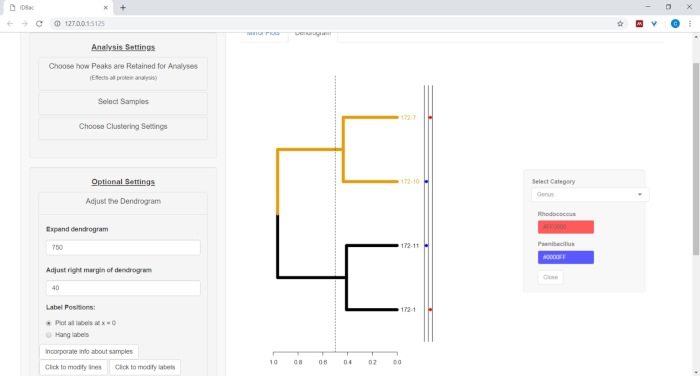
Figura 11: Incorporar información sobre muestras. Dentro del menú "Ajustar el Dendrogram" se encuentra la opción "Incorporar información sobre muestras". Si selecciona esta opción, podrá trazar información sobre muestras junto al dendrograma. La información de ejemplo se introduce en la página "Trabajar con experimentos anteriores". Haga clic aquí para ver una versión más grande de esta figura.
11. Inserte muestras de un experimento separado en el dendrograma
- Para insertar muestras de otro experimento, seleccione el botón de menú Insertar muestras de otro experimento. Siga las instrucciones del panel recién inaugurado (Figura12).
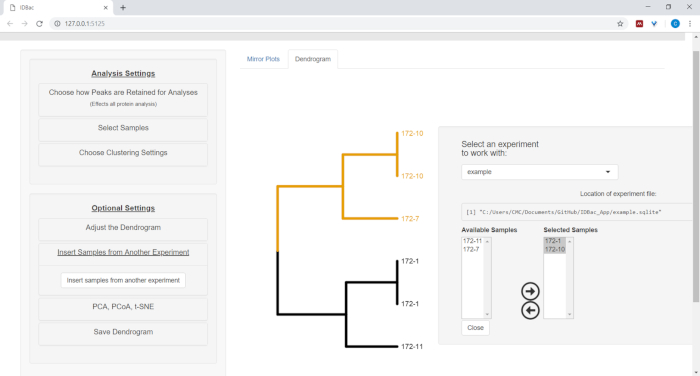
Figura 12: Insertar muestras desde otro menú de experimento. A veces es útil comparar muestras de otro experimento. Utilice el menú "Insertar muestras de otro experimento" para elegir los ejemplos que se incluirán en el dendrograma que se muestra actualmente. Haga clic aquí para ver una versión más grande de esta figura.
12. Análisis de datos de metabolitos especializados y redes de asociación de metabolitos (MUN)
- Continúe con la página Red de la Asociación de Metabolitos (Pequeña-Molécula). Esta página permite la visualización de datos por análisis de componentes principales (PCA) y MAN, que utilizan redes bipartitas para mostrar la correlación de valores m/z de moléculas pequeñas con muestras.
- Si se creó un dendrograma proteico (sección 9), también se mostrará en esta página. Haga clic y arrastre en el dendrograma para resaltar las muestras seleccionadas de interés que se van a analizar. Si no se resalta ninguna muestra o no se ha creado ningún dendrograma proteico, aparecerá un MAN de un subconjunto aleatorio o todas las muestras, respetuosamente (Figura13).
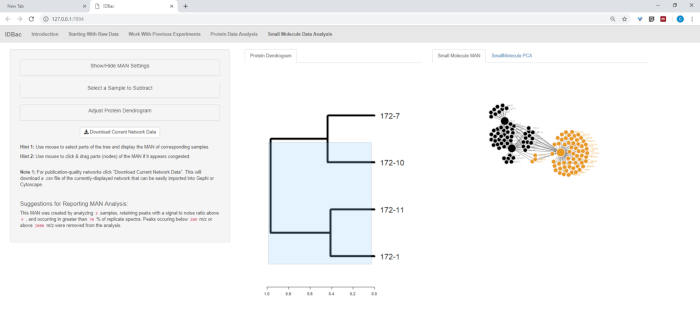
Figura 13: Página Análisis de datos de moléculas pequeñas". Si se creó un dendrograma a partir de espectros de proteínas, se mostrará en la página "Análisis de datos de moléculas pequeñas". Esta página también mostrará las redes asociadas de metabolito (MÁN) y el análisis de componentes principales (PCA) para los datos de moléculas pequeñas. Haga clic aquí para ver una versión más grande de esta figura.
- Para restar una matriz/media en blanco en el MAN, abra el menú Seleccionar una muestra para restar y elija la muestra adecuada para utilizarla como espacio en blanco.
- Abra el menú Mostrar/Ocultar configuración de MAN para seleccionar los valores deseados para el porcentaje de presencia máxima en réplicas, señal a ruido y cortes de masa superior e inferior, como se hizo para los espectros de proteínas en la Sección 9. Utilice las pequeñas gráficas de espejo de moléculas para guiar la selección de estos ajustes.
- Seleccione "Descargar datos de red actuales" para guardar los datos del MAN que se muestra actualmente. Estos datos se pueden utilizar en software de análisis de red que no sea IDBac.
- Para obtener informes de los resultados, copie el texto en el párrafo Sugerencias para informar el análisis DE MAN. Esto proporciona la configuración definida por el usuario utilizada para generar el MAN creado.
13. Compartir datos
- Cada "experimento" idBac se guarda como una sola base de datos SQLite. Contiene los espectros sin procesar mzML convertidos, picos detectados e información de entrada del usuario sobre las muestras. Por lo tanto, para compartir un experimento IDBac simplemente compartir el archivo SQlite que tiene el mismo nombre que el experimento (la ubicación del archivo se muestra en la página Trabajar con experimentos anteriores).
Resultados
Analizamos seis cepas de Micromonospora chokoriensis y dos cepas de Bacillus subtilis,que anteriormente se caracterizaron6,utilizando los datos disponibles en DOI: 10.5281/zenodo.2574096. Siguiendo las instrucciones en la pestaña Comenzando con datos sin procesar, seleccionamos la opción Haga clic aquí para convertir archivos Bruker y seguimos las instrucciones proporcionadas por IDBac para cada conjunto de datos (Figura14).
Después de que se completaron los pasos automatizados de conversión y preprocesamiento/pico, procedimos a crear un nuevo experimento combinado de IDBac transfiriendo muestras de los dos experimentos a un solo experimento que contiene tanto Bacillus como Muestras de micromonosoporas (Figura15). El análisis resultante implicó comparar espectros de proteínas utilizando gráficas de espejo, como se muestra en la Figura 16,que fue útil para evaluar la calidad de los espectros y ajustar la configuración de selección de picos. La Figura 17 muestra una captura de pantalla de los resultados de agrupación en clústeres de proteínas con la configuración predeterminada seleccionada. El dendrograma se coloreó ajustando el umbral en el trazado (aparece como una línea punteada). Cabe destacar la clara separación entre géneros, con M. chokoriensis y B. subtilis aíslan clustering por separado.
La Figura 18, la Figura 19y la Figura 20 resaltan la capacidad de generar MAN de regiones seleccionadas por el usuario haciendo clic y arrastrando a través del dendrograma de proteínas. Con esto pudimos crear rápidamente MANs para comparar sólo las cepas B. subtilis (Figura18), sólo las cepas M. chokoriensis (Figura19),y todas las cepas simultáneamente (Figura20). La función principal de estas redes es proporcionar a los investigadores una amplia visión general del grado de superposición de metabolitos especializados entre bacterias. Con estos datos en la mano, los investigadores ahora tienen la capacidad de tomar decisiones informadas a partir de sólo una pequeña cantidad de material raspado de una colonia bacteriana.
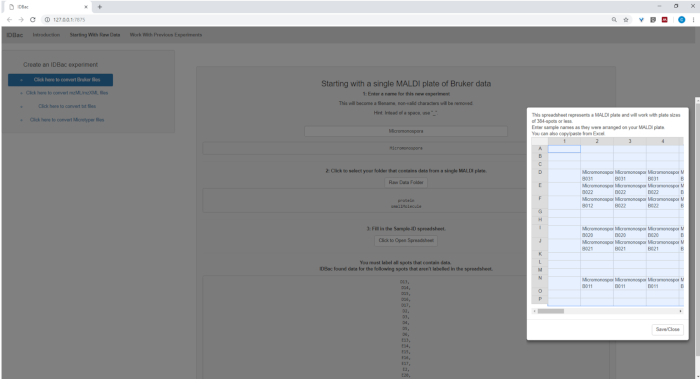
Figura 14: Procesamiento de espectros. Los espectros de Bruker autoFlex descargados se convirtieron y procesaron con IDBac. Haga clic aquí para ver una versión más grande de esta figura.
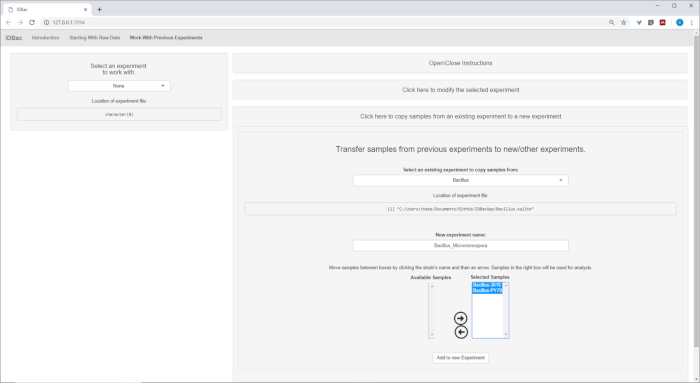
Figura 15: Experimento IDBac combinado. Debido a que los espectros Micromonospora y Bacillus fueron recogidos en diferentes placas de destino MALDI, los dos experimentos se combinaron posteriormente en un solo experimento: "Bacillus_Micromonsopora". Esto se hizo dentro de la pestaña "Trabajar con experimentos anteriores", siguiendo las instrucciones dentro del menú "Transferir muestras de experimentos anteriores a experimentos nuevos /otros". Haga clic aquí para ver una versión más grande de esta figura.
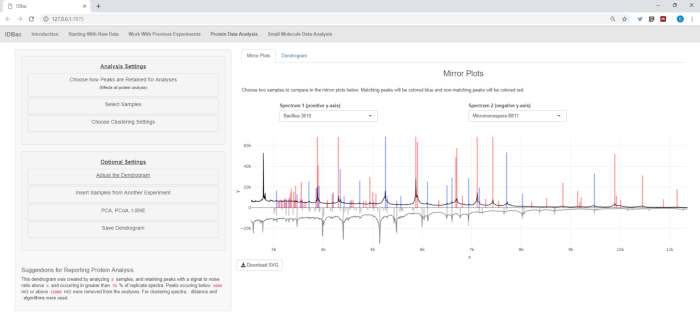
Figura 16: Comparación. Micromonspora y Bacillus spectra se compararon utilizando las gráficas de espejo dentro de la página "Análisis de datos de proteínas". En última instancia, se eligieron los ajustes de pico predeterminados. Haga clic aquí para ver una versión más grande de esta figura.
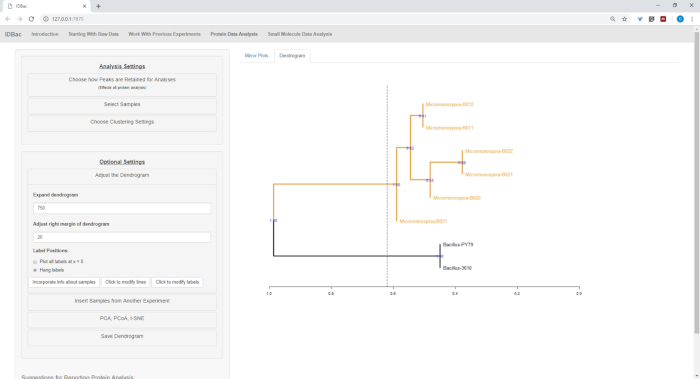
Figura 17: Agrupación jerárquica. La agrupación jerárquica, utilizando la configuración predeterminada, agrupa correctamente bacillus y micromonospora se aísla. El dendrograma fue coloreado por "cortar" el dendrograma a una altura arbitraria (mostrada como una línea discontinua) y 100 bootstraps utilizados para mostrar confianza en la ramificación. Haga clic aquí para ver una versión más grande de esta figura.
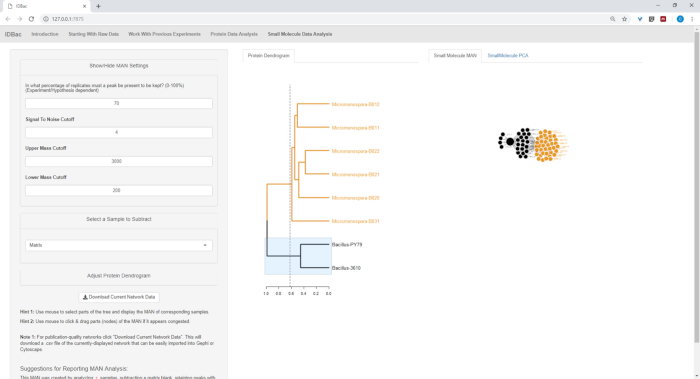
Figura 18: EL HOMBRE creado mediante la selección de las cepas de Bacillus sp. del dendrograma proteico mostró la producción diferencial de metabolitos especializados. Haga clic aquí para ver una versión más grande de esta figura.
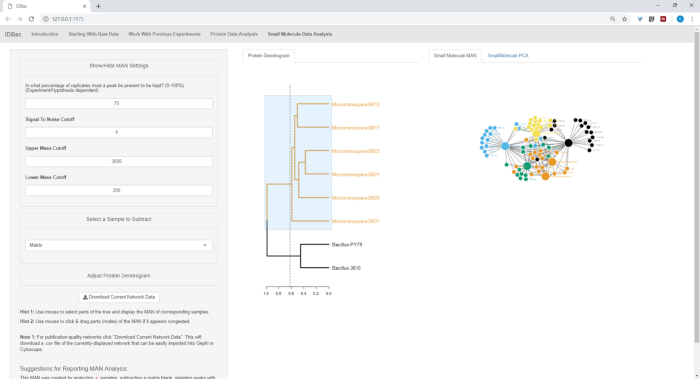
Figura 19: MAN creado mediante la selección de las seis cepas Micromonospora sp. del dendrograma proteico mostró la producción diferencial de metabolitos especializados. Haga clic aquí para ver una versión más grande de esta figura.
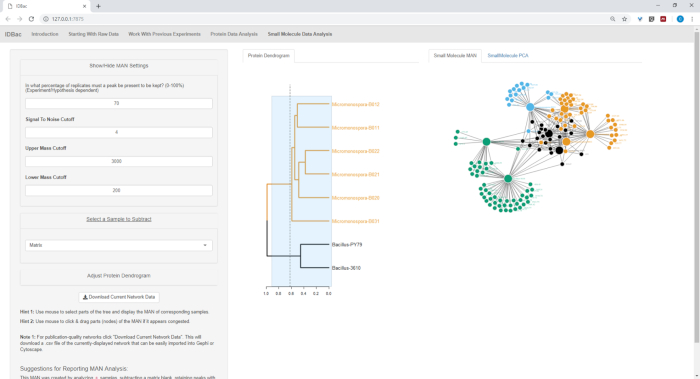
Figura 20: MAN de Bacillus sp. y Micromonospora sp. cepas que muestran una producción diferencial de metabolitos especializados. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo IDBac detalla la adquisición y análisis de datos de proteínas bacterianas y metabolitos especializados de hasta 384 aislados bacterianos en 4 h por un solo investigador. Con IDBac no hay necesidad de extraer ADN de aislados bacterianos o generar extractos de metabolitos especializados de caldos de fermentación líquida y analizarlos utilizando métodos cromatográficos. En su lugar, los datos de proteínas y metabolitos especializados se recopilan simplemente esparciendo material de colonias bacterianas directamente en una placa diana maldi. Esto reduce en gran medida el tiempo y el costo asociados con técnicasalternativas como la secuenciación del gen 16S rRNA y LCMS 9.
Es importante añadir una matriz en blanco y puntos de calibración a la placa MALDI, y recomendamos utilizar un número adecuado de réplicas para garantizar la reproducibilidad y la confianza estadística. El número de réplicas dependerá del experimento. Por ejemplo, si un usuario tiene la intención de diferenciar miles de colonias de una colección de placas de diversidad ambiental, pueden ser necesarias menos réplicas (nuestro laboratorio recoge tres réplicas técnicas por colonia). Alternativamente, si un usuario desea crear una base de datos personalizada de cepas de taxones bacterianos específicos para determinar rápidamente las clasificaciones de subespecies de aislados desconocidos, entonces más réplicas son apropiadas (nuestro laboratorio recoge ocho réplicas biológicas por tensión).
IDBac es una herramienta para diferenciar rápidamente aislados bacterianos altamente relacionados basados en información taxonómica putativa y la producción especializada de metabolitos. Puede complementar o servir como precursor de métodos ortogonales tales como análisis genéticos en profundidad, estudios que implican la producción y función de metabolitos, o la caracterización de la estructura de metabolitos especializados por espectroscopia de Resonancia Magnética Nuclear y/o LC-MS/MS.
La producción especializada de metabolitos (IDBac MANs) es altamente susceptible a las condiciones de crecimiento bacteriano, especialmente utilizando diferentes medios, que es una limitación potencial del método. Sin embargo, estos rasgos pueden ser explotados por el usuario, como IDBac puede generar fácilmente MANs mostrando las diferencias en la producción especializada de metabolitos bajo una variedad de condiciones de crecimiento. Es importante tener en cuenta que si bien las huellas dactilares de metabolitos especializados pueden variar según la condición de crecimiento, hemos demostrado previamente que las huellas dactilares de proteínas permanecen relativamente estables en estas variables (véase Clark et al.6). Cuando se trata de placas de diversidad ambiental, recomendamos purificar los aislados bacterianos antes del análisis con el fin de reducir las posibles contribuciones de la conversación cruzada bacteriana vecina.
Por último, la falta de una base de datos pública en la que se pueden realizar búsquedas de huellas dactilares de la proteína MS es una deficiencia importante en el uso de este método para clasificar bacterias ambientales desconocidas. Creamos IDBac con esto en mente, e incluimos la conversión automatizada de datos en un formato de código abierto aceptado por la comunidad (mzML)10,11,12 y diseñamos el software para permitir la búsqueda, el intercambio y la creación de bases de datos personalizadas. Estamos en proceso de crear una gran base de datos pública (>10.000 cepas plenamente caracterizadas), lo que permitirá la clasificación de algunos aislados a nivel de especie, incluyendo enlaces a números de adhesión de GenBank cuando estén disponibles.
IDBac es de código abierto y el código está disponible para que cualquiera pueda personalizar sus necesidades de análisis y visualización de datos. Recomendamos que los usuarios consulten un amplio cuerpo de literatura (Sauer et al.7, Silva et al.5) para ayudar a apoyar y diseñar sus objetivos experimentales. Organizamos un foro de discusión en: https://groups.google.com/forum/#!forum/idbac y un medio para informar de problemas con el software en: https://github.com/chasemc/IDBacApp/issues.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el Instituto Nacional de Ciencias Médicas Generales Grant R01 GM125943, National Geographic Grant CP-044R-17; Subvención 152336-051 del Fondo de Investigación de Islandia; y fondos de inicio de la Universidad de Illinois en Chicago. Además, agradecemos a los siguientes contribuyentes: la Dra. Amanda Bulman por la asistencia con los parámetros de adquisición de proteínas MALDI-TOF MS; Dr. Terry Moore y Dr. Atul Jain por recristalizar la matriz de ácido alfa-ciano-4-hidroxicinnamic (CHCA).
Materiales
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
Referencias
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados