Method Article
Utilizzo della pipeline OPEN-Source MALDI TOF-MS IDBac per l'analisi dei dati sulle proteine microbiche e sui metaboliti specializzati
In questo articolo
Riepilogo
IDBac è una pipeline di bioinformatica a base di spettrometria di massa open source che integra dati provenienti da proteine intatte e spettri di metaboliti specializzati, raccolti su materiale cellulare raschiato da colonie batteriche. La conduttura consente ai ricercatori di organizzare rapidamente centinaia di migliaia di colonie batteriche in gruppi tassonomici putativi e di differenziarle ulteriormente in base alla produzione specializzata di metaboliti.
Abstract
Per visualizzare la relazione tra la filoicina batterica e la produzione specializzata di metaboliti di colonie batteriche che crescono sull'agar nutritivo, abbiamo sviluppato IDBac, un sistema di desorpizzazione/ionizzazione del laser a matrice a basso costo e ad alta produttività spettrorometria di massa a tempo di volo (MALDI-TOF MS) pipeline bioinformatica. Il software IDBac è progettato per i non esperti, è liberamente disponibile e in grado di analizzare da poche a migliaia di colonie batteriche. Qui presentiamo le procedure per la preparazione di colonie batteriche per l'analisi MALDI-TOF MS, il funzionamento dello strumento MS e l'elaborazione e la visualizzazione dei dati in IDBac. In particolare, istruiamo gli utenti su come raggruppare i batteri in dendrogrammi sulla base delle impronte digitali delle proteine MS e creare interattivamente Metabolite Association Networks (MAN) da dati specializzati sui metaboliti.
Introduzione
Una delle principali barriere per i ricercatori che studiano la funzione batterica è la capacità di valutare rapidamente e simultaneamente l'identità tassonomica di un microrganismo e la sua capacità di produrre metaboliti specializzati. Ciò ha impedito progressi significativi nella comprensione della relazione tra filologia batterica e produzione specializzata di metaboliti nella maggior parte dei batteri isolati dall'ambiente. Sebbene i metodi basati sulla SM che utilizzano impronte proteiche per raggruppare e identificare i batteri siano ben descritti1,2,3,4, questi studi sono stati generalmente eseguiti su piccoli gruppi di isolati, in modo specifico per le specie. È importante sottolineare che le informazioni sulla produzione di metaboliti specializzati, uno dei principali fattori della funzione microbica nell'ambiente, sono rimaste non incorporate in questi studi. Silva et al.5 ha recentemente fornito una storia completa che descrive il sottoutilizzo di MALDI-TOF MS per analizzare i metaboliti specializzati e la carenza di software per alleviare gli attuali colli di bottiglia bioinformatica. Per far fronte a queste carenze, abbiamo creato IDBac, una pipeline di bioinformatica che integra modalità lineare e riflettore di MALDI-TOF MS6. Ciò consente agli utenti di visualizzare e differenziare rapidamente gli isolati batterici in base alle impronte digitali MS sia proteiche che specializzate.
IDBac è conveniente, ad alta velocità effettiva e progettato per l'utente laico. È liberamente disponibile (chasemc.github.io/IDBac) e richiede solo l'accesso a uno spettrometro di massa MALDI-TOF (la modalità riflettore sarà necessaria per l'analisi specialized dei metaboliti). La preparazione del campione si basa sul semplice metodo di "trasferimento diretto esteso"7,8 e i dati vengono raccolti con acquisizioni lineari e di riflettore consecutive su un singolo punto MALDI-target. Con IDBac, è possibile analizzare la produzione di filo putativo e metabolita specializzata di centinaia di colonie in meno di quattro ore, tra cui la preparazione del campione, l'acquisizione dei dati e la visualizzazione dei dati. Questo presenta un significativo vantaggio in termini di tempo e costi rispetto ai metodi tradizionali di identificazione dei batteri (come il sequenziamento genico) e all'analisi della produzione metabolica (spettrometria cromatometria-massa liquida [LCMS] e metodi cromatografici simili).
Utilizzando i dati ottenuti nell'analisi in modalità lineare, IDBac impiega il clustering gerarchico per rappresentare la parentela degli spettri proteici. Poiché gli spettri rappresentano per lo più proteine ribosomiche ionizzate, forniscono una rappresentazione della diversità filogenetica presente in un campione. Inoltre, IDBac incorpora dati in modalità riflettore per visualizzare le impronte digitali dei metaboliti specializzati come Metabolite Association Networks (MAN). I MAN sono reti bipartite che consentono una facile visualizzazione della produzione di metaboliti condivisi e unici tra isolati batterici. La piattaforma IDBac consente ai ricercatori di analizzare sia i dati sulle proteine che sui metaboliti specializzati in tandem, ma anche individualmente se viene acquisito un solo tipo di dati. È importante sottolineare che IDBac elabora i dati non elaborati degli strumenti Bruker e Xiamen, nonché txt, tab, csv, mzXML e mzML. Ciò elimina la necessità di conversione manuale e formattazione dei set di dati e riduce significativamente il rischio di errore dell'utente o di gestione errata dei dati MS.
Protocollo
1. Preparazione della matrice MALDI
- Preparare 10 mg/mL di acido MALDI- e/o riconfondibile di z-cyano-4-idrossicinnamici (CHCA) in solventi MS-grade: 50% acetonitrile (ACN), 47,5% di acqua (H2O), 2,5% di acido trifluoroacetico (TFA). Esempio: 100 soluzione ll : 50 ACN - 47,5 S L H2O
- Preparare almeno 1 l di soluzione a matrice per punto di piastra e vortice MALDI o sonicare fino a quando non è in soluzione (circa 5 minimi o nessun solido visibile).
PRUDENTE: TFA è un acido forte che deve essere maneggiato in un cappuccio del fumi chimico mentre indossa un'adeguata attrezzatura protettiva personale, in quanto può danneggiare la pelle, gli occhi e le vie aeree con contatto o inalazione.
NOTA: CHCA è igroscopica e sensibile alla luce e deve essere conservata in fiale d'ambra in un desiccatore. Sono disponibili molte opzioni di matrice MALDI. CHCA è più comune per la profilazione delle proteine dei batteri, ma funziona anche per l'analisi specializzata dei metaboliti. La selezione della matrice dipende dalle esigenze individuali dell'utente/esperimento.
- Preparare almeno 1 l di soluzione a matrice per punto di piastra e vortice MALDI o sonicare fino a quando non è in soluzione (circa 5 minimi o nessun solido visibile).
2. Preparazione di piastre bersaglio MALDI
NOTA: Vedere Sauer et al.7, per maggiori dettagli.
- Risciacquare la piastra MALDI con metanolo (grado HPLC o superiore) e asciugare con salviette di carta morbida. Non utilizzare spazzole abrasive durante la pulizia delle piastre bersaglio, in quanto ciò può danneggiare in modo permanente la superficie della piastra bersaglio.
- Assegnare macchie di calibro proteico e metabolita specializzato. Organizzare i punti di calibrazione in modo uniforme tra la popolazione campione, per tenere conto delle irregolarità della piastra MALDI e della deriva degli strumenti nel tempo. Assegnare un numero adeguato di punti media/matrice-vuoti per lo studio; questi spot conterranno solo supporti e matrici, o solo matrice.
- Utilizzando uno stuzzicadenti sterile, trasferire una piccola porzione di una colonia batterica nel punto appropriato della piastra MALDI. Diffondere la colonia batterica in modo uniforme sul posto. Il punto dovrebbe apparire il più piatto possibile.
NOTA: Sarà più facile appiattire le colonie batteriche che sono più mucoide/ amorfo. Per colonie più rigide/solide, evitare di lasciare gruppi visibili di massa cellulare nello spot MALDI (Figura 1).
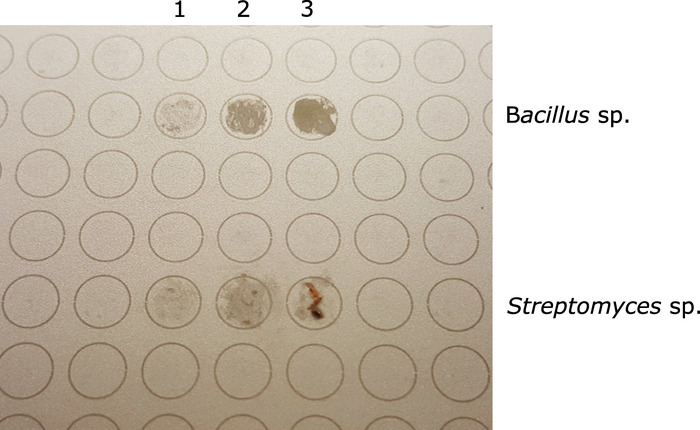
Figura 1: piastra bersaglio MALDI che mostra due diversi isolati prima di aggiungere acido formico e matrice MALDI (primi 3 punti - Bacillus sp.; 3 punti inferiori - Streptomyces sp.). Per entrambi, la colonna 3 rappresenta il campione in eccesso; la colonna 2 rappresenta la quantità appropriata di campione; colonna 1 rappresenta un campione insufficiente per l'analisi MALDI. Fare clic qui per visualizzare una versione più grande di questa figura.
- Preparare un controllo matrice/supporto utilizzando uno stuzzicadenti sterile per trasferire una quantità minima di agar/media sui punti appropriati sulla piastra MALDI.
- Sovrapporre 1 L di 70% di spettrometria di massa grado acido formico su ogni punto campione, compresi i punti di controllo della matrice. Lasciare asciugare completamente l'acido in una cappa di fumi chimici (circa 5 min).
AVVISO: L'acido formico è una sostanza chimica caustica e deve essere maneggiato in cappe di fumi chimici. Può danneggiare le vie respiratorie se inalato. - Aggiungere 1 l della soluzione di matrice MALDI preparata a ogni punto campione, nonché agli spot di controllo matrice/media. Lasciare asciugare completamente la soluzione a matrice (circa 5 min).
NOTA: È possibile conservare la piastra in un desiccatore, al buio, fino a quando non può essere analizzata su uno spettrometro di massa MALDI-TOF. I tempi di archiviazione consentiti possono variare a seconda della stabilità del campione. - Aggiungere il calibro da 0,5 a 1,0 l'area ai punti di calibrazione assegnati, seguito da una soluzione di matrice MALDI. Pipette la soluzione risultante su e giù per mescolare. Lasciare asciugare completamente tutti i punti prima dell'introduzione nello spettrometro di massa MALDI-TOF.
NOTA: La proteina e i calibratori dei metaboliti specializzati devono essere aggiunti entro 30 min dall'analisi MALDI, in quanto entrambi sono suscettibili alla degradazione.
3. Acquisizione dei dati
NOTA: i parametri generali per l'acquisizione dei dati sono elencati nella Tabella 1.
| parametro | proteina | Metabolita specializzato |
| Inizio massa (Da) | 1920 | 60 del sistema |
| Fine massa (Da) | 21000 | 2700 |
| Deflessione di massa (Da) | 1900 | 50 anni |
| Colpi | 500 | 1000 |
| Frequenza (Hz) | 2000 | 2000 |
| Dimensioni laser | l f inv | medio |
| MaxStdDev (ppm) | 300 | 30 milio |
Tabella 1.
- Seguendo i protocolli specifici per lo strumento utilizzato, impostare sia le calibrazioni proteiche che metabolite specializzate.
- Testare alcuni punti bersaglio separati per determinare la potenza ottimale del laser e il guadagno del rivelatore da utilizzare durante l'acquisizione di spettri (questo varierà giorno per giorno e per strumento).
NOTA: Figura 2A e Figura 3A mostrano spettri ottimali, mentre Figura 2D e Figura 3D sono esempi di spettri di scarsa qualità.
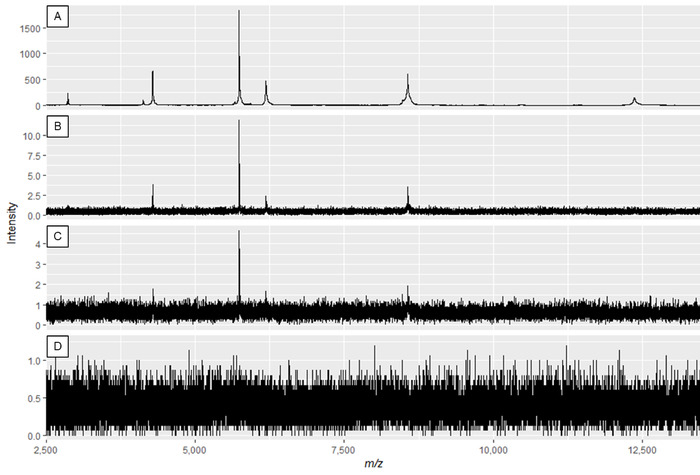
Figura 2: Esempio di spettri proteici che mostrano l'effetto della modifica della potenza laser e del guadagno del rivelatore. La qualità degli spettri è migliore nel pannello A, e diminuisce fino a quando insufficiente qualità degli spettri nei pannelli C e D. Mentre lo spettro nel pannello B può comportare picchi utilizzabili, il pannello A visualizza i dati ottimali. Fare clic qui per visualizzare una versione più grande di questa figura.
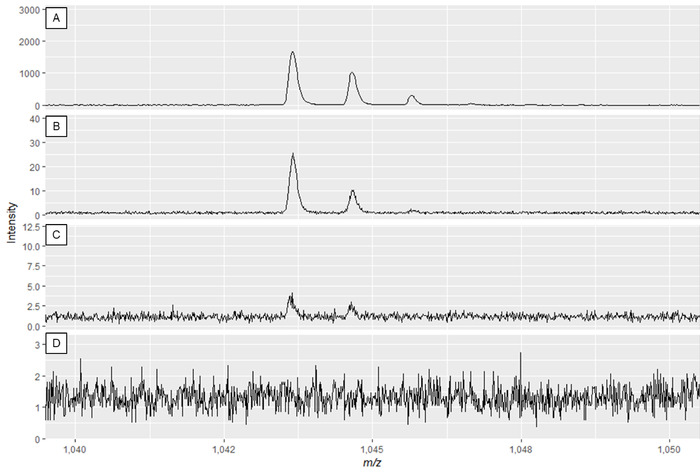
Figura 3: Esempio di spettri di metaboliti specializzati che mostrano l'effetto della modifica della potenza laser e del guadagno del rivelatore. La qualità degli spettri è migliore nel pannello A e diminuisce fino a quando non è insufficiente la qualità degli spettri nei pannelli C e D. Mentre lo spettro nel pannello B può comportare picchi utilizzabili, il pannello A visualizza i dati ottimali. Fare clic qui per visualizzare una versione più grande di questa figura.
- Acquisire spettri, salvare gli spettri proteici in una cartella e gli spettri di metaboliti specializzati in una seconda cartella separata.
4. Pulizia della piastra bersaglio MALDI (adattata da Sauer et al.7)
- Rimuovere la piastra bersaglio MALDI dal supporto e risciacquare con acetone.
- Lavare con un sapone liquido non abrasivo per rimuovere proteine traccia e lipidi, e salviette di carta morbida / spazzolino a setole morbide.
- Risciacquare con acqua de-ionizzata per circa 2 min per rimuovere completamente il sapone.
- Sonicare la piastra bersaglio in acqua (grado HPLC o superiore) per 5 min.
- Sciacquare la piastra bersaglio con acqua (grado HPLC o superiore).
- Risciacquare la piastra bersaglio con metanolo (grado HPLC o superiore).
5. Installazione del software IDBac
- Scaricare il software IDBac.
NOTA: per il download sono disponibili anche backup permanenti con versione (vedere la Tabella dei materiali). - Fare doppio clic sul scaricato "Install_IDBac.exe" per avviare il programma di installazione e seguire le istruzioni visualizzate.
6. A partire dai dati grezzi
NOTA: le spiegazioni dettagliate e le istruzioni di ogni fase di elaborazione dei dati sono incorporate all'interno di IDBac, tuttavia le principali analisi e input interattivi sono descritti di seguito.
- Fare doppio clic sul collegamento sul desktop IDBac per avviare IDBac. IDBac verrà aperto nella scheda Introduzione per impostazione predefinita.
- Utilizzare il pulsante Controlla aggiornamenti per assicurarsi che venga utilizzata la versione più recente di IDBac (richiede l'accesso a Internet). Se è disponibile una versione più recente, IDBac scaricherà e installerà automaticamente l'aggiornamento, dopo di che IDBac richiederà di riavviare.
- Fare clic sulla scheda Inizia con dati non elaborati e scegliere dal menu il tipo di dati da utilizzare con IDBac; continuare seguendo le istruzioni in-app.
- Durante l'impostazione della conversione e dell'elaborazione dei file di dati, immettere un nome descrittivo per l'esperimento nel caso richiesto (vedere la Figura4). Gli esperimenti verranno successivamente visualizzati in ordine alfabetico, pertanto una strategia utile consiste nell'iniziare i nomi degli esperimenti con un attributo di gruppo (ad esempio, "bacillus-trials_experiment-1"; "bacillus-trials_experiment-2").
Figura 4: passaggio di conversione e pre-elaborazione dei dati IDBac. IDBac converte gli spettri non elaborati nel formato mzML aperto e archivia mzML, elenchi di picco e informazioni di esempio in un database per ogni esperimento. Fare clic qui per visualizzare una versione più grande di questa figura.
7. Lavorare con esperimenti precedenti
- Dopo aver convertito i file e averli elaborazione con IDBac o in qualsiasi momento si desidera analizzare nuovamente un esperimento, passare alla pagina Utilizzo degli esperimenti precedenti e selezionare un esperimento con cui lavorare ( Figura5).
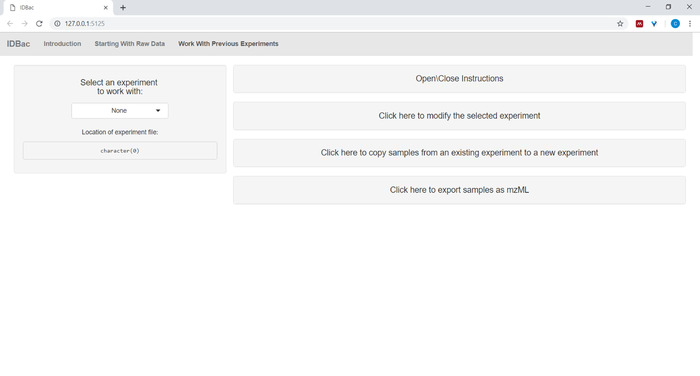
Figura 5: pagina "Lavorare con esperimenti precedenti". Utilizza la pagina "Lavora con esperimenti precedenti" di IDBac per selezionare un esperimento da analizzare o modificare. Fare clic qui per visualizzare una versione più grande di questa figura.
- (Facoltativo) Aggiungere informazioni sugli esempi utilizzando il menu Fare clic qui per modificare l'esperimentoselezionato. Immettere le informazioni nel foglio di calcolo compilato automaticamente e premere Salva (Figura 6). Questa opzione consente all'utente di colorare i dati durante le analisi.
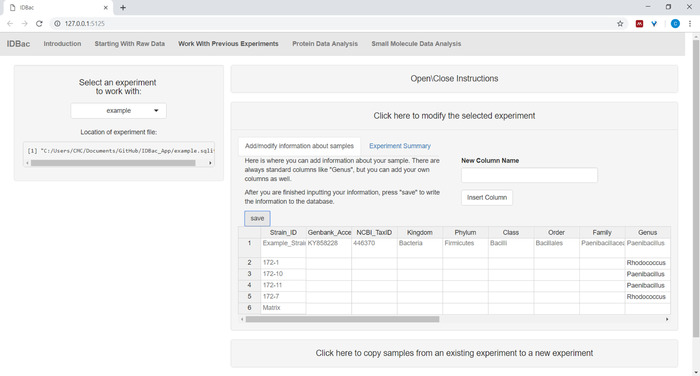
Figura 6: Immettere informazioni di esempio. All'interno della pagina "Utilizzo di esperimenti precedenti" gli utenti possono inserire informazioni su campioni come l'identità tassonomica, la posizione di raccolta, le condizioni di isolamento, ecc. Fare clic qui per visualizzare una versione più grande di questa figura.
- (Facoltativo) Trasferire tutti i campioni o un sottoinsieme di campioni in un esperimento nuovo o in un altro esperimento facendo clic su Trasferisci campioni da esperimenti precedenti a esperimenti nuovi/altri e seguendo le istruzioni fornite (Figura 7).
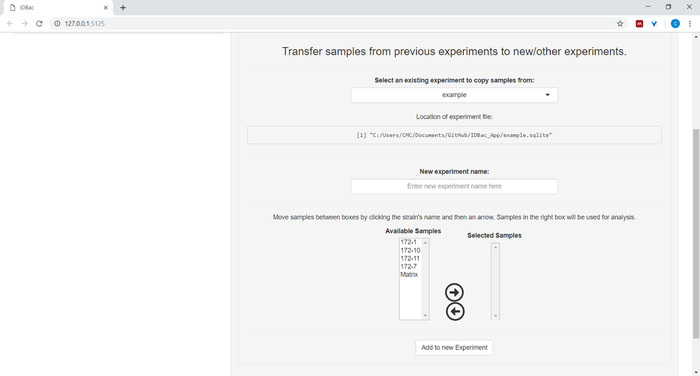
Figura 7: Trasferire i dati. La pagina "Utilizzo di esperimenti precedenti" contiene l'opzione per trasferire dati tra esperimenti esistenti e a nuovi esperimenti. Fare clic qui per visualizzare una versione più grande di questa figura.
- Quando si è pronti per iniziare l'analisi, assicurarsi che sia selezionato l'esperimento con cui lavorare. Selezionare Analisi dati proteine o Analisi dati molecola piccola.
8. Impostazione dell'analisi dei dati delle proteine e creazione di grafici a specchio
- Se si analizzano i dati delle proteine, passare innanzitutto alla pagina Analisi dei dati delle proteine. Scegliere le impostazioni di raccolta del picco e valutare gli spettri proteici dei campioni tramite i grafici a specchio visualizzati (Figura 8).
NOTA: Nei grafici a specchio, un picco rosso indica la presenza di tale picco solo nello spettro superiore, mentre i picchi blu rappresentano quelli che si verificano in entrambi gli spettri.
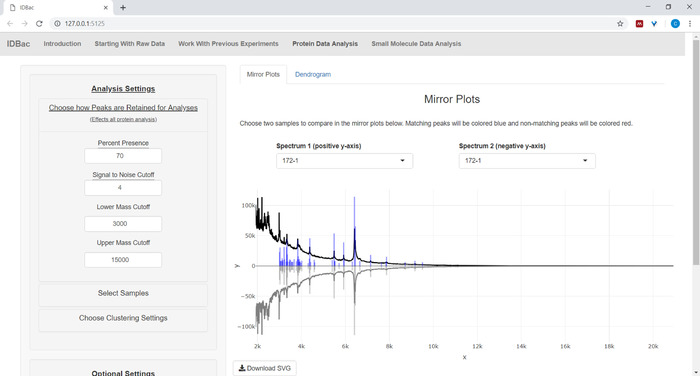
Figura 8: Scegliere la modalità di conservazione dei picchi per l'analisi. Dopo aver selezionato un esperimento da analizzare, visitando la pagina "Analisi dei dati proteici" e successivamente aprendo il menu "Scegli come vengono mantenuti i picchi per l'analisi" consente agli utenti di scegliere impostazioni come il rapporto segnale-rumore per mantenere i picchi. Il grafico speculare visualizzato (o dendrogramma) si aggiornerà automaticamente per riflettere le impostazioni scelte. Fare clic qui per visualizzare una versione più grande di questa figura.
- Regolare la percentuale di repliche in cui deve essere presente un picco per poter essere incluso per le analisi (ad esempio, se la soglia è impostata al 70% e si verifica un picco in almeno 7 repliche su 10, verrà incluso).
- Utilizzando le trame a specchio come guida visiva, regolare il segnale al taglio del rumore che mantiene i picchi più "genuini" e il minor rumore, notando che più repliche e un valore più alto di "presenza di picco percentuale" permetterà la selezione di un segnale inferiore al taglio del rumore.
- Specificare i tagli m/z inferiori e superiori, dettando l'intervallo di valori di massa all'interno di ogni spettro da utilizzare in ulteriori analisi da IDBac.
9. Clustering di campioni utilizzando i dati delle proteine
- Nella pagina Analisi dati proteine, selezionare la scheda Dendrogram. Ciò consente di raggruppare i campioni in un dendrogramma in base alle misure di distanza selezionate dall'utente e agli algoritmi di clustering.
- Fare clic su Seleziona campioni nel menu e seguire le istruzioni per selezionare i campioni da includere nelle analisi. Solo i campioni che contengono spettri proteici verranno visualizzati all'interno della casella Campioni disponibili (Figura9).
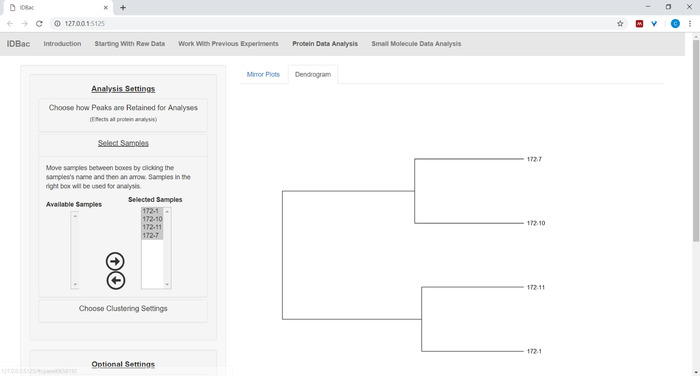
Figura 9: Selezionare i campioni dell'esperimento scelto da includere all'interno del dendrogramma visualizzato. Fare clic qui per visualizzare una versione più grande di questa figura.
- Utilizzare i valori predefiniti oppure, in Scegli impostazioni di clustering, selezionare gli algoritmi di distanza e clustering desiderati da applicare alla generazione del dendrogramma.
- Selezionare Presenza/Assenza come input. In alternativa, se si è certi delle altezze di picco dei campioni (ad esempio, dopo aver eseguito uno studio per valutare la variabilità dell'intensità di picco), selezionare Intensità come input.
NOTA: al momento della pubblicazione, IDBac offre flessibilità nelle impostazioni per il clustering, affidandosi agli utenti per scegliere le combinazioni appropriate. Se non ha familiarità con queste opzioni, si consiglia di accoppiare a: distanza "cosina" e clustering "media (UPGMA)"; o B: distanza "euclideo" e clustering "Ward.D2". - Per visualizzare i valori bootstrap nel dendrogramma, immettere un numero compreso tra 2 e 1000 in Bootstraps.
- Quando si segnalano i risultati, copiare il testo all'interno del paragrafo Suggerimenti per la segnalazione dell'analisi delle proteine. Fornisce le impostazioni definite dall'utente che hanno generato il dendrogramma specifico.
10. Personalizzazione del dendrogramma proteico
- Per iniziare a personalizzare il dendrogramma, aprire il menu Regola dendrogramma (Figura10).
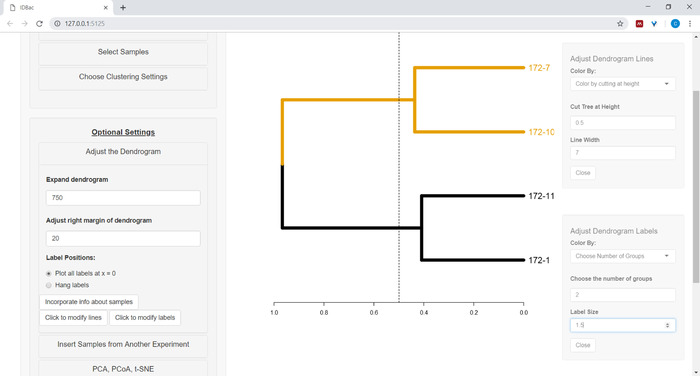
Figura 10: Regolare il dendrogramma. IDBac fornisce alcune opzioni per modificare l'aspetto del dendrogramma, questi possono essere trovati all'interno del menu "Regolare il dendrogramma". Ciò include la colorazione di rami ed etichette con k-mezzi, o "tagliando" il dendrogramma ad un'altezza fornita dall'utente. Fare clic qui per visualizzare una versione più grande di questa figura.
- Per colorare le linee e/o le etichette del dendrogramma selezionare il pulsante appropriato: fare clic per modificare le linee o Fare clic per modificare le etichette e selezionare le opzioni desiderate.
- Per stampare le informazioni dal foglio di calcolo accanto al dendrogramma (vedere il passaggio 7.2), selezionare il pulsante Incorpora informazioni sugli esempi. Si aprirà un pannello in cui una categoria (colonna nel foglio di calcolo) si autoposta in base ai valori immessi (Figura 11).
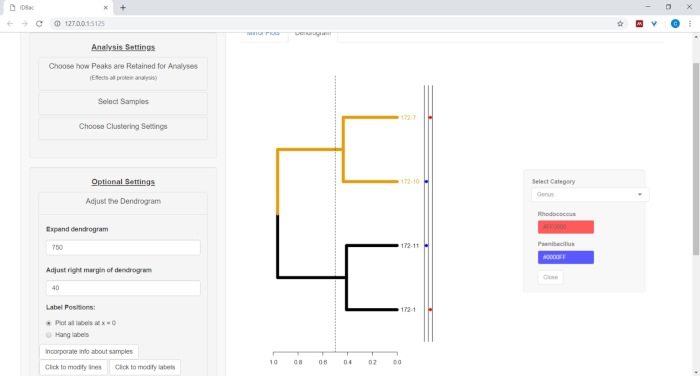
Figura 11: Incorporare informazioni sui campioni. All'interno del menu "Regola il Dendrogram" è l'opzione "Incorpora informazioni sui campioni". Selezionando questa opzione si consentiranno di tracciare le informazioni sui campioni accanto al dendrogramma. Le informazioni di esempio vengono immesse nella pagina "Utilizzo di esperimenti precedenti". Fare clic qui per visualizzare una versione più grande di questa figura.
11. Inserire campioni da un esperimento separato nel dendrogramma
- Per inserire campioni da un altro esperimento, seleziona il pulsante di menu Inserisci campioni da un altro esperimento. Seguire le istruzioni nel pannello appena aperto (Figura 12).
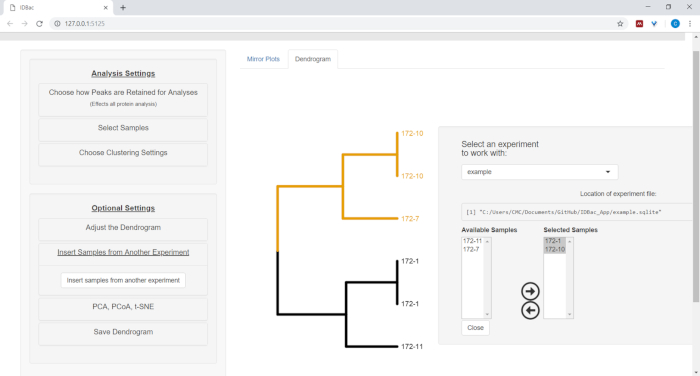
Figura 12: Inserire campioni dal menu Un altro esperimento. A volte è utile confrontare i campioni di un altro esperimento. Usa il menu "Inserisci campioni da un altro esperimento" per scegliere i campioni da includere all'interno del dendrogramma attualmente visualizzato. Fare clic qui per visualizzare una versione più grande di questa figura.
12. Analisi dei dati specializzati sui metaboliti e sulle reti di associazione dei metaboliti (MAN)
- Procedere alla pagina Metabolite Association Network (Small-Molecule). Questa pagina consente la visualizzazione dei dati mediante l'analisi dei componenti principali (PCA) e i MAN, che utilizzano reti bipartite per visualizzare la correlazione di piccoli valori di molecola m/z con campioni.
- Se è stato creato un dendrogramma proteico (sezione 9), verrà visualizzato anche in questa pagina. Fare clic e trascinare sul dendrogramma per evidenziare i campioni selezionati di interesse da analizzare. Se non vengono evidenziati campioni o non viene creato alcun dendrogramma proteico, verrà visualizzato un MAN di un sottoinsieme casuale o tutti i campioni rispettosamente (Figura 13).
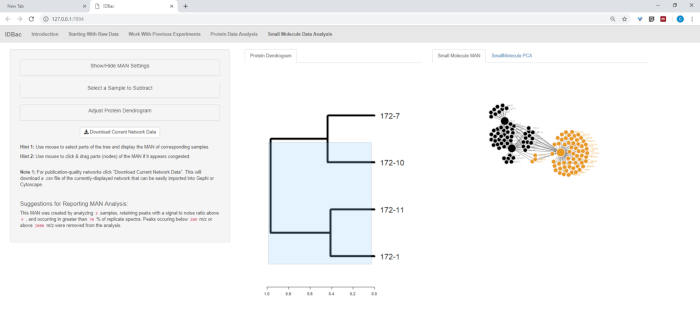
Figura 13: Pagina Analisi dei dati delle piccole molecole". Se un dendrogramma è stato creato da spettri proteici, verrà visualizzato all'interno della pagina "Small Molecule Data Analysis". Questa pagina mostrerà anche Metabolite Associate Networks (MAN) e Principle Components Analysis (PCA) per piccoli dati di molecola. Fare clic qui per visualizzare una versione più grande di questa figura.
- Per sottrarre una matrice/supporto vuoto nell'UOMO, aprire il menu Selezionare un campione da sottrarre e scegliere il campione appropriato da utilizzare come vuoto.
- Aprire il menu Mostra/Nascondi impostazioni MAN per selezionare i valori desiderati per la percentuale di presenza massima in repliche, segnale al rumore e tagli di massa superiori e inferiori di massa, come è stato fatto per gli spettri proteici nella Sezione 9. Utilizzare i piccoli grafici a specchio molecolare per guidare la selezione di queste impostazioni.
- Selezionare "Scarica dati di rete correnti" per salvare i dati dell'UOMO attualmente visualizzato. Questi dati possono essere utilizzati in software di analisi di rete diverso da IDBac.
- Per i risultati dei report, copiare il testo all'interno del paragrafo Suggerimenti per la creazione di report di analisi MAN. In questo modo vengono fornite le impostazioni definite dall'utente utilizzate per generare l'oggetto MAN creato.
13. Condivisione dei dati
- Ogni "esperimento" IDBac viene salvato come un singolo database SQLite. Contiene gli spettri non elaborati mzML convertiti, i picchi rilevati e tutte le informazioni di input dell'utente sui campioni. Pertanto, per condividere un esperimento IDBac è sufficiente condividere il file SQlite con lo stesso nome dell'esperimento (la posizione del file viene visualizzata nella pagina Utilizzo di esperimenti precedenti).
Risultati
Abbiamo analizzato sei ceppi di Micromonospora chokoriensis e due ceppi di Bacillus subtilis, precedentemente caratterizzati6, utilizzando i dati disponibili presso DOI: 10.5281/zenodo.2574096. Seguendo le istruzioni nella scheda A partire da dati non elaborati, è stata selezionata l'opzione Fare clic qui per convertire i file Bruker e sono seguite le istruzioni fornite da IDBac per ogni set di dati (Figura 14).
Dopo aver completato la conversione automatizzata e i passaggi di pre-elaborazione/picco, abbiamo proceduto alla creazione di un nuovo esperimento IDBac combinato trasferendo i campioni dei due esperimenti in un unico esperimento contenente sia Bacillus che Campioni di micromonosopora (Figura 15). L'analisi risultante ha comportato il confronto degli spettri proteici utilizzando grafici a specchio, come illustrato nella figura 16,che è stato utile per valutare la qualità degli spettri e regolare le impostazioni di selezione del picco. Figura 17 mostra uno screenshot dei risultati di clustering di proteine con le impostazioni predefinite selezionate. Il dendrogramma è stato colorato regolando la soglia sul grafico (appare come una linea tratteggiata). Da notare la netta separazione tra generi, con M. chokoriensis e B. subtilis isola il clustering separatamente.
Figura 18, Figura 19e Figura 20 evidenziare la possibilità di generare NOMI DI codice AD delle regioni selezionate dall'utente facendo clic e trascinando traslochi tra le proteine dendrogramma. Con questo siamo stati in grado di creare rapidamente MAN per confrontare solo i ceppi B. subtilis (Figura 18), solo i ceppi M. chokoriensis (Figura 19), e tutti i ceppi contemporaneamente (Figura 20). La funzione principale di queste reti è quella di fornire ai ricercatori un'ampia panoramica del grado di sovrapposizione dei metaboliti specializzati tra i batteri. Con questi dati in mano, i ricercatori hanno ora la capacità di prendere decisioni informate solo da una piccola quantità di materiale raschiato da una colonia batterica.
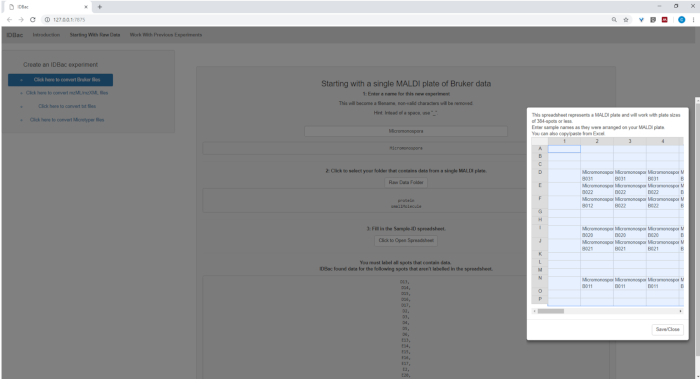
Figura 14: Elaborazione spectra. Gli spettri autoFlex di Bruker scaricati sono stati convertiti ed elaborati utilizzando IDBac. Fare clic qui per visualizzare una versione più grande di questa figura.
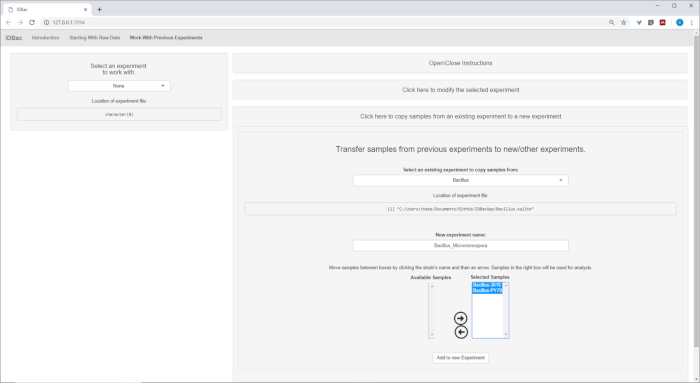
Figura 15: Esperimento IDBac combinato. Poiché gli spettri Micromonospora e Bacillus sono stati raccolti su diverse placche bersaglio MALDI, i due esperimenti sono stati successivamente combinati in un unico esperimento, "Bacillus_Micromonsopora". Questo è stato fatto all'interno della scheda "Lavorare con esperimenti precedenti", seguendo le istruzioni all'interno del menu "Trasferire campioni da esperimenti precedenti a nuovi/altri esperimenti". Fare clic qui per visualizzare una versione più grande di questa figura.
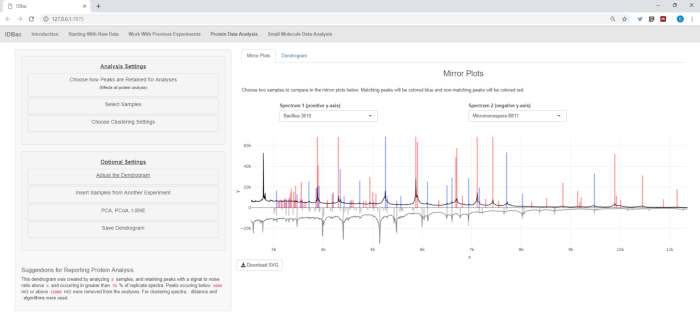
Figura 16: Confronto. Micromonspora e Bacillus spettri sono stati confrontati utilizzando i grafici a specchio all'interno della pagina "Protein Data Analysis". In definitiva, sono state scelte le impostazioni di picco predefinite. Fare clic qui per visualizzare una versione più grande di questa figura.
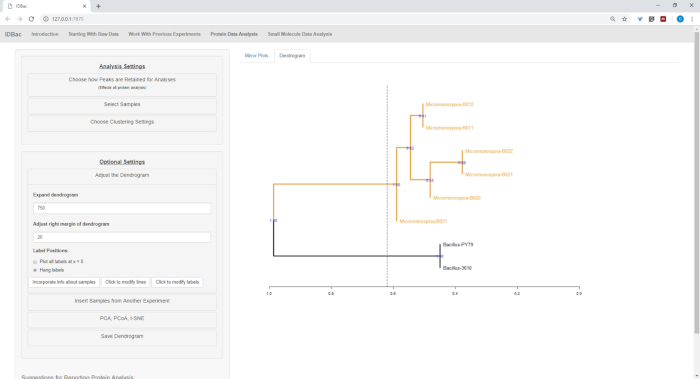
Figura 17: Clustering gerarchico. Clustering gerarchico, utilizzando le impostazioni predefinite, correttamente raggruppato Bacillus e Micromonospora isola. Il dendrogramma è stato colorato "tagliando" il dendrogramma ad un'altezza arbitraria (visualizzato come una linea tratteggiata) e 100 bootstrap utilizzati per mostrare la fiducia nella diramazione. Fare clic qui per visualizzare una versione più grande di questa figura.
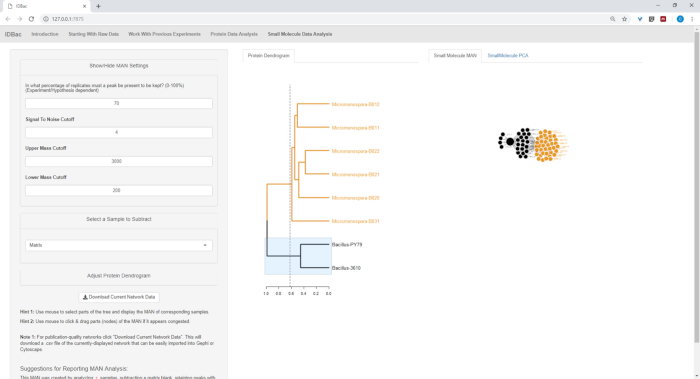
Figura 18: MAN creato selezionando i ceppi Bacillus sp. dal dendrogramma proteico ha mostrato la produzione differenziale di metaboliti specializzati. Fare clic qui per visualizzare una versione più grande di questa figura.
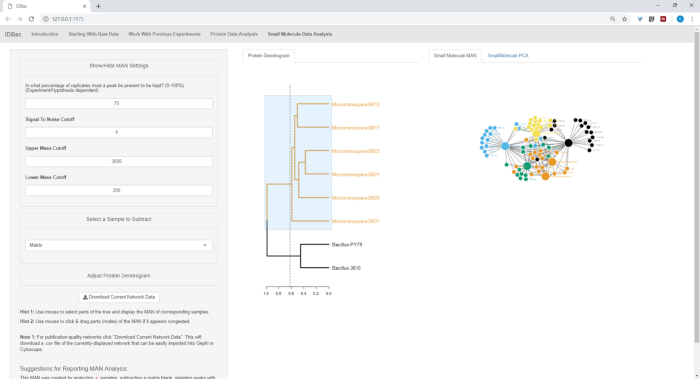
Figura 19: MAN creato selezionando i sei ceppi Micromonospora sp. dal dendrogramma proteico hanno mostrato la produzione differenziale di metaboliti specializzati. Fare clic qui per visualizzare una versione più grande di questa figura.
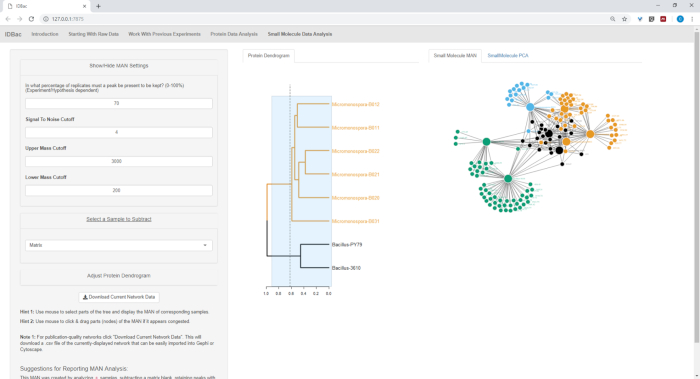
Figura 20: Ceppi MAN di Bacillus sp. e Micromonospora sp. che mostrano una produzione differenziale di metaboliti specializzati. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il protocollo IDBac descrive la proteina batterica e l'acquisizione e l'analisi di dati sui metaboliti specializzati fino a 384 isolati batterici in 4 h da un singolo ricercatore. Con IDBac non è necessario estrarre il DNA dagli isolati batterici o generare estratti di metaboliti specializzati dai brodi di fermentazione liquida e analizzarli utilizzando metodi cromatografici. Invece, i dati sulle proteine e sui metaboliti specializzati vengono raccolti semplicemente diffondendo materiale dalle colonie batteriche direttamente su una piastra bersaglio MALDI. Questo riduce notevolmente il tempo e i costi associati a tecniche alternative come il sequenziamento genico 16S rRNA e LCMS9.
È importante aggiungere una matrice vuota e punti di calibrazione alla piastra MALDI, e si consiglia di utilizzare un numero appropriato di repliche per garantire la riproducibilità e la fiducia statistica. Il numero di repliche dipenderà dall'esperimento. Ad esempio, se un utente intende differenziare migliaia di colonie da una raccolta di piastre di diversità ambientale, potrebbero essere necessarie meno repliche (il nostro laboratorio raccoglie tre repliche tecniche per colonia). In alternativa, se un utente desidera creare un database personalizzato di ceppi da specifici taxa batterici per determinare rapidamente le classificazioni sottospecie di isolati sconosciuti, allora più repliche sono appropriate (il nostro laboratorio raccoglie otto repliche biologiche per ceppo).
IDBac è uno strumento per differenziare rapidamente gli isolati batterici altamente correlati sulla base di informazioni tassonomiche putative e produzione di metaboliti specializzati. Può integrare o servire come precursore di metodi ortogonali come analisi genetiche approfondite, studi che coinvolgono la produzione e la funzione dei metaboliti, o la caratterizzazione della struttura di metaboliti specializzati mediante spettroscopia a risonanza magnetica nucleare e/o LC-MS/MS.
La produzione specializzata di metaboliti (MAN IDBac) è altamente suscettibile alle condizioni di crescita batterica, in particolare utilizzando diversi supporti, che è una potenziale limitazione del metodo. Tuttavia questi tratti possono essere sfruttati dall'utente, come IDBac può facilmente generare MAN mostrando le differenze nella produzione di metaboliti specializzati in una varietà di condizioni di crescita. È importante notare che, sebbene le impronte digitali dei metaboliti specializzati possono variare a seconda della condizione di crescita, abbiamo dimostrato in precedenza che le impronte digitali delle proteine rimangono relativamente stabili in queste variabili (vedi Clark et al.6). Quando si tratta di piastre di diversità ambientale, si consiglia di purificare gli isolati batterici prima dell'analisi al fine di ridurre i possibili contributi dal cross-talk batterico vicino.
Infine, la mancanza di un database pubblico ricercabile di impronte digitali delle proteine MS è una grave carenza nell'uso di questo metodo per classificare i batteri ambientali sconosciuti. Abbiamo creato IDBac con questo in mente, e incluso la conversione automatica dei dati in un formato open source accettato dalla comunità (mzML)10,11,12 e progettato il software per consentire la ricerca, la condivisione e la creazione di database personalizzati. Stiamo creando una grande banca dati pubblica (>10,000 ceppi completamente caratterizzati), che permetterà la classificazione di alcuni isolati a livello di specie, compresi i collegamenti ai numeri di adesione genBank quando disponibili.
IDBac è open source e il codice è disponibile per chiunque per personalizzare le proprie esigenze di analisi e visualizzazione dei dati. Consigliamo agli utenti di consultare un vasto corpo di letteratura (Sauer et al.7, Silva et al.5) per aiutare a sostenere e progettare i loro obiettivi sperimentali. Ospitiamo un forum di discussione all'indirizzo: https://groups.google.com/forum/#!forum/idbac e un mezzo per segnalare problemi con il software all'indirizzo: https://github.com/chasemc/IDBacApp/issues.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal National Institute of General Medical Sciences Grant R01 GM125943, National Geographic Grant CP-044R-17; Fondo di ricerca islandese sovvenzione 152336-051; e l'Università dell'Illinois presso i fondi per le startup di Chicago. Inoltre, ringraziamo i seguenti collaboratori: Dr. Amanda Bulman per l'assistenza con i parametri di acquisizione delle proteine MALDI-TOF MS; Dr. Terry Moore e Dr. Atul Jain per ricrystallizzare la matrice di acido alfa-ciano-4-idrossicininnamiche (CHCA).
Materiali
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
Riferimenti
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon