Method Article
Usando o pipeline de código aberto MALDI TOF-MS IDBac para análise de proteína microbiana e dados metabólito especializados
Neste Artigo
Resumo
IDBac é um pipeline de Bioinformática baseado em espectrometria de massas de código aberto que integra dados de proteínas intactas e espectros metabólito especializados, coletados em material celular raspado de colônias bacterianas. O pipeline permite que os pesquisadores organizem rapidamente centenas a milhares de colônias bacterianas em grupos taxonômicos putativos e diferenciem-se ainda mais com base na produção de metabólito especializado.
Resumo
A fim de visualizar a relação entre a filogenia bacteriana e a produção de metabólito especializado de colônias bacterianas que crescem em ágar nutriente, desenvolvemos IDBac — uma dessorção/ionização a laser assistida por matriz de baixo custo e alta taxa de transferência pipeline de Bioinformática de espectrometria de massas em tempo de voo (MALDI-TOF MS). IDBac software é projetado para não-especialistas, está disponível gratuitamente, e capaz de analisar alguns a milhares de colônias bacterianas. Aqui, apresentamos procedimentos para a preparação de colônias bacterianas para análise de MALDI-TOF MS, operação de instrumento MS e processamento e visualização de dados em IDBac. Em particular, Instruímos os usuários como agrupar bactérias em dendrogramas com base em impressões digitais de proteína MS e criar interativamente redes de associação de metabolito (MANs) de dados de metabólito especializados.
Introdução
Uma grande barreira para pesquisadores que estudam a função bacteriana é a capacidade de avaliar rapidamente e simultaneamente a identidade taxonômica de um microrganismo e sua capacidade de produzir metabólitos especializados. Isso impediu avanços significativos na compreensão da relação entre a filogenia bacteriana e a produção de metabólito especializado na maioria das bactérias isoladas do ambiente. Embora os métodos baseados em MS que usam impressões digitais de proteínas para agrupar e identificar bactérias sejam bem descritos1,2,3,4, esses estudos geralmente foram realizados em pequenos grupos de isolados, de forma específica da espécie. É importante ressaltar que as informações sobre a produção de metabólito especializado, um dos principais condutores de função microbiana no ambiente, permaneceram não incorporadas nesses estudos. Silva et al.5 forneceram recentemente uma história abrangente detalhando o baixo uso de MALDI-TOF MS para analisar metabólitos especializados e a escassez de softwares para aliviar os gargalos atuais de Bioinformática. A fim de abordar essas deficiências, criamos IDBac, um pipeline de Bioinformática que integra os modos linear e reflectron de MALDI-TOF MS6. Isto permite que os usuários visualizem e diferenciem rapidamente os isolados bacterianos baseados na proteína e nas impressões digitais especializadas do MS do metabolito, respectivamente.
IDBac é rentável, alta taxa de transferência, e projetado para o usuário leigo. Está disponível livremente (chasemc.github.io/IDBac), e exige somente o acesso a um espectrómetro maciço de MALDI-TOF (o modo do reflectron será exigido para a análise especializada do metabolito). A preparação da amostra depende do método simples de "transferência direta estendida"7,8 e os dados são coletados com aquisições consecutivas lineares e de reflectron em um único ponto do MALDI-alvo. Com IDBac, é possível analisar a filogenia putativa e a produção de metabólito especializado de centenas de colônias em menos de quatro horas, incluindo preparação de amostras, aquisição de dados e visualização de dados. Isto apresenta uma vantagem significativa do tempo e do custo sobre métodos tradicionais de identificar as bactérias (tais como o sequenciamento do gene), e analisando a saída metabólica (cromatografia líquida-espectrometria maciça [LCMS] e métodos cromatográficos similares).
Usando dados obtidos na análise de modo linear, IDBac emprega agrupamento hierárquico para representar a parentalidade dos espectros proteicos. Uma vez que os espectros representam principalmente proteínas ribossômicas ionizadas, eles fornecem uma representação da diversidade filogenética presente em uma amostra. Além disso, o idbac incorpora dados de modo reflectron para exibir as impressões digitais especializadas do metabolito como metabolito Association Networks (Mans). Mans são redes bipartido que permitem a fácil visualização da produção de metabólito compartilhada e única entre isolados bacterianos. A plataforma IDBac permite que os pesquisadores analisem os dados de proteínas e metabólito especializados em tandem, mas também individualmente, se apenas um tipo de dados for adquirido. Importante, IDBac processa dados brutos de instrumentos de Bruker e de Xiamen, assim como o txt, a aba, o CSV, o mzXML, e o mzML. Isso elimina a necessidade de conversão manual e formatação de conjuntos de dados, e reduz significativamente o risco de erro do usuário ou manipulação incorreta de dados MS.
Protocolo
1. preparação da matriz MALDI
- Prepare 10 mg/ml de MALDI-grade, e/ou ácido α-cyano-4-Hydroxycinnamic recristalizado (CHCA) em solventes da MS-classe: 50% acetonitrila (ACN), 47,5% água (H2O), 2,5% ácido trifluoroacético (TFA). Exemplo: 100 μL de solução = 50 μL de ACN + 47,5 μL H2O + 2,5 ΜL de TFA + 1 mg de CHCA
- Prepare pelo menos 1 μL de solução matricial por ponto de placa MALDI e vórtice ou proceda até em solução (aproximadamente 5 min de sonicação ou sem sólidos visíveis).
Cuidado: o TFA é um ácido forte que deve ser manuseado em uma capa de fumaça química enquanto usa equipamento de proteção pessoal adequado, pois pode danificar a pele, os olhos e as vias aéreas com contato ou inalação.
Nota: CHCA é higroscópico e sensível à luz e deve ser armazenado em frascos de âmbar em um dessecador. Existem muitas opções de matriz MALDI disponíveis. CHCA é mais comum para o perfil de proteínas de bactérias, mas também funciona para análise de metabolito especializado. A seleção de matrizes depende das necessidades individuais do usuário/experimento.
- Prepare pelo menos 1 μL de solução matricial por ponto de placa MALDI e vórtice ou proceda até em solução (aproximadamente 5 min de sonicação ou sem sólidos visíveis).
2. preparação de placas alvo MALDI
Nota: Veja Sauer et al.7, para mais detalhes.
- Enxague a placa MALDI com metanol (HPLC-grade ou superior) e seque com lenços de papel macios. Não use escovas abrasivas ao limpar as placas alvo, pois isso pode danificar permanentemente a superfície da placa alvo.
- Atribua pontos de calibração de proteínas e metabólito especializados. Organize pontos de calibração uniformemente em toda a população da amostra, para ter em conta a MALDI-Plate-irregularidades e a deriva do instrumento ao longo do tempo. Atribua um número apropriado de pontos de mídia/matriz-em branco para o estudo; esses pontos conterá apenas mídia e matriz, ou apenas matriz.
- Usando um palito de dente estéril, transfira uma pequena porção de uma colônia bacteriana para o local apropriado na placa MALDI. Espalhe a colônia bacteriana uniformemente sobre o local. O local deve aparecer o mais plano possível.
Nota: será mais fácil aplainar colônias bacterianas que são mais Mucoid/amorfos. Para colônias mais rígidas/sólidas, Evite deixar aglomerados visíveis de massa celular no ponto MALDI (Figura 1).
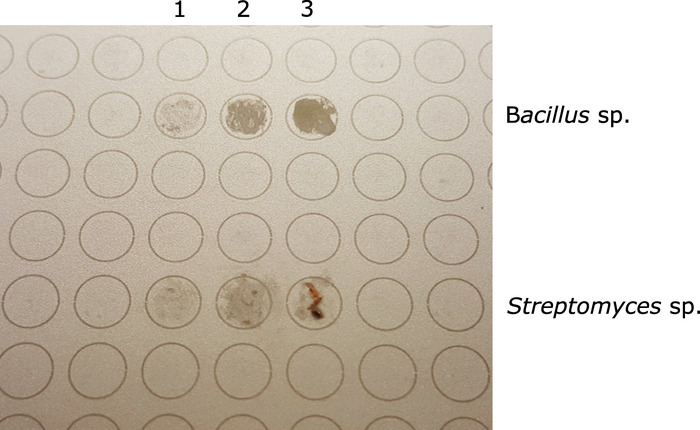
Figura 1: placa MALDI-alvo mostrando dois isolados diferentes antes da adição de ácido fórmico e matriz de MALDI (Top 3 spots- Bacillus SP.; fundo 3 manchas- Streptomyces SP.). Para ambos, a coluna 3 representa o excesso de amostra; coluna 2 representa a quantidade apropriada de amostra; coluna 1 representa amostra insuficiente para análise de MALDI. Por favor clique aqui para ver uma versão maior desta figura.
- Prepare uma matriz/controle de mídia usando um palito estéril para transferir uma quantidade mínima de agar/mídia para o local apropriado (s) na placa MALDI.
- Sobrepor 1 μL de ácido fórmico de grau de espectrometria de massa de 70% em cada ponto da amostra, incluindo os pontos de controle matricial. Deixe o ácido secar completamente em uma capa de fumaça química (aproximadamente 5 min).
PRECAUÇÃO: o ácido fórmico é um produto químico cáustico e deve ser manuseado em capas de fumos químicos. Pode danificar as vias respiratórias se inalado. - Adicione 1 μL da solução de matriz MALDI preparada para cada ponto da amostra, bem como para os pontos de controle de matriz/mídia. Permita a solução da matriz ao ar secar completamente (aproximadamente 5 minutos).
Nota: é possível armazenar a placa em um dessecador, no escuro, até que possa ser analisado em um espectrómetro de massa MALDI-TOF. Os tempos de armazenamento permitidos podem variar dependendo da estabilidade da amostra. - Adicione 0,5 – 1,0 μL de calibrante aos pontos de calibração atribuídos, seguidos de 1 μL de solução matricial MALDI. Pipeta a solução resultante para cima e para baixo para misturar. Permitir que todos os pontos de ar secar completamente antes da introdução no Espectrómetro de massa MALDI-TOF.
Nota: a proteína e os calibrantes metabólito especializados devem ser adicionados dentro de 30 min da análise de MALDI, pois ambos são suscetíveis à degradação.
3. aquisição de dados
Nota: os parâmetros gerais para aquisição de dados estão listados na tabela 1.
| Parâmetro | Proteína | Metabólito especializado |
| Início maciço (da) | 1920 | 60 |
| Massa final (da) | 21000 | 2700 |
| Deflexão maciça (da) | 1900 | 50 |
| Tiros | 500 | 1000 |
| Frequência (Hz) | 2000 | 2000 |
| Tamanho do laser | Grande | Médio |
| MaxStdDev (ppm) | 300 | 30 |
Tabela 1.
- Seguindo os protocolos específicos ao instrumento que está sendo usado, ajuste acima as calibrações proteínas e especializadas do metabolito.
- Teste alguns pontos de destino separados para determinar a potência ideal do laser e o ganho do detector para usar ao adquirir espectros (isso irá variar dia-a-dia e por instrumento).
Nota: a figura 2a e a Figura 3a mostram espectros ideais, enquanto a Figura 2D e a Figura 3D são exemplos de espectros de baixa qualidade.
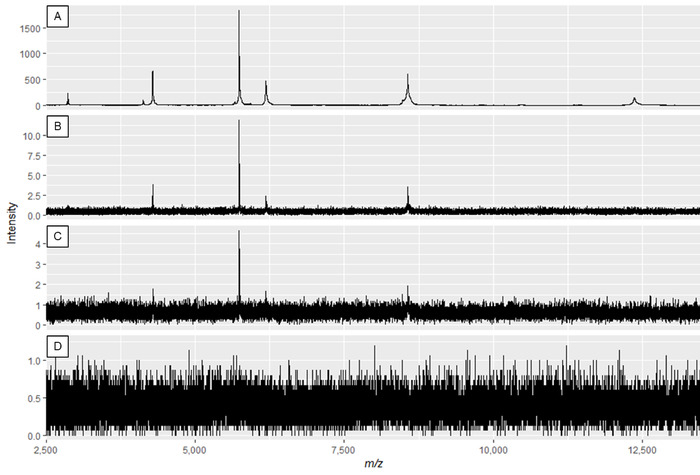
Figura 2: Exemplo de espectros proteicos que exibem o efeito de modificar a potência do laser e o ganho do detector. A qualidade dos espectros é melhor no painel A, e diminui até Aqualidade insuficiente dos espectros nos painéis C e D. Embora o espectro no painel B possa resultar em picos utilizáveis, o painel a exibe os dados ideais. Por favor clique aqui para ver uma versão maior desta figura.
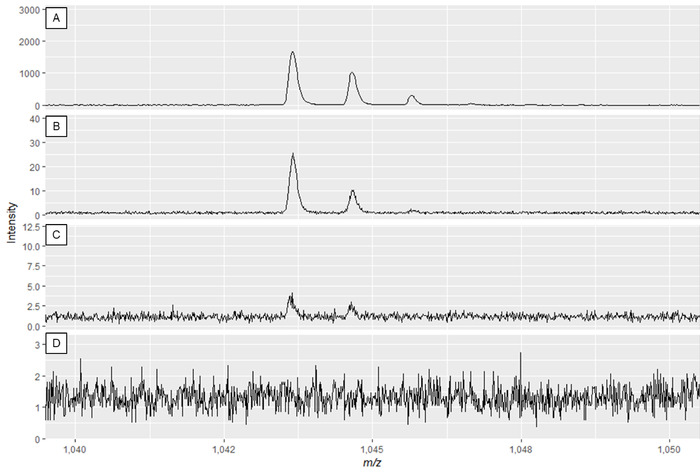
Figura 3: Espectros especializados do metabolito do exemplo que indicam o efeito de modificar o poder do laser e o ganho do detector. A qualidade dos espectros é melhor no painel A e diminui até A qualidade insuficiente dos espectros nos painéis C e D. Embora o espectro no painel B possa resultar em picos utilizáveis, o painel a exibe os dados ideais. Por favor clique aqui para ver uma versão maior desta figura.
- Adquira espectros, salvando espectros proteicos em uma pasta e espectros metabólito especializados em uma segunda pasta separada.
4. limpeza da placa alvo MALDI (adaptada de Sauer et al.7)
- Retire a placa alvo MALDI do seu suporte e enxague com acetona.
- Lave com um sabão líquido não abrasivo para remover proteínas de traço e lipídios, e lenços de papel macios/escova de dentes de cerdas macias.
- Enxágüe com água de-ionizada por aproximadamente 2 min para remover completamente o sabão.
- SONICATE a placa alvo em água (HPLC grau ou superior) para 5 min.
- Enxague a placa alvo com água (grau de HPLC ou superior).
- Enxague a placa alvo com metanol (grau de HPLC ou superior).
5. instalando o software IDBac
- Baixe o software IDBac.
Nota: os backups permanentes e com controle de versão também estão disponíveis para download (consulte a tabela de materiais). - Clique duas vezes no download "Install_IDBac. exe" para iniciar o instalador e siga as instruções na tela.
6. começando com dados brutos
Nota: explicações detalhadas e instruções de cada etapa de processamento de dados são incorporadas no IDBac, no entanto, as principais análises e entradas interativas são descritas abaixo.
- Clique duas vezes no atalho da área de trabalho IDBac para iniciar o IDBac. IDBac será aberto na guia introdução por padrão.
- Use o botão verificar atualizações para garantir que a versão mais atual do idbac esteja sendo usada (requer acesso à Internet). Se uma versão mais recente estiver disponível, o IDBac fará o download e instalará automaticamente a atualização, após o qual o IDBac solicitará a reiniciada.
- Clique na guia iniciando com dados brutos e escolha no menu o tipo de dados a serem usados com idbac; continuar seguindo as instruções no aplicativo.
- Ao configurar a conversão e o processamento de arquivos de dados, insira um nome descritivo para o experimento onde solicitado (consulte a Figura 4). Posteriormente, as experiências serão exibidas em ordem alfabética, portanto, uma estratégia útil é iniciar os nomes dos experimentos com um atributo de grupo (por exemplo, "Bacillus-trials_experiment-1"; "Bacillus-trials_experiment-2").
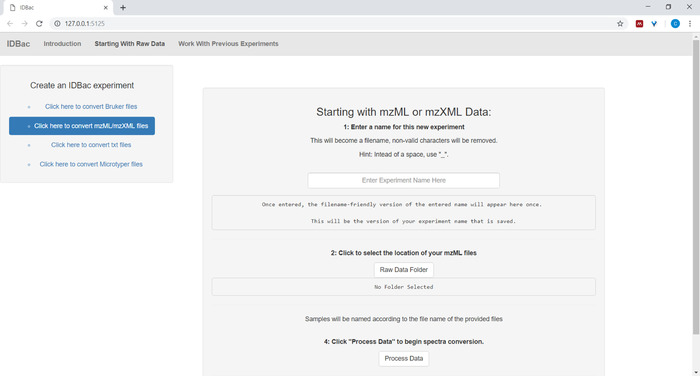
Figura 4: conversão de dados IDBac e etapa de pré-processamento. O IDBac converte espectros brutos no formato mzML aberto e armazena mzML, listas de pico e informações de amostra em um banco de dados para cada experimento. Por favor clique aqui para ver uma versão maior desta figura.
7. trabalhe com experimentos anteriores
- Depois de converter arquivos e processá-los com IDBac, ou a qualquer momento um deseja reanalisar uma experiência, navegue até a página trabalhar com experimentos anteriores e Selecione um experimento para trabalhar (Figura 5).
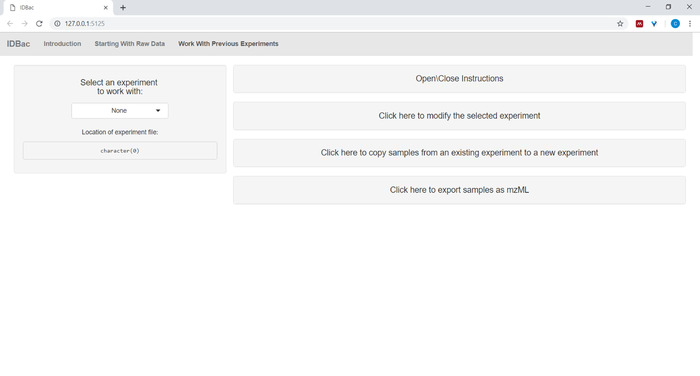
Figura 5: página "trabalhar com experimentos anteriores". Use a página "trabalhar com experimentos anteriores" do IDBac para selecionar um experimento para analisar ou modificar. Por favor clique aqui para ver uma versão maior desta figura.
- Opcional Adicione informações sobre amostras usando o menu clique aqui para modificar o experimento selecionado. Insira as informações na planilha preenchida automaticamente e pressione Save (Figura 6). Esta opção permite ao usuário dados de código de cores durante as análises.
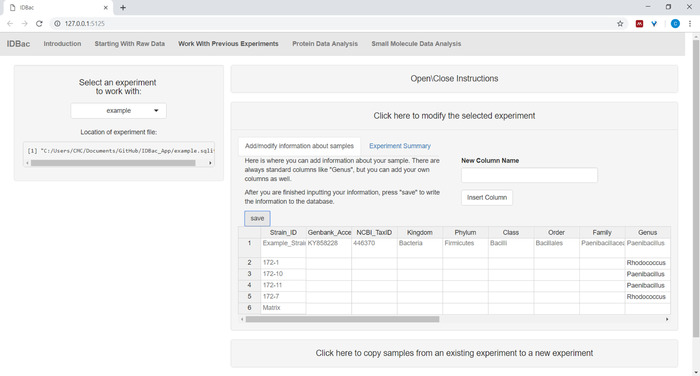
Figura 6: informações de exemplo de entrada. Dentro da página "trabalhar com experimentos anteriores", os usuários podem inserir informações sobre amostras como identidade taxonômica, localização de coleta, condições de isolamento, etc. clique aqui para ver uma versão maior desta figura.
- Opcional Transfira todos ou um subconjunto de amostras para um experimento novo ou outro clicando em transferir amostras de experimentos anteriores para novos/outros experimentos e seguindo as instruções fornecidas (Figura 7).
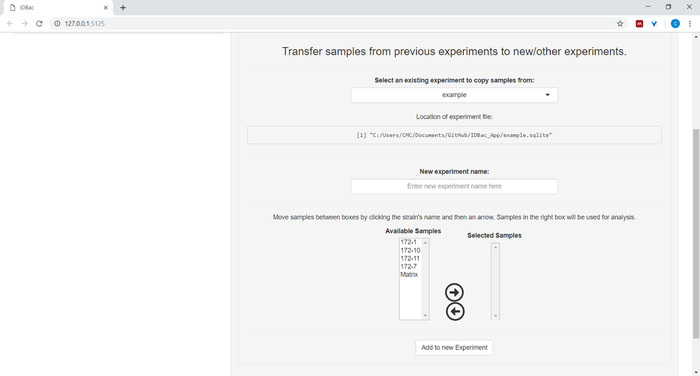
Figura 7: transferir dados. A página "trabalhar com experimentos anteriores" contém a opção de transferir dados entre experimentos existentes e novos experimentos. Por favor clique aqui para ver uma versão maior desta figura.
- Quando estiver pronto para começar a análise, assegure-se de que o experimento funcione com está selecionado. Selecione análise de dados de proteínas ou análise de dados de pequenas moléculas.
8. Configurando a análise de dados proteicos e criando parcelas espelhadas
- Se analisar dados de proteínas, primeiro navegue para a página de análise de dados de proteínas . Escolha as configurações de pico-picking e avalie os espectros proteicos das amostras através das parcelas espelhadas exibidas (Figura 8).
Nota: nas parcelas do espelho, um pico vermelho significa a presença desse pico somente no espectro superior, quando os picos azuis representarem aqueles que ocorrem em ambos os espectros.
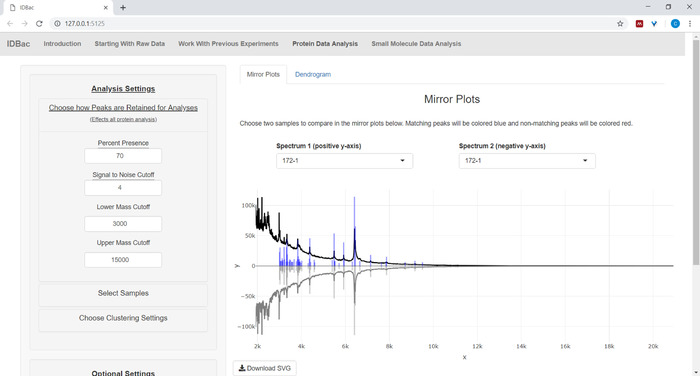
Figura 8: escolha como os picos são retidos para análise. Depois de selecionar um experimento para analisar, visitando a página "análise de dados de proteínas" e, subsequentemente, abrindo o menu "escolher como os picos são retidos para análise" permite que os usuários escolham configurações como a relação sinal-ruído para reter picos. A plotagem espelhada exibida (ou dendrograma) será atualizada automaticamente para refletir as configurações escolhidas. Por favor clique aqui para ver uma versão maior desta figura.
- Ajustar o percentual de repetições em que um pico deve estar presente para que ele seja incluído para análises (por exemplo, se o limiar é definido para 70% e um pico ocorre em pelo menos 7 de 10 repetições, ele será incluído).
- Usando as parcelas espelhadas como orientação Visual, ajuste o sinal para o corte de ruído que retém os picos mais "genuínos" e o mínimo de ruído, observando que mais repetições e um valor maior "presença de pico percentual" permitirá a seleção de um sinal mais baixo para o corte de ruído.
- Especifique os pontos de corte m/z inferiores e superiores, ditendo o intervalo de valores de massa dentro de cada espectro a ser usado em análises adicionais por idbac.
9. agrupamento de amostras utilizando dados proteicos
- Dentro da página de análise de dados de proteínas , selecione a guia dendrogram . Isso permite agrupar amostras em um dendrograma de acordo com as medidas de distância selecionadas pelo usuário e os algoritmos de clustering.
- Clique em selecionar amostras no menu e siga as instruções para selecionar amostras a serem incluídos nas análises. Somente amostras que contenham espectros proteicos serão exibidas dentro da caixa amostras disponíveis (Figura 9).
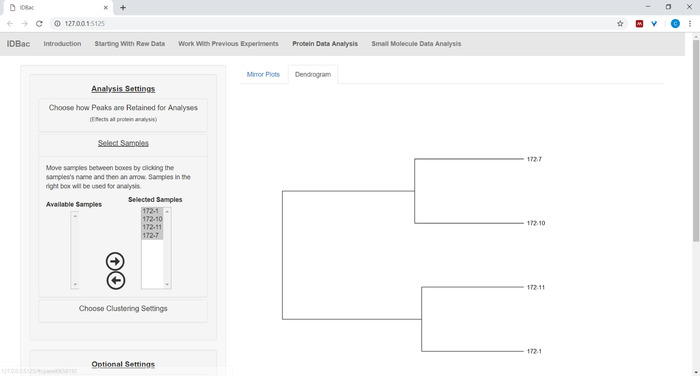
Figura 9: selecione amostras do experimento escolhido para incluir dentro do dendrograma exibido. Por favor clique aqui para ver uma versão maior desta figura.
- Use os valores padrão ou, em escolher configurações de clustering, selecione os algoritmos de distância e clustering desejados a serem aplicados à geração do dendrograma.
- Selecione presença/ausência como entrada. Alternativamente, se confiante sobre as alturas máximas das amostras (por exemplo, depois de realizar um estudo para avaliar a variabilidade da intensidade de pico), selecione intensidades como entrada.
Nota: no momento da publicação, o IDBac fornece flexibilidade nas configurações de clustering, confiando nos usuários para escolher as combinações apropriadas. Se não estiver familiarizado com essas opções, é sugerido para emparelhar a: distância "cosseno" e "média (UPGMA)" clustering; ou B: distância "euclidiana" e "Ward. D2" clustering. - Para exibir valores de Bootstrap no dendrogram, insira um número entre 2 e 1000 em bootstraps.
- Ao relatar resultados, copie o texto dentro das sugestões de relatório de análise de proteínas parágrafo. Isso fornece as configurações definidas pelo usuário que gerou o dendrograma específico.
10. Personalizando o dendrograma da proteína
- Para começar a personalizar o dendrograma, abra o menu ajustar o dendrograma (Figura 10).
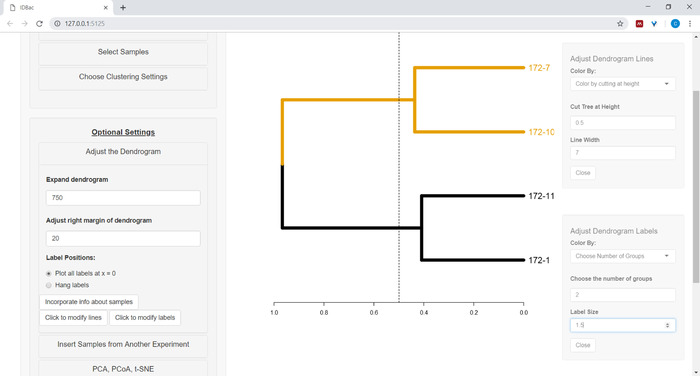
Figura 10: Ajuste o dendrograma. IDBac fornece algumas opções para modificar como o dendrograma parece, estes podem ser encontrados dentro do menu "ajustar o dendrograma". Isso inclui galhos e rótulos de coloração por k-Means, ou "cortando" o dendrograma em uma altura fornecida pelo usuário. Por favor clique aqui para ver uma versão maior desta figura.
- Para colorir as linhas e/ou rótulos do dendrograma, selecione o botão apropriado: clique para modificar linhas ou clique para modificar rótulos e selecione as opções desejadas.
- Para plotar informações da planilha ao lado do dendrograma (consulte a etapa 7,2), selecione o botão incorporar informações sobre amostras. Isso abrirá um painel onde uma categoria (coluna na planilha) será preenchida automaticamente com base nos valores inseridos (Figura 11).
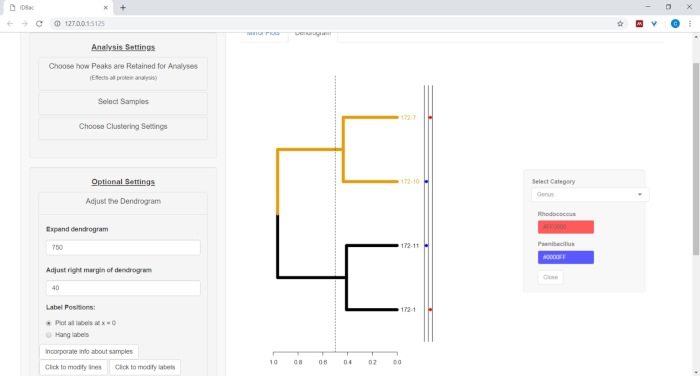
Figura 11: incorporar informações sobre amostras. Dentro do menu "ajustar o dendrograma" é a opção "incorporar informações sobre amostras". Selecionando isso permitirá plotar informações sobre amostras ao lado do dendrograma. Informações de exemplo são entradas dentro da página "trabalhar com experimentos anteriores". Por favor clique aqui para ver uma versão maior desta figura.
11. Inserir amostras de um experimento separado no dendrograma
- Para inserir amostras de outro experimento, selecione o botão de menu Inserir amostras de outro experimento. Siga as instruções no painel recém-aberto (Figura 12).
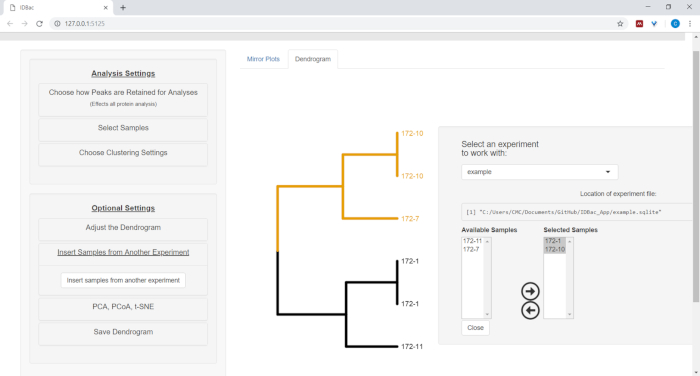
Figura 12: inserir amostras no menu outro experimento. Às vezes, é útil comparar amostras de outro experimento. Use o menu "inserir amostras de outro experimento" para escolher amostras a serem incluídos no dendrograma exibido no momento. Por favor clique aqui para ver uma versão maior desta figura.
12. analisar dados especializados de metabolito e redes de associação de metabolito (MANs)
- Prossiga para a página da rede de associação metabolito (molécula pequena) . Esta página permite a visualização de dados por análise de componentes principais (PCA) e Mans, que utilizam redes bipartido para exibir a correlação de valores de pequena molécula m/z com amostras.
- Se um dendrograma de proteína foi criado (seção 9), ele será exibido nesta página também. Clique e arraste no dendrograma para destacar amostras seleto de interesse a serem analisados. Se nenhuma amostra for destacada ou nenhum dendrograma de proteína foi criado, um homem de um subconjunto aleatório ou todas as amostras aparecerá, respeitosamente (Figura 13).
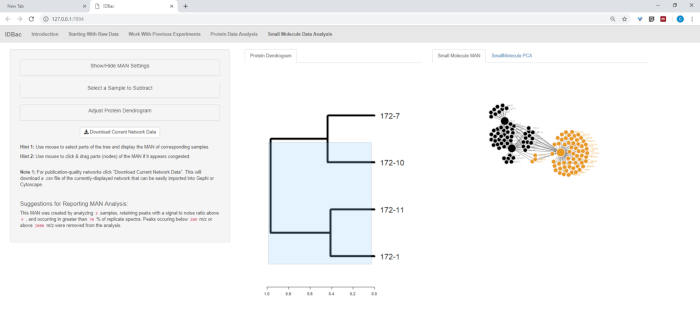
Figura 13: pequena molécula de análise de dados "página. Se um dendrograma foi criado a partir de espectros proteicos, ele será exibido dentro da página "Small molécula Data Analysis". Esta página também exibirá metabolite Associate Networks (MANs) e análise de componentes de princípio (PCA) para dados de pequenas moléculas. Por favor clique aqui para ver uma versão maior desta figura.
- Para subtrair uma matriz/mídia em branco no MAN, abra o menu Selecione um exemplo para subtrair e escolha o exemplo apropriado para usar como um espaço em branco.
- Abra o menu Mostrar/ocultar configurações do Man para selecionar os valores desejados para a porcentagem de presença de pico em repetições, sinal para ruído e cortes de massa superiores e inferiores, como foi feito para espectros proteicos na seção 9. Use as parcelas pequenas do espelho da molécula para guiar a seleção destas configurações.
- Selecione "transferir dados de rede actuais" para guardar os dados do MAN que está actualmente a ser apresentado. Esses dados podem ser usados em softwares de análise de rede que não sejam IDBac.
- Para relatar resultados, copie o texto dentro das sugestões para o parágrafo de análise de homem de relatório . Isso fornece as configurações definidas pelo usuário usadas para gerar o MAN criado.
13. compartilhando dados
- Cada IDBac "experimento" é salvo como um único banco de dados SQLite. Ele contém os espectros brutos mzML convertidos, picos detectados e todas as informações de entrada do usuário sobre amostras. Portanto, para compartilhar um experimento IDBac simplesmente compartilhe o arquivo SQlite que tem o mesmo nome que o experimento (o local do arquivo é exibido na página trabalhando com experimentos anteriores ).
Resultados
Foram analisadas seis cepas de Micromonospora chokoriensis e duas cepas de Bacillus subtilis, que foram caracterizadas anteriormente6, utilizando dados disponíveis em doi: 10.5281/zenodo. 2574096. Seguindo as instruções na guia iniciando com dados brutos , selecionamos a opção clique aqui para converter os arquivos do Bruker e seguimos a instrução fornecida pelo idbac para cada conjunto de dados (Figura 14).
Depois que as etapas automatizadas da conversão e do pré-processamento/pico-peaking foram terminadas, nós proseguimos criar um experimento combinado novo do IDBac Transferindo amostras dos dois experimentos em um único experimento que contem o bacilo e Amostras de micromonosopora (Figura 15). A análise resultante envolveu a comparação de espectros proteicos utilizando parcelas espelhadas, como ilustrado na Figura 16, que foi útil para avaliar a qualidade dos espectros e ajustar as configurações de pico-picking. A Figura 17 exibe uma captura de tela dos resultados de clustering de proteínas com as configurações padrão selecionadas. O dendrograma foi colorido ajustando o limiar no gráfico (aparece como uma linha pontilhada). Nota-se a clara separação entre gêneros, com os isolados de M. chokoriensis e B. subtilis em agrupamento separado.
Figura 18, Figura 19e Figura 20 destacam a capacidade de gerar Mans de regiões selecionadas pelo usuário clicando e arrastando através do dendrograma de proteínas. Com isso, pudemos criar rapidamente MANs para comparar apenas as cepas de B. subtilis (Figura 18), apenas as cepas de M. chokoriensis (Figura 19) e todas as cepas simultaneamente (Figura 20). A função preliminar destas redes é fornecer investigadores uma vista geral larga do grau de sobreposição especializada do metabolito entre bactérias. Com esses dados em mãos, os pesquisadores agora têm a capacidade de tomar decisões informadas a partir de apenas uma pequena quantidade de material raspado de uma colônia bacteriana.
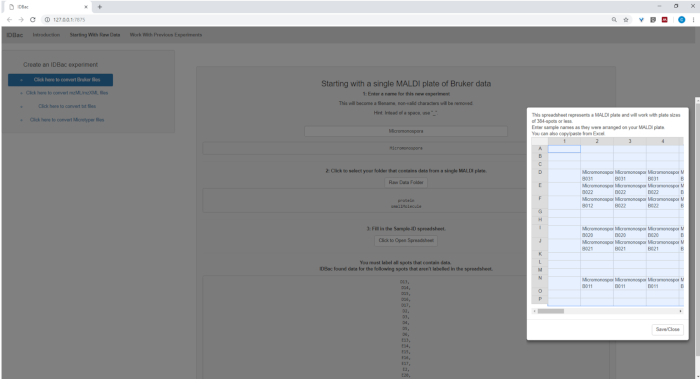
Figura 14: processamento de espectros. Os espectros baixados do autoFlex do Bruker foram convertidos e processados usando IDBac. Por favor clique aqui para ver uma versão maior desta figura.
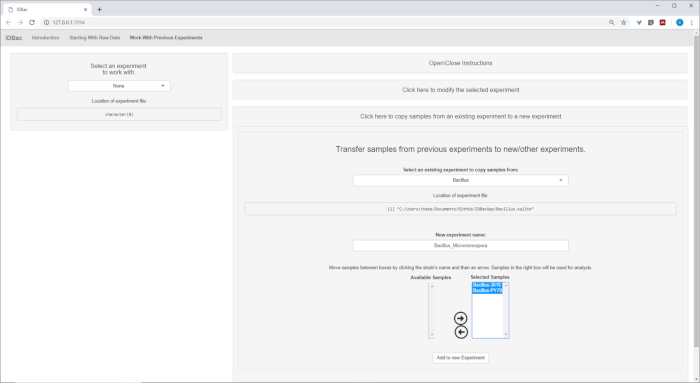
Figura 15: experimento de IDBac combinado. Porque os espectros da Micromonospora e do bacilo foram coletados em placas diferentes do alvo de MALDI, os dois experimentos foram combinados subseqüentemente em um único experimento-"Bacillus_Micromonsopora". Isso foi feito dentro da aba "trabalhar com experimentos anteriores", seguindo as instruções no menu "transferir amostras de experimentos anteriores para novos/outros experimentos". Por favor clique aqui para ver uma versão maior desta figura.
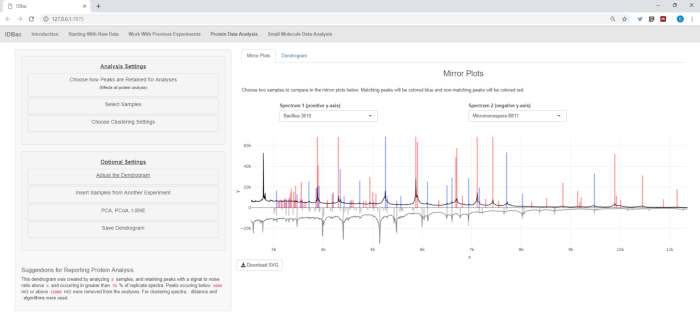
Figura 16: comparação. Micromonspora e Bacillus Spectra foram comparados utilizando as parcelas espelhadas na página "análise de dados proteicos". Em última análise, as configurações de pico padrão foram escolhidas. Por favor clique aqui para ver uma versão maior desta figura.
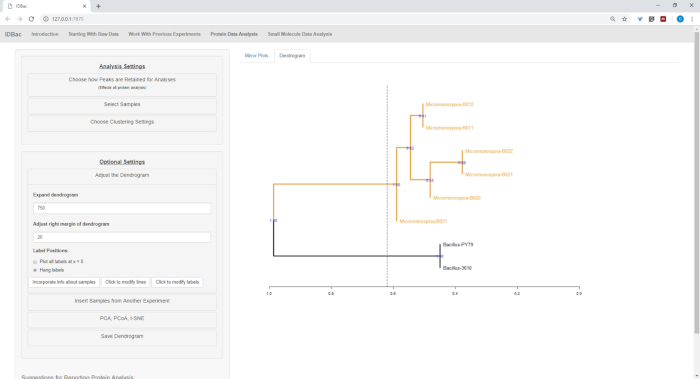
Figura 17: agrupamento hierárquico. Agrupamento hierárquico, usando configurações padrão, agrupados corretamente Bacillus e Micromonospora isolados. O dendrograma foi colorido por "cortar" o dendrograma em uma altura arbitrária (exibido como uma linha tracejada) e 100 bootstraps usados para mostrar confiança na ramificação. Por favor clique aqui para ver uma versão maior desta figura.
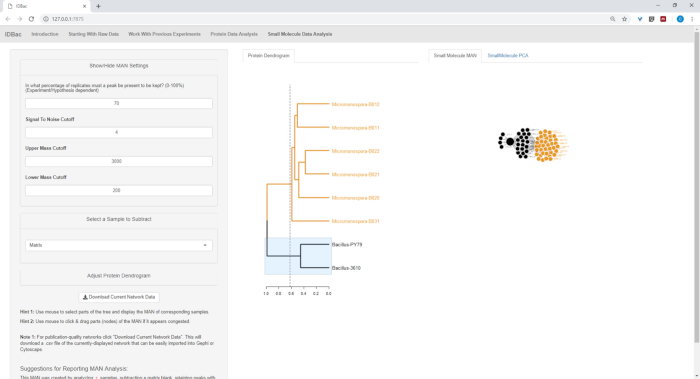
Figura 18: o homem criado selecionando as cepas de Bacillus SP. do dendrograma de proteínas mostrou produção diferencial de metabólitos especializados. Por favor clique aqui para ver uma versão maior desta figura.
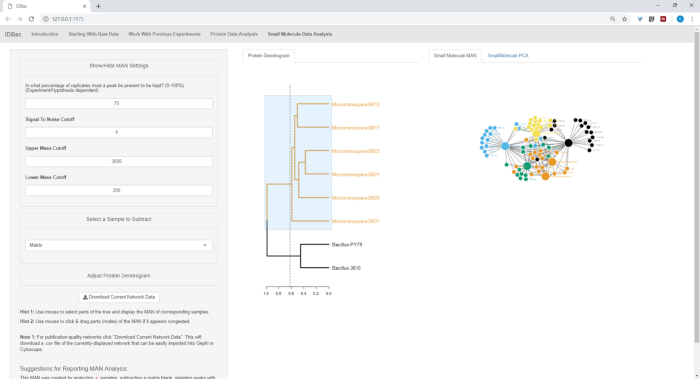
Figura 19: o homem criado selecionando as seis cepas de Micromonospora SP. do dendrograma de proteínas mostrou produção diferencial de metabólitos especializados. Por favor clique aqui para ver uma versão maior desta figura.
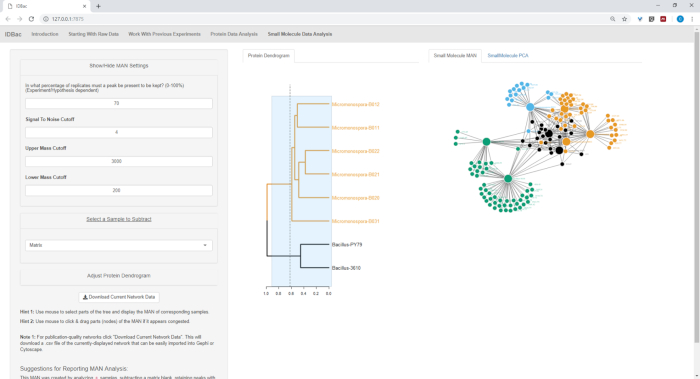
Figura 20: o homem das cepas de Bacillus SP. e Micromonospora SP. mostra uma produção diferencial de metabólitos especializados. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
O protocolo IDBac detalha a proteína bacteriana e a aquisição e análise de dados metabólito especializados de até 384 isolados bacterianos em 4 h por um único pesquisador. Com IDBac não há necessidade de extrair DNA de isolados bacterianos ou gerar extratos metabólitos especializados de caldos de fermentação líquida e analisá-los usando métodos cromatográficos. Em vez disso, os dados de proteínas e metabólito especializados são recolhidos simplesmente espalhando material de colônias bacterianas diretamente sobre uma placa alvo MALDI. Isto reduz extremamente o tempo e o custo associados com as técnicas alternativas tais como o sequenciamento do gene 16S rRNA e o LCMS9.
É importante adicionar uma matriz em branco e pontos de calibração à placa MALDI, e recomendamos o uso de um número adequado de repetições para garantir a reprodutibilidade e a confiança estatística. O número de repetições será dependente do experimento. Por exemplo, se um usuário pretende diferenciar milhares de colônias de uma coleção de placas de diversidade ambiental, menos repetições podem ser necessárias (nosso laboratório coleta três repetições técnicas por colônia). Alternativamente, se um usuário deseja criar um banco de dados personalizado de cepas de táxons bacterianos específicos para determinar rapidamente classificações de subespécies de isolados desconhecidos, mais réplicas são apropriadas (nosso laboratório coleta oito repetições biológicas por tensão).
O IDBac é uma ferramenta para diferenciar rapidamente os isolados bacterianos altamente relacionados baseados na informação taxonômica putativa e na produção especializada do metabolito. Pode complementar ou servir como um precursor de métodos ortogonais, tais como análises genéticas aprofundadas, estudos envolvendo produção e função do metabolito, ou caracterização da estrutura do metabolito especializado por espectroscopia de ressonância magnética nuclear e/ou LC-MS/MS.
A produção de metabolito especializado (IDBac MANs) é altamente suscetível a condições de crescimento bacteriano, especialmente utilizando diferentes meios de comunicação, o que é uma potencial limitação do método. No entanto, essas características podem ser exploradas pelo usuário, como IDBac pode prontamente gerar MANs mostrando as diferenças na produção de metabólito especializados uma variedade de condições de crescimento. É importante notar que, embora as impressões digitais do metabolito especializado possam variar de acordo com a condição de crescimento, mostramos previamente que as impressões digitais de proteínas permanecem relativamente estáveis nessas variáveis (ver Clark et al.6). Ao lidar com as placas de diversidade ambiental, recomendamos a purificação de isolados bacterianos antes da análise, a fim de reduzir possíveis contribuições de cross-talk bacteriano vizinho.
Finalmente, a falta de uma base de dados pública pesquisável de impressões digitais da proteína MS é um lacuna principal no uso deste método para classificar bactérias ambientais desconhecidas. Criamos o idbac com isso em mente e incluímos a conversão automatizada de dados em um formato de código aberto aceito pela Comunidade (mzml)10,11,12 e projetamos o software para permitir a pesquisa, compartilhamento e criação de bancos de dados personalizados. Estamos no processo de criação de um grande banco de dados público (> 10000 cepas totalmente caracterizadas), o que permitirá a classificação de alguns isolados para o nível de espécie, incluindo links para números de adesão GenBank quando disponível.
IDBac é fonte aberta e o código está disponível para qualquer pessoa personalizar suas necessidades de análise e visualização de dados. Recomendamos que os usuários consultem um extenso corpo de literatura (Sauer et al.7, Silva et al.5) para ajudar a apoiar e projetar seus objetivos experimentais. Nós hospedamos um fórum para discussão em: https://groups.google.com/forum/#!forum/idbac e um meio para relatar problemas com o software em: https://github.com/chasemc/IDBacApp/issues.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelo Instituto Nacional de ciências médicas gerais Grant R01 GM125943, National Geographic Grant CP-044R-17; Concessão do fundo de pesquisa Islandês 152336-051; e da Universidade de Illinois em Chicago fundos de inicialização. Além disso, agradecemos aos seguintes contribuidores: Dr. Amanda Bulman para assistência com os parâmetros de aquisição de proteínas MALDI-TOF MS; Dr. Terry Moore e Dr. Atul Jain para recristalização Alpha-cyano-4-Hydroxycinnamic Acid Matrix (CHCA).
Materiais
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
Referências
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados