Method Article
使用开源 MALDI TOF-MS IDBac 管道分析微生物蛋白和专用代谢物数据
摘要
IDBac 是一种基于开源质谱学的生物信息学管道,它整合了从完整蛋白质和专用代谢物光谱中收集的数据,这些数据来自从细菌菌落中刮取的细胞材料。该管道使研究人员能够迅速将数百到数千个细菌菌落组织成假定的分类组,并根据专门的代谢物生产进一步区分它们。
摘要
为了直观地了解生长在营养琼脂上的细菌菌落的细菌菌落的细菌菌落的专门代谢物生产之间的关系,我们开发了IDBac——一种低成本、高通量的基质辅助激光解吸/电化飞行时间质谱(MALDI-TOF MS)生物信息学管道。IDBac 软件专为非专家设计,可免费使用,能够分析数到数千个细菌菌落。在这里,我们介绍了为MALDI-TOF MS分析、MS仪器操作以及IDBac中的数据处理和可视化制备细菌菌落的程序。特别是,我们指导用户如何基于蛋白质MS指纹将细菌聚集到树状图中,并从专门的代谢物数据中交互地创建代谢物关联网络(MANs)。
引言
研究细菌功能的研究人员的一个主要障碍是能够快速同时评估微生物的分类特性及其产生专门代谢物的能力。这阻止了在了解细菌植理和从环境中分离的大多数细菌中专门代谢物生产之间的关系方面取得重大进展。虽然使用蛋白质指纹对细菌进行分组和识别的方法对1、2、3、4进行了很好的描述,但这些研究通常对一小群分离物进行了描述,以物种特定的方式。重要的是,关于专门代谢物生产的信息,是环境中微生物功能的主要驱动因素,在这些研究中仍未纳入其中。席尔瓦等人最近提供了一个全面的历史,详细介绍了MALDI-TOF MS在分析专业代谢物和软件短缺方面的不足,以缓解目前生物信息学的瓶颈。为了解决这些缺点,我们创建了IDBac,一种生物信息学管道,它集成了MALDI-TOF MS6的线性和反射线模式。这允许用户根据蛋白质和专用代谢物MS指纹快速可视化和区分细菌分离物。
IDBac 具有成本效益高、吞吐量高,专为非专业用户而设计。它可免费(chasemc.github.io/IDBac),并且只需要使用 MALDI-TOF 质谱仪(专用代谢物分析需要反射模式)。样品制备依赖于简单的"扩展直接传输"方法7,8,数据通过连续线性和反射子采集在单个MALDI目标点上采集。使用 IDBac,可以在四小时内分析数百个菌落的假定形态和专用代谢物生产,包括样品制备、数据采集和数据可视化。与传统识别细菌方法(如基因测序)和分析代谢输出(液相色谱-质谱 [LCMS] 和类似的色谱方法)相比,这具有显著的时间和成本优势。
IDBac利用线性模式分析中获得的数据,采用分层聚类来表示蛋白质光谱的相关性。由于光谱主要代表离子核糖体蛋白,它们提供了样品中存在的遗传多样性的表示。此外,IDBac 还集成了反射器模式数据,以将专门的代谢物指纹显示为代谢物关联网络 (MAN)。AN 是双部分网络,允许轻松可视化细菌分离物之间的共享和独特的代谢物生产。IDBac 平台允许研究人员同时分析蛋白质和专用代谢物数据,但如果只获得一种数据类型,则还可以单独分析这些数据。重要的是,IDBac 处理来自布鲁克和厦门仪器的原始数据,以及 txt、tab、csv、mzXML 和 mzML。这消除了对数据集进行手动转换和格式化的需要,并显著降低了用户错误或 MS 数据处理不当的风险。
研究方案
1. 编制MALDI矩阵
- 在MS级溶剂中制备10mg/mL MALDI级和/或再结晶的β--氰-4-羟基辛酸(CHCA):50%醋酸(ACN)、47.5%水(H2O)、2.5%三氟乙酸(TFA)。例如:100 μL 溶液 = 50 μL ACN = 47.5 μL H2O = 2.5 μL TFA = 1 mg CHCA
- 每个 MALDI 板点和涡流或声波至少制备 1 μL 的基质溶液,直到在溶液中(大约 5 分钟声波或无可见固体)。
注意:TFA 是一种强酸,在佩戴适当的个人防护设备时应在化学烟气罩中处理,因为它可能会因接触或吸入而损害皮肤、眼睛和气道。
注:CHCA具有吸湿性和光敏性,应存放在琥珀瓶中的干燥器中。有许多 MALDI 矩阵选项可用。CHCA是最常见的细菌蛋白质分析,但也工作为专门的代谢物分析。矩阵选择取决于单个用户/实验需求。
- 每个 MALDI 板点和涡流或声波至少制备 1 μL 的基质溶液,直到在溶液中(大约 5 分钟声波或无可见固体)。
2. MALDI目标板的准备
注:详情请参阅Sauer等人7。
- 用甲醇(HPLC 级或更高)冲洗 MALDI 板,并用软纸擦拭擦干。清洁目标板时不要使用磨料刷,因为这样会永久损坏目标板的表面。
- 分配蛋白质和专用代谢物校准点。在样本总体之间均匀组织校准点,以考虑 MALDI 板不规则值和仪器随时间漂移的情况。为研究分配适当数量的介质/矩阵空白点;这些点将只包含介质和矩阵,或仅包含矩阵。
- 使用无菌牙签,将细菌菌群的一小部分转移到 MALDI 板上的适当位置。将细菌菌群均匀地分布到点上。点应尽可能平坦。
注:将粘附/非晶状细菌菌落压平将更容易。对于更刚性/固体的菌落,避免在 MALDI 点上留下可见的细胞质量簇(图 1)。
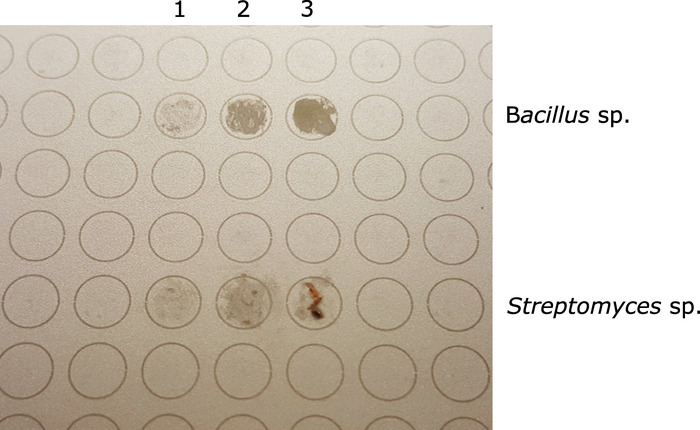
图1:MALDI目标板在添加甲酸和MALDI基质之前显示两个不同的分离物(前3个点- 杆菌,后3个斑点-链球菌。 对于两者,列3表示过量样本;第2列表示适当的样本量;第1列表示 MALDI 分析的样本不足。请点击此处查看此图的较大版本。
- 使用无菌牙签将少量琼脂/培养基转移到 MALDI 板上的适当位置,准备基质/介质控制。
- 将70%质谱级甲酸的1μL覆盖到每个样品点,包括基质控制点。让酸在化学烟气罩中完全干燥(约 5 分钟)。
注意:甲酸是一种腐蚀性化学物质,应在化学烟气罩中处理。吸入后会损害气道。 - 在每个采样点以及矩阵/介质控制点中添加 1 μL 制备的 MALDI 矩阵溶液。让基质溶液完全风干(约5分钟)。
注: 可以在黑暗中将板存放在干燥器中,直到可以在 MALDI-TOF 质谱仪上进行分析。允许的存储时间可能因样品稳定性而异。 - 向分配的校准点添加 0.5±1.0 μL 校准,然后添加 1 μL MALDI 基质溶液。上下移移溶液以混合。在引入 MALDI-TOF 质谱仪之前,让所有斑点完全风干。
注:蛋白质和专用代谢物校准剂应在MALDI分析的30分钟内加入,因为两者都容易降解。
3. 数据采集
注:数据采集的一般参数列在表1中。
| 参数 | 蛋白 | 专用代谢物 |
| 质量启动(Da) | 1920年 | 60 |
| 质量端(大) | 21000 | 2700 |
| 质量偏转(达) | 1900年 | 50 |
| 镜头 | 500 | 1000 |
| 频率(Hz) | 2000年 | 2000年 |
| 激光尺寸 | 大 | 中 |
| 最大StdDev(ppm) | 300 | 30 |
表 1.
- 遵循所使用仪器的特定协议,设置蛋白质和专用代谢物校准。
- 测试几个单独的目标点,以确定获取光谱时要使用的最佳激光功率和探测器增益(这将在日常和仪器上有所不同)。
注:图2A和图3A显示了最佳光谱,而图2D和图3D是质量差光谱的示例。
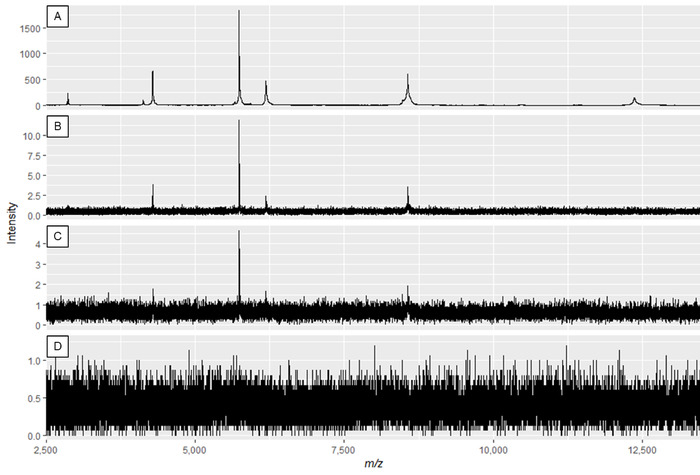
图 2:显示修改激光功率和探测器增益效果的蛋白质光谱示例。光谱质量在面板A中最好,并且下降,直到面板C和D的光谱质量不足。 虽然面板B中的频谱可能会导致可使用的峰值,但面板A显示最佳数据。请点击此处查看此图的较大版本。
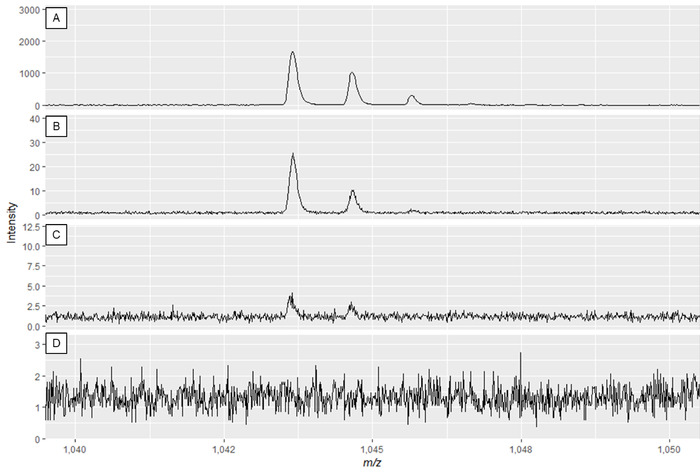
图 3:显示修改激光功率和探测器增益效果的专用代谢物光谱示例。光谱质量在面板A中最好,并且下降,直到面板C和D的光谱质量不足。 虽然面板B中的频谱可能会导致可使用的峰值,但面板A显示最佳数据。请点击此处查看此图的较大版本。
- 获取光谱,将蛋白质光谱保存到一个文件夹,将专门的代谢物光谱放入第二个单独的文件夹。
4. 清洁 MALDI 目标板(改编自绍尔等人7)
- 从支架上取下 MALDI 目标板,然后用丙酮冲洗。
- 用非磨蚀性液体肥皂清洗,去除微量蛋白质和脂质,以及软纸抹布/软毛牙刷。
- 用去离子水冲洗约2分钟,以完全去除肥皂。
- 将目标板在水中(HPLC 等级或更高)中声波 5 分钟。
- 用水(HPLC 等级或更高)冲洗目标板。
- 用甲醇(HPLC 等级或更高)冲洗目标板。
5. 安装 IDBac 软件
- 下载 IDBac 软件。
注:永久版本备份也可下载(参见材料表)。 - 双击下载的"安装_IDBac.exe"以启动安装程序并按照屏幕上的说明进行操作。
6. 从原始数据开始
注:每个数据处理步骤的详细说明和说明嵌入 IDBac 中,但主要分析和交互式输入如下所述。
- 双击 IDBac 桌面快捷方式以启动 IDBac。默认情况下,IDBac 将在"介绍"选项卡上打开。
- 使用"检查更新"按钮可确保使用最新版本的 IDBac(需要访问互联网)。如果有较新版本可用,IDBac 将自动下载并安装更新,之后 IDBac 将请求重新启动。
- 单击"从原始数据开始"选项卡,从菜单中选择要与 IDBac 一起使用的数据类型;继续遵循应用内说明。
- 设置数据文件的转换和处理时,在提示时为实验输入描述性名称(参见图 4)。实验稍后将按字母顺序显示,因此有用的策略是使用组属性开始实验名称(例如,"细菌试验-实验-1";"杆菌试验-实验-2")。
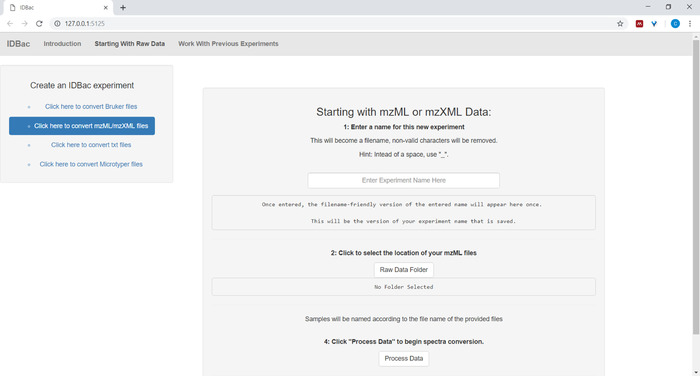
图4:IDBac数据转换和预处理步骤。IDBac 将原始光谱转换为开放的 mzML 格式,并将 mzML、峰值列表和示例信息存储在每个实验的数据库中。请点击此处查看此图的较大版本。
7. 使用以前的实验
- 转换文件并使用 IDBac 处理文件后,或者随时希望重新分析实验,导航到"使用以前的实验"页并选择要处理的实验(图 5)。
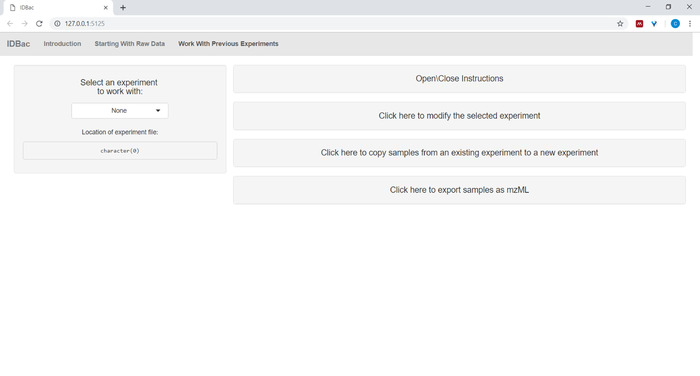
图 5:"使用以前的实验"页。使用 IDBac 的"使用以前的实验"页选择要分析或修改的实验。请点击此处查看此图的较大版本。
- (可选)使用菜单添加有关示例的信息单击此处修改所选实验。将信息输入到自动填充的电子表格中,然后按"保存"(图 6)。 此选项允许用户在分析期间对数据进行颜色编码。
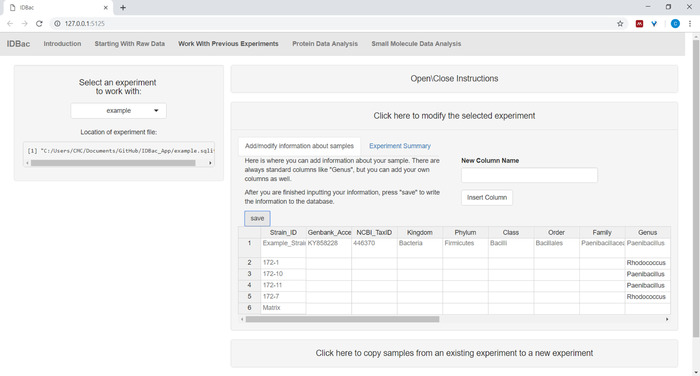
图 6:输入示例信息。在"使用以前的实验"页面中,用户可以输入有关样本的信息,如分类标识、集合位置、隔离条件等。
- (可选)通过单击将以前实验的样本转移到新/其他实验并按照提供的说明(图 7)将所有或样本子集传输到新的或另一个实验。
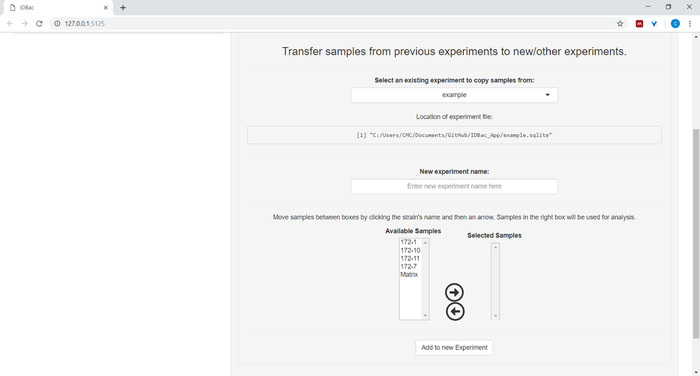
图 7:传输数据。"使用以前的实验"页包含在现有实验和新实验之间传输数据的选项。请点击此处查看此图的较大版本。
- 准备好开始分析时,确保选择要使用的实验。选择蛋白质数据分析或小分子数据分析。
8. 设置蛋白质数据分析和创建镜像图
- 如果分析蛋白质数据,首先导航到蛋白质数据分析页面。选择峰值拾取设置,并通过显示的镜像图评估样品的蛋白质光谱(图8)。
注:在镜像图中,红色峰仅表示该峰值存在于顶光谱中,而蓝色峰表示两个光谱中发生的峰值。
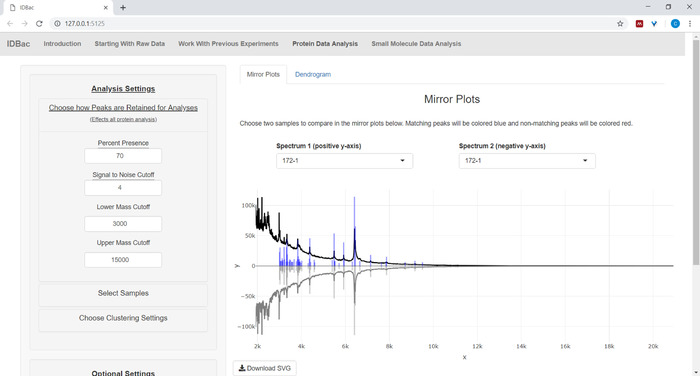
图 8:选择如何保留峰值进行分析。选择要分析的实验后,访问"蛋白质数据分析"页面,然后打开"选择如何保留峰值进行分析"菜单,允许用户选择诸如信噪比等设置来保留峰值。显示的镜像图(或方格拉姆)将自动更新以反映所选设置。请点击此处查看此图的较大版本。
- 调整必须存在峰值的复制百分比,以便将其包含在分析中(例如,如果阈值设置为 70%,并且峰值至少出现在 10 个复制中的 7 个中,则包括峰值)。
- 使用镜像图作为视觉引导,将信号调整为噪声截止,以保留最"正版"峰值和最小噪声,同时指出,更多的复制和更高的"百分比峰值存在"值将允许选择较低的噪声截止信号。
- 指定下部和上部m/z截止值,指定 IDBac 进一步分析中使用的每个频谱中的质量值范围。
9. 使用蛋白质数据对样品进行聚类
- 在蛋白质数据分析页中,选择"Dendrogram"选项卡。 这允许根据用户选择的距离度量和聚类算法将样本分组到树状图中。
- 单击菜单上的"选择样本",然后按照说明选择要包含在分析中的样本。只有含有蛋白质光谱的样品才会显示在可用样品框中(图9)。
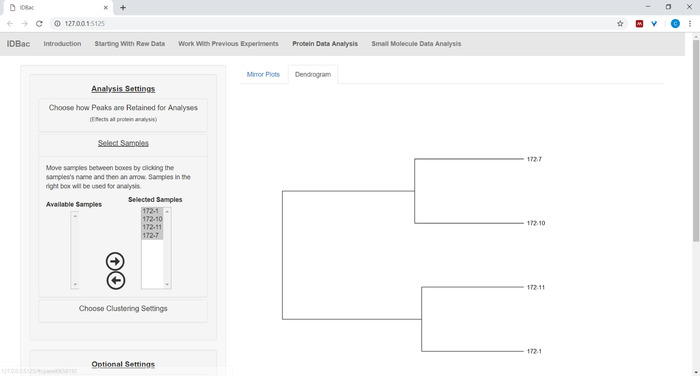
图 9:从所选实验中选择要包含在显示的树形图中的样本。请点击此处查看此图的较大版本。
- 使用默认值,或在"选择聚类设置"下,选择要应用于生成树状图所需的距离和聚类算法。
- 选择"存在/缺勤"作为输入。或者,如果对样本的峰值高度有信心(例如,在执行研究以评估峰值强度的可变性后),请选择强度作为输入。
注: 在发布时,IDBac 提供了群集设置的灵活性,依靠用户选择适当的组合。如果不熟悉这些选项,建议对 A:"后因"距离和"平均 (UPGMA)"聚类;或 B:"欧几里得"距离和"Ward.D2"聚类。 - 要在 dendrogram 上显示引导值,请在"引导"下输入介于 2 和 1000 之间的数字。
- 报告结果时,请复制"报告蛋白质分析建议"段落中的文本。这将提供生成特定树形图的用户定义的设置。
10. 定制蛋白质密度图
- 要开始自定义树形图,请打开"调整树形图"菜单(图 10)。
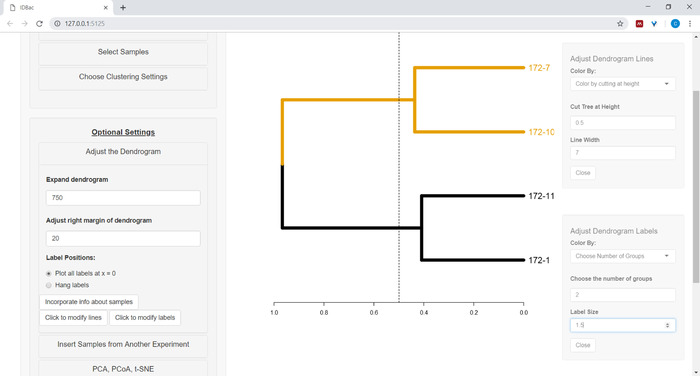
图 10:调整树状图。IDBac 提供了一些选项来修改树状图的外观,这些选项可以在菜单"调整 dendrogram"中找到。这包括通过 k 手段着色分支和标签,或以用户提供的高度"切割"树形图。请点击此处查看此图的较大版本。
- 要为树形图的线条和/或标签着色,请选择相应的按钮:单击以修改线条或单击以修改标注并选择所需的选项。
- 要绘制从电子表格中的信息,请参见步骤 7.2,请选择"包含有关示例的信息"按钮。这将打开一个面板,其中类别(电子表格中的列)将基于输入的值自行填充(图 11)。
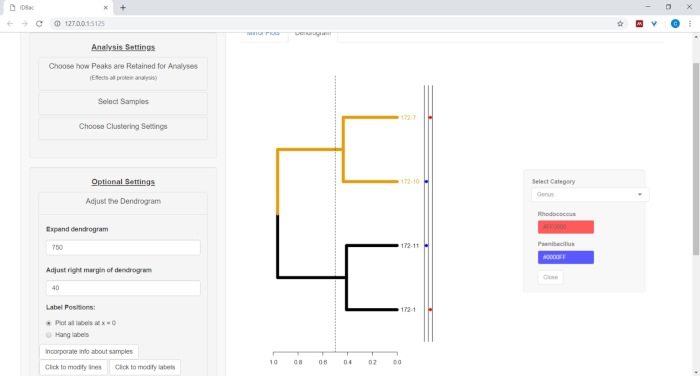
图 11:合并有关示例的信息。在"调整 Dendrogram"菜单中,有"包含有关样本的信息"选项。选择此选项将允许在树状图旁边绘制有关样本的信息。示例信息在"使用以前的实验"页中输入。请点击此处查看此图的较大版本。
11. 将单独实验的样品插入树状图
- 要插入来自其他实验的示例,请选择菜单按钮"插入另一个实验中的样本"。按照新打开的面板中的说明进行操作 (图 12)。
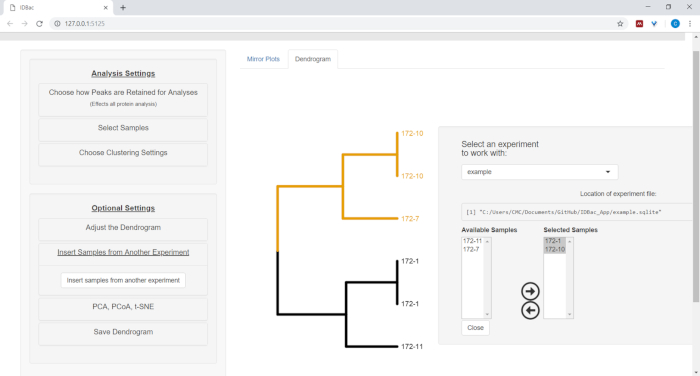
图 12:从另一个实验菜单中插入示例。有时比较另一个实验中的样本会很有帮助。使用"从另一个实验中插入样本"菜单选择要包含在当前显示的树状图中的样本。请点击此处查看此图的较大版本。
12. 分析专门的代谢物数据和代谢物关联网络
- 转到代谢物关联网络(小分子)页面。此页面允许通过主要分量分析 (PCA) 和 MAN 进行数据可视化,它们使用两部分网络来显示小分子m/z值与样本的相关性。
- 如果创建了蛋白质树突图(第 9 节),它也会显示在此页上。单击并拖动树角图以突出显示要分析的感兴趣样本。如果没有突出显示样品或未创建蛋白质树状图,则随机子集或所有样本的 MAN 将显示(图 13)。
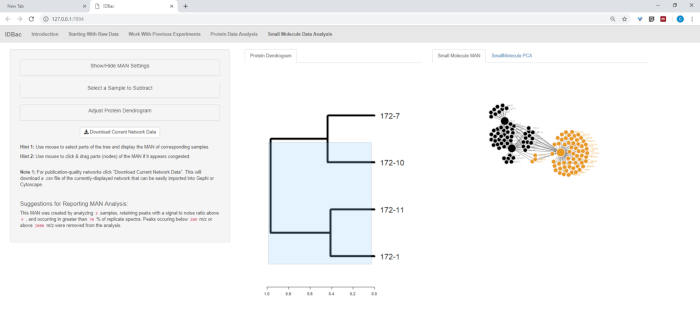
图13:小分子数据分析"页。如果根据蛋白质光谱创建树状图,它将显示在"小分子数据分析"页面中。本页还将显示小分子数据的代谢物关联网络 (MAN) 和原理组分分析 (PCA)。请点击此处查看此图的较大版本。
- 要减去 MAN 中的矩阵/媒体空白,请打开菜单"选择要减料的样本",然后选择用作空白的相应示例。
- 打开菜单"显示/隐藏 MAN 设置",以选择复制中峰值存在百分比、噪声信号以及上部和下质量截止值的所需值,如第 9 节中的蛋白质光谱所做的那样。使用小分子镜像图来指导这些设置的选择。
- 选择"下载当前网络数据"以保存当前显示的 MAN 的数据。这些数据可用于 IDBac 以外的网络分析软件。
- 有关报告结果,请复制"报告人分析建议"段落中的文本。这将提供用于生成创建的 MAN 的用户定义的设置。
13. 分享数据
- 每个 IDBac"实验"都保存为单个 SQLite 数据库。它包含转换的 mzML 原始光谱、检测到的峰值以及有关样品的所有用户输入信息。因此,要共享 IDBac 实验,只需共享与实验同名的 SQlite 文件(文件位置显示在"使用以前的实验"页上)。
结果
我们使用DOI上提供的数据:10.5281/zenodo.2574096分析了6个微单孢菌菌和两个以前被定性为6的杆菌菌株。按照"从原始数据开始"选项卡中的指示,我们选择了"单击此处转换布鲁克文件"选项,并遵循每个数据集的 IDBac 提供的说明(图 14)。
在自动转换和预处理/峰值步骤完成后,我们继续创建一个新的联合 IDBac 实验,将两个实验中的样本转移到一个包含杆菌和微单孢菌素样品(图15)。由此产生的分析涉及使用镜像图比较蛋白质光谱,如图16所示,这可用于评估光谱质量和调整峰分设置。图 17显示了蛋白质聚类结果的屏幕截图,并选择了默认设置。通过调整绘图上的阈值(显示为虚线)对树状图进行着色。值得注意的是,属间之间有明显的分离,M.chokoriensis和B.subtilis分离了分聚。
图18、图19和图20强调了通过单击和拖动蛋白质树突来生成用户选定区域的MAN的能力。有了这个,我们能够快速创建MAN来比较只有B.subtilis菌株(图18),只有M.胆碱菌株(图19),和所有菌株同时比较(图20)。这些网络的主要功能是向研究人员提供细菌之间专门代谢物重叠程度的广泛概述。有了这些数据,研究人员现在有能力从细菌菌群中刮掉的少量物质中做出明智的决定。
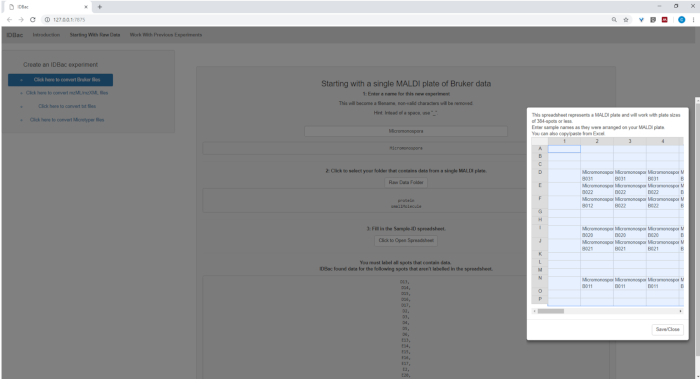
图14:光谱处理。下载的布鲁克自动Flex光谱被转换和处理使用IDBac。请点击此处查看此图的较大版本。
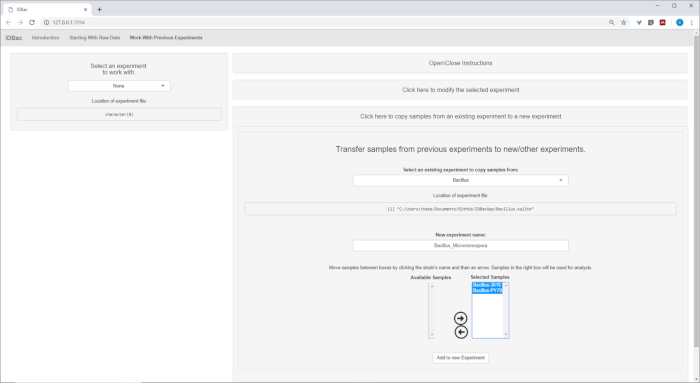
图 15:组合 IDBac 实验。由于微单孢菌和杆菌光谱被收集到不同的MALDI靶板上,因此这两个实验随后被合并为一个实验——"微球菌"。这是在"使用以前的实验"选项卡中完成的,按照菜单中"将以前实验的样本转移到新的/其他实验"中的指示进行。请点击此处查看此图的较大版本。
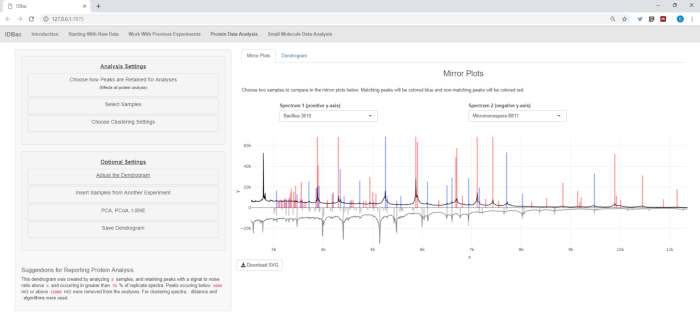
图16:比较。使用"蛋白质数据分析"页面中的镜像图对微蒙斯波拉和杆菌光谱进行了比较。最终,选择了默认峰值设置。请点击此处查看此图的较大版本。
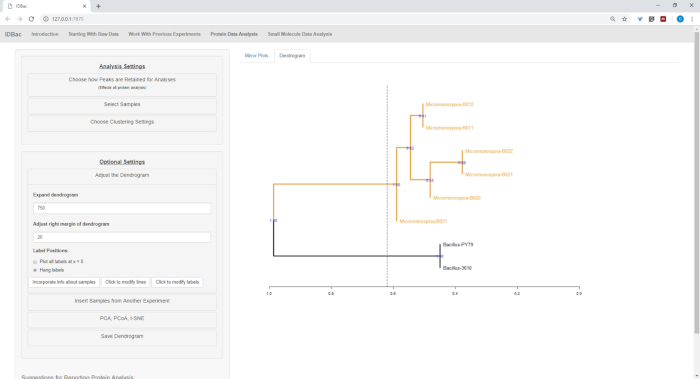
图 17:分层聚类。分层聚类,使用默认设置,正确分组杆菌和微单体孢子隔离。树突图的颜色是"切割"在任意高度(显示为虚线)和100引导用于显示对分支的信心的树形图。请点击此处查看此图的较大版本。
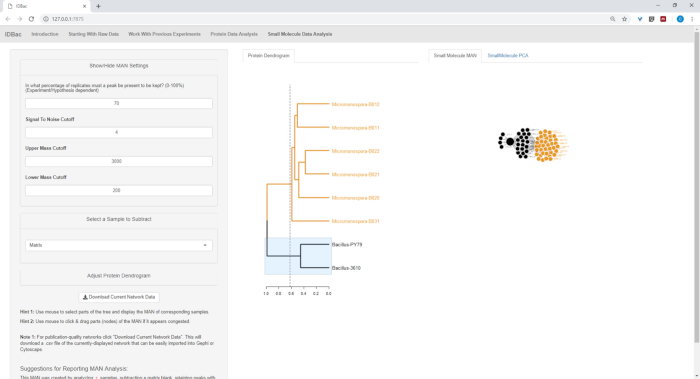
图18:MAN通过从蛋白质树状图中选择杆菌菌株而创建的,显示了特殊代谢物的差分生产。请点击此处查看此图的较大版本。
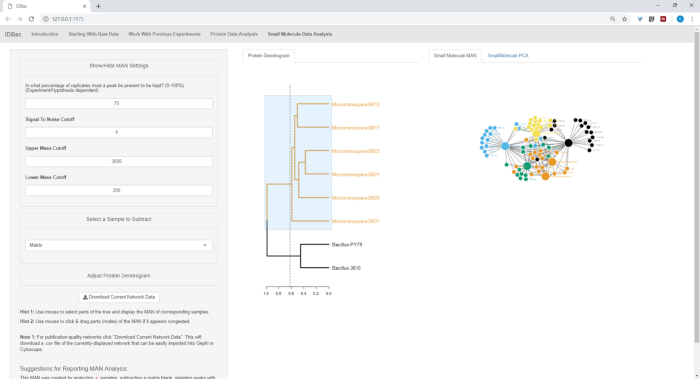
图19:MAN通过从蛋白质树突图中选择六个微单体孢子菌株而创建,显示了特殊代谢物的差分生产。请点击此处查看此图的较大版本。
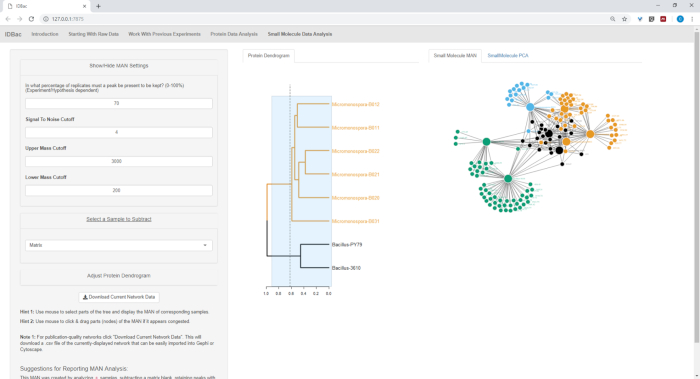
图20:杆菌和微单体孢子菌株的MAN,显示了特殊代谢物的差分生产。请点击此处查看此图的较大版本。
讨论
IDBac 协议详细介绍了细菌蛋白和专门的代谢物数据采集和分析,由单个研究人员在 4 小时内采集和分析多达 384 种细菌分离物。使用 IDBac,无需从细菌分离物中提取 DNA 或从液体发酵汤中提取专用代谢物提取物,并使用色谱方法对其进行分析。相反,蛋白质和专门的代谢物数据是通过简单地将细菌菌落中的材料直接传播到MALDI靶板上来收集的。这大大减少了与替代技术相关的时间和成本,如16S rRNA基因测序和LCMS9。
向 MALDI 板添加矩阵空白点和校准点非常重要,我们建议使用适当数量的复制,以确保可重现性和统计置信度。复制的数量将依赖于实验。例如,如果用户打算将数千个菌落与一组环境多样性板区分开来,则可能需要较少的复制(我们的实验室为每个菌落收集三个技术复制)。或者,如果用户希望创建特定细菌分类菌株的自定义数据库,以快速确定未知分离物的子物种分类,则需要更多的复制(我们的实验室每应变)。
IDBac 是一种基于假定分类学信息和专用代谢物生产快速区分高度相关细菌分离物的工具。它可以补充或作为正交方法的前体,如深入的遗传分析,涉及代谢物生产和功能的研究,或通过核磁共振光谱和/或鉴定专门的代谢物结构LC-MS/MS.
专门的代谢物生产(IDBac AN)极易受到细菌生长条件的影响,尤其是使用不同的培养基,这是该方法的潜在局限性。然而,这些特性可能被用户利用,因为IDBac可以很容易地生成MAN,显示在各种生长条件下在专用代谢物生产中的差异。需要注意的是,虽然专门的代谢物指纹可能因生长条件而异,但我们以前已经表明,蛋白质指纹在这些变量中保持相对稳定(见Clark等人6)。在处理环境多样性板时,我们建议在分析之前纯化细菌分离物,以减少邻近细菌串扰的可能贡献。
最后,缺乏可搜索的蛋白质MS指纹公共数据库是使用这种方法对未知环境细菌进行分类的一大缺陷。我们为此创建了 IDBac,包括将数据自动转换为社区接受的开源格式 (mzML)10、11、12,并设计了允许搜索、共享和创建自定义数据库。我们正在创建一个大型公共数据库(>10,000 个完全特征菌株),这将允许将一些分离物分类到物种级别,包括指向GenBank加入编号的链接(如果可用)。
IDBac 是开源的,任何人都可以自定义其数据分析和可视化需求。我们建议用户查阅大量文献(Sauer等人7,席尔瓦等人5),以帮助支持和设计他们的实验目标。我们主办了一个论坛讨论:https://groups.google.com/forum/#!forum/idbac和一种在:https://github.com/chasemc/IDBacApp/issues报告软件问题的方法。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家普通医学研究所资助R01 GM125943,国家地理赠款CP-044R-17;冰岛研究基金赠款152336-051;和伊利诺伊大学在芝加哥的创业基金。此外,我们感谢以下贡献者:阿曼达·布尔曼博士协助使用MALDI-TOF MS蛋白质采集参数;特里·摩尔博士和阿图尔·贾因博士用于重新结晶α-氰化物-4-羟基辛酸基质(CHCA)。
材料
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
参考文献
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。