Method Article
Использование трубопровода MALDI TOF-MS IDBac с открытым исходным кодом для анализа микробного белка и специализированных метаболитных данных
В этой статье
Резюме
IDBac является открытым исходным кодом масс-спектрометрии основе биоинформатики трубопровода, который объединяет данные из нетронутых белков и специализированных метаболитов спектра, собранные на клеточном материале Царапины из бактериальных колоний. Трубопровод позволяет исследователям быстро организовать сотни до тысячи бактериальных колоний в таксономические группы, и далее дифференцировать их на основе специализированного производства метаболита.
Аннотация
Для того, чтобы визуализировать связь между бактериальной филогенией и специализированным производством метаболитов бактериальных колоний, растущих на питательном агаре, мы разработали IDBac – недорогой и высокопроизводительный матричной матрицы при поддержке лазерной дезорпации/ионизации биоинформатика времен полета (MALDI-TOF MS). Программное обеспечение IDBac предназначено для неспециалистов, находится в свободном доступе и способно анализировать от нескольких до тысяч бактериальных колоний. Здесь мы представляем процедуры для подготовки бактериальных колоний для анализа MALDI-TOF MS, операции MS прибора, а также обработки и визуализации данных в IDBac. В частности, мы проинструктируем пользователей, как группировать бактерии в дендрограммы на основе отпечатков пальцев белка MS и интерактивно создавать метаболитассоциации сетей (MANs) из специализированных данных метаболита.
Введение
Основным препятствием для исследователей, изучающих бактериальную функцию, является способность быстро и одновременно оценивать таксономическую идентичность микроорганизма и его способность производить специализированные метаболиты. Это предотвратило значительные достижения в понимании взаимосвязи между бактериальной филогенией и специализированным производством метаболитов в большинстве бактерий, изолированных от окружающей среды. Хотя MS-методы, которые используют отпечатки пальцев белка для группы и выявления бактерий хорошо описаны1,2,3,4, эти исследования, как правило, были выполнены на небольших группах изолятов, в видовой манере. Важно отметить, что информация о специализированном производстве метаболитов, который является основным фактором микробной функции в окружающей среде, в этих исследованиях остается неинкорпорированной. Сильва и др.5 недавно представили всеобъемлющую историю с подробным описанием недостаточного использования MALDI-TOF MS для анализа специализированных метаболитов и нехватки программного обеспечения для облегчения текущих узких мест биоинформатики. Для устранения этих недостатков мы создали IDBac, конвейер биоинформатики, который интегрирует как линейные,так и отражающие режимы MALDI-TOF MS 6. Это позволяет пользователям быстро визуализировать и дифференцировать бактериальные изоляты на основе белка и специализированных метаболитных MS отпечатков пальцев, соответственно.
IDBac является экономически эффективным, высокой пропускной записи, и предназначен для неспециалистов. Он находится в свободном доступе (chasemc.github.io/IDBac) и требует доступа только к масс-спектрометру MALDI-TOF (режим отражательного режима потребуется для специализированного метаболита). Подготовка образцов основывается на простомметоде "расширенной прямой передачи" 7,8 иданные собираются с последовательными линейными и отражательными приобретениями на одном месте MALDI-целевой. С помощью IDBac можно проанализировать предохранение от филогении и специализированного производства метаболитов в сотнях колоний менее чем за четыре часа, включая подготовку образцов, получение данных и визуализацию данных. Это представляет собой значительное временное и затратное преимущество по сравнению с традиционными методами выявления бактерий (таких как секвенирование генов) и анализа метаболического выхода (жидкой хроматографии-масс-спектрометрии (LCMS) и аналогичных хроматографических методов).
Используя данные, полученные в линейном режиме анализа, IDBac использует иерархическую кластеризацию для представления родственности белковых спектров. Поскольку спектры в основном представляют ионизированные рибосомные белки, они обеспечивают представление филогенетического разнообразия, присутствующее в образце. Кроме того, IDBac включает данные отражательного режима для отображения специализированных метаболитных отпечатков пальцев в виде сетей метаболитной ассоциации (MANs). MAN представляют собой двухсторонние сети, которые позволяют легко визуализировать совместное и уникальное производство метаболитов между бактериальными изолятами. Платформа IDBac позволяет исследователям анализировать как белковые, так и специализированные метаболитовые данные в тандеме, но также индивидуально, если приобретен только один тип данных. Важно отметить, что IDBac обрабатывает необработанные данные с инструментов Bruker и Xiamen, а также txt, tab, csv, mzXML и mzML. Это устраняет необходимость ручного преобразования и форматирования наборов данных и значительно снижает риск ошибки пользователя или неправильного обращения с данными MS.
протокол
1. Подготовка матрицы MALDI
- Подготовка 10 мг/мл MALDI-класса, и / или рекристаллизованных -циано-4-гидроксициннамическая кислота (CHCA) в растворителях MS-класса: 50% ацетонитрила (ACN), 47,5% воды (H2O), 2,5% трифторорацетической кислоты (TFA). Пример: раствор 100 л.
- Приготовьте не менее 1 л матричного раствора на пятно пластины MALDI и вихрь или соните до раствора (приблизительно 5 мин sonication или нет видимых твердых веществ).
ВНИМАНИЕ: TFA является сильной кислотой, которая должна быть обработана в химическом капоте дыма во время ношения надлежащего индивидуального защитного оборудования, так как это может повредить кожу, глаза и дыхательные пути при контакте или вдыхании.
ПРИМЕЧАНИЕ: CHCA является гигроскопической и светочувствительной и должна храниться в янтарных флаконах в обезвоживании. Есть много вариантов матрицы MALDI доступны. CHCA является наиболее распространенным для профилирования белка бактерий, но и работает для специализированного анализа метаболита. Выбор матрицы зависит от индивидуальных потребностей пользователя/эксперимента.
- Приготовьте не менее 1 л матричного раствора на пятно пластины MALDI и вихрь или соните до раствора (приблизительно 5 мин sonication или нет видимых твердых веществ).
2. Подготовка целевых пластин MALDI
ПРИМЕЧАНИЕ: Смотрите Sauer и др.7, для более подробной информации.
- Промыть пластину MALDI с метанолом (HPLC-класса или выше) и протрите сухим мягкими бумажными салфетками. Не используйте абразивные щетки при чистке таргетовых пластин, так как это может навсегда повредить поверхность целевой пластины.
- Назначаем белковые и специализированные метаболитные калибровочные пятна. Организуйте калибровочные пятна равномерно по всей популяции выборки, с тем чтобы со временем учитывать неровности MALDI-plate и дрейф приборов. Назначить соответствующее количество носителей/матрицы-пустых пятен для исследования; эти пятна будут содержать только средства массовой информации и матрицы, или только матрицы.
- Используя стерильную зубочистку, перенесите небольшую часть бактериальной колонии в соответствующее место на пластине MALDI. Распределите бактериальную колонию равномерно над местом. Пятно должно выглядеть как можно более плоским.
ПРИМЕЧАНИЕ: Это будет легче сгладить бактериальных колоний, которые являются более слизистые / аморфные. Для более жестких / твердых колоний, не оставляйте видимые кластеры клеточной массы на месте MALDI(рисунок 1).
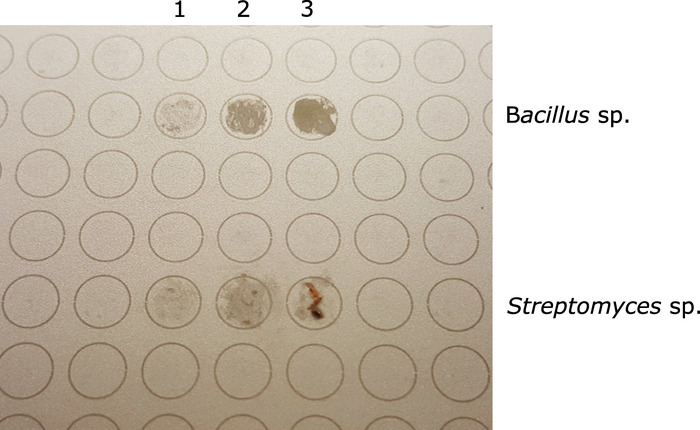
Рисунок 1: MALDI-целевая пластина, показывающая два различных изолята перед добавлением для кремновой кислоты и матрицы MALDI (топ 3 пятна - Bacillus sp.; нижние 3 пятна - Streptomyces sp.). Для обоих столбец 3 представляет собой избыточную выборку; Столбец 2 представляет соответствующее количество выборки; столбец 1 представляет собой недостаточную выборку для анализа MALDI. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Подготовьте матричной/медиа-контроль с помощью стерильной зубочистки для передачи минимального количества агара/носителя на соответствующее место (ы) на пластине MALDI.
- Наложение 1 Зл 70% масс-спектрометрии класса для мистовой кислоты на каждом месте образца, в том числе матричных контрольных пятен. Дайте кислоте полностью высохнуть в химическом капоте дыма (примерно 5 мин).
ВНИМАНИЕ: Formic кислота является едким химическим веществом и должны быть обработаны в химических вытяжки дыма. Это может повредить дыхательные пути при вдыхании. - Добавьте 1 зл и готового матричного решения MALDI к каждому месту образца, а также к точкам управления матрицами/медиа. Дайте матричного раствора полностью высохнуть (примерно 5 мин).
ПРИМЕЧАНИЕ: Можно хранить пластину в сушилке, в темноте, пока она не может быть проанализирована на масс-спектрометре MALDI-TOF. Допустимое время хранения может варьироваться в зависимости от стабильности образца. - Добавьте калибральное калибрантство 0,5–1,0 л к назначенным точкам калибровки, а затем к матритричному решению MALDI. Pipette результирующее решение вверх и вниз, чтобы смешать. Разрешить все пятна воздуха полностью высохнуть до введения в MALDI-TOF масс-спектрометр.
ПРИМЕЧАНИЕ: Белок и специализированные калибровки метаболита должны быть добавлены в течение 30 минут анализа MALDI, так как оба подвержены деградации.
3. Приобретение данных
ПРИМЕЧАНИЕ: Общие параметры для получения данных перечислены в таблице 1.
| Параметр | Белка | Специализированный метаболит |
| Масс-старт (Da) | 1920 год | 60 лет |
| Массовый энд (Da) | 21000 год | 2700 г. |
| Массовое отклонение (Da) | 1900 г. | 50 лет |
| Выстрелов | 500 | 1000 г. |
| Частота (Гц) | 2000 год | 2000 год |
| Размер лазера | Большой | Средний |
| MaxStdDev (ppm) | 300 г. | 30 год |
Таблица 1.
- Следуя протоколам, специфичным для используемого инструмента, налаждены как белковые, так и специализированные калибровки метаболита.
- Проверьте несколько отдельных целевых пятен, чтобы определить оптимальную лазерную мощность и увеличение детектора для использования при приобретении спектра (это будет меняться изо дня в день и по прибору).
ПРИМЕЧАНИЕ: Рисунок 2A и Рисунок 3A показывают оптимальные спектры, в то время как рисунок 2D и рисунок 3D являются примерами некачественных спектров.
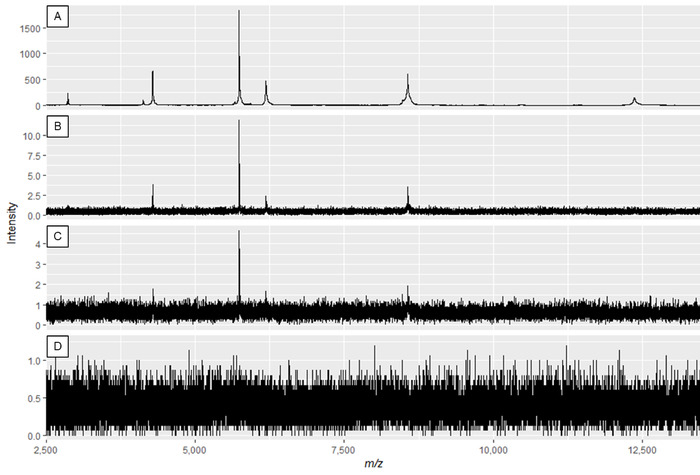
Рисунок 2: Пример белковых спектров, отображающих эффект изменения лазерной мощности и увеличения детектора. Качество спектра лучше всего в панели, и уменьшается до недостаточного качества спектров в панелях C и D. В то время как спектр в панели B может привести к использованию пиков, панель А отображает оптимальные данные. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
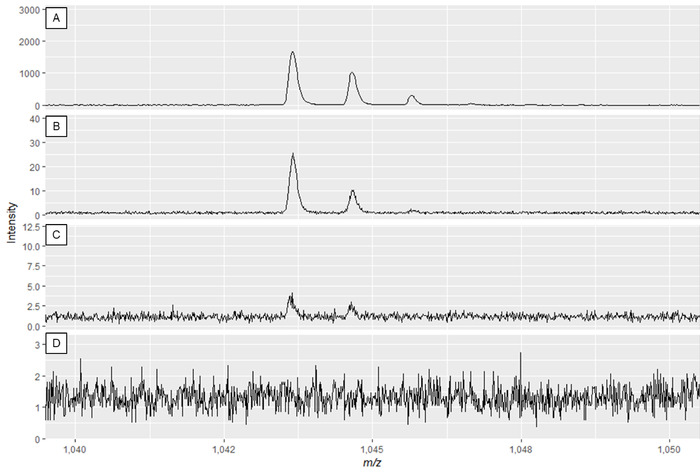
Рисунок 3: Пример специализированных метаболитных спектров, отображающих эффект изменения лазерной мощности и увеличения детектора. Качество спектра лучше всего в панели И уменьшается до недостаточного качества спектров в панелях C и D. В то время как спектр в панели B может привести к использованию пиков, панель А отображает оптимальные данные. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Приобретайте спектры, сохраняя белковые спектры в одну папку и специализированные метаболитные спектры во вторую, отдельную папку.
4. Очистка целевой пластины MALDI (адаптирована из Sauer et al.7)
- Снимите таргетную пластину MALDI с держателя и промыть ацетоном.
- Вымойте с неабразивным жидким мылом, чтобы удалить протеины и липиды следов, а также мягкие бумажные салфетки / мягкую щетку для зубов.
- Промыть деионизированной водой в течение примерно 2 минут, чтобы полностью удалить мыло.
- Снотворное целевую пластину в воде (класс HPLC или выше) в течение 5 мин.
- Промыть целевую пластину водой (класс HPLC или выше).
- Промыть целевую пластину метанолом (класс HPLC или выше).
5. Установка программного обеспечения IDBac
- Скачать программное обеспечение IDBac.
ПРИМЕЧАНИЕ: Постоянные, версии резервных копирования также доступны для скачивания (см. таблицу материалов). - Дважды щелкните загруженный "Install-IDBac.exe", чтобы инициировать установку и следовать инструкциям на экране.
6. Начиная с необработанных данных
ПРИМЕЧАНИЕ: Подробные объяснения и инструкции каждого шага обработки данных встроены в IDBac, однако основные анализы и интерактивные входы описаны ниже.
- Дважды щелкните ярлык рабочего стола IDBac для запуска IDBac. IDBac откроется на вкладке Введение по умолчанию.
- Используйте кнопку «Проверка обновлений», чтобы убедиться, что используется самая актуальная версия IDBac (требуется доступ в Интернет). Если доступна новая версия, IDBac автоматически загрузит и установит обновление, после чего IDBac запросит перезапустить его.
- Нажмите на вкладку «Начиная с необработанных данных» и выберите из меню тип данных, которые будут использоваться с IDBac; продолжить, следуя инструкциям в приложении.
- При настройке преобразования и обработки файлов данных вввейте описательное имя для эксперимента, где это вызвано (см. рисунок4). Эксперименты позже будут отображаться в алфавитном порядке, поэтому полезной стратегией является начало эксперимента с групповым атрибутом (например, "бациллы-испытания-эксперимент-1"; "бациллы-испытания-эксперимент-2").
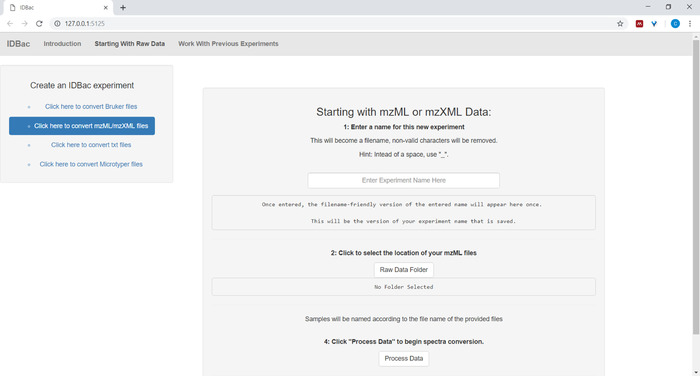
Рисунок 4: преобразование данных IDBac и этап предварительной обработки. IDBac преобразует сырые спектры в открытый формат mzML и хранит mzML, пиковые списки и выборочную информацию в базе данных для каждого эксперимента. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
7. Работа с предыдущими экспериментами
- После преобразования файлов и обработки их idBac, или в любое время, когда кто-либо хочет повторно проанализировать эксперимент, перейти к работе с предыдущими экспериментами страницы и выберите эксперимент для работы с (Рисунок 5).
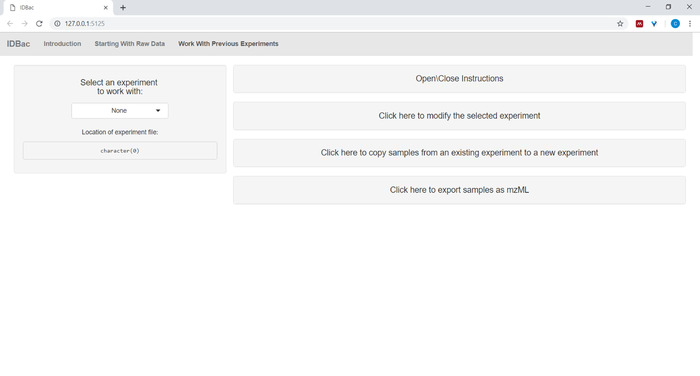
Рисунок 5: Страница "Работа с предыдущими экспериментами". Используйте страницу IDBac «Работа с предыдущими экспериментами», чтобы выбрать эксперимент для анализа или изменения. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- (Необязательно) Добавить информацию о образцах с помощью меню Нажмите здесь, чтобы изменить выбранный эксперимент. Ввод информации в автоматической заселенной электронной таблицы и нажмите Сохранить (Рисунок6). Эта опция позволяет пользователю цветового кода данных во время анализа.
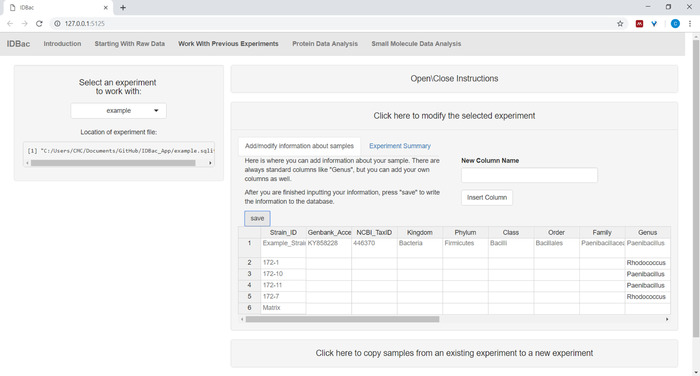
Рисунок 6: Информация о входных образцах. В рамках страницы "Работа с предыдущими экспериментами" пользователи могут ввести информацию о образцах, таких как таксономическая идентичность, расположение сбора, условия изоляции и т.д. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- (Необязательно) Перенесите все или подмножество образцов на новый или другой эксперимент, нажав на образцы передачи из предыдущих экспериментов на новые/другие эксперименты и следуя предоставленным инструкциям (рисунок7).
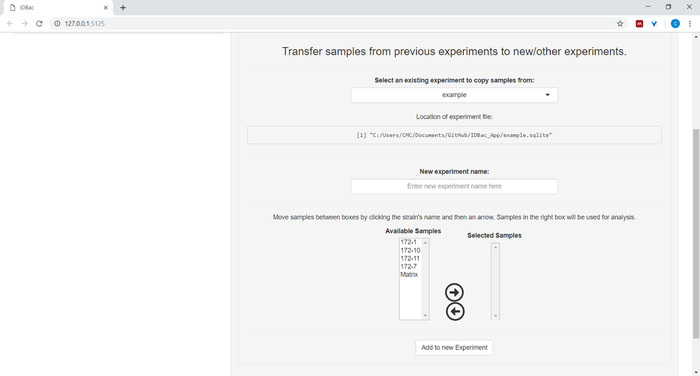
Рисунок 7: Данные о передаче. Страница "Работа с предыдущими экспериментами" содержит возможность передачи данных между существующими экспериментами и новыми экспериментами. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Когда готовы начать анализ, убедитесь, что эксперимент для работы с выбран. Выберите либо анализ данных о белках, либо анализ данных малыхмолекул.
8. Настройка анализа белковых данных и создание зеркальных участков
- При анализе белковых данных сначала перейдите на страницу анализа данных о белках. Выберите настройки пика и оцените белковые спектры образцов с помощью отображаемых зеркальных участков(рисунок8).
ПРИМЕЧАНИЕ: На зеркальных участках красный пик означает наличие этого пика только в верхнем спектре, в то время как синие пики представляют те, которые происходят в обоих спектрах.
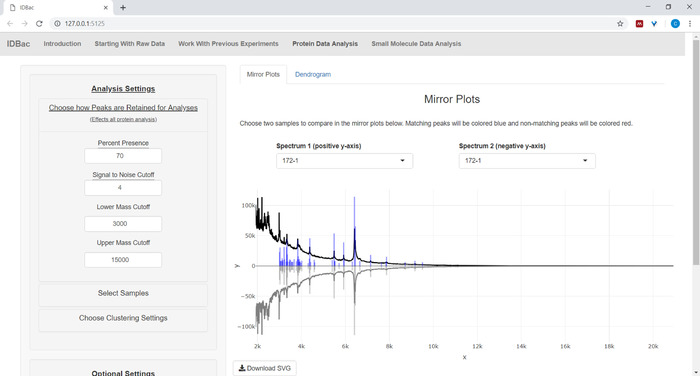
Рисунок 8: Выберите, как сохраняются пики для анализа. После выбора эксперимента для анализа, посещение страницы "Анализ данных о белках" и последующее открытие меню "Выбрать, как пики сохраняются для анализа" позволяет пользователям выбирать настройки, такие как соотношение сигнала к шуму для сохранения пиков. Отображаемый участок зеркала (или дендрограмма) будет автоматически обновляться, чтобы отразить выбранные настройки. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Отрегулируйте процент репликатов, в которых должен присутствовать пик, чтобы он был включен для анализа (например, если порог установлен на уровне 70% и пик наступает по крайней мере в 7 из 10 реплик, он будет включен).
- Используя зеркальные сюжеты в качестве визуального наведения, отрегулируйте сигнал на отсечение шума, которое сохраняет наиболее "подлинные" пики и наименьший шум, отмечая, что больше репликаций и более высокое значение "процентного пикового присутствия" позволит сделать выбор более низкого сигнала для отсечения шума.
- Укажите нижние и верхние м/з отсечения, диктуя диапазон значений массы в каждом спектре, которые будут использоваться в дальнейшем анализе IDBac.
9. Кластеризация образцов с использованием белковых данных
- На странице анализа данных белка выберите вкладку Dendrogram. Это позволяет группировать образцы в дендрограмму в соответствии с выбранными пользователем показателями расстояния и алгоритмами кластеризации.
- Нажмите Выберите образцы в меню и следуйте инструкциям, чтобы выбрать образцы, чтобы включить в анализы. Только образцы, содержащие белковые спектры, будут отображаться в поле «Доступные образцы» (рисунок9).
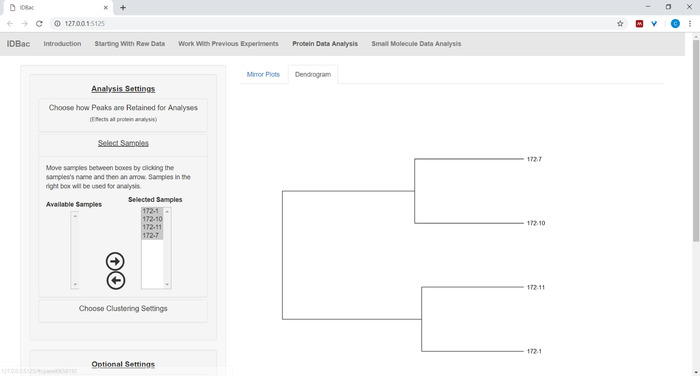
Рисунок 9: Выберите образцы выбранного эксперимента, чтобы включить в отображаемую дендрограмму. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Используйте значения по умолчанию или, в соответствии с настройками кластеризации,выберите желаемые алгоритмы расстояния и кластеризации, которые будут применены к генерации дендрограммы.
- Выберите Присутствие/Отсутствие в качестве ввода. Кроме того, если уверены в пиковых высотах образцов (например, после проведения исследования для оценки изменчивости пиковой интенсивности), выберите интенсивность в качестве ввода.
ПРИМЕЧАНИЕ: На момент публикации IDBac обеспечивает гибкость в настройках кластеризации, полагаясь на пользователей, чтобы выбрать соответствующие комбинации. Если не знакомы с этими вариантами, предлагается пара либо A: "cosine" расстояние, и "средний (UPGMA)" кластеризации; или B: "Евклидов" расстояние, и "Ward.D2" кластеризации. - Для отображения значений загрузки на дендрограмме, введите число от 2 до 1000 под Bootstraps.
- При представлении результатов копируйте текст в пункте «Предложения по анализу белка». Это обеспечивает пользовательские параметры, которые генерировали определенную дендрограмму.
10. Настройка белковой дендрограммы
- Чтобы начать настраивать дендрограмму, откройте меню dendrogram (рисунок 10).
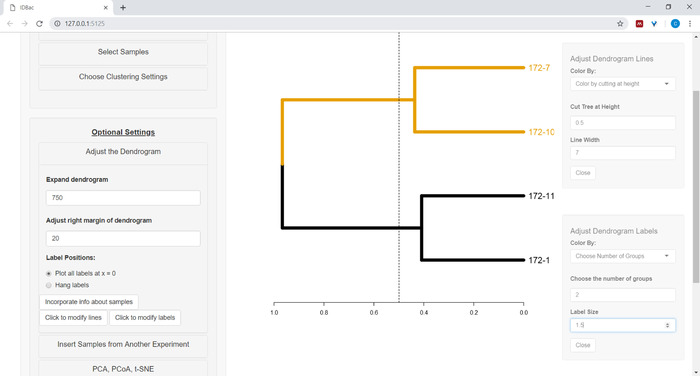
Рисунок 10: Отрегулируйте дендрограмму. IDBac предоставляет несколько вариантов для изменения, как дендрограмма выглядит, они могут быть найдены в меню "Отрегулируйте Дендрограмма". Это включает в себя окраску ветвей и этикеток k-means, или путем "резки" дендрограммы на пользовательской высоте. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Чтобы раскрасить линии dendrogram и/или метки, выберите соответствующую кнопку: нажмите, чтобы изменить линии или нажмите, чтобы изменить метки и выбрать нужные варианты.
- Чтобы участок информации из электронной таблицы рядом с дендрограммой (см. шаг 7.2, выберите кнопку Incorporate информация о образцах. Это откроет панель, где категория (столбец в таблице) будет самостоятельно заселяться на основе введенных значений(рисунок 11).
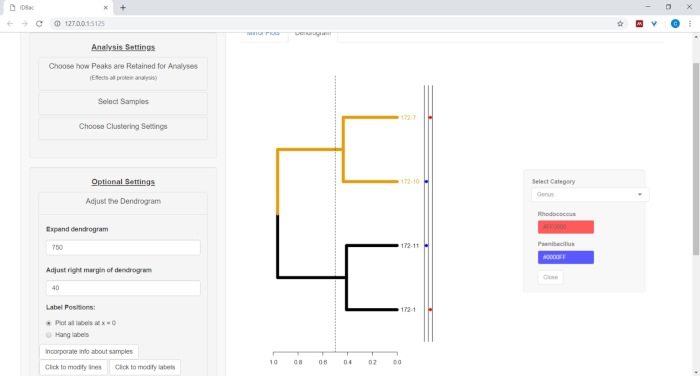
Рисунок 11: Включите информацию о образцах. В меню "Отрегулируйте Дендрограмму" есть опция "Инкорпорейд информация о образцах". Выбор этого позволит провести построение информации о образцах рядом с дендрограммой. Образец информации ввода в "Работа с предыдущими экспериментами" странице. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
11. Вставьте образцы из отдельного эксперимента в дендрограмму
- Чтобы вставить образцы из другого эксперимента, выберите кнопку меню Вставьте образцы из другогоэксперимента. Следуйте указаниям в недавно открывшейся панели(рисунок 12).
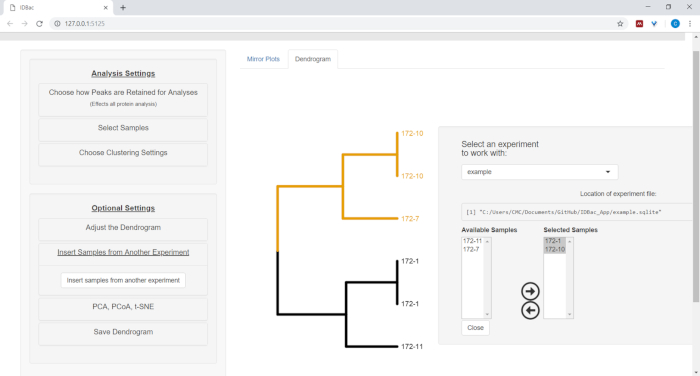
Рисунок 12: Вставьте образцы из другого меню эксперимента. Иногда полезно сравнить образцы другого эксперимента. Используйте меню "Вставьте образцы из другого эксперимента", чтобы выбрать образцы для включения в дедрограмму, отображаемую в настоящее время. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
12. Анализ специализированных метаболитных данных и сетей метаболитной ассоциации (MANs)
- Перейдите на страницу Метаболит-Ассоциации (Small-Molecule). Эта страница позволяет визуализировать данные с помощью анализа компонентов принципа (PCA) и MANs, которые используют двухсторонние сети для отображения корреляции значений малых молекул м/з с образцами.
- Если была создана белковая дендрограмма (раздел 9), она будет отображаться и на этой странице. Нажмите и перетащите дендрограмму, чтобы выделить избранные образцы интереса для анализа. Если образцы не выделены или не белка дендрограмма была создана, MAN либо случайных подмножество или все образцы будут появляться, почтительно(рисунок 13).
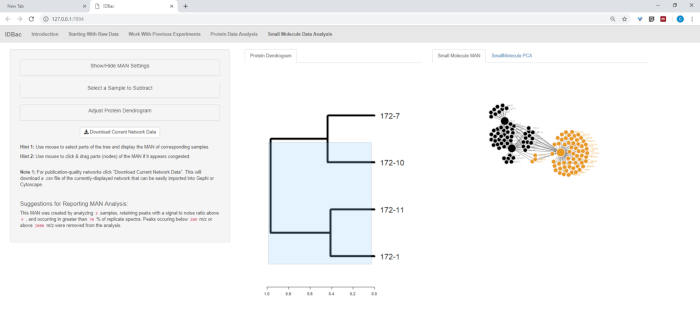
Рисунок 13: Малый молекулы анализа данных "страница. Если дендрограмма была создана из белковых спектров, она будет отображаться на странице "Малый анализ данных молекулы". На этой странице также будут отображаться метаболитные ассоциированные сети (MANs) и анализ компонентов принципа (PCA) для данных малых молекул. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Чтобы вычесть матрицу/медиа-пустоту в MAN, откройте меню Выберите образец, чтобы вычесть и выберите подходящий образец для использования в качестве пробела.
- Откройте меню Show/Hide MAN Настройки, чтобы выбрать желаемые значения для процента пикового присутствия в репликациях, сигнал к шуму, а также верхние и нижние отсечения массы, как это было сделано для белковых спектров в разделе 9. Используйте небольшие участки зеркала молекулы для руководства выбором этих параметров.
- Выберите "Загрузить текущие сетевые данные", чтобы сохранить данные MAN, которые в настоящее время отображаются. Эти данные могут быть использованы в программном обеспечении для сетевого анализа, кроме IDBac.
- Для получения отчетных результатов скопите текст в пункте «Предложения для отчета об анализе MAN». Это обеспечивает параметры, определяемые пользователем, используемые для генерации созданного MAN.
13. Обмен данными
- Каждый «эксперимент» IDBac сохраняется в виде единой базы данных S'Lite. Он содержит преобразованные mzML сырые спектры, обнаруженные пики, и все пользовательские вводимые сведения о образцах. Таким образом, для совместного участия в эксперименте IDBac просто поделитесь файлом S'lite с тем же названием, что и эксперимент (местоположение файла отображается на странице «Работа с предыдущими экспериментами»).
Результаты
Мы проанализировали шесть штаммов Micromonospora chokoriensis и два штамма Bacillus subtilis, которые ранее были охарактеризованы6, используя данные, имеющиеся в DOI: 10.5281/zenodo.2574096. Следуя указаниям в вкладке «Начало с необработанными данными», мы выбрали опцию Нажмите здесь, чтобы преобразовать файлы Bruker, и следовали инструкциям, предоставленным IDBac для каждого набора данных (рисунок14).
После завершения автоматизированной конверсии и предварительной обработки/пик-пиковых шагов, мы приступили к созданию нового комбинированного эксперимента IDBac путем переноса образцов из двух экспериментов в один эксперимент, содержащий как Bacillus, так и Образцы микромоносопора (рисунок 15). Полученный анализ включал в себя сравнение белковых спектров с использованием зеркальных участков, как показано на рисунке 16, который был полезен для оценки качества спектров и корректировки параметров пикового выбора. На рисунке 17 отображается скриншот результатов кластеризации белков с выбранными настройками по умолчанию. Дендрограмма была окрашена путем регулировки порога на участке (появляется как пунктирная линия). Следует отметить четкое разделение между родами, с м. chokoriensis и B. subtilis изолирует кластеризации отдельно.
Рисунок 18, Рисунок 19, и Рисунок 20 выделить способность генерировать MANs выбранных пользователем регионов, нажав и перетаскивая через дендрограмму белка. При этом мы смогли быстро создать MANs сравнить только штаммов B. subtilis (рисунок18), только штаммы M. chokoriensis (рисунок19), и все штаммы одновременно (Рисунок 20). Основная функция этих сетей заключается в предоставлении исследователям широкого обзора степени специализированного перекрытия метаболита между бактериями. С этими данными в руках, исследователи теперь имеют возможность принимать обоснованные решения только из небольшого количества материала Царапины из бактериальной колонии.
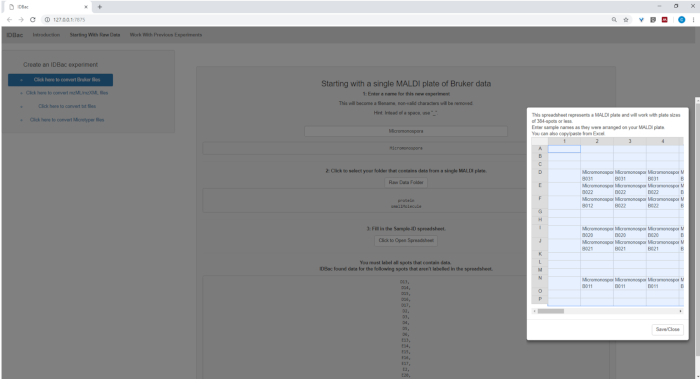
Рисунок 14: Спектробработка. Загруженные спектры Bruker autoFlex были преобразованы и обработаны с помощью IDBac. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
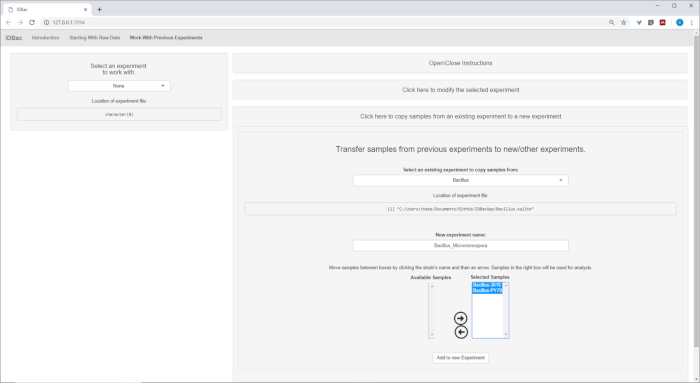
Рисунок 15: Комбинированный эксперимент IDBac. Поскольку спектры Micromonospora и Bacillus были собраны на различных целевых пластинах MALDI, эти два эксперимента были впоследствии объединены в один эксперимент - "Bacillus"Micromonsopora". Это было сделано в рамках вкладки "Работа с предыдущими экспериментами", следуя указаниям в меню "Передача образцов из предыдущих экспериментов в новые/другие эксперименты". Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
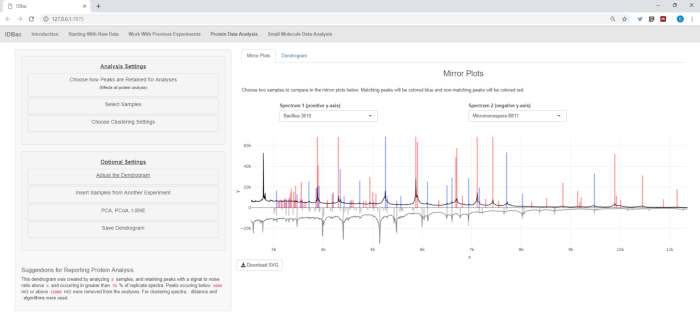
Рисунок 16: Сравнение. Микромонспора и спектры Bacillus были сопоставлены с использованием зеркальных участков на странице "Анализ данных о белках". В конечном счете были выбраны пиковые настройки по умолчанию. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
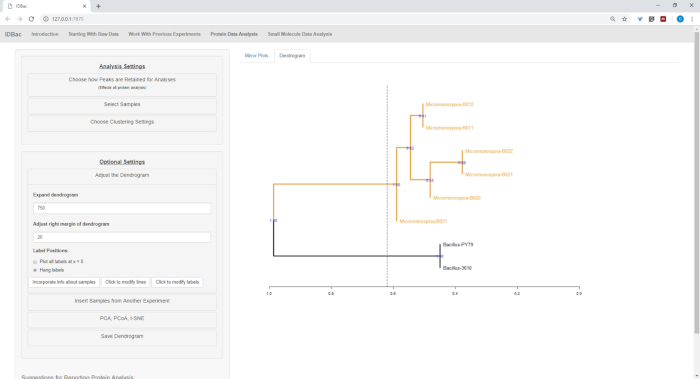
Рисунок 17: Иерархическая кластеризация. Иерархическая кластеризация с использованием параметров по умолчанию правильно сгруппировала изоляты Bacillus и Micromonospora. Дендрограмма была окрашена путем "резки" дендрограммы на произвольной высоте (отображается как пунктирной линии) и 100 загрузок, используемых, чтобы показать уверенность в ветвянии. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
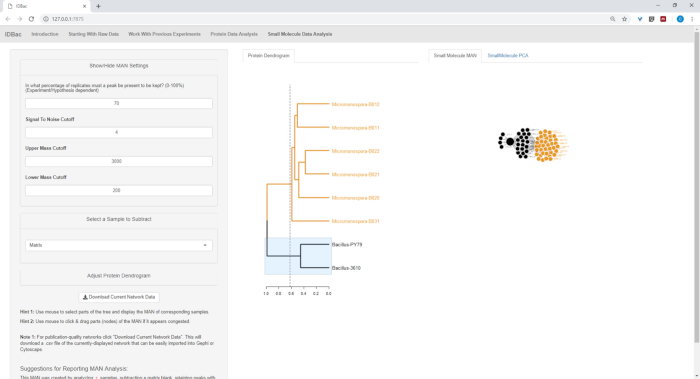
Рисунок 18: MAN, созданный путем выбора штаммов Bacillus sp. из белковой дендрограммы, показал дифференциальную выработку специализированных метаболитов. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
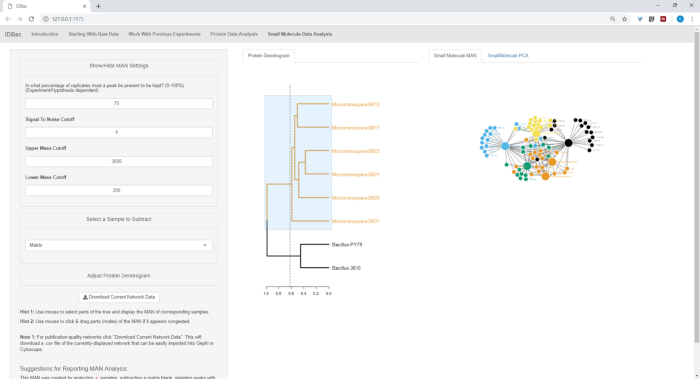
Рисунок 19: MAN, созданный путем выбора шести штаммов Micromonospora sp. из белковой дендрограммы, показали дифференциальную выработку специализированных метаболитов. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
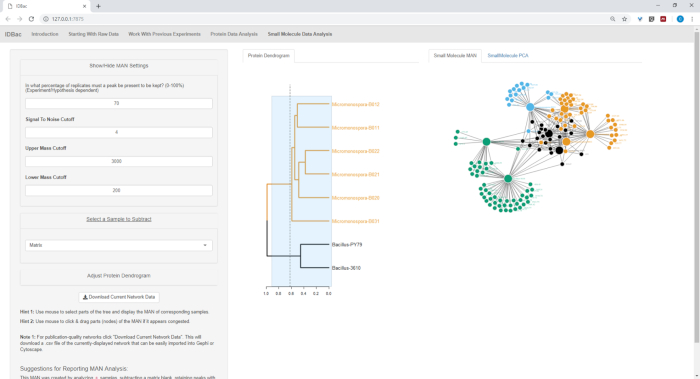
Рисунок 20: MAN Bacillus sp. и Micromonospora sp. штаммы, показывающие дифференциальную выработку специализированных метаболитов. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Обсуждение
Протокол IDBac детализирует бактериальный белок и специализированные метаболитные данные по сбору и анализу до 384 бактериальных изолятов в 4 ч одним исследователем. С IDBac нет необходимости извлекать ДНК из бактериальных изолятов или генерировать специализированные экстракты метаболита из жидкого брожения бульонов и анализировать их с помощью хроматографических методов. Вместо этого, белок и специализированные данные метаболита собираются просто распространяя материал из бактериальных колоний непосредственно на целевую пластину MALDI. Это значительно сокращает время и затраты, связанные с альтернативными методами, такимикак секвенирование генов 16S rRNA и LCMS 9.
Важно добавить в пластину MALDI матричные пустые и калибровочные пятна, и мы рекомендуем использовать соответствующее количество репликантов для обеспечения воспроизводимости и статистической достоверности. Количество репликатов будет зависеть от экспериментов. Например, если пользователь намеревается отличить тысячи колоний от коллекции тарелок экологического разнообразия, может потребоваться меньше репликаторов (наша лаборатория собирает три технических реплика на одну колонию). Кроме того, если пользователь хочет создать пользовательскую базу данных штаммов от конкретных бактериальных таксонов для быстрого определения подвидов классификаций неизвестных изолятов, то больше репликаций являются целесообразными (наша лаборатория собирает восемь биологических репликаторов на деформации).
IDBac является инструментом для быстрой дифференциализации высоко связанных бактериальных изолятов на основе ожидаемой таксономической информации и специализированного производства метаболитов. Он может дополнять или служить предшественником ортогональных методов, таких как углубленный генетический анализ, исследования, связанные с производством и функцией метаболита, или характеристика специализированной структуры метаболита с помощью ядерной магнитно-резонансной спектроскопии и/или LC-MS/MS.
Специализированное производство метаболитов (IDBac MANs) очень восприимчиво к бактериальным условиям роста, особенно с использованием различных носителей, что является потенциальным ограничением метода. Однако эти черты могут быть использованы пользователем, так как IDBac может легко генерировать MANs, показывающие различия в специализированном производстве метаболитов в различных условиях роста. Важно отметить, что в то время как специализированные метаболитовые отпечатки пальцев могут варьироваться в зависимости от состояния роста, мы ранее показали, что отпечатки белка остаются относительно стабильными по этим переменным (см. Clark et al.6). При работе с тарелость разнообразия окружающей среды, мы рекомендуем очищать бактериальные изоляты до анализа, чтобы уменьшить возможные вклады от соседних бактериальных перекрестных разговоров.
Наконец, отсутствие поисковой общедоступной базы данных отпечатков пальцев белка MS является одним из основных недостатков в использовании этого метода для классификации неизвестных экологических бактерий. Мы создали IDBac с учетом этого и включили автоматизированное преобразование данных в общепринятый формат с открытым исходным кодом (mzML)10,11,12 и разработали программное обеспечение, позволяющее осуществлять поиск, обмен и создание пользовательские базы данных. Мы находимся в процессе создания большой публичной базы данных (10 000 полностью охарактеризованных штаммов), которая позволит классифицировать некоторые изоляты на уровне видов, включая ссылки на номера присоединений GenBank при наличии.
IDBac является открытым исходным кодом, и код доступен для всех, чтобы настроить свои потребности анализа данных и визуализации. Мы рекомендуем пользователям проконсультироваться с обширным объемомлитературы (Sauer et al. 7, Silva et al.5),чтобы помочь поддержать и разработать свои экспериментальные цели. Мы проводим форум для обсуждения по адресу: https://groups.google.com/forum/#!forum/idbac и средства для сообщения о проблемах с программным обеспечением по адресу: https://github.com/chasemc/IDBacApp/issues.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана Национальным институтом общих медицинских наук Грант R01 GM125943, National Geographic Грант CP-044R-17; Исландский исследовательский фонд Грант 152336-051; и Иллинойский университет в Чикаго стартап-фондов. Кроме того, мы благодарим следующих участников: д-р Аманда Булман за помощь в приобретении белка MALDI-TOF MS; Д-р Терри Мур и д-р Атул Джайн для рекристаллизации альфа-циано-4-гидроксициннамической кислотной матрицы (CHCA).
Материалы
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
Ссылки
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены