A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
طريقة عالية الإنتاجية في الموقع لتقدير البليدي النووي الكبدي في الفئران
In This Article
Summary
نحن نقدم طريقة قوية وفعالة من حيث التكلفة ومرنة لقياس التغيرات في عدد الكبد وploidy النووية داخل عينات الأنسجة الثابتة / cryopreserved التي لا تتطلب قياس خلايا التدفق. نهجنا يوفر توقيع عينة قوية على نطاق واسع من علم خلايا الكبد مثالية لتتبع تطور إصابات الكبد والمرض.
Abstract
عندما يصاب الكبد، تنخفض أعداد خلايا الكبد، في حين أن حجم الخلية وحجمها النووي وزيادة البوليد. التوسع في الخلايا غير parenchymal مثل cholangiocytes، الخلايا العضلية، السلف والخلايا الالتهابية تشير أيضا إلى تلف الكبد المزمن، وإعادة عرض الأنسجة وتطور المرض. في هذا البروتوكول، نصف نهج بسيط عالي الإنتاجية لحساب التغيرات في التركيب الخلوي للكبد المرتبطة بالإصابة والأمراض المزمنة والسرطان. نعرض كيف يمكن استخدام المعلومات المستخرجة من أقسام الأنسجة ثنائية الأبعاد (2D) لقياس ومعايرة البوسيتي النووي الكبدي داخل عينة وتمكين المستخدم من تحديد موقع مجموعات فرعية محددة داخل الكبد في الموقع. تتطلب طريقتنا الوصول إلى مواد الكبد الثابتة /المجمدة، وكواشف الكيمياء المناعية الأساسية وأي منصة تصوير قياسية عالية المحتوى. وهو بمثابة بديل قوي لتقنيات قياس الخلايا القياسية للتدفق ، والتي تتطلب تعطيل الأنسجة التي تم جمعها حديثًا ، وفقدان المعلومات المكانية والتحيز المحتمل للتصنيف.
Introduction
يمكن أن تخضع خلايا الكبد في الكبد الثديي للخلايا المتوقفة لإنتاج خلايا ثنائية النووي ، وتكرار الحمض النووي لإنتاج نواة متعددة البلوية تحتوي على ما يصل إلى محتوى الحمض النووي 16N. زيادة إجمالية الخلوية والنووية في البوليد خلال تطور ما بعد الولادة، والشيخوخة واستجابة للضغوط الخلوية المتنوعة1. عملية تعدد البلح ديناميكية وعكسها2، على الرغم من أن وظيفتها البيولوجية الدقيقة لا تزال غير واضحة3. ويرتبط زيادة ploidy مع انخفاض القدرة التكاثرية4،والتنوع الوراثي2،والتكيف مع الإصابة المزمنة5 وحماية السرطان6. التعديلات بوكريات يبيدي تحدث نتيجة لإيقاع circadianالمعدلة 7, والفوط8. أبرزها, يتم تغيير الملف الجانبي بليفي للكبد من قبل الإصابة والمرض9,وتشير أدلة دامغة إلى أن تغييرات محددة ploidy, مثل زيادة ≥ 8N نواة أو فقدان خلايا الكبد 2N, توفير توقيعات مفيدة لتتبع مرض الكبد الدهني غير الكحولية (NAFLD) تطور3,,10,أو التأثير التفاضلي للعدوى الفيروسية11.
بشكل عام، ترتبط إصابة الكبد وتجديد مع زيادة حجم خلايا الكبد والمنطقة النووية12،جنبا إلى جنب مع انخفاض الأعداد الإجمالية من خلايا الكبد، ولا سيما تلك التي مع محتوى الحمض النووي 2N10،,11. كما أن إصابة Parenchymal في الكبد كثيرا ما يرافقه امكانية توسع الخلايا غير parenchymal (NPCs) ، بما في ذلك الخلايا العضلية سترومال ، والخلايا الالتهابية وخلايا السلف الكبد ية القدرات. طرق عالية الإنتاجية التي توفر لمحة كمية من الخلايا من عدد الخلايا parenchymal وploidy النووية، في حين المحاسبة أيضا للتغيرات في NPCs، وبالتالي لديها إمكانات كبيرة كأدوات بحثية وسريرية لتتبع استجابة الكبد أثناء الإصابة والمرض. مقنعة في الآونة الأخيرة تحليل في الموقع من أطياف ploidy في عينات بشرية من سرطان الكبد الخلوي أيضا تبين أن يتم زيادة كبيرة في البلبيدية النووية داخل الأورام ويتم تضخيمها على وجه التحديد في الأنواع الفرعية للورم أكثر عدوانية مع انخفاض التمايز وفقدان TP5313. وبالتالي، هناك احتمال قوي بأن يساعد التقدم المنهجي في التقييم الكمي للبوليدي النووي في التنميط التكهني لسرطان الكبد في المستقبل.
في هذا البروتوكول ، يتم وصف منهجية مرنة عالية الإنتاجية للتحليل المقارن لأقسام أنسجة كبد الماوس ، والتي توفر التنميط الخلوي التفصيلي لأرقام الخلايا الكبدية ، واستجابة المجلس الوطني للصحافة وطريقة معايرة داخليًا لتقدير ploidy النووي(الشكل 1). وتتميز خلايا الكبد عن الخلايا النووية من قبل عامل الكبد النووي 4 ألفا (HNF4α) وضع العلامات المناعية، قبل توصيف حجم نووي وقياس مورفومات النووية. يتم تقدير "الحد الأدنى من محتوى الحمض النووي" لجميع الأقنعة النووية الدائرية عن طريق دمج كثافة هويشت 33342 (وكيل لكثافة الحمض النووي) مع الحجم النووي ثلاثي الأبعاد (ثلاثي الأبعاد) المُبلغ به. ثم يتم معايرة الحد الأدنى من محتوى الحمض النووي الكبدي باستخدام NPCs لتوليد ملف تعريف ploidy النووية.
يتم إجراء الحصول على الصور والتقسيم النووي وتحليل الصور باستخدام التصوير عالي المحتوى ، مما يتيح فحص مساحات كبيرة من أقسام الكبد ثنائية الأبعاد (2D) التي تحتوي على عشرات الآلاف من الخلايا. يتم توفير برنامج مكتوب خصيصًا للمعالجة الآلية لبيانات تحليل الصور عالية المحتوى لإنتاج ملف تعريف بويدي على نطاق العينة لجميع نواة الكبد الدائرية. يتم تنفيذ هذا باستخدام البرمجيات مجانا لتحميل لحساب ploidy النووية على أساس تحليل الصور ستيريولوجي (SIA)10،11،14،15. وقد تم التحقق من منهجية SIA سابقا عن طريق قياس الخلايا التدفق كطريقة دقيقة، وإن كانت شاقة، لتقدير الكبد النووي ploidy في الكبد14،على افتراض مورفولوجيا نووية دائرية وعلاقة رتيبة بين الحجم النووي ومحتوى الحمض النووي. وفي هذا البروتوكول، تقاس كلا البارامترات النووية بتقييم قياس المورفولوجيا النووية ووضع العلامات على هويشت 33342. ويتبع حساب "الحد الأدنى من محتوى الحمض النووي" لكل قناع نووي معايرة للبوليد النووي الكبدي باستخدام مصادر القدرة النووية، التي لها محتوى معروف للحمض النووي 2-4N وبالتالي فهي بمثابة رقابة داخلية مفيدة.
وبالمقارنة بأساليب قياس الخلايا التقليدية في التدفق16، يتيح النهج الموصوف تقييم البوسيتي النووي الكبدي في الموقع ولا يتطلب الوصول إلى الأنسجة الطازجة أو أساليب التصنيف التي يمكن أن تحيّز النتائج ويصعب توحيدها. وكما هو الحال مع جميع النهج القائمة على المبادرة، فإن الفئات الفرعية النووية الخالية من المواد النووية > 2N ممثلة تمثيلاً ناقصاً بأخذ العينات من الأبعاد الثانية بسبب تقسيم النوى الأكبر خارج المستوى الاستوائي. كما يصف الملف الشخصي على نطاق الأنسجة الحد الأدنى من محتوى الحمض النووي لجميع الأقنعة النووية المنتقلة للكبد، ولا يميز بشكل مباشر بين خلايا الكبد أحادية النووي والخلايا النووية الثنائية التي تحتوي على نويتين منفصلتين ("غير مؤثرة") من نفس البوسيتي. ومع ذلك، فإن بساطة هذا البروتوكول تسمح بتكييف نطاق كبير لمراعاة بارامترات إضافية مثل التباعد بين النواة أو تحليل محيط الخلايا، من شأنها أن تسهل تحديد الخلايا النووية الثنائية التي توفر تقييمًا أكثر تفصيلاً للخلايا الخلوية.
Access restricted. Please log in or start a trial to view this content.
Protocol
وقد سبق أن وافقت لجنة الأخلاقيات التابعة للمؤسسة على جميع التجارب على الحيوانات. تم إيواء الفئران في منشأة خالية من مسببات الأمراض في مركز الاستثمار Príncipe فيليبي (فالنسيا، إسبانيا)، مسجلة كمربي الحيوانات التجريبية، والمستخدم، ومركز العرض (reg. لا. ES 46 250 0001 002) بموجب اللوائح الأوروبية والإسبانية الحالية لرعاية الحيوان (RD 53/2013).
1. حصاد الأنسجة وإعداد العينة
ملاحظة: يصف هذا البروتوكول كيفية تجميد الأنسجة دون التثبيت المسبق أو الحفظ بالتبريد. بالنسبة للعينات الثابتة السابقة/المحفوظة بالتبريد، تنتقل إلى القسم 2 وتحذف الخطوة 3-1. وقد أجريت جميع التحليلات باستخدام فئران أنثى بالغة تبلغ من العمر 12-16 أسبوعاً.
- التضحية بالحيوانات عن طريق حقن الفنتانيل / بنتوباربيتال داخل البيريبيتونية تليها خلع عنق الرحم. مع الماوس التي تواجه الجانب البطني لأعلى، وفتح تجويف البطن وفضح الكبد عن طريق فهم الجلد مع ملاقط وإجراء شق عمودي من قاعدة أسفل البطن إلى قاعدة القص باستخدام مقص الجراحية.
- إزالة المرارة بعناية باستخدام ملاقط غرامة، تشريح الكبد وشطف فصات الكبد المحدد في طبق 10 سم بيتري مليئة المالحة الفوسفات المخزنة (PBS).
ملاحظة: من المستحسن مقارنة نفس فص الكبد لكل الحيوان، في هذه الحالة تم استخدام الفص المتوسط. - ملء cryomold وصفت مع درجة حرارة القطع الأمثل (أكتوبر) المتوسطة في درجة حرارة الغرفة (RT). تجنب فقاعات أكتوبر. إذا ظهرت، ادفعها إلى حافة القالب باستخدام إبرة أو طرف ماصة.
- تضمين فص الكبد في cryomold OCT شغلها ووضعها على الفور على الجليد الجاف لضمان التجميد السريع. تخزين قوالب التبريد عند -80 درجة مئوية حتى تشريح التبريد.
2. التبريد
- نقل قوالب التبريد على الجليد الجاف لتجنب تدهور الأنسجة. قبل الاعتدال في قسم التبريد داخل cryostat تعيين إلى -20 درجة مئوية لمدة 20 دقيقة.
- إخراج العينة عن طريق الضغط على قاعدة cryomold البلاستيك. تطبيق السائل أكتوبر لقرص عينة دافئة في RT، والموقف في cryostat وإرفاق عينة الكبد جزءا لا يتجزأ من أكتوبر. تطبيق ضغط لطيف وانتظر 3 دقيقة لأكتوبر لتجميد ضمان العصي عينة على القرص.
ملاحظة: تجنب التعامل مع العينة بالأصابع قدر الإمكان للتهرب من تدهور الأنسجة. - قفل العينة في ذراع cryostat وضبط الاتجاه بحيث حافة العينة موازية مع شفرة cryostat. يُقطّع إلى العينة حتى يتم الوصول إلى الأنسجة.
- قسم العينة بسماكة 6 ميكرومتر. ضع شريحة مطلية بالبولي أميد على العينة لمدة 5 ق للسماح لعصا العينة على الشريحة. ضع الشريحة في RT لمدة 3−5 دقيقة، ثم، للحصول على أفضل النتائج، انتقل مباشرة إلى القسم 3.
ملاحظة: لمعالجة العديد من العينات الطازجة المجمدة تم الحصول على نتائج قابلة للاستنساخ عن طريق تخزين الشرائح مؤقتاً في صندوق شرائح على الجليد الجاف حتى تتم معالجة جميع العينات. عند استخدام هذا الأسلوب تسمح كافة الشرائح للتوازن إلى RT قبل الانتقال إلى القسم 3. يمكن استخدام عينات الفورمين الثابتة الرسمية (FFPE) ، على الرغم من زيادة autofluorescence الخلفية بهذه الطريقة. للشروع في عينات FFPE، القسم في 4 ميكرون جبل عن طريق اصطياد أقسام من حمام مائي 40 درجة مئوية على الشرائح المعالجة بالبولي أميد. الشرائح الحرارة لمدة 1 ساعة في 60 درجة مئوية، ثم deparaffinize بواسطة السحمة RT المسلسل (5 دقيقة) في الجرار كوبلين التي تحتوي على xylene (x2)، الإيثانول 100٪ (x2)، 96٪ (x2)، 70٪ (x1) و dH2O (x1). لفضح الشرائح مكان المستضدات في المخزن المؤقت التدرجات لمدة 20 دقيقة في 90 درجة مئوية قبل تخفيف الشرائح في برنامج تلفزيوني في RT. انتقل إلى الخطوة 3.2.
3- وضع العلامات المناعية للفلورسينس
- إصلاح أقسام الأنسجة في غطاء الدخان من خلال تطبيق 1 مل من 4٪ من بارافورمالديهايد (PFA) في برنامج تلفزيوني لمدة 10 دقيقة في RT. نقل الشرائح إلى جرة كوبلين مملوءة PBS ويغسل لمدة 3 دقيقة باستخدام الانفعالات لطيف (تكرار 3x).
ملاحظة: من الآن وحتى نهاية عملية المناعة، تجنب تجفيف العينة. - تجفيف المنطقة حول كل قسم من الأنسجة وتحيط باستخدام قلم كاره للماء. Permeabilize مع 0.5٪ السطحي غير الأيونية (أي تريتون X-100) في برنامج تلفزيوني لمدة 15 دقيقة في RT. ثم يغسل في PBS شغل جرة كوبلين لمدة 3 دقيقة باستخدام التحريض لطيف (كرر 2x).
- كتلة باستخدام محلول تصفية من 1٪ الزل مصل البقري (BSA), 5% مصل الخيل, 0.2% السطحي غير الأيونية في برنامج تلفزيوني (لمدة 1 ساعة على الأقل في RT).
- احتضان مع الأجسام المضادة HNF4α الأولية المخفف في منع العازلة على مدى الليل في 4 درجة مئوية في غرفة تلطيخ رطبة داكنة (انظر جدول المواد للأجسام المضادة والتخفيفات محددة).
- وضع الشرائح في جرة كوبلين مملوءة PBS ويغسل لمدة 3 دقيقة باستخدام الانفعالات لطيف (كرر 4x).
- احتضان مع اليكسا-488 اقتران الأجسام المضادة الثانوية وهويشت المخفف في تصفية 1٪ BSA و 0.2٪ السطحي غير ionic في PBS لمدة 2 ساعة في RT في غرفة تلطيخ رطبة داكنة (انظر جدول المواد للأجسام المضادة والتخفيفات محددة).
- وضع الشرائح في جرة كوبلين مملوءة PBS ويغسل لمدة 3 دقيقة باستخدام الانفعالات لطيف (كرر 4x). يغسل في ddH2O لمدة 3 دقيقة باستخدام الانفعالات لطيف (كرر 2x).
- جبل الشرائح عن طريق وضع اثنين من قطرات من الوسائط الفلورية تصاعد على coverslip (24 × 60 ملم) ووضع الشرائح أكثر من ذلك ، والقضاء على فقاعات عن طريق تطبيق ضغط لطيف. للتخزين على المدى الطويل، ختم غطاء على حواف مع طلاء الأظافر واضحة وتخزينها في الظلام في 4 درجة مئوية.
- قبل المتابعة، تحقق من الشرائح باستخدام مجهر الفلورسينس التقليدي لضمان التثبيت الجيد ووضع العلامات المناعية.
ملاحظة: راجع الشكل 2A, B للحصول على النتائج المتوقعة.
4- اكتساب صورة الفلورسينس
ملاحظة: لهذه الخطوة، مطلوب منصة تصوير عالية المحتوى(جدول المواد)التي تدعم اكتساب صورة الفلورسينس التلقائي.
- قم بتشغيل نظام التصوير وفتح بروتوكول استحواذ جديد.
- حدد الهدف 10x، لاحظ منطقة مجال الرؤية (في هذه الحالة 0.6 مم2).
- تعيين معلمات للحصول على صور الفلورسينس باستخدام مرشحات الإثارة والانبعاثات المناسبة (وفقا للخطوة 3.6). بالنسبة لقنوات Hoechst و Alexa-488 ، حدد قنوات "DAPI" و "GFP" مع 390/18 و 438/24 نانومتر وإثارة و432.5/48 و 475/24 نانومتر الانبعاثات ، على التوالي.
- ركز العينة وتأكد من أن كثافة الإشارة غير مشبعة. تأكد من أن التقاط الصور يتم بنفس وقت التعرض لجميع الصور أو استخدام نظام حيث يتم تصحيح شدة الفلورسآن لوقت التعرض.
- مسح العينة والحصول على صور كافية للحصول على تغطية كاملة لقسم الأنسجة (حوالي 20-50 حقل رؤية، اعتماداً على حجم العينة).
- مراجعة قاعدة بيانات الصور، والقضاء يدوياً على (1) الحقول سيئة التركيز، و(2) تلك الموجودة على حدود كل قسم من أقسام الأنسجة (لتجنب حسابات كثافة الخلايا المتحيزة)، و(3) تلك التي تحتوي على مناطق مطوية/متضررة مادياً من قسم الأنسجة إذا كانت موجودة.
5. تحليل صورة الفلورسينس الآلي
ملاحظة: تتطلب هذه الخطوة برامج تحليل الصور المناسبة(جدول المواد)قادرة على: (1) تحديد النوى الموسومة تلقائيًا في صور بسرعة 405 نانومتر (تجزئة نووية)، (2) تقييم الكثافة النووية الهويشت وقياس مورفومتر، و(3) تحليل العتبة لتحديد +/- حالة الفلور النووية عند 488 نانومتر (HNF4α). مطلوب بعض التدريب الأساسية المشغل / الخبرة لتقييم بصريا وضبط المعلمات تجزئة وعتبة داخل البرنامج لضمان أن النوى وHNF4α + / - حالة بوابة الأمثل(الشكل 2).
- في برنامج تحليل الصور، افتح ملف الاستحواذ الذي يحتوي على صور Hoechst (405 نانومتر) وHNF4α (488 نانومتر) من الخطوة 4.5، وأنشئ بروتوكول تحليل جديد.
- تحديد الأطوال الموجية التي ستستخدم للتجزئة النووية (Hoechst, 405 نانومتر) وتحليل عتبة الكبد/المجلس الوطني للصحافة (HNF4α, 488 نانومتر).
- تعديل معلمات تجزئة البرنامج النووي (مثل "الحد الأدنى من المجال النووي" والكشف النووي "الحساسية") لضمان الفصل الأمثل بين النوى.
ملاحظة: ينبغي إعطاء الأولوية لتجزئة جيدة من خلايا الكبد على تلك التي من NPCs. ونواة الكبد مدورة بشكل مميز (نطاق حجم interquartile: 40-64 ميكرومتر2). إن نواة المجلس الوطني للصحافة، مثل نواة الإندوثيوليا الجيوب الأنفية، مسطحة/بيضاويالشكل أو غير منتظمة الشكل، وأصغر حجماً ومعبأة بشكل وثيق عموماً من تلك الموجودة في خلايا الكبد (نطاق الحجم الورتورلي: 30-43 ميكرومتر2). بالنسبة لكبد الفأر، تم استخدام الحد الأدنى من المنطقة النووية ≥ 23 ميكرومتر2 والكشف عن "حساسية" 65٪ (انظر الشكل 2C، D للحصول على النتائج المتوقعة). تحدد الحساسية كيفية التعرف على مجموعات البكسل كنواة فردية استنادًا إلى كثافتها ويجب اختبارها تجريبيًا لكل عينة يحددها المستخدم قبل المتابعة في تحليل الصور التلقائي. - تعديل كثافة العتبة عند 488 نانومتر لضمان الجاتل الأمثل لخلايا الكبد (HNF4α+) والخلايا غير البقيمة (HNF4α-).
ملاحظة: راجع الشكل 2C, D للحصول على النتائج المتوقعة. قيمة كثافة العتبة نسبية وسوف تعتمد على كفاءة تلطيخ وإعدادات الاستحواذ مثل كثافة الليزر. ولذلك ينبغي أن يكون موحدا من قبل المستخدم. استخدام الخلايا المعروفة HNF4α مثل الخلايا الانبوبية وناقلات الطيور المحيطة باعتبارها التحكم السلبي الداخلي ونواة الكبد النووي ثنائي كمرجع إيجابي للتلطيخ. اختبار معلمات التحليل باستخدام عدد صغير من الصور لضمان تجزئة نووية جيدة والفصل عتبة الكثافة قبل تطبيق معلمات التحليل على مجموعة البيانات بأكملها. - حدد المعلمات النووية التالية التي سيتم قياسها كمياً: (1) المنطقة النووية القائمة على تلطيخ هويشت (ميكرومتر2)،(2) تعني كثافة هويشت النووية (RU)، (3) عامل التمطال النووي (متوسط نسبة المحور القصير للنواة إلى المحور الطويل للنواة، حيث يكون لجسم متناظر ة بين الوسط [غير ممدود] قيمة 1، (4) Nuc 1/(عامل الشكل)، يعني مؤشر "الاستدارة" النووي المحسوب حسب محيط 2/(4ο x المنطقة). تتراوح القيم من 1 إلى ما لا نهاية ، حيث 1 هو دائرة مثالية ، (5) حالة HNF4α (موجب -1 أو سالب-0) ، و (6) إحداثيات x /y النووية استنادًا إلى "مركز الجاذبية" (cg) ، وهي طريقة لتحديد مركز الكائن من الصور ذات المقياس الرمادي بدقة بكسل فرعي.
- قم بتشغيل التحليل لكافة مجموعات البيانات العينات وتصدير البيانات الرقمية من الخطوة 5.5 إلى برنامج جداول البيانات.
6 - تحليل البيانات
ملاحظة: يمكن تنفيذ خطوة تحليل البيانات باستخدام أي برنامج جدول بيانات قياسي.
- حساب خلايا الكبد وخلايا غير الكبد.
- حساب المساحة الإجمالية لقسم الكبد الذي تم تحليله لكل عينة عن طريق ضرب عدد حقول الرؤية حسب مساحة مجال الرؤية (الخطوة 4.2).

- العمل مع ملفات جداول البيانات التي تم إنشاؤها لكل قسم الكبد، قم بتصفية البيانات عن طريق تحديد نواة HNF4α+ فقط. حساب العدد الإجمالي للنواة HNF4α + تحليلها وتقسيم هذا على المساحة الإجمالية التي تم تحليلها للحصول على متوسط كثافة الكبد لكل عينة(الشكل 2F).

- إجراء نفس الحساب للخلايا غير parenchymal عن طريق تصفية جدول البيانات للخلايا HNF4α(الشكل 2E).
- حساب المساحة الإجمالية لقسم الكبد الذي تم تحليله لكل عينة عن طريق ضرب عدد حقول الرؤية حسب مساحة مجال الرؤية (الخطوة 4.2).
- حساب توزيع حجم الكبد النووي.
- باستخدام برنامج جداول البيانات، قم بتصفية البيانات لتحديد نواة HNF4α+ فقط.
- قيم المؤامرة من المنطقة النووية في الرسم البياني(الشكل 2G). تعيين عرض سلة المهملات إلى 5 ميكرون2.
ملاحظة: يمكن تصحيح قيم التردد للمنطقة (النوى/مم2)وفقاً للخطوة 6.1.1.
- إجراء تحليل الكبد ية النووية.
ملاحظة: يتم استخدام بيانات جدول البيانات من الخطوة 5.6 لإنشاء ملف تعريف بويدي نووي لكل عينة. وقد تم أتمتة هذه العملية ويمكن تنفيذها باستخدام برنامج مكتوب مخصص متاح مجانًا للتنزيل مع المعلومات الداعمة ومجموعات البيانات التجريبية فيhttps://github.com/lukeynoon(انظرملفات تكميلية). يتم توفير التعليمات البرمجية المصدر للمستخدمين الذين يرغبون في تكييف المنهجية. وفيما يلي وصف للخوارزمية، إلى جانب إرشادات التثبيت والاستخدام. يستخدم البرنامج بيانات جداول البيانات لفصل نواة الكبد تلقائيًا إلى مجموعتين؛ (1) تلك مع نواة دائرية "بسيطة" و (2) "معقدة" النوى غير الدائرية ممثل الخلايا الثنائية النووية مع >2c ploidy. ويتم بعد ذلك حساب الحد الأدنى من محتوى الحمض النووي (وظيفة المنطقة النووية وكثافة الحمض النووي) لجميع النوى "البسيطة". ثم يقوم خطوة لاحقة بمعايرة HNF4α+ hepatocyte النووية ploidy باستخدام HNF4α- نواة كرقابة داخلية معروفة 2-4N.- تحميل وتثبيت البرامج.
- تحميل التطبيق المعبأمن: https://github.com/lukeynoon
- إطلاق MATLAB. انتقل إلى علامة التبويب APP من toolstrip، انقر فوق تثبيت التطبيق وفتح التطبيق الذي تم تنزيله يطلق عليه"Ploidy_Application.mlappinstall". ستظهر رسالة لتأكيد التثبيت الناجح.
ملاحظة: التطبيق جاهز الآن للاستخدام وسيبقى في علامة التبويب APP من toolstrip.
- تنسيق بيانات الإدخال.
ملاحظة: قبل التحليل الآلي للبليدي النووي، يجب تخزين جميع ملفات جداول البيانات التي تحتوي على بيانات تصوير عالية المحتوى (الخطوة 5.6) وتنسيقها وفقًا للإرشادات التالية.- في كل ملف بيانات تم تصديره (. XLS 97-2004 المصنف) من الخطوة 5.6، وتشمل ورقة يطلق عليها "تدابير الخلية" تحتوي على جميع البيانات المطلوبة لتحليل ploidy المبينة في الأعمدة(الشكل 3ألف). تأكد من أن تخطيط جدول البيانات بما في ذلك أسماء رؤوس الأعمدة يبقى دون تغيير عن تخطيط الشكل 3A، لأن طريقة التحليل تجد بيانات العمود الصحيحة عن طريق البحث عن هذه الأسماء (انظر مجموعات بيانات العرض التوضيحي في الملفات التكميلية للرجوع إليها). إذا كان برنامج تحليل الصور عالي المحتوى على سبيل المثال لا ينتج عمود "تدفق خفيف"(الشكل 3A)،أدخل عمود "تدفق الضوء" يدويًا في نفس الموقع، أي العمود K وملئه بالأصفار.
- لكل حالة تجريبية (على سبيل المثال، "مصاب-d14")، توفير مجموعة بيانات التحكم، والتي سيتم استخدامها لحساب الرقابة الداخلية لمعايرة 2-4N النووية ploidy (الخطوة 6.3.4.3). هنا، حدد عينات الكبد من زملاء القمامة البالغين غير المعالجين ("Control-d0"؛ الشكل 3B−D).
- للنسخ المتماثلات البيولوجية (لكل حالة)، قم بتخزين كل جدول بيانات في مجلده الخاص (كما هو الحال في الشكل 3B). قم بتسمية بادئات المجلد بشكل تدريجي، على سبيل المثال، "Sample1، Sample2، Sample3... SampleN"، وفقاً لأسماء الملفات الواردة في. وبالتالي، يجب أن يحتوي كل مجلد مجموعة بيانات (على سبيل المثال، "Control-d0") على سلسلة من المجلدات الفرعية ("Sample1"، "Sample2"، وما إلى ذلك) يحتوي كل منها على ملف جدول بيانات بنفس الاسم المقابل.
- تشغيل التطبيق.
- داخل MATLAB، قم بتشغيل "Ploidy_Application" بالنقر على الرمز داخل علامة التبويب MY APPS من toolstrip(الشكل 3C). ستظهر واجهة المستخدم الرسومية Ploidy_Application (GUI)(الشكل 3C).
- انقر فوق زر المسار للتحكم في البيانات للانتقال إلى المجلد الذي تتواجد فيه بيانات التحكم (على سبيل المثال، "Control-d0"). سيظهر مسار البيانات هذا بعد ذلك في الواجهة (على سبيل المثال، /Users/Desktop/Control-d0).
- بعد ذلك، في "بادئة المجلد" اكتب الاسم الذي سيتم إعطاؤه لملفات الإخراج (على سبيل المثال، "نموذج").
ملاحظة: يمكن تغيير هذه البادئة إلى أي نص، شريطة أن تظل المجلدات وأسماء الملفات المسماة بشكل متزايد. - انقر فوق زر المسار إلى بيانات أخرى وانتقل إلى المجلد الذي تتواجد فيه البيانات المقارنة (على سبيل المثال، "الجرح-d14"). سيظهر مسار البيانات هذا بعد ذلك في الواجهة (على سبيل المثال، /Users/Desktop/Eded-d14).
- انقر فوق تشغيل!. عند اكتمال التحليل، سيقرأ شريط الحالة "اكتمال التحليل!..".
ملاحظة: سيبلغ التطبيق، لكل عينة، عن تقسيم النوى "البسيطة" إلى ≤ 2n و2n−4n و4n−8n و8n+ من حيث الأعداد المطلقة وكنسبة مئوية من المجموع(الشكل 3D). سيتم حفظ هذه الملفات تلقائيًا في كل مجلد عينة على النحو التالي:"Count_2n.txt"Count_2n_to_4n.txt" و"Count_4n_to_8n.txt" و"Count_8n_and_higher.txt" و"Percentage_2n.txt" و"Percentage_2nto4n.txt" و"Percentage_4nto8n.txt" و"Percentage_8n_and_higher.txt". سيقوم Ploidy_Application تلقائيًا بحفظ قائمة لكل عينة ، من جميع التقديرات الفردية لخلايا الكبد "البسيطة" ونواة غير الكبد في "Ploidy_All_Hepatocytes.txt" و "Ploidy_NonHepatocytes.txt". بالنسبة لمجموعة بيانات التحكم، يحفظ الأسلوب أيضًا الحد الأدنى من عتبات محتوى الحمض النووي المحسوبة للتقسيم الطبقي للبوليدي (انظر الخطوة 6.3.4.3.7) في ملف يسمى "Normalised_Thresholds_Control". وأخيراً، سيقوم التطبيق بإنتاج مجلد لكل من عنصر التحكم وبيانات الحالة المقارنة المحددة التي تسمى "ملخص". يحتوي هذا المجلد على مجلدين فرعيين، "Ploidy" و "التقسيم الطبقي" اللذين يحتويان على متوسطات كافة العينات المقدمة(الشكل 3D).
- وصف المنهجية.
ملاحظة: يصف القسم التالي بالتفصيل المنهجية التي يستخدمها برنامج تحليل Ploidy النووي. إذا اختار المستخدم عدم استخدام التطبيق، يمكن اتباع هذه الخطوات باستخدام برنامج جدول البيانات لحساب ملف تعريف ploidy النووي يدويًا.- نوى منفصلة إلى "بسيطة" أو "معقدة" وفقا لمورفوماتري النووي.
- حساب "مؤشر دائري" لجميع النوى، يعرف بأنه "عامل الإطالة" النووي مقسومًا على "عامل Nuc 1/(شكل)"، حيث تشير قيمة 1.0 إلى دائرة مثالية.
ملاحظة: "الإطالة النووية" و "Nuc 1/(عامل الشكل)" هما مقياسان منفصلان لـ "دائرية" الجسم التي تقيّم المعايير المورفولوجية التكميلية وغير المتداخلة. يقيس الأول المحاور الطويلة والقصيرة للجسم، بينما يقارن الثاني طول محيط الجسم بطول محور المنطقة. ولتعزيز تعريف التعميم النووي المستخدم في هذا البروتوكول، تم دمج هذين القياسين في "مؤشر دائري" واحد. نهج سابق لتقدير ploidy النووية باستخدام المنهجية الموصوفة تستخدم فقط استطالة النووية17. وفي حين تم الحصول على نتائج مقبولة باستخدام هذا النهج، لاحظ المؤلفون أن "مؤشر التعميم" المركب يحسن التمييز بين النوى المختارة يدوياً من خلايا الكبد أحادية النووي وثنائي النووي (البيانات غير مبينة). - تصنيف النوى مع مؤشر دائري ≤ 0.8 بأنها "معقدة" وتلك > 0.8 بأنها "بسيطة".
- حساب "مؤشر دائري" لجميع النوى، يعرف بأنه "عامل الإطالة" النووي مقسومًا على "عامل Nuc 1/(شكل)"، حيث تشير قيمة 1.0 إلى دائرة مثالية.
- تقدير "الحد الأدنى" من محتوى الحمض النووي (م) لجميع النوى "البسيطة".
- حساب نصف القطر النووي (ص) باستخدام الصيغة:

- حساب الحجم النووي (5) باستخدام حجم صيغة الكرة:
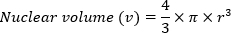
- إنشاء قيمة نسبية لمحتوى الحمض النووي الأدنى (م) باستخدام الصيغة:

- حساب نصف القطر النووي (ص) باستخدام الصيغة:
- معايرة مجموعة البيانات باستخدام نواة المجلس الوطني لنواب الشعب (HNF4α-) كتحكم داخلي 2-4N.
ملاحظة: تحتوي الـ NPCs على محتوى الحمض النووي 2-4N اعتمادًا على حالة دورة الخلية. وبالتالي ، فإن متوسط قيمة المجلس الوطني للصحابة "الحد الأدنى" من محتوى الحمض النووي (NPCm)يزيد مع الإصابة(الشكل 4A). يتم تقليل خطأ المعايرة عن طريق إنشاء حد أعلى من NPCm يمثل عتبة 4c(الشكل 4B).- ضمن جدول البيانات، حدد نواة NPC فقط مع قيم لـ "m" التي تقع ضمن انحراف معياري واحد (SD) من الوضع (هذا المرشحات من الضوضاء من خطأ التقسيم المحتملة).
- ضمن هذا النطاق الذي تمت تصفيته، قم بفحص المناطق النووية وكثافاتها المتوسطة المقابلة(الشكل 4C).
- تقدير أصغر منطقة نووية ضمن هذا النطاق الذي تمت تصفيته بأقصى كثافة نووية قصوى (أي النقطة التي يتغير فيها خط المنحنى في اتجاه مجموعة البيانات المصفاة كما يتضح من الدائرة الحمراء في الشكل 4C). تمثل هذه القيمة حالة انتقالية 2N-4N (t) فوقها التي تسود عينات من النوى 4c على نواة 2c ، مما يؤدي إلى أقصى حد من شدة Hoechst المتوسط.
ملاحظة: يتم تحديد هذه القيمة تلقائياً بواسطة البرنامج; ومع ذلك، يمكن لمستخدمي جدول البيانات تحديد هذه النقطة يدويًا كحجم انتقالي. - احسب الحد الأدنى من محتوى الحمض النووي الذي يمثله هذا الحجم الانتقالي (tm)باتباع الخطوة 6.3.4.2.
- لتقدير الكتف 4N من مجموعة بيانات NPCm، إضافة 1 SD إلى قيمة tm. الرقم الناتج(الشكل 4B)يصف الحد الأعلى من محتوى الحمض النووي الحد الأدنى من المجلس الوطني لنواب الشعب الصيني لاستخدامها في التقسيم الطبقي النووي (S4c).
- كرر الخطوات 6-3-4-3-6-3-4-3-5 لجميع عينات "التحكم".
ملاحظة: على سبيل المثال، في الشكل 3،يتم استخدام كبد التحكم غير المضرورة ("Control-d0") كشرط تحكم. - حساب متوسط عتبة التقسيم الطبقي 4c (S4c)لعينات "التحكم" واستخدام هذا لاستقراء 2c (S2c)و 8c (S8c)الحدود للحد الأدنى من محتوى الحمض النووي (م). يتم إنشاء عتبات التقسيم الطبقي تلقائيًا وتخزينها بواسطة البرنامج (الخطوة 6.3.3.3).
ملاحظة: اعتماداً على تصميم الدراسة، يمكن حساب متوسط قيم عتبة التقسيم الطبقي لكل حالة أو لظروف محددة (على سبيل المثال، الكبد التحكم الصحي). ومع ذلك، يتطلب برنامج تحليل Ploidy النووي أن يتم تعيين واحد من مجموعة من 2 ملفات كـ "تحكم" لأغراض حساب القيم ploidy النسبية. - حساب قيمة ploidy لجميع النوى باستخدام قيمة S2c ولدت في الخطوة 6.3.4.3.7 وفقا ل:

- طبقات "بسيطة" hepatocyte (HNF4α +) نواة في 2c/4c/8c/>8c الأقواس وفقا للمعايير التالية: "2c" HNF4α + = p ≤ 2; "4c" HNF4α + = 2 < p ≤ 4; "8c" HNF4α + = 4 < p ≤ 8; "> 8c" HNF4α + = 8 < p.
- لإعادة بناء النقش المكاني للمجموعات الفرعية البوليدية، قم بفصل البيانات النووية داخل كل جدول بيانات عينة وفقًا للحقول المقابلة التي تم الحصول عليها فيها. ثم استخدم إحداثيات x/y النووية المرتبطة بها (من الخطوة 5.5) لرسم المجموعات الفرعية البوليدية في 2D(الشكل 5C).
- نوى منفصلة إلى "بسيطة" أو "معقدة" وفقا لمورفوماتري النووي.
- تحميل وتثبيت البرامج.
Access restricted. Please log in or start a trial to view this content.
النتائج
وقد استخدمت هذه الطريقة لقياس تأثير الإصابة البَلْفة على كبد الفأر البالغ عن طريق تغذية الحيوانات لمدة 0-21 يوماً بنظام غذائي سام بالكبد يحتوي على 0.1% 3,5-diethoxycarbonyl-1,4-dihydrocollidine (DDC)17. وقد زادت نتائج التغذية المزمنة DDC في الإصابة الكبدية البلية توسع biportal من NPCs. يجب أ?...
Access restricted. Please log in or start a trial to view this content.
Discussion
يتم وصف نهج عالي المحتوى وعالي الإنتاجية لتحليل إعادة عرض الأنسجة وتقدير البوليد النووي الكبدي في كبد المورين. بمجرد معرفة الإجراء، يمكن للمستخدم معالجة عينات متعددة وصورتها وتحليلها في فترة 3-5 أيام، مما يؤدي إلى إنشاء مجموعات بيانات كبيرة قابلة للاختبار توفر توقيعًا مفصلًا لصحة الكبد. ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
تم تمويل هذا العمل من قبل حكومة ماينكو الإسبانية منح BFU2014-58686-P (LAN) وSAF-2017-84708-R (DJB). تم دعم الشبكة المحلية من قبل ماينكو رامون ذ كاخال الوطنية زمالة RYC-2012-11700 وجائزة خطة GenT (Comunitat Valenciana, CDEI-05/20-C), وFMN من قبل الإقليمية ValI +D الطلاب من الجنرالات فالنسيا ACIF/2016/020. ويود البرنامج أن ينوه بالبروفيسور إيوا ك. بالوش على التمويل. نشكر الدكتورة أليسيا مارتينيز روميرو (خدمة قياس الخلايا CIPF) على المساعدة في منصة IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
References
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved