Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un método in situ de alto rendimiento para la estimación de la ploideide nuclear de hepatocitos en ratones
En este artículo
Resumen
Presentamos un método robusto, rentable y flexible para medir los cambios en el número de hepatocitos y la ploidía nuclear dentro de muestras de tejido fijo/crioconservado que no requieren citometría de flujo. Nuestro enfoque proporciona una poderosa firma de citología hepática en toda la muestra ideal para rastrear la progresión de las lesiones y enfermedades hepáticas.
Resumen
Cuando el hígado se lesiona, el número de hepatocitos disminuye, mientras que el tamaño de la célula, el tamaño nuclear y la estratada aumentan. La expansión de las células no parénquimales como los colangiocitos, los miofibroblastos, los progenitores y las células inflamatorias también indican daño hepático crónico, remodelación tisular y progresión de la enfermedad. En este protocolo, describimos un enfoque simple de alto rendimiento para calcular los cambios en la composición celular del hígado que están asociados con lesiones, enfermedades crónicas y cáncer. Mostramos cómo la información extraída de las secciones de tejido bidimensional (2D) se puede utilizar para cuantificar y calibrar la ploideide nuclear de hepatocitos dentro de una muestra y permitir al usuario localizar subconjuntos específicos de ploidicia dentro del hígado in situ. Nuestro método requiere acceso a material hepático fijo/congelado, reactivos básicos de inmunocitoquímica y cualquier plataforma de imágenes estándar de alto contenido. Sirve como una poderosa alternativa a las técnicas estándar de citometría de flujo, que requieren la interrupción del tejido recién recolectado, la pérdida de información espacial y el posible sesgo de desagregación.
Introducción
Los hepatocitos en el hígado de los mamíferos pueden someterse a citoquinesis estancada para producir células binucleares, y endoreplicación de ADN para producir núcleos de poliploide que contienen hasta 16N de contenido de ADN. Aumento general de la ploideide celular y nuclear durante el desarrollo postnatal, envejecimiento y en respuesta a diversas tensiones celulares1. El proceso de poliploidización es dinámico y reversible2,aunque su función biológica precisa sigue sin estar clara3. El aumento de la ploideidey se asocia con una capacidad proliferativa reducida4,diversidad genética2,adaptación a la lesión crónica5 y protección contra el cáncer6. Las alteraciones de la estratadia de hepatocitos se producen como resultado de la alteración del ritmo circadiano7,y el destete8. En particular, el perfil de ploide del hígado se ve alterado por la lesión y la enfermedad9, y la evidencia convincente sugiere que cambios específicos de ploidey, tales como aumento de los núcleos 8N o pérdida de hepatocitos 2N, proporcionan firmas útiles para el seguimiento de la progresión delaenfermedad del hígado graso no alcohólico (NAFLD)3,10, o el impacto diferencial de las infecciones virales11.
En términos generales, la lesión hepática y la regeneración se asocian con el aumento del tamaño de las células de hepatocitos y el área nuclear12,junto con el número total reducido de hepatocitos, en particular aquellos con contenido de ADN 2N10,11. La lesión parenquimal en el hígado también se acompaña con frecuencia de la expansión de células no parénquimales (NNJ), incluyendo miofibroblastos estromales, células inflamatorias y células progenitoras hepáticas bipotentes. Los métodos de alto rendimiento que proporcionan un perfil citológico cuantitativo del número de células parénquimas y la ploidía nuclear, al tiempo que tienen en cuenta los cambios en los PNJ, tienen por lo tanto un potencial considerable como herramientas clínicas y de investigación para rastrear la respuesta del hígado durante la lesión y la enfermedad. El análisis in situ reciente conmovedor de espectros ploidy en muestras humanas de carcinoma hepatocelular también demuestra que la ploidicia nuclear aumenta dramáticamente dentro de los tumores y se amplifica específicamente en subtipos tumorales más agresivos con diferenciación reducida y pérdida de TP5313. Por lo tanto, existe una fuerte posibilidad de que los avances metodológicos en la evaluación cuantitativa de la ploidía nuclear ayuden en la elaboración de perfiles de pronóstico futuros del cáncer de hígado.
En este protocolo, se describe una metodología flexible de alto rendimiento para el análisis comparativo de secciones de tejido hepático de ratón, que proporciona perfiles citométricos detallados de los números de hepatocitos, la respuesta NPC y un método calibrado internamente para estimar la ploidía nuclear(Figura 1). Los hepatocitos se distinguen de los NNP por el inmunoetiquetado alfa del factor nuclear de hepatocitos 4 (HNF4), antes de la caracterización del tamaño nuclear y la morfometría nuclear. Se estima el "contenido mínimo de ADN" para todas las máscaras nucleares circulares integrando la intensidad media de Hoechst 33342 (un proxy para la densidad de ADN) con volumen nuclear tridimensional interpolado (3D). El contenido mínimo de ADN de los hepatocitos se calibra utilizando PNJ para generar un perfil de ploidy nuclear.
La adquisición de imágenes, la segmentación nuclear y el análisis de imágenes se realizan utilizando imágenes de alto contenido, lo que permite examinar grandes áreas de secciones hepáticas bidimensionales (2D) que contienen decenas de miles de células. Se proporciona un programa escrito a medida para el procesamiento posterior automatizado de datos de análisis de imágenes de alto contenido para producir un perfil de ploidy en toda la muestra para todos los núcleos de hepatocitos circulares. Esto se realiza utilizando software de descarga gratuita para calcular la ploidicia nuclear basada en el análisis de imágenes estereológicas (SIA)10,11,14,15. La metodología SIA ha sido previamente validada por la citometría de flujo como un método preciso, aunque laborioso, para estimar la ploidía nuclear de hepatocitos en el hígado14,asumiendo la morfología nuclear circular y una relación monotónica entre el tamaño nuclear y el contenido de ADN. En este protocolo, ambos parámetros nucleares se miden mediante la evaluación de la morfometría nuclear y el etiquetado Hoechst 33342. El cálculo del "contenido mínimo de ADN" para cada máscara nuclear es seguido por la calibración de la ploideide nuclear de hepatocitos utilizando PNJ, que tienen un contenido de ADN de 2 x 4N conocido y, por lo tanto, sirven como un control interno útil.
En comparación con los métodos convencionales de citometría de flujo16, el enfoque descrito permite evaluar in situ la ploidía nuclear de hepatocitos y no requiere acceso a métodos de tejido fresco o desagregación que puedan sesgar los resultados y ser difíciles de estandarizar. Al igual que con todos los enfoques basados en SIA, las subclases de ploidey nuclear >2N están subrepresentadas por el muestreo 2D debido a la sección de núcleos más grandes fuera del plano ecuatorial. El perfil de ploidy de todo el tejido también describe el contenido mínimo de ADN para todas las máscaras nucleares de hepatocitos circulares, y no discrimina directamente entre los hepatocitos mononucleares y las células binucleares que tienen dos núcleos discretos ("sin tocar") de la misma estratapidia. Sin embargo, la simplicidad de este protocolo permite adaptar un margen considerable para que tenga en cuenta parámetros adicionales como el espaciado internuclear o el análisis perimetral celular, que facilitarían la identificación de células binucleares que proporcionen una evaluación más detallada de la ploideideidería celular.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los experimentos con animales fueron aprobados previamente por el comité de ética del CIPF. Los ratones fueron alojados en una instalación libre de patógenos en el Centro de Investigación Príncipe Felipe (Valencia, España), registrado como criador, usuario y centro de suministro sin patógenos (reg. no. ES 46 250 0001 002) en virtud de la normativa europea y española vigente sobre bienestar animal (RD 53/2013).
1. Cosecha de tejidos y preparación de muestras
NOTA: Este protocolo describe cómo congelar el tejido sin fijación previa ni criopreservación. Para muestras previamente fijas/crioconservadas proceda a la sección 2 y omita el paso 3.1. Todos los análisis se han realizado utilizando ratones hembra adultas C57BL/6 de 12 a 16 semanas.
- Sacrificar animales por inyección intraperitoneal de fentanilo/pentobarbital seguida de luxación cervical. Con el ratón mirando hacia arriba, abra la cavidad abdominal y exponga el hígado agarrando la piel con pinzas y realizando una incisión vertical desde la base del abdomen inferior hasta la base del esternón utilizando tijeras quirúrgicas.
- Retire cuidadosamente la vesícula biliar con pinzas finas, diseccione el hígado y enjuague el lóbulo hepático seleccionado en una placa de plato Petri de 10 cm llena de solución salina con fosfato tampón (PBS).
NOTA: Se recomienda comparar el mismo lóbulo hepático para cada animal, en este caso se utilizó el lóbulo mediano. - Llene un criomold etiquetado con un medio de temperatura de corte óptima (OCT) a temperatura ambiente (RT). Evite las burbujas de OCT. Si aparecen, empújelos hasta el borde del molde con una aguja o una punta de pipeta.
- Incruste el lóbulo hepático en un criomold OCT lleno e colóquelo inmediatamente sobre hielo seco para asegurar una congelación rápida. Conservar criomoldes a -80oC hasta la criosección.
2. Criosección
- Transporte criomoldes sobre hielo seco para evitar la degradación de los tejidos. Antes de la criosección, el equilibrio en el interior del criostato se establece en -20 oC durante 20 min.
- Expulse la muestra aplicando presión sobre la base del criomold de plástico. Aplique OCT líquido al disco de muestra caliente en RT, colóquelo en criostato y adjunte una muestra de hígado incrustada de OCT. Aplique una presión suave y espere 3 minutos hasta que OCT se congele asegurando que la muestra se pegue al disco.
NOTA: Evite manipular la muestra con los dedos tanto como sea posible para evadir la degradación del tejido. - Bloquee la muestra en el brazo del criostato y ajuste la orientación para que el borde de la muestra sea paralelo a la hoja criostato. Cortar en la muestra hasta que se alcance el tejido.
- Seccione la muestra a un espesor de 6 m. Coloque un portaobjetos recubierto de poliamida etiquetado sobre la muestra durante 5 s para dejar que la muestra se pegue a la diapositiva. Coloque la diapositiva en RT durante 3 x 5 minutos, luego, para obtener mejores resultados, proceda directamente a la sección 3.
NOTA: Para el procesamiento de múltiples muestras congeladas se han obtenido resultados reproducibles almacenando temporalmente diapositivas en una caja de diapositivas sobre hielo seco hasta que todas las muestras han sido procesadas. Al utilizar este enfoque, permita que todas las diapositivas se equilibren en RT antes de continuar con la sección 3. Se pueden utilizar muestras de parafina fija de formalina (FFPE), aunque este método aumenta la autofluorescencia en segundo plano. Para proceder de las muestras de FFPE, sección a 4 m. Montar mediante la captura de secciones de baño de agua de 40 oC en toboganes tratados con poliamida. Toboganes de calor durante 1 h a 60 oC, luego desparaffinizar por lavados RT en serie (5 min) en frascos de Coplin que contienen xileno (x2), etanol 100% (x2), 96% (x2), 70% (x1) y dH2O (x1). Para exponer los antígenos, coloque las diapositivas en el tampón de citrato durante 20 minutos a 90 oC antes de templar las diapositivas en PBS en RT. Proceda al paso 3.2.
3. Inmunolacencia de fluorescencia
- Fije las secciones de tejido en una campana de humo aplicando 1 ml de 4% de paraformaldehído (PFA) en PBS durante 10 minutos en RT. Transfiera las diapositivas a un frasco de Coplín lleno de PBS y lávelo durante 3 minutos usando agitación suave (repetir 3x).
NOTA: Desde ahora hasta el final del proceso de inmunomancha, evite el secado de la muestra. - Seque el área alrededor de cada sección de tejido y rodéala con una pluma hidrófoba. Permeabilizar con tensioactivo no iónico al 0,5% (es decir, Triton X-100) en PBS durante 15 min a RT. Luego lave en frasco de Coplin lleno de PBS durante 3 minutos usando agitación suave (repetir 2x).
- Bloquear utilizando una solución filtrada de albúmina sérica bovina (BSA), 5% de suero de caballo, 0,2% tensioactivo no iónico en PBS (durante al menos 1 h en RT).
- Incubar con anticuerpos primarios HNF4 diluidos en tampón de bloqueo durante la noche a 4oC en una cámara de tinción húmeda oscura (ver Tabla de Materiales para anticuerpos y diluciones específicas).
- Coloque las diapositivas en un frasco de Coplín lleno de PBS y lave durante 3 minutos con agitación suave (repetir 4x).
- Incubar con anticuerposecundario conjugado Alexa-488 y Hoechst diluido en 1% de BSA filtrado y tensioactivo no iónico al 0,2% en PBS durante 2 horas a RT en una cámara de tinción húmeda oscura (ver Tabla de Materiales para anticuerpos y diluciones específicas).
- Coloque las diapositivas en un frasco de Coplín lleno de PBS y lave durante 3 minutos con agitación suave (repetir 4x). Lavar en ddH2O durante 3 min con agitación suave (repetir 2x).
- Monte los portaobjetos colocando dos gotas de medios de montaje fluorescentes en un cubreobjetos (24 x 60 mm) y colocando diapositivas sobre él, eliminando las burbujas aplicando una presión suave. Para un almacenamiento a largo plazo, cierre la tapa en los bordes con esmalte de uñas transparente y guárdelo en la oscuridad a 4 oC.
- Antes de continuar, compruebe las diapositivas utilizando un microscopio de fluorescencia convencional para garantizar una buena fijación e inmunoetiquetado.
NOTA: Consulte la Figura 2A,B para ver los resultados esperados.
4. Adquisición de imágenes de fluorescencia
NOTA: Para este paso, se requiere una plataforma de imágenes de alto contenido(Tabla de materiales)que admita la adquisición automática de imágenes de fluorescencia.
- Encienda el sistema de imágenes y abra un nuevo protocolo de adquisición.
- Seleccione el objetivo 10x, tenga en cuenta el área del campo de visión (en este caso 0,6 mm2).
- Establezca parámetros para adquirir imágenes de fluorescencia utilizando los filtros de excitación y emisión apropiados (según el paso 3.6). Para Hoechst y Alexa-488, seleccione los canales "DAPI" y "GFP" con excitación de 390/18 y 438/24 nm y emisiones de 432,5/48 y 475/24 nm, respectivamente.
- Enfoque la muestra y asegúrese de que la intensidad de la señal no se satura. Asegúrese de que la captura de imágenes se realiza con el mismo tiempo de exposición para todas las imágenes o utilice un sistema en el que se corrija la intensidad de la fluorescencia durante el tiempo de exposición.
- Escanee la muestra y adquiera imágenes suficientes para obtener una cobertura completa de la sección de tejido (aproximadamente 20 a 50 campos de visión, dependiendo del tamaño de la muestra).
- Revise la base de datos de imágenes, eliminando manualmente (i) los campos mal enfocados, (ii) los que están en los bordes de cada sección de tejido (para evitar sesgar los cálculos de densidad celular) y (iii) aquellos que contienen áreas plegadas/físicamente dañadas de la sección del tejido si están presentes.
5. Análisis automatizado de la imagen de fluorescencia
NOTA: Este paso requiere un software de análisis de imágenes adecuado(Tabla de Materiales)capaz de: (1) identificar automáticamente los núcleos etiquetados con Hoechst dentro de imágenes a 405 nm (segmentación nuclear), (2) evaluar la intensidad nuclear media de Hoechst y la morfometría, y (3) el análisis de umbral para determinar el estado +/- de la fluorescencia nuclear a 488 nm (HNF4). Se requiere cierta capacitación/experiencia básica del operador para evaluar y ajustar visualmente los parámetros de segmentación y umbral dentro del programa para garantizar que los núcleos y el estado de HNF4+/- estén bloqueados de manera óptima(Figura 2).
- En el software de análisis de imágenes, abra el archivo de adquisición que contiene las imágenes Hoechst (405 nm) y HNF4 (488 nm) del paso 4.5 y cree un nuevo protocolo de análisis.
- Definir las longitudes de onda que se utilizarán para la segmentación nuclear (Hoechst, 405 nm) y para el análisis del umbral de hepatocitos/NPC (HNF4, 488 nm).
- Ajuste los parámetros de segmentación nuclear del software (como "área nuclear mínima" y "sensibilidad" de detección nuclear) para garantizar que los núcleos estén segregados de forma óptima.
NOTA: Se debe priorizar una buena segmentación de los hepatocitos sobre la de los PNJ.2 Los núcleos NPC, como los de la endotelia sinusoidal, son aplanados/elípticos o de forma irregular y generalmente más pequeños y más estrechamente embalados que los de los hepatocitos (rango de tamaño intercuartil: 30-43 m2). Para el hígado de ratón, se utilizó un área nuclear mínima de 23 m2 y una "sensibilidad" de detección del 65% (ver Figura 2C,D para ver los resultados esperados). La sensibilidad determina cómo se reconocen los clústeres de píxeles como núcleos individuales en función de su intensidad y debe probarse empíricamente para cada muestra establecida por el usuario antes de continuar con el análisis automatizado de imágenes. - Modificar la intensidad del umbral a 488 nm para asegurar una gating óptima de los hepatocitos (HNF4 +) y las células no parénquimales (HNF4-).
NOTA: Consulte la Figura 2C,D para ver los resultados esperados. El valor de la intensidad del umbral es relativo y dependerá de la eficiencia de tinción y los ajustes de adquisición, como la intensidad del láser. Por lo tanto, debe ser estandarizado por el usuario. Utilice células conocidas de HNF4, como células endoteliales y PNJ periportal como un control negativo interno y núcleos de hepatocitos binucleares como referencia positiva para la tinción. Pruebe los parámetros de análisis utilizando un pequeño número de imágenes para garantizar una buena segmentación nuclear y una buena segregación del umbral de intensidad antes de aplicar parámetros de análisis a todo el conjunto de datos. - Seleccione los siguientes parámetros nucleares a cuantificar: (1) área nuclear basada en la tinción Hoechst (m2), (2) intensidad media de Hoechst nuclear (RU), (3) factor de alargamiento nuclear (relación media del eje corto del núcleo con el eje largo del núcleo, donde un objeto simétrico central [no alargado] tiene un valor de 1, (4) Nuc 1/(factor de forma), índice de "redondez" nuclear media calculado por perímetro 2/(4o x área). Los valores van de 1 a infinito, donde 1 es un círculo perfecto, (5) estado HNF4 (positivo-1 o negativo-0) y (6) coordenadas nucleares x/y basadas en "centro de gravedad" (cg), un método para localizar el centro del objeto a partir de imágenes en escala de grises con precisión de subpíxeles.
- Ejecute el análisis de todos los conjuntos de datos de ejemplo y exporte datos numéricos del paso 5.5 al software de hoja de cálculo.
6. Análisis de datos
NOTA: El paso de análisis de datos se puede realizar utilizando cualquier software de hoja de cálculo estándar.
- Calcule los números de células de hepatocitos y no hepatocitos.
- Calcular el área total de la sección hepática analizada para cada muestra multiplicando el número de campos de visión por el área del campo de visión (paso 4.2).

- Al trabajar con archivos de hoja de cálculo generados para cada sección hepática, filtre los datos seleccionando solo los núcleos HNF4+. Calcular el número total de núcleos HNF4 + analizados y dividirlo por el área total analizada para obtener la densidad media de hepatocitos para cada muestra(Figura 2F).

- Realice el mismo cálculo para las celdas no parenquimales filtrando la hoja de cálculo para las celdas HNF4-(Figura 2E).
- Calcular el área total de la sección hepática analizada para cada muestra multiplicando el número de campos de visión por el área del campo de visión (paso 4.2).
- Calcule la distribución del tamaño nuclear de los hepatocitos.
- Usando el software de hoja de cálculo, filtre los datos para seleccionar solo los núcleos HNF4 +.
- Valores de trazado del área nuclear en un histograma(Figura 2G). Establezca el ancho de la bandeja en 5 m2.
NOTA: Los valores de frecuencia se pueden corregir para el área (núcleos/mm2) según el paso 6.1.1.
- Realizar análisis de ploidey nuclear de hepatocitos.
NOTA: Los datos de hoja de cálculo del paso 5.6 se utilizan para generar un perfil de ploidy nuclear para cada muestra. Este proceso se ha automatizado y se puede realizar utilizando un software escrito personalizado que está disponible libremente para descargar con información de apoyo y conjuntos de datos de demostración enhttps://github.com/lukeynoon(verArchivos Suplementarios). El código fuente se proporciona a los usuarios que deseen adaptar la metodología. A continuación se describe una descripción del algoritmo, junto con las instrucciones de instalación y uso. El programa utiliza datos de hoja de cálculo para separar automáticamente los núcleos de hepatocitos en dos grupos; (1) aquellos con núcleos circulares "simples" y (2) núcleos no circulares "complejos" representativos de células binucleares con >2c ploidy. El contenido mínimo de ADN nuclear (una función del área nuclear y la densidad del ADN) se calcula a continuación para todos los núcleos "simples". A continuación, un paso posterior calibra automáticamente la ploidía nuclear de hepatocitos HNF4 + utilizando núcleos HNF4- como un control interno conocido de 2 x 4N.- Descargue e instale el software.
- Descargue la aplicación empaquetada desde: https://github.com/lukeynoon
- Inicie MATLAB. Vaya a la pestaña APP de la tira de herramientas, haga clic en Instalar aplicación y abra la aplicación descargada con el término"Ploidy_Application.mlappinstall". Aparecerá un mensaje para confirmar la instalación correcta.
NOTA: La aplicación ya está lista para su uso y permanecerá en la pestaña APP de la tira de herramientas.
- Dar formato a los datos de entrada.
NOTA: Antes del análisis automatizado de ploidey nuclear, todos los archivos de hoja de cálculo que contengan datos de imágenes de alto contenido (paso 5.6) deben almacenarse y formatearse de acuerdo con las siguientes instrucciones.- En cada archivo de datos exportado (. XLS 97-2004 libro de trabajo) del paso 5.6, incluye una hoja con el término "Medidas celulares" que contiene todos los datos necesarios para el análisis de ploidicia establecido en las columnas(Figura 3A). Asegúrese de que el diseño de hoja de cálculo, incluidos los nombres de encabezado de columna, permanece sin cambios con respecto al de la Figura 3A, ya que el método de análisis busca los datos de columna correctos buscando estos nombres (consulte conjuntos de datos de demostración en Archivos suplementarios como referencia). Si, por ejemplo, el software de análisis de imágenes de alto contenido no produce una columna "Flujo de luz"(Figura 3A),inserte manualmente una columna "Flujo de luz" en la misma ubicación, es decir, la columna K y rellénela con ceros.
- Para cada condición experimental (por ejemplo, "Lesionado-d14"), proporcione un conjunto de datos de control, que se utilizará para calcular el control interno para la calibración de ploidey nuclear de 2 x 4N (paso 6.3.4.3). Aquí, seleccione muestras de hígado de los compañeros de camada de adultos no tratados ("Control-d0"; Figura 3B-D).
- Para réplicas biológicas (por condición), almacene cada hoja de cálculo en su propia carpeta (como en la Figura 3B). Asigne un nombre incremental a los prefijos de carpeta, por ejemplo, "Sample1, Sample2, Sample3... SampleN", según los nombres de archivo contenidos en. Por lo tanto, cada carpeta del conjunto de datos (por ejemplo, "Control-d0") debe contener una serie de subcarpetas ("Sample1", "Sample2", etc.) cada una conteniendo un archivo de hoja de cálculo con el mismo nombre correspondiente.
- Ejecute la aplicación.
- En MATLAB, inicie el "Ploidy_Application" haciendo clic en el icono dentro de la pestaña MY APPS de la tira de herramientas(Figura 3C). Aparecerá la interfaz gráfica de usuario (GUI) Ploidy_Application (Figura 3C).
- Haga clic en el botón Ruta para controlar los datos para navegar a la carpeta en la que residen las réplicas de datos de control (por ejemplo, "Control-d0"). Esta ruta de acceso de datos aparecerá en la interfaz (por ejemplo, /Users/Desktop/Control-d0).
- A continuación, en "prefijo de carpeta", escriba el nombre que se dará a los archivos de salida (por ejemplo, "Muestra").
NOTA: Este prefijo se puede cambiar a cualquier texto, siempre que las carpetas y los nombres de archivo permanezcan con un nombre incremental. - Haga clic en el botón Ruta a otros datos y vaya a la carpeta en la que residen las réplicas de datos comparativos (por ejemplo, "Lesionado-d14"). Esta ruta de acceso de datos aparecerá en la interfaz (por ejemplo, /Users/Desktop/Injured-d14).
- Haga clic en Ejecutar!. Cuando se complete el análisis, la barra de estado mostrará "Análisis completo!..".
NOTA: La aplicación informará, para cada muestra, de la estratificación de los núcleos "simple" en 2n, 2n-4n, 4n-8n y 8n+ en términos de recuentos absolutos y como porcentaje del total(Figura 3D). Estos archivos se guardarán automáticamente en cada carpeta de ejemplo como: "Count_2n.txt", "Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". El Ploidy_Application guardará automáticamente una lista para cada muestra, de todas las estimaciones individuales de ploidey para núcleos "simple" de hepatocitos y no hepatocitos en "Ploidy_All_Hepatocytes.txt" y "Ploidy_NonHepatocytes.txt". Para el conjunto de datos de control, el método también guarda los umbrales mínimos de contenido de ADN calculados para la estratificación de la ploidición (consulte el paso 6.3.4.3.7) en un archivo denominado "Normalised_Thresholds_Control". Por último, la aplicación producirá una carpeta para el control y los datos de condición comparativa seleccionados llamados "Resumen". Esta carpeta contiene dos subcarpetas, "Ploidy" y "Stratification" que contienen los promedios de todas las muestras proporcionadas(Figura 3D).
- Descripción de la metodología.
NOTA: En la siguiente sección se describe en detalle la metodología utilizada por el software de análisis de loidición nuclear. Si el usuario decide no utilizar la aplicación, estos pasos se pueden seguir utilizando el software de hoja de cálculo para calcular el perfil de ploidy nuclear manualmente.- Separe los núcleos en "simple" o "complejo" según la morfometría nuclear.
- Calcular un "índice de circularidad" para todos los núcleos, definido como el "factor de alargamiento" nuclear dividido por el "Nuc 1/(factor de forma)", donde un valor de 1.0 indica un círculo perfecto.
NOTA: "Alargamiento nuclear" y "Nuc 1/(factor de forma)" son dos medidas discretas de la "circularidad" de un objeto que evalúan criterios morfométricos complementarios y no superpuestos. El primero mide los ejes largos y cortos de un objeto, mientras que el segundo compara la longitud del perímetro de un objeto con el de su área. Para reforzar la definición de circularidad nuclear utilizada en este protocolo, estas dos mediciones se han combinado en un único "índice de circularidad". Un enfoque anterior para estimar la ploidía nuclear utilizando la metodología descrita sólo utilizó el alargamiento nuclear17. Si bien se obtuvieron resultados aceptables utilizando este enfoque, los autores han observado que un "índice de circularidad" compuesto mejora la discriminación de los núcleos seleccionados manualmente de los hepatocitos mononucleares y binucleares (datos no mostrados). - Clasificar los núcleos con un índice de circularidad a 0,8 como "complejos" y los > 0,8 como "simple".
- Calcular un "índice de circularidad" para todos los núcleos, definido como el "factor de alargamiento" nuclear dividido por el "Nuc 1/(factor de forma)", donde un valor de 1.0 indica un círculo perfecto.
- Estimar el contenido de ADN "mínimo" (m) para todos los núcleos "simples".
- Calcule el radio nuclear (r) utilizando la fórmula:

- Calcular el volumen nuclear (v) utilizando el volumen de una fórmula de esfera:
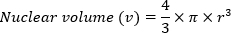
- Genere un valor relativo para el contenido mínimo de ADN (m) utilizando la fórmula:

- Calcule el radio nuclear (r) utilizando la fórmula:
- Calibre el conjunto de datos utilizando los núcleos NPC (HNF4-) como un control interno de 2 a 4N.
NOTA: Los PNJ tienen un contenido de ADN de 2 x 4N dependiendo del estado del ciclo celular. Por lo tanto, el valor medio del contenido de ADN "mínimo" de NPC (NPCm) aumenta con lesiones(Figura 4A). El error de calibración se minimiza estableciendo un límite superior de NPCm que representa un umbral 4c(Figura 4B).- Dentro de la hoja de cálculo, seleccione solo núcleos NPC con valores para "m" que se encuentran dentro de 1 desviación estándar (SD) del modo (esto filtra el ruido de un posible error de segmentación).
- Dentro de este rango filtrado, examine las áreas nucleares y sus correspondientes intensidades medias de Hoechst(Figura 4C).
- Estimar el área nuclear más pequeña dentro de este rango filtrado con la máxima intensidad nuclear Hoechst (es decir, el punto en el que la línea de la curva cambia de dirección en el dataset filtrado como se ilustra en el círculo rojo de la Figura 4C). Este valor representa un estado transitorio 2N-4N (t) por encima del cual predomina el muestreo de núcleos 4c sobre 2c, lo que resulta en un máximo de intensidad media de Hoechst.
NOTA: Este valor es determinado automáticamente por el software; sin embargo, los usuarios de hojas de cálculo pueden seleccionar manualmente este punto como el tamaño de transición. - Calcule el contenido mínimo de ADN representado por este tamaño de transición (tm) siguiendo el paso 6.3.4.2.
- Para estimar el hombro 4N del conjunto de datos NPCm, agregue 1 SD al valor de tm. El número resultante(Figura 4B) describe el límite superior del contenido mínimo de ADN de NPC que se utilizará para la estratificación de la ploidición nuclear (S4c).
- Repita los pasos 6.3.4.3.1-6.3.4.3.5 para todas las muestras de "control".
NOTA: Por ejemplo, en la Figura 3, los hígados de control no lesionados ("Control-d0") se utilizan como condición de control. - Calcular un umbral de estratificación promedio 4c (S4c) para muestras de "control" y utilizar esto para extrapolar los límites 2c (S2c) y 8c (S8c) para un contenido mínimo de ADN (m). Los umbrales de estratificación son generados y almacenados automáticamente por el software (paso 6.3.3.3).
NOTA: Dependiendo del diseño del estudio, los valores umbral estratificados promedio se pueden calcular para cada condición o para condiciones específicas (por ejemplo, hígado de control saludable). Sin embargo, el software de análisis de ploidicia nuclear requiere que uno de un conjunto de 2 archivos se designe como "control" a los efectos de calcular los valores de ploidy relativos. - Calcule un valor de ploidy para todos los núcleos utilizando el valor S2c generado en el paso 6.3.4.3.7 según:

- Estratificar núcleos de hepatocitos "simple" (HNF4+) en corchetes 2c/4c/8c/>8c de acuerdo con los siguientes criterios: "2c" HNF4o+ a p a 2; "4c" HNF4o+ á 2 < p a 4; "8c" HNF4o+ a 4 < p a 8; ">8c" HNF4 + a 8 < p.
- Para reconstruir el patrón espacial de los subgrupos ploidy, separe los datos nucleares dentro de cada hoja de cálculo de muestra de acuerdo con los campos correspondientes en los que fueron adquiridos. A continuación, utilice las coordenadas x/y nucleares asociadas (del paso 5.5) para trazar subgrupos ploidy en 2D(Figura 5C).
- Separe los núcleos en "simple" o "complejo" según la morfometría nuclear.
- Descargue e instale el software.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este método se ha utilizado para medir el impacto de la lesión colestásica en el hígado de ratón adulto mediante la alimentación de animales durante 0 a 21 días con una dieta hepatotóxica que contiene 0,1% 3,5-dietoxcarbonyl-1,4-dihidrocollidina (DDC)17. La alimentación crónica de DDC produce lesiones hepatocelulares que aumentan la ploimidia y la expansión periportal de los PNJ. El usuario debe ser consciente de que la tensión del ratón y las diferenc...
Access restricted. Please log in or start a trial to view this content.
Discusión
Se describe un enfoque de alto contenido y alto rendimiento para el análisis de la remodelación de tejidos y la estimación de la ploideide nuclear de hepatocitos en el hígado murino. Una vez familiarizado con el procedimiento, un usuario puede procesar, crear imágenes y analizar varias muestras en un período de 3 a 5 días, generando grandes conjuntos de datos comprobables que proporcionan una firma detallada de la salud del hígado. Dada la simplicidad del método de preparación de la muestra, junto con el gran n...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por las subvenciones del Gobierno español de MINECO BFU2014-58686-P (LAN) y SAF-2017-84708-R (DJB). LAN fue apoyada por una beca nacional MINECO Ramón y Cajal RYC-2012-11700 y el premio Plan GenT (Comunitat Valenciana, CDEI-05/20-C), y FMN por una beca regional ValI+D de la Generalitat Valenciana ACIF/2016/020. RP desea reconocer a la Prof. Ewa K. Paluch por su financiación. Agradecemos a la Dra. Alicia Martínez-Romero (servicio de citometría CIPF) por su ayuda con la plataforma IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Referencias
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados