A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
תפוקה גבוהה בשיטת באתרו להערכת הפטציט הגרעיני בעכברים
In This Article
Summary
אנו מציגים שיטה חזקה, חסכונית, וגמיש למדידת שינויים במספר hepatocyte ובכלי הנשק הגרעיני בתוך קבוע/הקפאה דגימות רקמות שאינו דורש זרימה cy, try. הגישה שלנו מספקת חתימה רבת עוצמה לדוגמה של ציטולוגיה בכבד עבור מעקב אחר ההתקדמות של פגיעה בכבד ומחלות.
Abstract
כאשר הכבד נפצע, מספרי הפטוציט מפחיתים, בעוד שגודל התאים, גודל הגרעין והעלייה בפלויידי. התרחבות של תאים שאינם מסוג מדומה, כגון המרה, מיופיברוציטים, ושלתי ותאים דלקתיים גם מצביעים על נזק כבד כרוני, שיפוץ רקמות והתקדמות המחלה. בפרוטוקול זה, אנו מתארים גישה פשוטה תפוקה גבוהה לחישוב שינויים בהרכב הסלולר של הכבד הקשורים לפציעה, מחלה כרונית וסרטן. אנו מראים כיצד מידע שהופק מסעיפים דו ממדיים (2D) רקמות ניתן להשתמש כדי לכמת ולכייל hepatocyte הגרעין הגרעיני בתוך מדגם ולאפשר למשתמש לאתר תת ערכות מסוימות פלואידיות בתוך הכבד באתרו. השיטה שלנו דורשת גישה לחומר כבד קבוע/קפוא, ריאגנטים חיסונית בסיסי וכלי כל פלטפורמת הדמיה גבוהה. היא משמשת חלופה רבת עוצמה לטכניקות הזרימה הסטנדרטיות של cy, אשר דורשות הפרעה של רקמה שנאספה לפני כן, אובדן מידע מרחבי והטיה פוטנציאלית לפירוק.
Introduction
הפטציטים בכבד היונקים יכולים לעבור נתקע ציטוקינזה כדי לייצר תאים binuclear, ו-DNA endoreplication לייצר גרעיני פוליפואיד המכילים עד 16N ה-DNA תוכן. הגדלת הפלואידיות התאית והגרעינית במהלך פיתוח הפוסט-לידה, הזדקנות ותגובה ללחצים סלולריים מגוונים1. תהליך הפוליפולואידיזציה הוא דינמי והפיך2, למרות שתפקידה הביולוגי המדויק אינו ברור3. מוגברת פלויידי הוא קשור עם קיבולת ההתרבות מופחתת4, מגוון גנטי2, הסתגלות לפציעה כרונית5 הגנה מפני סרטן6. השינוי הפטוציט הפלואידי מתרחש כתוצאה מקצב מעגלי משתנה7, והגמילהמשמונה. בעיקר, הפרופיל הפלואידי של הכבד משתנה על ידי פציעה ומחלה9, וראיות משכנעות מציע כי שינויים מסוימים פלואידיות, כגון הגדלת ≥ 8n גרעינים או אובדן של 2n hepatocytes, לספק חתימות שימושיות עבור מעקב אחר מחלת כבד שומני לא אלכוהוליים (nafld11) התקדמות3,10, או ה
במונחים כלליים, פגיעה בכבד והתחדשות קשורים עם גודל תא hepatocyte מוגבר שטח גרעיני12, יחד עם מספר כולל מופחת של hepatocyte, במיוחד אלה עם 2n תוכן DNA10,11. הפציעה בכבד מלווה גם לעתים קרובות על ידי התרחבות של תאים שאינם מתוך הכתוניים (NPCs), כולל סטרומה מיוסטרותקיעות, תאים דלקתיים ותאים מחולל בכבד biפוטנטי. שיטות תפוקה גבוהה המספקות פרופיל ציטולוגי כמותי של מספר תא מדומה ופלויידי גרעיני, ואילו גם חשבונאות לשינויים NPCs, ולכן יש פוטנציאל ניכר כמו מחקר כלים קליניים כדי לעקוב אחר תגובת הכבד במהלך הפציעה והמחלה. משכנעת לאחרונה בניתוח באתרו של הפלויידי ספקטרום בדגימות האדם של קרצינומה hepatocellular גם להדגים כי הפיידי הגרעינית הוא גדל באופן דרמטי בתוך גידולים והוא מוגבר במיוחד תת הגידול אגרסיבי יותר עם בידול מופחת ואובדן של TP5313. מכאן, קיימת אפשרות חזקה שהתקדמות מתודולוגיים בהערכה כמותית של הפלואידיות הגרעינית תסייע ביצירת פרופיל התחזיות העתידיות של סרטן הכבד.
בפרוטוקול זה, מתודולוגיה גמישה בתפוקה גבוהה עבור ניתוח השוואתי של מקטעי רקמת הכבד של העכבר מתוארים, אשר מספק פרופיל cy, מפורט של מספרי hepatocyte, התגובה NPC ושיטה מכויל פנימי להערכת הגרעין פלויידי (איור 1). הפטציטים נבדלים מ NPCs על ידי הגורם הגרעיני hepatocytes 4 אלפא (HNF4α) אימונויניום, לפני אפיון של גודל גרעיני וממורמטריה גרעינית. "תוכן ה-DNA מינימלי" מוערך עבור כל מסכות גרעיניות מעגלית על ידי שילוב ממוצע Hoechst 33342 עוצמה (proxy עבור צפיפות DNA) עם הנפח תלת מימדי (3D) הגרעיני. הפטציט תוכן די-אן-איי מינימלי מכויל לאחר מכן באמצעות NPCs כדי ליצור פרופיל של פלויידי גרעיני.
רכישת תמונה, פילוח גרעיני וניתוח תמונה מבוצעים באמצעות הדמיה בעלת תוכן גבוה, המאפשר אזורים גדולים של דו מימדי (2D) מקטעי כבד המכילים עשרות אלפי תאים להיות מוקרן. תוכנית מותאמת אישית מסופקת לצורך עיבוד אוטומטי של נתוני ניתוח תמונה באיכות גבוהה כדי להפיק פרופיל פלואידי רחב לדגימה לכל גרעין הפטוציט המעגלי. פעולה זו מבוצעת באמצעות חינם כדי להוריד את התוכנה כדי לחשב את הנשק הגרעיני מבוסס על ניתוח התמונה סטריאוולוגית (SIA)10,11,14,15. מתודולוגיית ה-SIA אומתה בעבר על ידי הזרמת cy, כשיטה מדויקת, אם כי מפרך, שיטה להערכת הפרופציט גרעינית בכבד14, בהנחה מורפולוגיה גרעינית מעגלית מערכת יחסים בין הגודל הגרעיני ותוכן ה-DNA. בפרוטוקול זה, שני הפרמטרים הגרעיניים נמדדים על ידי הערכה של מורמטריה גרעינית ו הופסט תוויות 33342. החישוב של "תוכן ה-DNA מינימלי" עבור כל מסכה גרעינית מלווה בכיול של hepatocyte הגרעין הגרעיני באמצעות NPCs, אשר יש הידוע 2 על התוכן DNA ולכן לשמש שליטה פנימית שימושי.
לעומת הזרם הקונבנציונלי cy, שיטות שירות16 הגישה המתוארת מאפשרת hepatocyte הגרעין הגרעיני כדי להיות מוערך באתרו ואינו דורש גישה לרקמה טרייה או שיטות מצבור שיכולים הטיה תוצאות ולהיות קשה לתקנן. כמו בכל הגישות המבוססות על SIA, מחלקות ההגנה הגרעינית > 2N מיוצגים באמצעות דגימת 2N בשל החשיפה של גרעין גדול מחוץ למישור המשוונית. הפרופיל ברקמות הפלואידיות מתאר גם את תוכן ה-DNA המינימלי לכל מסכות הגרעין העגולות, ואינו מפלה ישירות בין תאי הפטציטים והתאים הבינאוריים בעלי שני הגרעינים הנפרדים ("לא נוגעים") של אותו פלויידי. עם זאת, את הפשטות של פרוטוקול זה מאפשר היקף ניכר עבור אותו להיות מותאם לחשבון עבור פרמטרים נוספים כגון מרווח בין גרעיני או ניתוח היקפי התא, זה יקל על זיהוי של תאים binuclear מתן הערכה מפורטת יותר של הסלולר.
Access restricted. Please log in or start a trial to view this content.
Protocol
כל ניסויי החיות אושרו בעבר על ידי ועדת האתיקה של האו. עכברים שוכנו במתקן נטול הפתוגן במרכז החקירה של פרינסיפה פליפה (ולנסיה, ספרד), רשום בתור בעל חיים ניסיוני מגדל, משתמש, ואספקה (reg. no. ES 46 250 0001 002) תחת התנאים הנוכחיים של הרווחה האירופית והספרדית בעלי חיים (RD 53/2013).
1. קצירת רקמות והכנת דגימות
הערה: פרוטוקול זה מתאר כיצד להקפיא רקמות ללא קיבעון מוקדם או הקפאה מוקדמת. עבור דגימות שתוקנו/הקפאה בעבר, המשך לסעיף 2 והשמט את השלב 3.1. כל הניתוחים בוצעו באמצעות נקבה מבוגרת C57BL/6 עכברים בגילאי 12 עד 16 שבועות.
- הקרבת בעלי חיים על ידי הזרקת פנטניל/פנטוברביטל הצפק ואחריו פריקה צוואר הרחם. עם העכבר מול הצד הגחוני למעלה, לפתוח את חלל הבטן לחשוף את הכבד על ידי אוחז את העור עם מלקחיים וביצוע חתך אנכי מהבסיס של הבטן התחתונה לבסיס עצם החזה באמצעות מספריים כירורגי.
- בזהירות להסיר את כיס המרה באמצעות פינצטה עדין, לנתח את הכבד ולשטוף את הכבד הנבחר כדורית בצלחת 10 ס"מ צלחת פטרי ממולא מלוחים מואגור פוספט (PBS).
הערה: מומלץ להשוות את אותה אונה הכבד עבור כל חיה, במקרה זה האונה החציוני שימש. - מילוי קריומתויג עם טמפרטורת חיתוך (OCT) מיטבית בטמפרטורת החדר (RT). להימנע בועות OCT. אם הם מופיעים, לדחוף אותם לקצה של העובש באמצעות מחט או טיפ פיפטה.
- הטמע כדורית הכבד לתוך ממולא OCT הישן ומיד מניחים אותו על קרח יבש כדי להבטיח הקפאה מהירה. ב80 מעלות צלזיוס עד להקפאה.
2. קריוסיס1
- מובילים בקרח יבש על מנת. למנוע השפלה ברקמות לפני ההקפאה בתוך ההקפאה הגדר עד 20 ° c עבור 20 דקות.
- להוציא לדוגמה על ידי הפעלת לחץ על הבסיס של הקריומית פלסטיק. החל את OCT הנוזלי כדי לחמם את הדיסק לדוגמה ב-RT, מיקום בהקפאה וחבר דגימת כבד מוטבעת של OCT. החל לחץ עדין והמתן 3 דקות עבור OCT כדי להקפיא להבטיח את המדגם מקלות לדיסק.
הערה: הימנע מטיפול במדגם עם האצבעות ככל האפשר כדי להתחמק השפלה רקמות. - נעל את המדגם בזרועו של הקריוסטט וכוונן את הכיוון כך שקצה המדגם יהיה מקביל ללהב הקריוסטט. חותכים את הדגימה עד שהרקמה מגיעה.
- מקטע את המדגם בעובי של 6 יקרומטר. מניחים שקופית מצופה פוליאמיד המסומנת על המדגם עבור 5 s כדי לאפשר לדוגמה להיצמד לשקופית. מקם את השקופית ב-RT עבור 3-5 דקות, לאחר מכן, לקבלת תוצאות מיטביות, המשך ישירות לסעיף 3.
הערה: לעיבוד של מספר דוגמאות טריות מוקפאות מתקבלות על-ידי אחסון זמני של שקופיות בתיבת שקופיות בקרח יבש עד לעיבוד כל הדגימות. בעת שימוש בגישה זו, מאפשרים לכל השקופיות להגיע למצב של RT לפני שתמשיך בסעיף 3. Formalin קבוע פרפין מוטבע (FFPE) דגימות ניתן להשתמש, למרות הרקע autoפלואורסצנטית הוא גדל על ידי שיטה זו. כדי להמשיך מתוך דגימות FFPE, מקטע ב 4 μm. הר על ידי לכידת סעיפים מ 40 המים ° c בג על מגלשות מטופלים פוליאמיד. חום שקופיות עבור 1 h ב 60 ° c, לאחר מכן לאחר מכן על ידי שטיפת RT טורי (5 דקות) בצנצנות coplin המכילים קסילן (x2), אתנול 100% (x2), 96% (x2), 70% (x1) ו-dH2O (x1). כדי לחשוף שקופיות של מקום אנטיגנים במאגר ציטראט עבור 20 דקות ב-90 ° צ' לפני שקופיות הרפיה ב-PBS at RT. המשך לשלב 3.2.
3. אימונויניום
- תקן את מקטעי הרקמה בתוך מכסה המנוע על ידי החלת 1 מ ל 4% פאראפורמלדהיד (בתחתית) ב-PBS עבור 10 דקות ב-RT. העבר שקופיות לצנצנת PBS מילא ושטוף 3 דקות בעצבנות עדינה (חזור על 3x).
הערה: מעתה ועד סוף תהליך החיסוני, הימנע מייבוש המדגם. - יבש את האזור סביב כל חתך הרקמה ולהקיף באמצעות עט הידרופובי. החדירות עם 0.5% nonionic סורסטנט (כלומר, טריטון X-100) ב-PBS עבור 15 דקות ב-RT. לאחר מכן רוחצים ב-PBS בצנצנת Coplin במשך 3 דקות בעצבנות עדינה (חזור על 2x).
- בלוק שימוש בתמיסה מסוננת של 1% בסרום של שור (BSA), 5% סרום סוסים, 0.2% nonionic מבקר ב-PBS (לפחות 1 h ב RT).
- מודלת עם נוגדן HNF4α העיקרי מדולל במאגר חסימת בלילה ב 4 ° c בחדר כהה כתמים לחים (ראה טבלת חומרים עבור נוגדנים ומדלל ספציפי).
- הניחו שקופיות בצנצנת של מילוי הPBS ושטפו במשך 3 דקות בעצבנות עדינה (חזור על 4x).
- דגירה עם אלקסה-488 נוגדן משני מצוחים ו Hoechst מדולל מסונן 1% BSA ו 0.2% nonionic סורסטנט ב-PBS עבור 2 h ב RT ב לחות כהה תא כתמים (ראה טבלת חומרים עבור נוגדנים ומדלל ספציפי).
- הניחו שקופיות בצנצנת של מילוי הPBS ושטפו במשך 3 דקות בעצבנות עדינה (חזור על 4x). התרחץ ב-ddH2O עבור 3 דקות בעצבנות עדינה (חזור על 2x).
- הר שקופיות על ידי הצבת שתי טיפות של מדיה הרכבה פלורסנט על coverslip (24 x 60 מ"מ) והנחת שקופיות על זה, חיסול בועות על ידי הפעלת לחץ עדין. לאחסון לטווח ארוך, חותם שמיכות בקצוות עם לק ציפורניים ברור לאחסן בחושך ב 4 ° c.
- לפני שתמשיך, בדוק שקופיות באמצעות מיקרוסקופ פלואורסצנטית קונבנציונאלי כדי להבטיח קיבעון טוב immunolabeling.
הערה: ראה איור 2א, ב לקבלת תוצאות צפויות.
4. רכישת תמונה פלואורסצנטית
הערה: עבור שלב זה, פלטפורמת דימות בעלת תוכן גבוה (טבלת חומרים) נדרשת ותומכת ברכישת תמונות פלואורסצנטית אוטומטית.
- הפעל את מערכת ההדמיה ופתח פרוטוקול רכישה חדש.
- בחר את המטרה 10x, לשים לב לאזור של שדה התצוגה (במקרה זה 0.6 mm2).
- הגדר פרמטרים כדי לרכוש תמונות פלואורסצנטית באמצעות עירור המתאים ומסנני פליטה (כמו לכל שלב 3.6). עבור Hoechst ו אלקסה-488, בחר "DAPI" ו "GFP" ערוצים עם 390/18 ו 438/24 ננומטר ו 432.5/48 ו 475/24 פליטת ננומטר, בהתאמה.
- התמקד במדגם וודא שעוצמת האות אינה מרוויה. ודא כי לכידת תמונה נעשית עם אותו זמן חשיפה עבור כל התמונות או להשתמש במערכת שבה האינטנסיביות של הזריחה תוקנה לזמן חשיפה.
- לסרוק את המדגם ולרכוש תמונות מספיקות כדי לקבל כיסוי מלא של מקטע הרקמה (כ 20-50 שדות תצוגה, בהתאם לגודל המדגם).
- סקור את מסד הנתונים של התמונה, ביטול ידני (i) שדות ממוקדים היטב, (ii) אלה בגבולות של כל מקטע רקמות (כדי למנוע חישובים בצפיפות תאים ממתח), ו-(iii) אלה המכילים אזורים מקופלים/פיזית ניזוק של סעיף הרקמה אם קיים.
5. ניתוח אוטומטי של תמונה פלואורסצנטית
הערה: שלב זה דורש תוכנת ניתוח תמונהמתאימה (טבלת חומרים) המסוגלת: (1) זיהוי אוטומטי של הופסט גרעיני בתוך תמונות ב 405 ננומטר (מקטעי גרעין), (2) הערכה משמעות העוצמה הגרעינית hoechst, ו (3) בניתוח הסף כדי לקבוע את +/-מצב של זריחה גרעינית ב 488 ננומטר (HNF4α) כמה הכשרה מפעילה בסיסית/מומחיות נדרשת כדי להעריך ולהתאים את פילוח ופרמטרים סף בתוך התוכנית כדי להבטיח כי גרעיני ו HNF4α +/-מצב הם מגודרת בצורה אופטימלית (איור 2).
- בתוכנה לניתוח תמונות, פתח את קובץ הרכישה המכיל את התמונה (405 nm) ו HNF4α (488 nm) תמונות משלב 4.5, וליצור פרוטוקול ניתוח חדש.
- הגדר אורכי גל לשימוש עבור פילוח גרעיני (Hoechst, 405 nm) ועבור ניתוח הסף של hepatocyte/NPC (HNF4α, 488 nm).
- להתאים את הפרמטרים של התוכנה מפילוח גרעיני (כגון "שטח גרעיני מינימלי" וזיהוי גרעיני "רגישות") כדי להבטיח גרעינים הם הפרדה אופטימלית.
הערה: יש לקבל עדיפות גבוהה יותר מפילוח של הפטוציטים בגלל זה של npcs. גרעיני הפטציט מעוגל בדרך אופיינית (טווח בין רבעוני: 40-64 יקרומטר2). גרעיני NPC, כגון אלה של sinusoidal אנדותאליה, הם משוטחים/אליפטי או לא סדיר בצורה בדרך כלל קטן יותר וצפוף יותר מאשר אלה של hepatocytes (טווח הרביעון גודל: 30-43 יקרומטר2). עבור כבד העכבר, שטח גרעיני מינימלי ≥ 23 יקרומטר2 וזיהוי "רגישות" של 65% שימשו (ראה איור 2ג, D לקבלת תוצאות צפויות). רגישות קובעת כיצד אשכולות הפיקסלים מזוהים כגרעינים בודדים המבוססים על עוצמתם וצריכים להיבדק באופן אמפירי עבור כל דוגמה שנקבעה על-ידי המשתמש לפני שתמשיך בניתוח תמונה אוטומטי. - שינוי עוצמת הסף ב 488 ננומטר כדי להבטיח המשך הHNF4α של הפציטים (+) ותאים שאינם מתוך מכוניים (HNF4α-).
הערה: ראה איור 2ג, D לקבלת תוצאות צפויות. הערך של עוצמת הסף הוא יחסי ויהיה תלוי בצביעת יעילות והגדרות רכישה כגון עוצמת לייזר. לכן צריך להיות מתוקננת על ידי המשתמש. השתמש HNF4α-תאים מוכרים כגון תאים אנדותל ו-NPCs הperiportal כפקד פנימי שלילי ובין גרעיני hepatocyte כהתייחסות חיובית לצביעת. בדוק את פרמטרי הניתוח באמצעות מספר קטן של תמונות כדי להבטיח פילוח גרעיני טוב והפרדה בין הסף לפני החלת פרמטרי ניתוח על קבוצת הנתונים כולה. - בחרו את הפרמטרים הגרעיניים הבאים כדי לכמת: (1) אזור גרעיני המבוסס על כתמים הוקוסט (μm2), (2) פירושו חוזק גרעיני הופסט (RU), (3) גורם התארכות גרעינית (יחס ממוצע של הציר הקצר של הגרעין לציר הארוך של הגרעין, שבו לאובייקט מרכז סימטרי [לא מוארך] יש ערך של 1, (4) nuc 1/(מארז הטופס), משמעות גרעינית "מעוגלות" מדד מחושב על ידי היקפית 2/(4π x אזור). הערכים נעים בין 1 לאינסוף, כאשר 1 הוא מעגל מושלם, (5) HNF4α סטטוס (חיובי-1 או שלילי-0), ו (6) קואורדינטות x/y מבוסס על "מרכז הכבידה" (cg), שיטה לאיתור מרכז האובייקט מתמונות בקנה מידה אפור עם דיוק תת פיקסל.
- הפעל את הניתוח עבור כל ערכות הנתונים לדוגמה ויצא נתונים מספריים משלב 5.5 לתוכנת גיליון אלקטרוני.
6. ניתוח נתונים
הערה: ניתן לבצע את שלב ניתוח הנתונים באמצעות כל תוכנת גיליון אלקטרוני סטנדרטית.
- חשב את הפטציט ואת מספרי התאים הלא-מפאציט.
- חשב את האזור הכולל של מקטע הכבד שנותח עבור כל מדגם על-ידי הכפלת מספר שדות התצוגה על-ידי האזור של שדה התצוגה (שלב 4.2).

- עבודה עם קבצי גיליון אלקטרוני שנוצר עבור כל מקטע כבד, לסנן את הנתונים על ידי בחירת רק HNF4α + גרעיני. חשב את המספר הכולל של HNF4α + גרעיני מנותח וחילוק זה על-ידי האזור הכולל שנותח כדי להשיג דחיסות hepatocyte ממוצע עבור כל מדגם (איור 2F).

- בצע את החישוב זהה עבור תאים שאינם מופיעים על-ידי סינון הגיליון האלקטרוני עבור HNF4α-תאים (איור 2E).
- חשב את האזור הכולל של מקטע הכבד שנותח עבור כל מדגם על-ידי הכפלת מספר שדות התצוגה על-ידי האזור של שדה התצוגה (שלב 4.2).
- חשב את התפלגות הגודל הגרעיני של hepatocyte.
- באמצעות תוכנת גיליון אלקטרוני, סנן נתונים כדי לבחור רק HNF4α + גרעינים.
- התוויית ערכים של אזור גרעיני בהיסטוגרמה (איור 2G). הגדר את רוחב הסל עד 5 יקרומטר2.
הערה: ניתן לתקן ערכי תדירות עבור שטח (גרעינים/mm2) לפי שלב 6.1.1.
- בצעו ניתוח גרעיני. לחיפטציט
הערה: נתוני הגיליון האלקטרוני משלב 5.6 משמשים להפקת פרופיל משתמש גרעיני עבור כל דוגמה. תהליך זה היה אוטומטי וניתן לבצעה באמצעות תוכנה כתובה מותאמת אישית הזמינה באופן חופשי להורדה עם מידע תמיכה וערכות נתונים הדגמה בhttps://github.com/lukeynoon(ראה הוראותקבצים משלימים). קוד מקור מסופק עבור משתמשים המעוניינים להתאים את המתודולוגיה. תיאור של האלגוריתם, יחד עם הוראות התקנה ושימוש מתוארים להלן. התוכנית משתמשת בנתוני גיליון אלקטרוני כדי להפריד באופן אוטומטי גרעינים hepatocyte לשתי קבוצות; (1) אלה עם גרעיני "פשוט" מעגלי ו (2) "מורכב" הנציג של הגרעין הלא עגול של תאים binuclear עם > 2c פלויידי. התוכן המינימלי הגרעיני DNA (פונקציה של שטח גרעיני וצפיפות DNA) מחושב הבא עבור כל "פשוט" גרעיני. הצעד הבא מכייל באופן אוטומטי HNF4α + hepatocyte הגרעין הגרעיני באמצעות HNF4α-גרעיני כפקד פנימי 2-4N מוכר.- הורד והתקן תוכנה.
- הורד את היישום הארוז מ: https://github.com/lukeynoon
- . הפעל את MATLAB נווט אל הכרטיסייה APP של רצועת הכלים, לחץ על התקן app ופתח את היישום שהורדת כינה "Ploidy_Application. mlappinstall". תופיע הודעה שתאשר את ההתקנה המוצלחת.
הערה: היישום מוכן כעת לשימוש ויישאר בכרטיסייה APP של רצועת הכלים.
- עיצוב נתוני קלט.
הערה: לפני ניתוח מיידי של הגרעין הגרעיני, כל קבצי הגיליון האלקטרוני המכילים נתוני הדמיה של תוכן גבוה (שלב 5.6) צריכים להיות מאוחסנים ומעוצבים בהתאם להנחיות הבאות.- בכל קובץ נתונים מיוצא (. XLS 97-2004 חוברת עבודה) משלב 5.6, כלול גיליון הנקרא "מדדי תאים" המכיל את כל הנתונים הדרושים לניתוח הפלויידי שנקבעו בעמודות (איור 3א). ודא שפריסת הגיליון האלקטרוני כולל שמות של כותרות עמודה נותרת ללא שינוי מתוך הספרה 3A, מכיוון ששיטת הניתוח מאתרת את נתוני העמודה הנכונים על-ידי חיפוש שמות אלה (ראה ערכות נתונים של הדגמה בקבצים משלימים עבור הפניה). אם לדוגמה, תוכנה לניתוח תמונות באיכות גבוהה אינה מפיקה עמודת "שטף אור" (איור 3א), הכנס באופן ידני עמודת "שטף אור" באותו מיקום, כלומר, עמודה K ומלאו אותה באפסים.
- עבור כל מצב ניסיוני (למשל, "פצועים-d14"), מספקים ערכת נתונים של בקרה, שישמשו לחישוב השליטה הפנימית עבור 2 כיול גרעיני (שלב 6.3.4.3). כאן, בחר דגימות כבד מתוך כנות למבוגרים לא מטופלות ("בקרת-d0"; איור 3ב-ד).
- לשכפל ביולוגי (לכל תנאי), אחסן כל גיליון אלקטרוני בתיקיה משלה (כמו באיור 3ב). ציין את שם קידומות התיקיות בהפרשים קבועים, לדוגמה, "Sample1, Sample2, Sample3... SampleN ", לפי שמות הקבצים הכלולים בתוך. מכאן, כל תיקיית ערכת נתונים (למשל, "Control-d0") צריך להכיל סדרה של תיקיות משנה ("Sample1", "Sample2", וכו ') כל המכיל קובץ גיליון אלקטרוני של אותו שם מקביל.
- . תריץ את הבקשה
- בתוך MATLAB, הפעל את "Ploidy_Application" על ידי לחיצה על הסמל בתוך הכרטיסייה APPS שלי של רצועת הכלים (איור 3ג). ממשק המשתמש הגרפי Ploidy_Application (GUI) יופיע (איור 3ג).
- לחץ על הנתיב כדי לשלוט בלחצן נתונים כדי לנווט אל התיקיה שבה משכפל נתוני הפקד (לדוגמה, "control-d0"). נתיב נתונים זה יופיע לאחר מכן בממשק (לדוגמה, /Ds/deskpuerusuderul).
- לאחר מכן, ב "קידומת תיקיה" הקלד את השם שיינתן לקבצי הפלט (לדוגמה, "לדוגמה").
הערה: ניתן לשנות קידומת זו לכל טקסט, בתנאי שהתיקיות ושמות הקבצים יישארו מצטבר בשם. - לחץ על לחצן נתיב לנתונים אחרים ונווט אל התיקיה שבה משכפל הנתונים השוואתיים (לדוגמה, "פצועים-d14"). נתיב נתונים זה יופיע לאחר מכן בממשק (לדוגמה,/ Users/deskpsogoged-a).
- לחץ על הפעל!. לאחר השלמת הניתוח, שורת המצב תיקרא "ניתוח!.. השלמה".
הערה: היישום ידווח, עבור כל דוגמה, ריבוד של גרעין "פשוטים" לתוך ≤ 2n, 2n-4n, 4n-8n ו-8n + במונחים של ספירות מוחלטות וכאחוזים מכלל (איור 3ד). קבצים אלה יישמרו באופן אוטומטי בכל תיקיה לדוגמה כמו: "Count_2n. txt", "Count_2n_to_4n. txt", "Count_4n_to_8n. txt", "Count_8n_and_higher. txt", "Percentage_2n. txt", "Percentage_2nto4n. txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". הPloidy_Application תשמור באופן אוטומטי רשימה עבור כל דוגמה, של כל האומדנים הבודדים פלואידי עבור "פשוט" hepatocyte ו גרעיני לא-hepatocyte ב "Ploidy_All_Hepatocytes. txt" ו- "Ploidy_NonHepatocytes. txt". עבור ערכת הנתונים של הפקד, השיטה שומרת גם את ספי התוכן המינימלי של ה-DNA שחושבו לריבוד של פלויידי (ראה שלב 6.3.4.3.7) בקובץ בשם "Normalised_Thresholds_Control". לבסוף, היישום יפיק תיקיה עבור הפקד הן ונתוני התנאי השוואתי שנבחרו מכונים "תקציר". תיקיה זו מכילה שתי תיקיות ממשנה, "פלויידי" ו-"ריבוד" המכילות את הממוצעים של כל הדגימות שסופקו (איור 3ד).
- תיאור המתודולוגיה.
הערה: הסעיף הבא מתאר בפרוטרוט את המתודולוגיה המשמשת את תוכנת ניתוח הפלואידיות הגרעינית. אם המשתמש בוחר שלא להשתמש ביישום, ניתן לעקוב אחר שלבים אלה באמצעות תוכנת גיליון אלקטרוני כדי לחשב את פרופיל הפלואידי הגרעיני באופן ידני.- להפריד את הגרעינים לתוך "פשוט" או "מורכב" על פי מורמטריה גרעינית.
- חישוב "אינדקס מעגלי" עבור כל הגרעינים, המוגדרים כגורם גרעיני "פקטור התארכות" מחולק על-ידי "Nuc 1/(מארז טופס)", כאשר ערך של 1.0 מציין מעגל מושלם.
הערה: "התארכות גרעיניות" ו-"Nuc 1/(מארז הטופס") הם שני מדדים נפרדים של "מעגליות" של האובייקט, ההערכת קריטריונים משלימים, שאינם חופפים. הראשון מודד את הציר הארוך והקצר של העצם, בעוד האחרון משווה את אורך ההיקף של האובייקט לאזור. כדי לחזק את ההגדרה של מעגליות גרעיניות המשמשות בפרוטוקול זה, שתי מדידות אלה שולבו לתוך "אינדקס מעגלי" אחד. הגישה הקודמת להערכת הגרעין הגרעיני באמצעות המתודולוגיה המתוארת השתמשו רק התארכות גרעינית17. בעוד שתוצאות מקובלות התקבלו באמצעות גישה זו, המחברים ראו כי "מדד מעגלי" המורכב משפר את האפליה של גרעינים שנבחרו באופן ידני מתוך מוננוציטים ובין-בהיר (נתונים לא מוצגים). - סווג גרעינים עם מדד מעגלי ≤ 0.8 כ "מורכב" ואלה > 0.8 כמו "פשוט".
- חישוב "אינדקס מעגלי" עבור כל הגרעינים, המוגדרים כגורם גרעיני "פקטור התארכות" מחולק על-ידי "Nuc 1/(מארז טופס)", כאשר ערך של 1.0 מציין מעגל מושלם.
- הערכת "מינימלי" תוכן ה-DNA (m) עבור כל "פשוט" גרעיני.
- חשב את הרדיוס הגרעיני (r) באמצעות הנוסחה:

- חשב נפח גרעיני (v) באמצעות אמצעי האחסון של נוסחת הספירה:
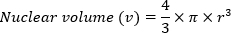
- צור ערך יחסי עבור תוכן DNA מינימלי (m) באמצעות הנוסחה:

- חשב את הרדיוס הגרעיני (r) באמצעות הנוסחה:
- כיול ערכת הנתונים באמצעות גרעין NPC (HNF4α) כפקד פנימי של 2-4N.
הערה: NPCs יש 2 התוכן DNA-4N בהתאם למצב מחזור התא. מכאן, ערך ממוצע של NPC "מינימלי" תוכן ה-DNA (npc) גדלעם פציעה (איור 4א). שגיאת הכיול ממוזערת על-ידי יצירת מגבלה עליונה של NPCm המייצגת סף 4c (איור 4ב).- בתוך הגיליון האלקטרוני, בחר רק גרעיני NPC עם ערכים עבור "m" זה לשקר בתוך 1 סטיית תקן (SD) של המצב (זה מסנן את הרעש מתוך שגיאת פילוח אפשרי).
- בתוך טווח מסונן זה, בחנו את האזורים הגרעיניים ואת עוצמות הכוונות המקבילות שלהם (איור 4ג).
- העריכו את השטח הגרעיני הקטן ביותר בתוך טווח מסונן זה בעוצמה הגרעינית המקסימלית האטומית (כלומר, הנקודה שבה קו העקומה משנה כיוון בערכת הנתונים המסוננת כפי שמודגם על-ידי העיגול האדום באיור 4ג). ערך זה מייצג מצב המעבר של 2N-4N (t) שמעליו מדגימה של גרעיני 4n בעיקר על פני גרעינים מדגם 2n, והתוצאה היא מקסימה של עוצמת ההואכסט.
הערה: ערך זה נקבע באופן אוטומטי על-ידי התוכנה; עם זאת, משתמשי גיליון אלקטרוני יכולים לבחור באופן ידני נקודה זו כגודל המעבר. - חשב את תוכן ה-DNA המינימלי המיוצג על-ידי גודל מעבר זה (tm) בעקבות שלב 6.3.4.2.
- כדי להעריך את הכתף 4N של ערכתהנתונים NPC , להוסיף 1 SD לערך של tm. המספר שהתקבל (איור 4ב) מתאר את הגבול העליון של התוכן NPC מינימלי DNA לשמש ריבוד הגרעינית (S4c).
- חזור על שלבים 6.3.4.3.1-6.3.4.3.5 עבור כל "שליטה" דגימות.
הערה: לדוגמה, באיור 3, כבדי שליטה שלא נפגעו ("control-d0") משמשים כתנאי בקרה. - לחשב את הממוצע 4c ריבוד סף (s4c) עבור "שליטה" דגימות ולהשתמש בזה כדי להסיק את 2 ג (s2 ג) ו 8c (s8c) גבולות עבור תוכן DNA מינימלי (m). ספי הריבוד נוצרים באופן אוטומטי ומאוחסנים על-ידי התוכנה (שלב 6.3.3.3).
הערה: בהתאם לעיצוב הלמידה, ניתן לחשב את ערכי הסף הריבוד הממוצעים עבור כל תנאי או עבור תנאים ספציפיים (לדוגמה, כבד בקרה בריא). עם זאת, תוכנת ניתוח התוכנה הגרעינית מחייבת שאחת מסדרה של 2 קבצים מוגדרת כ"שליטה" לצורך חישוב ערכים מדועתיים יחסיים. - חישוב ערך פלויידי עבור כל הגרעינים באמצעות ערך2 ג S שנוצר בשלב 6.3.4.3.7 לפי:

- בסוגריים מרובעים "פשוט" הפטציט (HNF4α +) הגרעיני לתוך 2c/4c/8c/> 8c, בהתאם לקריטריונים הבאים: "2c" HNF4α + = p ≤ 2; "4c" HNF4α + = 2 < p ≤ 4; "8c" HNF4α + = 4 < p ≤ 8; "> 8c" HNF4α + = 8 < p.
- כדי לשחזר את הדגימה המרחבית של תת-קבוצות הפלואידיות, הפרידו את הנתונים הגרעיניים בתוך כל גיליון אלקטרוני לדוגמה בהתאם לתחומים המתאימים שבהם הם הוכנסו. לאחר מכן השתמש בקואורדינטות x/y גרעיניות (משלב 5.5) כדי להתוות קבוצות מתים של פלואידיות ב-2D (איור 5ג).
- להפריד את הגרעינים לתוך "פשוט" או "מורכב" על פי מורמטריה גרעינית.
- הורד והתקן תוכנה.
Access restricted. Please log in or start a trial to view this content.
תוצאות
שיטה זו שימש כדי למדוד את ההשפעה של פציעה בכיס התינוק על הכבד של העכבר המבוגר על ידי האכלה בעלי חיים עבור 0-21 ימים עם דיאטה hepatotoxic המכיל 0.1% 3, 5-diethoxycarbonyl-1, 4-דיהידרוטסטוסטרון (DDC)17. תוצאות כרוניות DDC האכלה בפציעה hepatocellular גדל פלויידי והרחבת הפורטל של NPCs. המשתמש צריך ...
Access restricted. Please log in or start a trial to view this content.
Discussion
מתואר בגישה גבוהה ובתפוקה גבוהה לניתוח שיפוץ רקמות והערכת הערכה גרעינית של הפטוציט בכבד מורטין. ברגע שהוא מכיר את הנוהל, משתמש יכול לעבד, לצלם ולנתח דגימות מרובות ב-3-5 ימים של תקופה, יצירת ערכות נתונים של ברת גדול המספקים חתימה מפורטת של בריאות הכבד. בהינתן הפשטות של שיטת ההכנה לדוגמה, יחד ע...
Access restricted. Please log in or start a trial to view this content.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו ממומנת על ידי הממשלה הספרדית MINECO מענקים BFU2014-58686-P (LAN) ו-2017-84708-R (DJB). LAN נתמך על ידי האגודה הלאומית מינאקו רמון את החבר RYC-2012-11700 ומתכננת פרס (Comunitat ולנסיה, CDEI-05/20-C), ו-FMN על ידי האזור ValI ו-D שמאל של ולנסיה/2016/020. RP מעוניין להכיר בפרופ ' Ewa ק. פלך למימון. אנו מודים לדוקטור אלישיה מרטיז-רומרו (שירות השירות הסיילאנסה) לקבלת עזרה בפלטפורמת מנתח התאים.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
References
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved