Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une méthode in situ à haut débit pour l’estimation de la ploidy nucléaire hépatocyte chez les souris
Dans cet article
Résumé
Nous présentons une méthode robuste, rentable et flexible pour mesurer les changements dans le nombre d’hépatocytes et le stratagème nucléaire dans des échantillons de tissus fixes/cryoconservés qui ne nécessitent pas de cytométrie d’écoulement. Notre approche fournit une signature puissante à l’échelle de l’échantillon de cytologie hépatique idéale pour suivre la progression des lésions hépatiques et des maladies.
Résumé
Lorsque le foie est blessé, le nombre d’hépatocytes diminue, tandis que la taille des cellules, la taille nucléaire et le stratagème augmentent. L’expansion des cellules non parenchymales telles que les cholangiocytes, les myofibroblastes, les progéniteurs et les cellules inflammatoires indiquent également des lésions hépatiques chroniques, des remodelages tissulaires et la progression de la maladie. Dans ce protocole, nous décrivons une approche simple à haut débit pour calculer les changements dans la composition cellulaire du foie qui sont associés aux dommages, aux maladies chroniques et au cancer. Nous montrons comment les informations extraites des sections tissulaires bidimensionnelles (2D) peuvent être utilisées pour quantifier et calibrer le stratagème nucléaire hépatocyte dans un échantillon et permettre à l’utilisateur de localiser des sous-ensembles de ploidy spécifiques dans le foie in situ. Notre méthode nécessite l’accès à des matériaux hépatiques fixes/congelés, à des réactifs d’immunocytochimie de base et à toute plate-forme d’imagerie à haute teneur de série standard. Il sert d’alternative puissante aux techniques standard de cytométrie de débit, qui exigent la perturbation des tissus fraîchement recueillis, la perte d’information spatiale et le biais potentiel de désagrégation.
Introduction
Les hépatocytes dans le foie de mammifères peuvent subir la cytokinesis au point mort pour produire des cellules binucléaires, et l’endoreplication d’ADN pour produire des noyaux polyploïdes contenant jusqu’à 16N de contenu d’ADN. Augmentation globale du ploidy cellulaire et nucléaire pendant le développement postnatal, le vieillissement et en réponse à divers stress cellulaires1. Le processus de polyploidisation est dynamique et réversible2, bien que sa fonction biologique précise reste floue3. L’augmentation du stratagème est associée à une capacité de prolifération réduite4, à la diversité génétique2,à l’adaptation aux blessures chroniques5 et à la protection contre le cancer6. Les altérations de ploidy d’Hépatocyte se produisent en raison du rythme circadien altéré7, et du sevrage8. Plus particulièrement, le profil de ploidy du foie est modifié par des blessures et la maladie9, et des preuves convaincantes suggèrent que des changements spécifiques de ploidy, tels que l’augmentation des noyaux de 8N ou la perte des hépatocytes de 2N, fournissent des signatures utiles pour le suivi de la maladie du foie gras non-alcoolique (NAFLD) progression3,10, ou l’impact différentiel des infections virales11.
En termes généraux, les lésions hépatiques et la régénération sont associées à une augmentation de la taille des cellules hépatocytes et de la zone nucléaire12, ainsi qu’à une réduction du nombre total d’hépatocytes, en particulier ceux dont la teneur en ADN2N est 10,,11. Les lésions parenchymales dans le foie sont également fréquemment accompagnées par l’expansion des cellules non parenchymales (NPC), y compris les myofibroblastes stromaux, les cellules inflammatoires et les cellules progénitrices bipotentes de foie. Les méthodes à haut débit qui fournissent un profil cytologique quantitatif du nombre de cellules parenchymiques et du stratagème nucléaire, tout en tenant compte des changements dans les PNJ, ont donc un potentiel considérable en tant que recherche et outils cliniques pour suivre la réponse du foie pendant les blessures et les maladies. L’analyse in situ récente convaincante des spectres de ploidy dans des échantillons humains du carcinome hépatocellulaire démontrent également que le ploidy nucléaire est considérablement augmenté dans les tumeurs et est spécifiquement amplifié dans les sous-types de tumeur plus agressifs avec la différenciation et la perte réduites de TP5313. Par conséquent, il y a une forte possibilité que les progrès méthodologiques dans l’évaluation quantitative du stratagème nucléaire aideront dans le profilage pronostique futur du cancer du foie.
Dans ce protocole, une méthodologie flexible à haut débit pour l’analyse comparative des sections des tissus hépatiques de souris est décrite, qui fournit un profilage cytométrique détaillé des nombres d’hépatocytes, la réponse de PNJ et une méthode calibré interne pour estimer le stratagème nucléaire(figure 1). Les hépatocytes se distinguent des PNJ par l’immunolabelling hépatocyte du facteur nucléaire 4 alpha (HNF4MD), avant la caractérisation de la taille nucléaire et de la morphométrie nucléaire. Le « contenu minimal de l’ADN » est estimé pour tous les masques nucléaires circulaires en intégrant l’intensité moyenne hoechst 33342 (un indicateur de la densité d’ADN) avec le volume nucléaire tridimensionnel interpolé (3D). Le contenu minimal d’ADN d’Hépatocyte est alors calibré utilisant des PNJ pour générer un profil de stratagème nucléaire.
L’acquisition d’images, la segmentation nucléaire et l’analyse d’image sont effectuées à l’aide d’images à haute teneur, ce qui permet de dépister de vastes zones de sections hépatiques bidimensionnelles (2D) contenant des dizaines de milliers de cellules. Un programme écrit sur mesure est prévu pour le post-traitement automatisé des données d’analyse d’images à contenu élevé afin de produire un profil de stratagème à l’échelle de l’échantillon pour tous les noyaux hépatocytes circulaires. Ceci est effectué à l’aide d’un logiciel gratuit pour télécharger des logiciels pour calculer le ploidy nucléaire basé sur l’analyse d’image stereologique (SIA)10,11,14,15. La méthodologie SIA a été précédemment validée par cytométrie de flux comme méthode précise, quoique laborieuse, pour estimer le ploidy nucléaire hépatocyte dans le foie14, en supposant la morphologie nucléaire circulaire et une relation monotone entre la taille nucléaire et la teneur en ADN. Dans ce protocole, les deux paramètres nucléaires sont mesurés par l’évaluation de la morphométrie nucléaire et de l’étiquetage Hoechst 33342. Le calcul de la « teneur minimale en ADN » pour chaque masque nucléaire est suivi de l’étalonnage du stratagème nucléaire hépatocyte à l’aide des PNJ, qui ont une teneur connue en ADN de 2 à 4N et servent donc de contrôle interne utile.
Par rapport aux méthodes conventionnelles de cytométrie de débit16, l’approche décrite permet d’évaluer in situ le stratagème nucléaire hépatocyte et ne nécessite pas d’accès à des tissus frais ou à des méthodes de désagrégation qui peuvent biaiser les résultats et être difficiles à normaliser. Comme pour toutes les approches basées sur la SIA, les sous-classes de ploidy nucléaire sont sous-représentées par l’échantillonnage 2D en raison de la section des noyaux plus grands à l’extérieur du plan équatorial. Le profil de ploidy à l’échelle des tissus décrit également le contenu minimal de l’ADN pour tous les masques nucléaires hépatocytes circulaires, et ne fait pas directement de distinction entre les hépatocytes mononucléaires et les cellules binucléaires qui ont deux noyaux discrets (« non touchants ») du même stratagème. Cependant, la simplicité de ce protocole permet d’adapter une marge de manœuvre considérable pour tenir compte de paramètres supplémentaires tels que l’espacement internucléaire ou l’analyse du périmètre cellulaire, ce qui faciliterait l’identification des cellules binucléaires fournissant une évaluation plus détaillée de la ploidy cellulaire.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expériences sur les animaux ont déjà été approuvées par le comité d’éthique du CIPF. Des souris ont été logées dans une installation exempte d’agents pathogènes au Centro de Investigacion Proncipe Felipe (Valence, Espagne), enregistrée en tant qu’éleveur d’animaux expérimentaux, utilisateur et centre d’approvisionnement (reg. no. ES 46 250 0001 002) en vertu des réglementations européennes et espagnoles actuelles en matière de bien-être animal (RD 53/2013).
1. Récolte des tissus et préparation de l’échantillon
REMARQUE : Ce protocole décrit comment congeler les tissus sans fixation préalable ou cryoconservation. Pour les échantillons précédemment fixés/cryoconservés, passez à l’article 2 et ometz l’étape 3.1. Toutes les analyses ont été effectuées à l’aide de souris femelles adultes C57BL/6 âgées de 12 à 16 semaines.
- Sacrifiez les animaux par injection intraperitonéale de fentanyl/pentobarbital suivie d’une dislocation cervicale. Avec la souris face côté ventral vers le haut, ouvrir la cavité abdominale et exposer le foie en saisissant la peau avec des pincettes et l’exécution d’une incision verticale de la base de l’abdomen inférieur à la base du sternum à l’aide de ciseaux chirurgicaux.
- Retirez soigneusement la vésicule biliaire à l’aide d’une fine pince à épinerie, disséquez le foie et rincez le lobule hépatique sélectionné dans une assiette à vaisselle Petri de 10 cm remplie de saline tamponnée par phosphate (PBS).
REMARQUE : Il est recommandé de comparer le même lobe de foie pour chaque animal, dans ce cas le lobe médian a été employé. - Remplissez un cryomold étiqueté avec une température de coupe optimale (OCT) moyenne à température ambiante (RT). Évitez les bulles OCT. S’ils apparaissent, poussez-les au bord du moule à l’aide d’une aiguille ou d’une pipette.
- Encorps le lobule de foie dans un cryomold rempli d’OCT et placez-le immédiatement sur la glace sèche pour assurer la congélation rapide. Entreposez les cryomolds à -80 oC jusqu’à ce que la cryosection.
2. Cryosection
- Transportez des cryomolds sur de la glace sèche pour éviter la dégradation des tissus. Avant d’équilibrer l’équilibre à l’intérieur du cryostat réglé à -20 oC pendant 20 min.
- Échantillon d’éjection en appliquant une pression sur la base du cryomold en plastique. Appliquer le liquide OCT pour réchauffer le disque d’échantillon à RT, positionner dans le cryostat et attacher un échantillon de foie incrusté d’OCT. Appliquez une pression douce et attendez 3 min pour que l’OCT gèle en veillant à ce que l’échantillon colle au disque.
REMARQUE : Évitez de manipuler l’échantillon avec les doigts autant que possible pour éviter la dégradation des tissus. - Verrouillez l’échantillon dans le bras du cryostat et ajustez l’orientation de sorte que le bord de l’échantillon soit parallèle à la lame cryostat. Couper l’échantillon jusqu’à ce que le tissu soit atteint.
- Section de l’échantillon à 6 m d’épaisseur. Placer une diapositive étiquetée recouverte de polyamide sur l’échantillon pendant 5 s pour laisser l’échantillon coller sur la glissière. Placez la glissière à RT pendant 3 à 5 min, puis, pour obtenir de meilleurs résultats, passez directement à la section 3.
REMARQUE : Pour le traitement de plusieurs échantillons congelés frais, des résultats reproductibles ont été obtenus en stockant temporairement des diapositives dans une boîte à glissière sur de la glace sèche jusqu’à ce que tous les échantillons aient été traités. Lorsque vous utilisez cette approche, toutes les diapositives s’équilibrent vers LA RT avant de passer à l’article 3. Des échantillons de paraffine fixe formaline intégrés (FFPE) peuvent être utilisés, bien que l’autofluorescence de fond soit augmentée par cette méthode. Pour passer des échantillons de la FFPE, section à 4 m. Mont en attrapant des sections du bain d’eau de 40 oC sur des toboggans traités en polyamide. Diapositives de chaleur pour 1 h à 60 oC, puis déparaffinize par des lavages RT en série (5 min) dans des bocaux Coplin contenant du xylène (x2), de l’éthanol 100% (x2), 96% (x2), 70% (x1) et dH2O (x1). Pour exposer les antigènes placent des diapositives dans un tampon de citrate pendant 20 min à 90 oC avant de tempérer les diapositives dans PBS à RT. Procédez à l’étape 3.2.
3. Immunolabelling fluorescence
- Fixer les sections tissulaires dans un capot de fumée en appliquant 1 ml de paraformaldéhyde (PFA) de 4 % dans PBS pendant 10 min à RT. Transférer les diapositives dans un bocal Coplin rempli par PBS et laver pendant 3 min à l’aide d’agitation douce (répéter 3x).
REMARQUE : À partir de maintenant jusqu’à la fin du processus d’immunostachation, évitez le séchage de l’échantillon. - Séchez la zone autour de chaque section tissulaire et entourez-vous à l’aide d’un stylo hydrophobe. Perméabilize avec 0,5% surfactant non ionique (c.-à-d., Triton X-100) dans PBS pendant 15 min à RT. Ensuite, laver dans PBS rempli bocal Coplin pendant 3 minutes en utilisant l’agitation douce (répéter 2x).
- Bloquer à l’aide d’une solution filtrée de 1% de sérum bovin albumin (BSA), 5% sérum de cheval, 0,2% surfactant non ionique dans PBS (pour au moins 1 h à RT).
- Incubate avec l’anticorps primaire HNF4MD dilué dans le tampon de blocage pendant la nuit à 4 oC dans une chambre de coloration humide foncée (voir Tableau des matériaux pour les anticorps et les dilutions spécifiques).
- Placer les toboggans dans un bocal Coplin rempli par PBS et laver pendant 3 min à l’aide d’agitation douce (répéter 4x).
- Incubate avec Alexa-488 anticorps secondaires conjugués et Hoechst dilué dans filtré 1% BSA et 0,2% surfactant non ionique dans PBS pendant 2 h à RT dans une chambre de teinture humide foncé (voir Tableau des matériaux pour les anticorps et les dilutions spécifiques).
- Placer les toboggans dans un bocal Coplin rempli par PBS et laver pendant 3 min à l’aide d’agitation douce (répéter 4x). Laver en ddH2O pendant 3 min en utilisant de l’agitation douce (répéter 2x).
- Mount glisse en plaçant deux gouttes de support de montage fluorescent sur un coverlip (24 x 60 mm) et en posant des diapositives dessus, éliminant les bulles en appliquant une légère pression. Pour un stockage à long terme, sceller le housset sur les bords avec du vernis à ongles clair et entreposez-le dans l’obscurité à 4 oC.
- Avant de procéder, vérifiez les diapositives à l’aide d’un microscope à fluorescence classique pour assurer une bonne fixation et immuno-lbeling.
REMARQUE : Voir la figure 2A,B pour les résultats attendus.
4. Acquisition d’images de fluorescence
REMARQUE : Pour cette étape, une plate-forme d’imagerie à haute teneur (Tableau des matériaux) est nécessaire pour prendre en charge l’acquisition automatique d’images de fluorescence.
- Allumez le système d’imagerie et ouvrez un nouveau protocole d’acquisition.
- Sélectionnez l’objectif 10x, notez la zone du champ de vision (dans ce cas 0,6 mm2).
- Définissez des paramètres pour acquérir des images de fluorescence à l’aide des filtres d’excitation et d’émission appropriés (selon l’étape 3.6). Pour Hoechst et Alexa-488, sélectionnez les canaux "DAPI" et "GFP" avec des émissions de 390/18 et 438/24 nm et 432,5/48 et 475/24 nm émission, respectivement.
- Concentrez l’échantillon et assurez-vous que l’intensité du signal n’est pas saturante. Assurez-vous que la capture d’image se fait avec le même temps d’exposition pour toutes les images ou utilisez un système où l’intensité de la fluorescence est corrigée pendant le temps d’exposition.
- Numériser l’échantillon et acquérir suffisamment d’images pour obtenir une couverture complète de la section tissulaire (environ 20 à 50 champs de vision, selon la taille de l’échantillon).
- Examinez la base de données d’images, éliminant manuellement (i) les champs mal concentrés, (ii) ceux aux frontières de chaque section tissulaire (pour éviter les calculs de densité cellulaire biaisés), et (iii) ceux contenant des zones pliées/physiquement endommagées de la section tissulaire si elles sont présentes.
5. Analyse automatisée de l’image de fluorescence
REMARQUE : Cette étape nécessite un logiciel appropriéd’analyse d’images (Tableau des matériaux) capable de : (1) d’identifier automatiquement les noyaux étiquetés Hoechst dans les images à 405 nm (segmentation nucléaire), (2) l’évaluation de l’intensité et de la morphométrie nucléaires moyennes de Hoechst, et (3) l’analyse de seuil pour déterminer le statut de fluorescence nucléaire à 488 nm (HNF4MD). Une certaine formation/expertise de base de l’opérateur est nécessaire pour évaluer visuellement et ajuster les paramètres de segmentation et de seuil au sein du programme afin de s’assurer que les noyaux et le statut HNF4MD/- sont parfaitement fermés(figure 2).
- Dans le logiciel d’analyse d’images, ouvrez le fichier d’acquisition contenant des images Hoechst (405 nm) et HNF4MD (488 nm) de l’étape 4.5, et créez un nouveau protocole d’analyse.
- Définir les longueurs d’onde à utiliser pour la segmentation nucléaire (Hoechst, 405 nm) et pour l’analyse des seuils hépatocyte/NPC (HNF4MD, 488 nm).
- Ajuster les paramètres de segmentation nucléaire du logiciel (tels que la « zone nucléaire minimale » et la « sensibilité » de détection nucléaire) pour s’assurer que les noyaux sont parfaitement séparés.
REMARQUE : Une bonne segmentation des hépatocytes devrait être priorisée par rapport à celle des PNJ. Les noyaux d’hépatocyte sont typiquement arrondis (gamme de taille interquartile : 40 à 64 m2). Les noyaux de PNJ, tels que ceux de l’endothélie sinusoïdale, sont de forme aplatie/elliptique ou irrégulière et généralement plus petites et plus étroitement emballées que celles des hépatocytes (gamme de taille interquartile : 30 à 43 m2). Pour le foie de souris, une superficie minimale de 23 m2 et une « sensibilité » de détection de 65 % ont été utilisées (voir la figure 2C,D pour les résultats attendus). La sensibilité détermine comment les grappes de pixels sont reconnues comme des noyaux individuels en fonction de leur intensité et doivent être testées empiriquement pour chaque échantillon défini par l’utilisateur avant de procéder à l’analyse automatisée de l’image. - Modifier l’intensité du seuil à 488 nm pour assurer une gating optimale des hépatocytes (HNF4MD) et des cellules non parenchymales (HNF4MD-).
REMARQUE : Voir la figure 2C,D pour les résultats attendus. La valeur de l’intensité du seuil est relative et dépendra de l’efficacité de coloration et des paramètres d’acquisition tels que l’intensité laser. Il doit donc être standardisé par l’utilisateur. Utilisez les cellules HNF4- connues comme les cellules endothéliales et les PNJ périportaires comme un contrôle négatif interne et des noyaux binucléaires hépatocytes comme référence positive pour la coloration. Testez les paramètres d’analyse à l’aide d’un petit nombre d’images pour assurer une bonne segmentation nucléaire et une ségrégation des seuils d’intensité avant d’appliquer les paramètres d’analyse à l’ensemble du jeu de données. - Sélectionnez les paramètres nucléaires suivants à quantifier : (1) zone nucléaire basée sur la coloration Hoechst (m2), (2) le facteur d’élongation nucléaire (RU), (3) le facteur d’élongation nucléaire (rapport moyen de l’axe court du noyau à l’axe long du noyau, lorsqu’un objet centré symétrique [non allongé] a une valeur de 1, (4) Nuc 1/(facteur de forme), signifie l’indice de « rondeur » nucléaire calculé par périmètre 2/(4 x zone). Les valeurs vont de 1 à l’infini, où 1 est un cercle parfait, (5) le statut HNF4 (positif-1 ou négatif-0), et (6) les coordonnées nucléaires x/y basées sur le « centre de gravité » (cg), une méthode pour localiser le centre de l’objet à partir d’images à l’échelle grise avec une précision sous-pixel.
- Exécutez l’analyse pour tous les ensembles de données d’échantillons et exportez les données numériques de l’étape 5.5 au logiciel de tableur.
6. Analyse des données
REMARQUE : L’étape d’analyse des données peut être effectuée à l’aide de n’importe quel logiciel de tablement standard.
- Calculez les nombres de cellules hépatocytes et non hépatocytes.
- Calculer la superficie totale de la section hépatique analysée pour chaque échantillon en multipliant le nombre de champs de vue par la zone de vision (étape 4.2).

- En travaillant avec des fichiers de feuilles de calcul générés pour chaque section hépatique, filtrez les données en ne sélectionnant que les noyaux HNF4MD. Calculez le nombre total de noyaux HNF4MD analysés et divisez-le par la superficie totale analysée pour obtenir la densité moyenne des hépatocytes pour chaque échantillon(figure 2F).

- Effectuez le même calcul pour les cellules non parenchymales en filtrant la feuille de calcul des cellules HNF4MD(figure 2E).
- Calculer la superficie totale de la section hépatique analysée pour chaque échantillon en multipliant le nombre de champs de vue par la zone de vision (étape 4.2).
- Calculer la distribution de la taille nucléaire hépatocyte.
- À l’aide d’un logiciel de feuille de calcul, filtrez les données pour sélectionner uniquement les noyaux HNF4MD.
- Valeurs de parcelle de zone nucléaire dans un histogramme (figure 2G). Réglez la largeur du bac à 5 m2.
REMARQUE : Les valeurs de fréquence peuvent être corrigées pour la zone (noyau/mm2) par étape 6.1.1.
- Effectuez l’analyse du stratagème nucléaire hépatocyte.
REMARQUE : Les données de la feuille de calcul de l’étape 5.6 sont utilisées pour générer un profil de stratagème nucléaire pour chaque échantillon. Ce processus a été automatisé et peut être exécuté à l’aide d’un logiciel écrit personnalisé qui est librement disponible pour télécharger avec des informations à l’appui et des jeux de données de démonstration àhttps://github.com/lukeynoon(voirFichiers supplémentaires). Le code source est fourni aux utilisateurs qui souhaitent adapter la méthodologie. Une description de l’algorithme, ainsi que des instructions d’installation et d’utilisation sont décrites ci-dessous. Le programme utilise les données des feuilles de calcul pour séparer automatiquement les noyaux d’hépatocytes en deux groupes; (1) ceux avec des noyaux circulaires "simples" et (2) des noyaux non circulaires "complexes" représentatifs des cellules binucléaires avec un ploidy de 2c. La teneur minimale en ADN nucléaire (fonction de la zone nucléaire et de la densité de l’ADN) est ensuite calculée pour tous les noyaux « simples ». Une étape ultérieure calibre alors automatiquement le ploidy nucléaire hépatocyte HNF4MD en utilisant les noyaux HNF4MD comme un contrôle interne connu de 2 à 4N.- Téléchargez et installez un logiciel.
- Téléchargez l’application emballée à partir de: https://github.com/lukeynoon
- Lancement MATLAB. Naviguez vers l’onglet APP de la piste d’outils, cliquez sur Installer App et ouvrez l’application téléchargée appelée "Ploidy_Application.mlappinstall ". Un message apparaîtra pour confirmer l’installation réussie.
REMARQUE : L’application est maintenant prête à l’emploi et restera dans l’onglet APP de la recherche d’outils.
- Formatez les données d’entrée.
REMARQUE : Avant l’analyse automatisée du stratagème nucléaire, tous les fichiers de feuilles de calcul contenant des données d’imagerie à contenu élevé (étape 5.6) doivent être stockés et formatés selon les instructions suivantes.- Dans chaque fichier de données exporté (. XLS 97-2004 cahier de travail) de l’étape 5.6, comprennent une feuille appelée "Mesures cellulaires" contenant toutes les données requises pour l’analyse de stratagème énoncée dans les colonnes(figure 3A). Assurez-vous que la disposition de la feuille de calcul, y compris les noms d’en-tête de colonne, reste inchangée par rapport à celle de la figure 3A, car la méthode d’analyse trouve les données correctes de la colonne en recherchant ces noms (voir les jeux de données de démonstration dans les fichiers supplémentaires pour référence). Si, par exemple, le logiciel d’analyse d’images à contenu élevé ne produit pas de colonne « Flux lumineux »(figure 3A), insérez manuellement une colonne « Flux lumineux » au même endroit, c’est-à-dire colonne K et remplissez-la de zéros.
- Pour chaque condition expérimentale (p. ex., « Injured-d14 »), fournir un ensemble de données de contrôle, qui sera utilisé pour calculer le contrôle interne pour l’étalonnage du stratagème nucléaire de 2 à 4N (étape 6.3.4.3). Ici, sélectionnez des échantillons de foie de litières adultes non traitées (« Contrôle-d0 »; Figure 3B-D).
- Pour les répliques biologiques (par condition), conservez chaque feuille de calcul dans son propre dossier (comme dans la figure 3B). Nommez le dossier préfixe progressivement, par exemple, « Sample1, Sample2, Sample3... SampleN ", selon les noms de fichiers contenus à l’intérieur. Par conséquent, chaque dossier de jeu de données (p. ex., « Contrôle-d0 ») doit contenir une série de sous-plis (« Échantillon1 », « Échantillon2 », etc.) contenant chacun un fichier de feuille de calcul du même nom correspondant.
- Exécutez l’application.
- Au sein de MATLAB, lancez le « Ploidy_Application » en cliquant sur l’icône dans l’onglet MY APPS de la sortie d’outils(figure 3C). L’interface utilisateur graphique Ploidy_Application (GUI) apparaîtra(figure 3C).
- Cliquez sur le bouton Path to control data pour naviguer vers le dossier dans lequel les données de contrôle se réplique résident (par exemple, "Control-d0"). Ce chemin de données apparaîtra ensuite dans l’interface (p. ex., /Utilisateurs/Desktop/Control-d0).
- Ensuite, dans le type « préfixe de dossier », le nom à donner aux fichiers de sortie (p. ex., « Échantillon »).
REMARQUE : Ce préfixe peut être modifié à n’importe quel texte, à condition que les dossiers et les noms de fichiers restent progressivement nommés. - Cliquez sur le bouton Chemin vers d’autres données et naviguez vers le dossier dans lequel les données comparatives se reproduise résident (par exemple, «Injured-d14»). Ce chemin de données apparaîtra ensuite dans l’interface (p. ex., /Utilisateurs/Desktop/Injured-d14).
- Cliquez sur Run!. Lorsque l’analyse est terminée, la barre d’état se lit comme suit : « Analyse complète !.. ».
REMARQUE : La demande fera état, pour chaque échantillon, de la stratification des noyaux « simples » en 2n, 2n-4n, 4n-8n et 8n ' en termes de dénombrement absolu et en pourcentage du total(figure 3D). Ces fichiers seront automatiquement enregistrés dans chaque dossier d’échantillon comme: "Count_2n.txt", "Count_2n_to_4n.txt ", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". Le Ploidy_Application enregistrera automatiquement une liste pour chaque échantillon, de toutes les estimations de stratagèmes individuelles pour les noyaux « simples » hépatocytes et non-hépatocytes dans « Ploidy_All_Hepatocytes.txt » et « Ploidy_NonHepatocytes.txt ». Pour l’ensemble de données de contrôle, la méthode enregistre également les seuils minimaux de contenu d’ADN calculés pour la stratification du stratagème (voir l’étape 6.3.4.3.7) dans un fichier nommé "Normalised_Thresholds_Control". Enfin, l’application produira un dossier pour le contrôle et les données de l’état comparatif sélectionnée appelées « Résumé ». Ce dossier contient deux sous-plieurs, "Ploidy" et "Stratification" qui contiennent les moyennes de tous les échantillons fournis(figure 3D).
- Description de la méthodologie.
REMARQUE : La section suivante décrit en détail la méthodologie utilisée par le logiciel d’analyse du ploidy nucléaire. Si l’utilisateur choisit de ne pas utiliser l’application, ces étapes peuvent être suivies à l’aide d’un logiciel de feuille de calcul pour calculer manuellement le profil de stratagème nucléaire.- Séparer les noyaux en «simple» ou «complexe» selon la morphométrie nucléaire.
- Calculez un « indice de circularité » pour tous les noyaux, défini comme le « facteur d’allongement » nucléaire divisé par le « Nuc 1/(facteur de forme) », où une valeur de 1,0 indique un cercle parfait.
REMARQUE : « L’allongement nucléaire » et le « Nuc 1/(facteur de forme) » sont deux mesures distinctes de la « circularité » d’un objet qui évaluent des critères morphométriques complémentaires et non chevauchants. Le premier mesure les axes longs et courts d’un objet, tandis que le second compare la longueur du périmètre d’un objet à celle de sa zone. Pour renforcer la définition de la circularité nucléaire utilisée dans ce protocole, ces deux mesures ont été combinées en un seul « indice de circularité ». Une approche antérieure pour estimer la ploidie nucléaire à l’aide de la méthodologie décrite n’utilisait que l’allongement nucléaire17. Bien que des résultats acceptables aient été obtenus à l’aide de cette approche, les auteurs ont observé qu’un « indice de circularité » composite améliore la discrimination des noyaux choisis manuellement par des hépatocytes mononucléaires et binucléaires (données non montrées). - Classer les noyaux avec un indice de circularité de 0,8 comme «complexe» et ceux -gt; 0.8 comme «simple».
- Calculez un « indice de circularité » pour tous les noyaux, défini comme le « facteur d’allongement » nucléaire divisé par le « Nuc 1/(facteur de forme) », où une valeur de 1,0 indique un cercle parfait.
- Estimer le contenu « minimal » de l’ADN (m) pour tous les noyaux « simples ».
- Calculer le rayon nucléaire (r) à l’aide de la formule :

- Calculer le volume nucléaire (v) à l’aide du volume d’une formule de sphère :
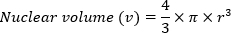
- Générer une valeur relative pour le contenu minimal de l’ADN (m) à l’aide de la formule :

- Calculer le rayon nucléaire (r) à l’aide de la formule :
- Calibrer l’ensemble de données à l’aide des noyaux NPC (HNF4MD) comme un contrôle interne de 2 à 4N.
REMARQUE : Les PNJ ont un contenu d’ADN de 2 à 4N selon l’état du cycle cellulaire. Par conséquent, la valeur moyenne du contenu génétique « minimal » de PNJ (PNJm)augmente avec les blessures(figure 4A). L’erreur d’étalonnage est minimisée en établissant une limite supérieure de PNJm représentant un seuil de 4c(figure 4B).- Dans la feuille de calcul, sélectionnez uniquement les noyaux de PNJ avec des valeurs pour "m" qui se trouvent dans 1 déviation standard (SD) du mode (ce filtre le bruit d’une éventuelle erreur de segmentation).
- Dans cette plage filtrée, examinez les zones nucléaires et leurs intensités moyennes correspondantes Hoechst(figure 4C).
- Estimez la plus petite zone nucléaire dans cette plage filtrée avec l’intensité maximale de Hoechst nucléaire (c.-à-d., le point où la ligne de la courbe change de direction dans le jeu de données filtré comme illustré par le cercle rouge de la figure 4C). Cette valeur représente un état de transition (t) de 2N à 4N au-dessus duquel l’échantillonnage des noyaux de 4c prédomine plus de 2c noyaux, ce qui entraîne une maxima d’intensité moyenne hoechst.
REMARQUE : Cette valeur est automatiquement déterminée par le logiciel ; toutefois, les utilisateurs de feuilles de calcul peuvent sélectionner manuellement ce point comme la taille transitoire. - Calculez le contenu minimal de l’ADN représenté par cette taille transitoire (tm) en suivant l’étape 6.3.4.2.
- Pour estimer l’épaule 4N du jeu de données NPCm, ajoutez 1 SD à la valeur de tm. Le nombre qui en résulte (figure 4B) décrit la limite supérieure de la teneur minimale en ADN de PNJ à utiliser pour la stratification du ploidy nucléaire (S4c).
- Étapes répétées 6.3.4.3.1-6.3.4.3.5 pour tous les échantillons de "contrôle".
REMARQUE : Par exemple, dans la figure 3,les foies témoins non blessés (« Contrôle-d0 ») sont utilisés comme condition de contrôle. - Calculer un seuil moyen de stratification de 4c (S4c) pour les échantillons de « contrôle » et l’utiliser pour extrapoler les limites 2c (S2c) et 8c (S8c) pour un contenu minimal d’ADN (m). Les seuils de stratification sont automatiquement générés et stockés par le logiciel (étape 6.3.3.3).
REMARQUE : Selon la conception de l’étude, les valeurs moyennes de seuil de stratification peuvent être calculées pour chaque condition ou pour des conditions spécifiques (p. ex., foie de contrôle sain). Cependant, le logiciel d’analyse du ploidy nucléaire exige que l’un des 2 fichiers soit désigné comme « contrôle » aux fins du calcul des valeurs relatives du stratagème. - Calculez une valeur de stratagème pour tous les noyaux en utilisant la valeur S2c générée à l’étape 6.3.4.3.7 selon :

- Stratifier les noyaux d’hépatocyte « simple » (HNF4MD) en tranches de 2c/4c/8c/8c selon les critères suivants : « 2c » HNF4MD "4c" HNF4MD à 2 lt; p 4; "8c" HNF4MD à 4 lt; p 8; HNF4md de 8 ans et 8 h 00
- Pour reconstruire le modelage spatial des sous-groupes de ploidy, séparer les données nucléaires dans chaque feuille de calcul de l’échantillon en fonction des champs correspondants dans lesquels ils ont été acquis. Ensuite, utilisez les coordonnées nucléaires x/y associées (de l’étape 5.5) aux sous-groupes de ploidy de parcelle en 2D(figure 5C).
- Séparer les noyaux en «simple» ou «complexe» selon la morphométrie nucléaire.
- Téléchargez et installez un logiciel.
Access restricted. Please log in or start a trial to view this content.
Résultats
Cette méthode a été utilisée pour mesurer l’impact des lésions cholestatiques sur le foie de souris adulte en nourrissant les animaux pendant 0-21 jours avec un régime hépatoxique contenant 0,1% 3,5-diethoxycarbonyl-1,4-dihydrocollidine (DDC)17. L’alimentation chronique de DDC a comme conséquence des dommages hépatocellulaires ont augmenté le stratagème et l’expansion périportaire des PNJ. L’utilisateur doit être conscient que la souche de sour...
Access restricted. Please log in or start a trial to view this content.
Discussion
Une approche à haut contenu et à haut débit pour l’analyse du remodelage et de l’estimation des tissus du ploidy nucléaire hépatocyte dans le foie murin est décrite. Une fois familier avec la procédure, un utilisateur peut traiter, imager et analyser plusieurs échantillons dans une période de 3 à 5 jours, générant de grands jeux de données testables qui fournissent une signature détaillée de la santé hépatique. Compte tenu de la simplicité de la méthode de préparation de l’échantillon, ainsi qu...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux ont été financés par le gouvernement espagnol MINECO subventions BFU2014-58686-P (LAN) et SAF-2017-84708-R (DJB). LAN a été soutenu par une bourse nationale MINECO Ramon y Cajal RYC-2012-11700 et plan GenT (Comunitat Valenciana, CDEI-05/20-C), et FMN par un étudiant régional ValI-D de la Generalitat ACIF de Valence/2016/020. RP tient à remercier le professeur Ewa K. Paluch pour son financement. Nous remercions la Dre Alicia Martinez-Romero (service de cytométrie CIPF) pour l’aide de la plate-forme IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Références
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon