È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un metodo ad alto throughput in Situ per la stima della ploidia nucleare epatica nei topi
In questo articolo
Riepilogo
Vi presentiamo un metodo robusto, conveniente e flessibile per misurare i cambiamenti nel numero di epatociti e nella ploidia nucleare all'interno di campioni di tessuto fisso/crioconservato che non richiede citometria di flusso. Il nostro approccio fornisce una potente firma a livello campione di citologia epatica ideale per monitorare la progressione delle lesioni epatiche e della malattia.
Abstract
Quando il fegato è ferito, il numero di epatociti diminuisce, mentre le dimensioni della cellula, le dimensioni del nucleare e l'aumento della ploidia. L'espansione di cellule non parenchychymal come cholangiociti, miofibromi, progenitori e cellule infiammatorie indicano anche danni epatici cronici, rimodellamento dei tessuti e progressione della malattia. In questo protocollo, descriviamo un semplice approccio ad alto contenuto di velocità per calcolare i cambiamenti nella composizione cellulare del fegato che sono associati a lesioni, malattie croniche e cancro. Mostriamo come le informazioni estratte dalle sezioni di tessuto bidimensionali (2D) possono essere utilizzate per quantificare e calibrare la ploidia nucleare degli epatociti all'interno di un campione e consentire all'utente di individuare specifici sottoinsiemi di ploidia all'interno del fegato in situ. Il nostro metodo richiede l'accesso a materiale epatico fisso/congelato, reagenti di immunocitochimica di base e qualsiasi piattaforma di imaging standard ad alto contenuto. Serve come una potente alternativa alle tecniche di citometria di flusso standard, che richiedono l'interruzione del tessuto appena raccolto, la perdita di informazioni spaziali e potenziale pregiudizio di disaggregazione.
Introduzione
Gli epatociti nel fegato dei mammiferi possono sottoporsi a citocinesi bloccata per produrre cellule binucleari, e l'endoreplicare del DNA per produrre nuclei poliploidi contenenti fino a 16N contenuto di DNA. Aumento complessivo della ploidia cellulare e nucleare durante lo sviluppo postnatale, l'invecchiamento e in risposta a diversi stress cellulari1. Il processo di poliploidizzazione è dinamico e reversibile2, anche se la sua precisa funzione biologica rimane poco chiara3. L'aumento della ploidia è associato alla ridotta capacità proliferiva4,alla diversità genetica2,all'adattamento alle lesioni croniche5 e alla protezione del cancro6. Le alterazioni della ploidia epatocita si verificano a seguito del ritmo circadiano alterato7e dello svezzamento8. In particolare, il profilo ploidiario del fegato è alterato da lesioni e malattia9, e prove convincenti suggeriscono che cambiamenti specifici ploidiari, come aumento dei nuclei di 8N dollari o perdita di 2N epatociti, forniscono firme utili per monitorare la progressione della malattia grassa non alcolica (NAFLD)3,10, o l'impatto differenziale delle infezioni virali11.
In termini generali, le lesioni e la rigenerazione del fegato sono associate all'aumento delle dimensioni delle cellule epatociti e dell'area nucleare12,insieme alla riduzione del numero complessivo di epatociti, in particolare quelli con 2N contenuto di DNA10,11. La lesione parenchimale nel fegato è spesso accompagnata anche dall'espansione di cellule non parenchymal (NPC), tra cui miofibromi stromali, cellule infiammatorie e cellule progenitrici epatiche bipotenti. I metodi ad alto contenuto di velocità di sviluppo che forniscono un profilo citologico quantitativo del numero di cellule parenchiche e della ploidia nucleare, pur tenendo conto dei cambiamenti nei PNG, hanno quindi un notevole potenziale come ricerca e strumenti clinici per monitorare la risposta del fegato durante lesioni e malattie. Icodraggi recenti analisi in situ degli spettri ploidi in campioni umani di carcinoma epatocellulare dimostrano anche che la ploidia nucleare è drammaticamente aumentata all'interno dei tumori ed è specificamente amplificata in sottotipi tumorali più aggressivi con ridotta differenziazione e perdita di TP5313. Pertanto, vi è una forte possibilità che i progressi metodologici nella valutazione quantitativa della ploidia nucleare contribuiscano alla profilazione prognostica futura del cancro del fegato.
In questo protocollo, viene descritta una metodologia flessibile ad alto rendimento per l'analisi comparativa delle sezioni del tessuto epatico del topo, che fornisce una profilazione citometrica dettagliata dei numeri di epatociti, la risposta NPC e un metodo calibrato internamente per la stima della ploidia nucleare (Figura 1). Gli epatociti si distinguono dai PNG per immunoetichettazione del fattore nucleare 4 alfa (HNF4) del fattore nucleare dell'epatocite, prima della caratterizzazione delle dimensioni nucleari e della morfometria nucleare. Il "contenuto minimo di DNA" è stimato per tutte le maschere nucleari circolari integrando l'intensità media di Hoechst 33342 (un proxy per la densità del DNA) con volume nucleare tridimensionale interpolato (3D). Il contenuto minimo di DNA di Epatocite viene quindi calibrato utilizzando i PNG per generare un profilo di ploidia nucleare.
L'acquisizione di immagini, la segmentazione nucleare e l'analisi delle immagini vengono eseguite utilizzando l'imaging ad alto contenuto, consentendo la screening di ampie aree di sezioni epatiche bidimensionali (2D) contenenti decine di migliaia di cellule. Viene fornito un programma scritto su misura per la post-elaborazione automatica di dati di analisi delle immagini ad alto contenuto per produrre un profilo di ploidia a livello di campione per tutti i nuclei circolari di epatociti. Questa operazione viene eseguita utilizzando software gratuito per scaricare per calcolare la ploidia nucleare basata sull'analisi stereologica delle immagini (SIA)10,11,14,15. La metodologia SIA è stata precedentemente convalidata dalla citometria di flusso come un metodo accurato, anche se laborioso, per stimare la ploidia nucleare epatocitica nel fegato14, assumendo una morfologia nucleare circolare e una relazione monotonica tra dimensione nucleare e contenuto di DNA. In questo protocollo, entrambi i parametri nucleari sono misurati mediante valutazione della morfometria nucleare e dell'etichettatura Hoechst 33342. Il calcolo del "contenuto minimo di DNA" per ogni maschera nucleare è seguito dalla calibrazione della ploidia nucleare epatocite utilizzando NPC, che hanno un noto 2-4N contenuto di DNA e quindi servono come un utile controllo interno.
Rispetto ai metodi convenzionali di citometria di flusso16, l'approccio descritto consente di valutare la ploidia nucleare epatocite in situ e non richiede l'accesso a tessuti nuovi o metodi di disaggregazione che possono biasizzare i risultati ed essere difficili da standardizzare. Come per tutti gli approcci basati su SIA, le sottoclassi di ploidia nucleare >2N sono sottorappresentate dal campionamento 2D a causa della sezionamento di nuclei più grandi al di fuori del piano equatoriale. Il profilo ploidiario a livello di tessuto descrive anche il contenuto minimo di DNA per tutte le maschere nucleari circolari degli epatociti e non discrimina direttamente tra epatociti mononucleari e cellule binucleari che hanno due nuclei discreti ("non toccanti") dello stesso ploidio. Tuttavia, la semplicità di questo protocollo consente di adattare in modo considerevole per tenere conto di parametri aggiuntivi come la spaziatura internucleare o l'analisi del perimetro cellulare, che faciliterebbero l'identificazione delle cellule binucleari fornendo una valutazione più dettagliata della ploidia cellulare.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti gli esperimenti sugli animali sono stati precedentemente approvati dal comitato etico del CIPF. I topi sono stati alloggiati in una struttura senza agenti patogeni presso il Centro de Investigaciàn Pràncipe Felipe (Valencia, Spagna), registrati come allevatore sperimentale di animali, utente e centro di approvvigionamento (reg n. n. ES 46 250 0001 002) ai sensi dell'attuale normativa europea e spagnola in materia di benessere degli animali (RD 53/2013).
1. Raccolta dei tessuti e preparazione del campione
NOTA: Questo protocollo descrive come congelare il tessuto senza previa fissazione o crioconservazione. Per i campioni precedentemente fissati/crioconservati procedere alla sezione 2 e omettere il passaggio 3.1. Tutte le analisi sono state eseguite utilizzando topi adulti C57BL/6 di età compresa tra 12 e 16 settimane.
- Sacrificare gli animali da iniezione intraperitoneale di fentanil/pentobarbitalseguita seguita da lussazione cervicale. Con il topo rivolto verso il lato ventrale verso l'alto, aprire la cavità addominale ed esporre il fegato afferrando la pelle con una pinzetta ed eseguendo un'incisione verticale dalla base dell'addome inferiore alla base dello sterno utilizzando forbici chirurgiche.
- Rimuovere con attenzione la cistifellea con una pinzetta fine, sezionare il fegato e sciacquare il lobulo di fegato selezionato in un piatto petri da 10 cm riempito con salina tamponata da fosfato (PBS).
NOTA: Si consiglia di confrontare lo stesso lobo epatico per ogni animale, in questo caso è stato utilizzato il lobo mediano. - Riempire un criomuffsi etichettato con una temperatura di taglio ottimale (OCT) media a temperatura ambiente (RT). Evitare le bolle dello Strumento di personalizzazione di Office. Se appaiono, spingerli al bordo dello stampo utilizzando una punta ago o pipetta.
- Incorporare il lobulo di fegato in un criomold RIEMPIto dello OCT e posizionarlo immediatamente sul ghiaccio secco per garantire un rapido congelamento. Conservare criomuffsi a -80 gradi centigradi fino a criosezione.
2. Criosezione
- Trasportare criomuffsi sul ghiaccio secco per evitare la degradazione dei tessuti. Prima della criosezione equilibrata all'interno della criostat impostare a -20 gradi centigradi per 20 minuti.
- Espellere il campione applicando pressione alla base del criomuffino di plastica. Applicare lo Strumento di personalizzazione di Office liquido sul disco campione riscaldato in RT, posizionare in criostato e collegare un campione di fegato incorporato dello Strumento di personalizzazione di Office. Applicare una leggera pressione e attendere 3 min per OCT per congelare assicurandosi che il campione si attacchi al disco.
NOTA: Evitare di maneggiare il campione con le dita il più possibile per eludere la degradazione del tessuto. - Bloccare il campione nel braccio del criostato e regolare l'orientamento in modo che il bordo del campione sia parallelo alla lama del criostato. Tagliare nel campione fino a raggiungere il tessuto.
- Sezionare il campione con uno spessore di 6 m. Posizionare un vetrino rivestito in poliammide etichettato sul campione per 5 s per far aderire il campione sul vetrino. Posizionare la diapositiva in corrispondenza di RT per 3-5 min, quindi, per ottenere i migliori risultati, procedere direttamente alla sezione 3.
NOTA: Per l'elaborazione di più campioni freschi, sono stati ottenuti risultati riproducibili conservando temporaneamente i vetrini in una scatola di diapositive su ghiaccio secco fino a quando tutti i campioni non sono stati elaborati. Quando si utilizza questo approccio, consentire a tutte le diapositive di raggiungere l'acognitazione a RT prima di procedere alla sezione 3. È possibile utilizzare campioni di formalina di paraffina fissa incorporata (FFPE), anche se l'autofluorescenza di sfondo viene aumentata con questo metodo. Per procedere da campioni di FFPE, sezione a 4 m. Monte catturando sezioni dal bagno d'acqua a 40 gradi centigradi su vetrini trattati con poliammide. Scivoli di calore per 1 h a 60 gradi centigradi, quindi deparaffinare da lavamenti CASUAL seriali (5 min) in vasetti Coplin contenenti xilene (x2), etanolo 100% (x2), 96% (x2), 70% (x1) e dH2O (x1). Per esporre gli antigeni, posizionare i vetrini nel tampone di citrati per 20 min a 90 gradi centigradi prima di temperare i vetrini in PBS a RT. Procedere al passaggio 3.2.
3. Immunolabelling con fluorescenza
- Fissare le sezioni del tessuto in un cappuccio fumatore applicando 1 mL del 4% di paraformaldeide (PFA) in PBS per 10 min a RT. Trasferire i vetrini in un barattolo Coplin riempito di PBS e lavare per 3 min utilizzando agitazione delicata (ripetere 3x).
NOTA: Da ora fino alla fine del processo di immunostaining, evitare l'essiccazione del campione. - Asciugare l'area intorno a ogni sezione di tessuto e circondare utilizzando una penna idrofobica. Permeabilizza con 0,5% di surfactant nonionico (cioè Triton X-100) in PBS per 15 min a RT. Quindi lavare in PBS riempito barattolo Coplin per 3 min utilizzando agitazione delicata (ripetere 2x).
- Bloccare utilizzando una soluzione filtrata di albumina di siero bovino (BSA), 5% siero di cavallo, 0,2% surfactant nonionico in PBS (per almeno 1 h a RT).
- Incubare con l'anticorpo primario HNF4, diluito nel buffer di blocco durante la notte a 4 gradi centigradi in una camera di colorazione umida scura (vedi Tabella dei materiali per anticorpi e diluizioni specifiche).
- Mettere i vetrini in un barattolo Coplin riempito con PBS e lavare per 3 min utilizzando agitazione delicata (ripetere 4x).
- Incubazione con Alexa-488 anticorpo secondario coniugato e Hoechst diluito in filtrato 1% BSA e 0.2% surfactant nonionico in PBS per 2 h a RT in una camera di colorazione umida scura (vedi Tabella dei materiali per anticorpi e diluizioni specifiche).
- Mettere i vetrini in un barattolo Coplin riempito con PBS e lavare per 3 min utilizzando agitazione delicata (ripetere 4x). Lavare in ddH2O per 3 min con agitazione delicata (ripetere 2x).
- Montare i vetrini posizionando due gocce di supporto di montaggio fluorescente su un coperchio (24 x 60 mm) e posando i vetrini su di esso, eliminando le bolle applicando una leggera pressione. Per la conservazione a lungo termine, sigillare coverslip ai bordi con smalto trasparente e conservare al buio a 4 gradi centigradi.
- Prima di procedere, controllare i vetrini utilizzando un microscopio a fluorescenza convenzionale per garantire una buona fissazione e immunolabeling.
NOTA: Vedere la figura 2A,B per i risultati previsti.
4. Acquisizione dell'immagine di fluorescenza
NOTA: per questo passaggio, è necessaria una piattaforma di imaging ad alto contenuto (Table of Materials) che supporti l'acquisizione automatica di immagini a fluorescenza.
- Accendere il sistema di imaging e aprire un nuovo protocollo di acquisizione.
- Selezionare l'obiettivo 10x, notare l'area del campo visivo (in questo caso 0,6 mm2).
- Impostare i parametri per acquisire immagini a fluorescenza utilizzando i filtri di eccitazione ed emissione appropriati (secondo il passaggio 3.6). Per Hoechst e Alexa-488, selezionare i canali "DAPI" e "GFP" rispettivamente con eccitazione 390/18 e 438/24 nm e 432,5/48 e 475/24 nm emission.
- Mettere a fuoco il campione e assicurarsi che l'intensità del segnale non sia saturante. Assicurarsi che l'acquisizione dell'immagine venga eseguita con lo stesso tempo di esposizione per tutte le immagini o utilizzare un sistema in cui l'intensità della fluorescenza viene corretta per il tempo di esposizione.
- Scansiona il campione e acquisisci immagini sufficienti per ottenere una copertura completa della sezione tissutale (circa 20-50 campi di vista, a seconda delle dimensioni del campione).
- Esaminare il database delle immagini, eliminando manualmente (i) i campi poco focalizzati, (ii) quelli ai bordi di ogni sezione tissutale (per evitare di inclinare i calcoli della densità delle cellule) e (iii) quelli che contengono aree piegate/fisicamente danneggiate della sezione dei tessuti, se presenti.
5. Analisi automatizzata delle immagini a fluorescenza
NOTA: questo passaggio richiede un adeguato software di analisi delle immagini (Tabella dei materiali) in grado di: (1) identificare automaticamente I nuclei etichettati da Hoechst all'interno di immagini a 405 nm (segmentazione nucleare), (2) valutare l'intensità e la morfometria nucleare di Hoechst e (3) per determinare lo stato di fluorescenza nucleare a 488 nm (HNF4). Per valutare e regolare visivamente i parametri di segmentazione e di soglia all'interno del programma, è necessaria una formazione/competenza dell'operatore di base per valutare e regolare visivamente i parametri di segmentazione e di soglia all'interno del programma, in modo da garantire che i nuclei e lo stato di HNF4//- siano perfettamente recintati (Figura 2).
- Nel software di analisi delle immagini aprire il file di acquisizione contenente le immagini di Hoechst (405 nm) e HNF4 (488 nm) del passaggio 4.5 e creare un nuovo protocollo di analisi.
- Definire le lunghezze d'onda da utilizzare per la segmentazione nucleare (Hoechst, 405 nm) e per l'analisi delle soglie di epatocite/NPC (HNF4, 488 nm).
- Regolare i parametri di segmentazione nucleare del software (come "area nucleare minima" e "sensibilità" di rilevamento nucleare) per garantire che i nuclei siano segregati in modo ottimale.
NOTA: Una buona segmentazione degli epatociti dovrebbe essere prioritaria rispetto a quella dei PNG.2 I nuclei NPC, come quelli degli endotili sinusoidali, sono appiattiti/ellittici o di forma irregolare e generalmente più piccoli e più imballati rispetto a quelli degli epatociti (gamma di dimensioni interquartile: 30-43m- 2). Per il fegato di topo, sono stati utilizzati un'area nucleare minima di 23 m2 e un rilevamento di "sensibilità" del 65% (vedere la Figura 2C,D per i risultati attesi). La sensibilità determina il modo in cui i cluster di pixel vengono riconosciuti come singoli nuclei in base alla loro intensità e deve essere testata empiricamente per ogni campione impostato dall'utente prima di procedere con l'analisi automatica delle immagini. - Modificare l'intensità della soglia a 488 nm per garantire un'analisi ottimale degli epatociti (HNF4) e delle celle non parenchymale (HNF4-).
NOTA: Vedere la figura 2C,D per i risultati attesi. Il valore dell'intensità di soglia è relativo e dipenderà dall'efficienza di colorazione e dalle impostazioni di acquisizione come l'intensità laser. Dovrebbe quindi essere standardizzato dall'utente. Utilizzare cellule note di HNF4, come cellule endoteliali e PNG periportali come controllo negativo interno e nuclei di epatocite binucleari come riferimento positivo per la colorazione. Testare i parametri di analisi utilizzando un numero ridotto di immagini per garantire una buona segmentazione nucleare e una buona segregazione della soglia di intensità prima di applicare i parametri di analisi all'intero set di dati. - Selezionare i seguenti parametri nucleari da quantificare: (1) area nucleare in base alla colorazione di Hoechst (z2),(2) intensità media di Hoechst nucleare (RU), (3) fattore di allungamento nucleare (rapporto medio tra l'asse corto del nucleo e l'asse lungo del nucleo, dove un oggetto centrato simmetrico [non allungato] ha un valore di 1, (4) Nuc 1/(fattore di forma), indice medio di "rotondità" nucleare calcolato dal perimetro 2/(4x x area). I valori vanno da 1 all'infinito, dove 1 è un cerchio perfetto, (5) stato HNF4, ovvero lo stato (positivo 1 o negativo-0) e (6) le coordinate x/y nucleari in base al "centro di gravità" (cg), un metodo per individuare il centro dell'oggetto da immagini in scala di grigi con precisione sub-pixel.
- Eseguire l'analisi per tutti i set di dati di esempio ed esportare i dati numerici dal passaggio 5.5 al software per fogli di calcolo.
6. Analisi dei dati
NOTA: la fase di analisi dei dati può essere eseguita utilizzando qualsiasi software di foglio di calcolo standard.
- Calcolare i numeri di cella epatociti e non epatociti.
- Calcolare l'area totale della sezione epatica analizzata per ogni campione moltiplicando il numero di campi di visualizzazione per l'area del campo visivo (passaggio 4.2).

- Lavorando con i file di fogli di calcolo generati per ogni sezione epatica, filtra i dati selezionando solo i nuclei HNF4. Calcolare il numero totale di nuclei HNF4 e dividerlo per l'area totale analizzata per ottenere densità media di epatociti per ogni campione (Figura 2F).

- Eseguire lo stesso calcolo per le celle non parenchymal filtrando il foglio di calcolo per le celle HNF4 -(Figura 2E).
- Calcolare l'area totale della sezione epatica analizzata per ogni campione moltiplicando il numero di campi di visualizzazione per l'area del campo visivo (passaggio 4.2).
- Calcolare la distribuzione delle dimensioni nucleari degli epatociti.
- Utilizzando il software per fogli di calcolo, filtrare i dati per selezionare solo i nuclei HNF4.
- Tracciare i valori dell'area nucleare in un istogramma (Figura 2G). Impostare la larghezza del contenitore su 5 m2.
NOTA: i valori di frequenza possono essere corretti per l'area (nuclei/mm2) secondo il punto 6.1.1.
- Eseguire l'analisi ploidia nucleare epatocite.
NOTA: i dati del foglio di calcolo del passaggio 5.6 vengono utilizzati per generare un profilo di ploidia nucleare per ogni campione. Questo processo è stato automatizzato e può essere eseguito utilizzando un software scritto personalizzato che è liberamente disponibile per il download con informazioni di supporto e set di dati dimostrativihttps://github.com/lukeynoon(vedereFile supplementari). Il codice sorgente è fornito per gli utenti che desiderano adattare la metodologia. Di seguito sono riportate una descrizione dell'algoritmo, insieme alle istruzioni per l'installazione e l'utilizzo. Il programma utilizza i dati del foglio di calcolo per separare automaticamente i nuclei di epatociti in due gruppi; (1) quelli con nuclei circolari "semplici" e (2) nuclei "complessi" non circolari rappresentativi di cellule binucleari con "2c ploidia. Il contenuto minimo di DNA nucleare (funzione dell'area nucleare e della densità del DNA) viene calcolato successivamente per tutti i nuclei "semplici". Un passo successivo poi calibra automaticamente la ploidia nucleare di HNF4 o l'epatocito usando l'HNF4- nuclei come un noto controllo interno di 2'4N.- Scaricare e installare il software.
- Scaricare l'applicazione in pacchetto da: https://github.com/lukeynoon
- Avviare MATLAB. Passare alla scheda APP della barra degli strumenti, fare clic su Installa app e aprire l'applicazione scaricata definita "Ploidy_Application.mlappinstall". Verrà visualizzato un messaggio per confermare la corretta installazione.
NOTA: L'applicazione è ora pronta per l'uso e rimarrà nella scheda APP della barra degli strumenti.
- Formattare i dati di input.
NOTA: prima dell'analisi automatizzata della ploidia nucleare, tutti i file di fogli di calcolo contenenti dati di imaging ad alto contenuto (passaggio 5.6) devono essere memorizzati e formattati in base alle seguenti istruzioni.- In ogni file di dati esportato (. La cartella di lavoro XLS 97-2004) del passaggio 5.6, includere un foglio chiamato "Misure di cella" contenente tutti i dati necessari per l'analisi ploidia di ploidia di tipo nelle colonne (Figura 3A). Assicurarsi che il layout del foglio di calcolo, inclusi i nomi delle intestazioni di colonna, rimanga invariato rispetto a quello della figura 3A, poiché il metodo di analisi trova i dati di colonna corretti cercando questi nomi (vedere i set di dati dimostrativi in File supplementari per riferimento). Se, ad esempio, un software di analisi delle immagini ad alto contenuto non produce una colonna "Flusso di luce"(Figura 3A), inserire manualmente una colonna "Flusso di luce" nella stessa posizione, ovvero la colonna K e riempirla con zeri.
- Per ogni condizione sperimentale (ad esempio, "Injured-d14"), fornire un set di dati di controllo, che verrà utilizzato per calcolare il controllo interno per la calibrazione della ploidia nucleare 2.4N (passaggio 6.3.4.3). Qui, selezionare i campioni di fegato da cucciolate adulte non trattate ("Control-d0"; Figura 3B-D).
- Per le repliche biologiche (per condizione), memorizzare ogni foglio di calcolo nella propria cartella (come illustrato nella figura 3B). Assegnare un nome incrementale ai prefissi delle cartelle, ad esempio "Sample1, Sample2, Sample3... SampleN", in base ai nomi di file contenuti all'interno. Di conseguenza, ogni cartella del set di dati (ad esempio, "Control-d0") deve contenere una serie di sottocartelle ("Sample1", "Sample2" e così via) ognuna contenente un file di foglio di calcolo con lo stesso nome corrispondente.
- Eseguire l'applicazione.
- All'interno di MATLAB, avviare il "Ploidy_Application" facendo clic sull'icona all'interno della scheda MY APPS della barra degli strumenti (Figura 3C). Verrà visualizzata l'interfaccia utente grafica (GUI) Ploidy_Application(Figura 3C).
- Fare clic sul pulsante Percorso dati controllo per passare alla cartella in cui si trovano i dati del controllo (ad esempio, "Control-d0"). Questo percorso dati verrà quindi visualizzato nell'interfaccia (ad esempio, /Users/Desktop/Control-d0).
- Successivamente, in "prefisso cartella" digitare il nome da assegnare ai file di output (ad esempio, "Esempio").
NOTA: questo prefisso può essere modificato in qualsiasi testo, a condizione che le cartelle e i nomi dei file rimangano denominati in modo incrementale. - Fare clic sul pulsante Percorso di altri dati e passare alla cartella in cui si trovano i dati comparativi (ad esempio, "Injured-d14"). Questo percorso dati verrà quindi visualizzato nell'interfaccia (ad esempio, /Users/Desktop/Injured-d14).
- Fare clic su Esegui!. Al termine dell'analisi, nella barra di stato verrà eseguito il messaggio "Analisi completata!..".
NOTA: l'applicazione segnalerà, per ogni campione, la stratificazione dei nuclei "semplici" in "2n", 2n-4n, 4n-8n e 8n in termini di conteggi assoluti e come percentuale del totale (Figura 3D). Questi file verranno salvati automaticamente in ogni cartella di esempio come: "Count_2n.txt", "Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". Il Ploidy_Application salverà automaticamente un elenco per ogni campione, di tutte le singole stime ploidiche per nuclei "semplici" epatitociti e non epatociti in "Ploidy_All_Hepatocytes.txt" e "Ploidy_NonHepatocytes.txt". Per il set di dati del controllo, il metodo salva anche le soglie minime del contenuto del DNA calcolate per la stratificazione della ploidy (vedere il passaggio 6.3.4.3.7) in un file denominato "Normalised_Thresholds_Control". Infine, l'applicazione produrrà una cartella sia per il controllo che per i dati delle condizioni comparative selezionati con il nome "Riepilogo". Questa cartella contiene due sottocartelle, "Ploidy" e "Stratification" che contengono le medie di tutti i campioni forniti (Figura 3D).
- Descrizione della metodologia.
NOTA: nella sezione seguente viene descritta in dettaglio la metodologia utilizzata dal software Nuclear Ploidy Analysis. Se l'utente sceglie di non utilizzare l'applicazione, questi passaggi possono essere seguiti utilizzando il software foglio di calcolo per calcolare manualmente il profilo di ploidia nucleare.- Separare i nuclei in "semplici" o "complessi" in base alla morfometria nucleare.
- Calcolare un "indice di circolarità" per tutti i nuclei, definito come il "fattore di allungamento" nucleare diviso per il "Nuc 1/(fattore di forma)", dove un valore di 1,0 indica un cerchio perfetto.
NOTA: "Allungamento nucleare" e "Nuc 1/(fattore di forma)" sono due misure discrete della "circolarità" di un oggetto che valutano criteri morfometrici complementari e non sovrapposti. Il primo misura gli assi lunghi e corti di un oggetto, mentre il secondo confronta la lunghezza del perimetro di un oggetto con quella della sua area. Per rafforzare la definizione di circolarità nucleare utilizzata in questo protocollo, queste due misurazioni sono state combinate in un unico "indice di circolarità". Un approccio precedente per stimare la ploidia nucleare utilizzando la metodologia descritta utilizzava solo l'allungamento nucleare17. Sebbene siano stati ottenuti risultati accettabili utilizzando questo approccio, gli autori hanno osservato che un "indice di circolarità" composito migliora la discriminazione dei nuclei selezionati manualmente da epatociti mononucleari ed binucleari (dati non mostrati). - Classificare i nuclei con un indice di circolarità 0,8 come "complessi" e quelli > 0,8 come "semplici".
- Calcolare un "indice di circolarità" per tutti i nuclei, definito come il "fattore di allungamento" nucleare diviso per il "Nuc 1/(fattore di forma)", dove un valore di 1,0 indica un cerchio perfetto.
- Stimare il contenuto di DNA "minimo" (m) per tutti i nuclei "semplici".
- Calcolare il raggio nucleare (r) utilizzando la formula:

- Calcolare il volume nucleare (v) utilizzando il volume di una formula sfera:
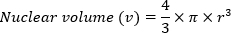
- Generare un valore relativo per il contenuto minimo di DNA (m) utilizzando la formula:

- Calcolare il raggio nucleare (r) utilizzando la formula:
- Calibrare il set di dati utilizzando i nuclei NPC (HNF4-) come controllo interno di 2-4N.
NOTA: i PNG hanno un contenuto di DNA di 2-4N a seconda dello stato del ciclo cellulare. Di conseguenza, il valore medio del contenuto di DNA "minimo" NPC (NPCm) aumenta con lesioni (Figura 4A). L'errore di calibrazione viene ridotto al minimo stabilendo un limite superiore di NPCm che rappresenta una soglia di 4c (Figura 4B).- All'interno del foglio di calcolo, selezionare solo i nuclei NPC con valori per "m" che si trovano all'interno di 1 deviazione standard (SD) della modalità (questo filtra il rumore da possibile errore di segmentazione).
- All'interno di questo intervallo filtrato, esaminare le aree nucleari e le corrispondenti intensità media di Hoechst (Figura 4C).
- Stimare l'area nucleare più piccola all'interno di questo intervallo filtrato con l'intensità massima nucleare di Hoechst (cioè il punto in cui la linea della curva cambia direzione nel set di dati filtrato come illustrato dal cerchio rosso nella Figura 4C). Questo valore rappresenta uno stato transitorio (t) al di sopra del quale il campionamento di nuclei 4c predomina su nuclei 2c, con conseguente massima di intensità media di Hoechst.
NOTA: questo valore viene determinato automaticamente dal software; tuttavia, gli utenti del foglio di calcolo possono selezionare manualmente questo punto come dimensione di transizione. - Calcolare il contenuto minimo del DNA rappresentato da questa dimensione transitoria (tm)seguendo il passaggio 6.3.4.2.
- Per stimare la spalla 4N del set di dati NPCm, aggiungere 1 SD al valore di tm. Il numero risultante (Figura 4B) descrive il limite superiore del contenuto minimo di DNA NPC da utilizzare per la stratificazione della ploidia nucleare (S4c).
- Ripetere i passaggi seguenti 6.3.4.3.1-6.3.4.3.5 per tutti i campioni di "controllo".
NOTA: Ad esempio, nella Figura 3,i fegati di controllo non danneggiati ("Control-d0") vengono utilizzati come condizione di controllo. - Calcolare una soglia media di stratificazione 4c (S4c) per i campioni di "controllo" e utilizzarla per estrapolare i limiti 2c (S2c)e 8c (S8c)per il contenuto minimo di DNA (m). Le soglie di stratificazione vengono generate e memorizzate automaticamente dal software (passaggio 6.3.3.3).
NOTA: a seconda del progetto dello studio, i valori medi della soglia di stratificazione possono essere calcolati per ogni condizione o per condizioni specifiche (ad esempio, fegato di controllo sano). Tuttavia, il software Nuclear Ploidy Analysis richiede che uno di un set di 2 file è designato come "controllo" ai fini del calcolo dei valori di ploidia relativa. - Calcolare un valore ploidio per tutti i nuclei utilizzando il valore S2c generato al punto 6.3.4.3.7 come da:

- Stratificare nuclei "semplici" epatociti (HNF4) in 2c/4c/8c/>8c staffe secondo i seguenti criteri: "2c" HNF4 ' "4c" HNF4 s.l.m. "8c" HNF4 s.l.m. ">8c" HNF4 s.l.m.
- Per ricostruire il patterning spaziale dei sottogruppi ploidi, separare i dati nucleari all'interno di ogni foglio di calcolo campione in base ai campi corrispondenti in cui sono stati acquisiti. Utilizzare quindi le coordinate x/y nucleari associate (dal passaggio 5.5) ai sottogruppi ploidi in 2D (Figura 5C).
- Separare i nuclei in "semplici" o "complessi" in base alla morfometria nucleare.
- Scaricare e installare il software.
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo metodo è stato utilizzato per misurare l'impatto della lesione colestatica sul fegato adulto del topo alimentando gli animali per 0-21 giorni con una dieta epatossica contenente lo 0,1% 3,5-diethoxycarbonyl-1,4-dicollididene (DDC)17. L'alimentazione cronica della DDC provoca lesioni epatocellulari aumento dell'espansione ploidia e periportalia dei PNG. L'utente deve essere consapevole che la tensione del topo e le differenze dipendenti dall'età possono esi...
Access restricted. Please log in or start a trial to view this content.
Discussione
Viene descritto un approccio ad alto contenuto e ad alto contenuto per l'analisi del rimodellamento dei tessuti e la stima della ploidia nucleare epatocite nel fegato murino. Una volta che ha familiarità con la procedura, un utente può elaborare, immagini e analizzare più campioni in un periodo di 3-5 giorni, generando set di dati testati di grandi dimensioni che forniscono una firma dettagliata dello stato di salute del fegato. Data la semplicità del metodo di preparazione del campione, insieme al gran numero di cel...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato dalla sovvenzione spagnola del governo MINECO BFU2014-58686-P (LAN) e SAF-2017-84708-R (DJB). LAN è stata sostenuta da un premio nazionale DI MineCO Ramàn y Cajal Fellowship RYC-2012-11700 e Plan GenT (Comunitat Valenciana, CDEI-05/20-C) e FMN da una studentessa regionale della Generalitat Valencian ACIF/2016/020. RP vorrebbe riconoscere il professor Ewa K. Paluch per il finanziamento. Ringraziamo la Dott.ssa Alicia Martanez-Romero (servizio CIPF Cytometry) per l'aiuto con la piattaforma IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Riferimenti
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon