このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
マウスにおける肝細胞核プロイドの推定に対するSitu法におけるハイスループット
要約
フローサイトメトリーを必要としない固定/凍結保存組織サンプル内で、肝細胞数および核プロイドの変化を測定するための堅牢で費用対効果の高い柔軟な方法を提示します。私たちのアプローチは、肝臓の損傷や病気の進行を追跡するのに理想的な肝臓細胞学の強力なサンプル全体の署名を提供します。
要約
肝臓が損傷すると、肝細胞数が減少し、細胞サイズ、核サイズ、策略が増加する。また、コリンギオサイト、筋線維芽細胞、前駆細胞、炎症細胞などの非実質細胞の拡張は、慢性肝障害、組織改変および疾患進行を示す。このプロトコルでは、傷害、慢性疾患および癌に関連する肝臓の細胞組成の変化を計算するための単純なハイスループットアプローチを説明する。我々は、2次元(2D)組織切片から抽出された情報を使用して、サンプル内の肝細胞核策略を定量化および較正し、ユーザーが肝臓内の特定のプロイドサブセットをその場で見つけることができる方法を示す。我々の方法は、固定/凍結された肝臓材料、基本的な免疫細胞化学試薬および任意の標準的な高含有画像化プラットフォームへのアクセスを必要とする。これは、新たに収集された組織の破壊、空間情報の損失および潜在的な崩壊バイアスを必要とする標準的なフローサイトメトリー技術に代わる強力な代替手段として機能します。
概要
哺乳類の肝臓の肝細胞は、双核細胞を産生するために失速したサイトカネシスを受け、DNA内在性を有して最大16NのDNA含量を含む多倍核を産生する。出生後の発達、老化、および多様な細胞ストレスへの応答における全体的な細胞および核の策略の増加1.多重化のプロセスは動的かつ可逆的である2、その正確な生物学的機能は不明のままであるが 3.増加したプロイディは、増殖能力の低下に関連付けられている 4 、 遺伝的多様性2、 慢性傷害5および癌保護への適応6.肝細胞の策略変化は、概日リズム7の変化の結果として起こり、8を引き上げ、最も顕著なのは、肝臓のプロイドプロファイルが傷害および疾患9によって変化し、そして説得力のある証拠は、≥8N核の増加または2N肝細胞の喪失などの特定の策略変化が、非アルコール性脂肪性肝疾患(NAFLD)進行3、10、10またはウイルス感染の3差有差影響を追跡するための有用な署名を提供することを示唆している。
一般に、肝損傷および再生は、肝細胞サイズの増加および核領域12に関連しており、肝細胞の全体数が減少すると共に、特に2NDNA含量10、11,11を有するものである。肝臓における実質損傷はまた、多くの場合、間質筋線維芽細胞、炎症性細胞およびバイポテンシー肝前駆細胞を含む非実質細胞(NPC)の拡張を伴う。パンキマル細胞数と核策略の定量的細胞学的プロファイルを提供する高スループット法は、NPCの変化を考慮しながら、傷害および疾患時の肝臓の応答を追跡する研究および臨床ツールとしてかなりの可能性を秘めています。肝細胞癌のヒトサンプルにおけるプロイドスペクトルの最新の説得力のある分析は、核プロイドが腫瘍内で劇的に増加し、TP5313の分化および損失を伴うより積極的な腫瘍サブタイプにおいて特異的に増幅されることを示している。したがって、核策略の定量的評価における方法論的進歩が、肝臓癌の将来の予後プロファイリングに役立つ可能性が高い。
本プロトコルでは、マウス肝組織切片の比較分析のための柔軟なハイスループット方法論が記載されており、これは肝細胞数の詳細な細胞量プロファイリング、NPC応答および核プロイドを推定するための内部的に較正された方法を提供する(図1)。肝細胞は、核サイズおよび核施設の特性評価に先立って、肝細胞核因子4α(HNF4α)免疫標識によってNPCと区別される。平均Hoechst 33342強度(DNA密度のプロキシ)と補間された3次元(3D)核体積を統合することによって、すべての円形核マスクに対して「最小DNA含有量」が推定される。その後、最小DNA含有量を測定し、NPCを使用して評価し、核プロイドプロファイルを生成します。
画像取得、核セグメンテーション、画像解析は高内容イメージングを用いて行われ、数万個の細胞を含む2次元(2D)肝臓切片の広い領域をスクリーニングすることが可能です。カスタム作成プログラムは、すべての円形肝細胞核のサンプル全体のプロイドプロファイルを生成するために、高コンテンツ画像解析データの自動後処理のために提供されます。これは、自由にソフトウェアをダウンロードして、立体画像解析(SIA)10、11、14、15に基づいて核策略10,11,14を15計算するために実行されます。SIAの方法論は、循環核形態と核サイズとDNA含有量の単調な関係を仮定して、肝臓14における肝細胞核策略を推定するための正確で、骨の折れる方法としてフローサイトメトリーによって以前に検証されてきた。この議定書では、両方の核パラメータは、核施設とHoechst 33342ラベリングの評価によって測定される。各核マスクの「最小DNA含有量」の計算に続いて、既知の2-4N DNA含有量を有するNPCを用いた肝細胞核プロイドの較正が行われ、有用な内部制御として機能する。
従来のフローサイトメトリー法16と比較して、説明したアプローチは、肝細胞核プロイドをその際に評価することを可能にし、結果を偏らさせ得る新鮮な組織または分解方法へのアクセスを必要とせず、標準化することが困難である。すべての SIA ベースのアプローチと同様に、核の小数部 >2N は、赤道平面の外側の大きな核の断面化により、2D サンプリングによって過小評価されます。組織全体の策略プロファイルはまた、すべての円形肝細胞核マスクの最小DNA含有量を記述し、同じ策略の2つの離散(「非接触」)核を有する単核肝細胞と双核細胞を直接区別しない。しかし、このプロトコルの単純さは、細胞の策略のより詳細な評価を提供する双核細胞の同定を容易にする核間間隔または細胞周囲分析などの追加パラメータを考慮するように適応するためのかなりの範囲を可能にする。
Access restricted. Please log in or start a trial to view this content.
プロトコル
すべての動物実験は、以前CIPF倫理委員会によって承認されました。マウスはセントロ・デ・インベスティガシオン・プリンシペ・フェリペ(スペイン・バレンシア)の病原体のない施設に収容され、実験動物のブリーダー、ユーザー、供給センターとして登録されました(reg.ES 46 250 0001 002)現在適用されるヨーロッパおよびスペインの動物福祉規則(RD 53/2013)。
1. 組織の収穫とサンプル調製
注:このプロトコルは、事前に固定または凍結保存せずに組織を凍結する方法を記述します。以前に固定/凍結保存されたサンプルについては、セクション 2 に進み、ステップ 3.1 を省略します。すべての分析は、12-16週齢の成体雌C57BL/6マウスを使用して行われました。
- フェンタニル/ペントバルビタール腹腔内注射による動物の犠牲、続いて子宮頸部脱臼。腹側にマウスを向けた状態で、腹腔を開き、ピンセットで皮膚をつかみ、下腹部の基部から胸骨の基部に外科的なはさみを使用して垂直切開を行うことによって肝臓を露出させる。
- 慎重に細かいピンセットを使用して胆嚢を除去し、肝臓を解剖し、リン酸緩衝生理食塩水(PBS)で満たされた10センチペトリ皿皿プレートで選択した肝臓の小葉を洗いすります。
注:この場合、中央値のローブが使用された場合、各動物について同じ肝臓の葉を比較することをお勧めします。 - ラベル付きのクリオマールを室温(RT)で最適な切削温度(OCT)媒体で満たします。OCTバブルを避けてください。表示された場合は、針またはピペットチップを使用して金型の端に押し込みます。
- 充填されたOCTクリオブオールに肝臓の葉を埋め込み、すぐにドライアイスの上に置いて急速な凍結を確実にします。クライオノルドは-80°Cで凍結切断するまで保管してください。
2. クライオセクション
- 乾燥した氷の上に凍結を輸送し、組織の劣化を避けます。クライオスタット内部を20分間-20°Cに平衡化する前に。
- プラスチックのクリオマールの基部に圧力をかけることによってサンプルを取り出す。液体OCTをRTの暖かいサンプルディスクに適用し、クライオスタットの位置に置き、OCT埋め込まれた肝臓サンプルを取り付けます。穏やかな圧力を加え、サンプルがディスクにくっつくように、OCTがフリーズするまで3分待ちます。
注:組織の劣化を回避するために、できるだけ指でサンプルを扱うことは避けてください。 - サンプルをクライオスタットのアームにロックし、サンプルの端がクライオスタットブレードと平行になるように向きを調整します。組織が到達するまでサンプルにカットします。
- サンプルを6μmの厚さに切り離します。ラベル付きポリアミドコーティングスライドをサンプルの上に5 s置き、サンプルをスライドに貼り付けます。RTに3~5分間スライドを置き、最良の結果を得るには、セクション3に直接進みます。
注:複数の新鮮な凍結サンプルの処理のために、すべてのサンプルが処理されるまでドライアイス上のスライドボックスにスライドを一時的に保存することによって再現可能な結果が得られました。この方法を使用すると、セクション 3 に進む前にすべてのスライドを RT に平衡化できます。ホルマリン固定パラフィン埋め込み(FFPE)サンプルを使用できますが、この方法によりバックグラウンド自己蛍光が増加します。FFPEサンプルから進むには、ポリアミド処理スライド上の40°C水浴からセクションをキャッチして4μmの取り付けでセクションを取り付けます。60°Cで1時間加熱し、キシレン(x2)、エタノール100%(x2)、96%(x2)、70%(x1)およびdH2O(x1)を含むコプリン瓶に連続RTの化傷(5分2)で脱パラフィン化する。抗原を露出させるために、RTでPBSでスライドを焼く前に90°Cで20分間クエン酸緩衝液にスライドを置く。
3. 蛍光免疫標識
- RTで10分間PBSに4%パラホルムアルデヒド(PFA)の1mLを塗布することにより、ヒュームフードの組織切片を固定します。
注:今から免疫染色プロセスの終わりまで、サンプルの乾燥を避けてください。 - 各組織セクションの周りの領域を乾燥させ、疎水性ペンを使用して囲みます。0.5%の非イオン性界面活性剤(すなわち、トリトンX-100)をPBSで15分間RTで透過させる。その後、PBSで満たされたコプリン瓶を3分間、穏やかな攪拌(2倍繰り返し)で洗います。
- ブロックは、1%ウシ血清アルブミン(BSA)、5%馬血清、0.2%のノニオン界面活性剤(RTで少なくとも1時間)の濾過溶液を使用した。
- 一次HNF4α抗体を用いて、暗湿染色チャンバーで4°Cで夜間にブロッキングバッファーに希釈したインキュベート(抗体および特異的希釈液については、材料表を参照)。
- スライドをPBSで満たされたコプリン瓶に入れ、穏やかな攪拌を使用して3分間洗浄します(4倍繰り返します)。
- Alexa-488コンジュゲート二次抗体と共役した2次抗体と、フィルターした1%BSAで希釈したHoechstと、暗い湿気の多い染色チャンバーでRTで2時間のPBSで2時間ノンイオン性界面活性剤を希釈したインキュベート(抗体および特異的希釈液の材料表を参照)。
- スライドをPBSで満たされたコプリン瓶に入れ、穏やかな攪拌を使用して3分間洗浄します(4倍繰り返します)。穏やかな攪拌を使用して3分間ddH2Oで洗浄します(2倍繰り返します)。
- カバースリップ(24 x 60 mm)に2滴の蛍光実装メディアを配置し、その上にスライドを敷設することで、穏やかな圧力を加えて泡を除去することで、スライドを取り付けます。長期保管の場合は、透明なマニキュア付きの端にカバースリップをシールし、4°Cで暗闇の中に保管してください。
- 先に進む前に、従来の蛍光顕微鏡を使用してスライドをチェックし、良好な固定と免疫標識を確保してください。
注: 期待される結果については、図 2A,Bを参照してください。
4. 蛍光画像取得
注: このステップでは、蛍光画像の自動取得をサポートする高コンテンツイメージングプラットフォーム(材料表)が必要です。
- イメージングシステムをオンにして、新しい取得プロトコルを開きます。
- 10x の目的を選択し、視野の面積をメモします(この場合は 0.6 mm2)。
- 適切な励起フィルターと発光フィルタを使用して蛍光画像を取得するためのパラメータを設定します(ステップ3.6)。Hoechst と Alexa-488 の場合は、390/18 および 438/24 nm 励起と 432.5/48 および 475/24 nm 放射をそれぞれ持つ「DAPI」および「GFP」チャネルを選択します。
- サンプルに焦点を当て、信号強度が非飽和であることを確認します。画像の撮影は、すべての画像に対して同じ露光時間で行うか、蛍光の強度が露光時間に補正されるシステムを使用してください。
- サンプルをスキャンし、組織セクションの完全なカバレッジを得るために十分な画像を取得します(サンプルサイズに応じて約20〜50の視野)。
- 画像データベースを見直し、(i)不十分な焦点を当てなかったフィールド、(ii)各組織セクションの境界にあるもの(偏った細胞密度計算を避けるために)、(iii)組織セクションの折り畳まれた/物理的に損傷した領域を含むものを手動で排除する。
5. 自動蛍光画像解析
注:このステップには、適切な画像分析ソフトウェア(材料表)が必要です:(1)405nmの画像内のHoechstラベル付き核を自動的に識別する(核セグメンテーション)、(2)平均ヘーヒトスト核強度とモーホメトリーを評価し、(3)488nm(HNα4)で核蛍光の+/-状態を決定するための閾値分析。プログラム内のセグメンテーションおよび閾値パラメータを視覚的に評価および調整し、核とHNF4α+/-ステータスが最適にゲートされていることを確認するには、基本的なオペレータトレーニング/専門知識が必要です(図2)。
- 画像解析ソフトウェアでは、ステップ 4.5 から Hoechst (405 nm) および HNF4α (488 nm) 画像を含む取得ファイルを開き、新しい解析プロトコルを作成します。
- 核セグメンテーション(Hoechst、405 nm)および肝細胞/NPC閾値解析(HNF4α、488nm)に使用する波長を定義します。
- 核が最適に分離されるように、ソフトウェアの核セグメンテーションパラメータ(「最小核面積」や核検出「感度」など)を調整します。
注: 肝細胞の良いセグメンテーションは、NPCのそれより優先されるべきです。2NPC核は、中弦波内皮のような、平坦化/楕円形または不規則な形状であり、一般的に肝細胞の核細胞のものよりも小さく、より密接に詰まっている(四分位間サイズ範囲:30-43 μm2)。マウス肝臓では、最小核面積≥23 μm2、検出「感度」65%が使用されました(期待される結果については図2C,Dを参照)。感度は、ピクセルクラスタが個々の核として認識される方法をその強度に基づいて決定し、自動画像解析を進める前に、ユーザーが設定した各サンプルについて経験的にテストする必要があります。 - 488 nmの閾値強度を変更して、肝細胞(HNF4α+)および非パレンキマル細胞(HNF4α-)の最適なゲーティングを確保します。
注 : 期待される結果については、図 2C,Dを参照してください。しきい値の強度の値は相対的であり、レーザー強度などの染色効率と取得設定に依存します。したがって、ユーザーによって標準化される必要があります。内皮細胞やペリポータルNPCなどの既知のHNF4-細胞を、内部陰性対照および二核肝細胞核として、染色のための陽性基準として用いる。データセット全体に解析パラメータを適用する前に、少数の画像を使用して解析パラメータをテストし、核セグメンテーションと強度しきい値の分離を確実に行います。 - 定量化する核パラメータを選択:(1)Hoechst染色(μm2)に基づく核面積(μm2)、(2)は、核の角流強度(RU)を意味し、(3)核伸長係数(核の長軸に対する核の短軸の平均比、 中心対称の[細長い]オブジェクトの値が1、(4)Nuc 1/(フォームファクター)は、周囲2/(4π x面積)で計算された核の「丸み」指数を意味します。値は1から無限大、(5)HNF4αステータス(正1または負の0)、(6)「重心」(cg)に基づく核x/y座標は、サブピクセル精度のグレースケール画像からオブジェクトの中心を見つける方法です。
- すべてのサンプル データセットの解析を実行し、ステップ 5.5 の数値データをスプレッドシート ソフトウェアにエクスポートします。
6. データ分析
注: データ分析のステップは、任意の標準的なスプレッドシート ソフトウェアを使用して実行できます。
- 肝細胞細胞および非肝細胞細胞数を計算する。
- 各サンプルについて分析した肝臓セクションの総面積を計算するには、視野の数と視野の面積を掛けます (ステップ 4.2)。

- 各肝セクションに対して生成されたスプレッドシートファイルを操作し、HNF4α+核のみを選択してデータをフィルタリングします。分析したHNF4α+核の総数を計算し、これを分析した全面積で割って各サンプルの平均肝細胞密度を得る(図2F)。

- HNF4α-セルのスプレッドシートをフィルタリングすることで、非数元細胞に対して同じ計算を行う(図2E)。
- 各サンプルについて分析した肝臓セクションの総面積を計算するには、視野の数と視野の面積を掛けます (ステップ 4.2)。
- 肝細胞の核サイズ分布を計算します。
- スプレッドシートソフトウェアを使用して、データをフィルタしてHNF4α+核のみを選択します。
- ヒストグラムにおける核面積のプロット値 (図 2G)。ビン幅を 5 μm2に設定します。
注: 周波数値は、ステップ 6.1.1 に従って面積(nuclei/mm2)を補正できます。
- 肝細胞核策略解析を行う。
注: ステップ 5.6 のスプレッドシート データは、各サンプルの核策略プロファイルを生成するために使用されます。このプロセスは自動化されており、サポート情報とデモデータセットと共に自由にダウンロードできるカスタムの書かれたソフトウェアを使用して実行できます。https://github.com/lukeynoon(を参照してください。補助ファイル).ソースコードは、方法論を適応させたいユーザーのために提供されます。アルゴリズムの説明とインストールと使用の手順を以下に概説します。プログラムは、自動的に2つのグループに肝細胞核を分離するためにスプレッドシートデータを使用しています。(1)「単純な」円形核と(2)2cのプロイドを有する双核細胞を代表する「複雑な」非円形核を有するもの。最小限の核DNA含有量(核領域とDNA密度の機能)は、次にすべての「単純な」核について計算されます。その後のステップでは、HNF4α+肝細胞核プロイドを知られた2−4N内部制御としてHNF4α-核を用いて自動的に校正する。- ソフトウェアをダウンロードしてインストールします。
- パッケージ化されたアプリケーションをダウンロードする方法: https://github.com/lukeynoon
- MATLAB を起動します。ツールトリップの [APP] タブに移動し、[アプリのインストール] をクリックして、ダウンロードしたアプリケーションを "Ploidy_Application.mlappinstall" と呼んで開きます。インストールが成功したことを確認するメッセージが表示されます。
注:アプリケーションは現在使用する準備ができて、ツールトリップのAPPタブに残ります。
- 入力データをフォーマットします。
注: 核計画の自動解析を行う前に、高コンテンツイメージング データを含むすべてのスプレッドシート ファイルを保存し、次の手順に従ってフォーマットする必要があります。- エクスポートされた各データ ファイル (.XLS 97-2004ワークブック)ステップ5.6から、列に示された策略分析に必要なすべてのデータを含む「セル測定」と呼ぶシートが含まれる(図3A)。分析方法では、これらの名前を検索して正しい列データを検出するため、列ヘッダー名を含むスプレッドシートのレイアウトが図 3Aのレイアウトと変わらないようにしてください (参照については、「補足ファイル」のデモ データセットを参照)。たとえば、高コンテンツ画像解析ソフトウェアが「光束」カラムを生成しない場合(図3A)、同じ位置に「光束」列を手動で挿入し、列Kをゼロで埋めます。
- 実験条件(例えば、「傷-d14」)ごとに、2-4N核策略校正の内部制御を計算するために使用される制御データセットを提供する(ステップ6.3.4.3)。ここでは、未処理の成人のゴミ捨て人(「Control-d0」)から肝臓サンプルを選択します。図 3B-D)。
- 生物学的複製 (条件ごと) の場合は、各スプレッドシートを専用のフォルダに保存します (図 3B)。フォルダーのプレフィックスにインクリメンタルで名前を付けます。例: "Sample1、Sample2、Sample3..SampleN" (含まれているファイル名に従って)。したがって、すべてのデータセットフォルダ(例えば、「Control-d0」)には、同じ名前のスプレッドシートファイルを含む一連のサブフォルダ(「Sample1」、「Sample2」など)を含める必要があります。
- アプリケーションを実行します。
- MATLAB 内で、ツールトリップの [マイ アプリ] タブ内のアイコンをクリックして 「Ploidy_Application」を起動します (図 3C)。グラフィカル・ユーザー・インターフェース (GUI) Ploidy_Applicationが表示されます (図 3C)。
- [コントロール データへのパス] ボタンをクリックして、コントロール データがレプリケートされるフォルダーに移動します (たとえば、"Control-d0")。このデータ パスはインターフェイスに表示されます (たとえば、/ユーザー/デスクトップ/コントロール d0)。
- 次に、「フォルダプレフィックス」に、出力ファイルに付ける名前(例えば「Sample」)を入力します。
注: このプレフィックスは、フォルダとファイル名が増分名前のままである場合、任意のテキストに変更できます。 - [他のデータへのパス] ボタンをクリックし、比較データがレプリケートされるフォルダーに移動します (たとえば、"負傷-d14")。このデータ パスはインターフェイスに表示されます (たとえば、/ユーザー/デスクトップ/負傷した d14)。
- [実行] をクリックします。分析が完了すると、ステータス バーに 「分析完了!..」と表示されます。
注:アプリケーションは、各サンプルについて、絶対数の観点から、および合計のパーセンテージとして、≤2n、2n-4n、4n-8nおよび8n+に「単純な」核の階層化を報告します(図3D)。これらのファイルは、各サンプル フォルダに自動的に保存されます:"Count_2n.txt"" "" """" "" "Count_2n_to_4n" "" "Count_4n_to_8n.txt"" "Count_8n_and_higher.txt" " "Percentage_2n.txt" " "Percentage_2nto4n.txt", "Percentage_4nto8n.txt" "Percentage_2n.txt" Percentage_8n_and_higher.txt"として保存されます。Ploidy_Applicationは、各サンプルのリストを自動的に保存し、"Ploidy_All_Hepatocytes.txt"および"Ploidy_NonHepatocytes.txt"の "単純な" 肝細胞および非肝細胞核に対するすべての個々の策略推定値を保存します。制御データセットの場合、このメソッドは、ploidy の階層化のために計算された最小の DNA コンテンツ閾値 (ステップ 6.3.4.3.7 を参照) を"Normalised_Thresholds_Control"という名前のファイルに保存します。最後に、アプリケーションは、コントロールと「概要」と呼ばれる選択した比較条件データの両方のフォルダを生成します。このフォルダには、提供されたすべてのサンプルの平均を含む"Ploidy"と"階層化"という 2 つのサブフォルダが含まれています (図 3D)。
- 方法論の説明。
注: 以下のセクションでは、核計画解析ソフトウェアで使用される方法について詳しく説明します。ユーザーがアプリケーションを使用しないことを選択した場合、これらの手順は、手動で核策略プロファイルを計算するスプレッドシートソフトウェアを使用して従うことができます。- 核穿動法に従って、核を「単純」または「複雑」に分離する。
- 核の「伸び率」を「Nuc 1/(フォームファクター)」で割った「円形度指数」を計算し、値が1.0の場合は完全な円を示します。
注:「核伸び」と「Nuc 1/(フォームファクター)」は、相補的で非重なり合う形態学的基準を評価するオブジェクトの「円形性」の2つの個別の尺度です。前者はオブジェクトの長軸と短軸を測定し、後者はオブジェクトの周長をその領域の長さと比較します。このプロトコルで使用される核循環の定義を強化するために、これら2つの測定は単一の「循環性指数」に結合された。記述された方法論を用いて核の策略を推定する以前のアプローチは、核伸長のみを用いた17.このアプローチを用いて許容可能な結果が得られたが、複合的な「循環指数」は、単核および双核肝細胞から手動で選択された核の識別を改善することを観察した(データは示していない)。 - 円形度指数 ≤ 0.8 の核を「複雑」、その核を「単純」と分類する。.
- 核の「伸び率」を「Nuc 1/(フォームファクター)」で割った「円形度指数」を計算し、値が1.0の場合は完全な円を示します。
- すべての「単純な」核について「最小」DNA含有量(m)を推定する。
- 次の式を使用して、核半径 (r) を計算します。

- 球体式の体積を使用して、核体積(v)を計算します。
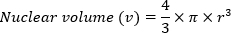
- 次の数式を使用して、最小 DNA コンテンツ (m) の相対値を生成します。

- 次の式を使用して、核半径 (r) を計算します。
- 内部 2-4N コントロールとして NPC (HNF4α-) 核を使用して、データセットを調整します。
注:NPCは細胞周期の状態に応じて2-4N DNA含有量を有します。したがって、NPCの「最小」DNA含有量(NPCm)の平均値は、傷害とともに増加する(図4A)。校正誤差は、4c閾値を表すNPCmの上限を設定することによって最小化される(図4B)。- スプレッドシート内で、モードの標準偏差(SD)内にある「m」の値を持つNPC核のみを選択します(これは、可能なセグメンテーションエラーからのノイズを除外します)。
- このフィルタリングされた範囲内で、核領域とそれに対応する平均のHoechst強度を調べる(図4C)。
- このフィルタリングされた範囲内の最小の核面積を最大核のHoechst強度で推定する(すなわち、図4Cの赤い円で示されるように、曲線の線がフィルタリングされたデータセットの方向を変化する点)。この値は、4c核のサンプリングが2c核を上回る2N-4N遷移状態(t)を表し、その結果、平均Hoechst強度の最大値になります。
注: この値は、ソフトウェアによって自動的に決定されます。ただし、スプレッドシートユーザーは、このポイントを移行サイズとして手動で選択できます。 - この過渡的なサイズ(tm)で表される最小のDNA含有量を、ステップ6.3.4.2に従って計算します。
- NPCmデータセットの 4N 肩を推定するには、1 SD を tmの値に追加します。結果の数(図4B)は、核策略層に用いられる最小DNA含有量のNPCの上限を記載する(S4c)。
- すべての「制御」サンプルについて、手順 6.3.4.3.1~6.3.4.3.5 を繰り返します。
注: たとえば、図 3では、コントロールの状態として、損傷していないコントロール肝臓 (「Control-d0」) が使用されています。 - 「制御」サンプルの平均4c層化閾値(S4c)を計算し、これを使用して最小DNA含有量(m)のために2c(S2c)および8c(S8c)境界を推定する。2c8c階層化のしきい値は、ソフトウェアによって自動的に生成され、保存されます(ステップ 6.3.3.3)。
注:研究の設計に応じて、平均階層化閾値は、各条件または特定の条件(例えば、健康なコントロール肝臓)について計算することができます。しかし、核プロイド解析ソフトウェアは、2つのファイルのセットのうちの1つが、相対的なプロイド値を計算する目的で「制御」として指定されることを要求する。 - ステップ 6.3.4.3.7 で生成された S2c値を使用して、すべての核のプロイド値を計算します。

- 次の基準に従って、2c /4c/8c/>8cブラケットに「単純な」肝細胞(HNF4α+)核を層状化する: "2c" HNF4α+ = p ≤ 2;"4c" HNF4α+ = 2 < p ≤ 4;"8c" HNF4α+ = 4 < p ≤ 8;">8c" HNF4α+ = 8 < p.
- 策略サブグループの空間パターン化を再構築するには、各サンプルスプレッドシート内の核データを、取得した対応するフィールドに従って分離します。次に、関連する核X/y座標(ステップ5.5から)を使用して、2Dでプロイドサブグループをプロットします(図5C)。
- 核穿動法に従って、核を「単純」または「複雑」に分離する。
- ソフトウェアをダウンロードしてインストールします。
Access restricted. Please log in or start a trial to view this content.
結果
この方法は、0.1%3,5-ジエトキシカルボニル-1,4-ジヒドロコリジン(DDC)17を含む肝毒性食を有する動物に0−21日間供給することにより、成人マウス肝臓に対する胆閉障害の17影響を測定するために使用されてきた。慢性DDCの摂食は、肝細胞損傷の結果、NPCのプロイディおよびポータル間の拡大を増加させた。ユーザーは、マウス株と年齢依存性の違いが核...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
マウス肝臓における肝細胞核プロイドの組織改修および推定の解析のための高含有、ハイスループットアプローチが記載されている。この手順に慣れると、ユーザーは3〜5日間で複数のサンプルを処理、画像化、分析し、肝臓の健康の詳細な署名を提供する大規模なテスト可能なデータセットを生成できます。サンプル調製方法のシンプルさを考えると、多数の細胞と組織領域が分析され(平?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは開示するものは何もない。
謝辞
この作業は、スペインのMINECO政府がBFU2014-58686-P(LAN)とSAF-2017-84708-R(DJB)を付与することによって資金提供されました。LANは、全国のミネコ・ラモン・イ・カハル・フェローシップRYC-2012-11700とプランGenT賞(コンニタット・バレンシアナ、CDEI-05/20-C)、およびバレンシア・ジェネラリタットACIF/2016/020の地域ValI +D学生シップによってFMNによって支えられました。RPは、エワ・K・パルチ教授の資金調達を認めたいと考えています。INセルアナライザプラットフォームの支援を受けたアリシア・マルティネス・ロメロ博士(CIPFサイトメトリー・サービス)に感謝します。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
参考文献
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved