É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um método de alta produtividade in situ para estimação da ploidiii y nuclear hepatocida em camundongos
Neste Artigo
Resumo
Apresentamos um método robusto, econômico e flexível para medir mudanças no número de hepatócitos e ploidy nuclear em amostras de tecido fixo/criopreservado que não requerem citometria de fluxo. Nossa abordagem fornece uma poderosa assinatura amostral de citologia hepática ideal para rastrear a progressão de lesões hepáticas e doenças.
Resumo
Quando o fígado está ferido, os números de hepatócitos diminuem, enquanto o tamanho da célula, o tamanho nuclear e a ploidy aumentam. A expansão de células não parenquiais como colangiocytes, miofibroblastos, progenitores e células inflamatórias também indicam danos crônicos no fígado, remodelação tecidual e progressão da doença. Neste protocolo, descrevemos uma abordagem simples de alto throughput para calcular mudanças na composição celular do fígado que estão associadas a lesões, doenças crônicas e câncer. Mostramos como as informações extraídas de seções de tecido bidimensional (2D) podem ser usadas para quantificar e calibrar ploidy nuclear hepatocito dentro de uma amostra e permitir que o usuário localize subconjuntos específicos de ploidy dentro do fígado in situ. Nosso método requer acesso a material hepático fixo/congelado, reagentes básicos de imunocitoquímica e qualquer plataforma de imagem padrão de alto conteúdo. Ele serve como uma poderosa alternativa às técnicas padrão de citometria de fluxo, que requerem interrupção do tecido recém-coletado, perda de informações espaciais e viés de desagregação potencial.
Introdução
Hepatócitos no fígado de mamíferos podem sofrer citocinese paralisada para produzir células binucleares, e endoreplicação de DNA para produzir núcleos poliplóides contendo até 16N conteúdo de DNA. Aumento geral da ploididy celular e nuclear durante o desenvolvimento pós-natal, envelhecimento e em resposta a diversos estresses celulares1. O processo de poliploidização é dinâmico e reversível2,embora sua função biológica precisa ainda não esteja clara3. O aumento da ploidia está associado à redução da capacidade proliferativa4, diversidade genética2, adaptação à lesão crônica5 e proteção contra o câncer6. Alterações de ploidy hepatocito ocorrem como resultado de um ritmo circadiano alterado7, e desmame8. Mais notavelmente, o perfil ploidiano do fígado é alterado por lesão e doença9, e evidências convincentes sugerem que alterações específicas de ploidia, como o aumento de núcleos ≥8N ou perda de hepatócitos 2N, fornecem assinaturas úteis para o rastreamento da progressão da doença hepática gordurosa não alcoólica (NAFLD)3,10, ou o impacto diferencial das infecções virais11.
Em termos gerais, a lesão hepática e a regeneração estão associadas ao aumento do tamanho das células hepatocitos e da área nuclear12,juntamente com o número global reduzido de hepatócitos, particularmente aqueles com teor de DNA 2N10,11. A lesão parenquimal no fígado também é frequentemente acompanhada pela expansão de células não parenquiais (NPCs), incluindo miofibroblastos estrômicos, células inflamatórias e células progenitoras hepáticas bipotentes. Métodos de alto throughput que fornecem um perfil citológico quantitativo de número de células parênquias e ploidia nuclear, ao mesmo tempo em que contabilizam mudanças nos NPCs, portanto, têm um potencial considerável como pesquisas e ferramentas clínicas para acompanhar a resposta do fígado durante lesões e doenças. Análises recentes convincentes in situ de espectroplóide em amostras humanas de carcinoma hepatocelular também demonstram que a ploidia nuclear é dramaticamente aumentada dentro dos tumores e é especificamente amplificada em subtipos tumorais mais agressivos com diferenciação reduzida e perda de TP5313. Assim, há uma forte possibilidade de que os avanços metodológicos na avaliação quantitativa da ploididia nuclear ajudem no futuro perfil prognóstico do câncer de fígado.
Neste protocolo, descreve-se uma metodologia flexível de alto desempenho para a análise comparativa das seções de tecido hepático do camundongo, que fornece perfis citométricos detalhados de números de hepatócitos, a resposta do NPC e um método previamente calibrado internamente para estimar ploidy nuclear (Figura 1). Os hepatócitos são distinguidos dos NPCs pela imunorotulagem do fator nuclear 4 alfa (HNF4α), antes da caracterização do tamanho nuclear e da morfometria nuclear. "Conteúdo mínimo de DNA" é estimado para todas as máscaras nucleares circulares, integrando a intensidade média de Hoechst 33342 (um proxy para a densidade de DNA) com volume nuclear tridimensional interpolado (3D). O conteúdo mínimo de DNA do hepatócito é então calibrado usando NPCs para gerar um perfil de ploidia nuclear.
Aquisição de imagens, segmentação nuclear e análise de imagens são realizadas por meio de imagens de alto conteúdo, permitindo que grandes áreas de seções hepáticas bidimensionais (2D) contendo dezenas de milhares de células sejam rastreadas. Um programa personalizado é fornecido para o pós-processamento automatizado de dados de análise de imagens de alto conteúdo para produzir um perfil ploidy em toda a amostra para todos os núcleos de hepatócitocircularcircular. Isto é realizado usando software gratuito para baixar software para calcular ploidia nuclear com base na análise de imagem estereológica (SIA)10,11,14,15. A metodologia SIA foi previamente validada pela citometria de fluxo como um método preciso, embora trabalhoso, para estimar ploidiy nuclear hepatocito no fígado14, assumindo morfologia nuclear circular e uma relação monótônica entre tamanho nuclear e conteúdo de DNA. Neste protocolo, ambos os parâmetros nucleares são medidos pela avaliação da mormetria nuclear e da rotulagem Hoechst 33342. O cálculo do "conteúdo mínimo de DNA" para cada máscara nuclear é seguido pela calibração da ploidia nuclear hepatocito usando NPCs, que têm um conteúdo conhecido de DNA 2-4N e, portanto, servem como um controle interno útil.
Em comparação com os métodos convencionais de citometria de fluxo16, a abordagem descrita permite que a ploidiiiiiide nuclear hepatocida seja avaliada in situ e não requer acesso a métodos de tecido fresco ou desagregação que podem influenciar os resultados e ser difíceis de padronizar. Como em todas as abordagens baseadas no SIA, as subclasses ploidiais nucleares >2N são sub-representadas pela amostragem 2D devido à secção de núcleos maiores fora do plano equatorial. O perfil ploidy em todo o tecido também descreve o conteúdo mínimo de DNA para todas as máscaras nucleares hepatocidas circulares, e não discrimina diretamente entre hepatocitos mononucleares e células binucleares que têm dois núcleos discretos ("não tocantes") da mesma ploidy. No entanto, a simplicidade deste protocolo permite um espaço considerável para que ele seja adaptado para responder por parâmetros adicionais, como espaçamento internuclear ou análise de perímetro celular, o que facilitaria a identificação de células binucleares, proporcionando uma avaliação mais detalhada da ploidiia celular.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os experimentos com animais foram previamente aprovados pelo comitê de ética do CIPF. Os camundongos foram alojados em uma instalação sem patógenos no Centro de Investigação Príncipe Felipe (Valência, Espanha), registrado como um criador de animais experimental, usuário e centro de abastecimento (reg. no. ES 46 250 0001 002) nos regulamentos de bem-estar animal europeus e espanhóis vigentes (RD 53/2013).
1. Colheita de tecidos e preparação da amostra
NOTA: Este protocolo descreve como congelar tecidos sem fixação prévia ou criopreservação. Para amostras previamente fixas/criopreservadas, prossiga para a seção 2 e omita a etapa 3.1. Todas as análises foram realizadas utilizando camundongos C57BL/6 adultos com idade entre 12 e 16 semanas.
- Animais de sacrifício por injeção intraperitoneal de fentanil/pentobarbital seguido de luxação cervical. Com o mouse voltado para o lado ventral para cima, abra a cavidade abdominal e exponha o fígado agarrando a pele com pinças e realizando uma incisão vertical desde a base do abdômen inferior até a base do esterno usando uma tesoura cirúrgica.
- Remova cuidadosamente a vesícula biliar usando pinças finas, disseque o fígado e enxágue o lóbulo do fígado selecionado em uma placa de placa de 10 cm de Petri cheia de solução salina tamponada de fosfato (PBS).
NOTA: Recomenda-se comparar o mesmo lóbulo hepático para cada animal, neste caso foi utilizado o lobo mediano. - Encha um criomolde rotulado com temperatura de corte ideal (OCT) média à temperatura ambiente (RT). Evite bolhas oct. Se eles aparecerem, empurre-os para a borda do molde usando uma agulha ou ponta de pipeta.
- Incorpore lobule de fígado em um criomold oct preenchido e coloque-o imediatamente em gelo seco para garantir o congelamento rápido. Armazene criomoldes a -80 °C até a criosecção.
2. Criosecção
- Transporte criomoldes em gelo seco para evitar a degradação do tecido. Antes da criosecção equilibrar o equilíbrio dentro do criostato definido para -20 °C por 20 min.
- Ejetar amostra aplicando pressão na base do criomolde plástico. Aplique oct líquido para aquecer o disco de amostra em RT, posicione-o em criostato e conecte uma amostra de fígado incorporada oct. Aplique pressão suave e espere 3 min para que o OCT congele, garantindo que a amostra grude no disco.
NOTA: Evite manusear a amostra com os dedos o máximo possível para evitar a degradação do tecido. - Bloqueie a amostra no braço do criostato e ajuste a orientação para que a borda da amostra seja paralela à lâmina criostato. Corte na amostra até que o tecido seja atingido.
- Seção da amostra a 6 μm de espessura. Coloque um slide revestido de poliamida rotulado sobre a amostra para 5 s para deixar a amostra grudar no slide. Coloque o slide em RT por 3-5 min, em seguida, para obter melhores resultados, siga diretamente para a seção 3.
NOTA: Para o processamento de múltiplas amostras recém-congeladas, os resultados reprodutíveis foram obtidos armazenando temporariamente slides em uma caixa de slides em gelo seco até que todas as amostras tenham sido processadas. Ao usar esta abordagem, permita que todos os slides se equilibrem com o RT antes de prosseguir para a seção 3. Podem ser utilizadas amostras de parafina fixa de formalina (FFPE), embora a autofluorescência de fundo seja aumentada por este método. Para proceder a partir de amostras de FFPE, seção a 4 μm. Monte capturando seções de banho de água de 40 °C em lâminas tratadas com poliamida. O calor desliza por 1 h a 60 °C, depois deparafina por lava-jatos RT serial (5 min) em frascos de Coplin contendo xileno (x2), etanol 100% (x2), 96% (x2), 70% (x1) e dH2O (x1). Para expor antígenos coloque slides no tampão citrato por 20 min a 90 °C antes de temperar slides em PBS em RT. Siga para o passo 3.2.
3. Imunorotulagem de fluorescência
- Fixar seções teciduals em uma capa de fumaça aplicando 1 mL de 4% de paraformaldeído (PFA) em PBS por 10 min em RT. Transfira slides para um frasco de Coplin cheio de PBS e lave por 3 min usando agitação suave (repetir 3x).
NOTA: A partir de agora até o final do processo de imunocoloração, evite a secagem da amostra. - Seque a área ao redor de cada seção de tecido e cerque usando uma caneta hidrofóbica. Permeabilizar com surfactante ninônico de 0,5% (ou seja, Triton X-100) em PBS por 15 min no RT. Em seguida, lave no pote de Coplin recheado da PBS por 3 min usando agitação suave (repita 2x).
- Bloqueie utilizando uma solução filtrada de albumina de soro bovino de 1% (BSA), 5% de soro de cavalo, 0,2% de surfactante nonionico em PBS (para pelo menos 1 h no RT).
- Incubar com anticorpo HNF4α primário diluído em tampão de bloqueio durante a noite a 4 °C em uma câmara de coloração úmida escura (ver Tabela de Materiais para anticorpos e diluições específicas).
- Coloque slides em um frasco de Coplin cheio de PBS e lave por 3 min usando agitação suave (repita 4x).
- Incubar com anticorpo secundário conjugado Alexa-488 e Hoechst diluído em 1% de BSA filtrado e 0,2% de surfactante nonionico em PBS por 2 h em RT em uma câmara de coloração úmida escura (ver Tabela de Materiais para anticorpos e diluições específicas).
- Coloque slides em um frasco de Coplin cheio de PBS e lave por 3 min usando agitação suave (repita 4x). Lave em ddH2O por 3 min usando agitação suave (repita 2x).
- Monte slides colocando duas gotas de mídia de montagem fluorescente em um deslizamento de cobertura (24 x 60 mm) e colocando slides sobre ele, eliminando bolhas aplicando pressão suave. Para armazenamento a longo prazo, solte a tampa nas bordas com esmalte claro e armazene no escuro a 4 °C.
- Antes de prosseguir, verifique os slides usando um microscópio convencional de fluorescência para garantir uma boa fixação e imunorotulagem.
NOTA: Consulte a Figura 2A,B para obter resultados esperados.
4. Aquisição de imagem de fluorescência
NOTA: Para esta etapa, é necessária uma plataforma de imagem de alto conteúdo(Tabela de Materiais)que suporte a aquisição automática de imagens de fluorescência.
- Ligue o sistema de imagem e abra um novo protocolo de aquisição.
- Selecione o objetivo 10x, observe a área do campo de visão (neste caso 0,6 mm2).
- Defina parâmetros para adquirir imagens de fluorescência usando os filtros apropriados de excitação e emissão (conforme etapa 3.6). Para Hoechst e Alexa-488, selecione os canais "DAPI" e "GFP" com emissão de 390/18 e 438/24 nm e emissão de 432,5/48 e 475/24 nm, respectivamente.
- Concentre a amostra e garanta que a intensidade do sinal não seja saturada. Certifique-se de que a captura de imagens seja feita com o mesmo tempo de exposição para todas as imagens ou use um sistema onde a intensidade da fluorescência seja corrigida para o tempo de exposição.
- Escaneie a amostra e adquira imagens suficientes para obter a cobertura completa da seção de tecidos (aproximadamente 20-50 campos de visão, dependendo do tamanho da amostra).
- Revise o banco de dados de imagens, eliminando manualmente (i) campos mal focados, (ii) aqueles nas bordas de cada seção de tecido (para evitar cálculos de densidade celular tendenciosos) e (iii) aqueles que contenham áreas dobradas/fisicamente danificadas da seção de tecidos, se presentes.
5. Análise automatizada de imagens de fluorescência
NOTA: Esta etapa requer um software de análise de imagem adequado (Tabela de Materiais) capaz de: (1) identificar automaticamente núcleos rotulados hoechst dentro de imagens a 405 nm (segmentação nuclear), (2) avaliar a intensidade nuclear média de Hoechst e a mormormetria e (3) análise de limiar para determinar o status +/- de fluorescência nuclear a 488 nm (HNF4α). Alguns treinamentos/conhecimentos básicos do operador são necessários para avaliar e ajustar visualmente parâmetros de segmentação e limiar dentro do programa para garantir que os núcleos e o status HNF4α+/- sejam otimizados(Figura 2).
- No software de análise de imagens, abra o arquivo de aquisição contendo imagens hoechst (405 nm) e HNF4α (488 nm) da etapa 4.5 e crie um novo protocolo de análise.
- Defina comprimentos de onda a serem utilizados para segmentação nuclear (Hoechst, 405 nm) e para análise de limiar hepatócito/NPC (HNF4α, 488 nm).
- Ajuste os parâmetros de segmentação nuclear do software (como "área nuclear mínima" e "sensibilidade" de detecção nuclear) para garantir que os núcleos sejam segregados de forma ideal.
NOTA: A boa segmentação dos hepatócitos deve ser priorizada sobre a dos NPCs. Os núcleos hepatócitos são caracteristicamente arredondados (faixa de tamanho interquartil: 40-64 μm2). Os núcleos NPC, como os da endotelia sinusoidal, são achatados/elípticos ou irregulares em forma e geralmente menores e mais embaladas do que as dos hepatócitos (faixa de tamanho interquartil: 30-43 μm2). Para o fígado de camundongos, foram utilizadas área nuclear mínima ≥23 μm2 e detecção de "sensibilidade" de 65% (ver Figura 2C,D para resultados esperados). A sensibilidade determina como os clusters de pixels são reconhecidos como núcleos individuais com base em sua intensidade e devem ser empiricamente testados para cada amostra definida pelo usuário antes de prosseguir com a análise automatizada de imagens. - Modifique a intensidade do limiar em 488 nm para garantir a gating ideal de hepatócitos (HNF4α+) e células não parênquias (HNF4α-).
NOTA: Consulte a Figura 2C,D para obter os resultados esperados. O valor da intensidade do limiar é relativo e dependerá da eficiência da coloração e das configurações de aquisição, como intensidade do laser. Portanto, deve ser padronizado pelo usuário. Use células HNF4α- conhecidas como células endoteliais e NPCs periportal como controle negativo interno e núcleos hepatocitos binucleares como referência positiva para a coloração. Teste os parâmetros de análise usando um pequeno número de imagens para garantir uma boa segmentação nuclear e segregação do limiar de intensidade antes de aplicar parâmetros de análise a todo o conjunto de dados. - Selecione os seguintes parâmetros nucleares a serem quantificados: (1) área nuclear baseada na coloração de Hoechst (μm2), (2) intensidade média de Hoechst nuclear (RU), (3) fator de alongamento nuclear (razão média do eixo curto do núcleo ao eixo longo do núcleo, onde um objeto centro-simétrico [não alongado] tem um valor de 1, (4) Nuc 1/(fator de forma), índice de "arredondação" nuclear médio calculado pelo perímetro 2/(4π x área). Os valores variam de 1 ao infinito, onde 1 é um círculo perfeito, (5) status HNF4α (positivo-1 ou negativo-0) e (6) coordenadas nucleares x/y baseadas em "centro de gravidade" (cg), um método para localizar o centro do objeto a partir de imagens em escala de cinza com precisão de subpixel.
- Execute a análise de todos os conjuntos de dados da amostra e exporte dados numéricos da etapa 5.5 para o software de planilha.
6. Análise de dados
NOTA: A etapa de análise de dados pode ser realizada usando qualquer software de planilha padrão.
- Calcular números de células hepatocitos e não hepatócitos.
- Calcular a área total da seção hepática analisada para cada amostra multiplicando o número de campos de visão pela área do campo de visão (etapa 4.2).

- Trabalhando com arquivos de planilha gerados para cada seção hepática, filtre os dados selecionando apenas núcleos HNF4α+. Calcular o número total de núcleos HNF4α+ analisados e dividi-lo pela área total analisada para obter densidade média de hepatócitos para cada amostra (Figura 2F).

- Realize o mesmo cálculo para células não parêmicas filtrando a planilha para hnf4α- células(Figura 2E).
- Calcular a área total da seção hepática analisada para cada amostra multiplicando o número de campos de visão pela área do campo de visão (etapa 4.2).
- Calcule a distribuição do tamanho nuclear do hepatócito.
- Usando software de planilha, filtre dados para selecionar apenas núcleos HNF4α+.
- Valores de enredo da área nuclear em um histograma (Figura 2G). Defina a largura da lixeira para 5 μm2.
NOTA: Os valores de frequência podem ser corrigidos para área (núcleos/mm2) conforme etapa 6.1.1.
- Faça análise de ploididy nuclear hepatocito.
NOTA: Os dados da planilha da etapa 5.6 são utilizados para gerar um perfil de ploididy nuclear para cada amostra. Este processo foi automatizado e pode ser realizado usando um software escrito personalizado que está livremente disponível para download com informações de suporte e conjuntos de dados de demonstração emhttps://github.com/lukeynoon(vejaArquivos Suplementares). O código fonte é fornecido para usuários que desejam adaptar a metodologia. Uma descrição do algoritmo, juntamente com as instruções de instalação e uso são descritas abaixo. O programa utiliza dados de planilhas para separar automaticamente núcleos hepatocitos em dois grupos; (1) aqueles com núcleos circulares "simples" e (2) núcleos não circulares "complexos" representativos de células binucleares com ploidiação >2c. O conteúdo mínimo de DNA nuclear (uma função da área nuclear e densidade de DNA) é o próximo calculado para todos os núcleos "simples". Um passo subseqüente então calibra automaticamente hnf4α+ hepatocyte ploidy nuclear usando hnf4α- núcleos como um controle interno 2−4N conhecido.- Baixe e instale software.
- Baixe o aplicativo embalado em: https://github.com/lukeynoon
- Lançar O MATLAB. Navegue até a aba APP da viagem de ferramentas, clique em Instalar app e abra o aplicativo baixado chamado "Ploidy_Application.mlappinstall". Uma mensagem aparecerá para confirmar a instalação bem sucedida.
NOTA: O aplicativo já está pronto para uso e permanecerá na aba APP da viagem de ferramentas.
- Formato de dados de entrada.
NOTA: Antes da análise automatizada da ploididi, todos os arquivos de planilha contendo dados de imagem de alto conteúdo (etapa 5.6) devem ser armazenados e formatados de acordo com as seguintes instruções.- Em cada arquivo de dados exportado (. A senha XLS 97-2004) da etapa 5.6, inclui uma planilha denominada "Medidas celulares" contendo todos os dados necessários para a análise ploidy definida nas colunas(Figura 3A). Certifique-se de que o layout da planilha, incluindo nomes de cabeçalho de coluna, permanece inalterado em relação ao da Figura 3A,porque o método de análise encontra os dados corretos da coluna pesquisando por esses nomes (consulte conjuntos de dados de demonstração em Arquivos Suplementares para referência). Se, por exemplo, o software de análise de imagens de alto conteúdo não produzir uma coluna "Fluxo de luz"(Figura 3A),insira manualmente uma coluna "Fluxo de luz" no mesmo local, ou seja, a coluna K e preencha-a com zeros.
- Para cada condição experimental (por exemplo, "Lesionado-d14"), forneça um conjunto de dados de controle, que será usado para calcular o controle interno para calibração de ploidia nuclear de 2-4N (passo 6.3.4.3). Aqui, selecione amostras de fígado de companheiros adultos não tratados ("Control-d0"; Figura 3B−D).
- Para réplicas biológicas (por condição), armazene cada planilha em sua própria pasta (como na Figura 3B). Nomeie os prefixos da pasta incrementalmente, por exemplo, "Sample1, Sample2, Sample3... SampleN", de acordo com os nomes de arquivos contidos dentro. Assim, cada pasta de conjunto de dados (por exemplo, "Control-d0") deve conter uma série de subpastas ("Sample1", "Sample2", etc.) cada uma contendo um arquivo de planilha com o mesmo nome correspondente.
- Execute a aplicação.
- Dentro do MATLAB, inicie o "Ploidy_Application" clicando no ícone dentro da guia MEUS APPS da toolstrip(Figura 3C). Aparecerá a Ploidy_Application interface gráfica do usuário (GUI)(Figura 3C).
- Clique no botão Caminho para controlar dados para navegar até a pasta em que os dados de controle se replicam (por exemplo, "Control-d0"). Esse caminho de dados aparecerá na interface (por exemplo, /Users/Desktop/Control-d0).
- Em seguida, no "prefixo de pasta" digite o nome a ser dado aos arquivos de saída (por exemplo, "Amostra").
NOTA: Este prefixo pode ser alterado para qualquer texto, desde que as pastas e nomes de arquivos permaneçam nomeados incrementalmente. - Clique no botão Caminho para outros dados e navegue até a pasta em que os dados comparativos se replicam (por exemplo, "Lesionado-d14"). Esse caminho de dados aparecerá na interface (por exemplo, /Users/Desktop/Injured-d14).
- Clique em Correr!. Quando a análise estiver concluída, a barra de status lerá "Análise Completa!..".
NOTA: A aplicação informará, para cada amostra, estratificação de núcleos "simples" em ≤2n, 2n−4n, 4n−8n e 8n+ em termos de contagem absoluta e como percentual do total(Figura 3D). Esses arquivos serão salvos automaticamente em cada pasta de amostra como: "Count_2n.txt", "Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". O Ploidy_Application salvará automaticamente uma lista para cada amostra, de todas as estimativas individuais de ploidy para núcleos hepatocitos "simples" e não hepatócitos em "Ploidy_All_Hepatocytes.txt" e "Ploidy_NonHepatocytes.txt". Para o conjunto de dados de controle, o método também salva os limites mínimos de conteúdo de DNA calculados para estratificação de ploidy (ver passo 6.3.4.3.7) em um arquivo chamado "Normalised_Thresholds_Control". Finalmente, o aplicativo produzirá uma pasta tanto para o controle quanto para os dados de condição comparativa selecionados denominados "Resumo". Esta pasta contém duas subpastas, "Ploidy" e "Estratificação" que contêm as médias de todas as amostras fornecidas (Figura 3D).
- Descrição da metodologia.
NOTA: A seção a seguir descreve detalhadamente a metodologia utilizada pelo software de Análise de Ploidy Nuclear. Se o usuário optar por não usar o aplicativo, essas etapas podem ser seguidas usando um software de planilha para calcular manualmente o perfil de ploidy nuclear.- Núcleos separados em "simples" ou "complexos" de acordo com a mormetria nuclear.
- Calcular um "índice de circularidade" para todos os núcleos, definido como o "fator de alongamento" nuclear dividido pelo "Nuc 1/(fator de forma)", onde um valor de 1,0 indica um círculo perfeito.
NOTA: "Alongamento nuclear" e "Nuc 1/(fator de forma)" são duas medidas discretas da "circularidade" de um objeto que avaliam critérios morfométricos complementares e não sobrepostos. O primeiro mede os eixos longos e curtos de um objeto, enquanto o segundo compara o comprimento do perímetro de um objeto ao de sua área. Para fortalecer a definição de circularidade nuclear utilizada neste protocolo, essas duas medidas foram combinadas em um único "índice de circularidade". Uma abordagem anterior para estimar a ploidia nuclear utilizando a metodologia descrita utilizou apenas alongamento nuclear17. Embora os resultados aceitáveis tenham sido obtidos por meio dessa abordagem, os autores observaram que um "índice de circularidade" composto melhora a discriminação de núcleos selecionados manualmente de hepatócitos mononucleares e binucleares (dados não mostrados). - Classificar núcleos com índice de circularidade ≤ 0,8 como "complexos" e aqueles > 0,8 como "simples".
- Calcular um "índice de circularidade" para todos os núcleos, definido como o "fator de alongamento" nuclear dividido pelo "Nuc 1/(fator de forma)", onde um valor de 1,0 indica um círculo perfeito.
- Estimar o conteúdo de DNA "mínimo" (m) para todos os núcleos "simples".
- Calcular o raio nuclear (r) usando a fórmula:

- Calcular o volume nuclear (v) utilizando o volume de uma fórmula de esfera:
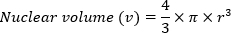
- Gerar um valor relativo para o conteúdo mínimo de DNA (m) usando a fórmula:

- Calcular o raio nuclear (r) usando a fórmula:
- Calibrar o conjunto de dados usando os núcleos NPC (HNF4α-) como um controle interno de 2-4N.
NOTA: Os NPCs têm um conteúdo de DNA de 2-4N, dependendo do status do ciclo celular. Assim, o valor médio do conteúdo de DNA "mínimo" do NPC (NPCm)aumenta com a lesão(Figura 4A). O erro de calibração é minimizado estabelecendo um limite superior de NPCm representando um limiar de 4c(Figura 4B).- Dentro da planilha, selecione apenas núcleos NPC com valores para "m" que estejam dentro de 1 desvio padrão (SD) do modo (isso filtra o ruído de um possível erro de segmentação).
- Dentro deste intervalo filtrado, examine as áreas nucleares e suas intensidades médias correspondentes de Hoechst(Figura 4C).
- Estimar a menor área nuclear dentro desta faixa filtrada com intensidade máxima nuclear de Hoechst (ou seja, o ponto em que a linha da curva muda de direção no conjunto de dados filtrado, conforme ilustrado pelo círculo vermelho na Figura 4C). Este valor representa um estado transitório de 2N−4N (t) acima do qual a amostragem de núcleos 4c predomina sobre núcleos 2c, resultando em uma máxima de intensidade média de Hoechst.
NOTA: Este valor é automaticamente determinado pelo software; no entanto, os usuários de planilha podem selecionar manualmente este ponto como o tamanho de transição. - Calcule o teor mínimo de DNA representado por esse tamanho transitório (tm) seguindo o passo 6.3.4.2.
- Para estimar o ombro 4N do conjunto de dados NPCm, adicione 1 SD ao valor de tm. O número resultante(Figura 4B) descreve o limite superior do conteúdo mínimo de DNA do NPC a ser usado para estratificação de ploidia nuclear (S4c).
- Repita as etapas 6.3.4.3.1−6.3.4.3.5 para todas as amostras de "controle".
NOTA: Por exemplo, na Figura 3,fígados de controle ilesos ("Control-d0") são usados como condição de controle. - Calcule um limiar médio de estratificação de 4c (S4c) para amostras de "controle" e use-o para extrapolar os limites 2c (S2c) e 8c (S8c) para o conteúdo mínimo de DNA (m). Os limiares de estratificação são gerados automaticamente e armazenados pelo software (etapa 6.3.3.3).
NOTA: Dependendo do desenho do estudo, os valores médios do limiar de estratificação podem ser calculados para cada condição ou para condições específicas (por exemplo, fígado de controle saudável). No entanto, o software Nuclear Ploidy Analysis requer que um de um conjunto de 2 arquivos seja designado como "controle" para fins de cálculo de valores ploidy relativos. - Calcule um valor ploidy para todos os núcleos usando o valor S2c gerado na etapa 6.3.4.3.7 conforme:

- Estrateie os núcleos hepatocitos "simples" (HNF4α+) em suportes 2c/4c/8c/>8c de acordo com os seguintes critérios: "2c" HNF4α+ = p ≤ 2; "4c" HNF4α+ = 2 < p ≤ 4; "8c" HNF4α+ = 4 < p ≤ 8; ">8c" HNF4α+ = 8 < p.
- Para reconstruir a padronização espacial dos subgrupos ploidiais, separe os dados nucleares dentro de cada planilha amostral de acordo com os campos correspondentes em que foram adquiridos. Em seguida, use coordenadas nucleares x/y associadas (a partir da etapa 5.5) para traçar subgrupos ploidiais em 2D (Figura 5C).
- Núcleos separados em "simples" ou "complexos" de acordo com a mormetria nuclear.
- Baixe e instale software.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este método tem sido utilizado para medir o impacto da lesão colestática no fígado de camundongos adultos, alimentando animais por 0-21 dias com uma dieta hepatotóxica contendo 0,1% 3,5-dietoxicarbonilo-1,4-dihidrocollidina (DDC)17. A alimentação crônica de DDC resulta em lesão hepatocelular aumento da ploidia e expansão periportal de NPCs. O usuário deve estar ciente de que a tensão do camundongo e as diferenças dependentes da idade podem existir na p...
Access restricted. Please log in or start a trial to view this content.
Discussão
Uma abordagem de alto teor e alto throughput para a análise da remodelagem de tecidos e estimativa da ploidiiiiiie nuclear hepatocito no fígado de murina é descrita. Uma vez familiarizado com o procedimento, o usuário pode processar, imagem e analisar várias amostras em um período de 3 a 5 dias, gerando grandes conjuntos de dados técíveis que fornecem uma assinatura detalhada da saúde hepática. Dada a simplicidade do método de preparação da amostra, juntamente com o grande número de células e área tecidua...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada para revelar.
Agradecimentos
Este trabalho foi financiado pelo Governo de Mineco espanhol concede bfu2014-58686-P (LAN) e SAF-2017-84708-R (DJB). A LAN foi apoiada pelo prêmio nacional MINECO Ramón y Cajal Fellowship RYC-2012-11700 e plan GenT (Comunitat Valenciana, CDEI-05/20-C) e FMN por uma estudantil regional ValI+D da Generalitat ACIF/2016/020. Rp gostaria de reconhecer o Prof. Ewa K. Paluch para financiamento. Agradecemos à Dra. Alicia Martínez-Romero (serviço citometria da CIPF) pela ajuda com a plataforma IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Referências
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados