Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Farelerde Hepatosit Nükleer Ploidi Tahmini için Yerinde Yüksek Throughput Yöntemi
Bu Makalede
Özet
Akış sitometrisi gerektirmeyen sabit/kriyokorunmuş doku örneklerinde hepatosit numarası ve nükleer ploidideki değişiklikleri ölçmek için sağlam, uygun maliyetli ve esnek bir yöntem saseril. Yaklaşımımız karaciğer hasarı ve hastalığın ilerlemesini izlemek için ideal karaciğer sitolojisi güçlü bir örnek çapında imza sağlar.
Özet
Karaciğer yaralandığında hepatosit sayıları azalır, hücre büyüklüğü ise, nükleer boyutu ve ploidi artar. Kolanjiyositler, miyofibroblastlar, atalar ve inflamatuar hücreler gibi parenkimal olmayan hücrelerin genişlemesi de kronik karaciğer hasarı, doku remodeling ve hastalığın ilerlemesini gösterir. Bu protokolde, karaciğerin hücresel bileşiminde yaralanma, kronik hastalık ve kanserle ilişkili değişiklikleri hesaplamak için basit bir yüksek iş ilişkisi yaklaşımını tanımlıyoruz. İki boyutlu (2D) doku bölümlerinden elde edilen bilgilerin, bir numune içinde hepatosit nükleer ploidiyi ölçmek ve kalibre etmek ve kullanıcının karaciğer içinde belirli ploidi alt kümeleri yerinde bulmasını sağlamak için nasıl kullanılabileceğini gösteriyoruz. Yöntemimiz sabit/dondurulmuş karaciğer materyaline, temel immünositokimya reaktiflerine ve herhangi bir standart yüksek içerikli görüntüleme platformuna erişim gerektirir. Bu taze toplanan doku bozulması, mekansal bilgi kaybı ve potansiyel disaggregasyon önyargı gerektiren standart akış sitometri teknikleri, güçlü bir alternatif olarak hizmet vermektedir.
Giriş
Memeli karaciğerindeki hepatositler, binükleer hücreler üretmek için durmuş sitokineze ve 16N DNA içeriği içeren poliploid çekirdekleri üretmek için DNA endorepasyonuna uğrayabilirler. Doğum sonrası gelişim sırasında genel hücresel ve nükleer ploidi artışı, yaşlanma ve çeşitli hücresel streslere yanıtolarak 1. Poliploidizasyon süreci dinamik ve geri dönüşümlü2, kesin biyolojik işlevi belirsiz kalırken3. Artmış ploidi azaltılmış proliferatif kapasite ile ilişkilidir4, genetik çeşitlilik2, kronik yaralanma adaptasyon5 ve kanserden korunma6. Hepatosit ploidi değişiklikler değişmiş sirkadiyenritimsonucu ortaya 7 , ve keserek8. En önemlisi, karaciğer ploidi profili yaralanma ve hastalıktarafındandeğiştirilir 9 , ve zorlayıcı kanıtlar belirli ploidi değişiklikler, artmış ≥8N çekirdekleri veya 2N hepatosit kaybı gibi, alkolsüz yağlı karaciğer hastalığı izlemek için yararlı imzalar sağlamak düşündürmektedir (NAFLD)3,10, veya viral enfeksiyonların diferansiyel etkisi11.
Genel anlamda, karaciğer hasarı ve rejenerasyon artmış hepatosit hücre büyüklüğü ve nükleer alan ile ilişkilidir12, hepatosit azaltılmış genel sayılar ile birlikte, özellikle 2N DNA içeriği10olanlar10 ,11. Karaciğerde parenkimal yaralanma da sıklıkla non-parenkimal hücrelerin genişlemesi eşlik (NPCs), stromal miyofibroblastlar da dahil olmak üzere, inflamatuar hücreler ve bipotent karaciğer ata hücreleri. Parenkimal hücre numarası ve nükleer ploidin nicel sitolojik profilini sağlayan yüksek iş parçacığı yöntemleri, nfc'lerde ki değişiklikleri de hesaba katarken, bu nedenle yaralanma ve hastalık sırasında karaciğerin yanıtını takip etmek için araştırma ve klinik araçlar olarak önemli bir potansiyele sahiptir. Hepatosellüler karsinom insan örneklerinde ploidy spektrumların yerinde analizi zorlayıcı son da nükleer ploidi önemli ölçüde tümörler içinde artar ve özellikle azaltılmış farklılaşma ve TP53 kaybı ile daha agresif tümör alt tipleri amplifiye olduğunu göstermektedir13. Bu nedenle, nükleer ploidi kantitatif değerlendirmede metodolojik gelişmeler karaciğer kanserinin gelecekteki prognostik profilleme yardımcı olacağını güçlü bir olasılık vardır.
Bu protokolde, fare karaciğer doku kesitlerinin karşılaştırmalı analizi için esnek bir yüksek iş lenme metodolojisi tanımlanmış olup, hepatosit sayılarının ayrıntılı sitometrik profillemesi, NPC yanıtı ve nükleer ploidiyi tahmin etmek için dahili olarak kalibre edilmiş bir yöntem(Şekil 1). Hepatositler ncd'lerden hepatosit nükleer faktör 4 alfa (HNF4α) immünetiketleme ile nükleer boyut ve nükleer morfometri karakterizasyonundan önce ayrılırlar. "Minimal DNA içeriği" ortalama Hoechst 33342 yoğunluğu (DNA yoğunluğu için bir proxy) interpole üç boyutlu (3D) nükleer hacim ile entegre ederek tüm dairesel nükleer maskeler için tahmin edilmektedir. Hepatosit minimal DNA içeriği daha sonra nükleer ploidi profili oluşturmak için NFC'ler kullanılarak kalibre edilir.
Görüntü edinimi, nükleer segmentasyon ve görüntü analizi yüksek içerikli görüntüleme kullanılarak gerçekleştirilir ve on binlerce hücreiçeren iki boyutlu (2D) karaciğer bölümlerinin geniş alanlarının taranmasını sağlar. Tüm dairesel hepatosit çekirdekleri için örnek geniş ploidi profili oluşturmak için yüksek içerikli görüntü analizi verilerinin otomatik olarak işlenmesi için özel olarak yazılmış bir program sağlanmaktadır. Bu stereoolojik görüntü analizi (SIA)10,11,,14,15dayalı nükleer ploidy hesaplamak için yazılım indirmek için ücretsiz kullanılarak gerçekleştirilir. SIA metodolojisi daha önce dairesel nükleer morfolojisi ve nükleer boyut ve DNA içeriği arasında monoton bir ilişki varsayarak, karaciğer14hepatosit nükleer ploidi tahmin etmek için doğru, zahmetli de olsa, doğru bir yöntem olarak akış sitometri tarafından doğrulanmıştır. Bu protokolde, her iki nükleer parametre de nükleer morfometri ve Hoechst 33342 etiketleme nin değerlendirilmesi ile ölçüldü. Her nükleer maske için "minimal DNA içeriğinin" hesaplanması, bilinen 2−4N DNA içeriğine sahip olan ve bu nedenle yararlı bir iç kontrol görevi gören NPT'ler kullanılarak hepatosit nükleer ploidin kalibrasyonu ile takip edilir.
Konvansiyonel akış sitometri yöntemleri ile karşılaştırıldığında16 açıklanan yaklaşım hepatosit nükleer ploidi yerinde değerlendirilmesini sağlar ve sonuçları önyargı ve standartlaştırmak zor taze doku veya disaggregasyon yöntemlerine erişim gerektirmez. Tüm SIA tabanlı yaklaşımlarda olduğu gibi, nükleer ploidi alt sınıfları >2N ekvator düzlemi dışında büyük çekirdeklerin kesiti nedeniyle 2B örnekleme ile yeterince temsil edilmiştir. Doku çapında ploidi profili de tüm dairesel hepatosit nükleer maskeler için minimum DNA içeriğini açıklar ve doğrudan mononükleer hepatositler ve aynı ploidi iki ayrık ("dokunmaz") çekirdekleri var binükleer hücreler arasında ayrım yapmaz. Ancak, bu protokolün basitliği, hücre içi ploidin daha ayrıntılı bir değerlendirmesini sağlayan iki nükleer hücrelerin belirlenmesini kolaylaştıracak, nükleer aralık lar veya hücre çevre analizi gibi ek parametrelere göre uyarlanması için önemli bir kapsam sağlar.
Access restricted. Please log in or start a trial to view this content.
Protokol
Tüm hayvan deneyleri daha önce CIPF etik komitesi tarafından onaylanmıştır. Fareler Centro de Investigación Príncipe Felipe'de (Valencia, İspanya) patojensiz bir tesiste, deneysel bir hayvan yetiştiricisi, kullanıcı ve tedarik merkezi olarak kayıtlı (reg. no. ES 46 250 0001 002) geçerli Avrupa ve İspanyol hayvan refahı düzenlemeleri (RD 53/2013) uyarınca.
1. Doku hasadı ve numune hazırlama
NOT: Bu protokol, önceden fiksasyon veya kriyopreservation olmadan doku dondurma nasıl açıklanır. Daha önce sabit/kriyokorunmuş örnekler için bölüm 2'ye geçin ve adım 3.1'i atlayın. Tüm analizler 12−16 haftalık yetişkin dişi C57BL/6 fareler kullanılarak yapılmıştır.
- Fentanil/pentobarbital intraperitoneal enjeksiyon ve ardından servikal çıkış ile kurban hayvanlar. Fare nin ventral tarafa dönük olmasıyla karın boşluğunu açın ve cildi cımbızla tutarak ve cerrahi makas kullanarak alt karın tabanından göğüs tabanına dikey bir kesi yaparak karaciğeri ortaya çıkarın.
- İnce cımbız kullanarak safra kesesini dikkatlice çıkarın, karaciğeri parçalayın ve seçilen karaciğer lobulesini fosfat tamponlu salin (PBS) ile doldurulmuş 10 cm'lik Petri kabında durulayın.
NOT: Her hayvan için aynı karaciğer lobu nun karşılaştırılması tavsiye edilir, bu durumda ortanca lob kullanılmıştır. - Etiketli bir kriyomold'u oda sıcaklığında (RT) optimum kesme sıcaklığı (OCT) ortamıyla doldurun. OCT kabarcıkları kaçının. Görünürlerse, bir iğne veya pipet ucu kullanarak kalıbın kenarına itin.
- Dolu bir OCT kriyomold içine karaciğer lobule gömmek ve hemen hızlı donma sağlamak için kuru buz üzerine yerleştirin. Kriyomoldları -80 °C'de kriyoseksiyona kadar saklayın.
2. Kriyoding
- Doku bozulmasını önlemek için kuru buz üzerinde cryomolds taşıma. Kriyodize den önce 20 dk için -20 °C olarak ayarlanmış kriyostat içinde denge.
- Plastik kriyomold tabanına basınç uygulayarak numune çıkarmak. RT'deki numune diskini ısıtmak için sıvı OCT uygulayın, kriyostat'ta konumlandırın ve OCT gömülü karaciğer örneğini takın. Hafif basınç uygulayın ve numunenin diske yapışmasını sağlamak için OCT'nin donması için 3 dakika bekleyin.
NOT: Doku bozulmasından kaçınmak için numuneyi mümkün olduğunca parmaklarla kullanmaktan kaçının. - Örneği kriyostat koluna kilitleyin ve yönü, numunenin kenarının kriyostat bıçakla paralel olacak şekilde ayarlayın. Doku ulaşılına kadar numuneyi kesin.
- Numuneyi 6 μm kalınlığında kesin. Numunenin slayta yapışmasını sağlamak için numunenin üzerine 5 s'lik etiketli poliamid kaplı bir slayt yerleştirin. Slaytı 3−5 dakika boyunca RT'ye yerleştirin ve en iyi sonuçlar için doğrudan bölüm 3'e gidin.
NOT: Birden fazla taze dondurulmuş numunenin işlenmesi için, tüm numuneler işlenene kadar slaytların kuru buz üzerinde geçici olarak bir slayt kutusunda saklanması ile tekrarlanabilir sonuçlar elde edilmiştir. Bu yaklaşımı kullanırken, bölüm 3'e geçmeden önce tüm slaytların RT'ye dengede olmasını bekleyin. Formalin sabit parafin gömülü (FFPE) örnekleri kullanılabilir, ancak arka plan otofloresansbu yöntemle artar. FFPE örneklerinden devam etmek için, 4 μm. Mount bölümünde poliamid ile işlenmiş slaytlar üzerinde 40 °C su banyosu bölümleri yakalayarak. Isı slaytlar için 1 saat 60 °C, daha sonra seri RT yıkama (5 dakika) ksilen içeren Coplin kavanoz (5 dakika), etanol 100% (x2), % 96 (x2), 70% (x1) ve dH2O (x1) ile deparaffinize. Antijenleri ortaya çıkarmak için, RT'de PBS'deki slaytları temperlemeden önce 90 °C'de 20 dakika boyunca sitrat tamponuna slayt yerleştirin.
3. Floresan immünetiketleme
- PBS'de 1 0 dakika boyunca %4 paraformaldehit (PFA) uygulayarak duman kaputundaki doku bölümlerini düzeltin.
NOT: Şu andan itibaren immünboyama işleminin sonuna kadar, numunenin kurumasını önleyebilirsiniz. - Kuru her doku bölümü etrafında alan ve bir hidrofobik kalem kullanarak çevreleyen. Rt'de 15 dakika boyunca PBS'de %0,5 noniyonik yüzey aktif madde (yani Triton X-100) ile permeabilize olun. Sonra pbs dolu Coplin kavanozda 3 dakika nazik ajitasyon (tekrar 2x) kullanarak yıkayın.
- %1 büyükbaş serum albumin (BSA), %5 at serumu, PBS'de %0.2 noniyonik yüzey aktif madde (RT'de en az 1 saat süreyle) filtrelenmiş bir solüsyon kullanarak bloke edin.
- Koyu nemli boyama odasında gece boyunca tampon uzerinde niskende seyreltilmiş primer HNF4α antikorlu inkübat (antikorlar ve spesifik seyreltmeler için Malzemeler Tablosuna bakınız).
- PBS dolu Coplin kavanozuna slaytlar yerleştirin ve nazik ajitasyon kullanarak 3 dakika yıkayın (tekrar 4x).
- Alexa-488 konjuge sekonder antikor ve Hoechst ile süzülen seyreltilmiş 1% BSA ve 0.2% niyonik yüzeyaktif pBS için 2 saat RT koyu nemli boyama odasında (antikorlar ve spesifik seyreltmeler için Malzemeler Tablosu bakınız).
- PBS dolu Coplin kavanozuna slaytlar yerleştirin ve nazik ajitasyon kullanarak 3 dakika yıkayın (tekrar 4x). Nazik ajitasyon kullanarak 3 dakika ddH2O yıkayın (tekrar 2x).
- Bir kapak üzerinde floresan montaj ortamı iki damla yerleştirerek slaytlar montaj (24 x 60 mm) ve üzerine slaytlar koyarak, hafif basınç uygulayarak kabarcıklar ortadan kaldırarak. Uzun süreli depolama için, açık oje ile kenarlarında mühür kapağı kayma ve 4 °C karanlıkta saklayın.
- Devam etmeden önce, iyi bir fiksasyon ve immün etiketleme sağlamak için geleneksel floresan mikroskobu kullanarak slaytları kontrol edin.
NOT: Beklenen sonuçlar için Şekil 2A,B'ye bakınız.
4. Floresan görüntü edinimi
NOT: Bu adım için, otomatik floresan görüntü edinimi destekleyen yüksek içerikli görüntüleme platformu(Malzeme Tablosu)gereklidir.
- Görüntüleme sistemini açın ve yeni bir satın alma protokolü açın.
- 10x hedefini seçin, görüş alanının alanını not edin (bu durumda 0,6 mm2).
- Uygun uyarma ve emisyon filtrelerini kullanarak floresan görüntüleri elde etmek için parametreleri ayarlayın (adım 3.6'ya göre). Hoechst ve Alexa-488 için sırasıyla 390/18 ve 438/24 nm uyarma ve 432,5/48 ve 475/24 nm emisyonlu "DAPI" ve "GFP" kanallarını seçin.
- Numuneye odaklanın ve sinyal yoğunluğunun doyup olmadığından emin olun. Görüntü yakalamanın tüm görüntüler için aynı pozlama süresiyle yapıldığından emin olun veya pozlama süresi için floresan yoğunluğunun düzeltildiği bir sistem kullanın.
- Örnek tarayıp doku bölümünün tam kapsamını elde etmek için yeterli görüntü elde edin (örnek boyutuna bağlı olarak yaklaşık 20−50 görüş alanı).
- Görüntü veritabanını gözden geçirin, (i) kötü odaklanmış alanları, (ii) her doku bölümünün sınırlarında bulunanları (hücre yoğunluğu hesaplamalarını önyargılı önlemek için) ve (iii) varsa doku bölümünün katlanmış/fiziksel hasarlı alanlarını içeren alanları el ile gözden geçirin.
5. Otomatik floresan görüntü analizi
NOT: Bu adım uygun görüntü analiz yazılımı(Tablo Malzemeler)yeteneğine sahiptir: (1) otomatik olarak 405 nm (nükleer segmentasyon) görüntüleri içinde Hoechst etiketli çekirdekleri tanımlayan, (2) ortalama Hoechst nükleer yoğunluk ve morfometri değerlendirme, ve (3) eşik analizi 488 nm (HNF4α) nükleer floresan +/- durumunu belirlemek için. Bazı temel operatör eğitimi/uzmanlığı, çekirdeklerin ve HNF4α+/- durumunun en iyi şekilde geçitli olmasını sağlamak için program daki segmentasyon ve eşik parametrelerini görsel olarak değerlendirmek ve ayarlamak için gereklidir(Şekil 2).
- Görüntü analizi yazılımında, Adım 4.5'ten Hoechst (405 nm) ve HNF4α (488 nm) görüntüleri içeren satın alma dosyasını açın ve yeni bir analiz protokolü oluşturun.
- Nükleer segmentasyon (Hoechst, 405 nm) ve hepatosit/NPC eşik analizi (HNF4α, 488 nm) için kullanılacak dalga boylarını tanımlayın.
- Çekirdeğin en iyi şekilde ayrılmasını sağlamak için yazılımın nükleer segmentasyon parametrelerini ("minimum nükleer alan" ve nükleer algılama "hassasiyeti" gibi) ayarlayın.
NOT: Hepatositlerin iyi segmentasyonu NPT'lere göre önceliklendirilmelidir.2 Sinüzoidal endotel çekirdekleri gibi NPC çekirdekleri düzleştirilmiş/eliptik veya düzensiz şeklindedir ve genellikle hepatositlerinkinden daha küçük ve daha yakın paketlenmiştir (interquartile boyut aralığı: 30−43 μm2). Fare karaciğeri için minimal nükleer alan ≥23 μm2 ve %65'lik bir "duyarlılık" saptandı (beklenen sonuçlar için Şekil 2C,D'ye bakınız). Duyarlılık, piksel kümelerinin yoğunluklarına göre nasıl ayrı çekirdekler olarak tanındığını belirler ve otomatik görüntü analizine geçmeden önce kullanıcı tarafından ayarlanan her örnek için ampirik olarak test edilmelidir. - Hepatositlerin (HNF4α+) ve parenkimal olmayan hücrelerin (HNF4α-) en iyi şekilde gating edilmesini sağlamak için eşik yoğunluğunu 488 nm olarak değiştirin.
NOT: Beklenen sonuçlar için Şekil 2C,D'ye bakınız. Eşik yoğunluğunun değeri görecelidir ve lazer yoğunluğu gibi boyama verimliliği ve edinme ayarlarına bağlıdır. Bu nedenle kullanıcı tarafından standartlaştırılmalıdır. Endotel hücreleri ve periportal NPC'ler gibi bilinen HNF4α hücreleri iç negatif kontrol ve binükleer hepatosit çekirdekleri boyama için olumlu bir referans olarak kullanın. Analiz parametrelerini tüm veri kümesine uygulamadan önce iyi bir nükleer segmentasyon ve yoğunluk eşiği ayrımı sağlamak için az sayıda görüntü kullanarak analiz parametrelerini test edin. - Ölçülmek için aşağıdaki nükleer parametreleri seçin: (1) Hoechst boyama (μm2),(2) ortalama nükleer Hoechst yoğunluğu (RU), (3) nükleer uzama faktörüne (çekirdeğin kısa ekseninin çekirdeğin uzun eksenine ortalama oranı) dayalı nükleer alan, bir merkez-simetrik [uzamış olmayan] nesnenin değeri 1, (4) Nuc 1/(form faktörü), çevre 2/(4π x alanı) tarafından hesaplanan ortalama nükleer "yuvarlaklık" indeksi. Değerler 1'in mükemmel bir daire olduğu 1'den sonsuza kadar değişir, (5) HNF4α durumu (pozitif-1 veya negatif-0) ve (6) nükleer x/y koordinatları "ağırlık merkezi" (cg) temel alınarak, nesnenin merkezini alt piksel hassasiyetiyle gri tonlu görüntülerden bulma yöntemidir.
- Tüm örnek veri kümeleri için çözümleme çalışması ve 5.5 adımından elektronik tablo yazılımına sayısal verileri dışa aktarın.
6. Veri analizi
NOT: Veri analizi adımı herhangi bir standart elektronik tablo yazılımı kullanılarak gerçekleştirilebilir.
- Hepatosit ve hepatosit olmayan hücre numaralarını hesaplayın.
- Her örnek için analiz edilen karaciğer bölümünün toplam alanını, görüş alanının alanıyla çarparak hesaplayın (adım 4.2).

- Her karaciğer bölümü için oluşturulan elektronik tablo dosyalarıyla çalışarak, yalnızca HNF4α+ çekirdeklerini seçerek verileri filtreleyin. Analiz edilen toplam HNF4α+ çekirdeği sayısını hesaplayın ve bunu analiz edilen toplam alana bölerek her bir örnek için ortalama hepatosit yoğunluğu elde edin (Şekil 2F).

- HNF4α- hücreleri için elektronik tabloyu filtreleyerek parenkimal olmayan hücreler için aynı hesaplamayı yapın (Şekil 2E).
- Her örnek için analiz edilen karaciğer bölümünün toplam alanını, görüş alanının alanıyla çarparak hesaplayın (adım 4.2).
- Hepatosit nükleer boyut dağılımını hesaplayın.
- Elektronik tablo yazılımLarını kullanarak, yalnızca HNF4α+ çekirdeklerini seçmek için verileri filtreleyin.
- Bir histogramdaki nükleer alanın çizim değerleri (Şekil 2G). Depo alanı genişliğini 5 μm2olarak ayarlayın.
NOT: Frekans değerleri 6.1.1 adıma göre alan (çekirdek/mm2)için düzeltilebilir.
- Hepatosit nükleer ploidi analizini yapın.
NOT: Adım 5.6'daki elektronik tablo verileri her örnek için nükleer ploidi profili oluşturmak için kullanılır. Bu işlem otomatikleştirilmiştir ve destekleyici bilgiler ve tanıtım veri kümeleri ile ücretsiz olarak indirilebilen özel bir yazılı yazılım kullanılarak gerçekleştirilebilirhttps://github.com/lukeynoon(bkz.Ek Dosyalar). Kaynak kodu, metodolojiyi uyarlamak isteyen kullanıcılar için sağlanır. Algoritmanın tanımı, kurulum ve kullanım talimatlarıyla birlikte aşağıda özetlenmiştir. Program, hepatosit çekirdeklerini otomatik olarak iki gruba ayırmak için elektronik tablo verilerini kullanır; (1) "basit" dairesel çekirdekleri ve (2) "karmaşık" dairesel olmayan çekirdekleri <2c ploidy ile binükleer hücrelerin temsilcisi olanlar. En az nükleer DNA içeriği (nükleer alan ve DNA yoğunluğu bir fonksiyonu) sonraki tüm "basit" çekirdekleri için hesaplanır. Daha sonraki bir adım, HNF4α- çekirdeklerini kullanarak HNF4α+ hepatosit nükleer ploidiyi otomatik olarak kalibre eder.- Yazılımı indirin ve yükleyin.
- Paket uygulamayı aşağıdaki: https://github.com/lukeynoon
- MATLAB'ı başlatın. Toolstrip'in APP sekmesine gidin, Uygulamayı Yükle'yi tıklatın ve indirilen"Ploidy_Application.mlappinstall" adlı uygulamayı açın. Başarılı yüklemeyi onaylamak için bir ileti görüntülenir.
NOT: Uygulama kullanıma hazırdır ve araçlar gezisinin APP sekmesinde kalır.
- Giriş verilerini biçimlendirin.
NOT: Otomatik nükleer ploidi analizinden önce, yüksek içerikli görüntüleme verileri (adım 5.6) içeren tüm elektronik tablo dosyaları aşağıdaki talimatlara göre depolanmalı ve biçimlendirilmelidir.- Dışa aktarılan her veri dosyasında (. XLS 97-2004 çalışma kitabı) adım 5.6, sütunlarda belirlenen ploidy analizi için gerekli tüm verileri içeren "Hücre ölçüleri" olarak adlandırdığı bir sayfa içerir(Şekil 3A). Çözümleme yöntemi bu adları arayarak doğru sütun verilerini bulduğundan, sütun üstbilgi adlarını içeren elektronik tablo düzeninin Şekil 3A'dakindendeğişmediğinden emin olun (bkz. başvuru için Ek Dosyalar'daki gösterim veri kümeleri). Örneğin, yüksek içerikli görüntü analizi yazılımı bir "Işık akısı" sütunu(Şekil 3A)oluşturmuyorsa, aynı konuma, yani K sütununa el ile bir "Işık akısı" sütunu ekleyin ve sıfırlarla doldurun.
- Her deneysel durum için (örneğin, "Yaralı-d14"), 2−4N nükleer ploidy kalibrasyonu (adım 6.3.4.3) için iç kontrolü hesaplamak için kullanılacak bir kontrol veri seti sağlar. Burada, tedavi edilmeyen yetişkin çöp arkadaşlarından ("Control-d0"; Şekil 3B−D).
- Biyolojik çoğaltmalar için (koşul başına), her elektronik tabloyu kendi klasöründe saklayın (Şekil 3B'dekigibi). Klasör önekleri artımlı olarak adlandırın, örneğin, "Örnek1, Örnek2, Örnek3... SampleN", içinde yer alan dosya adlarını göre. Bu nedenle, her veri kümesi klasörü (örneğin, "Control-d0") her biri aynı adı taşıyan bir elektronik tablo dosyası içeren bir dizi alt klasör ("Örnek1", "Örnek2", vb.) içermelidir.
- Uygulamayı çalıştırın.
- MATLAB içinde, araçlar gezisinin MY APPS sekmesindeki simgeye tıklayarak "Ploidy_Application" başlatın (Şekil 3C). Ploidy_Application grafik kullanıcı arabirimi (GUI) görünür (Şekil 3C).
- Denetim verilerinin çoğaltıldığı klasöre gitmek için veri düğmesini denetlemek için Yolu tıklatın (örn. "Control-d0"). Bu veri yolu daha sonra arabirimde görünür (örn. /Users/Desktop/Control-d0).
- Ardından, "klasör öneki" türünde çıktı dosyalarına verilecek ad (örneğin, "Örnek").
NOT: Bu önek, klasörlerin ve dosya adlarının artımlı olarak kalması koşuluyla herhangi bir metinle değiştirilebilir. - Diğer veri yolu düğmesini tıklatın ve karşılaştırmalı verilerin çoğaltıldığı klasöre gidin (örn. "Yaralı-d14"). Bu veri yolu daha sonra arabirimde görünür (örn. /Users/Desktop/Injured-d14).
- Çalıştır'ı tıklatın! Çözümleme tamamlandığında durum çubuğunda "Çözümleme Tamamlandı!.." okunur.
NOT: Uygulama, her örnek için ≤2n, 2n−4n, 4n−8n ve 8n+ içine "basit" çekirdeklerin tabakalaşmasını mutlak sayımlar açısından ve toplamın yüzdesi olarak rapor edecektir (Şekil 3D). Bu dosyalar her örnek klasöre otomatik olarak kaydedilir: "Count_2n.txt", "Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". Ploidy_Application otomatik olarak her örnek için bir liste kaydedecektir, "basit" hepatosit ve non-hepatosit çekirdekleri için tüm bireysel ploidy tahminleri "Ploidy_All_Hepatocytes.txt" ve "Ploidy_NonHepatocytes.txt". Kontrol veri kümesi için yöntem, "Normalised_Thresholds_Control"adlı bir dosyada ploidy tabakalaşması için hesaplanan en az DNA içerik eşiklerini de kaydeder (bkz. adım 6.3.4.3.7). Son olarak, uygulama hem denetim hem de "Özet" olarak adlandırılır seçili karşılaştırmalı durum verileri için bir klasör üretecektir. Bu klasör, sağlanan tüm örneklerin ortalamalarını içeren "Ploidy" ve "Tabakalaşma" olmak üzere iki alt klasör içerir(Şekil 3D).
- Metodolojinin tanımı.
NOT: Aşağıdaki bölümde Nükleer Ploidy Analizi yazılımı tarafından kullanılan metodoloji ayrıntılı olarak açıklanmaktadır. Kullanıcı uygulamayı kullanmamayı seçerse, nükleer ploidy profilini el ile hesaplamak için elektronik tablo yazılımı kullanılarak bu adımlar izlenebilir.- Çekirdekleri nükleer morfometriye göre "basit" veya "karmaşık" olarak ayırın.
- 1,0 değerinin mükemmel bir daireyi gösterdiği "Nuc 1/(form faktörü)" ile bölünen nükleer "uzama faktörü" olarak tanımlanan tüm çekirdekler için bir "dairesellik indeksi" hesaplayın.
NOT: "Nükleer uzama" ve "Nuc 1/(form faktörü)" bir nesnenin tamamlayıcı, örtüşen morfometrik kriterleri değerlendiren "daireselliği"nin iki ayrı ölçüsüdür. Eski, bir nesnenin uzun ve kısa eksenlerini ölçerken, ikincisi bir nesnenin çevresinin uzunluğunu kendi alanının ki ile karşılaştırır. Bu protokolde kullanılan nükleer dairesellik tanımını güçlendirmek için bu iki ölçüm tek bir "dairesellik indeksi" olarak birleştirilmiştir. Açıklanan metodoloji kullanarak nükleer ploidi tahmin etmek için bir önceki yaklaşım sadece nükleer uzama17kullanılır. Bu yaklaşım kullanılarak kabul edilebilir sonuçlar elde edilirken, yazarlar kompozit bir "dairesellik indeksi"nin mononükleer ve binükleer hepatositlerden elle seçilmiş çekirdeklerin ayrımcılığını artırdığını gözlemlemiştir (veriler gösterilmemiştir). - Çekirdekleri dairesellik indeksi ≤ 0,8 ile "karmaşık" ve bu > 0,8'i "basit" olarak sınıflandırın.
- 1,0 değerinin mükemmel bir daireyi gösterdiği "Nuc 1/(form faktörü)" ile bölünen nükleer "uzama faktörü" olarak tanımlanan tüm çekirdekler için bir "dairesellik indeksi" hesaplayın.
- Tüm "basit" çekirdekler için "minimal" DNA içeriğini (m) tahmin edin.
- Formülü kullanarak nükleer yarıçapı (r) hesaplayın:

- Bir küre formülünün hacmini kullanarak nükleer hacmi (v) hesaplayın:
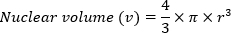
- Formülü kullanarak en az DNA içeriği (m) için göreli bir değer oluşturun:

- Formülü kullanarak nükleer yarıçapı (r) hesaplayın:
- NPC (HNF4α-) çekirdeklerini kullanarak veri kümesini dahili 2−4N kontrol olarak kalibre edin.
NOT: NDC hücre döngüsü durumuna bağlı olarak 2−4N DNA içeriğine sahiptir. Bu nedenle, NPC ortalama değeri "minimal" DNA içeriği (NPCm)yaralanma ile artar (Şekil 4A). Kalibrasyon hatası, 4c eşiğini temsil eden NPCm üst sınırı oluşturularak en aza indirilir (Şekil 4B).- Elektronik tabloda, modun 1 standart sapması (SD) içinde yer alan "m" değerlerine sahip yalnızca NPC çekirdeklerini seçin (bu durum olası segmentasyon hatasından gelen gürültüyü filtreler).
- Bu filtreli aralık içinde, nükleer alanları ve buna karşılık gelen ortalama Hoechst yoğunluklarını inceleyin(Şekil 4C).
- Bu filtrelenmiş aralıktaki en küçük nükleer alanı maksimal nükleer Hoechst yoğunluğuyla tahmin edin (yani, eğri çizgisinin Şekil 4C'dekikırmızı dairede gösterildiği gibi filtrelenmiş veri kümesinde yön değiştirdiği nokta). Bu değer, 4c çekirdeklerinin örneklemesinin 2c çekirdeklerin üzerinde baskın olduğu 2N−4N geçiş durumunu (t) temsil eder ve ortalama Hoechst yoğunluğunun bir maksimaile sonuçlanır.
NOT: Bu değer yazılım tarafından otomatik olarak belirlenir; ancak, elektronik tablo kullanıcıları bu noktayı geçiş boyutu olarak el ile seçebilir. - 6.3.4.2 adımını izleyerek bu geçiş boyutu (tm)ile temsil edilen minimum DNA içeriğini hesaplayın.
- NPCm veri kümesinin 4N omzunu tahmin etmek için tmdeğerine 1 SD ekleyin. Ortaya çıkan sayı(Şekil 4B),nükleer ploidi tabakalaşma için kullanılacak NPC minimal DNA içeriğinin üst sınırını açıklar (S4c).
- Tüm "kontrol" örnekleri için 6.3.4.3.1−6.3.4.3.5 adımlarını tekrarlayın.
NOT: Örneğin, Şekil3'te, yaralanmamış kontrol karaciğerleri ("Control-d0") kontrol koşulu olarak kullanılır. - "Kontrol" örnekleri için ortalama 4c tabakalaşma eşiğini (S4c)hesaplayın ve bunu en az DNA içeriği (m) için 2c (S2c)ve 8c (S8c)sınırlarını tahmin etmek için kullanın. Tabakalaşma eşikleri otomatik olarak oluşturulur ve yazılım tarafından depolanır (adım 6.3.3.3).
NOT: Çalışma tasarımına bağlı olarak, her durum için veya belirli koşullar için (örn. sağlıklı kontrol karaciğeri) ortalama tabakalaşma eşik değerleri hesaplanabilir. Ancak, Nükleer Ploidy Analizi yazılımı bir 2 dosya kümesinden biri göreli ploidy değerleri hesaplamak amacıyla "kontrol" olarak belirlenmiş gerektirir. - 6.3.4.3.7 adımında oluşturulan S2c değerini kullanarak tüm çekirdekler için ploidi değerini hesaplayın:

- "Basit" hepatosit (HNF4α+) çekirdeklerini 2c/4c/8c/>8c paranteziçine aşağıdaki kriterlere göre teslif edin: "2c" HNF4α+ = p ≤ 2; "4c" HNF4α+ = 2 < p ≤ 4; "8c" HNF4α+ = 4 < p ≤ 8; ">8c" HNF4α+ = 8 < p.
- Ploidi alt grupların mekansal desenlerini yeniden oluşturmak için, her bir örnek tablosundaki nükleer verileri, satın alındıkları ilgili alanlara göre ayırın. Daha sonra 2B ploidy alt grupları çizmek için ilişkili nükleer x/ y koordinatları (adım 5.5) kullanın (Şekil 5C).
- Çekirdekleri nükleer morfometriye göre "basit" veya "karmaşık" olarak ayırın.
- Yazılımı indirin ve yükleyin.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Bu yöntem, 0.1% 3,5-diethoxycarbonyl-1,4-dihidrocollidine (DDC)17içeren bir hepatotoksik diyet ile 0−21 gün boyunca hayvanları besleyerek yetişkin fare karaciğeri üzerinde kolestatik yaralanma etkisini ölçmek için kullanılmıştır. Kronik DDC besleme sonuçları hepatosellüler yaralanma artmış ploidi ve NPCs periportal genişleme. Kullanıcı, fare zorlanması ve yaşa bağlı farklılıkların nükleer ploidide mevcut olabileceğini ve tüm analiz...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Minür karaciğerde doku remodeling ve hepatosit nükleer ploidi tahmini analizi için yüksek içerikli, yüksek iş çıkışlı bir yaklaşım açıklanmıştır. Bir kez prosedür aşina, bir kullanıcı işleyebilir, görüntü ve 3−5 günlük bir süre içinde birden fazla örnek analiz, karaciğer sağlığı ayrıntılı bir imza sağlayan büyük test edilebilir veri kümeleri üreten. Örnek hazırlama yönteminin basitliği göz önüne alındığında, analiz edilen çok sayıda hücre ve doku alanı (ortal...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma İspanyol MINECO Hükümeti tarafından finanse edilmiştir BFU2014-58686-P (LAN) ve SAF-2017-84708-R (DJB). LAN ulusal MINECO Ramón y Cajal Bursu RYC-2012-11700 ve Plan GenT ödülü (Comunitat Valenciana, CDEI-05/20-C) ve FMN tarafından Valencian Generalitat ACIF/2016/020 bölgesel ValI +D öğrenciliği tarafından desteklendi. RP, Prof. Ewa K. Paluch'a fon için teşekkür etmek istiyorum. Biz IN Hücre Analizörü platformu ile yardım için Dr Alicia Martínez-Romero (CIPF Sitometri hizmeti) teşekkür ederiz.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Referanslar
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır