JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
마우스에서 간세포 핵 Ploidy의 추정을 위한 situ 방법에서 높은 처리량
요약
우리는 유세포 측정을 필요로하지 않는 고정 / 저온 보존 조직 샘플 내에서 간세포 수와 핵 ploidy의 변화를 측정하기위한 강력하고 비용 효율적이며 유연한 방법을 제시합니다. 우리의 접근 방식은 간 손상과 질병의 진행을 추적하는 데 이상적인 간 세포학의 강력한 샘플 전체 서명을 제공합니다.
초록
간이 손상되면 간세포 수가 감소하고 세포 크기, 핵 크기 및 ploidy가 증가합니다. 담관iocytes, myofibroblasts, 전구체 및 선동적인 세포와 같은 비 parenchymal 세포의 확장은 또한 만성 간 손상, 조직 개조 및 질병 진행을 표시합니다. 이 프로토콜에서는 부상, 만성 질환 및 암과 관련된 간 세포 구성의 변화를 계산하기 위한 간단한 높은 처리량 접근법을 설명합니다. 우리는 2 차원 (2D) 조직 섹션에서 추출 한 정보가 샘플 내에서 간세포 핵 ploidy를 정량화하고 교정하는 데 사용할 수있는 방법을 보여주고 사용자가 내부에서 간 내에서 특정 ploidy 하위 세트를 찾을 수 있습니다. 우리의 방법은 고정 / 냉동 간 재료, 기본 면역 세포 화학 시약 및 표준 고함량 이미징 플랫폼에 대한 액세스를 필요로한다. 그것은 표준 유동 세포 분석 기술에 대한 강력한 대안으로, 갓 수집 된 조직의 중단, 공간 정보 손실 및 잠재적 인 분리 편향을 필요로합니다.
서문
포유류 간에서 간세포는 이핵세포를 생성하기 위하여 실속된 사이토카인시스를 겪을 수 있고, DNA endoreplicai는 최대 16N DNA 내용을 포함하는 polyploid 핵을 생성하기 위하여. 전반적인 세포 및 핵 ploidy 증가 출생 후 개발 하는 동안, 노화 및 다양 한 세포 스트레스에 대 한 응답1. polyploidization의 과정은 그것의 정확한 생물학 기능이 불분명하게 남아 있더라도, 동적이고 뒤집을 수 있는2입니다3. 증가된 ploidy는 감소된 증식 용량 과 연관됩니다4,유전 적 다양성2,만성 상해에 적응5 및 암 보호6. 간세포 계략 변경은 변경 된 circadian 리듬7의결과로 발생 , 및 weaning8. 가장 주목할 만한, 간계 프로필은 상해 및 질병9에의해 변경되고, 설득력 있는 증거는 증가된 ≥8N 핵 또는 2N 간세포의 손실과 같은 특정 ploidy 변경이, 비 알콜 성 지방 간 질환 (NAFLD) 진행을 추적하기 위한 유용한 서명을 제공한다는 것을 건의합니다3,,10,또는 바이러스 감염의 차동 충격11.
일반적으로 간 손상 및 재생은 간세포 세포 크기 증가 및 핵영역(12)과연관되며, 간세포의 전체 수 감소와 함께, 특히 2N DNA함량이 10,,11인자. 간에서 의 자렌성 손상은 또한 자주 비 parenchymal 세포의 확장을 동반 (NPC), 기질 근섬유 아세포를 포함, 염증 성 세포와 이중 성 간 전구 세포. 또한 NPC의 변화를 고려하면서, 실치 세포 수와 핵 ploidy의 정량적 세포학적 프로파일을 제공하는 높은 처리량 방법은, 따라서 부상과 질병 동안 간 반응을 추적하는 연구 및 임상 도구로 상당한 잠재력을 가지고있다. 간세포암의 인간 샘플에서 계략 스펙트럼의 최근 의 결정적 스펙트럼분석은 또한 핵 계략이 종양 내에서 극적으로 증가하고 TP5313의감소된 분화 및 손실을 가진 더 공격적인 종양 특수형에서 구체적으로 증폭된다는 것을 보여준다. 따라서, 핵 ploidy의 정량적인 평가에 있는 방법론적 진보가 간암의 미래 예후 프로파일링에서 도움이 될 것이라는 강한 가능성이 있습니다.
이 프로토콜에서는, 마우스 간 조직 단면의 비교 분석을 위한 유연한 고처리량 방법론이 설명되며, 이는 간세포 수의 상세한 세포측정 프로파일링, NPC 반응 및 핵 경화추정을 위한 내부적으로 교정된 방법을제공한다(도 1). 간세포는 핵 크기 및 핵 모모시스템의 특성화 이전에 간세포 핵 인자 4 알파(HNF4α) 면역 라벨링에 의해 NPC와 구별된다. "최소 DNA 함량"은 평균 Hoechst 33342 강도(DNA 밀도에 대한 프록시)를 보간된 3차원(3D) 핵 부피를 통합하여 모든 원형 핵 마스크에 대해 추정됩니다. 간세포 최소한의 DNA 함량은 핵 ploidy 단면도를 생성하기 위하여 NPC를 사용하여 그 때 보정됩니다.
이미지 수집, 핵 세분화 및 이미지 분석은 고함량 이미징을 사용하여 수행되므로 수만 개의 세포를 포함하는 2차원(2D) 간 절개의 넓은 영역을 선별할 수 있습니다. 모든 원형 간세포 핵에 대한 샘플 전체 의 ploidy 프로파일을 생성하기 위해 고함량 이미지 분석 데이터의 자동 사후 처리를 위해 사용자 정의 작성 프로그램이 제공됩니다. 이것은 입체 이미지 분석 (SIA)10,,11,,14,,15에기초하여 핵 ploidy를 계산하는 소프트웨어를 다운로드 무료로 사용하여 수행됩니다. SIA 방법론은 이전에 원형 핵 형태와 핵 크기와 DNA 함량 사이의 단조로운 관계를 가정하여 간(14)에서간세포 핵 ploidy를 추정하기위한 정확하고 힘들기는하지만, 방법, 정확하고 힘들기는하지만 유세포 측정에 의해 검증되었습니다. 이 프로토콜에서, 두 핵 매개 변수는 핵 morphometry 및 Hoechst 의 평가에 의해 측정 된다 33342 라벨링. 각 핵 마스크에 대한 "최소 DNA 함량"의 계산은 알려진 2-4N DNA 함량을 가지고 따라서 유용한 내부 제어 역할을 NPC를 사용하여 간세포 핵 ploidy의 교정에 의해 선행된다.
종래의 유세포분석방법(16)에 비해 설명된 접근법은 간세포 핵 계략을 현장에서 평가할 수 있게 하고 결과를 편향시키고 표준화하기 어려운 신선한 조직 또는 분해 방법에 대한 접근을 필요로 하지 않는다. 모든 SIA 기반 접근법과 마찬가지로, 핵 계략 하위 클래스 >2N은 적도 평면 외부의 더 큰 핵의 단면화로 인해 2D 샘플링에 의해 과소 대표됩니다. 조직 전체 ploidy 단면도는 또한 모든 원형 간세포 핵 마스크에 대한 최소 DNA 함량을 설명하고, 동일한 ploidy의 두 개의 이산 ("비 접촉") 핵이 있는 단핵 간세포와 이핵세포 를 직접 구별하지 않습니다. 그러나, 이 프로토콜의 단순성은 핵간 간격 또는 세포 경계 분석과 같은 추가 파라미터를 고려하여 적응될 수 있게 하며, 이는 세포 계략에 대한 보다 상세한 평가를 제공하는 이핵전지의 식별을 용이하게 할 것이다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
모든 동물 실험은 이전에 CIPF 윤리위원회에 의해 승인되었습니다. 생쥐는 실험동물 사육자, 사용자 및 공급 센터로 등록된 센트로 데 Investigación Príncipe 펠리페(스페인 발렌시아)에 병원균이 없는 시설에 보관되었습니다( reg. no. ES 46 250 0001 002) 현재 적용되는 유럽 및 스페인 동물 복지 규정(RD 53/2013)에 따라.
1. 조직 수확 및 샘플 준비
참고 :이 프로토콜은 사전 고정 또는 동결 보존없이 조직을 동결하는 방법을 설명합니다. 이전에 고정/저온 보존된 시료의 경우 섹션 2로 진행하고 3.1 단계를 생략합니다. 모든 분석은 12-16 주 세 성인 여성 C57BL /6 마우스를 사용하여 수행되었습니다.
- 펜타닐/펜토바르비탈 복강 내 주사로 동물을 희생하고 자궁 경부 탈구를 하십시오. 마우스가 복부 쪽을 위로 향하게하면, 복강을 열고 핀셋으로 피부를 잡고 수술 용 가위를 사용하여 흉골의 기저부로 수직 절개를 수행하여 간을 노출시면 됩니다.
- 조심스럽게 미세 핀셋을 사용하여 담낭을 제거하고 간을 해부하고 인산완충 식염수 (PBS)로 채워진 10cm 페트리 접시 접시에 선택한 간 소엽을 헹구습니다.
참고 : 각 동물에 대해 동일한 간 엽을 비교하는 것이 좋습니다.이 경우 중간 엽이 사용되었습니다. - 실온(RT)에서 최적의 절삭 온도(OCT) 매개체로 라벨이 부착된 크라이오졸드를 채웁니다. OCT 거품을 피하십시오. 나타나면 바늘이나 파이펫 팁을 사용하여 금형 가장자리로 밀어 넣습니다.
- 가득 찬 OCT cryomold에 간 소엽을 넣고 즉시 드라이 아이스에 두어 급속한 동결을 보장하십시오. 냉동 절편이 될 때까지 -80 °C에서 저도졸을 저장합니다.
2. 냉동 부속
- 조직 저하를 피하기 위해 드라이 아이스에 저울을 운반하십시오. 저온 절제 하기 전에 20 분 동안 -20°C로 설정 된 저온 가트 내부평형.
- 플라스틱 크라이오졸드의 기저에 압력을 가하여 샘플을 배출합니다. RT에서 따뜻한 샘플 디스크에 액체 OCT를 적용, 저온 유지에 위치하고 OCT 임베디드 간 샘플을 부착. 부드러운 압력을 가하고 샘플이 디스크에 달라 붙도록 OCT가 동결될 때까지 3분 동안 기다립니다.
참고: 조직 저하를 피하기 위해 가능한 한 손가락으로 샘플을 취급하지 마십시오. - 샘플을 저온 해제의 팔에 고정하고 시료의 가장자리가 저온 블레이드와 평행하게 되도록 방향을 조정합니다. 조직에 도달 할 때까지 샘플로 잘라.
- 6 μm 두께로 샘플을 단면합니다. 표지가 있는 폴리아미드 코팅 슬라이드를 샘플 위에 5s로 놓아 샘플이 슬라이드에 달라붙도록 합니다. RT에서 3-5분 동안 슬라이드를 놓은 다음 최상의 결과를 얻으려면 섹션 3으로 직접 진행합니다.
참고: 여러 개의 신선 냉동 시료를 처리하기 위해 모든 시료가 처리될 때까지 드라이 아이스의 슬라이드 박스에 슬라이드를 일시적으로 저장하여 재현 가능한 결과를 얻을 수 있습니다. 이 방법을 사용하는 경우 섹션 3으로 진행하기 전에 모든 슬라이드가 RT와 평형을 사용할 수 있습니다. 포르말린 고정 파라핀 임베디드(FFPE) 샘플을 사용할 수 있지만, 배경 자동형광은 이 방법에 의해 증가된다. FFPE 샘플에서 진행하려면, 4 μm에서 섹션. 폴리 아미드 처리 슬라이드에 40 °C 수조에서 섹션을 잡아 마운트. 60°C에서 1시간 동안 열 슬라이드를 한 다음, 자일렌(x2), 에탄올 100% (x2), 96% (x2), 70% (x1) 및 dH2O (x1)를 포함하는 코플린 항아리에서 직렬 RT 와시 (5 분)에 의해 분리됩니다. 항원을 노출시키기 위해 90°C에서 20분 동안 구연산완충액에 슬라이드를 놓고 RT에서 PBS에서 슬라이드를 템퍼링하기 전에 3.2단계로 진행한다.
3. 형광 면역 라벨링
- RT에서 10분 동안 PBS에 4% 파라포름알데히드(PFA)의 1 mL을 적용하여 연기 후드의 티슈 부분을 고정시키고, 슬라이드를 PBS 채워진 코플린 항아리에 옮기고 부드러운 교반(반복 3x)을 사용하여 3분 동안 세척합니다.
참고 : 지금부터 면역 염색 과정이 끝날 때까지 시료의 건조를 피하십시오. - 각 조직 섹션 주변을 건조하고 소수성 펜을 사용하여 둘러싸십시오. RT에서 15 분 동안 PBS에서 0.5 % nonionic 계면 활성제 (즉, 트리톤 X-100)로 투과하십시오. 그런 다음 부드러운 교반 (반복 2x)을 사용하여 3 분 동안 PBS 충전 코플린 항아리에서 씻어.
- 1% 소 혈청 알부민(BSA), 5% 말 혈청, 0.2% nonionic 계면활성제의 여과용액을 이용하여 PBS(RT에서 적어도 1시간).
- 어두운 습한 염색 챔버에서 4 °C에서 밤에 차단 완충액으로 희석된 1차 HNF4α 항체와 배양합니다(항체 및 특이적 희석에 대한 재료 표 참조).
- 슬라이드를 PBS 가득 코플린 항아리에 넣고 부드러운 교반 (반복 4x)을 사용하여 3 분 동안 씻으십시오.
- 알렉사-488 컨쥬게이트 이차 항체와 Hoechst를 여과된 1% BSA 및 0.2% nonionic 계면활성제에서 어두운 습습 염색 챔버에서 RT에서 2시간 동안 PBS에서 희석한 배양(항체 및 특이적 희석에 대한 재료 표 참조).
- 슬라이드를 PBS 가득 코플린 항아리에 넣고 부드러운 교반 (반복 4x)을 사용하여 3 분 동안 씻으십시오. 부드러운 교반(반복 2x)을 사용하여 ddH2O로3분 동안 세척합니다.
- 커버슬립(24 x 60mm)에 형광 장착 매체 2방울을 떨어뜨리고 슬라이드를 그 위에 놓아 부드러운 압력을 가하여 거품을 제거합니다. 장기간 보관을 위해 뚜렷한 매니큐어로 가장자리에 덮개를 밀봉하고 4 °C의 어둠 속에서 보관하십시오.
- 진행하기 전에, 좋은 고정 및 면역 라벨을 보장하기 위해 기존의 형광 현미경을 사용하여 슬라이드를 확인합니다.
참고: 예상 결과는 그림 2A, B를 참조하십시오.
4. 형광 이미지 획득
참고: 이 단계에서는 자동 형광 이미지 수집을 지원하는 고함량 이미징플랫폼(재료 표)이필요합니다.
- 이미징 시스템을 켜고 새 수집 프로토콜을 엽니다.
- 10x 목표를 선택하고 시야 면적을 기록합니다(이 경우 0.6mm2).
- 적절한 여기 및 방출 필터를 사용하여 형광 이미지를 획득하도록 매개변수를 설정합니다(단계 3.6). Hoechst 및 Alexa-488의 경우 각각 390/18 및 438/24 nm 여기 및 432.5/48 및 475/24 nm 방출이 있는 "DAPI" 및 "GFP" 채널을 선택합니다.
- 샘플에 초점을 맞추고 신호 강도가 포화되지 않았는지 확인합니다. 모든 이미지에 대해 동일한 노출 시간으로 이미지 캡처가 수행되도록 하거나 노출 시간 동안 형광 강도가 보정되는 시스템을 사용합니다.
- 샘플을 스캔하고 조직 섹션의 완전한 커버리지를 얻기 위해 충분한 이미지를 획득합니다 (샘플 크기에 따라 약 20-50 개의 시야).
- 이미지 데이터베이스를 검토하고 (i) 제대로 초점을 맞추지 않은 필드, (ii) 각 조직 섹션의 경계에 있는 필드(편향된 세포 밀도 계산을 피하기 위해) 및 (iii) 조직 섹션의 접힌/물리적으로 손상된 영역을 포함하는 필드를 수동으로 제거합니다.
5. 자동 형광 이미지 분석
참고: 이 단계는 적절한 이미지 분석소프트웨어(1)405 nm(핵 분할)에서 이미지 내에서 Hoechst 표지 핵을 자동으로 식별하고, (2) 평균 Hoechst 핵 강도 및 모퍼링을 평가하고, (3) 임계값 분석을 통해 488 nm(HNF4α)에서 핵 형광의 +/-상태를 결정할 수 있어야 합니다. 핵과 HNF4α+/-상태가 최적으로 게이트되도록 프로그램 내에서 세분화 및 임계값 매개변수를 시각적으로 평가하고 조정하기 위해 일부 기본 작업자 교육/전문 지식이필요합니다(그림 2).
- 이미지 분석 소프트웨어에서, 4.5단계에서 Hoechst(405 nm) 및 HNF4α(488 nm) 이미지를 포함하는 수집 파일을 열고, 새로운 분석 프로토콜을 생성한다.
- 핵 분절(Hoechst, 405 nm) 및 간세포/NPC 임계값 분석(HNF4α, 488 nm)에 사용할 파장을 정의합니다.
- 소프트웨어의 핵 세분화 매개변수(예: "최소 핵 영역" 및 핵 탐지 "감도")를 조정하여 핵이 최적으로 분리되도록 합니다.
참고 : 간세포의 좋은 분절은 NPC보다 우선 순위를 매어야합니다.2 Sinusoidal 내피의 것과 같은 NPC 핵은, 모양에 있는 평평하게/타원형 또는 불규칙하고 일반적으로 간세포의 그것 보다는 더 작고 더 밀접하게 포장됩니다 (수족간 크기 범위: 30−43 μm2). 마우스 간, 최소 핵 영역 ≥23 μm2 및 검출 "감도" 65%를 사용하였다(예상 결과에 대한 도 2C,D 참조). 감도는 픽셀 클러스터가 강도에 따라 개별 핵으로 인식되는 방식을 결정하고 자동화된 이미지 분석을 진행하기 전에 사용자가 설정한 각 샘플에 대해 경험적으로 테스트해야 합니다. - 488 nm에서 임계 강도를 수정하여 간세포(HNF4α+) 및 비자말 세포(HNF4α-)의 최적의 게이팅을 보장합니다.
참고: 예상 결과는 그림 2C, D를 참조하십시오. 임계값 강도의 값은 상대적이며 레이저 강도와 같은 염색 효율 및 수집 설정에 따라 달라집니다. 따라서 사용자가 표준화해야 합니다. 내피 세포 및 회문 NPC와 같은 공지된 HNF4α-세포를 내부 음성 대조군 및 이중 핵간세포 핵을 염색에 대한 양성 기준으로 사용한다. 전체 데이터 집합에 분석 매개변수를 적용하기 전에 적은 수의 이미지를 사용하여 분석 매개변수를 테스트하여 핵 세분화 및 강도 임계값 분리가 양호하도록 합니다. - 정량화할 다음 핵 파라미터를 선택한다: (1) Hoechst 염색(μm2),(2) 평균 핵 회흐트 강도(RU), (3) 핵 연신률(핵의 긴 축에 대한 핵의 짧은 축의 평균 비) 중심 대칭 [길지 않는] 객체의 값이 1, (4) Nuc 1/(폼 팩터)인 경우 둘레 2/(4π x 영역)로 계산된 핵 "진도" 인덱스를 의미합니다. 값범위는 1에서 무한대까지이며, 여기서 1은 완벽한 원, (5) HNF4α 상태(양수-1 또는 음수-0) 및 (6) "무게 중심"(cg)을 기반으로 하는 핵 x/y 좌표, 하위 픽셀 정밀도로 그레이스케일 이미지에서 물체의 중심을 찾는 방법입니다.
- 모든 샘플 데이터 집합에 대한 분석을 실행하고 5.5단계에서 스프레드시트 소프트웨어로 수치 데이터를 내보냅니다.
6. 데이터 분석
참고: 데이터 분석 단계는 표준 스프레드시트 소프트웨어를 사용하여 수행할 수 있습니다.
- 간세포 및 비 간세포 세포 번호를 계산합니다.
- 각 샘플에 대해 분석된 간 섹션의 총 면적을 뷰 필드 면적에 곱하여 계산합니다(단계 4.2).

- 각 간 섹션에 대해 생성된 스프레드시트 파일로 작업하여 HNF4α+ 핵만 선택하여 데이터를 필터링합니다. 분석된 HNF4α+ 핵의 총 수를 계산하고 이를 분석된 총 면적으로 나누어 각 샘플에 대한 평균 간세포 밀도를얻었다(도 2F).

- HNF4α-셀에 대한 스프레드시트를 필터링하여 비실화 세포에 대해 동일한 계산을 수행합니다(그림2E).
- 각 샘플에 대해 분석된 간 섹션의 총 면적을 뷰 필드 면적에 곱하여 계산합니다(단계 4.2).
- 간세포 핵 크기 분포를 계산합니다.
- 스프레드시트 소프트웨어를 사용하여 데이터를 필터링하여 HNF4α+ 핵만 선택합니다.
- 핵 영역의 값을 히스토그램으로 플롯합니다(그림2G). 쓰레기통 너비를 5 μm2로설정합니다.
참고: 주파수 값은 6.1.1단계별로2면적(핵/mm2)에 대해 수정할 수 있습니다.
- 간세포 핵 ploidy 분석을 수행합니다.
참고: 5.6단계의 스프레드시트 데이터는 각 샘플에 대한 핵 프로파일을 생성하는 데 사용됩니다. 이 프로세스는 자동화되었으며 지원 정보 및 데모 데이터 집합을 사용하여 자유롭게 다운로드할 수 있는 사용자 지정 작성 소프트웨어를 사용하여 수행할 수 있습니다.https://github.com/lukeynoon(를 참조하십시오.추가 파일). 소스 코드는 방법론을 적용하려는 사용자를 위해 제공됩니다. 설치 및 사용에 대한 지침과 함께 알고리즘에 대한 설명은 아래에 설명되어 있습니다. 이 프로그램은 스프레드시트 데이터를 사용하여 간세포 핵을 두 그룹으로 자동으로 분리합니다. (1) "단순" 원형 핵및 (2) "복잡한" 비원형 핵을 가진 사람들은 2c ploidy를 가진 이핵포세포를 대표한다. 최소한의 핵 DNA 함량 (핵 영역과 DNA 밀도의 기능)은 모든 "간단한"핵에 대해 다음에 계산됩니다. 후속 단계는 HNF4α+핵계를 HNF4α-핵을 알려진 2−4N 내부 대조군으로 사용하여 HNF4α+ 간세포 핵 계략을 자동으로 교정합니다.- 소프트웨어를 다운로드하여 설치합니다.
- 패키지 된 응용 프로그램을 다운로드: https://github.com/lukeynoon
- MATLAB을 시작합니다. 도구 트립의 APP 탭으로 이동하여 앱 설치를 클릭하고"Ploidy_Application.mlappinstall"라는 다운로드 한 응용 프로그램을 엽니다. 성공적인 설치를 확인하는 메시지가 나타납니다.
참고 : 응용 프로그램은 이제 사용할 준비가되었으며 도구 트립의 APP 탭에 남아 있습니다.
- 입력 데이터의 서식을 지정합니다.
참고: 자동화된 핵 분석 전에 는 고콘텐츠 이미징 데이터(단계 5.6)가 포함된 모든 스프레드시트 파일을 다음 지침에 따라 저장하고 서식을 지정해야 합니다.- 내보낸 각 데이터 파일(. XLS 97-2004 통합 문서)단계 5.6에서, 열에 명시된 ploidy 분석에 필요한 모든 데이터를 포함하는 "셀 측정"이라는 시트를포함한다(그림 3A). 분석 메서드는 이러한 이름을 검색하여 올바른 열 데이터를 찾기 때문에 열 헤더 이름을 포함한 스프레드시트 레이아웃이 그림 3A의레이아웃과 변경되지 않도록 합니다(참조를 위해 보충 파일의 데모 데이터 집합 참조). 예를 들어, 고함량 이미지 분석 소프트웨어가 "광속"열(그림 3A)을생성하지 않는 경우, 동일한 위치에 "광속" 컬럼을 수동으로 삽입하고, 즉 K 열을 0으로 채웁니다.
- 각 실험 조건(예: "부상-d14")에 대해 2−4N 핵 쟁기 교정에 대한 내부 제어를 계산하는 데 사용되는 제어 데이터 세트를 제공합니다(단계 6.3.4.3). 여기에서, 처리되지 않은 성인 littermates에서 간 견본을 선택하십시오 ("Control-d0"; 그림 3B-D).
- 생물학적 복제(조건별)의 경우 각 스프레드시트를 자체 폴더에 저장합니다(그림 3B와같이). 폴더 접두사 이름을 증분(예: "Sample1, Sample1, Sample2, Sample3... SampleN", 내에 포함 된 파일 이름에 따라. 따라서 모든 데이터 집합 폴더(예: "Control-d0")에는 동일한 이름의 스프레드시트 파일이 포함된 일련의 하위 폴더("Sample1", "Sample2" 등)가 포함되어야 합니다.
- 응용 프로그램을 실행합니다.
- MATLAB 내에서 도구 트립의 내 앱 탭내 아이콘을 클릭하여 "Ploidy_Application"을시작합니다(그림 3C). Ploidy_Application 그래픽 사용자 인터페이스(GUI)가나타납니다(그림 3C).
- 경로 제어 데이터 단추를 클릭하여 제어 데이터가 복제되는 폴더(예: "Control-d0")로 이동합니다. 이 데이터 경로는 인터페이스(예: /Users/Desktop/Control-d0)에나타납니다.
- 다음으로 "폴더 접두사"에서 출력 파일에 부여할 이름을 입력합니다(예: "샘플").
참고: 폴더와 파일 이름이 점진적으로 명명된 경우 이 접두사는 모든 텍스트로 변경할 수 있습니다. - 다른 데이터 로가는 경로를 클릭하고 비교 데이터가 복제되는 폴더(예: "부상-d14")로 이동합니다. 이 데이터 경로는 인터페이스에 나타납니다(예: /Users/Desktop/부상-d14).
- 실행을 클릭합니다!. 분석이 완료되면 상태 표시줄에 "분석 완료!.."가 표시됩니다.
참고 : 응용 프로그램은, 각 샘플에 대해, ≤2n, 2n-4n, 4n-8n 및 8n +로 "간단한"핵의 계층화를 보고하고 절대 수의 관점에서 총의 백분율로(그림 3D). 이 파일은"Count_2n.txt","Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt"와같이 각 샘플 폴더에 자동으로 저장됩니다. Ploidy_Application "Ploidy_All_Hepatocytes.txt"와 "Ploidy_NonHepatocytes.txt"에서"간단한"간세포 및 비 간세포 핵에 대한 모든 개별 ploidy 추정치의 각 샘플에 대한 목록을 자동으로 저장합니다. 제어 데이터 집합의 경우 이 방법은 "Normalised_Thresholds_Control"라는파일에 ploidy의 계층화를 위해 계산된 최소 DNA 함량 임계값(단계 6.3.4.3.7 참조)을 저장합니다. 마지막으로 응용 프로그램은 컨트롤과 "요약"이라고 불리는 선택한 비교 조건 데이터에 대한 폴더를 생성합니다. 이 폴더에는 제공된 모든 샘플의 평균을 포함하는 두 개의 하위 폴더인 "Ploidy" 및 "계층화"가 포함되어있습니다(그림 3D).
- 방법론에 대한 설명입니다.
참고: 다음 섹션에서는 핵 Ploidy 분석 소프트웨어에서 사용하는 방법론을 자세히 설명합니다. 사용자가 응용 프로그램을 사용하지 않기로 선택한 경우 스프레드시트 소프트웨어를 사용하여 핵 프로파일을 수동으로 계산할 수 있습니다.- 핵을 핵모집에 따라 "단순" 또는 "복잡한" 것으로 분리한다.
- 모든 핵에 대해 "원형 지수"를 계산하며, 핵 "신장 률"을 "Nuc 1/(폼 팩터)"로 나눈 값으로 정의하며, 여기서 값은 1.0으로 완벽한 원을 나타냅니다.
참고: "핵 신장"과 "Nuc 1/(폼 팩터)"는 상호 보완적이고 겹치지 않는 형태 측정 기준을 평가하는 객체의 "원형"의 두 가지 개별 측정값입니다. 전자는 객체의 길고 짧은 축을 측정하고 후자는 객체의 둘레 길이를 해당 영역의 축과 비교합니다. 이 프로토콜에 사용되는 핵 순환성의 정의를 강화하기 위해 이 두 측정은 단일 "원형 지수"로 결합되었습니다. 기재된 방법론을 이용하여 핵쟁이를 추정하기 위한 이전의 접근법은 핵신장만을 사용하였다 17. 이 접근법을 사용하여 수용 가능한 결과를 얻었지만, 저자는 복합 "원형 지수"가 단일 핵 및 이핵성 간세포로부터 수동으로 선택된 핵의 차별을 향상시킨다는 것을 관찰했습니다(데이터는 표시되지 않음). - 원형 인덱스 ≤ 0.8을 "복합"으로 분류하고 그 > 0.8을 "단순"으로 분류합니다.
- 모든 핵에 대해 "원형 지수"를 계산하며, 핵 "신장 률"을 "Nuc 1/(폼 팩터)"로 나눈 값으로 정의하며, 여기서 값은 1.0으로 완벽한 원을 나타냅니다.
- 모든 "단순" 핵에 대해 "최소" DNA 함량(m)을 추정합니다.
- 수식을 사용하여 핵 반지름(r)을 계산합니다.

- 구 수식의 볼륨을 사용하여 핵 체적(v)을 계산합니다.
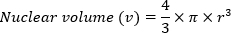
- 수식을 사용하여 최소 DNA 함량(m)에 대한 상대값을 생성합니다.

- 수식을 사용하여 핵 반지름(r)을 계산합니다.
- NPC(HNF4α-) 핵을 내부 2−4N 제어로 사용하여 데이터 세트를 교정합니다.
참고: NPC는 세포 주기 상태에 따라 2-4N DNA 함량을 갖습니다. 따라서, NPC의 평균 값 "최소" DNA 함량 (NPCm)은부상과 함께 증가한다(도 4A). 교정 오차는 4c 임계값을 나타내는 NPCm의 상한을 설정하여 최소화된다(도4B).- 스프레드시트 내에서 모드의 1표준 편차(SD) 내에 있는 "m"에 대한 값을 가진 NPC 핵만 선택합니다(가능한 세분화 오류로 인한 노이즈를 필터링함).
- 이 필터링된 범위 내에서 핵 영역과 그에 상응하는 평균 Hoechst 강도를 검사합니다(그림4C).
- 최대 핵 Hoechst 강도로 이 필터링된 범위 내의 가장 작은 핵 영역을 추정합니다(즉, 그림4C의빨간색 원으로 표시된 대로 곡선의 선이 필터링된 데이터 집합에서 방향을 변경하는 지점). 이 값은 4c 핵의 샘플링이 2c 핵을 지배하는 2N−4N 과도 상태(t)를 나타내며, 그 결과 평균 Hoechst 강도의 최대값을 생성합니다.
참고: 이 값은 소프트웨어에 의해 자동으로 결정됩니다. 그러나 스프레드시트 사용자는 이 점을 전환 크기로 수동으로 선택할 수 있습니다. - 6.3.4.2 단계 다음에 이 과도크기(t)로표현되는 최소 DNA 함량을 계산합니다.
- NPCm 데이터 집합의 4N 숄더를 추정하려면 tm값에 1 SD를 추가합니다. 생성된숫자(도 4B)는핵 계략 계층화(S4c)에사용되는 NPC 최소 DNA 함량의 상한을 설명한다.
- 모든 "제어" 샘플에 대해 6.3.4.3.1-6.3.4.3.5 단계를 반복합니다.
참고: 예를 들어, 그림 3에서,손상되지 않은 대조군 간("Control-d0")은 대조군 조건으로 사용된다. - "제어" 샘플에 대한 평균 4c 계층화 임계값(S4c)을계산하고 이를 사용하여 최소 DNA 함량(m)에 대한 2c(S2c)및8c(S 8c)경계를 추정합니다. 계층화 임계값은 소프트웨어에 의해 자동으로 생성되고 저장됩니다(6.3.3.3 단계).
참고: 연구 설계에 따라, 평균 계층화 임계값은 각 조건 또는 특정 조건(예: 건강한 대조간)에 대해 계산될 수 있습니다. 그러나 핵 Ploidy 분석 소프트웨어는 2개의 파일 집합 중 하나가 상대적인 플로이디 값을 계산하기 위해 "제어"로 지정되도록 요구합니다. - 단계 6.3.4.3.7에서 생성된 S2c 값을 사용하여 모든 핵에 대한 플로이디 값을 계산합니다.

- 다음 기준에 따라 "단순" 간세포(HNF4α+) 핵을 2c/4c/8c/>8c 브래킷으로 계층화합니다: "2c" HNF4α+ = p ≤ 2; "4c" HNF4α+ = 2 < p ≤ 4; "8c" HNF4α+ = 4 < p ≤ 8; ">8c" HNF4α+ = 8 < p.
- ploidy 하위 그룹의 공간 패터닝을 재구성하려면 각 샘플 스프레드시트 내의 핵 데이터를 획득한 해당 필드에 따라 분리합니다. 그런 다음 연관된 핵 x/y 좌표를 사용하여 (5.5단계에서) 2D(그림5C)에서플롯 된 하위 그룹을 플롯합니다.Figure 5
- 핵을 핵모집에 따라 "단순" 또는 "복잡한" 것으로 분리한다.
- 소프트웨어를 다운로드하여 설치합니다.
Access restricted. Please log in or start a trial to view this content.
결과
이 방법은 0.1% 3,5-디에톡시카보닐-1,4-디하이드로콜린(DDC)17을함유하는 간독성 식이요법으로 0-21일 동안 동물에게 먹이를 주어 성인 마우스 간에서 담낭성 손상의 영향을 측정하는데 사용되어 왔다. 만성 DDC 공급 결과 간 세포 손상 증가 ploidy 및 NPC의 periportal 확장. 사용자는 마우스 균주와 연령에 따른 차이가 핵 계략에 존재할 수 있으며 모든 분석은 12-16?...
Access restricted. Please log in or start a trial to view this content.
토론
뮤린 간에서 조직 리모델링 및 간세포 핵 계략의 분석에 대한 높은 함량, 높은 처리량 접근법이 설명된다. 이 절차에 익숙해지면 사용자는 3-5일 동안 여러 샘플을 처리, 이미지 및 분석하여 간 건강의 상세한 서명을 제공하는 대규모 테스트 가능한 데이터 세트를 생성할 수 있습니다. 샘플 전처리 방법의 단순성, 분석된 많은 수의 세포 및 조직 영역(평균 14mm 2/샘플)을 감안할 때, 결과는 견고하고 ?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 것이 없다.
감사의 말
이 작품은 스페인 MINECO 정부 보조금 BFU2014-58686-P (LAN) 및 SAF-2017-84708-R (DJB)에 의해 지원되었다. LAN은 국가 MINECO 라몬 y 카잘 펠로우십 RYC-2012-11700 및 계획 GenT 상 (Comunitat 발렌시아나, CDEI-05/20-C) 및 발렌시아 일반 ACIF /2016/020의 지역 ValI + D 학생에 의해 FMN에 의해 지원되었다. RP는 에와 K. 팔루치 교수에게 자금 지원을 인정하고 싶습니다. 우리는 IN 세포 분석기 플랫폼에 도움을 박사 알리시아 마르티네스 - 로메로 (CIPF 세포 분석 서비스)에 감사드립니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
참고문헌
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유