Method Article
تطوير واختبار المقايسات الكمية الخاصة ب PCR لتطبيقات الحمض النووي البيئي
* These authors contributed equally
In This Article
Summary
تتطلب اختبارات الحمض النووي البيئية تصميما واختبارا وتحسينا وتحققا دقيقا قبل بدء جمع البيانات الميدانية. هنا، نقدم بروتوكولا لأخذ المستخدمين من خلال كل خطوة لتصميم فحص qPCR خاص بالأنواع والقائم على المسبار للكشف عن الحمض النووي للأنواع المستهدفة وتحديد كميتها من العينات البيئية.
Abstract
ويجري تطوير أساليب جديدة غير جراحية للكشف عن وجود الأنواع ورصده للمساعدة في إدارة مصائد الأسماك وحفظ الحياة البرية. استخدام عينات الحمض النووي البيئي (eDNA) للكشف عن macrobiota هي واحدة من هذه المجموعة من الأساليب التي سرعان ما أصبحت شعبية ويجري تنفيذها في برامج الإدارة الوطنية. وهنا نركز على تطوير الأنواع المحددة من الأنواع المحددة المحددة للمبارس الكمي PCR (qPCR) التطبيقات. استخدام qPCR المستندة إلى التحقيق يوفر خصوصية أكبر مما هو ممكن مع التمهيديات وحدها. وعلاوة على ذلك، فإن القدرة على تحديد كمية الحمض النووي في العينة يمكن أن تكون مفيدة في فهمنا لإيكولوجيا الحمض النووي الناسف للطبيعة وتفسير أنماط الكشف عن الحمض النووي الدينا في الميدان. ويلزم النظر بعناية في وضع واختبار هذه المقايسات لضمان حساسية وخصوصية الكشف عن الأنواع المستهدفة من عينة بيئية. وفي هذا البروتوكول، سنخصص الخطوات اللازمة لتصميم واختبار المقايسات القائمة على التحقيق للكشف عن الأنواع المستهدفة؛ بما في ذلك إنشاء قواعد بيانات تسلسل وتصميم المقايسة واختيار المقايسة والتحسين واختبار أداء الفحص والتحقق من صحة الحقل. اتباع هذه الخطوات سوف يساعد على تحقيق كفاءة وحساسة، و المقايسة المحددة التي يمكن استخدامها بثقة. نحن نثبت هذه العملية مع المقايسة لدينا مصممة لسكان mucket (Actinonaias ligamentina)، وهو نوع بلح البحر في المياه العذبة وجدت في نهر كلينش، الولايات المتحدة الأمريكية.
Introduction
أصبح الباحثون والمديرون مهتمين بشكل متزايد باستخدام مقايسات الحمض النووي البيئية للكشف عن الأنواع. على مدى ثلاثة عقود، تم استخدام PCR الكمي أو الحقيقي (qPCR/rtPCR) في العديد من المجالات للكشف عن تسلسل محددة والكمي من الأحماض النووية1،2. وفي مجال بحوث ال eDNA الجديد نسبيا، أصبح استخدام هذه المقايسات ذات المنحنى القياسي للقياس الكمي لنسخ الحمض النووي المستهدف لكل حجم أو وزن لعينة من الحمض النووي الدينا ممارسة روتينية الآن. يتم استهداف تسلسل الحمض النووي الميتوكوندريا بشكل عام في مقايسات eDNA لأن جينوم الميتوكوندريا موجود في آلاف النسخ لكل خلية ، ولكن من الممكن أيضا إجراء فحوصات للحمض النووي أو تسلسل الحمض النووي الريبي. من الضروري أن نفهم أن المقايسات المنشورة لعينات eDNA ليست دائما متساوية في الأداء. قد تختلف موثوقية المقايسة في الكشف عن الحمض النووي للأنواع المستهدفة فقط (أي التحديد) والكشف عن الكميات المنخفضة من الحمض النووي المستهدف (أي الحساسية) بشكل كبير بسبب الاختلافات في كيفية تصميم المقايسة واختيارها وتحسينها واختبارها. التقارير المقاييس الكمية لأداء المقايسة قد تم تجاهلها في السابق إلى حد كبير، ولكن في الآونة الأخيرة معايير لتحسين الشفافية في تطوير المقايسة الناشئة3،4،5،6،7،8.
تحسين والإبلاغ عن المساعدات أداء المقايسة في تصميم الدراسة وتفسير نتائج المسح eDNA. ويمكن أن تؤدي المقايسات التي تتفاعل مع الحمض النووي للأنواع غير المستهدفة إلى اكتشافات إيجابية زائفة، في حين أن المقايسات ذات الحساسية الضعيفة قد تفشل في الكشف عن الحمض النووي للأنواع المستهدفة حتى عندما يكون موجودا في العينة (السلبيات الكاذبة). وسيساعد فهم حساسية المقايسة والانتقائية على توجيه جهود أخذ العينات اللازمة للكشف عن الأنواع النادرة. نظرا لوجود العديد من المصادر الطبيعية للتباين في eDNA ، يجب أن تحد الدراسات من مصادر الاختلاف التي يمكن التحكم فيها قدر الإمكان ، بما في ذلك التحسين الكامل وتميز مقايسة eDNA3.
الظروف التي تؤثر بشكل مباشر على خصوصية أو حساسية المقايسة ستغير أداء المقايسة. ويمكن أن يحدث ذلك في ظروف مختبرية مختلفة (أي الكواشف المختلفة والمستخدمين والآلات وما إلى ذلك). لذلك، يجب إعادة النظر في هذا البروتوكول عند تطبيق المقايسة في ظل شروط جديدة. حتى المقايسات التي تتميز جيدا في الأدب ينبغي اختبارها وتحسينها عند اعتمادها من قبل مختبر جديد أو عند استخدام الكواشف المختلفة (على سبيل المثال، حل المزيج الرئيسي)5،9. وقد تتغير خصوصية المقايسة عند تطبيقها على منطقة جغرافية مختلفة، لأن المقايسة تطبق على عينات من مجتمع حيوي جديد قد يشمل أنواعا غير مستهدفة لم يتم اختبار المقايسة ضدها، وقد يحدث تباين وراثي في الأنواع المستهدفة. ومرة أخرى، ينبغي إعادة تقييم المقايسة عند استخدامها في موقع جديد. وتختلف الظروف الميدانية عن الظروف المختبرية لأن مثبطات الفينول الخماسي الكلور في الميدان أكثر عرضة للتواجد في العينات. مثبطات PCR تؤثر بشكل مباشر على رد فعل التضخيم وبالتالي تؤثر على أداء المقايسة. لهذا السبب، مطلوب رقابة إيجابية داخلية عند تطوير اختبار eDNA.
وأخيرا، يمكن أن تؤثر الظروف البيئية في الميدان على جزيئات الحمض النووي للأنواع المستهدفة وقبضها من خلال تدهور الحمض النووي ونقله والاحتفاظ به. وعلاوة على ذلك، تختلف البروتوكولات المختلفة لجمع الحمض النووي واستخراجه من حيث كفاءتها وقدرتها على الاحتفاظ بالحمض النووي. ومع ذلك، من المهم ملاحظة أن هذه العمليات تؤثر على إمكانية اكتشاف eDNA ولكن ليس أداء المقايسات الجزيئية. وبالتالي، فإن إمكانية اكتشاف الحمض النووي من الأنواع المستهدفة في عينات الحقل هي وظيفة من الأداء التقني لكل من الفحص qPCR وكذلك الظروف الميدانية وبروتوكولات التجميع والتخزين والاستخراج. عند استخدام المقايسة ذات الخصائص الجيدة والأداء العالي ، يمكن للمستخدمين الشعور بالثقة في قدرات المقايسة . السماح للباحثين بالتركيز الآن على فهم عوامل الفحص الخارجية (أي المتغيرات البيئية والاختلافات في بروتوكولات الالتقاط أو الاستخراج) التي تؤثر على الكشف عن eDNA.
هنا نركز على وجه التحديد على الأداء الفني من خلال تقييم تصميم صارم والتحسين. نحن نثبت البروتوكول باستخدام الفحص القائم على التحقيق وضعت للكشف عن بلح البحر في المياه العذبة، وm mucket(Actinonaias ligamentina)،من المياه عينات في نهر كلينش، الولايات المتحدة الأمريكية. وفي الآونة الأخيرة، قدم تالينغر وآخرون (2020) مبادئ توجيهية للتحقق من صحة المقايسات المستهدفة ل eDNA. تصميم المقايسة بعد بروتوكولنا سيجلب المقايسة إلى مستوى Thalinger et al.s 4 بالإضافة إلى خطوة إضافية نحو المستوى 56. وعند هذه النقطة، سيتم تحسين الأداء التقني للمقاسين وسيكون جاهزا للاستخدام المنتظم في التطبيقات المختبرية والميدانية. ويمكن لمزيد من استخدام المقايسة في المختبر ، mesocosm ، والتجارب الميدانية ثم معالجة الأسئلة المتعلقة الكشف عن eDNA والعوامل التي تؤثر على detectability ، والخطوات النهائية للمستوى 5 التحقق من صحة6.
Protocol
1. إنشاء قاعدة بيانات تسلسلية لتسلسل الحمض النووي الميتوكوندريا من الأنواع المستهدفة وغير المستهدفة ذات الاهتمام
- تحديد السؤال والأهداف والنظام الذي يتم تناوله. تحديد الأنواع المستهدفة للكشف عن eDNA. تحديد النظام الجغرافي الذي سيتم فيه استخدام المقايسة. وضع قائمة من الأنواع ذات الاهتمام، بما في ذلك الأنواع المستهدفة، والأنواع الستاريك (التي تحدث بشكل مشترك) ضمن نفس التصنيف (عادة النظام أو مستوى الأسرة)، والأنواع allopatric ذات الصلة الوثيقة، تلك التي قد لا تكون في نفس الموقع الجغرافي كهدف(الشكل 1).
ملاحظة: هنا، تم استهداف مجموعات نهر كلينش من النوع A. ligamentina. - البحث وتحميل تسلسلات من مناطق الجينات متعددة للأنواع على القائمة من الخطوة 1. ويمكن استخدام قواعد بيانات تسلسل مثل NCBI (المركز الوطني لمعلومات التكنولوجيا الحيوية) ، BOLD (الباركود من قاعدة بيانات الحياة) ، EMBL (المختبر الأوروبي للبيولوجيا الجزيئية) وDDBJ (بنك بيانات الحمض النووي في اليابان). NCBI و EMBL و DDBJ جميع المعلومات تسلسل المشاركة.
- باستخدام قاعدة بيانات النيوكليوتيدات التابعة للهيئة الوطنية للبي، ابحث عن الكائن المستهدف (مثل أكتينوناياس اربيلينا) ومنطقةالجينات (مثل السيتوكروم c oxidase I (COI) أو NADH-dehydrogenase 1 (ND1)؛ مثال سلسلة البحث: أكتينوناياس ligamentina وND1)
- بعد ذلك، حدد كافة التسلسلات التي تطابق المواصفات وحدد إرسال إلى. اختر السجل الكامل، ملف وشكل التحميل إما GenBank أو FASTA ثم إنشاء ملف. يتم الآن حفظ هذه التسلسلات إلى الكمبيوتر.
- كرر هذه الخطوات لكافة الأنواع في القائمة المحددة في الخطوة 1. احتفظ بتسلسلات لكل منطقة جينية في ملف منفصل حيث سيتم تحليلها بشكل منفصل.
- تحميل جميع التسلسلات ذات الصلة (أو نسبة تمثيلية كبيرة من التسلسلات) للأنواع المستهدفة المحددة في الخطوة 1. تضمين المتغيرات الجغرافية إذا أمكن.
- كرر تسلسل البحث والتحميل للأنواع غير المستهدفة ذات الصلة والتكافلية من نفس المجموعة التصنيفية التي تم تحديدها في الخطوة 1 (على سبيل المثال، إذا كان النوع المستهدف هو متواليات التحميل(A. ligamentina)لجميع أنواع بلح البحر الأخرى في المياه العذبة في Family Unionidae التي تحدث في نظام الاهتمام).
- كرر البحث والتحميل عن الأنواع ذات الصلة الوثيقة ولكن allopatric (منفصلة جغرافيا) المدرجة في الخطوة 1.1.
ملاحظة: لن تكون جميع الأنواع (الأهداف وغير المستهدفة) متاحة في قواعد البيانات العامة. زيادة قاعدة البيانات المرجعية المحلية عن طريق تضخيم وتسلسل عينات تم التحقق منها تصنيفيا لأنواع ذات أهمية داخلية. إذا كان العمل مع الأنواع التي لديها تنوع وراثي عالي داخل الأنواع أو العمل في منطقة كبيرة جغرافيا حيث يمكن توقع المتغيرات الجغرافية، وجمع تسلسل من جميع أنحاء النطاق.
2. تصميم الفحص
- محاذاة تسلسلات من كل منطقة الجينات بشكل منفصل باستخدام برامج المحاذاة التي يمكن العثور عليها في مختلف تحرير التسلسل الجيني والبرامج المعلوماتية الحيوية. قم بهذا المحاذاة لكل منطقة من مناطق الجينات المختلفة.
- على سبيل المثال، باستخدام برنامج Geneious Prime (https://www.geneious.com) استيراد ملفات التسلسل التي تم تنزيلها إلى البرنامج.
- إنشاء مجلدات منفصلة لكل منطقة جينية.
- ضمن مجلد يحتوي على تسلسلات من منطقة جينية واحدة، حدد كافة التسلسلات.
- استخدم أداة المحاذاة المتعددة لإنشاء محاذاة النيوكليوتيدات للتسلسلات المحددة. قد يكون هناك العديد من الخيارات لنوع المحاذاة، وذلك باستخدام محاذاة جينيوس أو MUSCLE والمعلمات الافتراضية يعمل بشكل جيد.
- اختر المناطق الواعدة لتصميم المقايسة من خلال تصور بيانات التسلسل المنحازة. والمنطقة التي لديها الكثير من بيانات التسلسل المتاحة للأنواع ذات الاهتمام، متباعدة للغاية بين الأنواع، وتظهر تباينا منخفضا داخل الأنواع هي مرشح جيد. وهذا من شأنه أن يزيد من احتمال أن تكون اللتمهيدات والتحقيقات المصممة قادرة على التمييز بين الهدف والأنواع غير المستهدفة، مع ضمان أن المتغيرات داخل المحددة سوف تتضخم مع المقايسة.
- تصميم التمهيديات المقايسة والتحقيق.
- استخدم برنامج تصميم الفحص qPCR واتبع التعليمات. تم استخدام أداة PrimerQuest (https://www.idtdna.com/) IDT لتصميم 5 مجموعات من المقايسات qPCR هنا.
- لصق التسلسل المحدد في الخطوة 2.2 في مربع إدخال التسلسل. إذا كانت المحاذاة قد أنشأت مسافات، فاحذفها من التسلسل.
- حدد qPCR 2 التمهيديات + التحقيق في اختيار التصميم الخاص بك الخيار.
- قم بتنزيل المقايسات الموصى بها.
- نسخ التسلسلات من التمهيدي الأمامية من المقايسة الأولى والبحث عن هذا التسلسل التمهيدي في المحاذاة التي تم إنشاؤها في الخطوة 2.1.4. إذا كنت تستخدم Geneious Prime، فاستخدم أداة التعليق التوضيحي والتنبؤ لإضافة منطقة التمهيدي إلى المحاذاة. القيام بذلك لجميع مجموعات التمهيدي والتحقيق (الشكل 2).
- فحص هذه المناطق من المحاذاة للتباين داخل الأنواع المستهدفة وكذلك داخل الأنواع التي تحدث بشكل مشترك.
- إذا كان هناك اختلاف وراثي داخل المحدد، والبحث عن المقايسات حيث التمهيديات والتحقيق لا تقع داخل هذه المناطق.
- لمنع تضخيم الأنواع غير المستهدفة، ابحث عن عدم التطابق مع الأنواع غير المستهدفة. اختر المقايسات التي تحتوي على أكبر عدد من حالات عدم التطابق لمزيد من التحقق من الصحة. يقترح Currier et al. (2018) اختيار مجموعات مع اثنين على الأقل من المناطق الثلاث (التمهيديان ، أو التمهيدي والتحقيق) وجود عدم تطابقين على الأقل مع جميع الأنواع غير المستهدفة. ومع ذلك، نضع في اعتبارنا أن عدم التطابق في التحقيق تسهم أقل فيخصوصية 10.
ملاحظة: الاختلافات داخل 3 أزواج قاعدة من 3 'نهاية كل التمهيدي زيادة خصوصية أفضل من الاختلافات في نهاية 5 'من التمهيديات10.
- النظر في المعلمات الهامة التالية في تصميم المقايسة.
- تحديد درجات حرارة ذوبان وعلة من التمهيديات والتحقيق. من الناحية المثالية يجب أن تكون درجة حرارة ذوبان (TM) من التمهيديات بين 60-64 درجة مئوية وضمن 2 درجة مئوية من بعضها البعض ، وينبغي أن يكون TM من المسبار 6-8 درجات أعلى من TM من التمهيديات. تعيين درجة حرارة التلين (تا) من رد فعل qPCR 5 °C أقل من درجة حرارة ذوبان، حول 55-60 °C11.
- فحص محتوى GC. اختر بين 35 - 65٪ من محتوى GC، وتجنب المناطق التي بها 4 أو أكثر من Gs متتالية. وجود 1 أو 2 GS أو CS في القواعد الأخيرة 5 من نهاية 3 من التمهيدي (GC المشبك) قد تزيد من خصوصية لأنها من شأنها أن تساعد التمهيدي لجعل أقوى السندات12.
- البحث عن هياكل دبوس الشعر وخافض. اختبار التمهيديات والتحقيق لهياكل دبوس الشعر المتوقعة وd dimers باستخدام برنامج تحليل oligonucleotide (على سبيل المثال، OligoAnalyzer -IDT13؛ أوليغوكالكولوتور14). يمكن أن تتسبب هذه الهياكل في تضخيم غير مستهدف وكفاءة أقل. تجنب المقايسات التي من المتوقع أن تشكل هذه الهياكل.
- تحديد طول التمهيدي. تهدف للتمهيدي بين 18-25 قواعد في الطول وطول التحقيق بين 20-25 قواعد. قد يكون للمبرمجين والتحقيقات الأطول كفاءة تضخيم أقل.
- تحديد طول أمبيرليكون. يجب أن يكون بين ما يقرب من 100 و 250 زوجا أساسيا. هذا النطاق هو عموما قصيرة بما يكفي لكفاءة PCR عالية، ولكن طويلة بما يكفي لسهولة التحقق من قبل سانجر تسلسل4،15.
- تصميم المسابير. تأكد من أن المسابير ليس لديها قاعدة G في نهاية 5 ، لأنها يمكن أن تضعف الإشارة من الأصباغ الخضراء والصفراء11. لقد صممنا مسابير مزدوجة مروية، مع IDT 3IABkFQ ومرويات ZEN وفلوروفوريس FAM أو HEX.
ملاحظة: تحديد المسابير MGB: غالبا ما تستخدم المسابير TaqMan MGB (الموثق الأخدود طفيفة) لدراسات eDNA. ومع ذلك، لأن هذه المسابير قصيرة جدا، فإنها يمكن أن ترتبط غير أهداف حتى مع عدم تطابق زوج قاعدة 2 أو 310. - تحديد TM التحقيق. يجب أن تكون درجة حرارة ذوبان المسبار 6-8 درجة مئوية أعلى من التمهيديات. انخفاض درجات الحرارة يقلل من نجاح ملزمة للتحقيق.
- تحديد طول المسبار وموقعه. يجب أن يتراوح طول المسبار بين 20 و25 نقطة أساس ويقع بشكل مثالي بالقرب من موقع الربط التمهيدي على نفس الخيط دون تداخله.
3. فحص المقايسة والتحسين
- في تطوير اختبار السايساي سيليكو. قبل طلب مجموعات التمهيدي التحقيق، وتقييم خصوصية (التضخيم غير المستهدفة المحتملة) عن طريق اختبار تضخيم التمهيدي في السيليكو.
- اختبار التمهيديات من خلال NCBI التمهيدي الانفجار16 أو برامج مماثلة التي يمكن تحديد الأهداف غير المحتملة في قاعدة بيانات NCBI NT / NR التي قد تضخيم مع المقايسة. إذا كان استخدام التمهيدية انفجار لصق التمهيديات على استخدام بلدي التمهيدي مربع تحت المعلمات التمهيدي. في خيارات محددات التحقق من خصوصية الزوج التمهيدي، حدد nr كقاعدة بيانات واكتب ترتيب الكائن الحي الذي يهم (على سبيل المثال، "Unionida" أو "Unionoida") في مربع الكائن الحي.
- مواصلة تقييم مجموعات التمهيدي / التحقيق بصريا على بيانات تسلسل محاذاة.
- من أجل تقييم التمهيديات والتحقيقات في نفس الوقت في سيليكو ، وخلق سلسلة نصية من التمهيدي إلى الأمام ، 12 ن ، والتحقيق ، 12 ن ، والمكمل العكسي من التمهيدي العكسي. إذا كان تسلسل المسبار ضمن 12 زوجا أساسيا لأحد المبرمجين، فاستخدم عدد N المطابق لعدد أزواج القاعدة بين التمهيدي والمسبار.
- استخدام NCBI في البحث انفجار النيوكليوتيد (Blastn) للبحث ضد قاعدة البيانات NR17. استخدم علامة التبويب التصنيف للبحث عن أنواع غير مستهدفة مع عدم تطابق قليل; وينبغي اختبار هذه في المختبر أثناء التحسين المقايسة.
ملاحظة: في اختبار السيليكو يساعد على استبعاد المقايسات غير محددة، ولكن يجب اختبار المقايسات محددة محتملة تجريبيا (في المختبر)، كما ليس كل الأنواع لديها تسلسل في قواعد البيانات الجينية والتمهيدي والتحقيقات لا يزال يمكن ربط إلى غير الأهداف حتى لو اعتبرت غير محتملة من قبل البرنامج.
- اختر ثلاث إلى خمس مجموعات التمهيدي / التحقيق لاختبار في المختبر.
- طلب التمهيديات، والتحقيقات ومعيار الحمض النووي الاصطناعية، فضلا عن التمهيديات M13 الذيل إضافية لتسلسل amplicon.
- طلب الأوليغونوكليوتيد الاصطناعية التمهيديات والتحقيقات من الشركة التي تجعل أوليغوس. وتوصف المسابير بصبغة فلورية وquencher. يجب اختيار الفلوروفوريس المختلفة للمقايسات التي تحتاج إلى مضاعفة. تحقق من جهاز qPCR الخاص بك للحصول على قائمة بالفلوروفورات التي يمكن للآلة اكتشافها.
- تصميم وترتيب المبرمجين M13 الذيل للتحقق من الكشف عن qPCR مع تسلسل سانجر عن طريق إضافة تسلسل M13 إلى الأمام (-20) ، GTA AAA CGA CGG CCA GT ، إلى نهاية 5 من التمهيدي الأمامي ، وتسلسل M13 العكسي (-27) ، CAG GAA ACA ACAT ATG AC ، إلى نهاية 5 من التمهيدي العكسي.
- يحتوي معيار الحمض النووي الاصطناعي على التسلسل المستهدف (بما في ذلك مناطق التمهيدي) بتركيز معروف في النسخ / ميكرولتر. تحديد عينات غير معروفة استنادا إلى منحنى مصنوع بتركيزات معروفة من هذا المعيار (أي المنحنى القياسي). الحصول على المعيار الاصطناعي من نفس الشركة التي تصنع التمهيديات والتحقيق. اتبع توصيات الشركة المصنعة لإعادة الإنفاق والتخزين. معايير التخفيف في العازلة TE مع الناقل tRNA باستخدام الأواني البلاستيكية منخفضة الاحتفاظ للحد من التحلل المائي وملزمة للأسطح.
ملاحظة: إذا لم يكن أداء المنحنى القياسي جيدا (ضعف كفاءة PCR، راجع الخطوة 3.4.2)، حاول إعادة تعليق المعيار في الماء أو Tris-HCl. - تعليق التمهيديات والمسابير في المياه الخالية من النيوكليز، تريس-HCl، أو العازلة TE بتركيزات مريحة للاستخدام المقايسة. عموما، تمييع الأسهم العاملة 20 أضعاف في المزيج الرئيسي لتحقيق تركيز المقايسة النهائي الأمثل. تخزين oligos المعلقة في ثابت -20 درجة مئوية عندما لا تكون قيد الاستخدام.
- في المختبر (في المختبر) المقايسة الأمثل والاختبار. رفض المقايسات التي لها كفاءة ضعيفة، أو التفاعل مع الأنواع التي تحدث بشكل مشترك، أو التي لديها حساسية ضعيفة18. تضمين استخدام عنصر تحكم إيجابي داخلي (IPC) أثناء تطوير الفحص وكذلك عند تشغيل عينات فعلية.
- أولا، العثور على درجة الحرارة المثلى والتمهيدي / قيم تركيز التحقيق للمقاسة. بمجرد تحسين هذه المعلمات لكفاءة PCR (الخطوة 3.4.2) ، التفاعل المتبادل (الخطوة 3.4.3) والحساسية (الخطوة 3.4.4) ، تابع اختبار المقايسة باستخدام IPC متعدد المضاعفات (الخطوة 3.4.5).
- اختبار درجة الحرارة المثلى التلين (تا) لالتمهيدي والمسابير باستخدام تدرج درجة حرارة PCR تركزت 5° C تحت متوسط المتوقع التمهيدي TM.
- اختبار أفضل التمهيدي وتركيزات التحقيق. عادة، يتم اختبار تركيزات 200 nM و 400 nM و 800 nM التمهيدي و 75 nM و 125 nM و 200 nM.
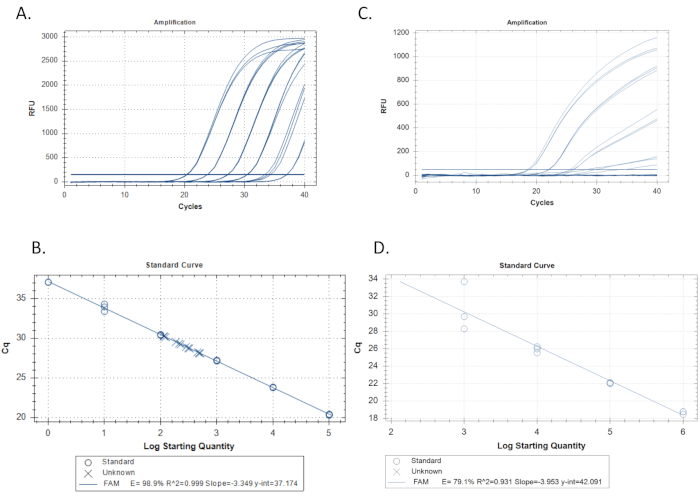
- إنشاء منحنى قياسي وتحديد الكفاءة والنطاق الخطي. اختبار ما لا يقل عن ستة 10 أضعاف التخفيف من معيار الحمض النووي الاصطناعية التي تحتوي على تسلسل الهدف، في ما يقرب من 100 نسخ / رد فعل على 105 نسخ / رد فعل (الشكل 3A).
- استخدم برنامج qPCR لرسم قيمة Cq (عتبة الدورة عند القياس الكمي) لكل معيار على المحور ص وقاعدة السجل 10 للتركيز القياسي الأولي في النسخ / التفاعل على المحور س. يجب أن يقوم برنامج qPCR تلقائيا بتشغيل انحدار خطي(الشكل 3B).
- حساب الكفاءة من منحدر الانحدار، E = -1 + 10(-1/المنحدر). على سبيل المثال، إذا كان الميل -3.4، E= -1 + 10(0.29) = 0.97 أو 97٪. تحقق أيضا من قيم r2 التي تشير إلى مدى ملاءمة النسخ المتماثلة القياسية على المنحنى. يجب أن يقوم برنامج qPCR بحساب هذا تلقائيا أيضا(الشكل 3B). الهدف من قيم الكفاءة بنسبة 100٪ (±10٪) وقيم r2 من ≥0.989،15،19،20،21،22.
- فحص بصريا منحنى القياسية للتحيز، وهذا هو، الانحرافات عن الانحدار في اتجاه ثابت أو لضعف الأداء منحنى القياسية كما تقاس بالكفاءة والقيم ص2 (الشكل 3C و 3D).
- خصوصية: تقييم التفاعل المتبادل مع الأنواع غير المستهدفة لتقليل فرصة حدوث إيجابيات كاذبة. عندما قد تؤدي عمليات الكشف عن eDNA إلى قرارات إدارية مكلفة، تحقق من عمليات الكشف الإيجابية عن طريق تسلسل amplicon.
- غير المستهدفة: إجراء الفحص ضد استخراج الحمض النووي الجينومي لعينات تم التحقق منها تصنيفيا للأنواع ذات الصلة والأنواع التي تحدث جغرافيا؛ مع أولوية قصوى هي لاختبار ضد الأنواع ذات الصلة الوثيقة، التي تحدث بشكل مشترك. استخدام تركيزات الحمض النووي الإجمالية مماثلة لكل من العينات المستهدفة وغير المستهدفة. وينبغي أن يسفر التركيز المختار عن تضخيم من عينات الأنواع المستهدفة بالقرب من منتصف النطاق الخطي للمنحنى القياسي. وينبغي ملاحظة التضخيم فقط مع الأنواع المستهدفة.
- إذا لوحظ التضخيم غير المستهدف، قم بتنظيف المنتج وتسلسله للتحقق من هويته. وليس من غير المألوف ملاحظة التلوث الناجم عن الأنواع المستهدفة في عينات الأنسجة من الأنواع غير المستهدفة، وبالتالي ينبغي التحقق من جميع التضخيمات في هذه المرحلة عن طريق التسلسل. إعادة ترطيب amplicons تنظيفها من اختبارات التحديد باستخدام التمهيديات M13 الذيل وتسلسل مع التمهيديات M13.
- في مختبر ما بعد PCR، نقل منتجات qPCR ليتم تسلسلها إلى أنابيب جديدة. إزالة التمهيديات المتبقية ومكونات رد الفعل مع مجموعة تنظيف (على سبيل المثال، MinElute PCR تنقية كيت).
- جعل 1:100 التخفيف من elutions وتضخيم 1 ميكرولتر من كل لمدة 30 دورة في رد فعل PCR 50 ميكرولتر مع التمهيديات ذيل M13 والبوليميراز عالية الدقة (على سبيل المثال، فوسيون عالية الدقة الحمض النووي بوليمراز).
- تشغيل 10 ميكرولتر من كل رد فعل على هلام agarose 1٪ للتحقق من وجود نطاق واحد من الحجم المتوقع. إذا لم يتم ملاحظة أي نطاق، قم بزيادة عدد الدورات أو مقدار العينة. إذا لوحظت نطاقات متعددة، هلام تنقية الفرقة من الحجم المتوقع.
- إزالة التمهيديات المتبقية ومكونات رد الفعل مع مجموعة تنظيف على النحو الوارد أعلاه وقياس تركيزات الحمض النووي من elutions.
- إعداد ردود الفعل التسلسل مع التمهيديات M13 وفقا لتعليمات مرفق التسلسل.
ملاحظة: لا تفتح عينات مكبرة في مختبر qPCR. إعداد عينات للتسلسل في مختبر مخصص لعينات ما بعد PCR.
- الحساسية: تؤثر الحساسية على فرصة حدوث سلبيات كاذبة، أو الفشل في الكشف عن الحمض النووي للأنواع المستهدفة عندما يكون موجودا. تقييم حد الكشف (LOD) والحد من القياس الكمي (LOQ) لكل مقايسة. وأخيرا، تشمل الرقابة الإيجابية الداخلية (IPC) لتقييم تثبيط PCR من العينات. تعدد واختبار هذا المقايسة IPC مع المقايسة المصممة لضمان لا تتداخل المقايستين مع بعضها البعض.
- LOD: جعل ستة 4 أضعاف المخففات التسلسلية من معيار الحمض النووي الاصطناعية، مع 8-24 يكرر لكل تخفيف القياسية(الشكل 4). حساب أدنى تركيز الأولي مع الكشف عن 95٪. يمكن إنشاء المؤامرات LOD و LOQ مع LOD / LOQ حاسبة R النصي5.
ملاحظة: لا يجب فرض رقابة على البيانات الموجودة أسفل LOD. وبسبب خصوصية PCR، لا يوجد حد أدنى للإيجابيات الحقيقية. ال LOD هو أعلى تركيز يمكن أن يتوقع حدوث السلبيات الكاذبة تحته. - LOQ: من نفس سلسلة التخفيف، احسب أدنى تركيز معياري أولي للحمض النووي قابل للقياس الكمي مع معامل الاختلاف (CV) أقل من 35٪.
ملاحظة: يجب الإبلاغ عن LOD و LOQ في نسخ / رد فعل. عند استخدام المقايسة التحقق من صحة وعينات الحقل تضخيم تحت LOQ، وينبغي الإبلاغ عن النتائج كما الكشف عن ٪ بدلا من تركيزات eDNA، لأنه لا يمكن قياس التركيز الدقيق بثقة5.
- LOD: جعل ستة 4 أضعاف المخففات التسلسلية من معيار الحمض النووي الاصطناعية، مع 8-24 يكرر لكل تخفيف القياسية(الشكل 4). حساب أدنى تركيز الأولي مع الكشف عن 95٪. يمكن إنشاء المؤامرات LOD و LOQ مع LOD / LOQ حاسبة R النصي5.
- استخدم عنصر تحكم إيجابي داخلي (IPC) لاختبار تثبيط PCR. يمكن أن يؤدي التثبيط إلى انخفاض في الحساسية والسلبيات الكاذبة. اختبار قدرة IPC المقايسة أن تكون متعددة مع المقايسة الهدف.
- يمكن مضاعفة المقايسة IPC مع الفحص المستهدف باستخدام مسبار بصبغة مراسل مختلفة عن المقايسة المستهدفة. يتكون هذا المقايسة من تسلسل الحمض النووي الاصطناعي القصير من نوع لا علاقة له بالتصنيف المستهدف ، مدمج في المزيج الرئيسي qPCR بتركيز منخفض يبلغ حوالي 102 نسخة / رد فعل ، إلى جانب التمهيدي والتحقيقات التي تكشفه. هذا التركيز أقل ضروري لتجنب المنافسة مع تسلسل الهدف للبوليميراز والنيوكليوتيدات23.
- مقارنة قيمة Cq قالب IPC الخاص بالعينة إلى قالب IPC في عنصر تحكم القالب لا. في عنصر تحكم القالب هذا (NTC) ، إدخال DNA فقط هو أن من قالب IPC. وينبغي أن يتضخم نموذج التصنيف الدولي للبراءات في رد الفعل هذا كما هو متوقع. إذا كان قالب IPC في عينة amplifies في 2 أو أكثر من دورات مختلفة عن ذلك من قالب IPC في NTC، يتم منع نموذج eDNA. يمكن تخفيف العينات التي تظهر تثبيط 1:10 وإعادة اختبارها. وإذا بقيت العينة مثبطة، فينبغي إزالة تلك العينة من التحليل.
- أولا، العثور على درجة الحرارة المثلى والتمهيدي / قيم تركيز التحقيق للمقاسة. بمجرد تحسين هذه المعلمات لكفاءة PCR (الخطوة 3.4.2) ، التفاعل المتبادل (الخطوة 3.4.3) والحساسية (الخطوة 3.4.4) ، تابع اختبار المقايسة باستخدام IPC متعدد المضاعفات (الخطوة 3.4.5).
- في وضع الاختبار الموقعي واختبار
- في المختبر: إذا كان الوصول إلى الكائن الحي في المختبر وكذلك الأنواع الستاريك متاح؛ أخذ عينات المياه من حاويات مع هذه الأنواع، ومعالجة العينات واختبار المقايسة ضد هذه العينات eDNA. تسلسل المنتجات على النحو الوارد أعلاه للتحقق من تضخيم الهدف المقصود باستخدام التمهيديات ذيل M13.
- في الميدان:
- تحديد المواقع التي من المعروف أن الكائن المستهدف يحدث ويعرف أنه لا يحدث. ومن الأفضل أن يكون هناك قدر من الوفرة في كل موقع تحدث فيه الأنواع المستهدفة.
- تحديد أحجام العينات وطرق جمع العينات (مثل الترشيح والطرد المركزي وما إلى ذلك).
- تضمين حقل فارغ أو تحكم سلبي في كل موقع، هذه هي المياه النظيفة التي تم إحضارها إلى موقع الحقل ومن ثم جمعها وإعدادها بنفس المعدات الميدانية والبروتوكولات المستخدمة لأخذ عينات eDNA24. والغرض من الحقل الفارغ هو الكشف عن تلوث معدات أخذ العينات والمعدات الميدانية التي يتم إحضارها إلى الموقع. خذ الحقل فارغا قبل معالجة عينات مياه الحقل.
- خذ عينات مياه متعددة لكل موقع، ويفضل أن تكون 3 عينات لكل موقع.
- مرة أخرى في المختبر، ومعالجة واستخراج العينات.
- قم بتشغيل المقايسة باستخدام لوحة تم إعدادها على غرار الشكل 5A وقارن تركيز eDNA وتكرار الكشف مع اختلافات الموقع المعروفة في الحدوث والوفرة. تأكد من جميع عمليات الكشف عن طريق تسلسل24،25.
ملاحظة: أعلاه التحقق من صحة الفحص من خلال المستوى 4 من الجدول Thalinger et al.s (2020)6 (تحسين الأداء الفني للمقايسة) والبدء في جمع البيانات التي تدعم التحقق من صحة الفحص المستوى 5. المستوى 5 يتضمن احتمال النمذجة واستخدام المقايسة لدراسات البيئة eDNA. ونرى أن هذا الأمر يتجاوز نطاق تطوير الفحص الأساسي، ولكننا نشجع هذه التطبيقات من الاختبارات المختبرية والميدانية التي تم فحصها لتحسين تصميم المقايسة وتفسير البيانات.
النتائج
في تصميم الأنواع محددة qPCR المقايسة لm mucket (A. ligamentina), تم تحميل تسلسل المتاحة من جميع الأنواع Unionidae في نهر كلينش. كما أدرجت أنواع وثيقة الصلة مثل السيليكويدية المصباحية في قاعدة البيانات المرجعية على الرغم من عدم وجودها في نفس النهر. لم يتم العثور على جميع الأنواع في نظام النهر المثير للاهتمام في GenBank ، لذلك تم تسلسل أنواع إضافية في المنزل. تم محاذاة التسلسلات باستخدام برنامج Geneious وتم استخدام برنامج Primer Quest (IDT) لتصميم عمليات قياس متعددة. تمت إضافة خمس مجموعات من التمهيديات والتحقيق إلى المحاذاة للتقييم البصري(الشكل 2). ثم تم اختبارها في سيليكو باستخدام التمهيدي الانفجار، وبعد ذلك أمرت لمزيد من الاختبار في المختبر. وفي المختبر، تم اختبار جميع المقايسات باستخدام استخراج الحمض النووي ل 27 نوعا متاحا للتحقق من التحديد. فحص واحد (A.lig.1) تضخيم بنجاح فقط الأنواع المستهدفة (الجدول 1; الجدول 2). هذا المقايسة تحركت إلى الأمام لمزيد من الاختبار من كفاءة الفحص، LOD وLOQ. لديها طول amplicon من 121 أزواج قاعدة. ويبين الجدول 3 التسلسل المستخدم في معيار الحمض النووي الاصطناعي A. ligamentina. الشكل 3A والشكل 3B تظهر نتائج تقييم ناجحة مع كفاءة جيدة والقيم ص2. الشكل 3C والشكل 3D تظهر المقايسة التي منحنى القياسية لديها كفاءة ضعيفة; تم تجاهل هذا المقايسة. تم العثور على LOD و LOQ للمقايسة المختارة (A.lig.1) على حد سواء لتكون 5.00 نسخ / رد فعل باستخدام طريقة منفصلة وصفها في Klymus وآخرون5. لم يؤثر IPC الذي تم multixed مع المقايسة(جداول 3-6)على منحنى A. ligamentina المقايسة القياسية. IPC نستخدمها هي جزء من نسخة HemT الماوس. تم تصميم هذا المقايسة مسبقا بواسطة IDT لتطبيق آخر ، لكننا قمنا بتعديل استخدامه ك IPC لتطبيقات eDNA الخاصة بالمختبر.
يجب أن يفي تشغيل qPCR الناجح بمعايير معينة لكل مقياس للأداء (أي تضخيم المنحنى القياسي ، والتحكم الإيجابي للحمض النووي الجينومي ، وعدم التحكم في القالب والتحكم الإيجابي الداخلي). يجب أن تحتوي معايير الفحص المستهدف على منحنيات تضخيم أسية. يجب أن تصل هذه المنحنيات إلى هضبة نقطة النهاية إذا سمح لها بتشغيل دورات كافية. وهذا يدل على أن المسبار الفلوري يستهلك بالكامل أثناء التفاعل، وتصل مستويات الفلورسينس إلى الحد الأقصى. قد لا تصل معايير تضخيم لاحقة إلى هضبة في 40 دورة. يجب أن يكون للضوابط الإيجابية (الحمض النووي الجينومي و IPC) نفس النمط. قد تكون المجهولات أو لا تضخم ، ولكن يجب أن يكون التضخيم في المجهولات أيضا نمط أسي وهضبة نقطة النهاية(الشكل 5).
في qPCR الجودة، وتضخيم التخفيف القياسية في Cq متباعدة بالتساوي من كل 3.3 دورات تقريبا لكل فرق 10 أضعاف في التركيز. كل تكرار لتخفيف القياسية تضخيم بطريقة مجمعة بإحكام وجود ما يقرب من نفس Cq (ممثلة بالقيم ص2). يجب أن تظهر جميع المخففات القياسية تضخيما (الشكل 3A). في qPCR الفقراء، قد تظهر المعايير شكل غير الأسي، والتباين متفاوتة في القيم Cq بين التخفيفات، لا تأتي إلى هضبة نقطة النهاية، أو بعض التخفيفات قد لا تضخيم على الإطلاق(الشكل 3D).
المعلمات الهامة للمنحنى القياسي هي الكفاءة، ص2،المنحدر، وy-اعتراض. يجب أن تنخفض الكفاءة بين 90٪ -110٪ مع القيم المثالية بالقرب من 100٪ وقيم r2 يجب أن تكون أعلى من 0.98 مع نتائج مثالية تقترب من 1.015،22. يجب أن تكون قيم الميل بين -3.2 و-3.5 مع نتائج مثالية بالقرب من -3.322. يجب أن تقع قيم تقاطع y بين Cq من 34-41 مع نتائج مثالية لها Cq من 37.0. تقاطع y هو Cq المتوقع لرد فعل مع نسخة واحدة من التسلسل المستهدف ، أصغر وحدة يمكن قياسها في qPCR واحد. المجهولات مع Cq أكبر من اعتراض ص من المرجح أن تكون مثبطة. وقد يكون من الضروري تشغيل أكثر من 40 دورة من PCR للكشف عن الهدف في حالة التثبيط أو مجموعة التمهيدي غير الفعالة ، ولكن القياس الكمي غير ممكن في ظل هذه الظروف ، وينبغي تشغيل ضوابط سلبية إضافية دون التسلسل المستهدف ، ولكن تحتوي على الحمض النووي الكلي مماثلة للمجهولات ، لاستبعاد التضخيم من مصادر غير محددة.
يجب مقارنة تضخيم الرقابة الإيجابية الداخلية (IPC) في عينات غير معروفة بنتائج التحكم السلبي في القالب IPC ، حيث لا توجد منافسة على الكواشف ولا توجد مثبطات. وينبغي اعتبار المجهولات التي تحتوي على IPC ذات دورتين أو أكبر من متوسط قيمة Cq للمجلس الوطني الانتقالي ، أو التي لا تضخمها مثبطة. إذا لم تكن هناك مثبطات موجودة في العينات ، فيجب أن يكون لجميع تضخيم IPC تجمع محكم في المؤامرة مع قيم Cq بالقرب من نفس NTC (الشكل 6).
وأخيرا، حدث اختبار في الموقع للمقاسة. تمت تصفية 20 عينة مياه من نهر كلينش وثلاث عينات فارغة من الحقل بين 25-26 سبتمبر 2019 على بعد 500 متر من سرير بلح البحر المعروف بألف أربطة. تمت تصفية ما يقرب من أربع عينات L 1 من المياه لكل موقع أخذ العينات. مواقع المواقع المدرجة في الجزء السفلي من سرير بلح البحر في تيار، أسفل سرير بلح البحر بالقرب من الشاطئ، 100 متر المصب من السرير في تيار، 500 متر المصب من السرير في تيار و 500 متر المصب من السرير بالقرب من الشاطئ (الشكل 7). مرة أخرى في المختبر، تم قطع كل مرشح إلى النصف وتم استخراج الحمض النووي من نصف مرشح فقط. تم تخزين النصف المتبقي لكل عينة في ثلاجة -80 درجة مئوية. ثم تم تشغيل العينات باستخدام A.lig.1 المقايسة متعددة مع IPC. ومن بين العينات ال 23، تبين أن خمس عينات قد تم تثبيطها. تم تخفيف هذه العينات 1:10 وأعيد تشغيل التخفيفات. تم تضخيم تسع عشرة عينة من العينات الميدانية العشرين باستخدام المقايسة المصممة. ومن بين هذه العينات ال 19، كانت خمس عينات فوق ال LOD وLOQ للمقايسة من 5 نسخ/رد فعل؛ بمعنى أن معظم العينات لديها الكشف عن eDNA ولكن على مستوى حيث من المرجح أن تحدث نتائج سلبية كاذبة وأن المقايسة لا يمكن أن تحدد بثقة رقم النسخ لتلك العينات 14. ومع ذلك، فإن 75 إلى 100 في المائة من الموقع البيولوجي الأربعة يكرر تضخيمه في كل موقع لأخذ العينات. وكانت اثنتان من الفراغات الميدانية الثلاثة سلبيتين، في حين أظهر حقل واحد فارغ تضخيما، مشددا على أهمية التقنية النظيفة في الميدان.

الشكل 1: سير العمل لبناء قاعدة بيانات تسلسل الحمض النووي الميتوكوندريا. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: تسلسل التحالفات لأنواع بلح البحر نهر كلينش مع التمهيديات المحتملين والمسابير لالرباط Actinonaias ND1 المقايسة. التمهيديات الأمامية باللون الأخضر الداكن، والتحقيق في التمهيدي الأحمر والعكس في الضوء الأخضر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الشكل 3: أمثلة الانحدار المنحنى الخطي والمنحنى القياسي. مثالعلى منحنى قياسي مقبول مشتق من تضخيم ثلاثة تكرارات لكل من ستة تمييعات قياسية. سلسلة تخفيف قياسية 10 أضعاف مع أعلى تركيز للمعيار على اليسار ، مع انخفاض التركيزات تتحرك إلى اليمين. الخط الأفقي عبور جميع الآثار هو عتبة لدورة في الكمية (Cq). حيث يعبر كل تتبع هذه العتبة حيث يتم تحديد Cq. B.الانحدار الخطي المصنوع من النسخ المتماثلة القياسية لل الشكل 3A. يتم رسم نسخ متماثلة من التخفيفات القياسية في دوائر ويتم رسم المجهولات (العينات) مع x. الكفاءة هي 98.9٪، ص2 تقترب من 1.0، والمنحدر من -3.349. ج.مثال على منحنى قياسي ضعيف مشتق من تضخيم ثلاثة تكرارات لكل من ستة تمييعات قياسية. د.انحدار خطي يشكل المنحنى القياسي للنسخ المتماثلة القياسية التي تم تضخيمها في المثال 3C. لاحظ ضعف الكفاءة وقيم r2. لاحظ أيضا أن 4 فقط من المعايير 6 تضخيمها. إذا بعد تكرار أشواط، منحنى القياسية لا يحسن، قد تكون المشكلة مع مجموعة التمهيدي / التحقيق الفقراء التي لا تضخيم الحمض النووي المستهدفة كما هو متوقع في هذه الحالة، لا ينبغي النظر في هذا المقايسة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
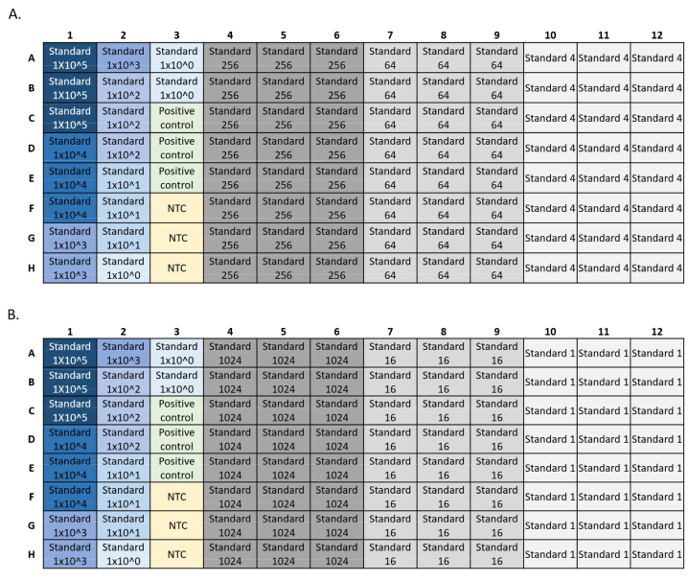
الشكل 4: أمثلة من الاجهزة لوحة لLOD وQQ qPCR القياسية يعمل. المعايير المستخدمة في المنحنى باللون الأزرق، وانخفاض التركيز القياسي من الأزرق الداكن إلى الأزرق الفاتح. التحكم الإيجابي للحمض النووي باللون الأخضر وعدم وجود عنصر تحكم في القالب (NTC) باللون الأصفر. تركيزات قياسية تجريبية باللون الرمادي تظهر 24 تكرارا لكل تخفيف قياسي. كانت سلسلة التخفيف مطلية عبر لوحتين (A، B)، كل منها بمنحنى قياسي، وتحكم إيجابي، وNTC. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
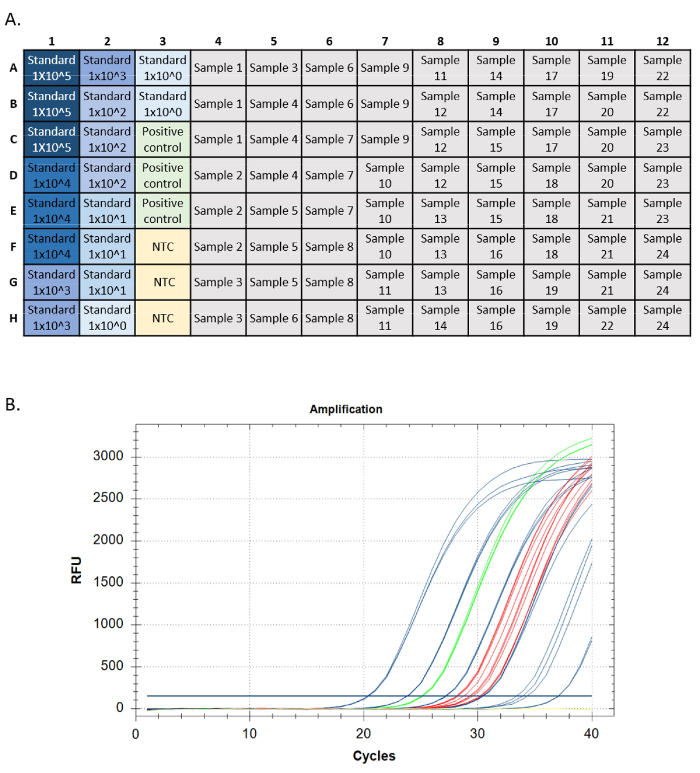
الشكل 5: إعداد لوحة وآثار التضخيم من تشغيل qPCR. A.إعداد لوحة، والمعايير المبينة باللون الأزرق، أغمق مما يدل على أعلى تركيز للمعيار. DNA التحكم الإيجابي في الأخضر، لا عناصر تحكم قالب باللون الأصفر (NTC)، عينة الأهداف باللون الرمادي. B.آثار التضخيم من تشغيل qPCR. المعايير الموضحة باللون الأزرق، والتحكم الإيجابي للحمض النووي باللون الأخضر، وعدم وجود عناصر تحكم في القالب باللون الأصفر، ومجهولين باللون الأحمر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
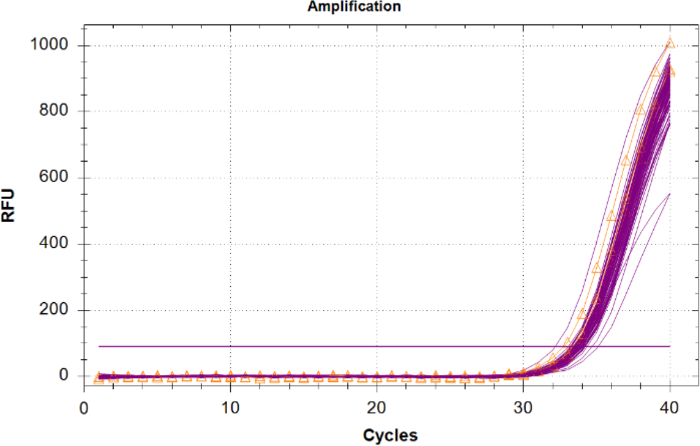
الشكل 6: آثار تضخيم للتحكم الإيجابي الداخلي (IPC). IPC تتبعات لكافة العينات غير معروف في أرجواني و IPC من عناصر تحكم قالب لا (NTCs) هو موضح باللون البرتقالي مع مثلثات. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
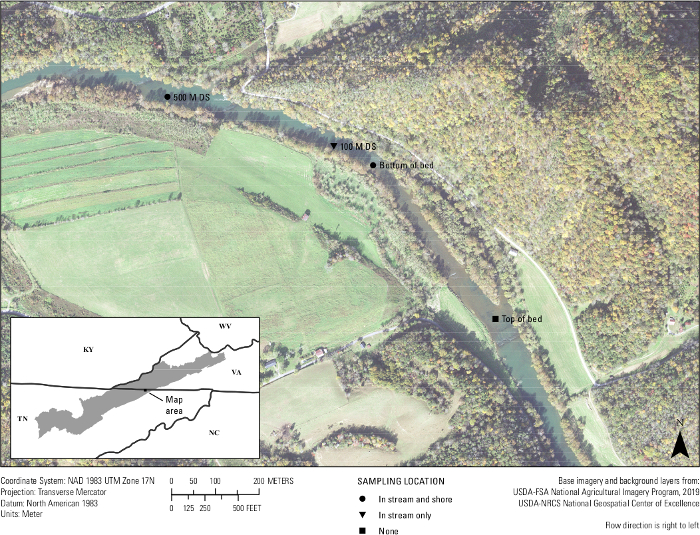
الشكل 7: خريطة تبين مواقع جمع eDNA لسرير بلح البحر في نهر كلينش على طول الحدود بين فيرجينيا وتينيسي. تم جمع عينات في والنز بيند في الجزء السفلي من السرير، 100 متر المصب من السرير، و 500 متر المصب من السرير. تم تجميع المواقع إما في منتصف المجرى (في التيار) أو ما يقرب من 1 - 2 متر من الشاطئ (الشاطئ). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
| مكون | اسم | تسلسل 5 ' – 3 ' | تسمية الفلورسنت | |
| التمهيدي الأمامي | A.lig.1-f | سيكتاتكاكجتاكاتاتكاتك | ||
| عكس التمهيدي | A.lig.1-r | غغاات جاغاكتاتاكاكتاكتا | ||
| مجس | A.lig.1 التحقيق | تي تيكتاغاكغاتاغاغاكتكغجت | فام | |
الجدول 1: المقايسة المصممة ل Actinonaias ligamentina qPCR (A.lig.1) بما في ذلك تسلسلات التمهيديات الأمامية والعكسية والتحقيق.
| جنس | تضخيم | في نهر كلينش |
| 1. أكتينونياس اربطة | نعم | نعم |
| 2. أكتينوناياس بيكروسا | لا | نعم |
| 3. أمبلما بليكاتا | لا | نعم |
| 4. كوربيكولا SPP. | لا | نعم |
| 5. كامبرلانديا مونودونتا | لا | نعم |
| 6. سيكلونياس درنة | لا | نعم |
| 7. سيبروجينيا ستغاريا | لا | نعم |
| 8. إليبتيو ديلاتاتا | لا | نعم |
| 9. إيبوبلاسما بريفيدنس | لا | نعم |
| 10. إيبوبالاسما كابسايس | لا | نعم |
| 11. إيببلاسما فلورنتينا أوريولا | لا | نعم |
| 12. إيبيوبلاسما تريكيترا | لا | نعم |
| 13. فوسكونايا كور | لا | نعم |
| 14. فوسكونايا سوبروتوندا | لا | نعم |
| 15. لامبسيليس أوفاتا | لا | نعم |
| 16. لامبسيليس سيليكويديا | لا | لا |
| 17. لاسميجونا كوستاتا | لا | نعم |
| 18. ليميوس ريموسوس | لا | نعم |
| 19- لكسينجتونيا دولابيلويدس | لا | نعم |
| 20. ميديونيدوس كونراديكوس | لا | نعم |
| 21. فيوريوس cyphyus | لا | نعم |
| 22. بليوروبيما بلنبل | لا | نعم |
| 23. فاسيولاريس Ptychobranchus | لا | نعم |
| 24. Ptychobranchus تحتية | لا | نعم |
| 25. كوادرولا بوستولوسا | لا | نعم |
| 26. ستروفيتوس متموجة | لا | نعم |
| 27. فيلوسا قزحية | لا | نعم |
الجدول 2: قائمة بالأنواع المستخدمة في اختبار خصوصية المختبر في المقايسة A.lig.1. وضخمت المقايسة الحمض النووي الجينومي للهدف(Actinonaias ligamentina)ولم تضخم أي من الأنواع غير المستهدفة.
| مكون | تسلسل 5 '-3' | ||||
| أكتينونياس ليجينيتينا القياسية | CCCTCATCACGTAC مكافحة الإرهابGTGTCغاتاكتاتكتكتكتشتغاكغاكتا | ||||
| AAGCCCTCGGGT أكتكاتكااتشاغاغككاتاتاغاغك كاتشاتكا | |||||
| كاكاكاتاجاجاتغاتكاتكاتاغكتاتككتكتغاكاكاكاكتكت | |||||
| كاهاكتاكاتاكاتكاتكاتاكاكاكاتغاتاوتاغاكتاغاكت | |||||
| غاكاتاتاكاتكاتكات كاتكاتاكتات ستاتغكتاتككتاتكت | |||||
| تغتاتاتكتكاتكتاكتاغاكتاتاكاكتاتاكاغاككتاكاكتاكاكتاكا | |||||
| أتاتGCCCTTAGGCTCGAGCCACCCAAACCATTTGAGGTTACAA | |||||
| تاك | |||||
| قالب IPC (هيم-T) | كاتكاتاغاغاكتاتاكاتكاتكاغاكتكتكتغاغتكتكغاكتكاغاكغكت | ||||
| GTAغاكاتكتكشتكتجتجاكاتاكتكغGCTCTATGTTCAGGACTGCAC | |||||
الجدول 3: تسلسل (5'-3') من معيار أكتينوناياس الرباط وقالب IPC (هيم-T) المستخدمة لهذا المقايسة. تسلسل للأمام وعكس التمهيديات هي في جريئة وم مائلة، ويتم التأكيد على أن من التحقيق.
| مكون | اسم | تسلسل 5 ' – 3 ' | تسمية الفلورسنت | |
| التمهيدي الأمامي | هيم تي-إف | تكتغاغتكتكتكغاكت | ||
| عكس التمهيدي | هيم تي آر | غاغاغاتغاكاكاتاغاغك | ||
| مجس | هيم تي بي | غاكاغتكتكتكتجتجتغاكاتكغ | Cy5 | |
الجدول 4: المقايسة الداخلية للرقابة الإيجابية (IPC) بما في ذلك تسلسلات للتمهيدي الأمامي والعكسي والتحقيق.
| حجم العينة الواحدة (ميكرولتر) | مكون |
| 10 | البيئة ماجستير ميكس |
| 1 | 20uM A. lig.1 F/ R مزيج |
| 1 | 2.5uM A. lig.1 التحقيق |
| 1 | 5uM IPC التمهيدي مزيج (HemT - F / R) |
| 0.75 | 2.5uM IPC التحقيق (HemT-P) |
| 1.5 | 1 X 103 تركيز قالب IPC |
| 2.75 | ح20 |
| 2 | عينة |
| 20 | إجمالي حجم التداول |
الجدول 5: مزيج PCR المستخدم في A.lig.1 المقايسة متعددة مع المقايسة IPC.
| درج | درجة الحرارة (°C ) | الوقت | |
| 1 | النشوة الأولية | 95 | 10 دقائق |
| 2 | النشوة | 95 | 15 ثانية |
| 3 | الصلب | 60 | دقيقة واحدة |
| 4 | انتقل إلى الخطوة 2، كرر 39X |
الجدول 6: شروط رد الفعل على المقايسة A.lig.1.
Discussion
كما هو الحال مع أي دراسة، تحديد السؤال الذي سيتم تناوله هو الخطوة الأولى وتصميم فحص eDNA يعتمد على نطاق الدراسة26. على سبيل المثال، إذا كان الهدف من البحث أو المسح هو الكشف عن نوع واحد أو عدد قليل، فإن الفحص المستهدف القائم على المسبار هو الأفضل. ومع ذلك، إذا كان الهدف هو تقييم مجموعة أكبر أو تجميع الأنواع، فإن المقايسات عالية الإنتاجية التسلسل metabarcoding هي أكثر ملاءمة. بمجرد تحديد النهج الذي يجب اتباعه ، يوصى بإجراء دراسة تجريبية بما في ذلك تصميم الفحص والاختبار والتحسين24. يبدأ تصميم المقايسة بقائمة من الأنواع كما هو موضح في الشكل 1. وستكون هذه القائمة هي الأساس لفهم مدى أداء المقايسة من حيث التحديد والنطاق الجغرافي الذي يمكن تطبيقه عليه6,10. ويشجع على تصميم المقايسة لمنطقة جغرافية محددة، مما يمكن المصمم من إجراء اختبار أفضل للفحص المتبادل للتفاعلات المتبادلة ضد الأنواع الأخرى في تلك المنطقة، وإدراك القيود التي يفرضها ذلك على توسيع نطاق الفحص ليشمل مناطق أخرى قد تحدث فيها أنواع مستهدفة24. وبمجرد الانتهاء من القائمة، يمكن تنزيل التسلسلات من قواعد البيانات الوراثية العامة. بما أن قواعد البيانات هذه غير مكتملة27، يجب على المرء تسلسل أكبر عدد ممكن من الأنواع في القائمة في المنزل لإكمال قاعدة البيانات المرجعية المحلية للتسلسلات التي سيتم استخدامها في تصميم المقايسة. إعطاء الأولوية للأنواع التي تحدث بشكل وثيق، لأن هذه هي على الأرجح غير الأهداف التي سوف تضخيم. التركيز على جميع الأنواع داخل نفس الجنس أو الأسرة مثل الأنواع المستهدفة هو مكان جيد للبدء. وستساعد المقارنات مع الأنواع ذات الصلة الوثيقة على تحديد مناطق التسلسل الفريدة من نوعها بالنسبة للأنواع المستهدفة. يمكن أن يساعد هذا في إعلام كيفية أداء الفحص في الأنظمة أو المواقع الأخرى. مناطق الميتوكوندريا هي الخيار المعتاد لتطوير المقايسة، وذلك لأن المزيد من المعلومات تسلسل من مجموعة متنوعة من الأنواع المتاحة في الجينات الميتوكوندريا التي استخدمت في الباركود من مشاريع الحياة، ولأن الحمض النووي الميتوكوندريا موجود بتركيز أكبر بكثير في نسخ / خلية من الحمض النووي24,28,29. وينبغي تقييم مناطق جينية متعددة من أجل زيادة تطوير المقايسة حيث تختلف تغطية التسلسل بين التصنيفات في قواعد بيانات المستودعات الجينية. بعد إنشاء قاعدة البيانات المحلية هذه من التسلسلات المرجعية ، يتم استخدام مزيج من التصور اليدوي لبيانات التسلسل المنحازة وبرامج الكمبيوتر لتصميم المقايسات التمهيدية / التحقيق. لا ينبغي للمرء أن يعتمد بشكل صارم على البرمجيات لتحديد أي المقايسات لاختبار. من المهم التحقق بصريا من المحاذاة حيث يجلس التمهيديون والمسابير على الأهداف وغير الأهداف للحصول على فهم أفضل لكيفية تصرفهم في PCR. وأخيرا فحص المقايسة والتحسين يشمل ثلاثة مستويات (في سيليكو، في المختبر وفي الموقع)6,7,24,25. في تصميم واختبار سيليكو مهمة لإنتاج قائمة قصيرة من المقايسات مع فرصة جيدة للنجاح، ولكن التجريبية (في المختبر) اختبار أمر بالغ الأهمية لاختيار المقايسة مع أفضل أداء فعلي. في المختبر الأمثل واختبار المقايسات تشمل قياس كفاءة رد الفعل وتحديد حساسية المقايسة وخصوصية. 10- إن حدود الكشف والتحديد الكمي هما معلمان كثيرا ما يغفلان في تطوير المقايسة ولكنهما مهمان لتفسير البيانات. من خلال تشغيل نسخ متماثلة متعددة من المنحنيات القياسية لإجراء الفحص ، يمكن بسهولة قياس LOD و LOQ1,5,30. تناقش دراسات قليلة النتائج فيما يتعلق ب LOD أو LOQ للمقاير ، ولكن Sengupta وآخرون (2019) يدمجون LOD و LOQ الخاصين بهم في تفسير البيانات والرسومات لفهم أوضح لنتائجهم31. يجب أن تكون الضوابط الإيجابية الداخلية متعددة في المقايسة المصممة أيضا. دون اختبار لتثبيط PCR في العينات، قد تحدث سلبيات كاذبة24,32. نقترح استخدام اختبار IPC متعدد الأجهزة مع المقايسة المستهدفة كأسهل طريقة لاختبار تثبيط PCR23. وأخيرا، من الضروري إجراء اختبار ميداني للمقاحصات من العينات الميدانية والمختبرية التي تم جمعها لضمان حدوث تضخيم مستهدف في العينات البيئية24.
وتوجد قيود على استخدام المقايسات الخاصة بالأنواع والقائمة على المسبار qPCR مع عينات من eDNA. على سبيل المثال، قد يكون تصميم العديد من المقايسات للاختبار محدودا بتوافر التسلسل، وقد يكون الحل الوسط ضروريا بشأن جوانب أداء الفحص. يجب أن تسترشد هذه الخيارات بأهداف الدراسة ويجب الإبلاغ عنها مع النتائج26. فعلى سبيل المثال، إذا كان الهدف هو الكشف عن أنواع نادرة ومن المتوقع حدوث عدد قليل من الإيجابيات، يمكن استخدام تقييم بخصوصية ناقصة (أي تضخيم الأنواع غير المستهدفة) إذا تم التحقق من جميع عمليات الكشف عن طريق التسلسل. إذا كان الهدف هو رصد النطاق الجغرافي للأنواع وليس هناك حاجة إلى بيانات تركيز eDNA ، يمكن استخدام الفحص بكفاءة ناقصة والبيانات المبلغ عنها فقط ككشف في المئة. وعلاوة على ذلك، ما لم يتم اختبار جميع الخصوصيات المحتملة في المختبر، وهو أمر نادر الحدوث، لا يمكن للمرء أن يعرف على وجه اليقين المطلق التحديد الحقيقي للمقايسة. فعلى سبيل المثال، تم تصميم الفحص واختباره ضد العديد من أنواع بلح البحر في المياه العذبة في نهر كلينش. لاستخدام هذا المقايسة في نظام نهر مختلف، سنحتاج إلى اختباره ضد مجموعة من الأنواع في الموقع الجديد. الاختلاف الجيني داخل الأنواع أو السكان التي لم يتم اختبارها أثناء تطوير المقايسة قد تؤثر أيضا على خصوصية. وأخيرا، حتى لو تم التحقق من أن المقايسة لها أداء تقني عال؛ تتغير الظروف عند العمل في الحقل . يمكن أن تغير الظروف غير المتعلقة بالتقيير مثل تدفق المياه، وhh، والسلوك الحيواني إمكانية الكشف عن eDNA كما يمكن استخدام بروتوكولات جمع واستخراج eDNA المختلفة. استخدام المقايسات التي يتم تحسينها ووصفها بشكل جيد سيساعد على تسهيل فهم تأثير هذه المعلمات على الكشف عن eDNA.
وينضج مجال نانا إلى ما بعد مرحلة التحليل الاستكشافي إلى زيادة توحيد الأساليب والتقنيات. هذه التطورات سوف تحسن فهمنا لتقنيات eDNA وقدراتها والقيود المفروضة عليها. تعمل عملية التحسين التي نوضحها أعلاه على تحسين حساسية الفحص وخصوصية والاستنساخ. الهدف النهائي من هذا التحسين وتوحيد أساليب eDNA هو تحسين قدرات الباحثين على إجراء استنتاجات استنادا إلى بيانات eDNA بالإضافة إلى زيادة ثقة المستخدم النهائي وصاحب الحصة في النتائج.
Disclosures
ولا يعلن صاحبا البلاغ عن وجود تضارب في المصالح. ولم يكن لمقدمي التمويل أي دور في تصميم الدراسة؛ في جمع البيانات أو تحليلها أو تفسيرها؛ في كتابة المخطوطة؛ أو في قرار نشر النتائج.
Acknowledgements
نشكر ألفي ودود وترودي فروست اللذين ساعدا في تطوير واختبار التمهيدي. تم توفير تمويل تصميم الفحص المبلغ عنه في هذه الدراسة من قبل وزارة الدفاع الاستراتيجية برنامج البحوث البيئية والتنمية (RC19-1156). أي استخدام لأسماء التجارة أو المنتجات أو الشركات هو لأغراض وصفية فقط ولا يعني موافقة حكومة الولايات المتحدة. تتوفر البيانات التي تم إنشاؤها خلال هذه الدراسة https://doi.org/10.5066/P9BIGOS5 إصدار بيانات USGS.
Materials
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
References
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved