Method Article
Разработка и тестирование специфических видов Количественные анализы ПЦР для применения ДНК окружающей среды
В этой статье
Резюме
Анализы ДНК окружающей среды требуют тщательного проектирования, тестирования, оптимизации и проверки до начала сбора полевых данных. Здесь мы представляем протокол, чтобы принять пользователей через каждый шаг проектирования конкретных видов, зонд основе qPCR анализ для обнаружения и количественной оценки ДНК целевого вида из экологических образцов.
Аннотация
Разрабатываются новые, неинвазивные методы обнаружения и мониторинга присутствия видов для оказания помощи в рыболовстве и охране дикой природы. Использование образцов ДНК окружающей среды (eDNA) для обнаружения макробиоты является одной из таких групп методов, которая быстро становится популярной и реализуется в национальных программах управления. Здесь мы сосредоточимся на разработке видов конкретных целевых анализов для зонда на основе количественных ПЦР (qPCR) приложений. Использование зонда на основе qPCR предлагает большую специфичность, чем это возможно только с грунтовки. Кроме того, способность количественно определить количество ДНК в образце может быть полезна в нашем понимании экологии eDNA и интерпретации моделей обнаружения eDNA в этой области. Необходимо тщательно рассмотреть вопрос о разработке и тестировании этих анализов для обеспечения чувствительности и специфичности обнаружения целевых видов из экологической выборки. В этом протоколе мы очертим шаги, необходимые для разработки и тестирования зондных анализов для обнаружения целевого вида; включая создание баз данных последовательности, проектирование анализа, выбор и оптимизацию анализа, производительность анализа тестирования и проверку поля. Следующие эти шаги помогут достичь эффективного, чувствительного и конкретного анализа, который может быть использован с уверенностью. Мы демонстрируем этот процесс с нашей анализа, предназначенные для популяций mucket (Actinonaias ligamentina), пресноводных мидий видов, найденных в реке Клинч, США.
Введение
Исследователи и менеджеры все больше интересуются использованием экологических анализов ДНК для обнаружения видов. В течение трех десятилетий, количественные или в режиме реального времени ПЦР (qPCR / rtPCR) был использован во многих областях для последовательности конкретных обнаружения и количественнойоценки нуклеиновых кислот 1,2. В относительно новой области исследований eDNA использование этих анализов со стандартной кривой для количественной оценки копий целевой ДНК на объем или вес образца eDNA в настоящее время стало обычной практикой. Митохондриальные последовательности ДНК, как правило, нацелены на анализ eDNA, потому что митохондриальный геном присутствует в тысячах копий на клетку, но также возможны анализы на ядерную ДНК или РНК. Важно понимать, что опубликованные анализы образцов eDNA не всегда равны по производительности. Надежность анализа в обнаружении только ДНК целевого вида (т.е. специфичность) и обнаружении небольших количеств целевой ДНК (т.е. чувствительности) может значительно различаться из-за различий в том, как анализ был разработан, выбран, оптимизирован и протестирован. Отчетность количественных показателей анализа производительности ранее в значительной степени упускается из виду, но в последнее время стандарты для повышения прозрачности ванализе развития появляются 3,4,5,6,7,8.
Оптимизация и отчетность по анализу эффективности помогает в разработке исследований и интерпретации результатов опроса eDNA. Анализы, что кросс-реакция с нецелесоприятной ДНК видов может привести к ложноположительных обнаружений, в то время как анализы с плохой чувствительностью может не обнаружить ДНК целевого вида, даже если он присутствует в образце (ложные негативы). Понимание чувствительности и избирательности анализа поможет определить усилия по отбору проб, необходимые для выявления редких видов. Поскольку существует много естественных источников вариаций в eDNA, исследования должны максимально ограничить контролируемые источники вариаций, включая полную оптимизацию и характеристику анализаeDNA 3.
Условия, непосредственно влияющие на специфику или чувствительность анализа, изменят производительность анализа. Это может происходить в различных лабораторных условиях (т.е. различных реагентов, пользователей, машин и т.д.). Поэтому этот протокол следует пересмотреть при применении анализа в новых условиях. Даже анализы, хорошо охарактеризованные в литературе, должны быть проверены и оптимизированы при принятии новой лабораторией или при использовании различных реагентов (например, мастер-микс решение)5,9. Специфика анализа может измениться при применении к другому географическому региону, потому что анализ применяется к образцам из нового биотического сообщества, которые могут включать нецелеприемные виды, против которые не были протестированы, и могут возникнуть генетические различия в целевых видах. Опять же, анализ должен быть повторно оценен при использовании в новом месте. Полевые условия отличаются от лабораторных условий тем, что в полевых условиях ингибиторы ПЦР с большей вероятностью присутствуют в образцах. Ингибиторы ПЦР непосредственно влияют на реакцию усиления и тем самым влияют на производительность анализа. По этой причине при разработке анализа eDNA необходим внутренний положительный контроль.
Наконец, условия окружающей среды в полевых условиях могут влиять на молекулы ДНК целевого вида и их захват в результате деградации, транспортировки и удержания ДНК. Кроме того, различные протоколы сбора и извлечения ДНК различаются по эффективности и способности удерживать ДНК. Однако важно отметить, что эти процессы влияют на обнаруживаемость eDNA, но не на производительность молекулярного анализа. Таким образом, обнаруживаемость ДНК целевого вида в полевых образцах является функцией как технических характеристик анализа qPCR, так и полевых условий и протоколов сбора, хранения и извлечения. При использовании хорошо охарактеризованных и высокую проготверхаем анализа, пользователи могут чувствовать себя уверенно в возможностях анализа; позволяя исследователям теперь сосредоточиться на понимании внешних факторов анализа (т.е. экологических переменных, различий в протоколах захвата или извлечения), влияющих на обнаружение eDNA.
Здесь мы ориентируемся конкретно на анализ технических характеристик путем тщательного проектирования и оптимизации. Мы демонстрируем протокол с помощью зонда на основе анализа, разработанного для обнаружения пресноводных мидий,mucket (Actinonaias ligamentina), из воды, пробы в реке Клинч, США. Недавно Thalinger et al. (2020) представила руководящие принципы для проверки целевых анализов eDNA. Анализ дизайна после нашего протокола принесет анализ на уровне Thalinger и др. плюс дополнительный шаг к уровню 56. На этом этапе технические характеристики анализа будут оптимизированы и он будет готов к регулярному использованию в лабораторных и полевых приложениях. Дальнейшее использование анализа в лабораторных, мезокосмных и полевых экспериментах может затем решить вопросы, касающиеся обнаружения eDNA и факторов, влияющих на обнаруживаемость, заключительные шаги для проверкиуровня 5 6.
протокол
1. Генерация последовательной базы данных митохондриальных последовательностей ДНК из целевых и нецелеприемных видов, представляющих интерес
- Определите вопрос, цели и систему, которая решается. Определите целевые виды для обнаружения eDNA. Определите географическую систему, в которой будет использоваться анализ. Составной список видов, представляющих интерес, в том числе целевых видов, симпатических (сопутствующих) видов в пределах той же таксы (обычно порядка или семейного уровня), и тесно связанных всеопатрических видов, те, которые не могут быть в том же географическом положении, как цель (Рисунок 1).
ПРИМЕЧАНИЕ: Здесь, clinch реки популяций вида A. ligamentina были мишенью. - Поиск и загрузка последовательностей из нескольких областей генов для видов в списке из шага 1. Можно использовать базы данных последовательности, такие как NCBI (Национальный центр биотехнологической информации), BOLD (Barcode of Life Database), EMBL (Европейская лаборатория молекулярной биологии) и DDBJ (Dna Data Bank of Japan). NCBI, EMBL и DDBJ делятся информацией о последовательности.
- Используя нуклеотидную базу данных NCBI, ищите целевой организм (например, эктинонайасский связкин)и область генов (например, цитохром c оксидазы I (COI) или NADH-дегидрогеназу 1 (ND1); Пример строки поиска: Actinonaias связки и ND1)
- Далее выберите все последовательности, которые соответствуют спецификациям, и выберите Отправить. Выберите Полную запись, файл и формат загрузки, как GenBank или FASTA, а затем создать файл. Эти последовательности теперь сохраняются на компьютере.
- Повторите эти шаги для всех видов в списке, определенном в шаге 1. Храните последовательности для каждого региона гена в отдельном файле, так как они будут анализироваться отдельно.
- Загрузите все соответствующие последовательности (или большую репрезентативную пропорцию последовательностей) для целевых видов, выявленных в шаге 1. Включите географические варианты, если это возможно.
- Повторите последовательности поиска и загрузки для связанных и симпатических нецелесообразных видов той же таксономической группы, которые были определены в шаге 1 (например, если целевым видом является mucket(A. ligamentina) скачать последовательности для всех других пресноводных видов мидий в Family Unionidae, которые происходят в системе интересов).
- Повторите поиск и загрузку для тесно связанных, но всеопатрических (географически отдельных) видов, перечисленных в шаге 1.1.
ПРИМЕЧАНИЕ: Не все виды (цели и не цели) будут доступны в общедоступных базах данных. Увеличьте лока локаную справочную базу данных путем усиления и секвенирования таксономически проверенных образцов видов, представляющих интерес для дома. Если вы работаете с видом, который имеет высокое генетическое разнообразие внутри видов или работает в географически большой области, где можно ожидать географических вариантов, соберите последовательности со всего диапазона.
2. Дизайн анализа
- Выравнивание последовательностей из каждой области гена отдельно с помощью выравнивания программного обеспечения, которое можно найти в различных генетических редактирования последовательности и биоинформатических программ. Сделай это выравнивание для каждого из различных областей генов.
- Например, с помощью программного обеспечения Geneious Prime (https://www.geneious.com) импортируют загруженные файлы последовательности в программу.
- Создание отдельных папок для каждого региона гена.
- В папке, которая содержит последовательности из одной области гена, выберите все последовательности.
- Используйте инструмент множественного выравнивания для создания нуклеотидного выравнивания выбранных последовательностей. Там может быть несколько вариантов для типа выравнивания, используя Geneious или MUSCLE выравнивания и параметры по умолчанию работает хорошо.
- Выберите перспективные регионы для анализа дизайна с помощью визуализации выровненных данных последовательности. Регион, который имеет много данных о последовательности, доступных для видов, представляющих интерес, сильно расходится между видами, и показывает низкие внутри вида изменения является хорошим кандидатом. Это увеличит вероятность того, что разработанные грунтовки и зонды смогут различать цель от нецелесоцелего вида, обеспечивая при этом усиление внутривидового варианта с помощью анализа.
- Дизайн анализов праймеров и зонда.
- Используйте программное обеспечение для анализа qPCR и следуйте инструкциям. Инструмент IDT Primer'st (https://www.idtdna.com/) для разработки 5 комплектов анализов qPCR был использован здесь.
- Вставьте последовательность, выбранную в шаге 2.2, в поле входа последовательности. Если выравнивание создало пробелы, удалите их из последовательности.
- Выберите qPCR 2 Праймеры и зонд в опции «Выберите свой дизайн».
- Скачать рекомендуемые анализы.
- Копировать последовательности из форвардной праймер первого анализа и искать эту последовательность грунтовки в выравнивании, созданном в шаге 2.1.4. При использовании Geneious Prime используйте инструмент Annotate and Predict, чтобы добавить область грунтовок в выравнивание. Сделай это для всех комбинаций грунтовок и зондов(рисунок 2).
- Проинспектировать эти области выравнивания для изменения в целевых видов, а также в рамках со-происходящих видов.
- Если есть внутриспецифические генетические изменения, поиск анализов, где грунтовки и зонд не попадают в эти регионы.
- Для предотвращения нецелевого усиления видов, поиск несоответствий с нецелесоцей видов. Выберите анализы с большинством несоответствий для дальнейшей проверки. Currier et al. (2018) предлагают выбрать наборы, по крайней мере, с двумя из трех регионов (два грунтовки, или грунтовка и зонд), имеющие по крайней мере два несоответствия со всеми нецелесоприимных видов. Однако имейте в виду, что несоответствия на зонде вносят меньший вклад вспецифичность 10.
ПРИМЕЧАНИЕ: Различия в пределах 3 базовых пар 3' конца каждой грунтовки увеличивают специфичность лучше, чем различия в конце 5' праймеров10.
- Рассмотрим следующие важные параметры в дизайне анализа.
- Определите температуру плавления и облагования грунтовок и зонда. В идеале температура плавления (Tm) грунтовок должна быть между 60-64 градусами По Цельсию и в пределах 2 градусов друг от друга, а Тм зонда должен быть на 6-8 градусов выше, чем Tm праймеров. Установите температуру аннеаляля (Ta) реакции qPCR на 5 градусов ниже температуры плавления, около 55-60 градусовпо Цельсию 11.
- Изучите содержимое GC. Выберите между 35 - 65% содержанием GC, и избегайте регионов с 4 или более последовательными Gs. Имея 1 или 2 Gs или Cs в 5 последних баз 3 'конец грунтовки (GC зажим) может увеличить специфичность, как это поможет грунтовки, чтобы сделать сильнеесвязи 12.
- Поиск шпильки и димер структур. Тест праймеры и зонд для прогнозируемых структур шпильки и димеры с помощью олигонуклеотида анализа программы (например, OligoAnalyzer-IDT13; ОлигоКальцулятор14). Эти структуры могут привести к нецелевому усилению и снижению эффективности. Избегайте анализов, которые, по прогнозам, образуют эти структуры.
- Определите длину грунтовок. Цель для грунтовки между 18-25 баз в длину и длина зонда между 20 -25 баз. Более длинные грунтовки и зонды могут иметь более низкую эффективность усиления.
- Определите длину ампликона. Она должна быть между примерно 100 и 250 базовых пар. Этот диапазон, как правило, достаточно короткий для высокой эффективности ПЦР, но достаточно долго для удобства проверки Sangerпоследовательности 4,15.
- Дизайн зондов. Убедитесь, что зонды не имеют базы G на 5 'конец, потому что это может ослабить сигнал от зеленых и желтых красителей11. Мы разработали двойные затухаемые зонды с флюорофорами IDT 3IABkF и ЗЕН, а также флюорофорами FAM или HEX.
ПРИМЕЧАНИЕ: Определите MGB зонды: TaqMan MGB (незначительный паз связующего) зонды часто используются для исследований eDNA. Однако, поскольку эти зонды очень короткие, они могут связываться с не-целей даже с 2 или 3 базовой пары несоответствие10. - Определить температуру зонда Tm. Температура плавления зонда должна быть на 6-8 градусов выше, чем у грунтовок. Более низкие температуры снижают связывающий успех зонда.
- Определите длину и местоположение зонда. Зонд должен быть между 20 и 25 bp в длину и идеально расположен близко к грунтовки связывания сайта на той же нити без перекрытия его.
3. Анализ скрининга и оптимизации
- В силико анализа разработки и тестирования. Перед заказом наборов грунтового зонда оцените специфичность (потенциальное нецелевое усиление) путем тестирования усиления грунтовок в силико.
- Тест праймеры через NCBI в Primer-Blast16 или аналогичных программ, которые могут определить потенциальные не-цели в ncBI nt/nr базы данных, которые могут усилить с анализом. При использовании Primer-Blast вставить грунтовки на использование моей собственной грунтовки поле под параметрами Праймер. В вариантах проверки параметров Primer Pair, выберите nr в качестве базы данных и ввярете Порядок организма, представляющий интерес (например, "Unionida" или "Unionoida") в поле Organism.
- Продолжайте оценивать наборы грунтовок/зондов визуально на выровненных данных последовательности.
- Для того, чтобы оценить грунтовки и зонды одновременно в силико, создайте текстовую строку вперед грунтовки, 12 N, зонд, 12 N, и обратное дополнение обратной грунтовки. Если последовательность зонда находится в пределах 12 базовых пар одной из грунтовок, используйте число N, соответствующее количеству базовых пар между грунтовки и зонда.
- Используйте NCBI Nucleotide Доменная поиск (Blastn) для поиска в отношении базы данныхNR 17. Используйте вкладку Таксономия для ищите нецелесоцей видов с несколькими несоответствиями; они должны быть протестированы в лаборатории во время оптимизации анализа.
ПРИМЕЧАНИЕ: В силико тестирование помогает исключить неспецифические анализы, но потенциально конкретные анализы должны быть проверены эмпирически (in vitro), так как не все виды имеют последовательности в генетических базах данных и грунтовки и зонды все еще могут связываться с не-целей, даже если считается маловероятным программного обеспечения.
- Выберите три-пять комбинаций грунтовка/зонд для тестирования в лаборатории.
- Закажите грунтовки, зонды и синтетический стандарт ДНК, а также дополнительные M13-хвостовые грунтовки для секвенирования ампликона.
- Закажите синтетические олигонуклеотидные грунтовки и зонды у компании, которая производит олиго. Зонды маркируются флуоресцентным красителем и утолением. Различные фторфоры должны быть выбраны для анализа, которые должны быть мультиплексированы. Проверьте свой qPCR инструмент для списка, из которых фторфоры инструмент может обнаружить.
- Проектирование и заказ M13-хвостовые грунтовки для проверки обнаружения qPCR с Sanger секвенирования, добавив M13 Вперед (-20) последовательность, GTA AAA CGA CGG CCA GT, к 5 'конец вперед грунтовки, и M13 Обратный (-27) последовательность, CAG GAA ACA GCT ATG AC, к 5 ' конец обратного грунта.
- Синтетический стандарт ДНК содержит целевую последовательность (включая области грунтовки) при известной концентрации в копиях/хл. Количественная оценка неизвестных образцов на основе кривой, сделанной известными концентрациями этого стандарта (т.е. стандартной кривой). Приобрети синтетический стандарт у той же компании, которая производит грунтовки и зонд. Следуйте рекомендациям производителя для повторного получения и хранения. Разбавить стандарты в буфере TE с помощью тРНК-носителя с использованием низкоухозяющего пластикового программного обеспечения для уменьшения гидролиза и связывания с поверхностями.
ПРИМЕЧАНИЕ: Если стандартная кривая не работает хорошо (плохая эффективность ПЦР, см. шаг 3.4.2), попробуйте повторно приостановить стандарт в воде или Tris-HCl. - Приостановить грунтовки и зонды в нуклеазной свободной воде, Tris-HCl, или TE буфера в удобных концентрациях для анализа использования. Как правило, разбавлять рабочие запасы 20 раз в мастер-микс для достижения оптимизированной окончательной концентрации анализа. Хранить приостановлено oligos при постоянной -20 градусов по Цельсию, когда не используется.
- In vitro (в лаборатории) анализ оптимизации и тестирования. Отклонить анализы, которые имеют низкую эффективность, перекрестное реагирование с ко-происходящих видов, или имеют плохую чувствительность18. Включите использование внутреннего положительного контроля (IPC) при разработке анализа, а также при запуске фактических образцов.
- Во-первых, найти оптимальную температуру и значения концентрации грунтовка/зонда для анализа. После оптимизации этих параметров для эффективности ПЦР (шаг 3.4.2), перекрестной реактивности (шаг 3.4.3) и чувствительности (шаг 3.4.4), приступить к тестированию анализа с мультиплексом IPC (шаг 3.4.5).
- Проверьте оптимальную температуру аннеалирования (Ta) для грунтовки и зондов с использованием градиента температуры ПЦР, центрированного на 5 градусов по Цельсию ниже прогнозируемого среднего грунтовки Tm.
- Проверьте оптимальную концентрацию грунтовки и зонда. Как правило, проверяются концентрации 200 нм, 400 нм и 800 нм грунтовки и 75 нм, 125 нм и 200 нм концентраций зондов.
- Создайте стандартную кривую и определите эффективность и линейный диапазон. Проверьте по крайней мере шесть 10-кратного разбавления синтетического стандарта ДНК, содержащего целевую последовательность, примерно в10 0 экземплярах/реакциидо 10 5 копий/реакции(рисунок 3A).
- Используйте программное обеспечение qPCR для построения значения Cq (порог цикла при количественной оценке) каждого стандарта на оси y и базы журнала 10 исходной стандартной концентрации в копиях/реакции на x-оси. Программное обеспечение qPCR должно автоматически запускать линейную регрессию(рисунок 3B).
- Рассчитайте эффективность наклона регрессии, E -1 и 10(-1/slope). Например, если наклон -3,4, эквалайзер -1 , 10(0,29) - 0,97 или 97%. Также проверьте значения r2, которые показывают, насколько хорошо стандартные репликации вписываются в кривую. Программное обеспечение qPCR должно автоматически вычислять это также(рисунок 3B). Цель для значения эффективности 100% (±10%) и r2 значения ≥0,989,15,19,20,21,22.
- Визуально проинспектировать стандартную кривую на предвзятость, то есть отклонения от регрессии в последовательном направлении или на плохую стандартную производительность кривой, измеряемуюэффективностью и значениями r2 (рисунок 3C и 3D).
- Специфика: Оцените кросс-реактивность с нецелемных видов, чтобы уменьшить вероятность ложных срабатываний. В тех случаях, когда обнаружение eDNA может привести к дорогостоящим управленческим решениям, проаналивация положительных обнаружений с помощью последовательности amplicon.
- Немишени: Прогон анализа геномной ДНК извлечения таксономически проверенных образцов родственных видов и географически сопутствующих видов; с наивысшим приоритетом является тестирование на тесно связанные, сопутствующие виды. Используйте аналогичные общие концентрации ДНК как для целевых, так и для нецелесочных образцов. Выбранная концентрация должна дать усиление из образцов целевых видов в середине линейного диапазона стандартной кривой. Усиление следует наблюдать только с целевыми видами.
- При наблюдении нецелевого усиления очистите и последовательность продукта для подтверждения его идентичности. Нередко наблюдается загрязнение целевых видов в образцах тканей нецелевых видов, поэтому все усиления на данном этапе должны быть проверены секвенированием. Reamplify очищены amplicons от специфики испытаний с использованием M13 хвостатые грунтовки и последовательности с M13 грунтовки.
- В лаборатории после ПЦР перенесите продукты кЗКПЧ, которые будут секвенированы, на свежие трубки. Удалите остаточные грунтовки и компоненты реакции с помощью комплекта очистки (например, комплект очистки ПЦР MinElute).
- Сделайте 1:100 разбавления элюций и усиливай 1 кл каждого в течение 30 циклов в 50 хл ПЦР реакции с M13 хвостатые грунтовки и высокой точностью полимеразы (например, Phusion High-Fidelity ДНК полимеразы).
- Вы запустите 10 йл каждой реакции на 1% агарозный гель, чтобы проверить на одну полосу ожидаемого размера. Если полоса не наблюдается, увеличьте количество циклов или количество выборки. При наблюдении нескольких полос гель очищает полосу ожидаемого размера.
- Удалите остаточные грунтовки и компоненты реакции с помощью комплекта очистки, как указано выше, и измерьте концентрацию ДНК элюционов.
- Настройка реакций секвенирования с помощью праймеров M13 в соответствии с инструкциями секвенирования объекта.
ПРИМЕЧАНИЕ: Никогда не ввешать усиленные образцы в лаборатории qPCR. Подготовка образцов для секвенирования в лаборатории, посвященной образцам пост-ПЦР.
- Чувствительность: Чувствительность влияет на вероятность ложных негативов, или неудачи для обнаружения ДНК целевого вида, когда она присутствует. Оцените предел обнаружения (LOD) и предел количественной оценки (ЛОЗ) для каждого анализа. Наконец, включите внутренний положительный контроль (МПК) для оценки ингибирования ПЦР образцов. Мультиплекс и проверить этот анализ IPC с разработанной анализа для обеспечения двух анализов не мешают друг другу.
- LOD: Сделайте шесть 4-кратных серийных разбавлений синтетического стандарта ДНК, с 8-24 репликациями на стандартное разбавление(рисунок 4). Рассчитайте самую низкую начальную концентрацию с обнаружением 95%. Участки LOD и LOЗ могут быть сгенерированы с помощью калькулятора LOD/LO' Rскрипта 5.
ПРИМЕЧАНИЕ: Данные ниже LOD не должны подвергаться цензуре. Из-за специфики ПЦР, нет нижнего предела для истинных срабатываний. LOD является самой высокой концентрацией, ниже которой можно ожидать ложных негативов. - ИЗ той же серии разбавления вычислите самую низкую начальную стандартную концентрацию ДНК, поддающуюся количественной оценке, с коэффициентом изменения (CV) ниже 35%.
ПРИМЕЧАНИЕ: LOD и ЛОЗ должны быть сообщены в копиях/реакции. При использовании проверенного анализа и полевых образцов усиливаются ниже ЛОЗ, результаты должны быть сообщены как % обнаружения, а не концентрации eDNA, потому что точная концентрация не может быть измерена суверенностью 5.
- LOD: Сделайте шесть 4-кратных серийных разбавлений синтетического стандарта ДНК, с 8-24 репликациями на стандартное разбавление(рисунок 4). Рассчитайте самую низкую начальную концентрацию с обнаружением 95%. Участки LOD и LOЗ могут быть сгенерированы с помощью калькулятора LOD/LO' Rскрипта 5.
- Используйте внутренний положительный контроль (IPC) для тестирования на ингибирование ПЦР. Ингибирование может привести к снижению чувствительности и ложных негативов. Проверьте способность анализа МПК быть мультиплексированы с целевым анализом.
- Анализ IPC может быть мультиплексирован с помощью анализа цели с помощью зонда с другим красителем репортера, чем анализ цели. Этот анализ IPC состоит из короткой синтетической последовательности ДНК от вида, не связанного с целевой таксой, включенной в мастер-микс qPCR при низкой концентрации приблизительно 102 экземпляров/реакции, наряду с грунтовой и зондами, которые обнаруживают его. Эта более низкая концентрация необходима, чтобы избежать конкуренции с целевой последовательностью полимеразы и нуклеотидов23.
- Сравните значение IPC шаблона Cq с значением шаблона IPC в элементе управления шаблоном. В этом нет шаблона управления (NTC), только вход ДНК является то, что шаблон IPC. Шаблон МПК в этой реакции должен усилиться, как и ожидалось. Если шаблон IPC в образце усиливается на 2 или более циклов, отличается от шаблона IPC в NTC, образец eDNA тормозится. Образцы, которые показывают ингибирование могут быть разбавлены 1:10 и повторно протестированы. Если образец остается ингибируется, этот образец должен быть удален из анализа.
- Во-первых, найти оптимальную температуру и значения концентрации грунтовка/зонда для анализа. После оптимизации этих параметров для эффективности ПЦР (шаг 3.4.2), перекрестной реактивности (шаг 3.4.3) и чувствительности (шаг 3.4.4), приступить к тестированию анализа с мультиплексом IPC (шаг 3.4.5).
- Разработка и тестирование на месте
- В лаборатории: Если доступ к организму в лаборатории, а также симпатрных видов имеется; взять пробы воды из вольеров с этими видами, обрабатывать образцы и проверить анализ на эти образцы eDNA. Последовательность продуктов, как указано выше, чтобы проверить усиление намеченной цели с помощью M13 хвостатые грунтовки.
- В полевых условиях:
- Определите места, где, как известно, происходит организм-мишень, и, как известно, этого не произойдет. Предпочтительно иметь некоторую меру изобилия в каждом месте, где встречается целевой вид.
- Решите, какие объемы выборки и методы сбора проб (например, фильтрация, центрифуга и т.д.) будут использоваться.
- Включите поле пустой или отрицательный контроль на каждом сайте, это чистая вода, которая была доставлена на место поля, а затем собраны и подготовлены с тем же оборудованием поля и протоколов, используемых для eDNAвыборки 24. Цель полевого пробела заключается в обнаружении загрязнения оборудования для отбора проб и полевого снаряжения, доставленного на объект. Возьмите поле пустым перед обработкой проб воды поля.
- Возьмите несколько проб воды на объект, предпочтительно 3 пробы на объект.
- Вернуться в лабораторию, процесс и извлечь образцы.
- Вы запустите анализ с помощью пластины, настроенной по аналогии с рисунком 5A, и сравните частоту концентрации и обнаружения eDNA с известными различиями в возникновении и изобилии сайта. Подтвердите все обнаружения с помощьюпоследовательности 24,25.
ПРИМЕЧАНИЕ: Вышеуказанная проверка анализа через уровень 4 шкалы Thalinger et al. (2020)(оптимизация технических характеристик анализа) и начнет собирать данные, подтверждающие проверку анализа уровня 5. Уровень 5 включает в себя моделирование вероятности и использование анализа для исследований экологии eDNA. Мы считаем, что это выходит за рамки базовой разработки анализа, но мы поощряем эти применения лабораторных и полевых проверенных анализов для улучшения анализа дизайна и интерпретации данных.
Результаты
При проектировании видового специфического qPCR-анализа для гадости(A. ligamentina),были загружены доступные последовательности всех видов Unionidae в реке Клинч. Близко связанные виды, такие как Lampsilis siliquoidea, также были включены в справочную базу данных, даже если они не находятся в одной реке. Не все виды в системе реки, представляющие интерес были найдены в GenBank, так что дополнительные виды были секвенированы в доме. Последовательности были выровнены с помощью программного обеспечения Geneious и программного обеспечения Primer Квест (IDT) был использован для разработки нескольких анализов. Пять наборов грунтовок и зонда были добавлены в выравнивание для визуальной оценки(рисунок 2). Затем они были протестированы в силико с помощью Primer-Blast, после чего они были заказаны для дальнейшего тестирования в пробирке. В лаборатории были протестированы все анализы с использованием ДНК-извлечения 27 доступных видов для проверки специфичности. Один анализ (A.lig.1) успешно усиливается только целевых видов(таблица 1; Таблица 2). Этот анализ продвинулся вперед для дальнейшего тестирования эффективности анализа, LOD и ЛОЗ. Он имеет длину ампликона 121 базовых пар. Таблица 3 показывает последовательность, используемую для стандарта синтетической ДНК A. ligamentina. Рисунок 3A и рисунок 3B показывают результаты успешного анализа с хорошей эффективностью и значениями r2. Рисунок 3C и рисунок 3D показывают анализ, стандартная кривая которого имеет низкую эффективность; этот анализ был отброшен. LOD и ЛОЗ для выбранного анализа (A.lig.1) были найдены, чтобы быть 5.00 копий / реакции с использованием дискретного метода, описанного в Klymus et al5. IPC, который был мультиплексирован с анализом(таблицы 3-6) не влияет на стандартную кривую анализа A. ligamentina. IPC мы используем фрагмент мыши HemT стенограммы. Этот анализ был предварительно разработанны IDT для другого приложения, но мы изменили его использование в качестве IPC для eDNA приложений нашей лаборатории.
Успешный запуск qPCR должен соответствовать определенным критериям для каждой меры производительности (т.е. стандартное усиление кривой, положительный контроль геномной ДНК, отсутствие контроля шаблонов и внутреннего положительного контроля). Целевые стандарты анализа должны иметь экспоненциальные кривые усиления. Эти кривые должны достичь плато конечных точь, если разрешено запустить достаточно циклов. Это свидетельствует о том, что флуоресцентный зонд полностью потребляется во время реакции, а уровень флуоресценции достигает максимального предела. Более поздние усиливающие стандарты могут не достичь плато в 40 циклов. Положительные элементы управления (геномная ДНК и IPC) должны иметь одинаковую модель. Неизвестные могут усиливаться или не усиливаться, но усиление в неизвестных также должно иметь экспоненциальную закономерность и плато конечной точки(рисунок 5).
В качественном qPCR стандартные разбавления усиливаются при равномерном пространстве Cq приблизительно каждые 3,3 цикла для каждой 10-кратной разницы в концентрации. Каждая репликация стандартного разбавления усиливается плотно сгруппированным образом, имея почти тот же Cq (представленный значениями r2). Все стандартные разбавления должны проявляться усилением(рисунок 3A). В плохом qPCR, стандарты могут проявлять не экспоненциальную форму, неравномерное изменение значений Cq между разбавлениями, не приходят к плато конечной точки, или некоторые разбавления не могут усилитьсявообще (рисунок 3D).
Важными параметрами стандартной кривой являются эффективность, r2,наклон и y-перехват. Эффективность должна падать между 90%-110% при идеальных значениях около 100% и r2 значения должны быть выше 0,98 с идеальными результатами приближается 1,015,22. Значения склона должны быть между -3.2 и -3.5 с идеальными результатами около -3.322. Значения y-intercept должны упасть между Cq 34-41 с идеальными результатами, имея Cq 37.0. Y-перехват является прогнозируемым Cq реакции с 1 копией целевой последовательности, самый маленький блок, который может быть измерен в одном qPCR. Неизвестные с Cq больше, чем у-перехвата, вероятно, будет тормозится. Запуск более 40 циклов ПЦР может быть необходимо для обнаружения цели в случае торможения или неэффективного набора грунтовки, однако количественная оценка невозможна в этих обстоятельствах и дополнительные негативные меры контроля без целевой последовательности, но содержащие общую ДНК, похожую на неизвестные, должны быть запущены, чтобы исключить усиление из неспецифических источников.
Усиление внутреннего положительного контроля (ИПК) в неизвестных образцах следует сравнивать с результатами отрицательного контроля шаблонов IPC, поскольку нет конкуренции за реагенты и нет ингибиторов. Неизвестные с IPC, имеющих Cq 2 циклов или больше, чем среднее значение Cq NTC, или которые не усиливают следует считать ингибируется. Если в образцах нет ингибиторов, то все усиление IPC должно иметь плотную группировку в участке с значениями Cq рядом с тем же, что и NTC(рисунок 6).
Наконец, на месте произошло тестирование анализа. Двадцать проб воды из реки Клинч и три полевых пустых образца были отфильтрованы в период с 25 по 26 сентября 2019 года в 500 метрах от мидии кровать, как известно, A. ligamentina. Приблизительно четыре пробы воды 1 л были отфильтрованы в месте отбора проб. Расположение сайтов включены в нижней части мидии кровать в потоке, дно мидии кровать возле берега, 100 м вниз по течению от кровати в потоке, 500 м вниз по течению от кровати в потоке и 500 м вниз по течению от кровати возле берега(рисунок 7). Вернувшись в лабораторию, каждый фильтр был разрезан пополам, а ДНК извлекается только из половины фильтра. Оставшаяся половина фильтра для каждого образца хранилась в морозильной камере -80 градусов по Цельсию. Образцы были затем запущены с помощью A.lig.1 анализ мультиплексов с IPC. Из 23 образцов было установлено, что пять проб были ингибированы. Эти образцы были разбавлены 1:10 и разбавления были повторно запущены. Девятнадцать из 20 образцов поля усилены с помощью разработанного анализа. Из этих 19 образцов пять были выше loD анализа и ЛОЗ 5 экземпляров/реакции; это означает, что большинство образцов имели обнаружение eDNA, но на уровне, когда могут иметь место ложные отрицательные результаты и что анализ не мог с уверенностью определить номер копии для этих 14 образцов. Тем не менее, от 75 до 100% из четырех биологических реплицирует участок, усиленный в каждом месте отбора проб. Два из трех пробелов поля были отрицательными, в то время как одно поле пустое действительно показывают усиление, подчеркивая важность чистой техники в этой области.

Рисунок 1: Рабочий процесс для строительства базы данных митохондриальной последовательности ДНК. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.

Рисунок 2: Последовательность выравнивания для clinch речных мидий видов с потенциальными грунтовки и зонды для Actinonaias ligamentina ND1 анализа. Вперед грунтовки в темно-зеленый, зонд в красном и обратном грунтовки в светло-зеленый. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
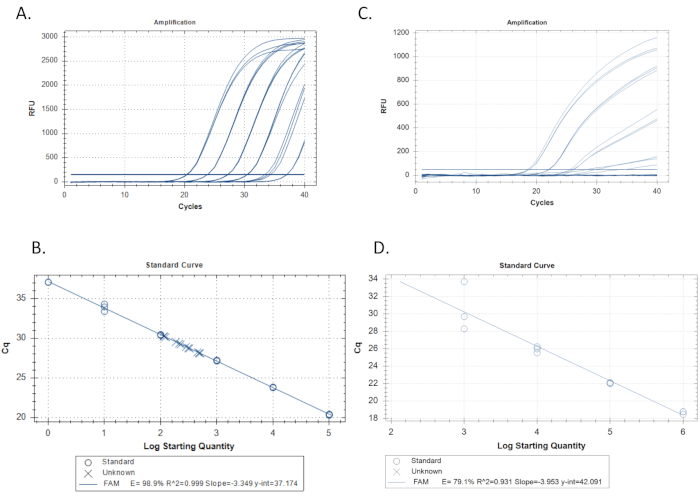
Рисунок 3: Примеры стандартной кривой и линейной регрессии. Примерприемлемой стандартной кривой, полученной из усиления трех репликаций каждого из шести стандартных разбавлений. 10-кратная стандартная серия разбавления с самой высокой концентрацией стандарта слева, с уменьшением концентраций, движущихся вправо. Горизонтальная линия, пересекающая все следы, является порогом цикла при количественной оценке (Cq). Где каждый след пересекает этот порог, где определяется Cq. B.Линейная регрессия, сделанная из стандартных репликаций рисунка 3A. Репликации стандартных разбавлений построены по кругу, а неизвестные (образцы) - с x's. Эффективность составляет 98,9%, r2 приближается к 1.0, и наклон -3.349. C.Пример плохой стандартной кривой, полученной от усиления трех репликаций каждого из шести стандартных разбавлений. D.Линейная регрессия, образуя стандартную кривую для стандартных репликаций, усиливается в примере 3C. Обратите внимание на низкую эффективность и значения r2. Также обратите внимание, что только 4 из 6 стандартов усиливается. Если после повторных запусков, стандартная кривая не улучшается, проблема может быть с плохой грунтовки / зонд набор, который не усиливает целевую ДНК, как ожидалось, в этом случае, этот анализ не следует рассматривать. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
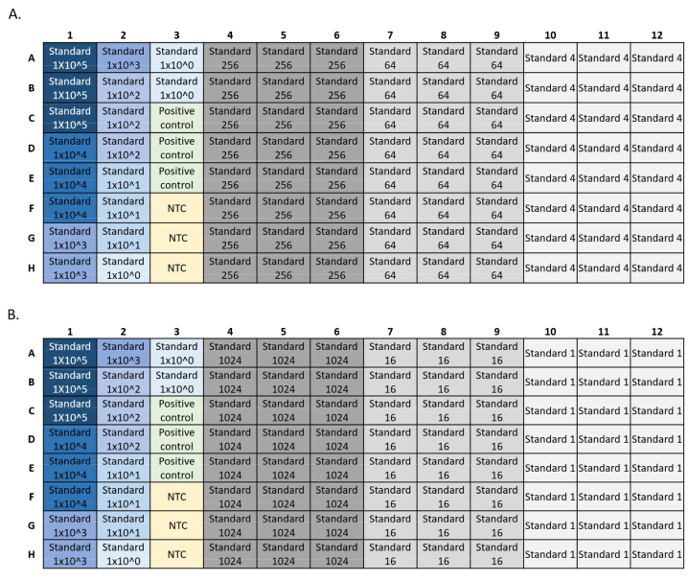
Рисунок 4: Примеры установок пластин для стандартных qPCR-запусков LOD и LO. Стандарты, используемые в кривой, в синем цвете, стандартная концентрация уменьшается от темно-синего до светло-голубого. Положительный контроль ДНК в зеленом цвете и отсутствие контроля шаблонов (NTC) желтым цветом. Экспериментальные стандартные концентрации серого цвета, показывающие 24 репликации для каждого стандартного разбавления. Серия разбавления была помына на две пластины (A, B), каждая со стандартной кривой, положительным контролем и NTC. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
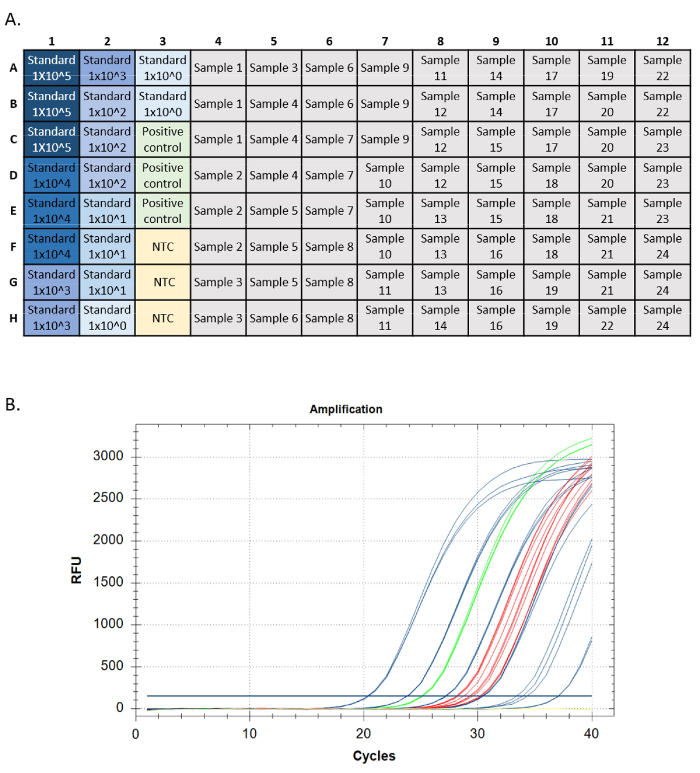
Рисунок 5: Настройка плиты и следы усиления от запуска qPCR. Установкаплиты, стандарты показаны синим, более темным цветом, указывающим на самую высокую концентрацию стандарта. Положительный контроль ДНК в зеленом цвете, отсутствие контроля шаблонов желтым цветом (NTC), выборка целей в сером цвете. B.Следы усиления от запуска qPCR. Стандарты, показанные синим цветом, положительный контроль ДНК в зеленом цвете, отсутствие элементов управления шаблоном в желтом и неизвестный красный. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
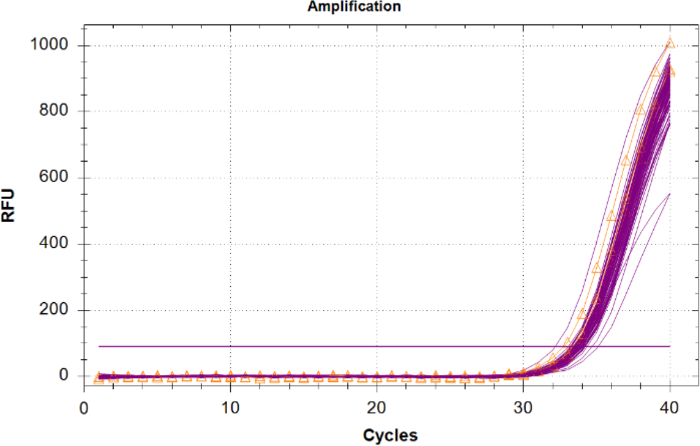
Рисунок 6: Следы усиления для внутреннего позитивного контроля (IPC). IPC следы для всех неизвестных образцов в пурпурный и IPC от нет шаблона управления (NTCs), показанный в оранжевом с треугольниками. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
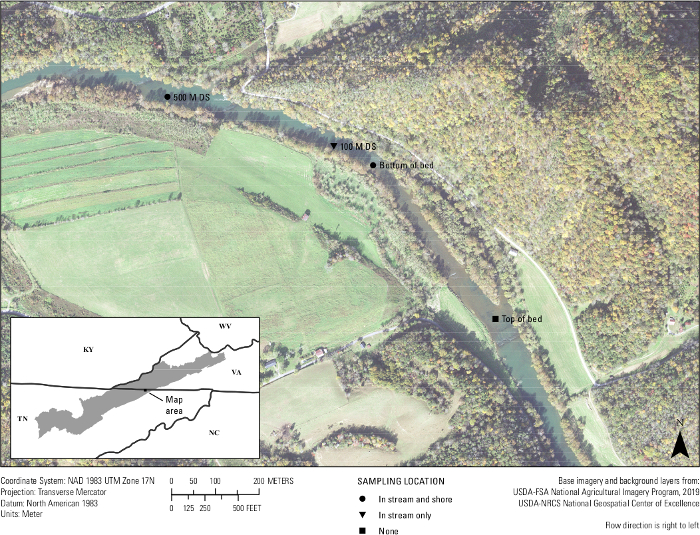
Рисунок 7: Карта, показывающая места сбора eDNA мидий в реке Клинч вдоль границы Вирджинии/Теннесси. Образцы были собраны в Wallens Bend в нижней части кровати, 100 м ниже по течению от кровати, и 500 м вниз по течению от кровати. Участки были либо собраны в середине ручья (в потоке), либо примерно в 1 - 2 метрах от береговой линии (берега). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| компонент | имя | Последовательность 5' - 3' | Флуоресцентная этикетка | |
| Форвард Праймер | A.lig.1-f | CCCTCATCACGTACCTCTTAATC | ||
| Обратный праймер | A.lig.1-r | ГГААТГККАТКАТКААКТТТА | ||
| зонд | Зонд A.lig.1 | TTCTTGAACGTAAAGCCCGGT | ФАМ | |
Таблица 1: Разработано Actinonaias ligamentina qPCR анализ (A.lig.1), включая последовательности для вперед и обратного грунтовки и зонда.
| вид | Усиливается | В реке Клинч |
| 1. Актинонайас связки | да | да |
| 2. Актинонайас пектороза | Нет | да |
| 3. Амблма plicata | Нет | да |
| 4. Корбикула spp. | Нет | да |
| 5. Камберлендия монодонта | Нет | да |
| 6. Циклонайас туберкулата | Нет | да |
| 7. Кипрогения стегария | Нет | да |
| 8. Эллиптио дилатата | Нет | да |
| 9. Эпиобласма бревиденс | Нет | да |
| 10. Эпиобласма капсаеформис | Нет | да |
| 11. Эпиобласма флорентина ауреола | Нет | да |
| 12. Эпиобласма трикетра | Нет | да |
| 13. Фусконайя кор | Нет | да |
| 14. Субротунда Фусконайя | Нет | да |
| 15. Лампсилис Оата | Нет | да |
| 16. Лампсилис силикуоидея | Нет | Нет |
| 17. Ласмигона costata | Нет | да |
| 18. Лемиокс Римосус | Нет | да |
| 19. Лексингтония долабеллойдес | Нет | да |
| 20. Медионидус конрадикус | Нет | да |
| 21. Плетобас цифий | Нет | да |
| 22. Пленум Плеробема | Нет | да |
| 23. Птихобранчус фасциоларис | Нет | да |
| 24. Подтентус Птихобранча | Нет | да |
| 25. Квадрула пустулоза | Нет | да |
| 26. Строфитус undulatus | Нет | да |
| 27. Вилоса радужная оболочка | Нет | да |
Таблица 2: Список видов, используемых для тестирования специфичности in vitro анализа A.lig.1. Анализ усиливает геномную ДНК цели(Actinonaias ligamentina) и не усиливает ни одного из нецелесообразных видов.
| компонент | Последовательность 5'-3' | ||||
| Стандарт Actinonaias ligementina | CCCTCATCACGTAC CTCTTAATCCTATTAGGTGTCGCATTTTTCACTCTTCTTGAACGTA | ||||
| AAGCCCTCGGGT ACTTTCAAATCCGAAAGGCCCAAATAAAGTTGAATTATGCATTC | |||||
| CCCAACCATTAGCAGATGCTCTAAAGCTCTCTGTAAAAGAAGAATGAGTAACACCAACCTCCT | |||||
| CAAACTACCTACCCTTCATCTTAACCCCAACCACTATTTAATTTTAGCACTACTACTTTTTTTTTTTTT | |||||
| ГАКААТТТККАТКТКТКАТКАТКАТКАТКАААТАНТТХТХТХТКТКТАТТЛТКТКТ | |||||
| TGTGTATCTCCCCCCTAGCTTTATACAACTTATAACAGGCTGAGCCTCAACTCCA | |||||
| AATATGCCCTTTTAGGCTATTGGAGCCATAGCCCAAACCATCTTATGAGGTTACAA | |||||
| ТААК | |||||
| Шаблон IPC (Hem-T) | CTACATAAGTAACACCCTCATCATCCAAAGCTCTCTGAGTGTCCCGAATCTCAGACGCT | ||||
| GTATGACAGTCTCCTTTTGTGAACATTCGGCTGCTCTATGTTCTCAAGGGCAC | |||||
Таблица 3: Последовательность (5'-3') стандарта Actinonaias ligamentina и шаблона IPC (Hem-T), используемого для этого анализа. Последовательность для вперед и обратного грунтовки в смелых и курсивом, и что зонд подчеркнул.
| компонент | имя | Последовательность 5' - 3' | Флуоресцентная этикетка | |
| Форвард Праймер | ГемТ-Ф | TCTGAGTGTCCCTCGAATCT | ||
| Обратный праймер | ГемТ-Р | ГКАГТККТГАГААКАТАГАГАГ | ||
| зонд | ГемТ-П | TGACAGTCTCCTTTCGTGAACATTCG | Cy5 | |
Таблица 4: Внутренний положительный контроль (IPC) анализ, включая последовательности для вперед и обратного грунтовки и зонда.
| Объем на образец (ОЛ) | компонент |
| 10 | Окружающая экологическая мастер микс |
| 1 | 20uM A. lig.1 F/R микс |
| 1 | 2.5uM A. lig.1 зонд |
| 1 | 5uM IPC грунтовка смесь (HemT-F / R) |
| 0.75 | 2.5uM IPC зонд (HemT-P) |
| 1.5 | 1 X 103 концентрация шаблона IPC |
| 2.75 | H20 |
| 2 | образец |
| 20 | Общий объем |
Таблица 5: Смесь ПЦР, используемая для анализа A.lig.1 с анализом IPC.
| шаг | Температура (КК) | Время | |
| 1 | Первоначальная денатура | 95 | 10 мин. |
| 2 | денатурировать | 95 | 15 сек |
| 3 | отжиг | 60 | 1 мин. |
| 4 | Перейти к шагу 2, повторить 39X |
Таблица 6: Условия реакции на анализ A.lig.1.
Обсуждение
Как и в любом исследовании, определение вопроса, который необходимо решить, является первым шагом, и разработка анализа eDNA зависит от объема исследования26. Например, если цель исследования или исследования состоит в том, чтобы обнаружить один или несколько видов, лучше всего провести целенаправленный анализ на основе зонда. Если, однако, цель состоит в том, чтобы оценить больший набор или сборку видов, высокая пропускная способность секвенирования метабаркодирования анализы лучше подходят. После того, как будет определено, какой подход следует принять, рекомендуется про экспериментальное исследование, включая проектирование, тестирование и оптимизацию анализа24. Дизайн анализа начинается со списка видов, описанных в Рисунок 1. Этот список станет основой для понимания того, насколько хорошо анализ выполняет с точки зрения специфики и географического диапазона он может быть применен к6,10. Рекомендуется разработать анализ для конкретной географической области, что позволяет дизайнеру лучше проверить анализ на перекрестное реактивность против других видов в этой области, и быть в курсе ограничений, которые это имеет на распространение анализа на другие области, где целевой вид может произойти24. После того, как список будет завершен, последовательности могут быть загружены из общедоступных генетических баз данных. Поскольку эти базы данных являются неполными27, следует последовательности столько видов в списке, как это возможно в доме, чтобы завершить локаную базу данных ссылок последовательностей, которые будут использоваться в анализе дизайна. Приоритеты со-происходящих тесно связанных видов, так как они являются наиболее вероятными не-целей, которые будут усиливаться. Сосредоточение внимания на всех видах одного рода или семьи, как целевой вид является хорошим местом для начала. Сравнение с близко родственными видами поможет определить области последовательности, уникальные для целевых видов. Это может помочь сообщить, как анализ может выполняться в других системах или местах. Митохондриальные регионы являются обычным выбором для развития анализа, потому что больше информации о последовательности от более широкого спектра видов доступна в митохондриальных генах, которые были использованы в штрих-коде жизненных проектов, и потому, что митохондриальная ДНК присутствует в гораздо большей концентрации в копиях/клетке, чем ядерная ДНК24,28,29. Несколько регионов генов должны быть оценены для дальнейшего развития анализа, поскольку охват последовательности варьируется между таксами в базах данных генетического репозитория. После создания локальной базы данных эталонных последовательностей для разработки анализов грунтовки/зонда используется комбинация ручной визуализации выровненных данных последовательности и компьютерных программ. Не следует полагаться строго на программное обеспечение, чтобы определить, какие анализы для тестирования. Важно визуально проверить на выравниваниях, где грунтовки и зонды сидят на мишенях и немишенях, чтобы лучше понять, как они могут действовать в ПЦР. Наконец, анализ скрининга и оптимизации включает в себя три уровня (в силико, in vitro и in situ)6,7,24,25. В silico дизайн и тестирование имеют важное значение для производства краткий список анализов с хорошими шансами на успех, но эмпирические (in vitro) тестирование имеет решающее значение для выбора анализа с лучшей фактической производительности. Оптимизация in vitro и тестирование анализов включают измерение эффективности реакции и определение чувствительности и специфичности анализа. Пределы обнаружения и количественной оценки являются двумя параметрами, которые часто упускаются из виду при разработке анализа, но важны для интерпретации данных. При запуске нескольких репликаций стандартных кривых для анализа, LOD и ЛОЗ могут быть легко измерены1,5,30. Немногие исследования обсуждают результаты в отношении LOD или LO, но Sengupta et al. (2019) включают LOD и LO' их анализа в их интерпретацию данных и графику для более четкого понимания их результатов31. Внутренний положительный контроль должен быть мультиплексирован в разработанный анализ, а также. Без тестирования на ингибирование ПЦР в образцах могут возникать ложные негативы24,32. Мы предлагаем использовать мультиплексный анализ IPC с целевым анализом как самый простой метод тестирования ингибирования ПЦР23. И наконец, для обеспечения целевого усиления в пробах окружающей среды необходимо провести тестирование анализов, взятых на местах и в лабораторных исследованиях, собранных на местах и в лабораторных лабораториях.24.
Существуют ограничения на использование видово-специфических, основанных на зондах qPCR анализов с образцами eDNA. Например, разработка нескольких анализов для тестирования может быть ограничена доступностью последовательности, и может потребоваться компромисс по аспектам анализа производительности. Эти варианты должны быть руководствуются целями исследования и должны быть сообщены с результатами26. Например, если целью является выявление редкого вида и ожидается небольшое количество положительных результатов, можно было бы использовать анализ с несовершенной специфичностью (т.е. усиление нецелесоприятных видов), если все обнаружения будут проверены секвенированием. Если цель заключается в мониторинге географического диапазона вида и данные о концентрации eDNA не требуется, анализ с несовершенной эффективностью может быть использован и данные сообщили только в процентах обнаружения. Кроме того, если все потенциальные конспецифические не будут протестированы в лаборатории, что редко возможно, нельзя с абсолютной уверенностью знать истинную специфику анализа. Например, анализ был разработан и протестирован против нескольких пресноводных видов мидий в реке Клинч. Чтобы использовать этот анализ в другой речной системе, мы должны проверить его на набор видов в новом месте. Генетические различия в видах или популяциях, которые не тестируются в ходе анализа развития, также могут повлиять на специфичность. Наконец, даже если было проверено, что анализ имеет высокую техническую производительность; условия меняются при работе в полевых условиях. Неаналимизируемые условия, такие как поток воды, рН и поведение животных, могут изменить обнаруживаемость eDNA, как и использование различных протоколов сбора и извлечения eDNA. Использование оптимизированных и хорошо описанных анализов поможет облегчить понимание влияния таких параметров на обнаружение eDNA.
Область eDNA созревает за пределами стадии исследовательского анализа для повышения стандартизации методов и методов. Эти разработки улучшат наше понимание методов, способностей и ограничений eDNA. Процесс оптимизации, который мы намекаем выше, улучшает чувствительность, специфичность и воспроизводимость анализа. Конечная цель такого уточнения и стандартизации методов eDNA заключается в улучшении способности исследователей делать выводы на основе данных eDNA, а также в повышении уверенности конечных пользователей и держателей акций в результатах.
Раскрытие информации
Авторы заявляют об отсутствие конфликта интересов. Спонсоры финансирования не играли никакой роли в разработке исследования; в сборе, анализе или интерпретации данных; в написании рукописи; или при принятии решения о публикации результатов.
Благодарности
Мы благодарим Альви Вадуда и Труди Фрост, которые помогали в разработке и тестировании грунтовки. Финансирование анализа дизайн сообщил в этом исследовании было предоставлено Министерство обороны Стратегические экологические исследования и разработки программы (RC19-1156). Любое использование торговых, продуктов или твердых наименований имеет только описательные цели и не означает одобрения со стороны правительства США. Данные, полученные в ходе этого исследования, доступны в качестве выпуска данных Геологической службы США https://doi.org/10.5066/P9BIGOS5.
Материалы
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
Ссылки
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены