Method Article
Desenvolvimento e teste de ensaios quantitativos de PCR específicos para aplicações de DNA ambiental
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Os ensaios de DNA ambiental requerem um design rigoroso, testes, otimização e validação antes que a coleta de dados de campo possa começar. Aqui, apresentamos um protocolo para levar os usuários através de cada etapa de projeto de um ensaio qPCR específico da espécie, baseado em sonda para a detecção e quantificação de dna de uma espécie-alvo a partir de amostras ambientais.
Resumo
Novos métodos não invasivos para detectar e monitorar a presença de espécies estão sendo desenvolvidos para auxiliar no manejo da pesca e da conservação da vida selvagem. O uso de amostras de DNA ambiental (eDNA) para detecção de macrobiota é um desses grupos de métodos que está rapidamente se tornando popular e sendo implementado em programas nacionais de gestão. Aqui focamos no desenvolvimento de ensaios específicos de espécies para aplicações de PCR quantitativos baseados em sonda (qPCR). O uso de qPCR baseado em teste oferece maior especificidade do que é possível apenas com primers. Além disso, a capacidade de quantificar a quantidade de DNA em uma amostra pode ser útil em nossa compreensão da ecologia do eDNA e na interpretação dos padrões de detecção de eDNA no campo. É necessária uma consideração cuidadosa no desenvolvimento e teste desses ensaios para garantir a sensibilidade e especificidade da detecção das espécies-alvo a partir de uma amostra ambiental. Neste protocolo, delinearemos as etapas necessárias para projetar e testar ensaios baseados em sondas para a detecção de uma espécie-alvo; incluindo criação de bancos de dados de sequência, design de ensaio, seleção e otimização de ensaios, desempenho de ensaio de teste e validação de campo. Seguir essas etapas ajudará a alcançar um ensaio eficiente, sensível e específico que possa ser usado com confiança. Demonstramos esse processo com nosso ensaio projetado para populações do mucket (Actinonaias ligamentina), uma espécie de mexilhões de água doce encontrada no rio Clinch, EUA.
Introdução
Pesquisadores e gestores estão cada vez mais interessados no uso de ensaios de DNA ambiental para detecção de espécies. Durante três décadas, o PCR quantitativo ou em tempo real (qPCR/rtPCR) tem sido usado em inúmeros campos para a detecção e quantificação específica da sequência de ácidos nucleicos1,2. Dentro do campo relativamente novo da pesquisa eDNA, o uso desses ensaios com uma curva padrão para quantificação de cópias de DNA alvo por volume ou peso da amostra de eDNA tornou-se agora prática de rotina. Sequências de DNA mitocondrial são geralmente alvo de ensaios eDNA porque o genoma mitocondrial está presente em milhares de cópias por célula, mas ensaios para sequências de DNA nuclear ou RNA também são possíveis. É vital entender que os ensaios publicados para amostras de eDNA nem sempre são iguais em desempenho. A confiabilidade de um ensaio na detecção apenas do DNA de uma espécie-alvo (ou seja, especificidade) e na detecção de baixas quantidades de DNA alvo (ou seja, sensibilidade) pode variar consideravelmente devido a diferenças na forma como o ensaio foi projetado, selecionado, otimizado e testado. As medidas quantitativas de relatórios de desempenho do ensaio foram previamente negligenciadas, mas recentemente as normas para melhorar a transparência no desenvolvimento dos ensaios estão surgindo3,4,5,6,7,8.
Otimização e relato de auxiliares de desempenho de ensaios no desenho do estudo e interpretação dos resultados da pesquisa eDNA. Ensaios que a reação cruzada com o DNA de espécies não-alvos podem levar a detecções falsas positivas, enquanto ensaios com pouca sensibilidade podem não detectar o DNA da espécie alvo mesmo quando ele está presente na amostra (falsos negativos). Uma compreensão da sensibilidade e seletividade do ensaio ajudará a informar o esforço de amostragem necessário para detectar espécies raras. Como existem muitas fontes naturais de variação no eDNA, os estudos devem limitar ao máximo as fontes controláveis de variação, incluindo a otimização total e caracterização do ensaio eDNA3.
Condições que afetam diretamente a especificidade ou sensibilidade de um ensaio mudarão o desempenho do ensaio. Isso pode ocorrer em diferentes condições laboratoriais (ou seja, diferentes reagentes, usuários, máquinas, etc.). Portanto, este protocolo deve ser revisto ao aplicar um ensaio sob novas condições. Mesmo ensaios bem caracterizados na literatura devem ser testados e otimizados quando adotados por um novo laboratório ou quando se utilizarem reagentes diferentes (por exemplo, solução master-mix)5,9. A especificidade do ensaio pode mudar quando aplicada a uma região geográfica diferente, porque o ensaio está sendo aplicado a amostras de uma nova comunidade biótica que pode incluir espécies não-alvo que o ensaio não foi testado, e a variação genética nas espécies-alvo pode ocorrer. Novamente, o ensaio deve ser reavaliado quando usado em um novo local. As condições de campo diferem das condições laboratoriais porque no campo os inibidores de PCR são mais propensos a estar presentes em amostras. Os inibidores do PCR afetam diretamente a reação de amplificação e, portanto, afetam o desempenho do ensaio. Por essa razão, é necessário um controle positivo interno ao desenvolver um ensaio eDNA.
Finalmente, as condições ambientais no campo podem afetar as moléculas de DNA da espécie alvo e sua captura através da degradação, transporte e retenção do DNA. Além disso, diferentes protocolos para coleta e extração de DNA variam em sua eficiência e capacidade de reter DNA. No entanto, é importante notar que esses processos afetam a detectabilidade do eDNA, mas não o desempenho de um ensaio molecular. Assim, a detectabilidade do DNA das espécies-alvo em amostras de campo é uma função tanto do desempenho técnico do ensaio qPCR quanto das condições de campo e protocolos de coleta, armazenamento e extração. Ao usar um ensaio bem caracterizado e de alto desempenho, os usuários podem se sentir confiantes nas capacidades do ensaio; permitindo que os pesquisadores agora se concentrem na compreensão dos fatores de ensaio externo (ou seja, variáveis ambientais, diferenças nos protocolos de captura ou extração) que afetam a detecção de eDNA.
Aqui focamos especificamente no desempenho técnico do ensaio através de design rigoroso e otimização. Demonstramos o protocolo usando um ensaio baseado em sonda desenvolvido para a detecção de um mexilhão de água doce, o mucket (Actinonaias ligamentina), da água amostrada no rio Clinch, EUA. Recentemente, Thalinger et al. (2020) apresentaram diretrizes para validação de ensaios direcionados eDNA. O design do ensaio seguindo nosso protocolo trará um ensaio para o nível 4 de Thalinger et al. mais um passo adicional para o nível 56. Neste ponto, o desempenho técnico de um ensaio será otimizado e estará pronto para uso regular em aplicações de laboratório e campo. O uso adicional do ensaio em experimentos de laboratório, mesocosmo e campo pode, então, abordar questões sobre detecção de eDNA e fatores que influenciam a detectabilidade, as etapas finais para validação nível5 6.
Protocolo
1. Geração de um banco de dados sequencial de sequências de DNA mitocondrial a partir de espécies de interesse alvo e não-alvo
- Defina a questão, os alvos e o sistema que está sendo abordado. Identifique as espécies-alvo para detecção de eDNA. Identifique o sistema geográfico no qual o ensaio será utilizado. Faça uma lista de espécies de interesse, incluindo as espécies-alvo, espécies simpátricas (co-ocorrendo) dentro da mesma taxa (geralmente ordem ou nível familiar), e espécies aopatricas intimamente relacionadas, aquelas que podem não estar na mesma localização geográfica que o alvo(Figura 1).
NOTA: Aqui, as populações do Rio Clinch da espécie A. ligamentina foram alvo. - Pesquise e baixe sequências de várias regiões genéticas para espécies na lista a partir do Passo 1. Bancos de dados de sequência como NCBI (National Center for Biotechnology Information), BOLD (Barcode of Life Database), EMBL (European Molecular Biology Laboratory Laboratory) e DDBJ (DNA Data Bank of Japan) podem ser usados. NCBI, EMBL e DDBJ compartilham informações de sequência.
- Utilizando o Banco de Dados Nucleotídeos do NCBI, procure o organismo alvo (por exemplo, Actinonaias ligamentina) e região genética (por exemplo, citocromo c oxidase I (COI) ou NADH-desidrogenase 1 (ND1); Exemplo de sequência de pesquisa: Actinonaias ligamentina e ND1)
- Em seguida, selecione todas as sequências que correspondem às especificações e selecione Enviar para. Escolha concluir o registro, arquivar e baixar o formato como GenBank ou FASTA e, em seguida, criar arquivo. Essas sequências estão agora salvas no computador.
- Repita estes passos para todas as espécies da lista definida na Etapa 1. Mantenha sequências para cada região genética em um arquivo separado, pois estas serão analisadas separadamente.
- Baixe todas as sequências relevantes (ou uma grande proporção representativa de sequências) para as espécies-alvo identificadas no Passo 1. Inclua variantes geográficas, se possível.
- Repita as sequências de pesquisa e download de espécies não-alvo relacionadas e simpátricas do mesmo grupo taxonômico que foram identificadas no Passo 1 (por exemplo, se a espécie alvo for a mucket (A. ligamentina) obter sequências de download para todas as outras espécies de mexilhões de água doce na Família Unionidae que ocorrem no sistema de interesse).
- Repita a pesquisa e baixe para espécies intimamente relacionadas, mas aopatricas (geograficamente separadas) listadas no Passo 1.1.
NOTA: Nem todas as espécies (alvos e não alvos) estarão disponíveis nas bases de dados públicas. Aumente o banco de dados de referência local amplificando e sequenciando tributicamente espécimes de espécies de interesse interna. Se trabalhar com uma espécie que tem alta diversidade genética dentro das espécies ou trabalhar em uma área geograficamente grande, onde variantes geográficas podem ser esperadas, reúna sequências de toda a gama.
2. Projeto de ensaio
- Alinhar sequências de cada região genética separadamente usando um software de alinhamento que pode ser encontrado em vários programas de edição de sequências genéticas e bioinformáticas. Faça esse alinhamento para cada uma das diferentes regiões genéticas.
- Por exemplo, usando o software Geneious Prime (https://www.geneious.com) importar os arquivos de sequência baixados para o programa.
- Crie pastas separadas para cada região genética.
- Dentro de uma pasta que contém sequências de uma região genética, selecione todas as sequências.
- Use a ferramenta de alinhamento múltiplo para criar um alinhamento nucleotídeo das sequências selecionadas. Pode haver várias opções para o tipo de alinhamento, usando os alinhamentos Geneioso ou MUSCULAR e os parâmetros padrão funcionam bem.
- Escolha regiões promissoras para design de ensaio através da visualização de dados de sequência alinhados. Uma região que tem um monte de dados de sequência disponíveis para as espécies de interesse, é altamente divergente entre as espécies, e mostra baixa variação dentro das espécies é um bom candidato. Isso aumentará a probabilidade de que os primers e sondas projetados sejam capazes de discriminar o alvo de espécies não-alvo, ao mesmo tempo em que garantem que as variantes intraespecíficas se amplificarão com o ensaio.
- Projeto de primers de ensaio e sonda.
- Use o software de design de ensaio qPCR e siga as instruções. A ferramenta PrimerQuest (https://www.idtdna.com/) do IDT para projetar 5 conjuntos de ensaios qPCR foi usada aqui.
- Cole a sequência selecionada na etapa 2.2 na caixa de entrada Sequência. Se o alinhamento tiver espaços criados, exclua-os da sequência.
- Selecione qPCR 2 Primers + Teste na opção Escolher seu design.
- Baixe os ensaios recomendados.
- Copie as sequências do primer dianteiro do primeiro ensaio e procure por esta sequência de primer no alinhamento criado na etapa 2.1.4. Se estiver usando o Geneious Prime, use a ferramenta Anotar e Prever para adicionar a região do primer ao alinhamento. Faça isso para todas as combinações de primer e sonda(Figura 2).
- Inspecione essas regiões do alinhamento para obter variação dentro das espécies-alvo, bem como dentro das espécies que ocorrem.
- Se houver variação genética intraespecífica, procure ensaios onde os primers e a sonda não se enquadram nessas regiões.
- Para evitar a amplificação de espécies não-alvo, procure incompatibilidades com espécies não-alvo. Escolha ensaios com mais incompatibilidades para posterior validação. Currier et al. (2018) sugerem escolher conjuntos com pelo menos duas das três regiões (os dois primers, ou uma cartilha e uma sonda) tendo pelo menos dois desencontros com todas as espécies não-alvo. No entanto, tenha em mente que os desencontros na sonda contribuem menos para a especificidade10.
NOTA: Diferenças dentro de 3 pares base do final de 3' de cada primer aumentam a especificidade melhor do que as diferenças no final de 5' dos primers10.
- Considere os seguintes parâmetros importantes no projeto do ensaio.
- Determine as temperaturas de fusão e ressaramento dos primers e da sonda. Idealmente, a temperatura de fusão (Tm) dos primers deve ser entre 60-64 °C e dentro de 2 °C um do outro, e o Tm da sonda deve ser 6-8 graus mais alto que o Tm dos primers. Defina a temperatura de ressaramento (Ta) da reação qPCR 5 °C abaixo da temperatura de fusão, em torno de 55-60 °C11.
- Examine o conteúdo gc. Escolha entre 35 – 65% de conteúdo GC e evite regiões com Gs 4 ou mais consecutivos. Ter 1 ou 2 Gs ou Cs nas últimas bases das 5 últimas bases do primer (grampo GC) pode aumentar a especificidade, pois ajudaria o primer a fazer um vínculo mais forte12.
- Procure por estruturas de grampos e mais fracos. Primers de teste e sonda para estruturas de grampos preditos e dimers usando um programa de análise de oligonucleotídeos (por exemplo, OligoAnalyzer -IDT13; OligoCalculador14). Essas estruturas podem causar amplificação não-alvo e menor eficiência. Evite ensaios que são previstos para formar essas estruturas.
- Determine o comprimento do primer. Aponte para primers entre 18-25 bases de comprimento e comprimento da sonda entre 20 -25 bases. Primers e sondas mais longas podem ter menor eficiência de amplificação.
- Determine o comprimento da amplicon. Deve ser entre aproximadamente 100 e 250 pares de base. Esta faixa é geralmente curta o suficiente para alta eficiência pcr, mas tempo suficiente para facilitar a verificação pelo sanger sequenciamento4,15.
- Sondas de design. Certifique-se de que as sondas não têm uma base G na extremidade de 5', pois poderia amortecer o sinal dos corantes verde e amarelo11. Projetamos sondas duplas saciadas, com iDT 3IABkFQ e quenchers ZEN e fluoroforos FAM ou HEX.
NOTA: Determine as sondas MGB: Os testes TaqMan MGB (aglutinante de ranhura menor) são frequentemente usados para estudos de eDNA. No entanto, como essas sondas são muito curtas, elas podem se ligar a não-alvos mesmo com um par de 2 ou 3 bases incompatíveis10. - Determine a sonda Tm. A temperatura de fusão da sonda deve ser 6-8°C mais alta que os primers. Temperaturas mais baixas diminuem o sucesso de ligação da sonda.
- Determine o comprimento e a localização da sonda. A sonda deve ter entre 20 e 25 bp de comprimento e, idealmente, localizada perto do local de ligação primer no mesmo fio sem sobrepor-a.
3. Triagem e otimização de ensaios
- No desenvolvimento e teste de ensaio silico. Antes de encomendar conjuntos de sondas de primer, avalie a especificidade (amplificação potencial não-alvo) testando a amplificação do primer em silico.
- Teste-se por meio do Primer-Blast16 da NCBI ou programas similares que podem identificar potenciais não-alvos no banco de dados NCBI nt/nr que podem se amplificar com o ensaio. Se usar primer-blast pasta primers na caixa de primer Use minha própria caixa de primer sob parâmetros Primer. Nas opções de Parâmetros de Verificação de Especificidade do Par Primer, selecione nr como banco de dados e digite a Ordem do organismo de interesse (por exemplo, "Unionida" ou "Unionoida") na caixa do Organismo.
- Continue avaliando conjuntos de primer/sonda visualmente em dados de sequência alinhados.
- Para avaliar primers e sondas ao mesmo tempo em silico, crie uma sequência de texto do primer dianteiro, 12 N's, a sonda, 12 N's, e o complemento inverso da primer reversa. Se a sequência da sonda estiver dentro de 12 pares base de um dos primers, use o número de N correspondente ao número de pares de base entre o primer e a sonda.
- Use a pesquisa Nucleotide Blast (Blastn) do NCBI para pesquisar contra o banco de dados nr17. Use a guia Taxonomia para procurar espécies não-alvo com poucas incompatibilidades; estes devem ser testados em laboratório durante a otimização do ensaio.
NOTA: No teste de silico ajuda a excluir ensaios não específicos, mas ensaios potencialmente específicos devem ser testados empiricamente (in vitro), pois nem todas as espécies têm sequências nos bancos de dados genéticos e primer e sondas ainda podem se ligar a não-alvos, mesmo que considerados improváveis pelo software.
- Escolha de três a cinco combinações de primer/sonda para testar em laboratório.
- Ordem primers, sondas e um padrão de DNA sintético, bem como primers adicionais de cauda M13 para sequenciamento de amplicon.
- Peça primers e sondas de oligonucleotídeos sintéticos de uma empresa que faz oligoses. As sondas são rotuladas com um corante fluorescente e uma saciador. Fluoroforos diferentes devem ser selecionados para ensaios que precisam ser multiplexados. Verifique o instrumento qPCR para obter uma lista de quais fluoroforos o instrumento pode detectar.
- Design e encomenda Primers com cauda M13 para verificação de detecções qPCR com sequência Sanger adicionando a sequência M13 Forward (-20), GTA AAA CGA CGG CCA GT, até o final de 5' do primer dianteiro, e a sequência M13 Reverse (-27), CAG GAA ACA GCT ATG AC, até o final de 5' do primer reverso.
- O padrão de DNA sintético contém a sequência de destino (incluindo as regiões de primer) em uma concentração conhecida em cópias/μL. Quantificar amostras desconhecidas com base em uma curva feita por concentrações conhecidas deste padrão (ou seja, a curva padrão). Adquira o padrão sintético da mesma empresa que fabrica os primers e a sonda. Siga as recomendações do fabricante para a ressuspensão e armazenamento. Diluir padrões no buffer TE com um portador de tRNA usando plásticos de baixa retenção para reduzir a hidrólise e a vinculação às superfícies.
NOTA: Se a curva padrão não tiver um bom desempenho (má eficiência do PCR, consulte o passo 3.4.2), tente suspender o padrão em água ou Tris-HCl. - Suspender primers e sondas em água livre de nuclease, tris-HCl ou tampão TE em concentrações convenientes para uso de ensaio. Geralmente, diluem os estoques de trabalho 20 vezes no mix mestre para alcançar a concentração de ensaio final otimizada. Armazene oligos suspensos a uma constante de -20 °C quando não estiver em uso.
- In vitro (em laboratório) ensaio otimização e teste. Rejeitar ensaios que têm baixa eficiência, reagem cruzadamente com espécies que ocorrem em co-ocorrem ou têm baixa sensibilidade18. Inclua o uso de um controle positivo interno (IPC) durante o desenvolvimento de ensaios, bem como ao executar amostras reais.
- Primeiro, encontre os valores de concentração de temperatura e primer/sonda ideal para o ensaio. Uma vez otimizados esses parâmetros para a eficiência do PCR (Etapa 3.4.2), reatividade cruzada (Passo 3.4.3) e sensibilidade (Passo 3.4.4), proceder ao teste do ensaio com um IPC multiplexado (Passo 3.4.5).
- Teste a temperatura de renascimento ideal (Ta) para primer e sondas usando um gradiente de temperatura PCR centrado 5° C abaixo do primer médio previsto Tm.
- Teste as concentrações ideais de primer e sonda. Normalmente, concentrações de 200 nM, 400 nM e 800 nM e concentrações de 75 nM, 125 nM e 200 nM são testadas.
- Crie uma curva padrão e determine eficiência e alcance linear. Teste pelo menos seis diluições 10 vezes de um padrão de DNA sintético contendo a sequência de destino, aproximadamente 100 cópias/reação a 105 cópias/reação(Figura 3A).
- Use o software qPCR para traçar o valor Cq (limiar para ciclo em quantificação) de cada padrão no eixo y e a base de registro 10 da concentração padrão inicial em cópias/reação no eixo x. O software qPCR deve executar automaticamente uma regressão linear(Figura 3B).
- Calcule a eficiência da inclinação da regressão, E = -1 + 10(-1/inclinação). Por exemplo, se a inclinação for -3,4, E= -1 + 10(0,29) = 0,97 ou 97%. Verifique também os valores r2 que indicam o quão bem as réplicas padrão se encaixam na curva. O software qPCR deve calcular isso automaticamente também(Figura 3B). Objetivo de valores de eficiência de 100% (±10%) e r2 valores de ≥0,989,15,19,20,21,22.
- Inspecione visualmente a curva padrão para viés, ou seja, desvios da regressão em direção consistente ou para baixo desempenho da curva padrão medida pela eficiência e pelos valores r2 (Figura 3C e 3D).
- Especificidade: Avaliar a reatividade cruzada com espécies não-específicas para diminuir a chance de falsos positivos. Quando as detecções de eDNA podem resultar em decisões de gerenciamento caras, verifique detecções positivas por sequenciamento amplicon.
- Não alvos: Executar o ensaio contra extrações genômicas de DNA de espécimes taxonomicamente verificados de espécies relacionadas e de espécies geograficamente co-ocorrendo; com a maior prioridade sendo testar contra espécies intimamente relacionadas, que ocorrem co-ocorrendo. Use concentrações totais de DNA semelhantes para amostras alvo e não-alvo. A concentração escolhida deve produzir amplificação a partir de amostras de espécies-alvo perto do meio da faixa linear da curva padrão. A amplificação deve ser observada apenas com a espécie alvo.
- Se for observada amplificação não-alvo, limpe e sequencie o produto para verificar sua identidade. Não é incomum observar a contaminação das espécies-alvo em amostras de tecidos de espécies não-alvo, portanto, todas as amplificações nesta fase devem ser verificadas por sequenciamento. Reamplifice amplicons limpos de testes de especificidade usando os primers de cauda M13 e sequência com primers M13.
- No laboratório pós-PCR, transfira os produtos qPCR para serem sequenciados para tubos frescos. Remova primers residuais e componentes de reação com um kit de limpeza (por exemplo, Kit de Purificação do PCR MinElute).
- Faça 1:100 diluições das eluções e amplie 1 μL de cada para 30 ciclos em uma reação PCR de 50 μL com os primers de cauda M13 e uma polimerase de alta fidelidade (por exemplo, Polimerase de DNA de alta fidelidade de Phusion).
- Execute 10 μL de cada reação em um gel de 1% de agarose para verificar se há uma única faixa do tamanho esperado. Se nenhuma banda for observada, aumente o número de ciclos ou a quantidade de amostra. Se várias bandas forem observadas, o gel purifica a banda do tamanho esperado.
- Remova primers residuais e componentes de reação com um kit de limpeza como acima e meça as concentrações de DNA das eluções.
- Configure reações de sequenciamento com os primers M13 de acordo com as instruções da instalação de sequenciamento.
NOTA: Nunca abra amostras amplificadas no laboratório qPCR. Prepare amostras para sequenciamento em um laboratório dedicado a amostras pós-PCR.
- Sensibilidade: A sensibilidade afeta a chance de falsos negativos, ou falhas na detecção do DNA da espécie alvo quando ela está presente. Avalie o limite de detecção (LOD) e o limite de quantificação (LOQ) para cada ensaio. Por fim, inclua um controle positivo interno (IPC) para avaliar a inibição do PCR das amostras. Multiplex e teste este ensaio IPC com o ensaio projetado para garantir que os dois ensaios não interfiram um com o outro.
- LOD: Faça seis diluições seriais de 4 vezes do padrão de DNA sintético, com 8-24 réplicas por diluição padrão(Figura 4). Calcule a menor concentração inicial com 95% de detecção. As parcelas LOD e LOQ podem ser geradas com uma calculadora LOD/LOQ R script5.
NOTA: Os dados abaixo do LOD não devem ser censurados. Devido à especificidade do PCR, não há limite menor para verdadeiros positivos. O LOD é a maior concentração abaixo da qual podem ocorrer falsos negativos. - LOQ: A partir da mesma série de diluição, calcule a menor concentração padrão inicial de DNA quantificável com um coeficiente de variação (CV) abaixo de 35%.
NOTA: LOD e LOQ devem ser reportados em cópias/reação. Ao utilizar um ensaio validado e amostras de campo amplificadas abaixo do LOQ, os resultados devem ser relatados como % detecções em vez de concentrações de eDNA, pois a concentração exata não pode ser medida com confiança5.
- LOD: Faça seis diluições seriais de 4 vezes do padrão de DNA sintético, com 8-24 réplicas por diluição padrão(Figura 4). Calcule a menor concentração inicial com 95% de detecção. As parcelas LOD e LOQ podem ser geradas com uma calculadora LOD/LOQ R script5.
- Use um controle positivo interno (IPC) para testar a inibição do PCR. A inibição pode levar a uma diminuição da sensibilidade e falsos negativos. Teste a capacidade do ensaio IPC a ser multiplexado com o ensaio de destino.
- Um ensaio IPC pode ser multiplexado com o ensaio de destino usando uma sonda com um corante repórter diferente do ensaio de destino. Este ensaio iPC consiste em uma pequena sequência de DNA sintético de uma espécie não relacionada ao imposto alvo, incorporada na mistura mestre qPCR a uma baixa concentração de aproximadamente10 2 cópias/reação, juntamente com primer e sondas que o detectam. Esta menor concentração é necessária para evitar a concorrência com a sequência de alvos para polimerase e nucleotídeos23.
- Compare o valor do Cq do modelo IPC da amostra com o do modelo IPC no controle sem modelo. Neste nenhum controle de modelo (NTC), a única entrada de DNA é a do modelo IPC. O modelo IPC nesta reação deve amplificar como esperado. Se o modelo IPC em uma amostra se amplificar a 2 ou mais ciclos diferentes do modelo IPC no NTC, a amostra eDNA será inibida. Amostras que mostram inibição podem ser diluídas 1:10 e retestadas. Se uma amostra permanecer inibida, essa amostra deve ser removida da análise.
- Primeiro, encontre os valores de concentração de temperatura e primer/sonda ideal para o ensaio. Uma vez otimizados esses parâmetros para a eficiência do PCR (Etapa 3.4.2), reatividade cruzada (Passo 3.4.3) e sensibilidade (Passo 3.4.4), proceder ao teste do ensaio com um IPC multiplexado (Passo 3.4.5).
- In situ ensaio desenvolvimento e teste
- Em laboratório: Se houver acesso ao organismo em laboratório, bem como espécies simpatricas; colher amostras de água de compartimentos com essas espécies, processar as amostras e testar o ensaio contra essas amostras de eDNA. Produtos de sequência acima para verificar a amplificação do alvo pretendido usando os primers de cauda M13.
- No campo:
- Identifique locais onde o organismo alvo é conhecido por ocorrer e conhecido por não ocorrer. É preferível ter alguma medida de abundância em cada local onde as espécies-alvo ocorrem.
- Decida quais serão utilizados volumes amostrais e métodos de coleta de amostras (por exemplo, filtração, centrifugação, etc.)
- Inclua um controle em branco ou negativo em cada local, esta é a água limpa que foi trazida para o local do campo e, em seguida, coletada e preparada com os mesmos equipamentos de campo e protocolos utilizados para amostragem eDNA24. O objetivo do campo em branco é detectar a contaminação dos equipamentos de amostragem e equipamentos de campo trazidos ao local. Leve o campo em branco antes de processar amostras de água de campo.
- Pegue várias amostras de água por local, de preferência 3 amostras por local.
- De volta ao laboratório, processe e extraia amostras.
- Execute o ensaio usando uma placa configurada semelhante à Figura 5A e compare a concentração e frequência de detecção eDNA com diferenças de local conhecidas na ocorrência e abundância. Confirme todas as detecções sequenciando24,25.
NOTA: O acima validaria um ensaio através do Nível 4 da escala6 (otimização do desempenho técnico do ensaio) e começaria a coletar dados com suporte à validação do ensaio nível 5. O nível 5 incorpora modelagem de probabilidade e uso do ensaio para estudos de ecologia eDNA. Achamos que isso está além do escopo do desenvolvimento básico de ensaios, mas encorajamos essas aplicações de ensaios de laboratório e campo controlados para melhorar o design de ensaios e a interpretação de dados.
Resultados
Ao projetar um ensaio qPCR específico para a mucket(A. ligamentina),foram baixadas sequências disponíveis de todas as espécies de Unionidae no rio Clinch. Espécies intimamente relacionadas, como Lampsilis siliquoidea, também foram incluídas no banco de dados de referência, embora não sejam encontradas no mesmo rio. Nem todas as espécies do sistema de interesse do rio foram encontradas no GenBank, então espécies adicionais foram sequenciadas em casa. As sequências foram alinhadas usando o software Geneious e o software Primer Quest (IDT) foi usado para projetar vários ensaios. Cinco conjuntos de primers e sonda foram adicionados ao alinhamento para avaliação visual(Figura 2). Eles foram então testados em silico usando Primer-Blast, após o qual eles foram ordenados para novos testes in vitro. Em laboratório, todos os ensaios foram testados utilizando extrações de DNA de 27 espécies disponíveis para verificar a especificidade. Um ensaio (A.lig.1) ampliou com sucesso apenas as espécies-alvo (Tabela 1; Tabela 2). Este ensaio avançou para novos testes de eficiência de ensaio, LOD e LOQ. Tem um comprimento de amplicon de 121 pares de base. A Tabela 3 mostra a sequência usada para o padrão de DNA sintético A. ligamentina. Figura 3A e Figura 3B mostram os resultados de um ensaio bem sucedido com boa eficiência e valores r2. A Figura 3C e a Figura 3D mostram um ensaio cuja curva padrão tem uma eficiência ruim; este ensaio foi descartado. O LOD e o LOQ para o ensaio selecionado (A.lig.1) foram encontrados como sendo 5,00 cópias/reação usando o método discreto descrito em Klymus et al5. O IPC que foi multiplexado com o ensaio (Tabelas 3-6) não afetou a curva padrão do ensaio A. ligamentina. O IPC que usamos é um fragmento da transcrição hemT do mouse. Este ensaio foi pré-assinado pelo IDT para outra aplicação, mas modificamos seu uso como IPC para aplicações eDNA do nosso laboratório.
Uma execução de qPCR bem sucedida deve atender a certos critérios para cada medida de desempenho (ou seja, amplificação de curva padrão, controle positivo de DNA genômico, sem controle de modelo e controle positivo interno). Os padrões de ensaio de destino devem ter curvas de amplificação exponenciais. Essas curvas devem atingir um patamar de ponto final se permitidos a rodar ciclos suficientes. Isso indica que a sonda fluorescente está sendo completamente consumida durante a reação, e os níveis de fluorescência atingem um limite máximo. Padrões de amplificação posteriores podem não atingir um patamar em 40 ciclos. Os controles positivos (DNA genômico e IPC) devem ter o mesmo padrão. Desconhecidos podem ou não amplificar, mas a amplificação em desconhecidos também deve ter um padrão exponencial e um platô de ponto final(Figura 5).
Em um qPCR de qualidade, as diluições padrão amplificam em CQ espaçado uniformemente de aproximadamente cada 3,3 ciclos para cada diferença de 10 vezes na concentração. Cada réplica de uma diluição padrão amplifica-se de forma bem agrupada tendo quase o mesmo Cq (representado pelos valores r2). Todas as diluições padrão devem exibir amplificação(Figura 3A). Em um qPCR ruim, as normas podem exibir forma não exponencial, variação desigual nos valores do CQ entre diluições, não chegar a um patamar de ponto final, ou algumas diluições podem não amplificar em tudo(Figura 3D).
Os parâmetros importantes para uma curva padrão são eficiência, r2,inclinação e y-intercept. A eficiência deve cair entre 90%-110% com valores ideais próximos a 100% e os valores r2 devem estar acima de 0,98 com resultados ideais aproximando-se de 1,015,22. Os valores de inclinação devem ser entre -3.2 e -3.5 com resultados ideais próximos a -3.322. Os valores de y-intercept devem cair entre um Cq de 34-41 com resultados ideais com um Cq de 37,0. O y-intercept é o Cq previsto de uma reação com 1 cópia da sequência de destino, a menor unidade que pode ser medida em um único qPCR. Desconhecidos com CQ maior do que o y-intercept são susceptivos de serem inibidos. A execução de mais de 40 ciclos de PCR pode ser necessária para detectar o alvo em caso de inibição ou um conjunto de primer ineficiente, porém a quantificação não é possível nessas circunstâncias e controles negativos adicionais sem a sequência de destino, mas contendo DNA total semelhante às incógnitas, deve ser executado para excluir a amplificação de fontes não específicas.
A amplificação do Controle Positivo Interno (IPC) em amostras desconhecidas deve ser comparada com os resultados do controle de modelo negativo IPC, pois não há concorrência para reagentes e não há inibidores. Desconhecidos com um IPC com um CQ de 2 ciclos ou superior ao valor médio de Cq do NTC, ou que não amplificem devem ser considerados inibidos. Se não houver inibidores nas amostras, então toda a amplificação do IPC deve ter um agrupamento apertado na trama com valores de Cq próximos aos mesmos do NTC(Figura 6).
Finalmente, no situ o teste do ensaio ocorreu. Vinte amostras de água do rio Clinch e três amostras em branco de campo foram filtradas entre 25 e 26 de setembro de 2019 a 500 metros de um leito de mexilhões conhecido por ter A. ligamentina. Aproximadamente quatro amostras de 1 L de água foram filtradas por local de amostragem. Locais de localização incluídos no fundo do leito de mexilhões no córrego, fundo do leito de mexilhões perto da costa, 100 metros rio abaixo do leito em córrego, 500 metros a jusante do leito próximo à costa(Figura 7). De volta ao laboratório, cada filtro foi cortado ao meio e o DNA foi extraído de apenas metade de um filtro. A metade restante do filtro para cada amostra foi armazenada em um congelador de -80 °C. As amostras foram então executadas usando o ensaio A.lig.1 multiplexed com o IPC. Das 23 amostras, cinco foram inibidas. Estas amostras foram diluídas 1:10 e diluições foram re-executadas. Dezenove das 20 amostras de campo amplificadas usando o ensaio projetado. Dessas 19 amostras, cinco estavam acima do LOD do ensaio e loq de 5 cópias/reação; o que significa que a maioria das amostras teve uma detecção de eDNA, mas em um nível onde os resultados falsos negativos provavelmente ocorrerão e que o ensaio não poderia quantificar com confiança o número da cópia para essas 14 amostras. No entanto, 75 a 100% dos quatro locais biológicos se replicam amplificados em cada local de amostragem. Dois dos três espaços em branco foram negativos, enquanto um campo em branco mostrou amplificação, enfatizando a importância da técnica limpa no campo.

Figura 1: Fluxo de trabalho para construção do banco de dados de sequência de DNA mitocondrial. Clique aqui para ver uma versão maior desta figura.

Figura 2: Alinhamentos de sequência para espécies de mexilhões do rio Clinch com primers e sondas prospectivas para o ensaio Actinonaias ligamentina ND1. Primers avançados em verde escuro, sonda em vermelho e primer reverso em verde claro. Clique aqui para ver uma versão maior desta figura.
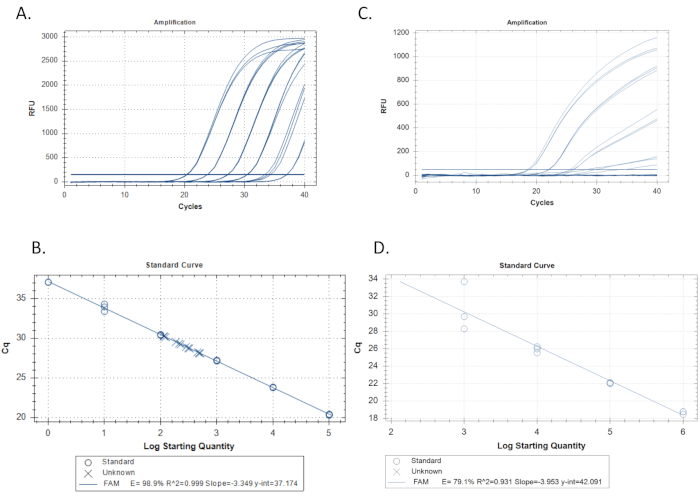
Figura 3: Exemplos de curva padrão e regressão linear. A. Exemplo de uma curva padrão aceitável derivada da amplificação de três réplicas cada uma das seis diluições padrão. Uma série de diluição padrão de 10 vezes com a maior concentração do padrão à esquerda, com concentrações decrescentes movendo-se para a direita. A linha horizontal que cruza todos os traços é o limiar para ciclo em quantitação (Cq). Onde cada traço cruza este limiar é onde o Cq é determinado. B. Regressão linear feita a partir das réplicas padrão da Figura 3A. As réplicas das diluições padrão são plotadas em círculos e as incógnitas (amostras) são plotadas com x's. A eficiência é de 98,9%, r2 se aproximando de 1,0 e inclinação de -3,349. C. Exemplo de uma curva padrão ruim derivada da amplificação de três réplicas cada uma das seis diluições padrão. D. Uma regressão linear formando a curva padrão para as réplicas padrão amplificadas no exemplo 3C. Note a baixa eficiência e os valores r2. Observe também que apenas 4 dos 6 padrões foram amplificados. Se após a repetição de corridas, a curva padrão não melhorar, o problema pode ser com um conjunto de primer/sonda ruim que não amplifica o DNA alvo como esperado nesse caso, este ensaio não deve ser considerado. Clique aqui para ver uma versão maior desta figura.
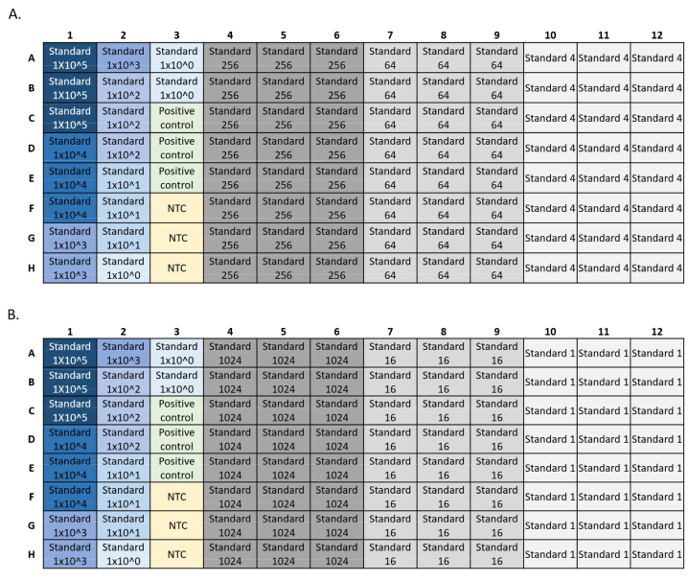
Figura 4: Exemplos de configurações de placas para corridas qPCR padrão LOD e LOQ. Os padrões utilizados na curva são em azul, a concentração padrão diminui de escuro para azul claro. Controle positivo de DNA em verde e sem controle de modelo (NTC) em amarelo. Concentrações padrão experimentais em cinza mostrando 24 réplicas para cada diluição padrão. A série de diluição foi emplaada em duas placas (A, B), cada uma com uma curva padrão, controle positivo e NTC. Clique aqui para ver uma versão maior desta figura.
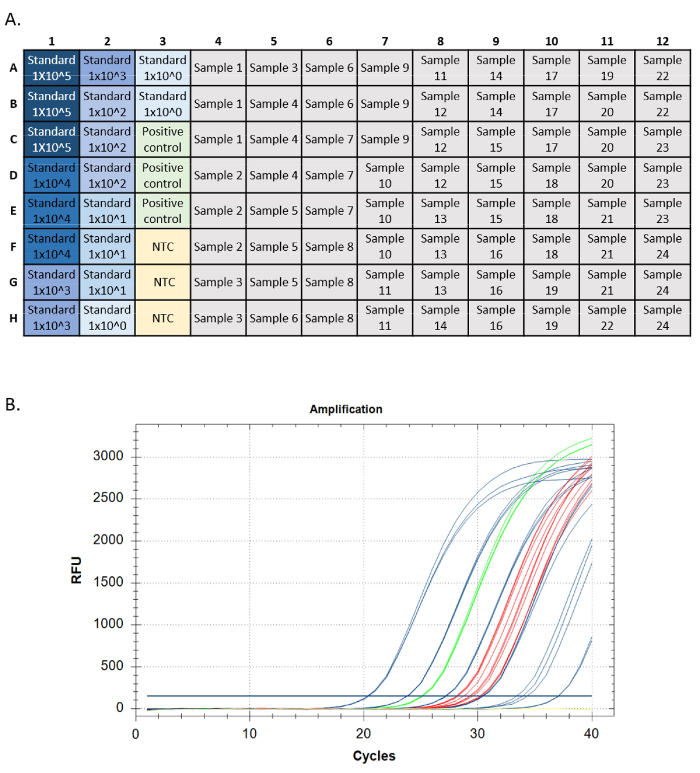
Figura 5: Os traços de configuração e amplificação da placa a partir de uma execução qPCR. A. Configuração da placa, padrões mostrados em azul, cor mais escura indicando a maior concentração do padrão. Controle positivo de DNA em verde, sem controles de modelo em amarelo (NTC), alvos de amostra em cinza. B. Traços de amplificação a partir de uma execução qPCR. Padrões mostrados em azul, controle positivo de DNA em verde, sem controles de modelo em amarelo, e desconhecidos em vermelho. Clique aqui para ver uma versão maior desta figura.
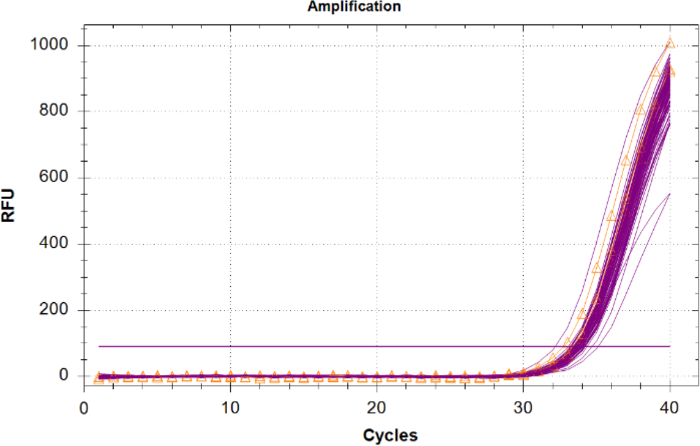
Figura 6: Traços de amplificação para o Controle Positivo Interno (IPC). Traços de IPC para todas as amostras desconhecidas em magenta e iPC a partir dos controles de não modelo (NTCs) mostrados em laranja com triângulos. Clique aqui para ver uma versão maior desta figura.
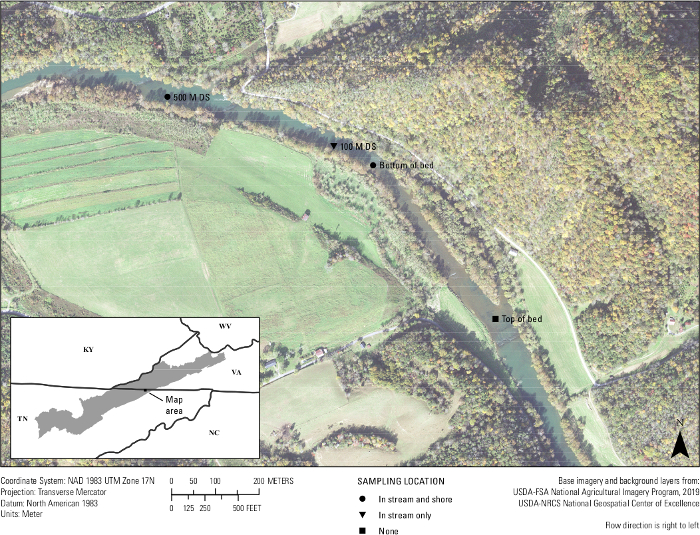
Figura 7: Mapa mostrando os locais de coleta eDNA de um leito de mexilhões no rio Clinch ao longo da fronteira Virginia/Tennessee. Amostras foram coletadas em Wallens Bend, no fundo da cama, 100 metros rio abaixo da cama e 500 metros rio abaixo da cama. Os locais foram coletados no meio do córrego (em córrego) ou a cerca de 1 – 2 metros da costa (costa). Clique aqui para ver uma versão maior desta figura.
| componente | nome | Sequência 5' – 3' | Rótulo fluorescente | |
| Primer avançado | A.lig.1-f | CCCTCATCACGTACCTCTTAATC | ||
| Primer reverso | A.lig.1-r | GGAATGCCCATAATTCCAACTTTA | ||
| sondar | Sonda A.lig.1 | TTCTTGAACGTAAAGCCCTCGGGT | Fam | |
Tabela 1: O ensaio actinonaias ligamentina qPCR projetado (A.lig.1) incluindo sequências para os primers dianteiros e invertidos e a sonda.
| espécie | Amplificado | No rio Clinch |
| 1. Actinonaias ligamentina | sim | sim |
| 2. Actinonaias pectorosa | Não | sim |
| 3. Amblema plicata | Não | sim |
| 4. Corbicula spp. | Não | sim |
| 5. Cumberlandia monodonta | Não | sim |
| 6. Ciclonaias tuberculata | Não | sim |
| 7. Cyprogenia stegaria | Não | sim |
| 8. Elítio dilatata | Não | sim |
| 9. Epioblasma brevidens | Não | sim |
| 10. Epioblasma capsaeformis | Não | sim |
| 11. Epioblasma florentina aureola | Não | sim |
| 12. Epioblasma triquetra | Não | sim |
| 13. Fusconaia cor | Não | sim |
| 14. Fusconaia subrotunda | Não | sim |
| 15. Lampsilis ovata | Não | sim |
| 16. Lampsilis siliquoidea | Não | Não |
| 17. Lasmigona costata | Não | sim |
| 18. Lemiox rimosus | Não | sim |
| 19. Lexingtonia dolabelloides | Não | sim |
| 20. Medionidus conradicus | Não | sim |
| 21. Plethobasus cyphyus | Não | sim |
| 22. Pleurobema | Não | sim |
| 23. Ptychobranchus fasciolaris | Não | sim |
| 24. Ptychobranchus subtentus | Não | sim |
| 25. Quadrula pustulosa | Não | sim |
| 26. Strophitus undulatus | Não | sim |
| 27. Íris de Villosa | Não | sim |
Tabela 2: Uma lista de espécies utilizadas para o teste de especificidade in vitro do ensaio A.lig.1. O ensaio ampliou o DNA genômico do alvo (Actinonaias ligamentina) e não ampliou nenhuma das espécies não-alvo.
| componente | Sequência 5'-3' | ||||
| Padrão actinonaias ligementina | CCCTCATCACGTAC CTCTTAATCCTATTAGGTGTCCCCCCCCCCCCCCCTTCACTCTTCTTGAACGTA | ||||
| AAGCCCTCGGGT ACTTTCAAATCCGAAAAAGGCCCAAATAAAGTTGGAATTATGGGCATTC | |||||
| CCCAACCATTAGCAGATGCTCTAAAGCTCTTCGTAAAAGAATGAGAACACCAACCTCCT | |||||
| CAAACTACCTACCCCTCCATCTTAACCCCAACCACTATATTTAATTTTAGCACTACTACTTT | |||||
| GACAATTATTTCCATCCTTTATANTATCATCCCAAATANTTTTTGGTATGCTCCTATTCTCTCTCTCTCT | |||||
| TGTGTATCTCCCTCTGTTTACAACACAACATAACAGGCTGAGCCTCAAACTCCA | |||||
| AATATGCCCTTTTAGGAGCTATTCGAGCCACCCAAACCATTTCTTATGAGGTTACAA | |||||
| TAAC | |||||
| Modelo IPC (Hem-T) | CTACATAAGTAACACCCTCATCATGTCCAAAGCTCTCTGAGTCCCTCGAATCTCAGACGCT CAGACGCT | ||||
| GTATGACAGTCTCCTTTCGTGAACATTCGGCTATGTTCTCAAGGACTGCAC | |||||
Tabela 3: Sequência (5'-3') do padrão Actinonaias ligamentina e do modelo IPC (Hem-T) utilizado para este ensaio. A sequência para os primers dianteiros e invertidos são em negrito e itálico, e a da sonda é sublinhada.
| componente | nome | Sequência 5' – 3' | Rótulo fluorescente | |
| Primer avançado | HemT-F | TCTGAGTGTCCCTCGAATCT | ||
| Primer reverso | HemT-R | GCAGTCCTTGAGAACATAGAGC | ||
| sondar | HemT-P | TGACAGTCTCCTTTCGTGAACATTCG | Cy5 | |
Tabela 4: O ensaio do Controle Positivo Interno (IPC) incluindo sequências para os primers dianteiros e invertidos e a sonda.
| Volume por amostra (μL) | componente |
| 10 | Mistura de Mestre Ambiental |
| 1 | 20uM A. lig.1 F/R mix |
| 1 | 2.5uM A. lig.1 sonda |
| 1 | Mix de primer IPC 5uM (HemT-F/ R) |
| 0.75 | Sonda IPC 2.5uM (HemT-P) |
| 1.5 | 1 X 103 de concentração do modelo IPC |
| 2.75 | H20 |
| 2 | amostra |
| 20 | Total Volume |
Tabela 5: O mix PCR usado para o ensaio A.lig.1 multiplexed com o ensaio IPC.
| passo | Temperatura (°C) | Hora | |
| 1 | Denaturação Inicial | 95 | 10 min. |
| 2 | Desnaturar | 95 | 15 seg |
| 3 | recozimento | 60 | 1 min |
| 4 | Vá para o Passo 2, repita 39X |
Tabela 6: Condições de reação para o ensaio A.lig.1.
Discussão
Como em qualquer estudo, definir a questão a ser abordada é o primeiro passo e o desenho do ensaio eDNA depende do escopo do estudo26. Por exemplo, se o objetivo da pesquisa ou pesquisa é detectar uma ou algumas espécies, um ensaio baseado em sonda direcionada é o melhor. Se, no entanto, o objetivo é avaliar uma suíte maior ou conjunto de espécies, ensaios de metabarcocote de alto rendimento são mais adequados. Uma vez determinado qual abordagem tomar, recomenda-se um estudo piloto, incluindo design de ensaio, testes e otimização24. O design de ensaios começa com uma lista de espécies descritas em Figura 1. Esta lista será a base para entender o quão bem um ensaio funciona em termos de especificidade e a faixa geográfica a que pode ser aplicado6,10. Ele é encorajado a projetar o ensaio para uma área geográfica específica, permitindo que o designer teste melhor um ensaio para reatividade cruzada contra outras espécies nessa área, e esteja ciente das limitações que isso tem em estender um ensaio para outras áreas onde uma espécie-alvo pode ocorrer24. Uma vez que a lista esteja completa, as sequências podem ser baixadas em bancos de dados genéticos públicos. Uma vez que esses bancos de dados estão incompletos27, deve-se sequenciar o maior número possível de espécies da lista em casa para completar o banco de dados de referência local de sequências que serão usadas no projeto do ensaio. Priorize espécies relacionadas a co-ocorrência, pois estas são as não-metas mais prováveis que irão amplificar. Focar em todas as espécies dentro do mesmo gênero ou família como a espécie alvo é um bom lugar para começar. Comparações com espécies intimamente relacionadas ajudarão a identificar regiões sequenciais exclusivas das espécies-alvo. Isso pode ajudar a informar como o ensaio pode funcionar em outros sistemas ou locais. As regiões mitocondriais são a escolha usual para o desenvolvimento de ensaios, porque mais informações sequenciais de uma variedade mais ampla de espécies estão disponíveis em genes mitocondriais que têm sido usados em códigos de barras de projetos de vida, e porque o DNA mitocondrial está presente em uma concentração muito maior em cópias/células do que o DNA nuclear24,28,29. Várias regiões genéticas devem ser avaliadas para maior desenvolvimento de ensaios, uma vez que a cobertura de sequência varia entre os impostos nos bancos de dados do repositório genético. Depois que este banco de dados local de sequências de referência é criado, uma combinação de visualização manual de dados de sequência alinhadas e programas de software de computador é usada para projetar os ensaios de primer/teste. Não se deve confiar estritamente em software para determinar quais ensaios testar. É importante verificar visualmente sobre alinhamentos onde os primers e sondas se sentam nos alvos e não-alvos para obter uma melhor compreensão de como eles podem agir em um PCR. Finalmente, a triagem e otimização de ensaios inclui três níveis (em silico, in vitro e in situ)6,7,24,25. No design e testes de silico são importantes para produzir uma pequena lista de ensaios com uma boa chance de sucesso, mas testes empíricos (in vitro) são cruciais para selecionar o ensaio com o melhor desempenho real. A otimização in vitro e os testes de ensaios incluem medir a eficiência da reação e definir a sensibilidade e especificidade do ensaio. Limites de detecção e quantificação são dois parâmetros muitas vezes negligenciados no desenvolvimento de ensaios, mas importantes para a interpretação de dados. Ao executar várias réplicas das curvas padrão para um ensaio, LOD e LOQ podem ser facilmente medidos1,5,30. Poucos estudos discutem resultados com relação ao LOD ou LOQ do ensaio, mas Sengupta et al. (2019) incorporam o LOD e LOQ de seus ensaios em sua interpretação de dados e gráficos para uma compreensão mais clara de seus resultados31. Os controles positivos internos também devem ser multiplexados no ensaio projetado. Sem testes para inibição de PCR nas amostras, falsos negativos podem ocorrer24,32. Propomos o uso de um ensaio de IPC multiplexado com o ensaio de destino como o método mais fácil para testes de inibição de PCR23. Por fim, é necessário testar in situ o ensaio a partir de amostras coletadas em campo e laboratório para garantir que a amplificação do alvo ocorra em amostras ambientais24.
Existem limitações para o uso de ensaios qPCR específicos da espécie, baseados em sondas, com amostras de eDNA. Por exemplo, o design de vários ensaios para testes pode ser limitado pela disponibilidade de sequência, e o compromisso pode ser necessário em aspectos do desempenho do ensaio. Essas escolhas devem ser orientadas pelas metas do estudo e devem ser relatadas com os resultados26. Por exemplo, se o objetivo é a detecção de uma espécie rara e poucos pontos positivos são esperados, um ensaio com especificidade imperfeita (ou seja, amplificação de espécies não-alvo) poderia ser usado se todas as detecções forem verificadas por sequenciamento. Se o objetivo é monitorar a faixa geográfica de uma espécie e os dados de concentração de eDNA não forem necessários, um ensaio com eficiência imperfeita poderia ser usado e dados relatados apenas como por cento de detecção. Além disso, a menos que todos os potenciais conspecíficos sejam testados em laboratório, o que raramente é possível, não se pode saber com absoluta certeza a verdadeira especificidade de um ensaio. Por exemplo, o ensaio foi projetado e testado contra várias espécies de mexilhões de água doce no rio Clinch. Para usar este ensaio em um sistema fluvial diferente, precisaríamos testá-lo contra um conjunto de espécies no novo local. A variação genética dentro da espécie ou população que não é testada durante o desenvolvimento do ensaio também pode afetar a especificidade. Finalmente, mesmo que tenha sido verificado um ensaio ter alto desempenho técnico; as condições mudam ao trabalhar no campo. Condições relacionadas a não ensaios, como fluxo de água, pH e comportamento animal podem alterar a detectabilidade do eDNA, assim como o uso de diferentes protocolos de coleta e extração de eDNA. O uso de ensaios otimizados e bem descritos ajudará a facilitar a compreensão da influência que tais parâmetros têm na detecção de eDNA.
O campo do eDNA está amadurecendo além do estágio da análise exploratória para aumentar a padronização de métodos e técnicas. Esses desenvolvimentos melhorarão nossa compreensão das técnicas, habilidades e limitações do eDNA. O processo de otimização que delineamos acima melhora a sensibilidade, especificidade e reprodutibilidade de um ensaio. O objetivo final deste refinamento e padronização dos métodos eDNA é melhorar as habilidades dos pesquisadores para fazer inferências baseadas em dados eDNA, bem como aumentar a confiança do usuário final e do titular da estaca nos resultados.
Divulgações
Os autores não declaram conflito de interesses. Os patrocinadores do financiamento não tiveram papel na concepção do estudo; na coleta, análises ou interpretação dos dados; na escrita do manuscrito; ou na decisão de publicar os resultados.
Agradecimentos
Agradecemos a Alvi Wadud e Trudi Frost que ajudaram no desenvolvimento e teste de primer. O financiamento para o projeto de ensaio relatado neste estudo foi fornecido pelo Programa Estratégico de Pesquisa e Desenvolvimento Ambiental do Departamento de Defesa (RC19-1156). Qualquer uso de nomes comerciais, produtos ou firmais é apenas para fins descritivos e não implica endosso do Governo dos EUA. Os dados gerados durante este estudo estão disponíveis como uma https://doi.org/10.5066/P9BIGOS5 de liberação de dados do USGS.
Materiais
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
Referências
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados