Method Article
Sviluppo e test di test quantitativi PCR specifici per specie per applicazioni del DNA ambientale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I test del DNA ambientale richiedono una progettazione, un test, un'ottimizzazione e una convalida rigorosi prima che possa iniziare la raccolta dei dati sul campo. Qui, presentiamo un protocollo per portare gli utenti attraverso ogni fase della progettazione di un saggio qPCR specifico per specie e basato su sonda per il rilevamento e la quantificazione di un DNA di specie bersaglio da campioni ambientali.
Abstract
Sono in fase di sviluppo nuovi metodi non invasivi per individuare e monitorare la presenza delle specie per aiutare nella pesca e nella gestione della conservazione della fauna selvatica. L'uso di campioni di DNA ambientale (eDNA) per rilevare macrobiota è uno di questi gruppi di metodi che sta rapidamente diventando popolare e viene implementato nei programmi di gestione nazionali. Qui ci concentriamo sullo sviluppo di saggi mirati specifici per specie per applicazioni PCR (qPCR) quantitative basate su sonda. L'utilizzo di qPCR basato su sonda offre una specificità maggiore di quella possibile con i soli primer. Inoltre, la capacità di quantificare la quantità di DNA in un campione può essere utile nella nostra comprensione dell'ecologia dell'eDNA e nell'interpretazione dei modelli di rilevamento eDNA sul campo. È necessaria un'attenta considerazione nello sviluppo e nella sperimentazione di questi saggi per garantire la sensibilità e la specificità dell'individuazione delle specie bersaglio da un campione ambientale. In questo protocollo diseticheremo i passaggi necessari per progettare e testare saggi basati su sonde per il rilevamento di una specie bersaglio; tra cui la creazione di database di sequenza, la progettazione di saggi, la selezione e l'ottimizzazione dei saggi, le prestazioni dei test e la convalida sul campo. Seguire questi passaggi aiuterà a ottenere un saggio efficiente, sensibile e specifico che può essere utilizzato con sicurezza. Dimostriamo questo processo con il nostro saggio progettato per le popolazioni del mucket(Actinonaias ligamentina),una specie di cozze d'acqua dolce che si trova nel fiume Clinch, negli Stati Uniti.
Introduzione
Ricercatori e manager si stanno sempre più interessando all'uso di test del DNA ambientale per il rilevamento delle specie. Per tre decenni, la PCR quantitativa o in tempo reale (qPCR/rtPCR) è stata utilizzata in numerosi campi per il rilevamento e la quantificazione specifici della sequenza degli acidi nucleici1,2. Nell'ambito relativamente nuovo della ricerca eDNA, l'uso di questi saggi con una curva standard per la quantificazione di copie del DNA bersaglio per volume o peso del campione di eDNA è diventato una pratica di routine. Le sequenze di DNA mitocondriale sono generalmente mirate nei saggi eDNA perché il genoma mitocondriale è presente in migliaia di copie per cellula, ma sono possibili anche saggi per sequenze di DNA nucleare o RNA. È fondamentale comprendere che i test pubblicati per i campioni di eDNA non sono sempre uguali nelle prestazioni. L'affidabilità di un saggio nel rilevare solo il DNA di una specie bersaglio (cioè la specificità) e nel rilevamento di basse quantità di DNA bersaglio (cioè sensibilità) può variare considerevolmente a causa delle differenze nel modo in cui il saggio è stato progettato, selezionato, ottimizzato e testato. La segnalazione di misure quantitative delle prestazioni dei saggi è stata in precedenza ampiamente trascurata, ma recentemente stanno emergendo standard per migliorare la trasparenza nello sviluppo dei saggi3,4,5,6,7,8.
Ottimizzazione e reporting degli ausili per le prestazioni dei saggi nella progettazione e nell'interpretazione degli studi dei risultati dell'indagine eDNA. I saggi che reagiscono incrociatamente con il DNA delle specie non bersaglio potrebbero portare a falsi rilevamenti positivi, mentre i test con scarsa sensibilità potrebbero non riuscire a rilevare il DNA della specie bersaglio anche quando è presente nel campione (falsi negativi). La comprensione della sensibilità e della selettività dei saggi aiuterà a informare lo sforzo di campionamento necessario per rilevare specie rare. Poiché ci sono molte fonti naturali di variazione nell'eDNA, gli studi devono limitare il più possibile le fonti controllabili di variazione, inclusa la piena ottimizzazione e caratterizzazione del saggio eDNA3.
Le condizioni che influiscono direttamente sulla specificità o sulla sensibilità di un saggio cambieranno le prestazioni del saggio. Ciò può verificarsi in diverse condizioni di laboratorio (ad esempio, reagenti diversi, utenti, macchine, ecc.). Pertanto, questo protocollo dovrebbe essere rivisitato quando si applica un saggio in nuove condizioni. Anche i saggi ben caratterizzati in letteratura devono essere testati e ottimizzati quando adottati da un nuovo laboratorio o quando si utilizzano diversi reagenti (ad esempio, soluzione master-mix)5,9. La specificità del saggio può cambiare se applicata a una regione geografica diversa, perché il saggio viene applicato a campioni di una nuova comunità biotica che possono includere specie non bersaglio contro le quali il saggio non è stato testato e possono verificarsi variazioni genetiche nelle specie bersaglio. Anche in questo caso, il saggio deve essere rivalutazione se utilizzato in una nuova posizione. Le condizioni sul campo differiscono dalle condizioni di laboratorio perché nel campo gli inibitori della PCR hanno maggiori probabilità di essere presenti nei campioni. Gli inibitori della PCR influenzano direttamente la reazione di amplificazione e quindi influenzano le prestazioni del saggio. Per questo motivo, è necessario un controllo positivo interno quando si sviluppa un saggio eDNA.
Infine, le condizioni ambientali sul campo possono influenzare le molecole di DNA della specie bersaglio e la loro cattura attraverso la degradazione, il trasporto e la ritenzione del DNA. Inoltre, diversi protocolli per la raccolta e l'estrazione del DNA variano nella loro efficienza e capacità di trattenere il DNA. Tuttavia, è importante notare che questi processi influenzano la rilevabilità dell'eDNA ma non le prestazioni di un saggio molecolare. Pertanto, la rilevabilità del DNA dalle specie bersaglio nei campioni di campo è una funzione sia delle prestazioni tecniche del saggio qPCR, sia delle condizioni sul campo e dei protocolli di raccolta, stoccaggio ed estrazione. Quando si utilizza un saggio ben caratterizzato e altamente performante, gli utenti possono sentirsi sicuri delle capacità del saggio; consentendo ai ricercatori di concentrarsi ora sulla comprensione dei fattori di dosaggio esterni (ad esempio, variabili ambientali, differenze nei protocolli di cattura o estrazione) che influenzano il rilevamento dell'eDNA.
Qui ci concentriamo specificamente sulle prestazioni tecniche di dosaggio attraverso una progettazione e un'ottimizzazione rigorose. Dimostriamo il protocollo utilizzando un saggio a base di sonda sviluppato per il rilevamento di una cozza d'acqua dolce, il mucket (Actinonaias ligamentina), dall'acqua campionata nel fiume Clinch, USA. Recentemente Thalinger et al. Il design del saggio seguendo il nostro protocollo porterà un saggio al livello 4 diThalinger et al. A questo punto le prestazioni tecniche di un saggio saranno ottimizzate e saranno pronte per l'uso regolare in applicazioni di laboratorio e sul campo. L'ulteriore utilizzo del saggio in laboratorio, mesocosmo ed esperimenti sul campo può quindi affrontare le domande riguardanti il rilevamento dell'eDNA e i fattori che influenzano la rilevabilità, i passaggi finali per la convalida di livello5 6.
Protocollo
1. Generazione di una banca dati di sequenza di sequenze di DNA mitocondriale da specie di interesse target e non bersaglio
- Definire la domanda, gli obiettivi e il sistema in corso di definizione. Identificare le specie bersaglio per il rilevamento dell'eDNA. Identificare il sistema geografico in cui verrà utilizzato il saggio. Fare un elenco delle specie di interesse, comprese le specie bersaglio, le specie simpattrici (co-presenti) all'interno degli stessi taxa (di solito ordine o livello familiare) e le specie allopatrica strettamente correlate, quelle che potrebbero non essere nella stessa posizione geografica del bersaglio (Figura 1).
NOTA: Qui sono state prese di mira le popolazioni del fiume Clinch della specie A. ligamentina. - Cerca e scarica sequenze da più regioni geniche per le specie nell'elenco del passaggio 1. È possibile utilizzare database di sequenze come NCBI (National Center for Biotechnology Information), BOLD (Barcode of Life Database), EMBL (European Molecular Biology Laboratory) e DDBJ (DNA Data Bank of Japan). NCBI, EMBL e DDBJ condividono tutte le informazioni sulla sequenza.
- Utilizzando la banca dati nucleotidica dell'NCBI, cercare l'organismo bersaglio (ad esempio, Actinonaias ligamentina) e la regione genica (ad esempio, citocromo c ossidasi I (COI) o NADH-deidrogenasi 1 (ND1); Stringa di ricerca di esempio: Actinonaias ligamentina E ND1)
- Selezionare quindi tutte le sequenze che corrispondono alle specifiche e selezionare Invia a. Scegliere Record completo, Formato file e download come GenBank o FASTA, quindi Crea file. Queste sequenze vengono ora salvate nel computer.
- Ripetere questi passaggi per tutte le specie nell'elenco definito nel passaggio 1. Conservare le sequenze per ogni regione genica in un file separato in quanto queste verranno analizzate separatamente.
- Scaricare tutte le sequenze rilevanti (o una grande proporzione rappresentativa di sequenze) per le specie bersaglio identificate nel passaggio 1. Includi varianti geografiche, se possibile.
- Ripetere le sequenze di ricerca e download per le specie non bersaglio correlate e simpatiche dello stesso gruppo tassonomico identificate nel passaggio 1 (ad esempio, se la specie bersaglio è la mucket (A. ligamentina) sequenze di download per tutte le altre specie di mitili d'acqua dolce nella famiglia Unionidae che si verificano nel sistema di interesse).
- Ripetere la ricerca e il download di specie strettamente correlate ma allopatrica (geograficamente separate) elencate nel passaggio 1.1.
NOTA: non tutte le specie (obiettivi e non target) saranno disponibili nelle banche dati pubbliche. Aumentare la banca dati di riferimento locale amplificando e sequenziando esemplari di specie di interesse interno verificati tassonomicamente. Se si lavora con una specie che ha un'elevata diversità genetica all'interno delle specie o si lavora in un'area geograficamente vasta in cui ci si può aspettare varianti geografiche, raccogliere sequenze da tutto l'areale.
2. Progettazione del saggio
- Allineare le sequenze da ogni regione genica separatamente utilizzando un software di allineamento che può essere trovato in vari programmi di editing di sequenze genetiche e bioinformatici. Eseguire questo allineamento per ciascuna delle diverse regioni geniche.
- Ad esempio, utilizzando il software Geneious Prime (https://www.geneious.com) importare i file di sequenza scaricati nel programma.
- Creare cartelle separate per ogni area genica.
- All'interno di una cartella che contiene sequenze da un'area genica, selezionare tutte le sequenze.
- Utilizzate lo strumento Allineamento multiplo per creare un allineamento nucleotidico delle sequenze selezionate. Ci possono essere diverse opzioni per il tipo di allineamento, utilizzando gli allineamenti Geneious o MUSCLE e i parametri predefiniti funzionano bene.
- Scegli le aree promettenti per la progettazione del test attraverso la visualizzazione di dati di sequenza allineati. Una regione che ha molti dati di sequenza disponibili per le specie di interesse, è molto divergente tra le specie e mostra che una bassa variazione all'interno delle specie è un buon candidato. Ciò aumenterà la probabilità che primer e sonde progettati siano in grado di discriminare il bersaglio da specie non bersaglio, garantendo al contempo che le varianti intraspecifiche si amplificano con il saggio.
- Progettazione di primer e sonde di dosaggio.
- Utilizzare il software di progettazione del test qPCR e seguire le istruzioni. Lo strumento PrimerQuest (https://www.idtdna.com/) di IDT per progettare 5 set di test qPCR è stato utilizzato qui.
- Incollare la sequenza selezionata nel passaggio 2.2 nella casella di entrata Sequenza. Se l'allineamento ha creato spazi, eliminarli dalla sequenza.
- Selezionate qPCR 2 Primers + Probe nell'opzione Scegli progettazione (Choose Your Design).
- Scarica i saggi consigliati.
- Copiare le sequenze dal primer in avanti del primo saggio e cercare questa sequenza di primer nell'allineamento creato nel passaggio 2.1.4. Se usate Prime geneioso(Geneious Prime), utilizzate lo strumento Annota (Annotate) e Previsione (Predict) per aggiungere l'area del primer all'allineamento. Eseguire questa questa questa questa situazione per tutte le combinazioni di primer e sonda (Figura 2).
- Ispezionare queste regioni dell'allineamento alla ricerca di variazioni all'interno delle specie bersaglio e all'interno delle specie co-presenti.
- Se c'è una variazione genetica intraspecifica, cerca saggi in cui i primer e la sonda non rientrano in queste regioni.
- Per prevenire l'amplificazione delle specie non bersaglio, cercare disallineamenti con specie non bersaglio. Scegli i test con il maggior numero di disallineamenti per un'ulteriore convalida. (2018) suggeriscono di scegliere insiemi con almeno due delle tre regioni (i due primer, o un primer e una sonda) con almeno due disallineamenti con tutte le specie non bersaglio. Tuttavia, tenere presente che le discrepanze alla sonda contribuiscono meno alla specificità10.
NOTA: Le differenze all'interno di 3 coppie di basi dell'estremità 3' di ogni primer aumentano la specificità meglio delle differenze all'estremità 5 ' dei primer10.
- Considerare i seguenti parametri importanti nella progettazione del saggio.
- Determinare le temperature di fusione e ricottura dei primer e della sonda. Idealmente la temperatura di fusione (Tm) dei primer dovrebbe essere compresa tra 60-64 °C ed entro 2 °C l'uno dall'altro, e il Tm della sonda dovrebbe essere superiore di 6-8 gradi rispetto al Tm dei primer. Impostare la temperatura di ricottura (Ta) della reazione qPCR 5 °C al di sotto della temperatura di fusione, intorno a 55-60 °C11.
- Esaminare il contenuto gc. Scegli tra il contenuto GC 35 - 65% ed evita le aree con 4 o più G consecutivi. Avere 1 o 2 G o C nelle 5 ultime basi dell'estremità 3 ' del primer (morsetto GC) potrebbe aumentare la specificità in quanto aiuterebbe il primer a fare un legame più forte12.
- Cerca strutture per tornanti e dimeri. Primer di prova e sonda per strutture e dimeri di forcine previsti utilizzando un programma di analisi oligonucleotidi (ad esempio, OligoAnalyzer -IDT13; OligoCalculatore14). Queste strutture possono causare amplificazione non bersaglio e minore efficienza. Evitare saggi che si prevede formino queste strutture.
- Determinare la lunghezza del primer. Puntare per primer tra 18-25 basi di lunghezza e lunghezza della sonda tra 20 -25 basi. Primer e sonde più lunghi possono avere una minore efficienza di amplificazione.
- Determinare la lunghezza dell'amplicon. Dovrebbe essere compreso tra circa 100 e 250 coppie di basi. Questa gamma è generalmente abbastanza breve per un'elevata efficienza PCR, ma abbastanza lunga da facilitare la verifica con il sequenziamento Sanger4,15.
- Progettare sonde. Assicurarsi che le sonde non abbiano una base G all'estremità di 5', perché potrebbe smorzare il segnale dai coloranti verdi e gialli11. Abbiamo progettato sonde a doppia tempra, con tempra IDT 3IABkFQ e ZEN e fluorofori FAM o HEX.
NOTA: Determinare le sonde MGB: le sonde TaqMan MGB (legante a scanalatura minore) sono spesso utilizzate per studi eDNA. Tuttavia, poiché queste sonde sono molto corte, possono legarsi a non-bersagli anche con una mancata corrispondenza della coppia di basi 2 o3 10. - Determinare la sonda Tm. La temperatura di fusione della sonda deve essere superiore di 6-8°C rispetto ai primer. Temperature più basse riducono il successo di legame della sonda.
- Determinare la lunghezza e la posizione della sonda. La sonda deve avere una lunghezza compresa tra 20 e 25 bp e si trova in posizione ideale vicino al sito di attacco del primer sullo stesso filamento senza sovrapporla.
3. Screening e ottimizzazione del saggio
- Nello sviluppo e nel test del saggio silico. Prima di ordinare i set di sonde primer, valutare la specificità (potenziale amplificazione non bersaglio) testando l'amplificazione del primer in silico.
- Testare i primer attraverso primer-blast16 di NCBI o programmi simili in grado di identificare potenziali non-bersagli nel database NT/nr NCBI che potrebbero amplificarsi con il saggio. Se utilizzate primer primer-blast incollare i primir nella casella Usa il mio primer in Parametri primer. Nelle opzioni Parametri di verifica della specificità della coppia primer, selezionare nr come database e digitare l'Ordine dell'organismo di interesse (ad esempio, "Unionida" o "Unionoida") nella casella Organismo.
- Continuare a valutare visivamente i set di primer/probe sui dati di sequenza allineati.
- Per valutare primer e sonde contemporaneamente in silico, creare una stringa di testo del primer in avanti, 12 N, la sonda, 12 N e il complemento inverso del primer inverso. Se la sequenza della sonda si trova entro 12 coppie di basi di uno dei primer, utilizzare il numero di N corrispondente al numero di coppie di basi tra il primer e la sonda.
- Utilizzare la ricerca Nucleotide Blast (Blastn) di NCBI per cercare nel database nr17. Utilizzare la scheda Tassonomia per cercare specie non bersaglio con poche discrepanze; questi devono essere testati in laboratorio durante l'ottimizzazione del test.
NOTA: Nel silico i test aiutano a escludere saggi non specifici, ma i saggi potenzialmente specifici devono essere testati empiricamente (in vitro), poiché non tutte le specie hanno sequenze nei database genetici e primer e sonde possono ancora legarsi a non-target anche se ritenuti improbabili dal software.
- Scegli da tre a cinque combinazioni primer/sonda da testare in laboratorio.
- Ordina primer, sonde e uno standard di DNA sintetico, nonché primir aggiuntivi a coda M13 per il sequenziamento dell'amplicon.
- Ordina primer e sonde oligonucleotidi sintetici da un'azienda che produce oligoni. Le sonde sono etichettate con un colorante fluorescente e un quencher. Diversi fluorofori devono essere selezionati per i test che devono essere multiplexati. Controllare lo strumento qPCR per un elenco dei fluorofori che lo strumento è in grado di rilevare.
- Progettare e ordinare primer a coda M13 per la verifica dei rilevamenti qPCR con sequenziamento Sanger aggiungendo la sequenza M13 Forward (-20), GTA AAA CGA CGG CCA GT, all'estremità 5' del primer in avanti, e la sequenza M13 Reverse (-27), CAG GAA ACA GCT ATG AC, all'estremità 5' del primer inverso.
- Lo standard del DNA sintetico contiene la sequenza bersaglio (comprese le regioni primer) ad una concentrazione nota in copie/μL. Acquisire lo standard sintetico dalla stessa azienda che produce i primer e la sonda. Seguire le raccomandazioni del produttore per la sospensione e lo stoccaggio. Diluire gli standard nel tampone TE con un supporto per tRNA utilizzando stoviglie a bassa ritenzione per ridurre l'idrolisi e legarsi alle superfici.
NOTA: Se la curva standard non funziona bene (scarsa efficienza PCR, vedere il passaggio 3.4.2), provare a sospendere di nuovo lo standard in acqua o Tris-HCl. - Sospendere primer e sonde in acqua priva di nucleasi, Tris-HCl o tampone TE a concentrazioni convenienti per l'uso di test. Generalmente, diluire le scorte di lavoro 20 volte nel mix principale per ottenere la concentrazione ottimizzata del saggio finale. Conservare gli oligo sospesi a una costante di -20 °C quando non sono in uso.
- Ottimizzazione e test dei saggi in vitro (in laboratorio). Rifiutare i saggi che hanno scarsa efficienza, reagiscono incrociatamente con specie co-presenti o hanno scarsa sensibilità18. Includere l'uso di un controllo positivo interno (IPC) durante lo sviluppo del saggio e durante l'esecuzione di campioni effettivi.
- In primo luogo, trovare la temperatura ottimale e i valori di concentrazione di primer / sonda per il saggio. Una volta ottimizzati questi parametri per l'efficienza della PCR (Fase 3.4.2), la reattività incrociata (Passaggio 3.4.3) e la sensibilità (Passaggio 3.4.4), procedere alla prova del test con un IPC multiplexato (Passaggio 3.4.5).
- Testare la temperatura di ricottura ottimale (Ta) per primer e sonde utilizzando un gradiente di temperatura PCR centrato di 5 ° C al di sotto del primer medio previsto Tm.
- Testare le concentrazioni ottimali di primer e sonda. Tipicamente, vengono testate concentrazioni di primer da 200 nM, 400 nM e 800 nM e 75 nM, 125 nM e 200 nM di concentrazione di sonda.
- Creare una curva standard e determinare l'efficienza e l'intervallo lineare. Testare almeno sei diluizioni di 10 volte di uno standard di DNA sintetico contenente la sequenza bersaglio, a circa10 0 copie/reazione a 105 copie/reazione (Figura 3A).
- Utilizzare il software qPCR per tracciare il valore Cq (soglia per il ciclo alla quantificazione) di ogni standard sull'asse y e la base di log 10 della concentrazione standard iniziale in copie/reazione sull'asse x. Il software qPCR deve eseguire automaticamente una regressione lineare (Figura 3B).
- Calcolare l'efficienza dalla pendenza della regressione, E = -1 + 10(-1/pendenza). Ad esempio, se la pendenza è -3,4, E= -1 + 10(0,29) = 0,97 o 97%. Controllare anche i valori r2 che indicano quanto bene le repliche standard si adattano alla curva. Anche il software qPCR deve calcolare automaticamente anche questo (Figura 3B). Puntare a valori di efficienza del 100% (±10%) e r2 valori di ≥0,989,15,19,20,21,22.
- Ispezionare visivamente la curva standard per la distorsione, o cioè le deviazioni dalla regressione in una direzione coerente o per le scarse prestazioni della curva standard misurate in base all'efficienza e ai valori r2 (Figura 3C e 3D).
- Specificità: valutare la reattività incrociata con specie non bersaglio per ridurre la probabilità di falsi positivi. Laddove i rilevamenti di eDNA possano comportare costose decisioni di gestione, verificare i rilevamenti positivi mediante sequenziamento dell'amplicon.
- Non-target: Eseguire il test contro le estrazioni genomiche di DNA di esemplari verificati tassonomicamente di specie correlate e di specie geograficamente co-presenti; con la massima priorità è testare contro specie strettamente correlate e co-presenti. Utilizzare concentrazioni totali di DNA simili sia per campioni bersaglio che non bersaglio. La concentrazione scelta dovrebbe produrre amplificazione da campioni di specie bersaglio vicino al centro dell'intervallo lineare della curva standard. L'amplificazione deve essere osservata solo con le specie bersaglio.
- Se si osserva un'amplificazione non bersaglio, pulire e sequenziare il prodotto per verificarne l'identità. Non è raro osservare la contaminazione delle specie bersaglio in campioni di tessuto di specie non bersaglio, pertanto tutte le amplificazioni in questa fase dovrebbero essere verificate sequenziando. Ricampionare gli ampli puliti dai test di specificità utilizzando i primer a coda M13 e la sequenza con primer M13.
- Nel laboratorio post-PCR, trasferire i prodotti qPCR da sequenziare in tubi freschi. Rimuovere i primer residui e i componenti di reazione con un kit di pulizia (ad esempio, MinElute PCR Purification Kit).
- Effettuare diluizioni 1:100 delle eluizioni e amplificare 1 μL di ciascuno per 30 cicli in una reazione PCR da 50 μL con i primer a coda M13 e una polimerasi ad alta fedeltà (ad esempio, DNA polimerasi ad alta fedeltà phusion).
- Eseguire 10 μL di ogni reazione su un gel di agarosio dell'1% per verificare la presenza di una singola banda della dimensione prevista. Se non viene osservata alcuna banda, aumentare il numero di cicli o la quantità di campione. Se si osservano più bande, il gel purifica la banda della dimensione prevista.
- Rimuovere i primer residui e i componenti di reazione con un kit di pulizia come sopra e misurare le concentrazioni di DNA delle eluizioni.
- Impostare le reazioni di sequenziamento con i primer M13 secondo le istruzioni della struttura di sequenziamento.
NOTA: Non aprire mai campioni amplificati nel laboratorio qPCR. Preparare campioni per il sequenziamento in un laboratorio dedicato ai campioni post-PCR.
- Sensibilità: La sensibilità influisce sulla possibilità di falsi negativi, o fallimenti nel rilevare il DNA della specie bersaglio quando è presente. Valutare il limite di rivelazione (LOD) e il limite di quantificazione (LOQ) per ciascun saggio. Infine, includere un controllo positivo interno (IPC) per valutare l'inibizione pcr dei campioni. Multiplex e testare questo saggio IPC con il saggio progettato per garantire che i due test non interferiscano tra loro.
- LOD: Effettuare sei diluizioni seriali 4 volte dello standard del DNA sintetico, con 8-24 repliche per diluizione standard (Figura 4). Calcola la concentrazione iniziale più bassa con il rilevamento del 95%. I grafici LOD e LOQ possono essere generati con uno script R5della calcolatrice LOD/LOQ.
NOTA: I dati sotto il LOD non devono essere censurati. A causa della specificità della PCR, non esiste un limite inferiore per i veri positivi. Il LOD è la più alta concentrazione al di sotto della quale ci si può aspettare che si verifichino falsi negativi. - LOQ: Dalla stessa serie di diluizione, calcolare la concentrazione standard di DNA iniziale più bassa quantificabile con un coefficiente di variazione (CV) inferiore al 35%.
NOTA: LOD e LOQ devono essere riportati in copie/reazione. Quando si utilizza un saggio convalidato e campioni di campo amplificano al di sotto del LOQ, i risultati devono essere segnalati come rilevamenti in % anziché come concentrazioni di eDNA, perché la concentrazione esatta non può essere misurata con fiducia5.
- LOD: Effettuare sei diluizioni seriali 4 volte dello standard del DNA sintetico, con 8-24 repliche per diluizione standard (Figura 4). Calcola la concentrazione iniziale più bassa con il rilevamento del 95%. I grafici LOD e LOQ possono essere generati con uno script R5della calcolatrice LOD/LOQ.
- Utilizzare un controllo positivo interno (IPC) per testare l'inibizione della PCR. L'inibizione può portare a una diminuzione della sensibilità e ai falsi negativi. Testare la capacità del saggio IPC di essere multiplexato con il saggio di destinazione.
- Un test IPC può essere multiplexato con il saggio bersaglio usando una sonda con un colorante reporter diverso dal saggio di destinazione. Questo saggio IPC consiste in una breve sequenza di DNA sintetico da una specie non correlata al taxa bersaglio, incorporata nel mix principale qPCR ad una bassa concentrazione di circa 102 copie / reazione, insieme a primer e sonde che lo rilevano. Questa minore concentrazione è necessaria per evitare la concorrenza con la sequenza bersaglio per polimerasi e nucleotidi23.
- Confrontare il valore Cq del modello IPC dell'esempio con quello del modello IPC nel controllo no template. In questo nessun controllo modello (NTC), l'unico input di DNA è quello del modello IPC. Il modello IPC in questa reazione dovrebbe amplificarsi come previsto. Se il modello IPC in un campione si amplifica a 2 o più cicli diversi da quello del modello IPC nell'NTC, il campione eDNA viene inibito. I campioni che mostrano inibizione possono essere diluiti 1:10 e ri-testati. Se un campione rimane inibito, tale campione deve essere rimosso dall'analisi.
- In primo luogo, trovare la temperatura ottimale e i valori di concentrazione di primer / sonda per il saggio. Una volta ottimizzati questi parametri per l'efficienza della PCR (Fase 3.4.2), la reattività incrociata (Passaggio 3.4.3) e la sensibilità (Passaggio 3.4.4), procedere alla prova del test con un IPC multiplexato (Passaggio 3.4.5).
- Sviluppo e test del saggio in situ
- In laboratorio: Se è disponibile l'accesso all'organismo in laboratorio e alle specie simpatiche; prelevare campioni d'acqua da recinti con queste specie, elaborare i campioni e testare il saggio su questi campioni di eDNA. Prodotti di sequenza come sopra per verificare l'amplificazione del bersaglio previsto utilizzando i primer a coda M13.
- Sul campo:
- Identificare i siti in cui è noto che l'organismo bersaglio si verifica e noto che non si verifica. È preferibile avere una certa misura di abbondanza in ogni sito in cui si verifica la specie bersaglio.
- Decidere quali volumi di campioni e metodi di raccolta dei campioni (ad esempio filtrazione, centrifugazione, ecc.) verranno utilizzati.
- Includere un controllo vuoto o negativo in ogni sito, si tratta di acqua pulita che è stata portata nel sito sul campo e quindi raccolta e preparata con le stesse attrezzature sul campo e protocolli utilizzati per il campionamento eDNA24. Lo scopo del campo vuoto è quello di rilevare la contaminazione delle attrezzature di campionamento e degli attrezzi da campo portati nel sito. Togliere il campo vuoto prima di elaborare i campioni d'acqua del campo.
- Prelevare più campioni d'acqua per sito, preferibilmente 3 campioni per sito.
- Di nuovo in laboratorio, elaborare ed estrarre campioni.
- Eseguire il saggio utilizzando una piastra impostata in modo simile alla figura 5A e confrontare la concentrazione e la frequenza di rilevamento dell'eDNA con le note differenze di sito in occorrenza e abbondanza. Confermare tutti i rilevamenti sequenziando24,25.
NOTA: Quanto sopra convaliderebbe un saggio attraverso il livello 4 della scala6 di Thalinger et al.s (2020) (ottimizzazione delle prestazioni tecniche del saggio) e inizierebbe a raccogliere dati a supporto della convalida del saggio di livello 5. Il livello 5 incorpora la modellazione della probabilità e l'uso del saggio per gli studi di ecologia eDNA. Riteniamo che ciò esula dall'ambito dello sviluppo di test di base, ma incoraggiamo queste applicazioni di saggi controllati in laboratorio e sul campo per migliorare la progettazione dei saggi e l'interpretazione dei dati.
Risultati
Nel progettare un saggio qPCR specifico per specie per il mucket (A. ligamentina), sono state scaricate le sequenze disponibili di tutte le specie Unionidae nel fiume Clinch. Anche specie strettamente imparentate come Lampsilis siliquoidea sono state incluse nel database di riferimento anche se non si trovano nello stesso fiume. Non tutte le specie del sistema fluviale di interesse sono state trovate in GenBank, quindi altre specie sono state sequenziate in casa. Le sequenze sono state allineate utilizzando il software Geneious e il software Primer Quest (IDT) è stato utilizzato per progettare più test. Cinque serie di primer e sonde sono stati aggiunti all'allineamento per la valutazione visiva (Figura 2). Sono stati quindi testati in silico utilizzando Primer-Blast, dopo di che sono stati ordinati per ulteriori test in vitro. In laboratorio, tutti i test sono stati testati utilizzando estrazioni di DNA di 27 specie disponibili per verificare la specificità. Un saggio (A.lig.1) ha amplificato con successo solo le specie bersaglio(tabella 1; La tabella 2). Questo test è andato avanti per ulteriori test sull'efficienza del test, LOD e LOQ. Ha una lunghezza dell'amplicon di 121 coppie di basi. La tabella 3 mostra la sequenza utilizzata per lo standard del DNA sintetico A. ligamentina. La figura 3A e la figura 3B mostrano i risultati di un saggio di successo con una buona efficienza e valori r2. La figura 3C e la figura 3D mostrano un saggio la cui curva standard ha una scarsa efficienza; questo saggio è stato scartato. Il LOD e il LOQ per il saggio selezionato (A.lig.1) sono stati entrambi trovati in 5,00 copie/reazione utilizzando il metodo discreto descritto in Klymus et al5. L'IPC che è stato multiplexato con il saggio (tabelle 3-6) non ha influenzato la curva standard del saggio A. ligamentina. L'IPC che usiamo è un frammento della trascrizione HemT del mouse. Questo test è stato preprogettato da IDT per un'altra applicazione, ma ne abbiamo modificato l'uso come IPC per le applicazioni eDNA del nostro laboratorio.
Una corsa qPCR di successo dovrebbe soddisfare determinati criteri per ogni misura delle prestazioni (ad esempio, amplificazione della curva standard, controllo genomico positivo del DNA, nessun controllo del modello e controllo positivo interno). Gli standard di dosaggio target dovrebbero avere curve di amplificazione esponenziali. Queste curve dovrebbero raggiungere un plateau del punto finale se consentito di eseguire cicli sufficienti. Questo è indicativo del fatto che la sonda fluorescente viene completamente consumata durante la reazione e che i livelli di fluorescenza raggiungono un limite massimo. Standard successivi amplificanti potrebbero non raggiungere un altopiano in 40 cicli. I controlli positivi (DNA genomico e IPC) dovrebbero avere lo stesso modello. Le incognite possono amplificarsi o meno, ma l'amplificazione in incognite dovrebbe avere anche un modello esponenziale e un plateau di endpoint (Figura 5).
In un qPCR di qualità, le diluizioni standard amplificano a Cq uniformemente distanziato di circa ogni 3,3 cicli per ogni differenza di concentrazione di 10 volte. Ogni replica di una diluizione standard si amplifica in modo strettamente raggruppato avendo quasi lo stesso Cq (rappresentato dai valori r2). Tutte le diluizioni standard devono presentare amplificazione (Figura 3A). In un qPCR scadente, gli standard possono mostrare una forma non esponenziale, variazioni irregolari nei valori Cq tra le diluizioni, non arrivare a un plateau di endpoint o alcune diluizioni potrebbero non amplificarsi affatto (Figura 3D).
I parametri importanti per una curva standard sono efficienza, r2, pendenza e intercetta y. L'efficienza dovrebbe scendere tra il 90% e il 110% con valori ideali vicini al 100% e i valori r2 dovrebbero essere superiori a 0,98 con risultati ideali che si avvicinano a 1,015,22. I valori di pendenza devono essere compreso tra -3,2 e -3,5 con risultati ideali vicini a -3,322. I valori dell'intercetta y dovrebbero scendere tra un Cq di 34-41 con risultati ideali con un Cq di 37,0. L'intercetta y è il Cq previsto di una reazione con 1 copia della sequenza bersaglio, l'unità più piccola che può essere misurata in un singolo qPCR. Le incognite con Cq maggiore dell'intercetta y sono suscettibili di essere inibite. Per rilevare il bersaglio in caso di inibizione o di un set di primer inefficiente può essere necessario eseguire più di 40 cicli di PCR, tuttavia la quantificazione non è possibile in queste circostanze e ulteriori controlli negativi senza la sequenza bersaglio, ma contenenti DNA totale simile alle incognite, dovrebbero essere eseguiti per escludere l'amplificazione da fonti non specifiche.
L'amplificazione del controllo positivo interno (IPC) in campioni sconosciuti deve essere confrontata con i risultati dell'IPC di controllo del modello negativo, in quanto non vi è competizione per i reagenti e non sono presenti inibitori. Le incognite con un IPC con un Cq di 2 cicli o superiore al valore Cq medio dell'NTC, o che non amplificano dovrebbero essere considerate inibite. Se nei campioni non sono presenti inibitori, tutta l'amplificazione IPC deve avere un raggruppamento stretto nel grafico con valori Cq vicini allo stesso ntc (Figura 6).
Infine, si è verificato un test in situ del saggio. Venti campioni d'acqua del fiume Clinch e tre campi vuoti sono stati filtrati tra il 25 e il 26 settembre 2019 entro 500 metri da un letto di cozze noto per avere A. ligamentina. Circa quattro campioni d'acqua da 1 L sono stati filtrati per luogo di campionamento. Siti di localizzazione inclusi nella parte inferiore del letto di cozze nel torrente, fondo del letto di cozze vicino alla riva, 100 m a valle del letto nel torrente, 500 m a valle del letto nel torrente e 500 m a valle del letto vicino alla riva(figura 7). Tornato in laboratorio, ogni filtro è stato tagliato a metà e il DNA è stato estratto solo dalla metà di un filtro. La metà del filtro rimanente per ciascun campione è stata conservata in un congelatore a -80 °C. I campioni sono stati quindi eseguiti utilizzando il saggio A.lig.1 multiplexato con l'IPC. Dei 23 campioni, cinque sono stati trovati inibiti. Questi campioni sono stati diluiti 1:10 e le diluizioni sono state rimesse. Diciannove dei 20 campioni di campo amplificati utilizzando il saggio progettato. Di questi 19 campioni, cinque erano al di sopra del LOD e del LOQ del saggio di 5 copie/reazione; il che significa che la maggior parte dei campioni aveva un rilevamento eDNA, ma a un livello in cui è probabile che si verifichino risultati falsi negativi e che il saggio non è stato in grado di quantificare con sicurezza il numero di copia per quei 14 campioni. Tuttavia, dal 75 al 100% dei quattro siti biologici si replica amplificato in ogni luogo di campionamento. Due dei tre spazi vuoti di campo erano negativi, mentre un campo vuoto mostrava amplificazione, sottolineando l'importanza della tecnica pulita nel campo.

Figura 1: Flusso di lavoro per la costruzione di database di sequenze di DNA mitocondriale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Allineamenti di sequenza per le specie di cozze del fiume Clinch con primir prospettici e sonde per il saggio Actinonaias ligamentina ND1. Primer in avanti in verde scuro, sonda in rosso e primer inverso in verde chiaro. Clicca qui per visualizzare una versione più grande di questa figura.
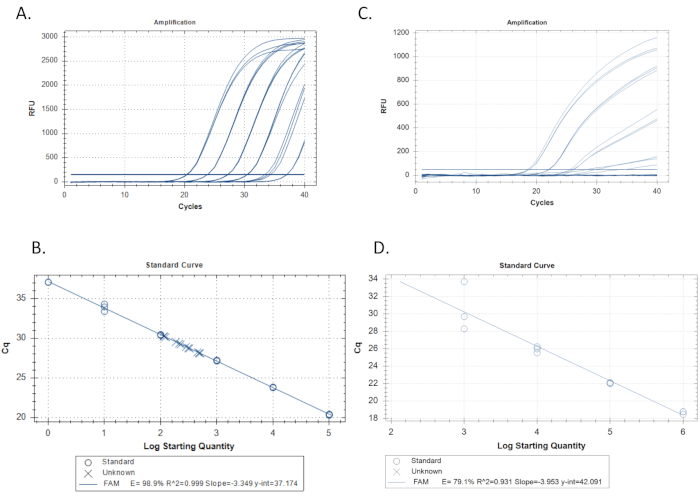
Figura 3: Esempi standard di curva e regressione lineare. A. Esempio di curva standard accettabile derivata dall'amplificazione di tre repliche ciascuna delle sei diluizioni standard. Una serie di diluizione standard di 10 volte con la più alta concentrazione dello standard a sinistra, con concentrazioni decrescenti che si spostano a destra. La linea orizzontale che attraversa tutte le tracce è la soglia per il ciclo alla quantificazione (Cq). Dove ogni traccia supera questa soglia è dove viene determinato il Cq. B. Regressione lineare effettuata dalle repliche standard della figura 3A. Le repliche delle diluizioni standard sono tracciate in cerchi e le incognite (campioni) vengono tracciate con x. L'efficienza è del 98,9%, r2 si avvicina all'1,0 e pendenza di -3.349. C. Esempio di una curva standard scadente derivata dall'amplificazione di tre repliche ciascuna delle sei diluizioni standard. D. Una regressione lineare che forma la curva standard per le repliche standard amplificata nell'esempio 3C. Si noti la scarsa efficienza e ivalori r 2. Si noti inoltre che solo 4 dei 6 standard sono stati amplificati. Se dopo le corse ripetute, la curva standard non migliora, il problema potrebbe essere con un set di primer / sonda scadente che non amplifica il DNA bersaglio come previsto nel qual caso, questo saggio non dovrebbe essere considerato. Clicca qui per visualizzare una versione più grande di questa figura.
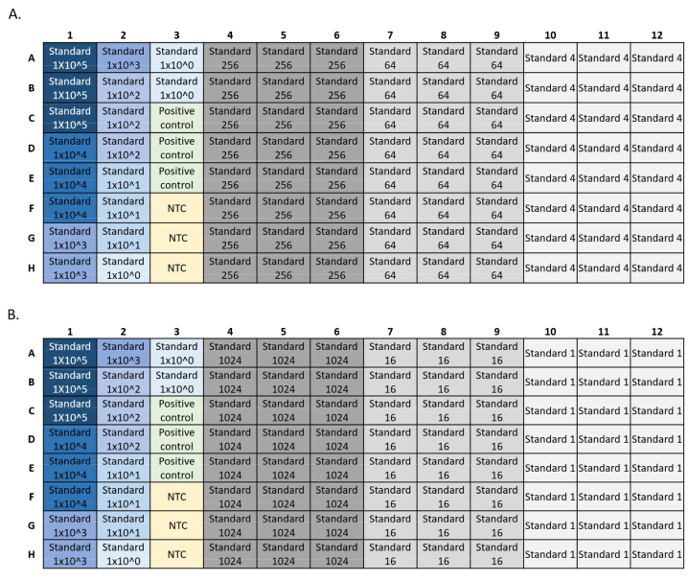
Figura 4: Esempi di configurazioni di lastre per corse qPCR standard LOD e LOQ. Gli standard utilizzati nella curva sono in blu, la concentrazione standard diminuisce dal buio all'azzurro. Controllo positivo del DNA in verde e nessun controllo del modello (NTC) in giallo. Concentrazioni standard sperimentali in grigio che mostrano 24 repliche per ogni diluizione standard. La serie di diluizione era placcata su due piastre (A, B), ognuna con una curva standard, un controllo positivo e NTC. Clicca qui per visualizzare una versione più grande di questa figura.
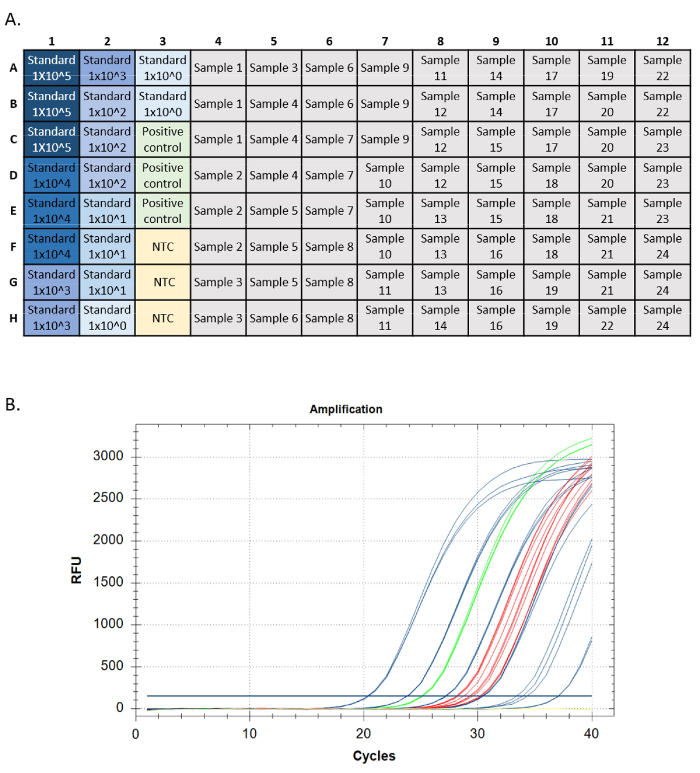
Figura 5: Tracce di configurazione e amplificazione della piastra da una corsa qPCR. A. Impostazione piastra, standard mostrati in blu, colore più scuro che indica la più alta la concentrazione dello standard. Controllo positivo del DNA in verde, nessun controllo modello in giallo (NTC), bersagli campione in grigio. B. Tracce di amplificazione da una corsa qPCR. Standard mostrati in blu, controllo positivo del DNA in verde, nessun controllo del modello in giallo e incognite in rosso. Clicca qui per visualizzare una versione più grande di questa figura.
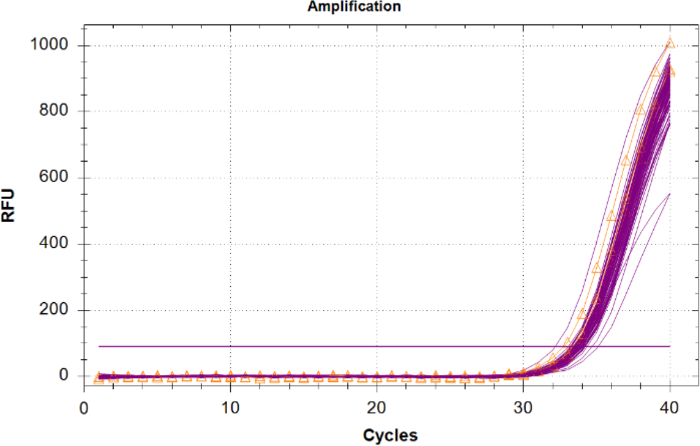
Figura 6: Tracce di amplificazione per il controllo positivo interno (IPC). Tracce IPC per tutti gli esempi sconosciuti in magenta e IPC dai controlli no template (NTC) visualizzati in arancione con triangoli. Clicca qui per visualizzare una versione più grande di questa figura.
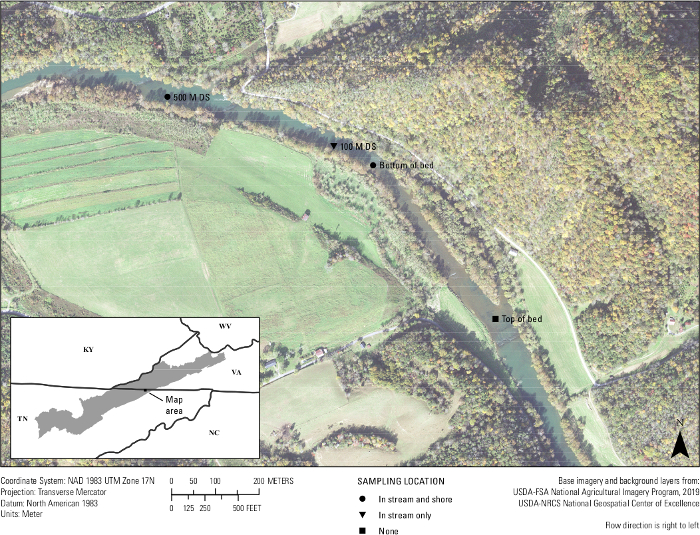
Figura 7: Mappa che mostra i siti di raccolta dell'eDNA di un letto di mitili nel fiume Clinch lungo il confine tra Virginia e Tennessee. I campioni sono stati raccolti a Wallens Bend nella parte inferiore del letto, 100 m a valle del letto e 500 m a valle del letto. I siti sono stati raccolti al centro del torrente (in torrente) o a circa 1 - 2 metri dal litorale (riva). Clicca qui per visualizzare una versione più grande di questa figura.
| componente | nome | Sequenza 5' – 3' | Etichetta fluorescente | |
| Primer avanti | A.lig.1-f | CCCTCATCACGTACCTCTTAATC | ||
| Primer inverso | A.lig.1-r | GGAATGCCCATAATTCCAACTTTA | ||
| sonda | Sonda A.lig.1 | TTCTTGAACGTAAAGCCCTCGGGT | Fam | |
Tabella 1: Il saggio actinonaias ligamentina qPCR progettato (A.lig.1) comprese le sequenze per i primer avanti e indietro e la sonda.
| specie | amplificato | Nel fiume Clinch |
| 1. Actinonaia ligamentina | sì | sì |
| 2. Actinonaia pettorale | No | sì |
| 3. Amblema plicata | No | sì |
| 4. Corbicula spp. | No | sì |
| 5. Cumberlandia monodonta | No | sì |
| 6. Ciclonaie tuberculata | No | sì |
| 7. Cyprogenia stegaria | No | sì |
| 8. Elliptio dilatata | No | sì |
| 9. Epioblasma brevidens | No | sì |
| 10. Epioblasma capsaeformis | No | sì |
| 11. Epioblasma florentina aureola | No | sì |
| 12. Epioblasma triquetra | No | sì |
| 13. Fusconaia cor | No | sì |
| 14. Fusconaia subrotunda | No | sì |
| 15. Lampsilis ovata | No | sì |
| 16. Lampsilis siliquoidea | No | No |
| 17. Lasmigona costata | No | sì |
| 18. Lemiox rimoso | No | sì |
| 19. Lexingtonia dolabelloides | No | sì |
| 20. Medionidus conradicus | No | sì |
| 21 Plethobasus cyphyus | No | sì |
| 22. Pleurobema plenum | No | sì |
| 23. Ptychobranchus fasciolaris | No | sì |
| 24. Ptychobranchus subtentus | No | sì |
| 25. Quadrula pustulosa | No | sì |
| 26. Strofito undulato | No | sì |
| 27. Iride Villosa | No | sì |
Tabella 2: Elenco delle specie utilizzate per la prova di specificità in vitro del saggio A.lig.1. Il saggio ha amplificato il DNA genomico del bersaglio (Actinonaias ligamentina) e non ha amplificato nessuna delle specie non bersaglio.
| componente | Sequenza 5'-3' | ||||
| Actinonaias ligementina standard | CCCTCATCACGTAC CTCTTAATCCTATTAGGTGTCGCATTTCACTCTTCTTGAACGTA | ||||
| AAGCCCTCGGGT ACTTTCAAATCCGAAAGGCCCAAATAAAGTTGGAATTATGGGCATTC | |||||
| CCCAACCATTAGCAGATGCTCTAAAGCTCTTCGTAAAAGAATGAGTAACACCAACCTCCT | |||||
| CAAACTACCTACCCTTCATCTTAACCCCAACCACTATGTTAATTTTAGCACTTAGACTTT | |||||
| GACAATTATTTCCATCCTTTATANTATCATCCCAAATANTTTTTGGTATGCTCCTATTCT | |||||
| TGTGTATCTCCTCCCTAGCTGTTTATACAACACTTATAACAGGCTGAGCCTCAAACTCCA | |||||
| AATATGCCCTTTTAGGAGCTATTCGAGCCATAGCCCAAACCATTTCTTATGAGGTTACAA | |||||
| TAAC | |||||
| Modello IPC (Hem-T) | CTACATAAGTAACACCTTCTCATGTCCAAAGCTCTCTGAGTGTCCCTCGAATCTCAGACGCT | ||||
| GTATGACAGTCTCCTTTCGTGTGTGAACATTCGGCT GCTATGTTCTCAAGGACTGCAC | |||||
Tabella 3: Sequenza (5'-3') dello standard Actinonaias ligamentina e modello IPC (Hem-T) utilizzato per questo saggio. La sequenza per i primer avanti e indietro è in grassetto e corsivo e quella della sonda è sottolineata.
| componente | nome | Sequenza 5' – 3' | Etichetta fluorescente | |
| Primer avanti | HemT-F | TCTGAGTGTCCCTCGAATCT | ||
| Primer inverso | HemT-R | GCAGTCCTTGAGAACATAGAGC | ||
| sonda | HemT-P | TGACAGTCTCCTTGTGTGTGAACATTCG | Ci5 | |
Tabella 4: Il saggio Internal Positive Control (IPC), comprese le sequenze per i primer avanti e indietro e la sonda.
| Volume per campione (μL) | componente |
| 10 | Mix di master ambientali |
| 1 | 20uM A. lig.1 F/R mix |
| 1 | 2.5uM A. lig.1 sonda |
| 1 | Mix di primer IPC da 5uM (HemT-F/ R) |
| 0.75 | Sonda IPC da 2,5u M (HemT-P) |
| 1.5 | 1 X 103 concentrazione del modello IPC |
| 2.75 | H20 |
| 2 | campione |
| 20 | Volume totale |
Tabella 5: La miscela PCR utilizzata per il saggio A.lig.1 multiplexato con il saggio IPC.
| passo | Temperatura (°C) | Ore | |
| 1 | Denaturazione iniziale | 95 | 10 min |
| 2 | denaturare | 95 | 15 secondi |
| 3 | ricottura | 60 | 1 min |
| 4 | Passare al passaggio 2, ripetere 39X |
Tabella 6: Condizioni di reazione per il saggio A.lig.1.
Discussione
Come per qualsiasi studio, definire la domanda da affrontare è il primo passo e la progettazione del saggio eDNA dipende dall'ambito dello studio26. Ad esempio, se l'obiettivo della ricerca o dell'indagine è quello di rilevare una o poche specie, un saggio mirato basato su sonde è il migliore. Se, tuttavia, l'obiettivo è valutare una suite più grande o un assemblaggio di specie, i test di decodifica metabarcodi ad alta produttività sono più adatti. Una volta determinato quale approccio adottare, si consiglia uno studio pilota che includa la progettazione, il test e l'ottimizzazione dei saggi24. Il design del saggio inizia con un elenco di specie come descritto in Figura 1. Questo elenco sarà la base per comprendere le prestazioni di un saggio in termini di specificità e l'intervallo geografico a cui potrebbe essere applicato6,10. È incoraggiato a progettare il saggio per una specifica area geografica, consentendo al progettista di testare meglio un saggio di reattività incrociata contro altre specie in quell'area, e di essere consapevole dei limiti che ciò ha nell'estendere un saggio ad altre aree in cui può verificarsi una specie bersaglio24. Una volta completato l'elenco, le sequenze possono essere scaricate da database genetici pubblici. Poiché queste basi di dati sono incomplete27, si dovrebbe sequenziare il maggior numero possibile di specie nell'elenco internamente per completare il database di riferimento locale delle sequenze che verranno utilizzate nella progettazione del saggio. Dare priorità alle specie strettamente correlate che si verificano, poiché queste sono le più probabili non-bersagli che si amplificano. Concentrarsi su tutte le specie all'interno dello stesso genere o famiglia della specie bersaglio è un buon punto di partenza. I confronti con specie strettamente correlate aiuteranno a identificare le regioni di sequenza uniche per le specie bersaglio. Questo può aiutare a informare come il saggio può funzionare in altri sistemi o luoghi. Le regioni mitocondriali sono la scelta abituale per lo sviluppo del saggio, perché più informazioni sulla sequenza di una più ampia varietà di specie sono disponibili nei geni mitocondriali che sono stati utilizzati nei progetti di codice a barre della vita e perché il DNA mitocondriale è presente a una concentrazione molto maggiore in copie / cellule rispetto al DNA nucleare24,28,29. Più regioni geniche dovrebbero essere valutate per un ulteriore sviluppo del saggio, poiché la copertura delle sequenze varia tra i taxa nelle banche dati del repository genetico. Dopo aver creato questo database locale di sequenze di riferimento, viene utilizzata una combinazione di visualizzazione manuale dei dati di sequenza allineati e dei programmi software per computer per progettare i test di primer / probe. Non si dovrebbe fare affidamento rigorosamente sul software per determinare quali test testare. È importante verificare visivamente gli allineamenti in cui i primer e le sonde si trovano sui bersagli e sui non bersagli per ottenere una migliore comprensione di come potrebbero agire in una PCR. Infine lo screening e l'ottimizzazione del saggio comprendono tre livelli (in silico, in vitro e in situ)6,7,24,25. Nella progettazione e nel test del silico sono importanti per produrre un breve elenco di test con buone possibilità di successo, ma i test empirici (in vitro) sono cruciali per selezionare il saggio con le migliori prestazioni effettive. L'ottimizzazione in vitro e il test dei saggi includono la misurazione dell'efficienza di reazione e la definizione della sensibilità e della specificità del saggio. I limiti di rilevamento e quantificazione sono due parametri spesso trascurati nello sviluppo del saggio ma importanti per l'interpretazione dei dati. Eseguendo più repliche delle curve standard per un saggio, LOD e LOQ possono essere facilmente misurati1,5,30. Pochi studi discutono i risultati rispetto al LOD o ALQ del saggio, ma Sengupta et al.31. Anche i controlli positivi interni devono essere multiplexati nel saggio progettato. Senza testare l'inibizione della PCR nei campioni, possono verificarsi falsi negativi24,32. Proponiamo l'uso di un saggio IPC multiplexato con il saggio target come metodo più semplice per i test di inibizione PCR23. Infine, è necessario testare in situ il saggio da campioni raccolti sul campo e in laboratorio per garantire che l'amplificazione del bersaglio avvenga in campioni ambientali24.
Esistono limitazioni per l'uso di test qPCR specifici per specie e basati su sonda con campioni di eDNA. Ad esempio, la progettazione di più test per i test può essere limitata dalla disponibilità della sequenza e può essere necessario un compromesso su aspetti delle prestazioni del saggio. Queste scelte devono essere guidate dagli obiettivi dello studio e devono essere riportate con i risultati26. Ad esempio, se l'obiettivo è il rilevamento di una specie rara e sono attesi pochi aspetti positivi, un saggio con specificità imperfetta (cioè l'amplificazione di specie non bersaglio) potrebbe essere utilizzato se tutti i rilevamenti saranno verificati sequenziando. Se l'obiettivo è monitorare l'intervallo geografico di una specie e non sono necessari dati sulla concentrazione di eDNA, è possibile utilizzare un test con efficienza imperfetta e i dati riportati solo come rilevamento percentuale. Inoltre, a meno che tutti i potenziali conspecifici non siano testati in laboratorio, il che è raramente possibile, non si può conoscere con assoluta certezza la vera specificità di un saggio. Ad esempio, il saggio è stato progettato e testato contro diverse specie di cozze d'acqua dolce nel fiume Clinch. Per utilizzare questo saggio in un sistema fluviale diverso, dovremmo testarlo contro una suite di specie nella nuova posizione. Anche la variazione genetica all'interno della specie o della popolazione che non viene testata durante lo sviluppo del saggio potrebbe influire sulla specificità. Infine, anche se è stato verificato che un saggio ha elevate prestazioni tecniche; le condizioni cambiano quando si lavora sul campo. Condizioni non correlate al test come il flusso d'acqua, il pH e il comportamento degli animali possono cambiare la rilevabilità dell'eDNA come può utilizzare diversi protocolli di raccolta ed estrazione dell'eDNA. L'uso di saggi ottimizzati e ben descritti aiuterà a facilitare la comprensione dell'influenza che tali parametri hanno sul rilevamento eDNA.
Il campo dell'eDNA sta maturando oltre lo stadio dell'analisi esplorativa per aumentare la standardizzazione dei metodi e delle tecniche. Questi sviluppi miglioreranno la nostra comprensione delle tecniche, delle abilità e dei limiti dell'eDNA. Il processo di ottimizzazione descritto sopra migliora la sensibilità, la specificità e la riproducibilità di un saggio. L'obiettivo finale di questo perfezionamento e standardizzazione dei metodi eDNA è migliorare le capacità dei ricercatori di effettuare deduzioni basate sui dati eDNA e aumentare la fiducia degli utenti finali e dei titolari di palo nei risultati.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi. Gli sponsor del finanziamento non hanno avuto alcun ruolo nella progettazione dello studio; nella raccolta, analisi o interpretazione dei dati; nella scrittura del manoscritto; o nella decisione di pubblicare i risultati.
Riconoscimenti
Ringraziamo Alvi Wadud e Trudi Frost che hanno contribuito allo sviluppo e ai test dei primer. I finanziamenti per la progettazione del saggio riportati in questo studio sono stati forniti dal Dipartimento della Difesa Strategic Environmental Research and Development Program (RC19-1156). Qualsiasi uso di nomi commerciali, di prodotti o di società è solo a scopo descrittivo e non implica l'approvazione da parte del governo degli Stati Uniti. I dati generati durante questo studio sono disponibili come scheda di rilascio dei dati USGS https://doi.org/10.5066/P9BIGOS5.
Materiali
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
Riferimenti
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon