Method Article
ÇevreSEL DNA Uygulamaları için Türlere Özgü Nicel PCR Testlerinin Geliştirilmesi ve Testi
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Çevresel DNA tahlilleri, alan verilerinin toplanmasına başlamadan önce titiz bir tasarım, test, optimizasyon ve doğrulama gerektirir. Burada, çevresel örneklerden hedef tür DNA'sının tespiti ve nicelleştirilmesi için türe özgü, prob tabanlı bir qPCR testi tasarlamanın her adımında kullanıcıları götürecek bir protokol sunuyoruz.
Özet
Balıkçılık ve doğal hayatı koruma yönetimine yardımcı olmak için tür varlığını tespit etmek ve izlemek için yeni, invaziv olmayan yöntemler geliştirilmektedir. Makrobiyotayı tespit etmek için çevresel DNA (eDNA) örneklerinin kullanılması, hızla popüler hale gelen ve ulusal yönetim programlarında uygulanan bu tür yöntemlerden biridir. Burada prob bazlı nicel PCR (qPCR) uygulamaları için türlere özgü hedefli tahlillerin geliştirilmesine odaklanıyoruz. Prob tabanlı qPCR kullanmak, tek başına astarlarla mümkün olandan daha fazla özgüllük sunar. Ayrıca, bir numunedeki DNA miktarını ölçme yeteneği, eDNA'nın ekolojisini ve alandaki eDNA algılama kalıplarının yorumlanmasını anlamamızda yararlı olabilir. Hedef türlerin çevresel bir numuneden tespit edilmesi hassasiyetini ve özgüllüğünü sağlamak için bu tahlillerin geliştirilmesinde ve testinde dikkatli bir değerlendirmeye ihtiyaç vardır. Bu protokolde, hedef bir türün tespiti için prob tabanlı tahlillerin tasarlanması ve test edilmesi için gereken adımları tespit edeceğiz; sıra veritabanlarının oluşturulması, tahlil tasarımı, tahlil seçimi ve optimizasyonu, test tahlil performansı ve alan doğrulaması dahil. Bu adımların ardından, güvenle kullanılabilecek verimli, hassas ve spesifik bir test elde edilmesine yardımcı olacaktır. Bu süreci, ABD'nin Clinch Nehri'nde bulunan tatlı su midyesi türü olan mucket popülasyonları (Actinonaias ligamentina) popülasyonlarıiçin tasarlanmış tahlillerimizle gösteriyoruz.
Giriş
Araştırmacılar ve yöneticiler, tür tespiti için çevresel DNA testlerinin kullanımıyla giderek daha fazla ilgilenmeye başladı. Otuz yıldır, nicel veya gerçek zamanlı PCR (qPCR / rtPCR) nükleik asitlerin diziye özgü tespiti ve nicelleştirilmesi için çok sayıda alanda 1,2. EDNA araştırmasının nispeten yeni alanında, bu tahlillerin, eDNA örneğinin hacmi veya ağırlığı başına hedef DNA kopyalarının ölçülmesi için standart bir eğri ile kullanılması artık rutin bir uygulama haline gelmiştir. Mitokondriyal DNA dizileri genellikle eDNA tahlillerinde hedeflenir, çünkü mitokondriyal genom hücre başına binlerce kopyada bulunur, ancak nükleer DNA veya RNA dizileri için tahliller de mümkündür. eDNA örnekleri için yayınlanan testlerin performansta her zaman eşit olmadığını anlamak çok önemlidir. Bir testin yalnızca bir hedef türün DNA'sını (yani özgüllüğü) tespit etmedeki güvenilirliği ve düşük miktarda hedef DNA'nın (yani hassasiyet) tespit edilmesi, testin nasıl tasarlandığı, seçildiği, optimize edildiği ve test edildiği konusundaki farklılıklar nedeniyle önemli ölçüde değişebilir. Test performansının nicel önlemlerinin rapor edildiği daha önce büyük ölçüde göz ardı edilmiştir, ancak son zamanlarda tahlil gelişiminde şeffaflığı artırmaya yönelik standartlar ortaya çıkıyor 3 ,4,5,6,7,8.
EDNA anket sonuçlarının eDNA anket sonuçlarının incelenmesi ve yorumlanmasında tahlil performans yardımcılarının optimizasyonu ve raporlanması. Hedef olmayan tür DNA'sı ile çapraz reaksiyon gösteren tahliller yanlış pozitif tespitlere yol açabilirken, hassasiyeti zayıf olan tahliller örnekte mevcut olsa bile hedef tür DNA'sını tespit edemeyebilir (yanlış negatifler). Test hassasiyeti ve seçicilik anlayışı, nadir türleri tespit etmek için gereken örnekleme çabasını bilgilendirmeye yardımcı olacaktır. eDNA'da birçok doğal varyasyon kaynağı olduğundan, çalışmalar kontrol edilebilir varyasyon kaynaklarını mümkün olduğunca sınırlamalıdır, eDNA testini tamamen optimize etmek ve karakterize etmek de dahil olmak üzere3.
Bir tahlilin özgüllüğünü veya hassasiyetini doğrudan etkileyen koşullar, tahlilin performansını değiştirecektir. Bu, farklı laboratuvar koşullarında (örneğin, farklı reaktifler, kullanıcılar, makineler vb.) oluşabilir. Bu nedenle, yeni koşullar altında bir tahlil uygulanırken bu protokol yeniden ele alınmalıdır. Literatürde iyi karakterize edilen tahliller bile, yeni bir laboratuvar tarafından benimsendiğinde veya farklı reaktifler (örneğin, ana karışım çözeltisi)5,9. Test farklı bir coğrafi bölgeye uygulandığında test özgüllüğü değişebilir, çünkü test, testin test edilmemiş olduğu hedef olmayan türleri içerebilecek yeni bir biyotik topluluktan alınan örneklere uygulanmaktadır ve hedef türlerde genetik varyasyon meydana gelebilir. Yine, test yeni bir konumda kullanıldığında yeniden değerlendirilmelidir. Saha koşulları laboratuvar koşullarından farklıdır, çünkü sahada PCR inhibitörlerinin numunelerde bulunma olasılığı daha yüksektir. PCR inhibitörleri amplifikasyon reaksiyonu doğrudan etkiler ve böylece tahlil performansını etkiler. Bu nedenle, bir eDNA tahlili geliştirirken dahili bir pozitif kontrol gereklidir.
Son olarak, alandaki çevresel koşullar, hedef türün DNA moleküllerini ve DNA bozulması, taşınması ve tutulması yoluyla yakalanmasını etkileyebilir. Ayrıca, DNA toplama ve çıkarma için farklı protokoller, DNA'yı tutma verimliliklerine ve yeteneklerine göre değişir. Bununla birlikte, bu süreçlerin eDNA'nın tespit edilebilirliğini etkilediğini, ancak moleküler bir tahlil performansını etkilemediğini belirtmek önemlidir. Bu nedenle, alan örneklerinde hedef türlerden DNA'nın tespit edilebilirliği, hem qPCR testinin teknik performansının hem de saha koşullarının ve toplama, depolama ve çıkarma protokollerinin bir işlevidir. İyi karakterize edilmiş ve yüksek performanslı bir tahlil kullanırken, kullanıcılar tahlil yeteneklerinden emin hissedebilirler; araştırmacıların artık eDNA algılamasını etkileyen dış tahlil faktörlerini (yani çevresel değişkenler, yakalama veya çıkarma protokollerindeki farklılıklar) anlamaya odaklanmalarını sağlar.
Burada özellikle titiz tasarım ve optimizasyon yoluyla teknik performansı test etmeye odaklanıyoruz. Protokolü, ABD'nin Clinch Nehri'nde örnek alınan sudan bir tatlı su midyesi olan mucket'in(Actinonaias ligamentina)tespiti için geliştirilen sonda bazlı bir tahlil kullanarak gösteriyoruz. Son zamanlarda Thalinger ve arkadaşları (2020), hedeflenen eDNA testlerinin doğrulanması için yönergeler sundu. Protokolümüzü takip eden tahlil tasarımı Thalinger ve arkadaşlarının seviye 4'üne bir test artı seviye 56'yadoğru ek bir adım getirecektir. Bu noktada bir tahlil teknik performansı optimize edilecek, laboratuvar ve saha uygulamalarında düzenli kullanıma hazır hale gelecektir. Tahlillerin laboratuvar, mezokozm ve saha deneylerinde daha fazla kullanılması, eDNA tespiti ve tespit edilebilirliği etkileyen faktörler, seviye 5 doğrulama6için son adımlar ile ilgili soruları ele alabilir.
Protokol
1. Hedef ve hedef olmayan ilgi türlerinden mitokondriyal DNA dizilerinin bir dizi veritabanının üretilmesi
- Ele alınmak üzere soruyu, hedefleri ve sistemi tanımlayın. eDNA algılaması için hedef türleri tanımlayın. Tahlillerin kullanılacağı coğrafi sistemi tanımlayın. Hedef türler, aynı takson içindeki sympatrik (birlikte oluşan) türler (genellikle düzen veya aile düzeyi) ve hedefle aynı coğrafi konumda olmayabilecek allopatrik türler de dahil olmak üzere ilgi çekici türlerin bir listesini yapın (Şekil 1).
NOT: Burada A. ligamentina türünün Clinch Nehri popülasyonları hedeflenmiştir. - Adım 1'den listedeki türler için birden fazla gen bölgesinden dizileri arayın ve indirin. NCBI (Ulusal Biyoteknoloji Bilgi Merkezi), BOLD (Barkod of Life Database), EMBL (Avrupa Moleküler Biyoloji Laboratuvarı) ve DDBJ (Japonya DNA Veri Bankası) gibi sıra veritabanları kullanılabilir. NCBI, EMBL ve DDBJ sıra bilgilerini paylaşır.
- NCBI'nin Nükleotid Veritabanını kullanarak, hedef organizmayı (örneğin, Actinonaias ligamentina)ve gen bölgesini (örneğin, sitokrom c oksidaz I (COI) veya NADH-dehidrogenaz 1 (ND1) arayın; Örnek arama dizesi: Actinonaias ligamentina VE ND1)
- Ardından, belirtimlerle eşleşen tüm sıraları seçin ve Gönder'i seçin. Kaydı tamamla, Dosya ve indirme biçimini GenBank veya FASTA olarak seçin ve dosya oluştur 'u seçin. Bu sıralar artık bilgisayara kaydedilir.
- Adım 1'de tanımlanan listedeki tüm türler için bu adımları yineleyin. Her gen bölgesi için dizileri ayrı bir dosyada tutun, çünkü bunlar ayrı ayrı analiz edilecektir.
- Adım 1'de tanımlanan hedef türler için ilgili tüm dizileri (veya dizilerin büyük, temsili bir oranını) indirin. Mümkünse coğrafi varyantları ekleyin.
- Adım 1'de tanımlanan aynı taksonomik grubun ilgili ve sympatrik olmayan türleri için arama ve indirme sıralarını tekrarlayın (örneğin, hedef tür mucket ise (A. ligamentina) Family Unionidae'deki diğer tüm tatlı su midye türleri için ilgi çekici sistemde meydana gelen dizileri indir).
- Adım 1.1'de listelenen yakından ilişkili ancak alopatrik (coğrafi olarak ayrı) türler için aramayı ve indirmeyi tekrarlayın.
NOT: Tüm türler (hedefler ve hedef olmayanlar) genel veritabanlarında bulunmaz. Şirket içinde ilgi gören türlerin taksonomik olarak doğrulanmış örneklerini yükselterek ve sıralayarak yerel referans veritabanını artırın. Türler içinde genetik çeşitliliği yüksek bir türle çalışıyorsanız veya coğrafi varyantların beklenebilir olduğu coğrafi olarak geniş bir alanda çalışıyorsanız, aralıktaki dizileri toplayın.
2. Tahlil tasarımı
- Çeşitli genetik dizi düzenleme ve biyoinformatik programlarda bulunabilen hizalama yazılımını kullanarak her gen bölgesinden dizileri ayrı ayrı hizalayın. Bu hizalamayı farklı gen bölgelerinin her biri için yapın.
- Örneğin, Geneious Prime yazılımını (https://www.geneious.com) kullanarak indirilen sıra dosyalarını programa aktarın.
- Her gen bölgesi için ayrı klasörler oluşturun.
- Bir gen bölgesinden diziler içeren bir klasörde, tüm dizileri seçin.
- Seçili dizilerin nükleotid hizalamasını oluşturmak için Çoklu hizalama aracını kullanın. Geneious veya MUSCLE hizalamalarını kullanarak hizalama türü için çeşitli seçenekler olabilir ve varsayılan parametreler iyi çalışır.
- Hizalanmış sıra verilerinin görselleştirilmesi yoluyla tahlil tasarımı için umut verici bölgeler seçin. İlgi gören türler için çok fazla sıra verisi olan, türler arasında oldukça farklı olan ve türler içinde düşük varyasyon gösteren bir bölge iyi bir adaydır. Bu, tasarlanan astarların ve probların hedefi hedef olmayan türlerden ayırt edebilme olasılığını artırırken, aynı zamanda spesifik olmayan varyantların testle birlikte artmasını sağlayacaktır.
- Test astarları ve prob tasarımı.
- qPCR tahlil tasarım yazılımını kullanın ve talimatları izleyin. IDT'nin 5 set qPCR tahlilleri tasarlamak için primerquest aracı (https://www.idtdna.com/) burada kullanıldı.
- 2.2. adımda seçilen sırayı Sıra giriş kutusuna yapıştırın. Hizalama boşluk oluşturduysa, bunları diziden silin.
- Tasarımınızı Seçin seçeneğinden qPCR 2 Primer + Probe'u seçin.
- Önerilen tahlilleri indirin.
- İlk tahlilin ileri astarından dizileri kopyalayın ve 2.1.4 adımında oluşturulan hizalamada bu astar sırasını arayın. Geneious Prime kullanıyorsanız, astar bölgesini hizalamaya eklemek için Açıklama ve Tahmin aracını kullanın. Bunu tüm astar ve prob kombinasyonları için yapın (Şekil 2).
- Hizalamanın bu bölgelerini hedef türler içinde ve birlikte oluşan türler içinde varyasyon açısından inceleyin.
- İntraspesifik genetik varyasyon varsa, astarların ve probun bu bölgelere girmediği tahlilleri arayın.
- Hedef olmayan türlerin amplifikasyonu önlemek için hedef olmayan türlerle uyumsuzluklar arayın. Daha fazla doğrulama için en uyumsuzlara sahip tahlilleri seçin. Currier ve ark. (2018), hedef olmayan tüm türlerle en az iki uyumsuzluk olan üç bölgeden en az ikisini (iki astar veya bir astar ve bir prob) seçmeyi önerir. Ancak, probdaki uyuşmazlıkların özgüllük10'adaha az katkıda bulunduğunu unutmayın.
NOT: Her astarın 3' ucunun 3 baz çifti içindeki farklılıklar, astarların 5' ucundaki farklılıklardan daha iyi özgüllüğü artırır10.
- Tahlil tasarımında aşağıdaki önemli parametreleri göz önünde bulundurun.
- Astarların ve probun erime ve tavlama sıcaklıklarını belirleyin. İdeal olarak astarların erime sıcaklığı (Tm) 60-64 ° C arasında ve birbirine 2 ° C içinde olmalı ve probun Tm'si astarların Tm'sinden 6-8 derece daha yüksek olmalıdır. qPCR reaksiyonunun tavlama sıcaklığını (Ta) erime sıcaklığının 5 °C altına, yaklaşık 55-60 °C11ayarlayın.
- GC içeriğini inceleyin. %35 - 65 GC içeriği arasında seçim yapın ve 4 veya daha fazla ardışık Gs olan bölgelerden kaçının. Astarın 3' ucunun (GC kelepçesi) 5 son tabanında 1 veya 2 G veya Cs olması, astarın daha güçlü bir bağ yapmasına yardımcı olacağı için özgüllüğü artırabilir12.
- Saç tokası ve daha soluk yapılar arayın. Bir oligonükleotid analiz programı kullanarak tahmin edilen saç tokası yapıları ve dimerler için astarları ve probu test edin (örneğin, OligoAnalyzer -IDT13; OligoCalculator14). Bu yapılar hedef dışı amplifikasyona ve daha düşük verimliliğe neden olabilir. Bu yapıları oluşturmuş olduğu tahmin edilen tahlillerden kaçının.
- Astar uzunluğunu belirleyin. 18-25 taban arasında astarlar ve 20-25 taban arasında prob uzunluğu hedefleyin. Daha uzun astarlar ve problar daha düşük amplifikasyon verimliliğine sahip olabilir.
- Amplicon uzunluğunu belirleyin. Yaklaşık 100 ila 250 baz çift arasında olmalıdır. Bu aralık genellikle yüksek PCR verimliliği için yeterince kısadır, ancak Sanger dizilimi4,15tarafından doğrulama kolaylığı için yeterince uzundur.
- Sondalar tasarlay. Probların 5' ucunda bir G tabanı olmadığından emin olun, çünkü yeşil ve sarı boyalardan gelen sinyali sönümleyebilir11. IDT 3IABkFQ ve ZEN söndürücüler ve FAM veya HEX floroforları ile çift söndürülen problar tasarladık.
NOT: MGB problarını belirleyin: TaqMan MGB (küçük oluk bağlayıcı) probları genellikle eDNA çalışmaları için kullanılır. Ancak, bu problar çok kısa olduğundan, 2 veya 3 baz çifti uyuşmazlığı10ile bile hedef olmayanlara bağlanabilirler. - Probu belirleyin Tm. Probun erime sıcaklığı astarlardan 6-8°C daha yüksek olmalıdır. Daha düşük sıcaklıklar probun bağlama başarısını azaltır.
- Prob uzunluğunu ve konumunu belirleyin. Prob 20 ila 25 bp uzunluğunda olmalı ve ideal olarak üst üste binmeden aynı iplikçik üzerindeki astar bağlama bölgesine yakın bir yerde bulunmalıdır.
3. Tahlil taraması ve optimizasyonu
- Silico tahlil geliştirme ve test. Astar prob setlerini sipariş etmeden önce, silikoda astar amplifikasyonu test ederek özgüllüğü (potansiyel hedef dışı amplifikasyon)değerlendirin.
- NCBI nt/nr veritabanındaki olası hedef olmayanları tanımlayabilen NCBI'nin Primer-Blast16 veya benzeri programları aracılığıyla astarları test edin. Primer-Blast yapıştırıcı astarlarını kullanıyorsanız Primer parametreleri altında kendi astar kutumu kullanın. Astar Çifti Özgüllük Denetimi Parametreleri seçeneklerinde, Veritabanı olarak nr'yi seçin ve organizma kutusuna ilgi çekici organizmanın sırasını (örneğin, "Unionida" veya "Unionoida") yazın.
- Hizalanmış sıra verilerinde astar/prob kümelerini görsel olarak değerlendirmeye devam edin.
- Silikoda astarları ve probları aynı anda değerlendirmek için ileri astarın bir metin dizesini, 12 N'leri, probu, 12 N'leri ve ters astarın ters tamamlayıcısını oluşturun. Prob sırası astarlardan birinin 12 baz çifti içindeyse, astar ve prob arasındaki taban çiftlerinin sayısına karşılık gelen N sayısını kullanın.
- NR veritabanı17'yiaramak için NCBI'nin Nucleotide Blast aramasını (Blastn) kullanın. Birkaç uyuşmazlık olan hedef olmayan türleri aramak için Taksonomi sekmesini kullanın; bunlar tahlil optimizasyonu sırasında laboratuvarda test edilmelidir.
NOT: Siliko testinde spesifik olmayan tahliller ekarte edilmeye yardımcı olur, ancak potansiyel olarak spesifik tahliller ampirik olarak test edilmelidir (in vitro), çünkü tüm türlerin genetik veritabanlarında dizileri ve astarı ve probları yazılım tarafından olası görülmese bile hedef dışılara bağlanabilir.
- Laboratuvarda test etmek için üç ila beş astar/prob kombinasyonu seçin.
- Amplicon dizilimi için astarlar, problar ve sentetik DNA standardının yanı sıra ek M13 kuyruklu astarlar sipariş edin.
- Oligo yapan bir şirketten sentetik oligonükleotid astarlar ve problar sipariş edin. Problar floresan boya ve quencher ile etiketlenmiştir. Çoklayıcı olması gereken tahliller için farklı floroforlar seçilmelidir. QPCR cihazınızı, cihazın algılayabileceği floroforların listesi için kontrol edin.
- M13 Forward (-20) dizisi GTA AGA CGA CGG CCA GT'yi ileri astarın 5' ucuna ve M13 Reverse (-27) dizisi CAG GAA ACA GCT ATG AC'yi ters astarın 5' ucuna ekleyerek Sanger dizilimi ile qPCR algılamalarının doğrulanması için M13 kuyruklu astarları tasarlayın ve sipariş edin.
- Sentetik DNA standardı, kopyalarda/μL'de bilinen bir konsantrasyonda hedef diziyi (astar bölgeleri dahil) içerir. Astarları ve probu üreten aynı şirketten sentetik standardı alın. Yeniden depolama ve depolama için üretici önerilerine uyun. Hidrolizi ve yüzeylere bağlanmayı azaltmak için düşük muhafazalı plastik eşyalar kullanarak te tampondaki standartları bir tRNA taşıyıcı ile seyreltin.
NOT: Standart eğri iyi performans göstermezse (zayıf PCR verimliliği, bkz. adım 3.4.2), su veya Tris-HCl'deki standardı yeniden askıya almaya çalışın. - Nuclease içermeyen su, Tris-HCl veya TE tamponundaki astarları ve probları tahlil kullanımı için uygun konsantrasyonlarda askıya alın. Genellikle, optimize edilmiş son test konsantrasyonuna ulaşmak için ana karışımda çalışma stoklarını 20 kat seyreltin. Askıya alınan oligoları kullanılmadığında sabit -20 °C'de saklayın.
- In vitro (laboratuvarda) test optimizasyonu ve testi. Verim düşük, birlikte oluşan türlerle çapraz tepki veren veya hassasiyeti zayıf olan tahlilleri reddedin18. Test geliştirme sırasında ve gerçek örnekleri çalıştırırken dahili pozitif kontrol (IPC) kullanımını ekleyin.
- İlk olarak, test için en uygun sıcaklık ve astar / prob konsantrasyon değerlerini bulun. Bu parametreler PCR verimliliği (Adım 3.4.2), çapraz reaktivite (Adım 3.4.3) ve duyarlılık (Adım 3.4.4) için optimize edildikten sonra, testi çoklanmış bir IPC (Adım 3.4.5) ile test etmeye devam edin.
- Tahmin edilen ortalama astar Tm'nin 5° C altında ortalanmış bir PCR sıcaklık gradyanı kullanarak astar ve problar için optimum tavlama sıcaklığını (Ta) test edin.
- Optimum astar ve prob konsantrasyonlarını test edin. Tipik olarak, 200 nM, 400 nM ve 800 nM astar konsantrasyonları ve 75 nM, 125 nM ve 200 nM prob konsantrasyonları test edilir.
- Standart bir eğri oluşturun ve verimliliği ve doğrusal aralığı belirleyin. Hedef sırayı içeren sentetik bir DNA standardının en az altı adet 10 kat seyreltmesini yaklaşık 10 0 kopya/reaksiyonda10 5 kopya/reaksiyona test edin (Şekil 3A).
- y eksenindeki her standardın Cq değerini (nicelikte döngü eşiği) ve x eksenindeki kopyalarda / reaksiyonda ilk standart konsantrasyonun günlük tabanı 10'u çizmek için qPCR yazılımını kullanın. qPCR yazılımı otomatik olarak doğrusal bir regresyon çalıştırmalıdır (Şekil 3B).
- Verimliliği regresyon eğiminden hesaplayın, E = -1 + 10(-1/eğim). Örneğin, eğim -3,4 ise, E= -1 + 10(0,29) = 0,97 veya%97' dir. Ayrıca, standardın eğriye ne kadar iyi uyduğunu gösteren r2 değerlerini de kontrol edin. qPCR yazılımı bunu da otomatik olarak hesaplamalıdır (Şekil 3B). %100 (%±10) verimlilik değerlerini hedefleyin ve r2 değerleri ≥0,989,15,19,20,21,22.
- Standart eğriyi önyargı, yani tutarlı bir yönde gerilemeden sapmalar veya verimlilik ve r 2 değerleri(Şekil 3C ve3D)ile ölçülen düşük standart eğri performansı için görsel olarak inceleyin.
- Özgüllük: Yanlış pozitif olasılığını azaltmak için hedef olmayan türlerle çapraz reaktiviteyi değerlendirin. eDNA algılamalarının maliyetli yönetim kararlarına neden olabileceği durumlarda, amplicon dizileme ile pozitif algılamaları doğrulayın.
- Hedef olmayanlar: Testi, ilgili türlerin ve coğrafi olarak birlikte bulunan türlerin taksonomik olarak doğrulanmış örneklerinin genomik DNA ekstraksiyonlarına karşı çalıştırın; en yüksek önceliğe sahip, yakından ilişkili, birlikte oluşan türlere karşı test etmek. Hem hedef hem de hedef olmayan örnekler için benzer toplam DNA konsantrasyonları kullanın. Seçilen konsantrasyon, standart eğrinin doğrusal aralığının ortasına yakın hedef tür örneklerinden amplifikasyon vermelidir. Amplifikasyon sadece hedef türlerle gözlenmelidir.
- Hedef dışı amplifikasyon gözlenirse, ürünü temizleyin ve kimliğini doğrulamak için sıralayın. Hedef olmayan türlerin doku örneklerinde hedef türlerden kaynaklanan kirlenmeyi gözlemlemek nadir değildir, bu nedenle bu aşamadaki tüm amplifikasyonlar sıralama ile doğrulanmalıdır. M13 kuyruklu astarları kullanarak temizlenmiş ampliconları özgüllük testlerinden yeniden örnekle ve M13 astarlarla sıralayın.
- PCR sonrası laboratuvarda, sıralanacak qPCR ürünlerini taze tüplere aktarın. Artık astarları ve reaksiyon bileşenlerini temizleme kiti (örneğin, MinElute PCR Arıtma Kiti) ile çıkarın.
- Elutionların 1:100 seyreltilmesini yapın ve M13 kuyruklu astarlar ve yüksek kaliteli polimeraz (örneğin, Phusion Yüksek Kaliteli DNA Polimeraz) ile 50 μL PCR reaksiyonunda 30 döngü için her birinin 1 μL'sini yükseltin.
- Beklenen boyutta tek bir bant olup olmadığını kontrol etmek için her reaksiyonun 10 μL'sini% 1 agarose jel üzerinde çalıştırın. Bant gözlenmezse, döngü sayısını veya örnek miktarını artırın. Birden fazla bant gözlenirse, jel beklenen boyuttaki bandı arındırır.
- Yukarıdaki gibi bir temizleme kiti ile artık astarları ve reaksiyon bileşenlerini çıkarın ve elutionların DNA konsantrasyonlarını ölçün.
- Sıralama tesisinin talimatlarına göre M13 astarları ile sıralama reaksiyonları ayarlayın.
NOT: qPCR laboratuvarında asla güçlendirilmiş numuneler açmayın. PCR sonrası numunelere adanmış bir laboratuvarda sıralama için numuneler hazırlayın.
- Duyarlılık: Duyarlılık, yanlış negatiflerin veya hedef tür DNA'sının mevcut olduğunda tespit edilmemesi olasılığını etkiler. Her test için algılama sınırını (LOD) ve nicelik sınırını (LOQ) değerlendirin. Son olarak, örneklerin PCR inhibisyonunu değerlendirmek için bir iç pozitif kontrol (IPC) ekleyin. Multiplex ve iki testin birbirine karışmamasını sağlamak için tasarlanan test ile bu IPC testini test edin.
- LOD: Sentetik DNA standardının altı adet 4 katlı seri seyreltmesini, standart seyreltme başına 8-24 çoğaltma ile yapın (Şekil 4). %95 algılama ile en düşük başlangıç konsantrasyonu hesaplayın. LOD ve LOQ çizimleri bir LOD / LOQ hesap makinesi R komut dosyası5ile oluşturulabilir.
NOT: LOD'un altındaki veriler sansürlenmemelidir. PCR'nin özgüllüğü nedeniyle, gerçek pozitifler için alt sınır yoktur. LOD, yanlış negatiflerin oluşmasının beklenebileceği en yüksek konsantrasyondur. - LOQ: Aynı seyreltme serisinden, %35'in altında bir değişim katsayısı (CV) ile ölçülebilir en düşük ilk DNA standart konsantrasyonunu hesaplayın.
NOT: LOD ve LOQ kopya/reaksiyon olarak bildirilmelidir. Doğrulanmış bir test kullanırken ve alan örnekleri LOQ'nun altında yükseltilirken, sonuçlar eDNA konsantrasyonları yerine % algılamaları olarak bildirilmelidir, çünkü tam konsantrasyon güvenle ölçülemez5.
- LOD: Sentetik DNA standardının altı adet 4 katlı seri seyreltmesini, standart seyreltme başına 8-24 çoğaltma ile yapın (Şekil 4). %95 algılama ile en düşük başlangıç konsantrasyonu hesaplayın. LOD ve LOQ çizimleri bir LOD / LOQ hesap makinesi R komut dosyası5ile oluşturulabilir.
- PCR inhibisyonunu test etmek için dahili pozitif kontrol (IPC) kullanın. İnhibisyon duyarlılıkta azalmaya ve yanlış negatiflere yol açabilir. IPC testinin hedef testle çoklanmış olma yeteneğini test edin.
- Bir IPC tahlili, hedef testten farklı bir muhabir boyasına sahip bir prob kullanılarak hedef test ile çoklanabilir. Bu IPC testi, hedef taksonla ilgisi olmayan bir türden kısa bir sentetik DNA dizisinden oluşur, qPCR ana karışımına yaklaşık 102 kopya / reaksiyon düşük konsantrasyonda dahil edilir ve bunu algılayan astar ve problar. Bu düşük konsantrasyon, polimeraz ve nükleotitler için hedef dizi ile rekabeti önlemek içingereklidir 23.
- Örneğin IPC şablonunun Cq değerini şablon yok denetimdeki IPC şablonuyla karşılaştırın. Bu şablon denetimi yok (NTC), tek DNA girişi IPC şablonudur. Bu reaksiyondaki IPC şablonu beklendiği gibi yükseltilmelidir. Bir örnekteki IPC şablonu NTC'deki IPC şablonundan farklı olarak 2 veya daha fazla döngüde artarsa, eDNA örneği engellenmiştir. İnhibisyon gösteren örnekler 1:10'da seyreltilebilir ve yeniden test edilebilir. Bir örnek inhibe edilirse, bu örnek analizden çıkarılmalıdır.
- İlk olarak, test için en uygun sıcaklık ve astar / prob konsantrasyon değerlerini bulun. Bu parametreler PCR verimliliği (Adım 3.4.2), çapraz reaktivite (Adım 3.4.3) ve duyarlılık (Adım 3.4.4) için optimize edildikten sonra, testi çoklanmış bir IPC (Adım 3.4.5) ile test etmeye devam edin.
- Yerinde tahlil geliştirme ve test
- Laboratuvarda: Laboratuvardaki organizmaya ve sempatrik türlere erişim varsa; bu türlerle birlikte muhafazalardan su örnekleri alın, numuneleri işleyin ve testi bu eDNA örneklerine karşı test edin. M13 kuyruklu astarları kullanarak hedeflenen hedefin amplifikasyonunun doğrulanması için ürünleri yukarıdaki gibi sıralayın.
- Alanında:
- Hedef organizmanın oluştuğu bilinen ve gerçekleşmediği bilinen bölgeleri tanımlayın. Hedef türlerin meydana geldiği her yerde bir miktar bolluk ölçüsüne sahip olmak tercih edilir.
- Hangi numune hacimlerinin ve numune toplama yöntemlerinin (örneğin filtrasyon, santrifüjleme vb.) kullanılacağına karar verin.
- Her sahada bir alan boş veya negatif kontrol ekleyin, bu saha sahasına getirilen ve daha sonra eDNA örnekleme için kullanılan aynı saha ekipmanı ve protokolleri ile toplanıp hazırlanan temizsudur 24. Alanın boş olmasının amacı, alana getirilen numune alma ekipmanının ve saha ekipmanlarının kirlenmesini tespit etmektir. Alan suyu numunelerini işlemeden önce alanı boş alın.
- Saha başına birden fazla su numunesi alın, tercihen saha başına 3 numune alın.
- Laboratuvara geri dönün, numuneleri işleyin ve çıkarın.
- Şekil 5A'ya benzer bir plaka kullanarak tahlil çalıştırın ve eDNA konsantrasyonu ve algılama sıklığını bilinen site farklılıkları ve bolluk ile karşılaştırın. 24 , 25sırasını alarak tüm algılamaları onaylayın.
NOT: Yukarıdakiler, Thalinger ve arkadaşlarının (2020) ölçek 6'nın Seviye 4'ü(tahlil teknik performansının optimizasyonu) aracılığıyla bir tahlil doğrular ve Seviye 5 test doğrulamasını destekleyen verileri toplamaya başlar. Seviye 5, eDNA ekoloji çalışmaları için olasılık modellemesini ve testin kullanımını içerir. Bunun temel tahlil geliştirme kapsamının dışında olduğunu hissediyoruz, ancak test tasarımını ve veri yorumlamasını geliştirmek için laboratuvar ve saha incelemeli tahlillerin bu uygulamalarını teşvik ediyoruz.
Sonuçlar
Mucket (A. ligamentina) için türe özgü bir qPCR tahlili tasarlanırken, Clinch nehrindeki tüm Unionidae türlerinin mevcut dizileri indirildi. Lampsilis siliquoidea gibi yakından ilişkili türler de aynı nehirde bulunmasalar da referans veritabanına dahil edildi. Nehir sistemindeki tüm türler GenBank'ta bulunamadı, bu nedenle ek türler evde sıralandı. Diziler Geneious yazılımı kullanılarak hizalandı ve birden fazla tahlil tasarlamak için Primer Quest (IDT) yazılımı kullanıldı. Görsel değerlendirme için hizalamaya beş astar ve prob seti eklendi (Şekil 2). Daha sonra Primer-Blast kullanılarak silikoda test edildiler, daha sonra in vitro'yu daha fazla test etmeleriemredildi. Laboratuvarda, tüm testler özgüllüğü doğrulamak için mevcut 27 türün DNA ekstraksiyonları kullanılarak test edildi. Bir tahlil (A.lig.1) sadece hedef türleri başarıyla yükseltmiş (Tablo 1; Tablo 2). Bu test, test verimliliği, LOD ve LOQ'nun daha fazla test için ileriye taşındı. 121 baz çift amplicon uzunluğuna sahiptir. Tablo 3, A. ligamentina sentetik DNA standardı için kullanılan sırayı göstermektedir. Şekil 3A ve Şekil 3B, iyi verimlilik ve r2 değerleri ile başarılı bir tahlil sonuçlarını göstermektedir. Şekil 3C ve Şekil 3D, standart eğrisi düşük verimliliğe sahip bir test gösterir; bu test atıldı. Seçilen test için LOD ve LOQ (A.lig.1) her ikisi de Klymus ve ark 5'te açıklanan ayrık yöntem kullanılarak5.00kopya / reaksiyon olarak bulunmuştur. Test ile çoklanan IPC (Tablo 3-6) A. ligamentina testinin standart eğrisini etkilemedi. Kullandığımız IPC fare HemT transkriptinin bir parçası. Bu test, IDT tarafından başka bir uygulama için önceden tasarlanmıştı, ancak laboratuvarımızın eDNA uygulamaları için IPC olarak kullanımını değiştirdik.
Başarılı bir qPCR çalışması, her performans ölçüsü için belirli kriterleri karşılamalıdır (örneğin, standart eğri amplifikasyonu, genomik DNA pozitif kontrolü, şablon kontrolü ve dahili pozitif kontrol). Hedef test standartları üstel amplifikasyon eğrilerine sahip olmalıdır. Bu eğriler, yeterli döngü çalıştırmasına izin verilirse bir bitiş noktası platosunda ulaşmalıdır. Bu, floresan probun reaksiyon sırasında tamamen tüketildiğinin ve floresan seviyelerinin maksimum sınıra ulaştığının göstergesidir. Daha sonra yükseltici standartlar 40 döngüde bir platoya ulaşamayabilir. Pozitif kontroller (genomik DNA ve IPC) aynı örüntüye sahip olmalıdır. Bilinmeyenler yükseltilebilir veya yükseltilebilir, ancak bilinmeyenlerdeki amplifikasyonun da üstel bir deseni ve bir uç nokta platosu olmalıdır (Şekil 5).
Kaliteli bir qPCR'de, standart seyreltmeler, konsantrasyondaki her 10 kat fark için yaklaşık her 3,3 döngüde eşit aralıklı Cq'da yükseltilir. Standart bir seyreltmenin her bir çoğaltması, neredeyse aynı Cq'ya (r2 değerleriyle temsil edilir) sahip sıkı bir şekilde gruplandırılmış bir şekilde güç verir. Tüm standart seyreltmeler amplifikasyon sergilemelidir (Şekil 3A). Zayıf bir qPCR'de standartlar üstel olmayan şekil, seyreltmeler arasındaki Cq değerlerinde düzensiz varyasyon gösterebilir, uç nokta platosunda gelmeyebilir veya bazı seyreltmeler hiç yükseltmeyebilir(Şekil 3D).
Standart eğri için önemli parametreler verimlilik, r2, eğim ve y-kesişimdir. Verimlilik% 90-% 110 arasında düşmeli ve ideal değerler% 100'e yakın olmalıdır ve r2 değerleri 1.0 15,22'yeyaklaşan ideal sonuçlarla 0.98'in üzerinde olmalıdır. Eğim değerleri -3.2 ile -3.5 arasında olmalı ve ideal sonuçlar -3.322 'yeyakın olmalıdır. y-intercept değerleri 34-41 Cq arasında düşmeli ve ideal sonuçlar 37.0 Cq'ya sahip olmalıdır. y-intercept, tek bir qPCR ile ölçülebilen en küçük birim olan hedef sıranın 1 kopyasına sahip bir reaksiyonun tahmin edilen Cq'sudur. Cq'ların y-kesişiminden daha büyük olduğu bilinmeyenler muhtemelen engellenecektir. 40'tan fazla PCR döngüsü çalıştırmak, inhibisyon veya verimsiz bir astar kümesi durumunda hedefi tespit etmek için gerekli olabilir, ancak bu koşullar altında niceleme mümkün değildir ve hedef sırası olmadan ek negatif kontroller, ancak bilinmeyenlere benzer toplam DNA içeren, spesifik olmayan kaynaklardan amplifikasyonu ekarte etmek için çalıştırılmalıdır.
Bilinmeyen örneklerde İç Pozitif Kontrol (IPC) amplifikasyonu, reaktifler için bir rekabet olmadığı ve inhibitör bulunmadığından, negatif şablon kontrolü IPC sonuçları ile karşılaştırılmalıdır. 2 döngü CQ'su olan veya NTC'nin ortalama Cq değerinden daha büyük olan veya yükseltilmeyen bilinmeyenler inhibe edilmiş olarak kabul edilmelidir. Örneklerde inhibitör yoksa, tüm IPC amplifikasyonu, NTC ile aynı Cq değerlerine sahip arsada sıkı bir gruplandırmaya sahip olmalıdır (Şekil 6).
Son olarak, testin yerinde testi gerçekleşti. Clinch Nehri'nden yirmi su örneği ve üç alan boş numunesi, 25-26 Eylül 2019 tarihleri arasında A. ligamentinaolduğu bilinen bir midye yatağından 500 metre içinde filtrelenmiştir. Örnekleme yeri başına yaklaşık dört adet 1 L su numunesi filtrelendi. Deredeki midye yatağının dibinde, kıyıya yakın midye yatağının dibinde, deredeki yatağın 100 m aşağı akıntısında, deredeki yatağın 500 m aşağı akışında ve kıyıya yakın yatağın 500 m aşağı akışında yer alan konum siteleri (Şekil 7). Laboratuvarda, her filtre ikiye bölündü ve bir filtrenin sadece yarısından DNA çıkarıldı. Her numune için kalan filtre yarısı -80 °C dondurucuda saklandı. Örnekler daha sonra IPC ile katlanmış A.lig.1 tahlili kullanılarak çalıştırıldı. 23 numuneden beşinin inhibe edildiği tespit edildi. Bu örnekler 1:10'da seyreltildi ve seyreltmeler yeniden çalıştırıldı. Tasarlanan test kullanılarak 20 alan örneğinden 19'u güçlendirildi. Bu 19 numuneden beşi, 5 kopya/reaksiyonun test lod ve LOQ'sunun üzerindeydi; yani örneklerin çoğunda bir eDNA tespiti vardı, ancak yanlış olumsuz sonuçların ortaya çıkması muhtemel bir seviyede ve test bu 14 örnek için kopya numarasını güvenle ölçemedi. Bununla birlikte, dört biyolojik alanın% 75 ila 100'ü her örnekleme yerinde yükseltilir. Üç alan boşluğundan ikisi negatif çıkarken, bir alan boşluğu alanda temiz tekniğin önemini vurgulayarak amplifikasyon gösterdi.

Şekil 1: Mitokondriyal DNA dizisi veritabanı yapımı için iş akışı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: Clinch nehri midye türleri için sıra dizilimleri, Actinonaias ligamentina ND1 tahlili için prob astarları ve probları ile. Koyu yeşil renkte ileri astarlar, kırmızı prob ve açık yeşilde ters astar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
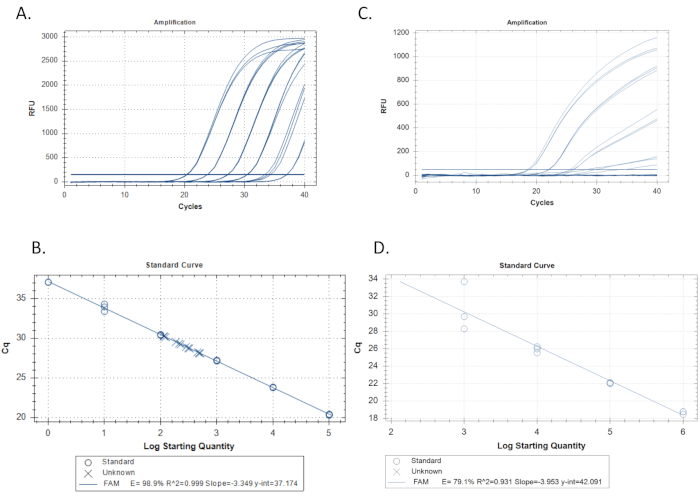
Şekil 3: Standart eğri ve doğrusal regresyon örnekleri. A. Üç büyütmeden elde edilen kabul edilebilir bir standart eğri örneği, altı standart seyreltmenin her birini çoğaltır. Soldaki standardın en yüksek konsantrasyonuna sahip 10 kat standart seyreltme serisi, azalan konsantrasyonlar sağa hareket eder. Tüm izlemeleri geçen yatay çizgi, nicellikte döngü eşiğidir (Cq). Her izlemenin bu eşiği geçtiği yer, Cq'nun belirlendiği yerdir. B. Şekil 3A'nın standart kopyalarından yapılan doğrusal regresyon. Standart seyreltmelerin kopyaları daireler halinde çizilir ve bilinmeyenler (örnekler) x'lerle çizilir. Verimlilik% 98.9, r2 1.0'a yaklaşıyor ve eğim -3.349. C. Üçnün amplifikasyonundan elde edilen zayıf standart eğri örneği, altı standart seyreltmenin her birini çoğaltır. D. Standart eğriyi oluşturan doğrusal regresyon, örnek 3C'de yükseltilmiş olarak çoğaltılır. Düşük verimlilik ve r2 değerlerine dikkat edin. Ayrıca, 6 standartın sadece 4'ünin yükseltildiğini unutmayın. Tekrar çalıştırıldıktan sonra standart eğri düzelmezse, sorun beklendiği gibi hedef DNA'sını yükseltmeyen zayıf bir astar / prob kümesi ile olabilir, bu test dikkate alınmamalıdır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 4: LOD ve LOQ standart qPCR çalıştırmaları için plaka kurulum örnekleri. Eğride kullanılan standartlar mavidir, standart konsantrasyon koyudan açık maviye düşer. Yeşilde DNA pozitif kontrolü ve sarıda şablon kontrolü (NTC) yoktur. Her standart seyreltme için 24 kopya gösteren gri deneysel standart konsantrasyonlar. Seyreltme serisi, her biri standart eğri, pozitif kontrol ve NTC ile iki plaka (A, B) boyunca kaplandı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
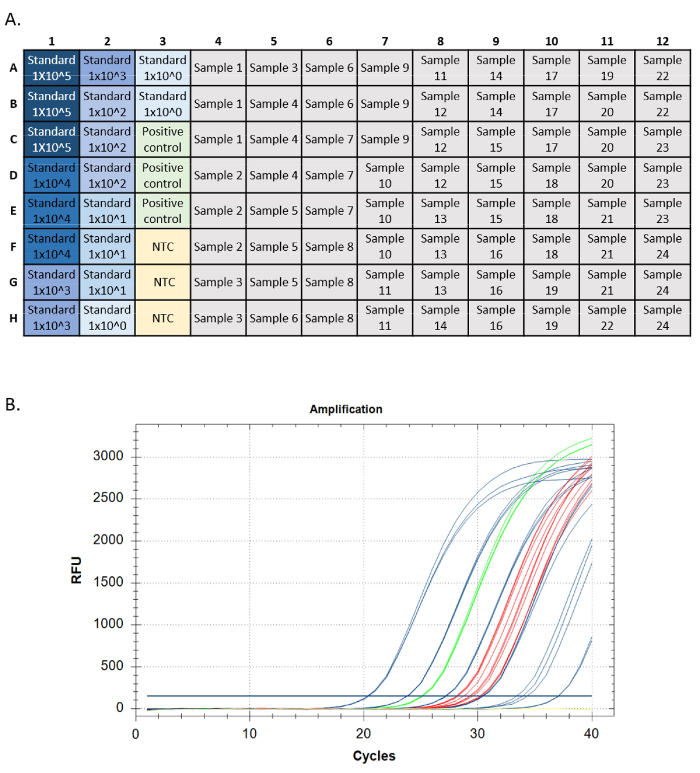
Şekil 5: Plaka kurulumu ve bir qPCR çalıştırmasından amplifikasyon izleri. A. Plaka kurulumu, standardın konsantrasyonunun en yüksek olduğunu gösteren mavi, daha koyu renkte gösterilen standartlar. Yeşilde DNA pozitif kontrolü, sarıda şablon kontrolü yok (NTC), gri renkte örnek hedefler. B. Bir qPCR çalıştırmasından amplifikasyon izleri. Mavi renkte gösterilen standartlar, yeşilde DNA pozitif kontrolü, sarıda şablon kontrolü yok ve kırmızıda bilinmeyenler. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
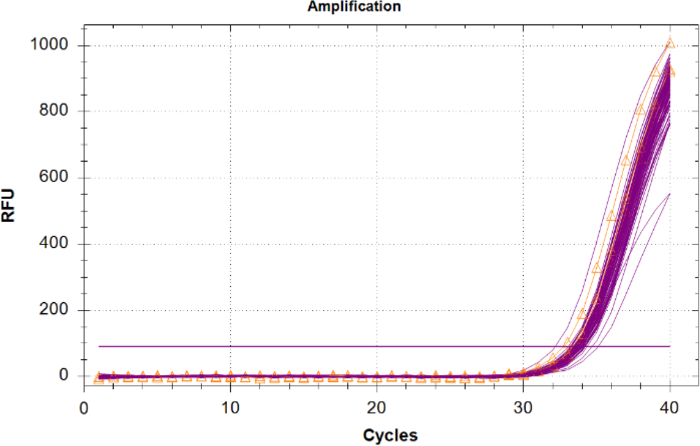
Şekil 6: İç Pozitif Kontrol (IPC) için amplifikasyon izleri. Üçgenlerle turuncu renkte gösterilen şablonsuz denetimlerden (NTC' ler) macenta ve IPC'deki bilinmeyen tüm örnekler için IPC izlemeleri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
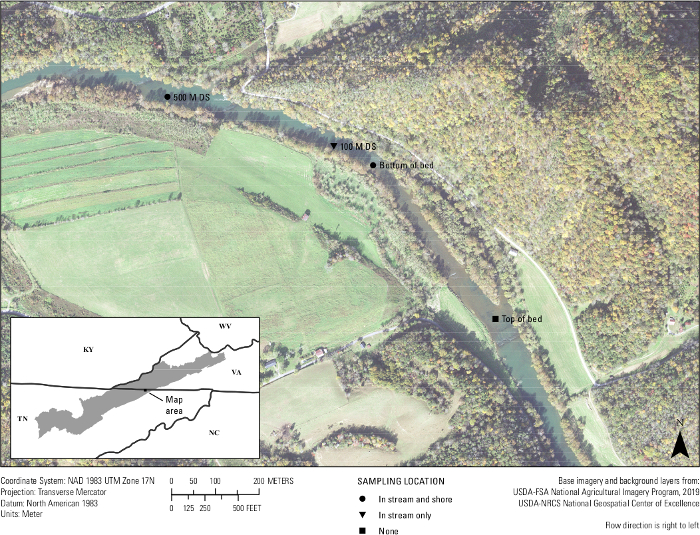
Şekil 7: Virginia/Tennessee sınırı boyunca Clinch Nehri'nde bir midye yatağının eDNA toplama alanlarını gösteren harita. Numuneler yatağın dibindeki Wallens Bend'de, yatağın 100 m aşağısında ve yatağın 500 m aşağı akışında toplanarak alındı. Siteler ya derenin ortasında (dere içinde) ya da kıyı şeridinden (kıyı) yaklaşık 1 – 2 metre uzakta toplandı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| parça | ad | Sıra 5' – 3' | Floresan etiketi | |
| İleri Astar | A.lig.1-f | CCCTCATCACGTACCTCTTAATC | ||
| Ters Astar | A.lig.1-r | GGAATGCCCATAATTCCAACTTTA | ||
| sonda | A.lig.1 prob | TTCTTGAACGTAAAGCCCTCGGGT | FAM | |
Tablo 1: İleri ve ters astarlar ve prob için diziler de dahil olmak üzere tasarlanan Actinonaias ligamentina qPCR tahlil (A.lig.1).
| tür | Güçlendirilmiş | Clinch Nehri'nde |
| 1. Actinonaias ligamentina | evet | evet |
| 2. Actinonaias pektorosa | Hayır | evet |
| 3. Amblema plicata | Hayır | evet |
| 4. Corbicula spp. | Hayır | evet |
| 5. Cumberlandia monodonta | Hayır | evet |
| 6. Cyclonaias tüberküla | Hayır | evet |
| 7. Cyprogenia stegaria | Hayır | evet |
| 8. Elliptio dilatata | Hayır | evet |
| 9. Epioblasma brevidens | Hayır | evet |
| 10. Epioblasma capsaeformis | Hayır | evet |
| 11. Epioblasma florentina aureola | Hayır | evet |
| 12. Epioblasma triquetra | Hayır | evet |
| 13. Fusconaia cor | Hayır | evet |
| 14. Fusconaia subrotunda | Hayır | evet |
| 15. Lampsilis ovata | Hayır | evet |
| 16. Lampsilis siliquoidea | Hayır | Hayır |
| 17. Lasmigona costata | Hayır | evet |
| 18. Lemiox rimosus | Hayır | evet |
| 19. Lexingtonia dolabelloides | Hayır | evet |
| 20. Medionidus conradicus | Hayır | evet |
| 21. Plethobasus cyphyus | Hayır | evet |
| 22. Pleurobema plenum | Hayır | evet |
| 23. Ptychobranchus fasciolaris | Hayır | evet |
| 24. Ptychobranchus subtentus | Hayır | evet |
| 25. Quadrula pustulosa | Hayır | evet |
| 26. Strophitus undulatus | Hayır | evet |
| 27. Villosa iris | Hayır | evet |
Tablo 2: A.lig.1 testinin in vitro özgüllük testi için kullanılan türlerin listesi. Test, hedefin genomik DNA'sını(Actinonaias ligamentina)güçlendirdi ve hedef olmayan türlerin hiçbirini güçlendirmedi.
| parça | Sıra 5'-3' | ||||
| Actinonaias ligementina standardı | CCCTCATCACGTAC CTCTTAATCCTATTAGGTGTCGCATTTCACTCTTCTTGAACGTA | ||||
| AAGCCCTCGGGT ACTTTCAAATCCGAAAAGGCCCAAATAAAGTTGGAATTATGCATTC | |||||
| CCCAACCATTAGCAGATGCTAAGCTCTTCGTAAAAGAATGAGTAACACCAACCTCCT | |||||
| CAAACTACACCCTTCATCTTAACCCCAACCACTATGTTAATTTTAGCACTTAGACTTT | |||||
| GACAATTATTTCCATCCTTTATANTATCATCCCAAATANTTTTTGGTATGCTCCTATTCT | |||||
| TGTGTATCTCCTCCCTAGCTGTTTATAACACTTATAACAGGCTGAGCCTCAAACTCCA | |||||
| AATATGCCCTTTTAGGAGCTATTCGAGCCATAGCCCAAACCATTTCTTATGAGGTTACAA | |||||
| TAAC | |||||
| IPC şablonu (Hem-T) | CTACATAAGTAACACCTTCTCATGTCCAAAGCTCTCTGAGTTCCCGAATCTCAGACGCT | ||||
| GTATGACAGTCTCCTTTCGTGAACATTCGGCTGCTCTATGTTCTCAAGGACTGCAC | |||||
Tablo 3: Actinonaias ligamentina standardının sırası (5'-3') ve bu test için kullanılan IPC şablonu (Hem-T). İleri ve ters astarların sırası kalın ve italiktir ve probunkinin altı çizilir.
| parça | ad | Sıra 5' – 3' | Floresan etiketi | |
| İleri Astar | HemT-F | TCTGAGTGTCCCGAATCT | ||
| Ters Astar | HemT-R | GCAGTCCTTGAGAACATAGAGC | ||
| sonda | HemT-P | TGACAGTCTCCTTTCGTGAACATTCG | Cy5 | |
Tablo 4: İç Pozitif Kontrol (IPC) tahlilleri, ileri ve ters astarlar ve prob için diziler de dahil olmak üzere.
| Numune başına hacim (μL) | parça |
| 10 | Çevre Master Mix |
| 1 | 20uM A. lig.1 F/R karışımı |
| 1 | 2.5uM A. lig.1 prob |
| 1 | 5uM IPC astar karışımı (HemT-F/ R) |
| 0.75 | 2.5uM IPC probu (HemT-P) |
| 1.5 | 1 X 103 IPC şablonu konsantrasyonu |
| 2.75 | H20 |
| 2 | örnek |
| 20 | Toplam Birim |
Tablo 5: A.lig.1 tahlil için kullanılan PCR karışımı, IPC testiyle çoklanmıştır.
| adım | Sıcaklık (°C ) | Saat | |
| 1 | İlk Denatüre | 95 | 10 dk |
| 2 | Denatüre | 95 | 15 sn |
| 3 | Tavlama | 60 | 1 dk |
| 4 | Adım 2'ye gidin, 39X'i yineleyin |
Tablo 6: A.lig.1 tahlilinin reaksiyon koşulları.
Tartışmalar
Her çalışmada olduğu gibi, ele alınacak soruyu tanımlamak ilk adımdır ve eDNA testinin tasarımı çalışmanın kapsamına bağlıdır.26. Örneğin, araştırmanın veya anketin amacı bir veya birkaç türü tespit etmekse, hedeflenen bir sonda tabanlı test en iyisidir. Bununla birlikte, amaç türlerin daha büyük bir paketini veya montajını değerlendirmekse, yüksek verimli sıralama metabarkodlama tahlilleri daha uygundur. Hangi yaklaşımın benimseyeceği belirlendikten sonra, tahlil tasarımı, test ve optimizasyon içeren bir pilot çalışma önerilir.24. Tahlil tasarımı, Şekil 1. Bu liste, bir tahlilin özgüllük ve uygulanabileceği coğrafi aralık açısından ne kadar iyi performans gösterdiğini anlamanın temelini oluşturacaktır.6,10. Testin belirli bir coğrafi alan için tasarlanması, tasarımcının o bölgedeki diğer türlere karşı çapraz reaktivite için bir testi daha iyi test etmesini ve bunun bir testin hedef türün oluşabileceği diğer alanlara genişletilmesindeki sınırlamaların farkında olmasını sağlamak teşvik edilir.24. Liste tamamlandıktan sonra, diziler genel genetik veritabanlarından indirilebilir. Bu veritabanları eksik olduğundan27, tahlil tasarımında kullanılacak dizilerin yerel referans veritabanını tamamlamak için listedeki mümkün olduğunca çok türü evde sıralamalıdır. Birlikte meydana gelen yakından ilişkili türlere öncelik vermek, çünkü bunlar en olası hedef olmayanlardır. Hedef türle aynı cins veya aile içindeki tüm türlere odaklanmak başlamak için iyi bir yerdir. Yakından ilişkili türlerle yapılan karşılaştırmalar, hedef türlere özgü sıra bölgelerini belirlemeye yardımcı olacaktır. Bu, tahlillerin diğer sistemlerde veya konumlarda nasıl performans gösterebileceğini bildirmeye yardımcı olabilir. Mitokondriyal bölgeler tahlil gelişimi için olağan seçimdir, çünkü yaşam projelerinin barkodunda kullanılan mitokondriyal genlerde daha çeşitli türlerden daha fazla dizi bilgisi mevcuttur ve mitokondriyal DNA, kopyalarda / hücrede nükleer DNA'dan çok daha fazla konsantrasyonda bulunur.24,28,29. Dizi kapsamı genetik depo veritabanlarındaki takson arasında değiştiği için daha fazla test gelişimi için birden fazla gen bölgesi değerlendirilmelidir. Bu yerel başvuru dizileri veritabanı oluşturulduktan sonra, astar/prob tahlillerini tasarlamak için hizalanmış sıra verilerinin ve bilgisayar yazılım programlarının manuel görselleştirilmesinin bir kombinasyonu kullanılır. Hangi tahlillerin test edilmesi gerektiğini belirlemek için kesinlikle yazılıma güvenmemek gerekir. Bir PCR'de nasıl davranabileceklerini daha iyi anlamak için astarların ve probların hedeflere ve hedef olmayanlara oturduğu hizalamalarda görsel olarak doğrulamak önemlidir. Son olarak tahlil tarama ve optimizasyon üç seviye içerir (siliko, in vitro ve in situ)6,7,24,25. Siliko tasarımında ve testlerinde başarı şansı yüksek kısa bir test listesi üretmek önemlidir, ancak ampirik (in vitro) test, tahlilleri en iyi gerçek performansla seçmek için çok önemlidir. In vitro optimizasyonu ve tahlillerin test edilmesi, reaksiyon verimliliğinin ölçülmesi ve testin hassasiyetinin ve özgüllüğünün tanımlanmasını içerir. Algılama ve niceleme sınırları, tahlil geliştirmede sıklıkla göz ardı edilen ancak veri yorumlama için önemli olan iki parametredir. Bir test için standart eğrilerin birden fazla çoğaltmasını çalıştırarak, LOD ve LOQ kolayca ölçülebilir1,5,30. Çok az çalışma, testin LOD veya LOQ'su ile ilgili sonuçları tartışır, ancak Sengupta ve arkadaşları (2019), sonuçlarının daha net anlaşılması için testlerinin LOD ve LOQ'larını veri yorumlama ve grafiklerine dahil eder.31. dahili pozitif kontroller de tasarlanan teste çoklayıcı olmalıdır. Örneklerde PCR inhibisyonu testi yapılmadan, yanlış negatifler oluşabilir24,32. PCR inhibisyon testi için en kolay yöntem olarak hedef test ile çok yönlü bir IPC testinin kullanılmasını öneriyoruz23. Son olarak, saha ve laboratuvardan toplanan numunelerden yapılan testin yerinde test edilmesi, çevresel örneklerde hedef amplifikasyonun gerçekleşmesini sağlamak için gereklidir.24.
Türlere özgü, prob tabanlı qPCR testlerinin eDNA örnekleriyle kullanımı için sınırlamalar mevcuttur. Örneğin, test için birden fazla tahlil tasarımı sıra kullanılabilirliği ile sınırlı olabilir ve test performansının yönleri üzerinde uzlaşma gerekebilir. Bu seçimler çalışmanın hedeflerine göre yönlendirilmeli ve sonuçlarla birlikte bildirilmelidir26. Örneğin, amaç nadir bir türün tespiti ise ve birkaç pozitif bekleniyorsa, tüm algılamalar sıralama ile doğrulanacaksa kusurlu özgüllüğe sahip bir tahlil (yani hedef olmayan türlerin amplifikasyonu) kullanılabilir. Amaç bir türün coğrafi aralığını izlemekse ve eDNA konsantrasyon verilerine gerek yoksa, kusurlu verimliliğe sahip bir test kullanılabilir ve veriler yalnızca yüzde tespiti olarak bildirilebilir. Ayrıca, tüm potansiyel spesifikler laboratuvarda test edilmediği sürece, ki bu nadiren mümkündür, bir testin gerçek özgüllüğünü kesin olarak bilemez. Örneğin, test Clinch Nehri'ndeki birkaç tatlı su midye türüne karşı tasarlanmış ve test edilmiştir. Bu testi farklı bir nehir sisteminde kullanmak için, yeni yerdeki bir tür paketine karşı test etmemiz gerekir. Test gelişimi sırasında test olmayan türler veya popülasyon içindeki genetik varyasyon da özgüllüğü etkileyebilir. Son olarak, bir tahlil yüksek teknik performansa sahip olduğu doğrulanmış olsa bile; alanda çalışırken koşullar değişir. Su akışı, pH ve hayvan davranışı gibi testle ilgili olmayan koşullar, farklı eDNA toplama ve çıkarma protokollerinin kullanılabileceği gibi eDNA algılanabilirliğini değiştirebilir. Optimize edilmiş ve iyi tanımlanmış tahlillerin kullanılması, bu tür parametrelerin eDNA algılaması üzerindeki etkisinin anlaşılmasını kolaylaştıracaktır.
eDNA alanı, keşif analizi aşamasının ötesinde, yöntem ve tekniklerin standardizasyonunu artırmaya kadar olgunlaşmaktır. Bu gelişmeler eDNA tekniklerini, yeteneklerini ve sınırlamalarını anlamamızı geliştirecektir. Yukarıda özetlediğimiz optimizasyon süreci, bir analizin hassasiyetini, özgüllüğünü ve tekrarlanabilirliğini geliştirir. Bu iyileştirmenin ve eDNA yöntemlerinin standardizasyonunun nihai amacı, araştırmacıların eDNA verilerine dayalı çıkarımlar yapma yeteneklerini geliştirmek ve sonuçlara olan son kullanıcı ve bahis sahibi güvenini artırmaktır.
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyanda bulunun. Fon sponsorlarının çalışmanın tasarımında hiçbir rolü yoktu; verilerin toplanmasında, analizlerinde veya yorumlanmasında; yazının yazımında; veya sonuçların yayımlanması kararında.
Teşekkürler
Astar geliştirme ve testlerinde yardımcı olan Alvi Wadud ve Trudi Frost'a teşekkür ederiz. Bu çalışmada bildirilen tahlil tasarımı için fon Savunma Bakanlığı Stratejik Çevre Araştırma ve Geliştirme Programı (RC19-1156) tarafından sağlanmıştır. Ticari, ürün veya firma adlarının herhangi bir şekilde kullanılması yalnızca açıklayıcı amaçlar içindir ve ABD Hükümeti tarafından onay anlamına gelmez. Bu çalışma sırasında oluşturulan veriler USGS veri yayınlama https://doi.org/10.5066/P9BIGOS5 olarak mevcuttur.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
Referanslar
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır