Method Article
環境DNA応用に関する種特異的定量PCRアッセイの開発と試験
要約
環境DNAアッセイでは、現場データの収集を開始する前に、厳格な設計、テスト、最適化、検証が必要です。ここでは、環境試料から標的種DNAの検出と定量を行うための、種特異的なプローブベースのqPCRアッセイを設計する各ステップを通じてユーザーを取るプロトコルを提示する。
要約
漁業や野生動物保護管理を支援するために、種の存在を検出し、監視するための新しい非侵襲的な方法が開発されています。マクロビオタを検出するための環境DNA(eDNA)サンプルの使用は、急速に普及し、国家管理プログラムで実施されている方法の一つです。ここでは、プローブベースの定量PCR(qPCR)アプリケーションに対する種特異的標的アッセイの開発に焦点を当てています。プローブベースのqPCRを使用すると、プライマー単独で可能な限り特異性が高くなります。さらに、サンプル中のDNAの量を定量化する能力は、eDNAの生態と、現場におけるeDNA検出パターンの解釈を理解するのに役立ちます。環境試料から標的種を検出する感度と特異性を確保するために、これらのアッセイの開発と試験には慎重に検討が必要です。このプロトコルでは、標的種の検出のためのプローブベースのアッセイを設計およびテストするために必要な手順を説明します。シーケンスデータベースの作成、アッセイ設計、アッセイの選択と最適化、テストアッセイ性能、フィールド検証など。これらの手順に従うと、効率的で、敏感で、かつ、信頼できる特定のアッセイを実現するのに役立ちます。このプロセスは、米国クリンチ川で見られる淡水ムール貝種であるマケット(アクチナガイアス・シネラシナ)の集団のために設計されたアッセイで実証しています。
概要
研究者や管理者は、種検出のための環境DNAアッセイの使用にますます関心を持つようになっています。30年にわたり、核酸1,2の配列特異的検出および定量化のために、定量的またはリアルタイムPCR(qPCR/rtPCR)が多数の分野で使用されてきました。eDNA研究の比較的新しい分野では、eDNAサンプルの体積または重量当たりの標的DNAのコピーを定量化するための標準曲線を用いたアッセイの使用が、今や日常的な実践となっている。ミトコンドリアのDNA配列は、ミトコンドリアゲノムが細胞当たり数千部に存在するため、一般にeDNAアッセイで標的とされているが、核DNAまたはRNA配列のアッセイも可能である。eDNAサンプルに対して公開されたアッセイは、必ずしも性能が等しいとは限らないということを理解することが重要です。標的種のDNA(特異性)と低量の標的DNA(すなわち、感度)の検出におけるアッセイの信頼性は、アッセイの設計、選択、最適化、試験方法の違いにより大きく異なる可能性があります。アッセイ性能の定量的尺度報告は以前はほとんど見落とされてきたが、最近ではアッセイ開発の透明性を向上させる基準が3、4、5、6、7、8に浮上している。
eDNA調査結果の設計と解釈の研究に役立つアッセイ性能の最適化と報告。非標的種DNAとの交差反応は偽陽性検出につながる可能性があるが、感度の低いアッセイは、サンプル中に存在する場合でも標的種DNAを検出できない可能性がある(偽陰性)。アッセイ感度と選択性を理解することは、希少種の検出に必要なサンプリング作業を知らせるのに役立ちます。eDNAには多くの自然な変動源があるため、eDNAアッセイ3を完全に最適化し、特性を付け込むなど、制御可能な変動源を可能な限り制限する必要があります。
アッセイの特異性や感度に直接影響する条件は、アッセイの性能を変えます。これは、異なる実験室の条件下で発生する可能性があります (すなわち、異なる試薬、ユーザー、機械など)。したがって、このプロトコルは、新しい条件下でアッセイを適用する際に再検討する必要があります。文献に良く特徴付けられるアッセイでさえ、新しい実験室で採用されたとき、または異なる試薬(例えば、マスターミックス溶液)5,9を使用する場合に、試験および最適化されるべきである。アッセイ特異性は、異なる地理的領域に適用されると変化する可能性があり、アッセイが検査されていない非標的種を含む新しい生物学的コミュニティからのサンプルにアッセイが適用されているため、標的種の遺伝的変異が生じる可能性がある。再び、アッセイは新しい場所で使用する場合に再評価されるべきである。フィールド PCR 阻害剤はサンプルに存在する可能性が高いため、フィールド条件は実験室の条件とは異なります。PCR阻害剤は、増幅反応に直接影響を与え、アッセイ性能に影響を与えます。このため、eDNAアッセイを開発する際には、内部の正のコントロールが必要です。
最後に、現場の環境条件は、標的種のDNA分子に影響を与え、DNA分解、輸送および保持を通じて捕捉する可能性があります。さらに、DNAの収集と抽出のための異なるプロトコルは、その効率とDNAを保持する能力が異なります。しかし、これらのプロセスはeDNAの検出可能性に影響を与えますが、分子アッセイの性能には影響しないことに注意することが重要です。したがって、フィールドサンプル中の標的種からのDNAの検出可能性は、qPCRアッセイの技術的性能と、フィールド条件および収集、保存、抽出プロトコルの両方の機能である。よく特徴付け、高度に実行アッセイを使用する場合、ユーザーはアッセイの能力に自信を持つことができます。研究者は、eDNA検出に影響を与える外部アッセイ因子(すなわち、環境変数、捕獲または抽出プロトコルの違い)の理解に集中できるようになりました。
ここでは、厳密な設計と最適化によるアッセイ技術性能に特に焦点を当てています。米国クリンチ川でサンプリングした水から淡水ムール貝(アクチナガイアス・靭帯)を検出するために開発されたプローブベースのアッセイを用いてプロトコルを実証します。最近、Thalingerら(2020)は、標的eDNAアッセイの検証のためのガイドラインを提示した。私たちのプロトコルに従ったアッセイ設計は、Thalingerらのレベル4にアッセイに加えて、レベル5 6に向けた追加のステップをもたらすでしょう。この時点で、アッセイの技術的性能が最適化され、実験室および現場でのアプリケーションでの定期的な使用に備える。実験室でのアッセイのさらなる使用、メソコスム、およびフィールド実験は、次に、eDNA検出および検出性に影響を与える因子に関する質問に対処することができる、レベル5検証6のための最終ステップ。
プロトコル
1. 標的種および非標的種からのミトコンドリアDNA配列の配列データベースの生成
- 対処する質問、ターゲット、システムを定義します。eDNA検出のターゲット種を特定します。アッセイが使用される地理的システムを特定します。対象種を含む対象種のリストを作成し、同じ分類体内の交配(共生)種(通常はオーダーまたはファミリーレベル)、および密接に関連する同種の同じ地理的位置に存在し得るもの(図1)。
注:ここでは、種 A.靭帯の クリンチ川の集団が標的にされました。 - ステップ1からリスト上の種の複数の遺伝子領域から配列を検索してダウンロードします。NCBI(国立バイオテクノロジー情報センター)、BOLD(バーコード・オブ・ライフデータベース)、EMBL(欧州分子生物学研究所)、DDBJ(日本のDNAデータバンク)などの配列データベースを使用できます。NCBI、EMBL、および DDBJ は、すべてシーケンス情報を共有します。
- NCBIのヌクレオチドデータベースを使用して、標的生物(例えば、 アクチナイアス・シネアシナ)および遺伝子領域(例えば、シトクロムcオキシダーゼI(COI)またはNADH-デヒドロゲナーゼ1(ND1)を検索する。検索文字列例: アクチナアイアス・靭帯およびND1)
- 次に、仕様に一致するすべてのシーケンスを選択し、[送信] を選択します。[完全なレコード]、[ ファイル]、および [ファイル] を選択し、[ファイルの作成] を選択します。これらのシーケンスは、コンピューターに保存されます。
- ステップ 1 で定義したリストのすべての種について、これらの手順を繰り返します。各遺伝子領域の配列は別々のファイルに保存し、個別に分析します。
- ステップ 1 で特定されたターゲット種に関連するすべてのシーケンス(または、シーケンスの代表比率)をすべてダウンロードします。可能であれば、地理的バリアントを含めます。
- ステップ1で同定された同じ分類群の関連および同伴的な非標的種の検索とダウンロードシーケンスを繰り返します(例えば、標的種がマケット(A.靭帯)である場合)、対象系で発生するファミリーユニオン科の他のすべての淡水ムール貝種のダウンロードシーケンス。
- ステップ 1.1 に記載されている、密接に関連するアソパトリ (地理的に別々の) 種の検索とダウンロードを繰り返します。
注: すべての種(ターゲットとターゲット以外)が公開データベースで利用できるわけではありません。国内で関心のある種の分類学的に検証された標本を増幅し、シーケンシングすることにより、ローカル参照データベースを増やします。種内の遺伝的多様性が高い種で作業したり、地理的変異体が予想される地理的に広い地域で働いている場合は、その範囲全体から配列を収集します。
2. アッセイ設計
- 様々な遺伝子配列編集およびバイオインフォマティクスプログラムで見つけることができるアライメントソフトウェアを使用して、各遺伝子領域からの配列を別々に整列させます。異なる遺伝子領域のそれぞれについてこのアライメントを行います.
- たとえば、Geneious Prime ソフトウェア (https://www.geneious.com) を使用すると、ダウンロードしたシーケンス ファイルがプログラムにインポートされます。
- 遺伝子領域ごとに別々のフォルダーを作成します。
- 1 つの遺伝子領域の配列を含むフォルダー内で、すべての配列を選択します。
- 選択した配列のヌクレオチドアライメントを作成するには、 複数 配置ツールを使用します。 [Geneious] または [MUSCLE] のアライメントを使用して、アライメントの種類にはいくつかのオプションがあり、デフォルトのパラメータが適切に機能します。
- 整列配列データの視覚化を通じてアッセイ設計のための有望な領域を選択します。対象となる種に対して利用可能な多くの配列データを持ち、種間で非常に発散し、種内変動が低いことを示す領域が良い候補である。これにより、設計されたプライマーとプローブがターゲットを非標的種から区別できる可能性が高まり、同時に特異的な変異体がアッセイで増幅される可能性も高まります。
- アッセイプライマーとプローブの設計。
- qPCRアッセイ設計ソフトウェアを使用し、指示に従ってください。ここでは、5セットのqPCRアッセイを設計するためのIDTのプライマークエストツール(https://www.idtdna.com/)を使用しました。
- ステップ 2.2 で選択したシーケンスをシーケンス入力ボックスに貼り付けます。線形によってスペースが作成された場合は、シーケンスからスペースを削除します。
- [デザインを選択]オプションでqPCR 2プライマー+プローブを選択します。
- 推奨アッセイをダウンロードします。
- 第1アッセイの順次プライマーから配列をコピーし、ステップ2.1.4で作成したアライメント内でこのプライマー配列を検索する。[Geneious Prime] を使用する場合は、[アコージン]ツールと[予測]ツールを使用して、プライマー領域をアライメントに追加します。すべてのプライマーとプローブの組み合わせに対してこれを行います(図2)。
- これらのアライメント領域で、ターゲット種内および共生種内の変動がないか調べます。
- 特異性の遺伝子変異がある場合は、プライマーとプローブがこれらの領域内に含まれていないアッセイを検索します。
- 非標的種の増幅を防ぐには、非ターゲット種との不一致を検索します。さらに検証するために、最も不一致のアッセイを選択します。Currier et al. (2018) すべての非標的種と少なくとも2つの不一致を有する3つの領域(2つのプライマー、またはプライマーおよびプローブ)のうちの少なくとも2つを有するセットを選択することを示唆している。ただし、プローブでの不一致は特異性10に影響を与える可能性が低い点に注意してください。
注:各プライマーの3'末端の3塩基対内の違いは、プライマー10の5'末端の違いよりも特異性を高めます。
- アッセイ設計における次の重要なパラメータを考慮する。
- プライマーとプローブの融解とアニーリング温度を決定します。理想的には、プライマーの融解温度(Tm)は60〜64°Cの間と互いの2°C以内にする必要があり、プローブのTmはプライマーのTmよりも6〜8度高くする必要があります。qPCR反応の焼鈍温度(Ta)を溶解温度より5°C以下、55~60°C11付近に設定する。
- GC コンテンツを調べます。35~65%のGCコンテンツを選択し、4つ以上の連続したGsを持つ領域を避けてください。プライマーの3'末端の5個の最後の塩基に1または2のGsまたはCsを有する(GCクランプ)は、プライマーが強い結合12を作るのを助けるので特異性を増加させるかもしれない。
- ヘアピンとダイマー構造を検索します。オリゴヌクレオチド分析プログラムを用いて、予測されたヘアピン構造およびダイマーのプライマーおよびプローブをテストする(例えば、OligoAnalyzer -IDT13;オリゴ電卓14.これらの構造は、非標的増幅および低効率を引き起こす可能性があります。これらの構造を形成すると予測されるアッセイは避けてください。
- プライマーの長さを決定します。長さ18~25基のプライマーと20~25基のプローブ長を目指します。プライマーおよびプローブが長いほど、増幅効率が低い場合があります。
- アンプリコンの長さを決定します。約 100 ~ 250 の基本ペアを指定します。この範囲は、一般的に高いPCR効率のために十分に短いですが、サンガーシーケンシング4、15による検証の容易さのために十分な長さです。
- プローブを設計します。プローブは、緑と黄色の染料11からの信号を減衰させる可能性があるため、5'の端にGベースを持たないようにしてください。私たちは、IDT 3IABkFQとZENクエンチャーとFAMまたはHEXフルオロフォアを備えた二重急クションプローブを設計しました。
注: MGB プローブの決定: TaqMan MGB (マイナーグルーブ バインダー) プローブは、eDNA の調査によく使用されます。しかし、これらのプローブは非常に短いため、2または3のベースペアのミスマッチ10でも非ターゲットに結合することができます。 - プローブのTm.融解温度を決定し、プライマーより6〜8°C高くする必要があります。温度が低いと、プローブの結合成功が減少します。
- プローブの長さと位置を確認します。プローブは、長さが20~25bpの間で、重ねることなく同じストランド上のプライマー結合部位の近くに理想的に位置する必要があります。
3. アッセイのスクリーニングと最適化
- シリコアッセイの開発とテストで。プライマープローブセットを注文する前に、インシリコでプライマー増幅をテストして特異性(潜在的な非標的増幅)を評価する。
- NCBIのプライマーブラスト16 または同様のプログラムを介してプライマーをテストし、アッセイで増幅する可能性のあるNCBI nt/nrデータベース内の潜在的な非標的を特定することができます。プライマー-ブラストペーストプライマーを使用する場合は、プライマーパラメータの下に 自分のプライマー ボックスを使用します。プライマーペア特異性チェックパラメータオプションで、データベースとして nr を選択し、対象生物の順序(例えば、「Unionida」または「Unionoida」)を [生物 ]ボックスに入力します。
- 位置合わせされたシーケンスデータのプライマー/プローブセットを視覚的に評価し続けます。
- インシリコでプライマーとプローブを同時に評価するために、フォワードプライマー、12 N、プローブ、12 N、および逆プライマーの逆補体のテキスト文字列を作成します。プローブシーケンスがプライマーの1つの基本ペアの12塩基対内にある場合は、プライマーとプローブの間の塩基対数に対応するNの数を使用します。
- NCBIのヌクレオチドブラスト検索(Blastn)を使用して、nrデータベース17に対して検索します。[分類] タブを使用して、不一致の少ない対象外の種を探します。これらは、アッセイの最適化中に実験室でテストする必要があります。
注:シリコ試験では、非特異的なアッセイを排除するのに役立ちますが、すべての種が遺伝的データベースに配列を持っているわけではないので、潜在的に特定のアッセイを経験的にテストする必要があります(in vitro)、プライマーとプローブは、ソフトウェアによって可能性が低いと判断された場合でも、非ターゲットに結合することができます。
- 実験室でテストするために3から5プライマー/プローブの組み合わせを選択してください。
- オーダープライマー、プローブおよび合成DNA標準だけでなく、アンプリコンシーケンシングのための追加のM13尾プライマー。
- オリゴを作る会社から合成オリゴヌクレオチドプライマーおよびプローブを注文する。プローブは蛍光染料とクエンチャーで標識されています。多重化する必要があるアッセイには異なる蛍光ホルを選択する必要があります。qPCR機器で、計測器が検出できる蛍光ホルのリストを確認します。
- M13フォワード(-20)シーケンス、GTA AAA CGA CGG CCA GT、フォワードプライマーの5'末端、およびM13リバース(-27)シーケンス、CAG GAA ACA GCT ATG ACを追加することにより、サンガーシーケンシングによるqPCR検出の検証のためのM13テールプライマーを設計および注文します。
- 合成DNA標準は、コピー/μLの既知の濃度で標的配列(プライマー領域を含む)を含み、この標準の既知の濃度(すなわち標準曲線)によって作られた曲線に基づいて未知のサンプルを定量化する。プライマーとプローブを製造する同じ会社から合成規格を取得します。再中断とストレージの製造元の推奨事項に従ってください。低保持プラスチックウェアを使用して、加水分解を低減し、表面に結合するtRNAキャリアを有するTEバッファーの希釈規格。
注: 標準曲線がうまく動作しない場合(PCR効率が悪く、ステップ3.4.2を参照)、水またはTris-HClで標準を再中断してみてください。 - ヌクレアーゼフリー水、Tris-HCl、またはTEバッファー内のプライマーおよびプローブを、アッセイ用に都合のよい濃度で懸濁します。一般的に、マスターミックスで20倍の希釈加工ストックを使用して、最適化された最終アッセイ濃度を達成する。使用しない場合は、一定の-20°Cで懸濁オリゴを保存します。
- インビトロ(実験室で)アッセイの最適化とテスト。効率が悪い、共同発生種とクロス反応する、感度が低いアッセイを拒絶する18.アッセイの開発中や実際のサンプルを実行する際に、内部陽性制御(IPC)の使用を含めます。
- まず、アッセイの最適温度とプライマー/プローブ濃度の値を求めます。これらのパラメータが PCR 効率(ステップ 3.4.2)、クロス反応性(ステップ 3.4.3)、感度(ステップ 3.4.4)に最適化されたら、多重化された IPC でのアッセイのテストに進みます(ステップ 3.4.5)。
- PCR温度勾配を中心としたPCR温度勾配を使用して、予測平均プライマーTmを5°C下回るプローブの最適アニーリング温度(Ta)を試験します。
- 最適なプライマーとプローブ濃度をテストします。典型的には、200 nM、400 nM、および800 nMプライマー濃度および75 nM、125 nM、および200 nMプローブ濃度が試験される。
- 標準曲線を作成し、効率と線形範囲を決定します。標的配列を含む合成DNA標準の少なくとも6つの10倍希釈を、約100 コピー/反応で105 コピー/反応(図3A)で試験する。
- qPCR ソフトウェアを使用して、各標準の Cq 値 (定量時のサイクルのしきい値) を y 軸に、x 軸上のコピー/リアクションの初期標準濃度の対数ベース 10 をプロットします。qPCR ソフトウェアは、線形回帰を自動的に実行する必要があります (図 3B)。
- 回帰の勾配から効率を計算する E = -1 + 10(-1/傾斜)を計算します。たとえば、傾斜角が -3.4 の場合、E= -1 + 10(0.29) = 0.97 または 97% です。また、標準の反復がカーブにどれだけ適合するかを示す r2の値も確認してください。qPCR ソフトウェアは、この計算も自動的に行う必要があります (図 3B)。100%の効率値を目指す(±10%)r2値 ≥0.989,15 ,19,20, 21,22.
- 標準曲線の偏り、つまり、一貫した方向の回帰からの偏差、または効率とr2値で測定された標準曲線性能の低下を視覚的に調べる(図3Cおよび3D)。
- 特異性:標的でない種との交差反応性を評価して、偽陽性の可能性を減らす。eDNA検出によって管理上の意思決定がコストのかかる場合は、アンプリコンシーケンシングによる正の検出を検証します。
- 非標的:関連種および地理的に同時発生する種の分類学的に検証された標本のゲノムDNA抽出に対するアッセイを実行する。最も優先順位が高いのは、密接に関連する、共同発生種に対してテストすることです。ターゲットサンプルと非ターゲットサンプルの両方に同様の総DNA濃度を使用します。選択された濃度は、標準曲線の線形範囲の中間付近の標的種サンプルから増幅を得る必要があります。増幅は標的種でのみ観察されるべきである。
- 非標的増幅が観察された場合は、製品をクリーンにして配列し、そのアイデンティティを検証します。非標的種の組織サンプル中の標的種からの汚染を観察することは珍しくないため、この段階でのすべての増幅はシーケンシングによって検証されるべきである。M13尾プライマーとM13プライマーとシーケンスを使用して特異性試験から洗浄された増幅器を再増幅します。
- PCR後の実験室で、新しい管に配列されるqPCRプロダクトを移す。クリーンアップキット(MinElute PCR精製キットなど)を使用して残留プライマーおよび反応成分を除去します。
- 溶出の1:100希釈を行い、M13テールプライマーと高忠実度ポリメラーゼ(例えば、フシオンハイフィデリティDNAポリメラーゼ)との50 μL PCR反応で30サイクルずつそれぞれ1μLを増幅します。
- 1%アガロースゲルで各反応の10 μLを実行し、予想されるサイズの単一バンドを確認します。バンドが観測されない場合は、サイクル数またはサンプル量を増やします。複数のバンドが観察される場合、ゲルは予想されるサイズのバンドを浄化する。
- 上記のようにクリーンアップキットで残留プライマーと反応成分を除去し、溶出のDNA濃度を測定します。
- シーケンシング施設の指示に従って、M13プライマーとのシーケンシング反応を設定します。
注: qPCR ラボで増幅されたサンプルを開かないでください。PCR後のサンプル専用の実験室でシーケンシング用サンプルを準備します。
- 感度:感度は、偽陰性の可能性、または存在する場合に標的種DNAを検出する失敗の可能性に影響を与えます。各アッセイの検出限界(LOD)と定量(LOQ)の限界を評価します。最後に、サンプルのPCR阻害を評価する内部陽性制御(IPC)を含む。多重と2つのアッセイが互いに干渉しないことを確認するために設計されたアッセイを用いてこのIPCアッセイをテストする。
- LOD: 合成DNA標準の6倍の4倍の連続希釈を行い、標準希釈ごとに8〜24回の複製を行います(図4)。95%の検出で最も低い初期濃度を計算します。LOD および LOQ プロットは、LOD/LOQ 計算機 R スクリプト5で生成できます。
注: LOD の下のデータは検閲しないでください。PCR の特異性のため、真陽性の下限はありません。LODは、偽陰性が発生すると予想される最も高い濃度です。 - LOQ:同じ希釈系列から、35%未満の変動係数(CV)で定量可能な最も低い初期DNA標準濃度を計算します。
注: LOD と LOQ は、コピー/リアクションで報告する必要があります。検証済みのアッセイとフィールドサンプルを使用する場合、正確な濃度は信頼できる5で測定できないため、eDNA濃度ではなく%検出として結果を報告する必要があります。
- LOD: 合成DNA標準の6倍の4倍の連続希釈を行い、標準希釈ごとに8〜24回の複製を行います(図4)。95%の検出で最も低い初期濃度を計算します。LOD および LOQ プロットは、LOD/LOQ 計算機 R スクリプト5で生成できます。
- 内部陽性コントロール (IPC) を使用して PCR 阻害をテストします。阻害は、感度の低下と偽陰性につながることができます。.標的アッセイで多重化されるIPCアッセイの能力を試験する。
- IPCアッセイは、ターゲットアッセイとは異なるレポーター色素を用いたプローブを用いて、標的アッセイで多重化することができる。このIPCアッセイは、標的分類とは無関係の種からの短い合成DNA配列から成り、それを検出するプライマーおよびプローブと共に、約102 コピー/反応の低濃度でqPCRマスターミックスに組み込まれる。この低濃度は、ポリメラーゼおよびヌクレオチド23の標的配列との競合を避けるために必要である。
- サンプルの IPC テンプレートの Cq 値を、テンプレートなしコントロールの IPC テンプレートの Cq 値と比較します。このテンプレートコントロールなし(NTC)では、唯一のDNA入力はIPCテンプレートの入力です。この反応の IPC テンプレートは期待どおりに増幅する必要があります。.サンプル中の IPC テンプレートが、NTC の IPC テンプレートとは異なる 2 サイクル以上で増幅する場合、eDNA サンプルは禁止されます。阻害を示すサンプルは、1:10に希釈し、再試験することができる。サンプルが禁止されたままの場合は、そのサンプルを分析から削除する必要があります。
- まず、アッセイの最適温度とプライマー/プローブ濃度の値を求めます。これらのパラメータが PCR 効率(ステップ 3.4.2)、クロス反応性(ステップ 3.4.3)、感度(ステップ 3.4.4)に最適化されたら、多重化された IPC でのアッセイのテストに進みます(ステップ 3.4.5)。
- Situアッセイの開発とテスト
- 研究室で: 実験室で生物へのアクセスだけでなく、共生種が利用可能な場合;これらの種のエンクロージャから水サンプルを採取し、サンプルを処理し、これらのeDNAサンプルに対してアッセイをテストします。上記のように製品を配列し、M13尾プライマーを用いて意図したターゲットの増幅を検証する。
- フィールドで:
- 標的生物が発生することが知られており、発生しないことが知られている部位を特定する。標的種が生じる部位ごとに何らかの量合いを有することが好ましい。
- 使用するサンプル量とサンプル収集方法(例えば、ろ過、遠心分離など)を決定します。
- 各サイトにブランクまたはネガティブコントロールのフィールドを含めて、これは、フィールドサイトに持ち込まれたきれいな水であり、eDNAサンプリング24に使用されるのと同じフィールド機器とプロトコルで収集および準備される。フィールドブランクの目的は、現場に持ち込まれたサンプリング装置やフィールドギアの汚染を検出することです。フィールド水サンプルを処理する前に、フィールドを空白にします。
- サイトごとに複数の水サンプルを採取し、好ましくはサイトあたり3サンプルを採取する。
- 実験室に戻って、サンプルを処理し、抽出する。
- 図5Aに類似したプレートを使用してアッセイを実行し、eDNA濃度と検出頻度を既知の部位の出現と存在量の違いと比較します。24、25をシーケンスですべての検出を確認します。
注:上記は、Thalingerらのレベル4(2020)スケール6( アッセイの技術的性能の最適化)を通じてアッセイを検証し、レベル5のアッセイ検証をサポートするデータの収集を開始します。レベル5は、eDNA生態学研究のための確率モデリングとアッセイの使用を組み込んでいます。基本的なアッセイ開発の範囲を超えていると感じていますが、アッセイの設計とデータ解釈を改善するために、実験室や現場での審査アッセイの応用を奨励しています。
結果
マケット(A.靭帯内)の種特異的なqPCRアッセイを設計する際に、クリンチ川内のすべてのUnionidae種の利用可能な配列がダウンロードされました。ランプシリス・シリコイデアのような密接に関連する種も、同じ川に見つからないにもかかわらず、参照データベースに含まれていました。GenBankでは、関心のある河川系のすべての種が見つかったわけではないので、追加の種が社内で配列されました。配列はGeneiousソフトウェアを使用して整列し、プライマークエスト(IDT)ソフトウェアは複数のアッセイを設計するために使用されました。視覚評価のために5組のプライマーとプローブをアライメントに追加した(図2)。その後、プライマーブラストを使用してシリコでテストされ、その後、彼らはインビトロでさらなるテストのために命じられました.研究室では、全てのアッセイを27種のDNA抽出を用いて試験し、特異性を検証した。1つのアッセイ(A.lig.1)は標的種のみを正常に増幅した(表1;表 2.このアッセイは、アッセイ効率、LODおよびLOQのさらなるテストのために前進しました。それは121塩基対のアンプリコンの長さを有する。表3は、A.靭帯が合成DNA標準に使用する配列を示す。図3Aおよび図3Bは、良好な効率とr2値を有するアッセイが成功した結果を示す。図3Cと図3Dは、標準曲線の効率が悪いアッセイを示しています。このアッセイは破棄されました。選択アッセイ用のLODおよびLOQ(A.lig.1)は、いずれもKlymusら5に記載の離散法を用いて5.00コピー/反応であることが判明した。アッセイで多重化されたIPC(表3-6)は、A.靭帯アッセイの標準曲線に影響を与えなかった。私たちが使用するIPCは、マウスHemTトランスクリプトの断片です。このアッセイは、別のアプリケーション用にIDTによって事前に設計されましたが、ラボのeDNAアプリケーション用のIPCとしての使用を変更しました。
成功したqPCRの実行は、パフォーマンスの各尺度(すなわち、標準の曲線増幅、ゲノムDNA陽性制御、テンプレート制御および内部陽性制御)の一定の基準を満たすべきである。ターゲットアッセイ基準は、指数増幅曲線を有する必要があります。これらの曲線は、十分なサイクルを実行できる場合は、終点の高原に達する必要があります。これは、反応中に蛍光プローブが完全に消費され、蛍光レベルが最大限界に達していることを示しています。後で増幅規格は40サイクルで高原に達しない場合があります。陽性のコントロール(ゲノムDNAとIPC)は同じパターンを持っている必要があります。未知数は増幅するかもしれないし、増幅しないかもしれないが、未知の増幅は指数パターンおよび終点高原を有するべきである(図5)。
品質qPCRでは、標準希釈液は濃度の10倍の差ごとに約3.3サイクルの等間隔のCqで増幅します。標準的な希釈の各反復は、ほぼ同じCq(r2値で表される)を有する緊密にグループ化された方法で増幅する。すべての標準的な希釈は増幅を示すべきである(図3A)。悪くないqPCRでは、規格は非指数形状を示し、希釈間のCq値の不均一な変動、エンドポイント高原に来ない、またはいくつかの希釈物が全く増幅しない可能性がある(図3D)。
標準曲線の重要なパラメータは、効率、r2、傾斜角、および y-切片です。効率は、100%に近い理想的な値で90%から110%の間で低下し、r2値は0.98を超え、理想的な結果は1.0 15、22に近づくはずです。勾配値は -3.2 ~ -3.5 で、理想的な結果は -3.322の近くにある必要があります。y 切片値は 34-41 の Cq の間に入り、37.0 の Cq を有する理想的な結果を示す。y-切片は、ターゲットシーケンスの1コピーでの反応の予測Cqであり、単一のqPCRで測定できる最小単位である。Cqがy-インターセプトより大きい未知数は、阻害される可能性が高い。PCRの40サイクルを超える実行は、阻害または非効率的なプライマーセットの場合にターゲットを検出するために必要な場合がありますが、これらの状況下では定量は不可能であり、標的配列なしで追加の陰性制御は不可能ですが、未知と同様の全DNAを含むが、非特異的な供給源からの増幅を排除するために実行されるべきである。
未知のサンプルにおける内部陽性制御(IPC)増幅は、試薬の競合がなく、阻害剤が存在しないので、陰性テンプレート制御IPCの結果と比較されるべきである。2サイクル以上のCqを有するIPCを有する未知の、または増幅しない、または増幅しないものは、阻害されると考えるべきである。サンプルに阻害剤が存在しない場合、すべての IPC 増幅は、NTC と同じ Cq 値の近くにプロット内の緊密なグループ化を持つ必要があります (図 6)。
最後に、アッセイの検査が行われた。クリンチ川からの20の水サンプルと3つのフィールドブランクサンプルは 、A.靭帯を持つ知られているムール貝のベッドから500メートル以内に2019年9月25-26日の間に濾過されました。約4つの1Lサンプルの水をサンプリング場所ごとに濾過した。ストリームのムール貝ベッドの底に含まれる場所サイト, 海岸近くのムール貝ベッドの底, ストリームでベッドの下流の 100 m, 小川のベッドの下流と海岸近くのベッドの下流 500 m (図 7).研究室に戻ると、各フィルターを半分に切断し、フィルターの半分からDNAを抽出した。各サンプルの残りのフィルター半分を-80°Cの冷凍庫に保管した。次いで、サンプルをIPCで多重化したA.lig.1アッセイを用いて実行した。23個のサンプルのうち、5個が阻害されていることがわかった。これらのサンプルを1:10希釈し、希釈を再実行した。20のフィールドサンプルのうち19個は、設計されたアッセイを使用して増幅した。これらの19個のサンプルのうち、5個はアッセイのLODとLOQの5コピー/反応を上回っていた。つまり、サンプルのほとんどはeDNA検出を持っていましたが、偽陰性の結果が発生する可能性が高く、アッセイがこれらの14個のサンプルのコピー番号を自信を持って定量化できなかったレベルです。それにもかかわらず、4つの生物学的部位の75〜100%が各サンプリング位置で増幅された複製を複製する。3つのフィールドブランクのうち2つは負で、1つのフィールドブランクは増幅を示し、フィールドにおけるクリーンなテクニックの重要性を強調しました。

図1 ミトコンドリアDNA配列データベース構築のワークフローこの図の大きなバージョンを表示するには、ここをクリックしてください。

図2:アクチナイアス靭帯線 ND1アッセイの将来のプライマーとプローブを持つクリンチ川ムール貝種の配列アライメント。濃い緑色のフォワードプライマー、赤でプローブ、ライトグリーンのリバースプライマー。 この図の大きなバージョンを表示するには、ここをクリックしてください。
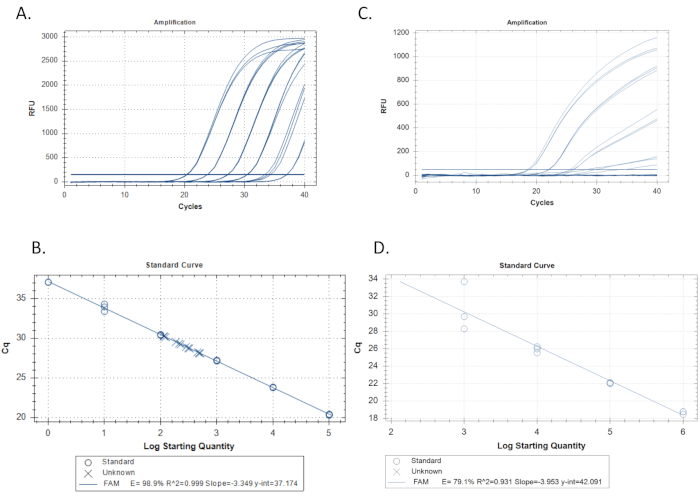
図3:標準曲線と線形回帰の例A.3つの増幅に由来する許容可能な標準曲線の例は、それぞれ6つの標準希釈を複製する。左側の標準濃度が最も高く、濃度が右に移動する10倍の標準希釈シリーズ。すべてのトレースを横切る水平線は、定量時のサイクルのしきい値 (Cq) です。各トレースがこのしきい値を超える場所は、Cq が決定される場所です。図3Aの標準複製から作られた線形回帰。標準希釈の反復は円でプロットされ、未知数(サンプル)はxでプロットされます。効率は98.9%、r2は1.0に近く、斜面は-3.349です。C.3つの増幅に由来する悪い標準曲線の例は、それぞれ6つの標準希釈を複製する。D.標準曲線を形成する線形回帰であり、例3Cで増幅された複製を行う。効率が悪く、r2値に注意してください。また、6規格のうち4規格のみが増幅されることにも注意してください。繰り返し実行した後、標準曲線が改善しない場合、問題は、その場合に期待どおりに標的DNAを増幅しない貧弱なプライマー/プローブセットで、このアッセイは考慮されるべきではありません。この図の大きなバージョンを表示するには、ここをクリックしてください。
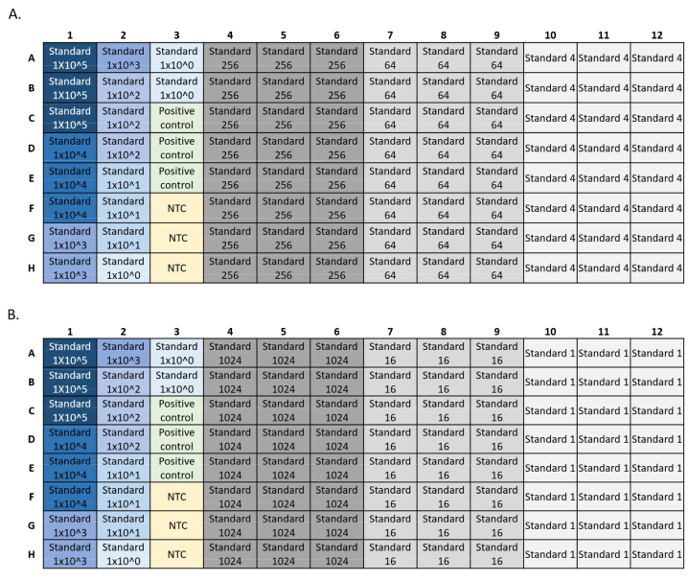
図 4: LOD および LOQ 標準 qPCR 実行のプレート設定の例曲線に使用される標準は青色で、標準濃度は暗い青色から水色に減少します。緑色のDNA陽性コントロールと無テンプレートコントロール(NTC)黄色。各標準希釈について24の反復を示す灰色の実験的標準濃度。希釈シリーズは、標準曲線、正の制御、およびNTCを持つ2つのプレート(A、B)にメッキされました。 この図の大きなバージョンを表示するには、ここをクリックしてください。
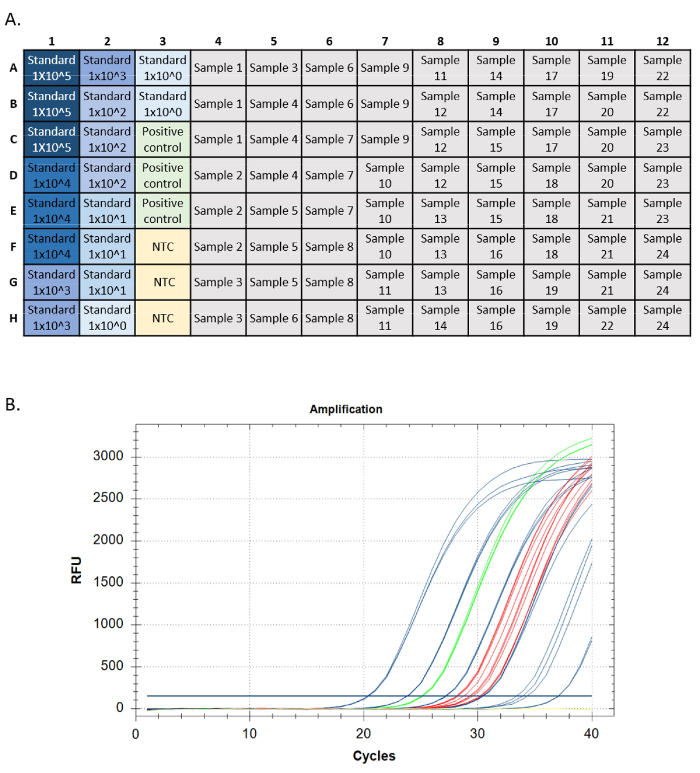
図 5: qPCR 実行からのプレートのセットアップと増幅トレースプレートの設定、標準の最も高い濃度を示す青色、暗い色で示す標準。緑色のDNA陽性コントロール、黄色(NTC)のテンプレートコントロールなし、サンプルターゲットは灰色です。B.qPCR 実行からの増幅トレース。青色、緑色のDNA陽性対照、黄色のテンプレートコントロールなし、赤で未知数で示された標準。この図の大きなバージョンを表示するには、ここをクリックしてください。
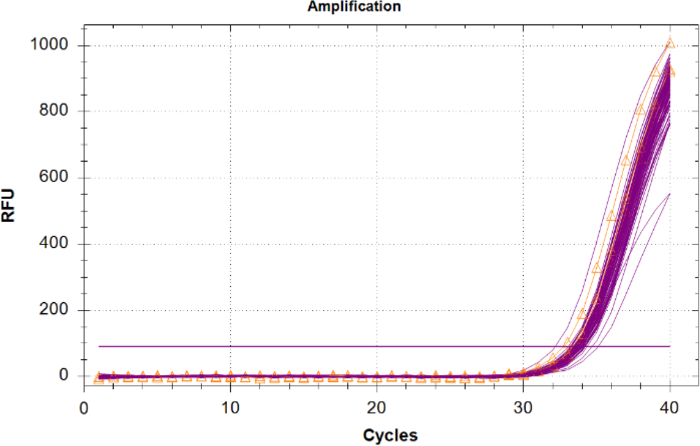
図 6: 内部ポジティブ コントロール (IPC) の増幅トレースマゼンタのすべての未知のサンプルの IPC トレースと、三角形付きのオレンジ色で示されているテンプレートコントロール (NTC) なしからの IPC。 この図の大きなバージョンを表示するには、ここをクリックしてください。
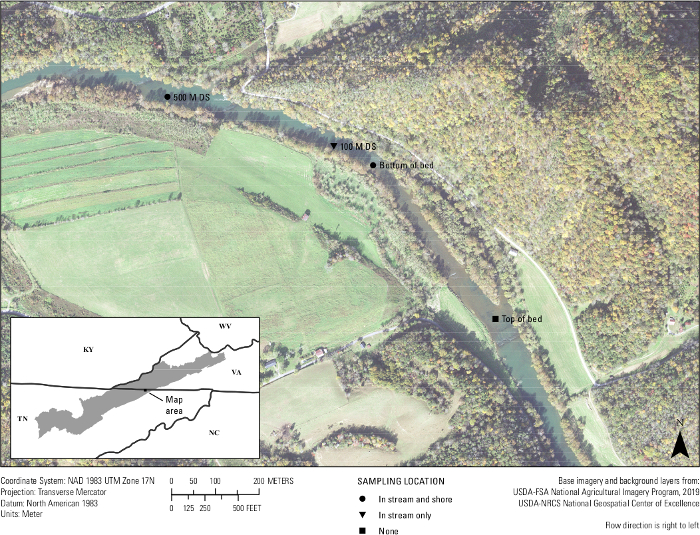
図 7: バージニア州/テネシー州の国境沿いのクリンチ川にあるムール貝のベッドのeDNA採取サイトを示す地図。サンプルは、ベッドの底部、ベッドの下流100m、ベッドの下流500mのウォーレンスベンドで採取した。サイトは、河川の中央(流れ)または海岸線(海岸)からおよそ1〜2メートルで収集されました。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| コンポーネント | 名前 | シーケンス 5' – 3' | 蛍光標識 | |
| フォワードプライマー | A.lig.1-f | CCCTCATCACGTAccTTTAATC | ||
| リバースプライマー | A.lig.1-r | ガガートハッカタータートコッチャッタ | ||
| プローブ | A.lig.1 プローブ | TTCTTGAACGTAAAGCCCCGT | FAM | |
表1:前方および逆プライマーおよびプローブの配列を含む設計された アクチナイアス・靭帯 (A.lig.1)のqPCRアッセイ(A.lig.1)。
| 種 | 増幅 | クリンチ川で |
| 1. アクチナシアナ | はい | はい |
| 2. アクチナ症ペクロサ | いいえ | はい |
| 3. アンブレマ・プリカータ | いいえ | はい |
| 4. コルビキュラのpp. | いいえ | はい |
| 5. カンバーランディア・モノドンタ | いいえ | はい |
| 6. シクロナイアス結核 | いいえ | はい |
| 7. シプロゲニアステガリア | いいえ | はい |
| 8. エリプティオ・ディラタータ | いいえ | はい |
| 9. エピオブラスマブレビデン | いいえ | はい |
| 10. エピオブラスマカプセフォルマ | いいえ | はい |
| 11. エピオブラスマ フロレンティナ アウレオラ | いいえ | はい |
| 12. エピオブラスマトリケトラ | いいえ | はい |
| 13. フスコナヤ・コル | いいえ | はい |
| 14. フスコナイア・サブロタンダ | いいえ | はい |
| 15. ランプシリス・オバタ | いいえ | はい |
| 16. ランプシリス・シリコイデア | いいえ | いいえ |
| 17. ラスミゴナ・コスタタ | いいえ | はい |
| 18. レミオックス・リモサス | いいえ | はい |
| 19. レキシントニア・ドラベルノイド | いいえ | はい |
| 20. メディオンドゥス・コンラディカス | いいえ | はい |
| 21. プレトバサスのシフィウス | いいえ | はい |
| 22. プレウベマプレナム | いいえ | はい |
| 23. プティコブランチス・ファショラリス | いいえ | はい |
| 24. プティコブランチスサブテンタス | いいえ | はい |
| 25. 四重四重膿疱 | いいえ | はい |
| 26. ストロフィトゥス・アンジュラトゥス | いいえ | はい |
| 27. ビロサアイリス | いいえ | はい |
表2:A.lig.1アッセイのインビトロ特異性試験に用いる種のリスト。 アッセイは標的のゲノムDNA(アクチノサイアス・シネシナ)を増幅し、非標的種のいずれも増幅しなかった。
| コンポーネント | シーケンス 5'-3' | ||||
| アクチナアイアス・リゲメンティナ 標準 | CCCTCATCACGTACCTCTTAATTATTTGTCGCATTTTCACTCTTCTTGAACGTA | ||||
| アググアッグツクググGTアクトカアックッチャグアアッグク・カーアアアクト・トゥッガタットタットガッガッグGCATAtTC | |||||
| CCCAACCATTAGCAATGCTCTAAAAAAGAGCTTTTTAAAAGAATGAGTAカッチャクカアッカアッケクト | |||||
| カーアクトアクタックタックトカッテッタハッハカアカアクトクトトゥットッタGCACTACTTTT | |||||
| ガカアットットコッカッテクトッタタンタカタカタチャターサアタントTTTGGTATGCTCCTATTTTTTCT | |||||
| TGTCTCTCCCタグクトクトッタタカアカアクトターガククトガクトガグッカクトカアクッカ | |||||
| アタツグムツタッタグガグクトタッチャッカタッカッカッカッカラッハッタクトクトガグクトタカア | |||||
| タアク | |||||
| IPC テンプレート (ヘム-T) | CTACATAGTACTCTCTCTCTCCAアグクトククテクト・トクトゲクト・カガクトクト | ||||
| GTATGACAGTクトカガクトGCAC | |||||
表3:このアッセイに使用される アクチオナシアシアチナ 標準およびIPCテンプレート(Hem-T)の配列(5'--3')。 前方および逆プライマーの配列は太字と斜体で、プローブのシーケンスには下線が付いています。
| コンポーネント | 名前 | シーケンス 5' – 3' | 蛍光標識 | |
| フォワードプライマー | ヘムト-F | TCTGAGTCCCTCGAATCT | ||
| リバースプライマー | ヘムト-R | GCAGTCCTGCTガガカタガグ | ||
| プローブ | ヘムト-P | ガカクトクトグガーキャットCG | サイ5 | |
表4:内部正極制御(IPC)アッセイは、前方および逆プライマーおよびプローブの配列を含む。
| サンプルあたりの体積(μL) | コンポーネント |
| 10 | 環境マスターミックス |
| 1 | 20uM A. リグ1 F/Rミックス |
| 1 | 2.5uM A. lig.1 プローブ |
| 1 | 5uM IPC プライマーミックス(ヘムト-F/R) |
| 0.75 | 2.5uM IPC プローブ (ヘムト-P) |
| 1.5 | IPC テンプレートの 1 X 103 濃度 |
| 2.75 | H20 |
| 2 | 見本 |
| 20 | 総ボリューム |
表5:IPCアッセイで多重化したA.lig.1アッセイに使用するPCRミックス。
| 歩 | 温度 (°C) | 時間 | |
| 1 | 初期変性 | 95 | 10分 |
| 2 | 変性 | 95 | 15秒 |
| 3 | アニーリング | 60 | 1分 |
| 4 | ステップ 2 に進み、39 倍の時間を繰り返します。 |
表6:A.lig.1アッセイの反応条件。
ディスカッション
他の研究と同様に、対処する質問を定義することは最初のステップであり、eDNAアッセイの設計は研究の範囲に依存する26.例えば、研究または調査の目的が1種または数種を検出することである場合、標的プローブベースのアッセイが最善である。しかし、より大きな組み合えまたは種の集合体を評価することが目標である場合、高スループットシーケンシングメタバーコーディングアッセイの方が適しています。どのアプローチを取る必要が決定されたら、検定の設計、テスト、最適化を含むパイロットスタディを推奨します。24.アッセイの設計は、次の説明に従って種のリストから始まります。 図 1.このリストは、アッセイが特異性と適用される可能性のある地理的範囲の観点から、アッセイがどの程度優れたパフォーマンスを発揮するかを理解するための基礎となります。6,10.特定の地理的領域に対するアッセイを設計し、設計者がその地域の他の種に対する交差反応性のアッセイをより良くテストすることを可能にし、標的種が発生する可能性のある他の地域にアッセイを拡張することに関する限界を認識することが奨励される24.リストが完成すると、パブリック遺伝データベースからシーケンスをダウンロードできます。これらのデータベースは不完全であるため27、アッセイ設計で使用される配列のローカル参照データベースを完了するために、一つは、家の中でリスト上のできるだけ多くの種をシーケンスする必要があります。これらは増幅する最も可能性の高い非標的であるとして、密接に関連する種の共生を優先する。ターゲット種と同じ属または家族内のすべての種に焦点を当てることは、開始する良い場所です。密接に関連する種との比較は、標的種に特有の配列領域を同定するのに役立つ。これは、アッセイが他のシステムまたは場所でどのように実行されるかを知らせるのに役立ちます。ミトコンドリア領域は、より多様な種からのより多くの配列情報が、バーコード・オブ・ライフ・プロジェクトで使用されてきたミトコンドリア遺伝子で入手可能であり、ミトコンドリアDNAが核DNAよりもコピー/細胞にはるかに高い濃度で存在するため、アッセイ開発のための通常の選択である24,28,29.配列カバレッジは、遺伝的リポジトリデータベース内の分類によって異なるため、複数の遺伝子領域をさらなるアッセイ開発について評価する必要があります。この参照シーケンスのローカルデータベースが作成された後、配列配列データとコンピュータソフトウェアプログラムの手動可視化を組み合わせてプライマー/プローブアッセイを設計します。どのアッセイをテストするかを決定するために、ソフトウェアに厳密に依存すべきではありません。プライマーとプローブがターゲットと非ターゲットに座っている位置合わせを視覚的に検証して、PCRでどのように作用するかをよりよく理解することが重要です。最後にアッセイスクリーニングと最適化は、3つのレベル(インシリコ、インビトロおよびinitu)を含む6,7,24,25.シリコの設計とテストは、成功の可能性が高いアッセイの短いリストを生成するために重要ですが、経験的な(インビトロ)テストは、最高の実際のパフォーマンスでアッセイを選択するために重要です。インビトロでの最適化とアッセイのテストには、反応効率の測定とアッセイの感度と特異性の定義が含まれます。検出と定量の限界は、アッセイの開発において見落とされがちであるが、データ解釈にとって重要な2つのパラメータである。アッセイ用の標準曲線の複数の反復を実行することにより、LODとLOQを簡単に測定することができます1,5,30.アッセイのLODまたはLOQに関する結果について議論する研究はほとんどありませんが、Senguptaら(2019)は、アッセイのLODとLOQをデータ解釈とグラフィックスに組み込み、結果を明確に理解します。31.内部の肯定的な制御は、同様に設計されたアッセイに多重化されるべきである。サンプル内の PCR 阻害をテストしないと、偽陰性が発生する可能性があります。24,32.PCR阻害試験の最も簡単な方法として、標的アッセイによる多重化IPCアッセイの使用を提案する23.最後に、現場および実験室から採取したサンプルからのアッセイの現場試験では、環境試料中でターゲット増幅が確実に起こる必要がある24.
eDNAサンプルを用いた種特異的なプローブベースのqPCRアッセイの使用には制限があります。例えば、テスト用の複数のアッセイの設計は、配列の可用性によって制限され、アッセイ性能の面で妥協が必要な場合があります。これらの選択は、研究の目標によって導かれ、結果26と報告されなければならない。例えば、希少種の検出が目標であり、陽性が少ない場合、すべての検出がシーケンシングによって検証されるならば、不完全な特異性(すなわち、非標的種の増幅)を伴うアッセイを使用することができる。目標が種の地理的範囲を監視する場合、eDNA濃度データが不要な場合、不完全な効率を持つアッセイを使用し、データはパーセント検出としてのみ報告されます。さらに、可能な限り不可能な実験室ですべての潜在的な特異性がテストされない限り、アッセイの真の特異性を絶対的な確実性をもって知ることはできません。例えば、アッセイはクリンチ川のいくつかの淡水ムール貝種に対して設計され、テストされました。このアッセイを別の河川システムで使用するには、新しい場所の種のスイートに対してテストする必要があります。アッセイの発生中に試験されない種または集団内の遺伝的変異も特異性に影響を及ぼす可能性がある。最後に、アッセイが高い技術的性能を有することが確認された場合でも。フィールドで作業すると、条件が変化します。水流、pH、動物の挙動などの非アッセイ関連条件は、異なるeDNA採取および抽出プロトコルを使用できるようにeDNA検出可能性を変化させる可能性があります。最適化され、よく説明されたアッセイを使用すると、そのようなパラメータがeDNA検出に及ぼす影響を理解しやすくなります。
eDNAの分野は、探索的分析の段階を超えて、方法や技術の標準化を高める段階で成熟しています。これらの開発により、eDNAの技術、能力、および制限に関する理解が深まりつつあります。上記の最適化プロセスにより、アッセイの感度、特異性、再現性が向上します。eDNA法の改良と標準化の最終目標は、eDNAデータに基づいて推論を行う研究者の能力を向上させるとともに、結果に対するエンドユーザーとステークホルダーの信頼を高めることです。
開示事項
著者らは利益相反を宣言しない。資金提供者は、研究の設計に何の役割も持っていなかった。データの収集、分析、または解釈に含まれる。原稿の執筆に。または結果を公開する決定に含まれます。
謝辞
私たちは、プライマーの開発とテストを支援したアルヴィ・ワドゥドとトルディ・フロストに感謝します。本研究で報告されたアッセイ設計のための資金は、国防総省戦略環境研究開発プログラム(RC19-1156)によって提供されました。貿易、製品、または会社名の使用は記述目的のものであり、米国政府による承認を意味するものではありません。この調査中に生成されたデータは、USGS データ・リリース https://doi.org/10.5066/P9BIGOS5 として入手できます。
資料
| Name | Company | Catalog Number | Comments |
| 96 Place Reversible Racks with Covers | Globe Scientific | 456355AST | |
| Clean gloves (ie. latex, nitrile, etc.) | Kimberly-Clark | 43431, 55090 | |
| CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855196 | |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 5408129 | |
| Fisherbrand Premium Microcentrifuge Tubes: 2.0mL | Fisher Scientific | 2681332 | |
| Hard-Shell 96-Well PCR Plates, low profile, thin wall, skirted, white/clear | Bio-Rad | #HSP9601 | |
| IPC forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| IPC PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| IPC Ultramer DNA Oligo synthetic template | Integrated DNA Technologies, Inc. | none | custom product |
| Labnet MPS 1000 Compact Mini Plate Spinner Centrifuge for PCR Plates | Labnet | C1000 | |
| Microcentrifuge machine | Various | - | Any microcentrifuge machine that hold 1.5mL and 2.0mL tubes is typically okay. |
| Microseal 'B' PCR Plate Sealing Film, adhesive, optical | Bio-Rad | MSB1001 | |
| Nuclease-Free Water (not DEPC-Treated) | Invitrogen | AM9932 | |
| Pipette Tips GP LTS 1000 µL F 768A/8 | Rainin | 30389272 | |
| Pipette Tips GP LTS 20 µL F 960A/10 | Rainin | 30389274 | |
| Pipette Tips GP LTS 200 µL F 960A/10 | Rainin | 30389276 | |
| Pipettes | Rainin | Various | Depending on lab preference, manual or electronic pipettes can be used at various maximum volumes. |
| TaqMan Environmental Master Mix 2.0 | Thermo Fisher Scientific | 4396838 | |
| Target forward and reverse primers | Integrated DNA Technologies, Inc. | none | custom product |
| Target PrimeTime qPCR Probes | Integrated DNA Technologies, Inc. | none | custom product |
| Target synthetic gBlock gene fragment | Integrated DNA Technologies, Inc. | none | custom product. used for qPCR standard dilution series |
| TE Buffer | Invitrogen | AM9849 | |
| VORTEX-GENIE 2 VORTEX MIXER | Fisher Scientific | 50728002 |
参考文献
- Kubista, M., et al. The real-time polymerase chain reaction. Mol Aspects Med. 27 (2-3), 95-125 (2006).
- Higuchi, R. D., Walsh, P. S., Griffith, R. Simultaneous amplification and detection of specific DNA sequences. Biotechnology. 10, 5(1992).
- Mauvisseau, Q., et al. Influence of accuracy, repeatability and detection probability in the reliability of species-specific eDNA based approaches. Scientific Reports. 9 (1), 580(2019).
- Hernandez, C., et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA. , (2020).
- Klymus, K. E., et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA. , (2019).
- Thalinger, B., et al. A validation scale to determine the readiness of environmental DNA assays for routine species monitoring. bioRxiv. , (2020).
- Helbing, C. C., Hobbs, J. Environmental DNA Standardization Needs for Fish and Wildlife Population Assessments and Monitoring. CSA Group. , (2019).
- Sepulveda, A. J., Nelson, N. M., Jerde, C. L., Luikart, G. Are Environmental DNA Methods Ready for Aquatic Invasive Species Management. Trends in Ecology & Evolution. , (2020).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Wilcox, T. M., et al. Robust detection of rare species using environmental DNA: the importance of primer specificity. PLoS One. 8 (3), 59520(2013).
- Prediger, E. How to design primers and probes for PCR and qPCR. IDT. , Available from: http://www.idtdna.cco/pages/education/decoded/article/designing-pcr-primers-and-probes (2020).
- Thornton, B., Basu, C. Real-time PCR (qPCR) primer design using free online software. Biochemistry and Molecular Biology Education. 39, 145-154 (2011).
- Owczarzy, R., et al. IDT SciTools: a suite for analysis and design of nucleic acid oligomers. Nucleic Acids Research. 36, Web Server issue 163-169 (2008).
- Kibbe, W. A. OligoCalc: an online oligonucleotide properties calculator. Nucleic Acids Research. 35, Web Server issue 43-46 (2007).
- Taylor, S. C., et al. The Ultimate qPCR Experiment: Producing Publication Quality, Reproducible Data the First Time. Trends in Biotechnology. 37 (7), 761-774 (2019).
- Ye, J., et al. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13 (134), 11(2012).
- Altschul, S. F., Gish, W., Miller, W., Myers, E. W., Lipman, D. J. Basic Local Alignment Search Tool. Journal of Molecular Biology. 215, 403-410 (1990).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Bio-Rad. Bio-Rad Vol. 5279. , ed Bio-Rad (2020).
- Bio-Rad. Bio-Rad Vol. 6894. , Bio-Rad (2020).
- Eurogentec. Eurogentec. Vol. 0708-V2. , ed Eurogentec (2020).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Hoorfar, J., et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 42 (5), 1863-1868 (2004).
- Goldberg, C. S., et al. Critical considerations for the application of environmental DNA methods to detect aquatic species. Methods in Ecology and Evolution. 7 (11), 1299-1307 (2016).
- Guan, X., et al. Environmental DNA (eDNA) Assays for Invasive Populations of Black Carp in North America. Transactions of the American Fisheries Society. 148 (6), 1043-1055 (2019).
- Mosher, B. A., et al. Successful molecular detection studies require clear communication among diverse research partners. Frontiers in Ecology and the Environment. 18 (1), 43-51 (2019).
- Kwonga, S., Srivathsana, A., Meier, R. An update on DNA barcoding: low species coverage and numerous unidentified sequences. Cladistics. 28, 6(2012).
- Rees, H. C., et al. REVIEW: The detection of aquatic animal species using environmental DNA - a review of eDNA as a survey tool in ecology. Journal of Applied Ecology. 51 (5), 1450-1459 (2014).
- Evans, N. T., Lamberti, G. A. Freshwater fisheries assessment using environmental DNA: A primer on the method, its potential, and shortcomings as a conservation tool. Fisheries Research. 197, 60-66 (2018).
- Forootan, A., et al. Methods to determine limit of detection and limit of quantification in quantitative real-time PCR (qPCR). Biomolecular Detection and Quantification. 12, 1-6 (2017).
- Sengupta, M. E., et al. Environmental DNA for improved detection and environmental surveillance of schistosomiasis. Proceedings of the National Academy of Sciences of the United States of America. 116 (18), 8931-8940 (2019).
- Klymus, K. E., Richter, C. A., Chapman, D. C., Paukert, C. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. 183, 77-84 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved