Method Article
تقييم تأثير التكسير الهيدروليكي على الجداول باستخدام التوقيعات الجزيئية الميكروبية
In This Article
Summary
هنا، نقدم بروتوكولا للتحقيق في آثار التكسير الهيدروليكي على الجداول القريبة من خلال تحليل مجتمعاتهم الميكروبية للمياه والرواسب.
Abstract
يستخدم التكسير الهيدروليكي (HF)، الذي يطلق عليه عادة "التكسير"، مزيجا من الماء عالي الضغط والرمل والمواد الكيميائية لكسر الصخور وإطلاق النفط والغاز. أحدثت هذه العملية ثورة في صناعة الطاقة في الولايات المتحدة، حيث أنها تتيح الوصول إلى الموارد التي لم يكن من الممكن الحصول عليها من قبل وتنتج الآن ثلثي إجمالي الغاز الطبيعي في الولايات المتحدة. على الرغم من أن التكسير كان له تأثير إيجابي على الاقتصاد الأمريكي، إلا أن العديد من الدراسات سلطت الضوء على آثاره البيئية الضارة. ومما يثير القلق بوجه خاص تأثير التكسير على مجاري مياه الرأس، التي لها أهمية خاصة بسبب تأثيرها الكبير بشكل غير متناسب على صحة مستجمعات المياه بأكملها. يمكن استخدام البكتيريا داخل تلك الجداول كمؤشرات على صحة التيار ، حيث من المتوقع أن تختلف البكتيريا الموجودة ووفرتها في تيار مضطرب عن تلك الموجودة في تيار قابل للمقارنة ولكن دون عائق. لذلك ، يهدف هذا البروتوكول إلى استخدام المجتمع البكتيري لتحديد ما إذا كانت الجداول قد تأثرت بالتكسير. وتحقيقا لهذه الغاية، يجب جمع عينات من الرواسب والمياه، من تيارات قريبة من التكسير (يحتمل أن تتأثر) ومن المنبع أو في مستجمعات مياه مختلفة من نشاط التكسير (دون أية أهمية). ثم تخضع تلك العينات لاستخراج الحمض النووي، وإعداد المكتبة، والتسلسل للتحقيق في تكوين المجتمع الميكروبي. ويمكن بعد ذلك استخدام نماذج التحليل الارتباطي والتعلم الآلي لتحديد السمات التي هي مخططة للتباين في المجتمع، فضلا عن تحديد المؤشرات الحيوية التنبؤية لتأثير التكسير. ويمكن لهذه الأساليب أن تكشف عن مجموعة متنوعة من الاختلافات في المجتمعات الميكروبية بين مجاري مياه الرأس، على أساس القرب من التكسير، وأن تكون بمثابة أساس للتحقيقات المستقبلية بشأن الأثر البيئي لأنشطة التكسير.
Introduction
التكسير الهيدروليكي (HF)، أو "التكسير"، هو وسيلة لاستخراج الغاز الطبيعي، والتي أصبحت سائدة بشكل متزايد مع استمرار ارتفاع الطلب على الوقود الأحفوري. وتتكون هذه التقنية من استخدام معدات الحفر عالية الطاقة لحقن مزيج من الماء والرمل والمواد الكيميائية في رواسب الصخر الزيتي الغنية بالميثان، وعادة لإطلاق الغازات المحاصرة1.
ولأن تقنيات الحصاد غير التقليدية هذه جديدة نسبيا، فمن المهم التحقيق في آثار هذه الممارسات على الممرات المائية القريبة. وتفويض أنشطة التكسير بتطهير مساحات كبيرة من الأراضي لنقل المعدات وبناء منصة الآبار. يجب تطهير ما يقرب من 1.2-1.7 هكتار من الأراضي لكل منصة بئر2، مما قد يؤثر على الجريان السطحي وجودة المياه في النظام3. هناك نقص في الشفافية المحيطة بالتركيب الكيميائي الدقيق لسائل التكسير ، بما في ذلك المبيدات الحيوية المستخدمة. بالإضافة إلى ذلك ، تميل مياه الصرف الصحي التكسيرية إلى أن تكون ملحية للغاية2. وعلاوة على ذلك، قد تحتوي مياه الصرف الصحي على معادن ومواد مشعة تحدث بشكل طبيعي2. ولذلك، فإن احتمال حدوث تسربات وانسكابات من سائل التكسير بسبب خطأ بشري أو عطل في المعدات أمر مثير للقلق.
ومن المعروف أن النظم الإيكولوجية للجداول حساسة جدا للتغيرات في المناظر الطبيعية المحيطة4 وهي مهمة للحفاظ على التنوع البيولوجي5 وركوب الدراجات المغذية المناسبة6 داخل مستجمعات المياه بأكملها. الميكروبات هي الكائنات الأكثر وفرة في مجاري المياه العذبة، وبالتالي، ضرورية لركوب الدراجات المغذيات، والتحلل البيولوجي، والإنتاج الأولي. تكوين المجتمع الميكروبي ووظيفته بمثابة أدوات كبيرة للحصول على معلومات عن النظام البيئي بسبب حساسيتها للاضطرابات ، وأظهرت الأبحاث الحديثة تحولات متميزة في التجمعات البكتيرية الملاحظة على أساس القرب من نشاط التكسير7،8. على سبيل المثال، Beijerinckia، Burkholderia، و Methanobacterium تم تحديدها على أنها غنية في تيارات بالقرب من التكسير في حين Pseudonocardia، Nitrospira، وRhdobacter تم إثراء في تيارات لا بالقرب من التكسير7.
الجيل القادم من تسلسل الجين RNA ريبوسومال 16S (rRNA) هو وسيلة بأسعار معقولة لتحديد تكوين المجتمع البكتيري الذي هو أسرع وأرخص من تسلسل الجينوم كله النهج9. ممارسة شائعة في مجال الإيكولوجيا الجزيئية هو استخدام منطقة V4 متغير للغاية من الجين 16S rRNA لتسلسل القرار، وغالبا وصولا الى مستوى جنس مع نطاق واسع من تحديد9،كما أنها مثالية لعينات بيئية لا يمكن التنبؤ بها. وقد تم تنفيذ هذه التقنية على نطاق واسع في الدراسات المنشورة واستخدمت بنجاح لتحديد تأثير عمليات التكسير على البيئات المائية7،8. ومع ذلك ، تجدر الإشارة إلى أن البكتيريا لديها أرقام نسخ مختلفة من جين 16S rRNA ، مما يؤثر على وفرة المكتشفة10. هناك عدد قليل من الأدوات لحساب هذا، ولكن فعاليتها مشكوك فيها10. ممارسة أخرى تنمو بسرعة في الانتشار وتفتقر إلى هذا الضعف هي التسلسل الميتاترانسكريبتومي ، حيث يتم تسلسل جميع الحمض النووي الريبي ، مما يسمح للباحثين بتحديد البكتيريا النشطة والتعبير عن جيناتهم.
لذلك ، على النقيض من الأساليب في الدراسات المنشورة سابقا7،8،11،12، يغطي هذا البروتوكول أيضا جمع العينات والحفاظ عليها ومعالجتها وتحليلها للتحقيق في وظيفة المجتمع الميكروبي (metatranscriptomics). تسمح الخطوات المفصلة هنا للباحثين برؤية تأثير التكسير، إن وجد، على الجينات والمسارات التي تعبر عنها الميكروبات في تياراتها، بما في ذلك جينات مقاومة مضادات الميكروبات. وعلاوة على ذلك، تم تحسين مستوى التفاصيل المقدمة لجمع العينات. على الرغم من أن العديد من الخطوات والملاحظات قد تبدو واضحة للباحثين ذوي الخبرة ، إلا أنها يمكن أن تكون لا تقدر بثمن لأولئك الذين بدأوا الأبحاث للتو.
هنا، نقوم بوصف طرق جمع العينات ومعالجتها لتوليد البيانات الوراثية البكتيرية كوسيلة للتحقيق في تأثير التكسير على الجداول القريبة استنادا إلى خبرة مختبراتنا التي تبلغ عدة سنوات. يمكن استخدام هذه البيانات في تطبيقات المصب لتحديد الاختلافات المقابلة لحالة التكسير.
Protocol
1. جمع عينات الرواسب لاستخراج الحمض النووي
- غمر أنبوب مخروطي معقم سعة 50 مل في مياه المجرى. ارتداء قفازات أثناء جمع العينات لتجنب إدخال التلوث البشري غير المرغوب فيه. تنفيذ هذه الخطوة إما من الشاطئ أو تواجه المنبع إذا كان في الماء.
- بينما يتم غمر الأنبوب المخروطي، قم بإزالة الغطاء، واستخدمه لاستخراج ما يقرب من 3 مل من الرواسب من عمق 1 إلى 3 سم في الأنبوب المخروطي.
- إزالة أنبوب مخروطي من الماء وتفريغ جميع المياه، باستثناء طبقة رقيقة تغطي عينة الرواسب (حوالي 1 مل).
- باستخدام ماصة 1000 ميكرولتر ونصائح ماصة مناسبة ، أضف 3 مل من المواد الحافظة للحمض النووي / الحمض النووي الريبي (انظر جدول المواد لمواصفات المواد الحافظة) إلى العينة التي تم جمعها. الحفاظ على نصائح ماصة في مربع تلميح ماصة معقمة وإرفاقها فقط مباشرة قبل الاستخدام والتخلص منها بعد الاستخدام. عكس أنبوب مخروطي توج 10 مرات لضمان المواد الحافظة وعينة مختلطة تماما.
ملاحظة الخطوة 1.4 غير ضرورية، ولكن يوصى بشدة إذا كان الحمض النووي الريبي هو أن يستخرج من الرواسب في وقت لاحق. - ضع العينات على الجليد لبقية جمع العينات. عند العودة من جمع، وتخزينها في الثلاجة في -20 درجة مئوية إذا كانت العينات لاستخدامها لتحليل 16S (الحمض النووي)، أو -70 درجة مئوية، إذا كان لاستخدامها لتحليل metatranscriptomics (الجيش الملكي النيبالي).
2. تصفية جمع لاستخراج الحمض النووي
- إزالة غطاء زجاجة 1 لتر معقمة. أثناء مواجهة المنبع أو من الشاطئ ، املأ الزجاجة بمياه التيار إلى الأعلى ثم قم بالتخلص منها. كرر هذه العملية مرتين أخريين لتكرار الزجاجة. ملء زجاجة كاملة للمرة الرابعة وسقف عليه.
ملاحظة: إذا إعادة استخدام زجاجة 1 لتر، يمكن تعقيمها عن طريق الشطف مع تبييض 10٪ لمدة 2 دقيقة، تليها الشطف ثلاث مرات مع الماء deionized ثم مرة واحدة مع الإيثانول 70٪، وأخيرا autoclaving مع إعدادات: 30 دقيقة وقت التعرض في 121.1 درجة مئوية و 15 دقيقة وقت التجفيف. أثناء الالاستعباد التلقائي ، يجب أن يكون الغطاء على الزجاجة فضفاضا جدا لتجنب ضغط الزجاجة في هذه العملية. - مرة واحدة على سطح مستقر، واستخدام حقنة قفل لوير معقمة ووضع حجم كامل. ثم قم بتوصيل المحاقن بمصفاة بولي إيثرسولفون معقمة وخالية من الحمض النووي/الحمض النووي الريبي قطرها 1.7 سم بحجم مسام 0.22 ميكرومتر ودفع مستوى الصوت بأكمله من خلال الفلتر عن طريق الضغط على المكبس على طول الطريق لأسفل. كرر هذه العملية حتى يتم دفع إجمالي حجم جمعها في زجاجة (1 لتر) من خلال مرشح.
ملاحظة: يمكن أن يكون حجم المحاقن متغيرا، إذا تم تعقب إجمالي كمية المياه التي يتم دفعها عبر الفلتر. ومع ذلك، عموما، يفضل 60 مل. في حين أن 1 L مثالي ، على سبيل السرد ، فإن حجم لا يقل عن 200 مل من المرجح أن لا يزال يجمع ما يكفي من الكتلة الحيوية (على افتراض ~ 20000 خلية لكل مل) لاستخراج الحمض النووي والجيش الملكي النيبالي. - إزالة المياه الزائدة من مرشح عن طريق رسم ما يقرب من 20 مل قيمة الهواء في الحقنة ودفعه من خلال مرشح.
ملاحظة: سيساعد هذا في منع فقدان المواد الحافظة إذا تم تنفيذ الخطوة 2.4. - باستخدام P1000 micropipette، إضافة 2 مل من الحمض النووي / الحمض النووي الريبي الحافظة عن طريق تفريغه من خلال فتحة أكبر للمرشح (حيث تم إرفاقه إلى الحقنة) في حين عقد مرشح أفقيا. يجب أن يكون طرف المصصة داخل برميل الفلتر عندما يكون المصصة مكتئبا لضمان دخول المادة الحافظة إلى الفلتر. تغيير تلميح بعد كل استخدام.
ملاحظة: كما هو الحال مع جمع الرواسب، هذه الخطوة ليست ضرورية، ولكن ينصح بشدة لزيادة غلة حمض النوى في وقت لاحق، وخاصة بالنسبة للجيش الملكي النيبالي. - قشر قبالة مربع واحد من فيلم البارافين والتفاف عليه بإحكام حول كل افتتاح / نهاية للمرشح لختم. ضع فيلم البارافين ملفوفا في كيس عينة معقم ثم ضع الكيس بأكمله على الثلج أثناء التجميع.
ملاحظة: تأكد من أن الجانب المستخدم في لف الفلتر عقيم، أي أنه لم يتعرض للبيئة من قبل. - عند العودة من أخذ العينات، تخزن المرشحات عند -20 درجة مئوية ل 16S أو -70 درجة مئوية لبيئة النسخ الفوقية.
3. استخراج الحمض النووي وتحديد كمي
- تنظيف منطقة العمل مع 10٪ التبييض و 70٪ الإيثانول قبل البدء في نقل العينة.
- بالنسبة للرواسب (من الخطوة 1.5)، استخدم بشكل عام ~ 0.25 غرام من العينة. اللهب تعقيم أداة معدنية عن طريق غمسه في كوب من الإيثانول 70٪ وحرق الإيثانول قبالة بين العينات.
- بالنسبة للفلاتر (من الخطوة 2.6)، قم بنقل ورق التصفية إلى أنبوب معقم لاستخراجه. للقيام بذلك اتبع الخطوات التالية.
- إنشاء معقمة، الحمض النووي والجيش الملكي النيبالي السطح الحر عن طريق طي رقائق الألومنيوم بحيث لا يتعرض الجزء الداخلي من حظيرة للبيئة الخارجية وautclaving قطعة مطوية مع الإعدادات: 121.1 درجة مئوية و 5 دقائق وقت التجفيف.
- تعقيم قبضة فيس مع الإيثانول 70٪ ولهب مفتوح. ثم استخدم قبضة اللزوجة لكسر فتح غلاف الفلتر على السطح العقيم وإزالة اللب من الغلاف.
- استخدام مشرط عقيمة لقطع ورقة تصفية بعيدا عن جوهر عن طريق تشريح في أعلى وأسفل ومن ثم على طول التماس. قم بطي ورق الفلتر باستخدام ملاقط معقمة ثم اقطع الفلتر إلى قطع صغيرة باستخدام المشرط.
- ضع قطع الفلتر في أنبوب طرد مركزي صغير لاستخراجه. تأكد من أن ورقة التصفية لا تلامس أي أسطح غير معقمة أو يمكن أن تحتوي على حمض نووي موجود ، لأن هذا سيؤدي إلى تلوث غير مرغوب فيه للعينة.
- إجراء عزل الحمض النووي كما هو موضح سابقا13 أو باستخدام مجموعة متوفرة تجاريا المستندة إلى العمود (انظر جدول المواد). وترد أدناه وصفا موجزا لخطوات المجموعة التجارية المدرجة.
- Lyse الخلايا داخل العينة عن طريق نقله إلى أنبوب حبة وإخضاعه لتعطيل الخلية بسرعة عالية لمدة 5 دقائق على الأقل. الطرد المركزي ونقل supernatant إلى أنبوب الطرد المركزي الدقيق العقيمة.
- إضافة تحلل المخزن المؤقت إلى supernatant (1:1 وحدة التخزين) ونقل إلى عامل التصفية المتوفرة (الأصفر). طرد مركزي للمرشح.
- نقل المرشح إلى أنبوب طرد مركزي صغير معقم جديد. أضف المخزن المؤقت للإعداد (400 ميكرولتر) والطرد المركزي وتجاهل التدفق من خلاله.
- أضف مخزن الغسيل المؤقت (700 ميكرولتر) والطرد المركزي وتجاهل التدفق من خلاله. ثم أضف المخزن المؤقت للغسيل (400 ميكرولتر) والطرد المركزي وتجاهل التدفق مرة أخرى.
- نقل المرشح إلى أنبوب طرد مركزي صغير معقم جديد. Elute مع 50 ميكرولتر من المياه الحرة DNase / RNase والسماح للجلوس لمدة 5 دقائق في درجة حرارة الغرفة قبل الطرد المركزي.
- خلال تلك الفترة في التكعيب، وإعداد مرشح الثالث-HRC عن طريق وضعه في أنبوب جمع وإضافة الحل الإعدادية مجلس حقوق الإنسان (600 ميكرولتر) إليها، تليها خطوة الطرد المركزي من 3 دقائق في 8000 × ز.
- حرك الفلتر المعد إلى أنبوب طرد دقيق معقم. نقل الحمض النووي eluted من الخطوة 3.4.5 لهذا المرشح والطرد المركزي في 16000 س ز لمدة 3 دقائق. التدفق من خلال يحتوي على الحمض النووي المستخرج.
- تخزين مستخلصات الحمض النووي لكل من الرواسب والمرشحات في -20 درجة مئوية.
ملاحظة: يمكن تخزين مستخلصات الحمض النووي لمدة 8 سنوات تقريبا عند -20 درجة مئوية على افتراض درجة حرارة مستقرة ، والتعرض المحدود للضوء ، وعدم وجود ملوثات ضارة14. - تنفيذ عزل الحمض النووي الريبي وفقا لبروتوكول الشركة المصنعة. تخزين مقتطفات الحمض النووي الريبي في -80 درجة مئوية.
- Lyse الخلايا داخل العينة عن طريق نقله إلى أنبوب حبة وإخضاعه لتعطيل الخلية بسرعة عالية لمدة خمس دقائق على الأقل. الطرد المركزي ونقل supernatant إلى أنبوب الطرد المركزي الدقيق العقيمة.
- إضافة مخزن تحلل المؤقت إلى supernatant (1:1 وحدة التخزين) ونقل إلى العمود المتوفر (الأصفر). طرد مركزي للعمود.
- إضافة حجم متساو من الإيثانول 95-100٪ إلى تدفق من خلال وخلط عن طريق pipetting صعودا وهبوطا خمس مرات.
- ضع عمود IICG (أخضر) على أنبوب طرد مركزي دقيق معقم. نقل الحل المختلط إلى العمود والطرد المركزي.
- أضف مخزن الغسيل المؤقت (400 ميكرولتر) والطرد المركزي وتجاهل التدفق من خلاله.
- أضف 5 ميكرولتر من DNase I و 75 ميكرولتر من مخزن هضم الحمض النووي إلى العمود واحتضنه في درجة حرارة الغرفة لمدة 15 دقيقة.
- إضافة المخزن المؤقت الإعدادية (400 ميكرولتر) ، الطرد المركزي ، وتجاهل تدفق من خلال.
- أضف مخزن الغسيل المؤقت (700 ميكرولتر) والطرد المركزي وتجاهل التدفق من خلاله. ثم أضف المخزن المؤقت للغسيل (400 ميكرولتر) والطرد المركزي وتجاهل التدفق مرة أخرى.
- نقل العمود إلى أنبوب طرد مركزي صغير معقم جديد. Elute مع 50 ميكرولتر من DNase / RNase المياه الحرة والسماح للجلوس لمدة 5 دقائق قبل الطرد المركزي.
- خلال فترة الحضانة تلك، قم بإعداد فلتر III-HRC عن طريق وضعه في أنبوب تجميع وإضافة محلول إعداد HRC (600 ميكرولتر) إليه، تليها خطوة طرد مركزي من 3 دقائق عند 8000 × ز.
- حرك الفلتر المعد إلى أنبوب طرد دقيق معقم. نقل الحمض النووي الريبي من الخطوة 3.6.9 إلى هذا المرشح والطرد المركزي في 16000 x ز لمدة 3 دقائق. التدفق من خلال يحتوي على الحمض النووي الريبي المستخرج.
ملاحظة: يمكن تخزين مستخلصات الحمض النووي الريبي لمدة عام واحد فقط قبل أن تبدأ في التحلل15. تتحلل كل من مستخلصات الحمض النووي والجيش الملكي النيبالي عن طريق تكرار ذوبان الجليد. تسمح بعض البروتوكولات لاستخراج كل من الحمض النووي والحمض النووي الريبي من نفس العينة16،17.
- قياس عينات الحمض النووي الحمض النووي الحمض النووي الريبي المستخرج باستخدام مقياس الفلور أو مطياف. انظر الجدول 1 على سبيل المثال قيم تركيز الحمض النووي للفلورومتر. للحصول على مثال بروتوكول قياس كمي مطياف، راجع المرجع18. تتراوح قيم تركيز الحمض النووي للرواسب مع المجموعة المدرجة في جدول المواد بشكل عام من 1 إلى 40 نانوغرام/ميكرولتر، في حين أن قيم تركيز الحمض النووي المرشح تميل إلى أن تتراوح بين 0.5 إلى 10 نانوغرام/ميكرولتر. تتراوح عادة من 0.5 إلى 5 نانوغرام / ميكرولتر.
4. 16S rRNA إنشاء مكتبة
- تنظيف منطقة العمل مع 10٪ التبييض و 70٪ الإيثانول. يجب أن تكون منطقة العمل مساحة مغلقة قادرة على إنتاج ظروف تدفق صفح (غطاء تدفق صفح).
- استخدم مستخلصات الحمض النووي (من الخطوة 3.5) واستعد عينات لتسلسل 16S rRNA amplicon مع بروتوكول PCR قياسي ، مثل البروتوكول الموصوف على موقع Microbiome للأرض الذي يضخم منطقة V4 شديدة التباين من 16S rRNA19 في ظل ظروف تدفق صفح.
- إعداد هلام agarose 2٪ كما هو موضح سابقا والسماح لها ترسيخ17. مزيج 7 ميكرولتر من منتج PCR و 13 ميكرولتر من المياه الخالية من DNase. إضافة صبغة تحميل هلام إلى تركيز النهائي من 1x. مرة واحدة يتم ترسيخ agarose، تحميل هذا مزيج منتجات PCR على هلام agarose 2٪.
ملاحظة: بدلا من ذلك، يمكن استخدام هلام ما قبل الزهر بدلا من ذلك، حيث تعمل هذه المواد الهلامية بشكل أسرع وتأتي مسبقة الصنع. - تشغيل هلام في 90 V لمدة 60-90 دقيقة للتحقق من حجم الفرقة من 386 كما تضخيم ناجحة ل16S rRNA V4 amplicons، وذلك باستخدام بروتوكول ميكروبيوم الأرض.
5. تنقية مكتبة الحمض النووي 16S rRNA
- تجمع 10 ميكرولتر من منتجات PCR للعينات التي أسفرت عن عصابات مشرق و 13 ميكرولتر للعينات التي أسفرت عن نطاقات خافتة في أنبوب الطرد المركزي الدقيق المعقمة الحجم المناسب.
- تحقق من تركيز بركة الناتجة باستخدام مقياس الفلور أو مطياف وإعداد هلام agarose 2٪ كما كان من قبل. ومن الناحية المثالية، ينبغي أن يكون تركيز المجمع لا يقل عن 10 نانوغرام/ميكرولتر، وكان ينبغي أن يكون تركيز معظم العينات حوالي 25 نانوغرام/ميكرولتر.
- التركيز وحجم السماح، وتحميل حوالي 150-200 نانوغرام في بئر من هلام agarose 2٪.
- تشغيل هلام لمدة 60-90 دقيقة في 90 فولت.
- تنقية المكتبة المجمعة عن طريق تشغيل هلام agarose 2٪.
- استئصال الفرقة الحمض النووي 386 bp من هلام وتنقية المكتبة المجمعة باستخدام عدة المتاحة تجاريا كما هو موضح سابقا20. Elute الحمض النووي المنقى مع 30 ميكرولتر من 10 mM تريس-Cl (درجة الحموضة 8.5). تنفيذ هذه الخطوة في منطقة مختلفة عن استخراج الحمض النووي أو الحمض النووي الريبي لمنع التلوث في المستقبل، وقطع هلام سوف تنتشر amplicons PCR على كل من المجرب والمنطقة المحيطة بها.
- تحقق من تركيز المسبح المنقى باستخدام مقياس الفلور أو مطياف. إذا سارت عملية التنقية على ما يرام، يجب أن يكون تركيزها على الأقل نصف حمام السباحة غير المنقوط. عموما، يجب أن يتراوح التركيز النهائي من 5 إلى 20 نانوغرام/ميكرولتر.
- إرسال المكتبات المنقى لتسلسل الجيل القادم. تأكد من أنها تبقى باردة أثناء النقل عن طريق تضمين الجليد الجاف في حاوية الشحن.
6. إنشاء مكتبة الجيش الملكي النيبالي وتنقية
- ويمكن استخدام عدة مجموعات تجارية لإنشاء مكتبات الجيش الملكي النيبالي. لأيهما يتم استخدامه، اتبع بروتوكول الشركة المصنعة كما هو مكتوب أثناء العمل في بيئة تدفق صفح معقمة. ويرد أدناه21نسخة موجزة جدا من بروتوكول لعدة في جدول المواد.
- جعل أول مزيج سيد توليف حبلا cDNA (8 ميكرولتر من المياه الخالية من النيوكليز و 2 ميكرولتر من أول ستراند توليف Enyzme ميكس) وإضافته إلى العينة. ضع العينة في مدورة حرارية مع الشروط المحددة في البروتوكول.
- جعل ثاني حبلا cDNA توليف مزيج رئيسي (8 ميكرولتر من الثانية ستراند توليف التفاعل العازلة, 4 ميكرولتر الثاني ستراند توليف مزيج الانزيم, و 48 ميكرولتر من المياه الخالية من النيوكليز) على الجليد وإضافته إلى العينة. ضعه في دراجة حرارية تم ضبطها على 16 درجة مئوية لمدة ساعة واحدة.
- تنقية رد الفعل عن طريق إضافة الخرز المقدمة (144 ميكرولتر) وأداء اثنين من يغسل الإيثانول 80٪ (200 ميكرولتر).
- Elute مع العازلة TE المقدمة (53 ميكرولتر) ونقل 50 ميكرولتر من supernatant إلى أنبوب PCR نظيفة. ضع أنبوب PCR على الجليد.
- جعل نهاية الإعدادية الرئيسية مزيج (7 ميكرولتر من نهاية الإعدادية رد فعل العازلة و 3 ميكرولتر من نهاية الإعدادية انزيم ميكس) على الجليد وإضافته إلى أنبوب PCR. ضع أنبوب PCR في دراجة حرارية مع الشروط المحددة في البروتوكول.
- اخلط محول المخفف (2.5 ميكرولتر)، وربط ماستر ميكس (30 ميكرولتر) ومحسن الربط (1 ميكرولتر) حلول على الجليد. أضف الحلول المختلطة للعينة وضعها في دراجة حرارية لمدة 15 دقيقة عند 20 درجة مئوية.
- تنقية رد الفعل عن طريق إضافة الخرز المقدمة (87 ميكرولتر) وأداء يغسل الإيثانول (200 ميكرولتر) والجيل كما كان من قبل، باستثناء إضافة فقط 17 ميكرولتر من TE.
- إضافة مؤشرات (10 ميكرولتر) و Q5 ماجستير ميكس (25 ميكرولتر) الحل ووضعها في thermocycler مع الشروط الموضحة في البروتوكول.
- تنقية رد الفعل عن طريق إضافة الخرز المقدمة (45 ميكرولتر) وأداء إضافة اثنين يغسل الإيثانول (200 ميكرولتر) وelute مع 23 ميكرولتر من TE. نقل 20 ميكرولتر إلى أنبوب PCR نظيفة.
- تحقق من المكتبات بحثا عن تركيزات الحمض النووي الريبي القابلة للكشف باستخدام التحليل الحيوي أو مقياس الفلور أو مطياف.
- تجمع المكتبات الميتاترانسكربتوميك في نسبة متساوية تقريبا.
- تنقية المكتبة باتباع نفس البروتوكول لتنقية مكتبة 16S ، باستثناء أجزاء المكوس بين 250 و 400 bp. في حين أن مكتبة 16S كان لها نطاق متميز يمثل المنطقة المضخمة ، فإن النتيجة هنا هي تشويه.
- تحقق من تركيز المكتبة المنقى كما كان من قبل.
- شحن المكتبة المنقى مع الجليد الجاف إلى مرفق التسلسل.
ملاحظة: بدلا من ذلك، يمكن إرسال مقتطفات الحمض النووي الريبي إلى جامعة أو شركة خاصة لإعداد المكتبة وتسلسلها.
7. تحليل المجتمع الميكروبي
- بمجرد اكتمال التسلسل، قم بالوصول إلى نموذج البيانات. تحميل البرنامج على جهاز كمبيوتر قابل للاستخدام.
ملاحظة: بشكل مثالي، يجب أن يكون الجهاز على الأقل 16 غيغابايت من ذاكرة الوصول العشوائي. لمناقشة متطلبات الحوسبة (لQiime2)، راجع https://forum.qiime2.org/t/recommended-specifications-to-run-qiime2/9808. - استخدم برامج مثل mothur و QIIME2 و R لتحليل بيانات rRNA 16S. انظر هنا https://docs.qiime2.org/2020.11/tutorials/moving-pictures/ للحصول على مثال QIIME2 16S تحليل البرنامج التعليمي.
- بالنسبة لبيانات metatranscriptomics (RNA)، استخدم HUMAnN2 و ATLAS لتحديد الجينات والمسارات الموجودة في العينات.
ملاحظة: مثال خط أنابيب metatranscriptomics يتوج بالتنوع ويتم تقديم تحليل الغابات العشوائية في ملف المعلومات التكميلية. يتم تشغيل كافة الأوامر من خلال سطر الأوامر، على سبيل المثال، المحطة الطرفية لمستخدمي Mac.
النتائج
يمكن تقييم نجاح استخراج الحمض النووي والحمض النووي الريبي باستخدام مجموعة متنوعة من المعدات والبروتوكولات. عموما، يعتبر أي تركيز يمكن الكشف عنها من أي كافية لاستنتاج أن استخراج كانت ناجحة. وعند فحص الجدول 1، سيطلق على جميع عمليات الاستخراج، باستثناء واحدة، اسم ناجح. غالبا ما يرجع الفشل في هذه الخطوة إلى انخفاض الكتلة الحيوية الأولية أو ضعف الحفاظ على العينة أو الخطأ البشري أثناء الاستخراج. في حالة المرشحات، قد يكون الاستخراج ناجحا حتى لو كان التركيز أقل من الكشف. إذا كانت تلك المقتطفات لا تسفر عن نطاقات لPCR (إذا كان القيام 16S) أو تركيز يمكن الكشف عنها بعد إعداد المكتبة (metatranscriptomics)، ثم أنها لم تفشل على الأرجح حقا.
إذا تم اتباع بروتوكول 16S ، فإن النطاقات الساطعة التالية لتضخيم PCR ، كما رأينا في الآبار 4 و 6 في الشكل 1، تشير إلى النجاح ، في حين يشير نقص النطاقات ، كما رأينا في الآبار الأخرى في الصف العلوي ، إلى الفشل. وعلاوة على ذلك، فإن وجود نطاق ساطع في حارة الجل يحتوي على عنصر تحكم سلبي في الفينول الخماسي الكلور سيشير أيضا إلى حدوث عطل لأنه سيكون من الخطر افتراض أن التلوث الذي يؤثر على السيطرة السلبية (الرقابة) لم يؤثر على العينات.
بالنسبة لكل من 16S و metatranscriptomics ، يمكن تقييم نجاح التسلسل من خلال النظر في عدد التسلسلات التي تم الحصول عليها(الشكل 2). يجب أن يكون الحد الأدنى لعينات 16S 1000 تسلسل ، مع ما لا يقل عن 5000 مثالية(الشكل 2A). وبالمثل، ينبغي أن يكون عينات metatranscriptomics ما لا يقل عن 500،000 تسلسل، مع ما لا يقل عن 2،000،000 يجري المثالي(الشكل 2B). وينبغي عدم استخدام العينات ذات التسلسلات الأقل من تلك الحدود الدنيا للتحليلات، لأنها قد لا تمثل بدقة مجتمعها البكتيري. ومع ذلك، لا يزال من الممكن استخدام العينات التي تقع بين الحد الأدنى والمثالي على الرغم من أنه ينبغي تفسير النتائج بحذر أكبر إذا سقطت العديد من العينات في هذا النطاق.
ويمكن تحديد نجاح التحليل النهائي اللاحق ببساطة على أساس ما إذا كان قد تم الحصول على ملفات الإخراج المتوقعة أم لا. وعلى أية حال، ينبغي للبرامج، مثل QIIME2 و R(الشكل 3)،أن تسمح بتقييم الاختلافات الكبيرة المحتملة بين المجتمعات البكتيرية القائمة على التكسير. تم الحصول على بيانات الشكل 3 عن طريق جمع عينات من الرواسب من 21 موقعا مختلفا في ثلاثة عشر مجاري مختلفة لتحليل 16S و metatranscriptomics. ومن بين هذه المواقع ال 21، كان اثنا عشر موقعا منها في المصب من نشاط التكسير وصنفت على أنها HF+، وكان تسعة منها إما في المنبع من نشاط التكسير أو في مستجمعات مياه لا يحدث فيها التكسير؛ تم تصنيف هذه الجداول كHF-. وإلى جانب وجود نشاط التكسير، كانت الجداول قابلة للمقارنة.
ويمكن أن تتخذ هذه الاختلافات شكل تحولات تكوينية متسقة على أساس حالة التكسير. وإذا كان الأمر كذلك، يتوقع أن تتجمع عينات HF+ وHF- بعيدا عن بعضها البعض في قطعة أرض PCoA، كما هو الحال في الشكل 3A والشكل 3B. للتأكد من أن هذه التحولات الظاهرة ليست مجرد قطعة أثرية من طريقة التنسيق ، هناك حاجة إلى مزيد من التحليل الإحصائي. على سبيل المثال، كشف اختبار PERMANOVA22 على مصفوفة المسافة التي يستند إليها الشكل 3A والشكل 3B عن تجمع كبير استنادا إلى حالة التكسير، مما يعني أن الفصل الملاحظ في المؤامرة يتسق مع الاختلافات بين المجتمعات البكتيرية للعينات، بدلا من قطعة أثرية من التنسيق. ومن النتائج الهامة التي تمخضت عنها PERMANOVA أو ANOSIM وجود إشارة قوية إلى وجود اختلافات ثابتة بين عينات HF+ وHF- ، مما يشير إلى أن عينات HF + تأثرت بالتكسير ، في حين أن القيمة العالية P تشير إلى أن العينات لم تتأثر. يمكن أيضا تصور البيانات الميتاترانسكريبتومية وتقييمها باستخدام نفس الطرق.
ويمكن أن يكشف فحص السمات التفاضلية (الميكروبات أو الوظائف) عن أدلة على أن العينات قد تأثرت أيضا. طريقة واحدة لتحديد ميزات تفاضلية هو إنشاء نموذج مجموعة تفرعات عشوائية. يمكن استخدام نموذج الغابة العشوائي لمعرفة مدى جودة تصنيف حالة تكسير العينات بشكل صحيح. إذا كان أداء النموذج أفضل مما كان متوقعا عن طريق الصدفة، فإن ذلك سيكون دليلا إضافيا على الاختلافات التي تعتمد على حالة التكسير. وعلاوة على ذلك، فإن أهم التوقعات تكشف عن الميزات الأكثر أهمية للتمييز بشكل صحيح بين العينات(الشكل 3C). تلك الميزات أيضا ثم كان لها قيم مختلفة باستمرار على أساس حالة التكسير. وبمجرد تحديد هذه الميزات التفاضلية، يمكن مراجعة الأدبيات لمعرفة ما إذا كانت قد ارتبطت سابقا بالتكسير. ومع ذلك، قد يكون من الصعب العثور على الدراسات التي حددت وظائف تفاضلية، لأن معظمها قد استخدمت فقط 16S rRNA البيانات التركيبية. ولذلك، من أجل تقييم الآثار المترتبة على الوظائف التفاضلية، فإن إحدى الطرق الممكنة هي معرفة ما إذا كانت قد ارتبطت في السابق بمقاومة محتملة للمبيدات الحيوية المستخدمة عادة في سائل التكسير أو ما إذا كان يمكن أن تساعد في تحمل الظروف الملحية للغاية. وعلاوة على ذلك، فإن دراسة الملامح الوظيفية للتصنيف من الفائدة يمكن أن تكشف عن أدلة على تأثير التكسير(الشكل 3D). على سبيل المثال، إذا تم تحديد التصنيف على أنه تفاضلي من خلال نموذج الغابة العشوائي، يمكن مقارنة ملف مقاومته المضادة للميكروبات في عينات HF+ بملفه الشخصي في عينات HF- وإذا اختلفت بشكل كبير، يمكن أن يشير ذلك إلى أن سائل التكسير الذي يحتوي على مبيدات حيوية دخل المجرى.
| معرف العينة | التركيز (نانوغرام/ميكرولتر) |
| 1 | 1.5 |
| 2 | 1.55 |
| 3 | 0.745 |
| 4 | 0.805 |
| 5 | 7.82 |
| 6 | 0.053 |
| 7 | 0.248 |
| 8 | 0.945 |
| 9 | 1.82 |
| 10 | 0.804 |
| 11 | 0.551 |
| 12 | 1.69 |
| 13 | 4.08 |
| 14 | Below_Detection |
| 15 | 7.87 |
| 16 | 0.346 |
| 17 | 2.64 |
| 18 | 1.15 |
| 19 | 0.951 |
الجدول 1: مثال تركيزات الحمض النووي على أساس قياس الفلورومتر 1x DS الحمض النووي عالية الحساسية المقايسة. وتعتبر عمليات استخراج جميع هذه العينات، باستثناء 14 عينة، ناجحة بسبب وجود كميات يمكن اكتشافها من الحمض النووي.
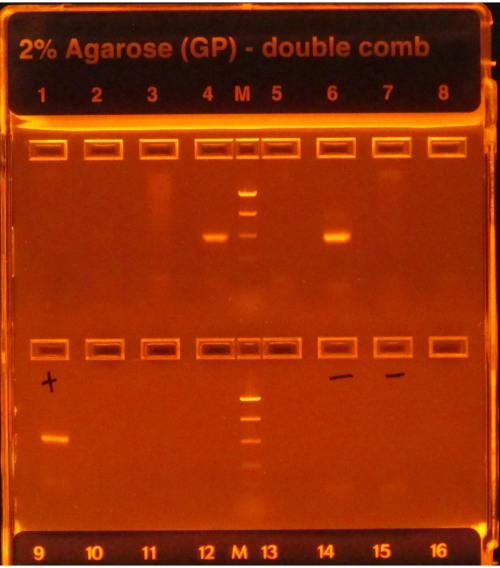
الشكل 1: مثال هلام إلكتروني مع منتجات PCR. كان الجل ملطخا مسبقا ومتخيلا تحت ضوء الأشعة فوق البنفسجية ، مما تسبب في توهج أي حمض نووي موجود عليه. عملت PCR للعينات في الآبار 4 و 6 في الصف الأول ، حيث كان لكليهما نطاق مشرق واحد من الحجم المتوقع (استنادا إلى السلم). وفشلت العينات في الآبار الست الأخرى، لأنها لم تنتج أي نطاقات. وكان للتحكم الإيجابي (البئر الأول والصف الثاني) نطاق ساطع يشير إلى أن ال PCR تم تنفيذه بشكل صحيح، ولم يكن للضوابط السلبية (البئران 6 و7، الصف الثاني) أي نطاقات، مما يشير إلى أن العينات لم تكن ملوثة. ولو كان للنفي نطاق ساطع مثل العينات، لكان PCR يعتبر فشلا لأنه سيكون من الخطر افتراض أن العينات تحتوي على أمبليكونات ليست مجرد نتيجة للتلوث. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
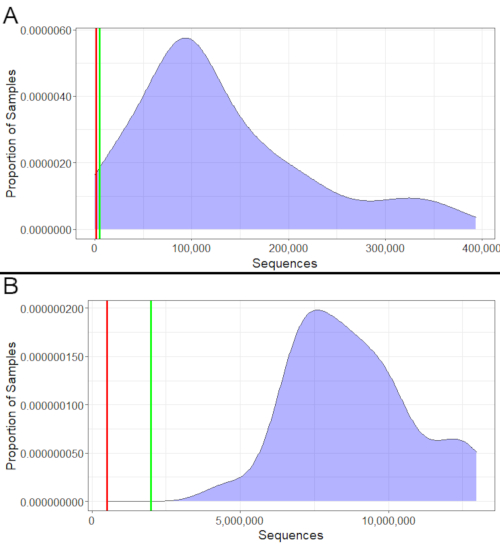
الشكل 2: المثال عدد التسلسل. (أ) 16S المثال تسلسل التهم. تقريبا كل هذه العينات 16S كان أكثر من 1000 تسلسل. وينبغي استبعاد القلة القليلة جدا التي يقل تسلسلها عن 000 1 تسلسل من التحليلات النهائية، لأن تسلسلها لا يكفي لتمثيل مجتمعاتها البكتيرية بدقة. وكان عدد من التسلسلات يتراوح بين 000 1 و 000 5 تسلسل؛ في حين أنها ليست مثالية ، فإنها لا تزال صالحة للاستخدام لأنها تتجاوز الحد الأدنى ، وغالبية العينات تتجاوز الحد الأدنى المثالي من 5000 كذلك. (ب) حساب المثال ميتاترانسكريبتوميك. وتجاوزت جميع العينات الحد الأدنى (000 500) والحد الأدنى المثالي (000 000 2) من التسلسلات. ولذلك، كان التسلسل ناجحا بالنسبة لهم جميعا، ويمكن استخدامها جميعا في التحليل النهائي. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
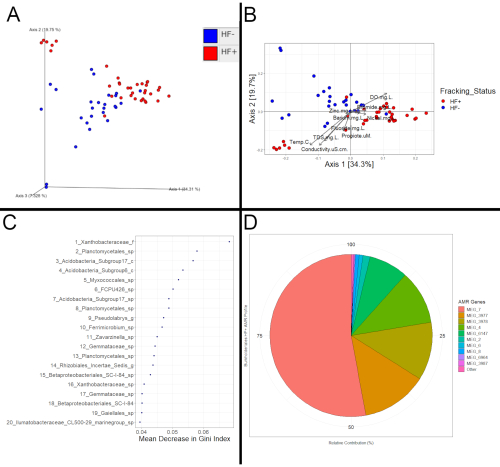
الشكل 3: تحليل المثال. (أ) مخطط PCoA استنادا إلى الإحداثيات المحسوبة بمصفوفة مسافة Unifrac مرجحة تم إنشاؤها وتصورها من خلال QIIME2. (B) مخطط PCoA استنادا إلى الإحداثيات المحسوبة بمصفوفة المسافة Unifrac المرجحة المصدرة من QIIME2. تم تصور الإحداثيات باستخدام حزم Phyloseq و ggplot2 في ناقلات البيانات الوصفية R. تم تركيبها على المؤامرة باستخدام حزمة Vegan. تمثل كل نقطة المجتمع البكتيري للعينة ، مع نقاط أقرب تشير إلى تكوينات مجتمعية أكثر تشابها. ولوحظ وجود تجمع على أساس حالة التكسير لعينات الرواسب 16S هذه (PERMANOVA, p=0.001). وعلاوة على ذلك، تكشف المتجهات أن عينات HF+ تميل إلى أن تكون مستويات أعلى من الباريوم والبروميد والنيكل والزنك، والتي تتوافق مع تكوين المجتمع البكتيري المختلفة مقارنة مع عينات HF- . (ج) مؤامرة من أفضل التوقعات لنموذج الغابات العشوائية التي اختبرت حيث وفرة البكتيرية يمكن استخدامها للتنبؤ حالة التكسير بين العينات. تم إنشاء نموذج الغابة العشوائية من خلال R باستخدام حزمة randomForest. وتظهر أعلى 20 تنبؤات وكذلك الانخفاضات الناتجة في النجاسة (قياس عدد عينات HF + وHF - مجمعة معا) في شكل انخفاض متوسط في مؤشر جيني عندما يتم استخدامها لفصل العينات. (D) مخطط دائري يوضح ملف تعريف مقاومة مضادات الميكروبات لملف تعريف البيركهولدريالات استنادا إلى بيانات metatranscriptomic. تم شرح التسلسلات لأول مرة مع Kraken2 لتحديد التصنيف الذي ينتمون إليه. ثم استخدم الانفجار مع تلك التسلسلات المشروحة وقاعدة بيانات MEGARes 2.0 لتحديد جينات مقاومة مضادات الميكروبات (في شكل "MEG_#") التي يتم التعبير عنها بنشاط. ثم تم استخراج جينات مقاومة مضادات الميكروبات التي أعرب عنها أعضاء بيركهولدريال لمعرفة الجينات الأكثر انتشارا بين تلك التصنيفات. في حين أن أكثر تكلفة وتستغرق وقتا طويلا، metatranscriptomics لا تسمح لتحليل وظيفي، مثل هذا الذي لا يمكن القيام به مع بيانات 16S. وتجدر الإشارة إلى أن كراكين2 استخدم لتحليل هذا المثال، بدلا من HUMAnN2. كراكين2 أسرع من HUMAnN2; ومع ذلك ، فإنه يخرج فقط المعلومات التركيبية ، بدلا من التكوين والمساهمة والوظائف (الجينات) ومسارات مثل HUMAnN2 لا. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
ملف تكميلي: مثال خط أنابيب metatranscriptomics. الرجاء الضغط هنا لتحميل هذا الملف.
Discussion
تم تطوير الأساليب الموصوفة في هذه الورقة وصقلها على مدار العديد من الدراسات التي نشرتها مجموعتنا بين عامي 2014 و 20187و8و10 وتم توظيفها بنجاح في مشروع تعاوني للتحقيق في آثار التكسير على المجتمعات المائية في مشروع مدته ثلاث سنوات سيقدم قريبا ورقة للنشر. وسيستمر استخدام هذه الأساليب خلال الفترة المتبقية من المشروع. بالإضافة إلى ذلك ، تصف الأدبيات الحالية الأخرى التي تحقق في تأثير التكسير على الجداول والنظم الإيكولوجية أساليب مماثلة لجمع العينات ومعالجتها وتحليلها7و8و10و11. ومع ذلك، لم تستخدم أي من تلك الأوراق التحليل الميتاترانسكريبتومي، مما يجعل هذه الورقة الأولى لوصف كيفية استخدام هذه التحليلات لتوضيح تأثير التكسير على الجداول القريبة. وعلاوة على ذلك، فإن الأساليب المعروضة هنا لجمع العينات أكثر تفصيلا، وكذلك الخطوات المتخذة لتجنب التلوث.
واحدة من أهم الخطوات في بروتوكولنا هي جمع العينات الأولية والحفاظ عليها. يأتي أخذ العينات الميدانية وجمعها مع بعض التحديات، حيث أن الحفاظ على بيئة معقمة أو معقمة أثناء الجمع قد يكون صعبا. خلال هذه الخطوة، من الضروري تجنب تلويث العينات. للقيام بذلك، يجب ارتداء قفازات، وينبغي السماح فقط حاويات وأدوات معقمة لتلامس مع العينات. كما ينبغي وضع العينات على الجليد على الفور بعد جمعها للتخفيف من تدهور الحمض النووي. إضافة مادة حافظة حمض النوى التجارية عند جمع يمكن أيضا زيادة غلة حمض النيوكليك والسماح للعينات ليتم تخزينها لفترات أطول من الزمن بعد جمعها. كلما يتم تنفيذ استخراج الحمض النووي، من المهم استخدام الكمية المناسبة من العينة، والكثير يمكن أن تسد مرشحات تدور المستخدمة لاستخراج (لتلك البروتوكولات التي تستخدم منها) ولكن القليل جدا يمكن أن يؤدي إلى انخفاض الغلة. تأكد من اتباع الإرشادات الخاصة بأي مجموعة يتم استخدامها.
وعلى غرار جمع الحقول، فإن تجنب التلوث أو تقليله إلى أدنى حد مهم أيضا أثناء استخراج الحمض النووي وإعداد العينات، لا سيما عند العمل مع عينات منخفضة من غلة الحمض النووي، مثل عينات الرواسب دون المستوى الأمثل (عينات تحتوي على كمية كبيرة من الحصى أو الصخور) أو عينات المياه. لذلك ، كما هو الحال مع جمع العينات ، يجب ارتداء قفازات خلال جميع هذه الخطوات للحد من التلوث. بالإضافة إلى ذلك ، يجب تعقيم جميع أسطح العمل المستخدمة أثناء إجراءات المختبر مسبقا عن طريق المسح بمحلول تبييض بنسبة 10٪ ، يليه محلول الإيثانول بنسبة 70٪. بالنسبة لخطوات الأنابيب (3-6) ، يجب استخدام نصائح التصفية لتجنب التلوث بسبب المصصة نفسها ، مع تغيير النصائح في كل مرة تلمس فيها سطحا غير معقم. يجب مسح جميع الأدوات المستخدمة في العمل المختبري ، بما في ذلك المصامح ، قبل وبعد حلول التبييض والإيثانول. لتقييم التلوث، يجب تضمين فراغات الاستخراج والسلبيات (السائل المعقم) خلال كل مجموعة من عمليات استخراج الحمض النووي وتفاعلات PCR. إذا كشف القياس الكمي بعد الاستخراج عن كمية يمكن اكتشافها من الحمض النووي / الحمض النووي الريبي في السلبيات ، يمكن تكرار عمليات الاستخراج إذا كان هناك عينة كافية متبقية. إذا أظهرت عينات سالبة ل PCR تضخيم، يجب إجراء استكشاف الأخطاء وإصلاحها لتحديد المصدر ومن ثم يجب إعادة تشغيل العينات. ولحصر مستويات التلوث المنخفضة، يوصى بتسلسل فراغات الاستخراج وسلبيات PCR بحيث يمكن تحديد الملوثات وإزالتها، إذا لزم الأمر، أثناء التحليل الحسابي. وعلى العكس من ذلك، يمكن أن يفشل تضخيم PCR أيضا بسبب مجموعة متنوعة من الأسباب. بالنسبة للعينات البيئية ، غالبا ما يكون تثبيط تفاعل PCR هو الجاني ، والذي يمكن أن يكون بسبب مجموعة متنوعة من المواد التي تتداخل مع Taq polymerase23. إذا اشتبه في تثبيط، يمكن استخدام المياه الصف PCR (انظر جدول المواد)لتمييع مقتطفات الحمض النووي.
هذا البروتوكول لديه بعض القيود الملحوظة والصعوبات المحتملة. ويمكن أن يشكل جمع العينات تحديا بالنسبة لعينات المياه والرواسب على حد سواء. من أجل الحصول على ما يكفي من الكتلة الحيوية، من الناحية المثالية 1 لتر من مياه المجرى يحتاج إلى دفع من خلال مرشح. المسام من مرشح تحتاج إلى أن تكون صغيرة لالتقاط الميكروبات ولكن يمكن أيضا فخ الرواسب. إذا كان هناك الكثير من الرواسب في الماء بسبب هطول الأمطار مؤخرا ، يمكن للمرشح تسد مما يجعل من الصعب دفع الحجم بأكمله من خلال الفلتر. وبالنسبة لجمع الرواسب، قد يكون من الصعب تقدير عمق الرواسب أثناء جمعها. وعلاوة على ذلك، من المهم ضمان أن تكون الرواسب التي يتم جمعها في الغالب تربة، لأن الحصى والصخور ستؤدي إلى انخفاض غلة الحمض النووي وقد لا تكون تمثيلا دقيقا للمجتمع الميكروبي. وأخيرا، من الضروري أيضا أن يتم الاحتفاظ بالعينات على الجليد بعد جمعها، خاصة إذا لم يتم استخدام مادة حافظة.
على الرغم من أن هذا البروتوكول يغطي بروتوكولات الميثاترانسكريبتوميك و16S المعملية، يجب التأكيد على أن هاتين الطريقتين مختلفتان جدا في كل من العملية وفي نوع البيانات التي توفرها. الجين 16S rRNA هو منطقة مستهدفة بشكل شائع ، محفوظة بشكل كبير في البكتيريا والعطرة ، ومفيدة لوصف المجتمع البكتيري في عينة. وعلى الرغم من اتباع نهج محدد ومحدد، فإن استبانة مستوى الأنواع غالبا ما تكون بعيدة المنال، ومن الصعب وصف الأنواع أو السلالات المتباينة حديثا. على العكس من ذلك ، metatranscriptomics هو نهج أوسع يلتقط جميع الجينات النشطة والميكروبات الموجودة داخل العينة. في حين أن 16S يوفر بيانات فقط لتحديد الهوية ، يمكن أن يوفر metatranscriptomics بيانات وظيفية مثل الجينات المعرب عنها والمسارات الأيضية. كلاهما قيمة وعندما مجتمعة، فإنها يمكن أن تكشف عن البكتيريا الموجودة والجينات التي يعبرون عنها.
تصف هذه الورقة طرق جمع الحقول ومعالجة العينات لكل من التحليلات 16S rRNA و metatranscriptomic في سياق دراسة التكسير. بالإضافة إلى ذلك ، فإنه تفاصيل أساليب جمع الحمض النووي عالية الجودة / الحمض النووي الريبي من عينات الكتلة الحيوية المنخفضة والتخزين على المدى الطويل. الأساليب الموصوفة هنا هي تتويج لتجاربنا مع جمع العينات ومعالجتها في جهودنا لمعرفة كيفية تأثير التكسير على الجداول القريبة من خلال دراسة بنية ووظيفة مجتمعاتهم الميكروبية. تستجيب الميكروبات بسرعة للاضطرابات، وبالتالي، يمكن للميكروبات الموجودة والجينات التي تعبر عنها أن توفر معلومات حول آثار التكسير على النظم الإيكولوجية. وعموما، يمكن أن تكون هذه الأساليب لا تقدر بثمن في فهمنا لكيفية تأثير التكسير على هذه النظم الإيكولوجية الهامة.
Disclosures
وليس لدى صاحبي البلاغ ما يكشفان عنه.
Acknowledgements
ويود المؤلفون أن يعترفوا بمصادر تمويل المشاريع التي أدت إلى تطوير هذه الأساليب، مع وجود هذه المصادر: معهد هوارد هيوز الطبي (http://www.hhmi.org) من خلال برنامج التعليم العلمي قبل الجامعي والجامعي، وكذلك من قبل المؤسسة الوطنية للعلوم (http://www.nsf.gov) من خلال جوائز NSF DBI-1248096 وCBET-1805549.
Materials
| Name | Company | Catalog Number | Comments |
| 200 Proof Ethanol | Thermo Fisher Scientific | A4094 | 400 mL need to be added to Buffer PE (see Qiagen QIAQuck Gel Extraction kit protocol) and 96 mL needs to be added to the DNA/RNA Wash Buffer (see ZymoBIOMICS DNA/RNA Miniprep kit protocol). Additional ethanol is needed for the ZymoBIOMICS DNA/RNA Miniprep and NEBNext® Ultra™ II RNA Library Prep with Sample Purification Beads kits. |
| Agarose | Thermo Fisher Scientific | BP1356-100 | 100 g per bottle. 0.6 g of agarose would be needed to make one 2% 30 mL gel. |
| Disinfecting Bleach | Walmart (Clorox) | No catalog number | Use a 10% bleach solution for cleaning the work area before and after lab procedures |
| DNA gel loading dye | Thermo Fisher Scientific | R0611 | Each user-made (i.e. non-e-gel) should include loading dye with all of the samples in the ratio of 1 µL dye to 5 µL sample |
| DNA ladder | MilliporeSigma | D3937-1VL | A ladder should be run on every gel/e-gel |
| DNA/RNA Shield (2x) | Zymo Research | R1200-125 | 3 mL per sediment sample (50 mL conical) and 2 mL per water sample (filter) |
| Ethidium bromide | Thermo Fisher Scientific | BP1302-10 | Used for staining user-made e-gels |
| Forward Primer | Integrated DNA Technologies (IDT) | 51-01-19-06 | 0.5 µL per PCR reaction |
| Isopropanol | MilliporeSigma | 563935-1L | Generally less than 2 mL per library. Volume needed varies by mass of excised gel fragment (see Qiagen QIAQuick Gel Extraction kit protocol). |
| PCR-grade water | MilliporeSigma | 3315932001 | 13 µL per PCR reaction (assuming 1 µL of sample DNA template is used) |
| Platinum Hot Start PCR Master Mix (2x) | Thermo Fisher Scientific | 13000012 | 10 µL per PCR reaction |
| Reverse Primer | Integrated DNA Technologies (IDT) | 51-01-19-07 | 0.5 µL per PCR reaction |
| TBE Buffer (Tris-borate-EDTA) | Thermo Fisher Scientific | B52 | 1 L of 10x TBE buffer (30 mL of 1x TBE buffer would be needed to make one 30 mL gel) |
| 1 L bottle | Thermo Fisher Scientific | 02-893-4E | One needed per stream (the same bottle can be used for multiple streams if it is sterilized between uses) |
| 1.5 mL Microcentrifuge tubes | MilliporeSigma | BR780400-450EA | 5 microcentrifuge tubes are needed per DNA extraction and an additional 3 are needed to purify RNA (see ZymoBIOMICS DNA/RNA Miniprep kit protocol) |
| 2% Agarose e-gel | Thermo Fisher Scientific | G401002 | Each gel can run 10 samples (so 9 with a PCR negative and 8 if the extraction negative is run on the same gel) |
| 50 mL Conicals | CellTreat | 229421 | 1 50 mL conical needed per sediment samples |
| 500 mL Beaker | MilliporeSigma | Z740580 | Only 1 needed (for flame sterilization) |
| Aluminum foil | Walmart (Reynolds KITCHEN) | No number | Aluminum foil can be folded and autoclaved. The part not exposed to the environment can then be used as a sterile, DNA and RNA free surface for processing filters (one folded piece per filter to avoid cross-contamination) |
| Autoclave | Gettinge | LSS 130 | Only one needed |
| Centrifuge | MilliporeSigma | EP5404000138-1EA | Only 1 needed |
| Cooler | ULINE | S-22567 | Just about any cooler can be used. This one is listed due to being made of foam, making it lighter and thus easier to take along for field sampling. |
| Disruptor Genie | Bio-Rad | 3591456 | Only one needed |
| Electrophoresis chamber | Bio-Rad | 1664000EDU | Only 1 needed |
| Electrophoresis power supply | Bio-Rad | 1645050 | Only 1 needed |
| Freezer (-20 C) | K2 SCIENTIFIC | K204SDF | One needed to store DNA extracts |
| Freezer (-80 C) | K2 SCIENTIFIC | K205ULT | One needed to store RNA extracts |
| Gloves | Thermo Fisher Scientific | 19-020-352 | The catalog number is for Medium gloves. |
| Heat block | MilliporeSigma | Z741333-1EA | Only one needed |
| Lab burner | Sterlitech | 177200-00 | Only one needed |
| Laminar Flow Hood | AirClean Systems | AC624LFUV | Only 1 needed |
| Library purification kit | Qiagen | 28704 | One kit has enough for 50 reactions |
| Magnet Plate | Alpaqua | A001219 | Only one needed |
| Microcentrifuge | Thermo Fisher Scientific | 75004061 | Only one needed |
| Micropipette (1000 µL volume) | Pipette.com | L-1000 | Only 1 needed |
| Micropipette (2 µL volume) | Pipette.com | L-2 | Only 1 needed |
| Micropipette (20 µL volume) | Pipette.com | L-20 | Only 1 needed |
| Micropipette (200 µL volume) | Pipette.com | L-200R | Only 1 needed |
| NEBNext Ultra II RNA Library Prep with Sample Purification Beads | New England BioLabs Inc. | E7775S | One kit has enough reagents for 24 samples. |
| Parafilm | MilliporeSigma | P7793-1EA | 2 1" x 1" squares are needed per filter |
| PCR Tubes | Thermo Fisher Scientific | AM12230 | One tube needed per reaction |
| Pipette tips (for 1000 µL volume) | Pipette.com | LF-1000 | Pack of 576 tips |
| Pipette tips (for 20 µL volume) | Pipette.com | LF-20 | Pack of 960 tips |
| Pipette tips (for 200 µL volume) | Pipette.com | LF-250 | Pack of 960 tips |
| PowerWulf ZXR1+ computer cluster | PSSC Labs | No number | This is just an example of a supercomputer powerful enough to perform metatranscriptomics analysis in a timely manner. Only one needed. |
| Qubit fluorometer starter kit | Thermo Fisher Scientific | Q33239 | Comes with a Qubit 4 fluorometer, enough reagent for 100 DNA assays, and 500 Qubit tubes |
| Scoopula | Thermo Fisher Scientific | 14-357Q | Only one needed |
| Sterile blades | AD Surgical | A600-P10-0 | One needed per filter |
| Sterivex-GP Pressure Filter Unit | MilliporeSigma | SVGP01050 | 1 filter needed per water sample |
| Thermocycler | Bio-Rad | 1861096 | Only one needed |
| Vise-grip | Irwin | 2078500 | Only one needed (for cracking open the filters) |
| Vortex-Genie 2 | MilliporeSigma | Z258415-1EA | Only 1 needed |
| WHIRL-PAK bags | ULINE | S-22729 | 1 needed per filter |
| ZymoBIOMICS DNA/RNA Miniprep kit | Zymo Research | R2002 | One kit has enough reagents for 50 samples. |
References
- The process of unconventional natural gas production. US EPA Available from: https://www.epa.gov/uog/process-unconventional-natural-gas-production (2013)
- Brittingham, M. C., Maloney, K. O., Farag, A. M., Harper, D. D., Bowen, Z. H. Ecological risks of shale oil and gas development to wildlife, aquatic resources, and their habitats. Environmental Science & Technology. 48 (19), 11034-11047 (2014).
- McBroom, M., Thomas, T., Zhang, Y. Soil erosion and surface water quality impacts of natural gas development in East Texas, USA. Water. 4 (4), 944-958 (2012).
- Maloney, K. O., Weller, D. E. Anthropogenic disturbance, and streams: land use and land-use change affect stream ecosystems via multiple pathways. Freshwater Biology. 56 (3), 611-626 (2011).
- Meyer, J. L., et al. The contribution of headwater streams to biodiversity in river networks1. JAWRA Journal of the American Water Resources Association. 43 (1), 86-103 (2007).
- Alexander, R. B., Boyer, E. W., Smith, R. A., Schwarz, G. E., Moore, R. B. The role of headwater streams in downstream water quality. Journal of the American Water Resources Association. 43 (1), 41-59 (2007).
- Ulrich, N., et al. Response of aquatic bacterial communities to hydraulic fracturing in Northwestern Pennsylvania: A five-year study. Scientific Reports. 8 (1), 5683 (2018).
- Chen See, J. R., et al. Bacterial biomarkers of Marcellus shale activity in Pennsylvania. Frontiers in Microbiology. 9, 1697 (2018).
- Rausch, P., et al. Comparative analysis of amplicon and metagenomic sequencing methods reveals key features in the evolution of animal metaorganisms. Microbiome. 7 (1), 133 (2019).
- Louca, S., Doebeli, M., Parfrey, L. W. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem. Microbiome. 6 (1), 41 (2018).
- Trexler, R., et al. Assessing impacts of unconventional natural gas extraction on microbial communities in headwater stream ecosystems in Northwestern Pennsylvania. Frontiers in Microbiology. 5, 522 (2014).
- Mumford, A. C., et al. Shale gas development has limited effects on stream biology and geochemistry in a gradient-based, multiparameter study in Pennsylvania. Proceedings of the National Academy of Sciences. 117 (7), 3670-3677 (2020).
- JoVE Core Biology DNA Isolation. Journal of Visualized Experiments Available from: https://www.jove.com/cn/science-education/10814/dna-isolation (2020)
- Oxford Gene Technology DNA Storage and Quality. OGT Available from: https://www.ogt.com/resources/literature/403_dna_storage_and_quality (2011)
- ThermoFisher SCIENTIFIC Technical Bulletin #159: Working with RNA. Thermoscientific Available from: https://www.thermofisher.com/us/en/home/references/ambion-tech-support/nuclease-enzymes/general-articles/working-with-rna.html (2020)
- QIAGEN AllPrep DNA/RNA Mini Kit. Qiagen Available from: https://www.qiagen.com/us/products/discovery-and-translational-research/dna-rna-purification/multianalyte-and-virus/allprep-dnarna-mini-kit/#orderinginformation (2020)
- ZymoBIOMICS DNA/RNA Miniprep Kit. Zymo Research Available from: https://www.zymoresearch.com/products/zymobiomics-dna-rna-miniprep-kit (2020)
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- 16S Illumina amplicon protocol: Earth microbiome project. Earth microbiome project Available from: https://earthmicrobiome.org/protocols-and-standards/16s/ (2018)
- Gel Purification: Binding, washing and eluting a sample | Protocol. Journal of Visualized Experiments Available from: https://www.jove.com/v/5063/gel-purification (2020)
- New England Biolabs protocol for the use with NEBNext Poly(A) mRNA magnetic isolation module (E7490) and NEBNext Ultra II RNA library prep kit for Illumina (E7770, E7775). New England Biolabs Available from: https://www.neb.com/protocols/2017/03/04/protocol-for-use-with-purified-mrna-or-rrna-depleted-rna-and-nebnext-ultra-ii-rna-library-prep-ki (2020)
- Anderson, M. J. Permutational multivariate analysis of variance (PERMANOVA). Wiley StatsRef: Statistics Reference Online. , 1-15 (2017).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved