Method Article
미생물 분자 서명을 사용하여 스트림에 유압 골절의 영향 평가
요약
여기에서는 물과 퇴적물 미생물 커뮤니티를 분석하여 인근 개울에 유압 파쇄가 미치는 영향을 조사하는 프로토콜을 제시합니다.
초록
일반적으로 "파쇄"라고 불리는 유압 파쇄(HF)는 고압 물, 모래 및 화학 물질을 혼합하여 바위를 파괴하고 석유와 가스를 방출합니다. 이 프로세스는 이전에는 얻을 수 없었던 자원에 대한 액세스를 제공하고 현재 미국의 전체 천연 가스의 3 분의 2를 생산하기 때문에 미국 에너지 산업에 혁명을 일으켰습니다. 파쇄는 미국 경제에 긍정적인 영향을 미쳤지만, 여러 연구는 해로운 환경 효과를 강조했습니다. 특히 우려되는 것은 전체 분수령의 건강에 불균형적으로 큰 영향을 미치기 때문에 특히 중요한 헤드 워터 스트림에 파쇄하는 효과입니다. 그 스트림 내의 박테리아는 스트림 건강의 지표로 사용할 수 있습니다. 따라서 이 프로토콜은 세균 커뮤니티를 사용하여 스트림이 파쇄에 의해 영향을 되었는지 확인하는 것을 목표로 합니다. 이를 위해 파쇄(잠재적으로 영향을 받을 수 있음) 및 상류 또는 다른 파쇄 활동(영향을 받지 않음)의 다른 분수령에서 온 퇴적물과 물 샘플을 수집해야 합니다. 그 견본은 그 때 미생물 지역 사회 조성을 조사하기 위하여 핵산 추출, 도서관 준비 및 순서를 복종합니다. 상관 관계 분석 및 기계 학습 모델을 사용하여 커뮤니티의 변화에 대한 설명과 파쇄의 영향을 위한 예측 바이오마커 식별을 식별할 수 있습니다. 이러한 방법은 파쇄에 근접하여 헤드워터 스트림 중 미생물 커뮤니티의 다양한 차이를 나타낼 수 있으며 파쇄 활동의 환경 영향에 대한 향후 조사의 토대가 될 수 있습니다.
서문
유압 파쇄(HF) 또는 "파쇄"는 화석 연료에 대한 수요가 계속 증가함에 따라 점점 더 널리 퍼지고 있는 천연 가스 추출 방법입니다. 이 기술은 고성능 드릴링 장비를 사용하여 메탄이 풍부한 셰일 퇴적물에 물, 모래 및 화학 물질을 혼합하여 일반적으로 갇힌 가스1을방출하는 것으로 구성됩니다.
이러한 틀에 얽매이지 않는 수확 기술은 비교적 새로운 것이기 때문에 이러한 관행이 인근 수로에 미치는 영향을 조사하는 것이 중요합니다. 파쇄 활동은 장비 운송 및 잘 패드 건설을위한 토지의 큰 swaths의 청소를 의무화. 약 1.2-1.7 헥타르의 토지는 각 웰 패드2에대해 지워야하며 잠재적으로 시스템3의유출 및 수질에 영향을 미칩니다. 어떤 생물제가 사용되는지 포함하여 파쇄 유체의 정확한 화학 적 조성을 둘러싼 투명성이 부족합니다. 또한, 폐수를 파쇄하는 경향이 높은 식수2. 더욱이, 폐수는 금속 및 자연발생 방사성 물질을 포함할 수 있다2. 따라서 사람의 실수나 장비 오작동으로 인한 파쇄유체의 누출 및 유출 가능성에 관한 것입니다.
스트림 생태계는 주변 풍경 의 변화에 매우 민감한 것으로 알려져 있습니다4 전체 분수령 내에서 생물 다양성5 적절한 영양소 사이클링6을 유지하는 데 중요합니다. 미생물은 담수 스트림에서 가장 풍부한 유기체이므로 영양 순환, 생분해 및 1 차 생산에 필수적입니다. 미생물 공동체 구성 및 기능은 과소에 대한 민감성으로 생태계에 대한 정보를 얻을 수 있는 훌륭한 도구역할을 하며, 최근 연구에 따르면 파쇄 활동7,8에근접하여 관찰된 세균 조립체의 뚜렷한 변화를 보이고 있다. 예를 들어, 베이제린키아, 버크홀더리아, 메탄노박테리움은 파쇄 근처의 개울에서 농축된 것으로 확인되었으며, 슈도노카르디아, 니트로스피라, 로도박터는 파쇄7에가깝지 않은 개울에서 농축되었다.
16S 리보소말 RNA(rRNA) 유전자의 차세대 염기서열 분석은 전체 게놈 염기서열 분석 접근법9보다빠르고 저렴세균공동체 조성을 결정하는 저렴한 방법이다. 분자 생태분야 내의 일반적인 사례는 16S rRNA 유전자의 고가변 V4 영역을 시퀀싱 해상도로, 종종 식별9의넓은 범위의 속 수준까지, 예측할 수 없는 환경 샘플에 이상적이기 때문에 이다. 이 기술은 출판된 연구에서 널리 구현되었으며 파쇄 작업이 수생 환경에 미치는 영향을 식별하는 데 성공적으로 활용되었습니다7,8. 그러나, 박테리아는 그들의 검출된 풍부도10에영향을 미치는 16S rRNA 유전자의 다양한 사본 번호를 가지고 있다는 것을 주목할 가치가 있습니다. 이를 설명하는 몇 가지 도구가 있지만 효능은의심스럽습니다 10. 보급에서 급속하게 증가하고 이 약점이 결여되는 또 다른 사례는 모든 RNA가 순서되는 형이상전사 순서입니다, 연구원이 활성 박테리아와 그들의 유전자 발현 둘 다 확인할 수 있도록.
따라서, 이전에 발표된연구에서7,8,11,12의방법과 는 달리, 이 프로토콜은 미생물 공동체 기능(metatranscriptomics)을 조사하기 위한 샘플 수집, 보존, 처리 및 분석을 다룹니다. 여기에 상세한 단계는 연구원이 항균 저항 유전자를 포함하여 그들의 스트림에 있는 세균에 의해 표현된 유전자 및 통로에 어떤 충격, 있는 경우에, 파쇄가 있었다는 것을 볼 수 있습니다. 또한 샘플 수집을 위해 제시된 세부 수준이 향상됩니다. 비록 몇 가지 단계와 노트 경험 많은 연구자에 게 분명 보일 수 있습니다., 그들은 단지 연구를 시작 하는 사람들에 게 귀중 한 수 있습니다.
본 명세서에서는 실험실의 수년간의 경험을 바탕으로 인근 스트림에 파쇄가 미치는 영향을 조사하기 위한 수단으로 세균 유전 데이터를 생성하는 샘플 수집 및 처리 방법을 설명합니다. 이러한 데이터는 다운스트림 응용 프로그램에서 파쇄 상태에 해당하는 차이를 식별하는 데 사용할 수 있습니다.
프로토콜
1. 핵산 추출을 위한 퇴적물 시료 의 수집
- 멸균 50mL 원콘튜브를 하천 물에 담급한다. 원치 않는 인간의 오염을 도입하지 않도록 샘플 수집 중에 장갑을 착용하십시오. 해안에서 또는 물 에서 상류를 향하고 중 하나를 수행합니다.
- 원문 튜브가 침수되는 동안 캡을 제거하고 원문 튜브에 1 ~ 3cm 깊이에서 약 3 mL의 퇴적물을 스쿱하는 데 사용합니다.
- 퇴적물 시료(약 1mL)를 덮는 얇은 층을 제외하고, 물에서 원물 튜브를 제거하고 모든 물을 덤프합니다.
- 1000 μL 파이펫과 적절한 파이펫 팁을 사용하여 수집된 샘플에 DNA/RNA 방부제 3mL(방부제 사양용 재료 표 참조)을 추가합니다. 파이펫 팁을 멸균 파이펫 팁 상자에 넣고 사용 후 폐기하기 직전에 만 부착하십시오. 방부제와 시료가 철저히 혼합되도록 캡된 원추형 튜브를 10번 반전시하십시오.
참고 단계 1.4는 필요하지 않지만 RNA를 나중에 퇴적물에서 추출하는 경우 강력히 권장됩니다. - 샘플 수집의 나머지 부분에 대한 얼음에 샘플을 배치합니다. 수집에서 반환하면, 샘플이 16S 분석 (DNA) 또는 -70 °C에 사용되는 경우 - 20 °C에서 냉동실에 저장, 그들은 형이상학 분석 (RNA)에 사용되는 경우.
2. 핵산 추출을 위한 필터 수집
- 멸균 1 L 병의 캡을 제거합니다. 상류 또는 해안에서 마주보고있는 동안, 상단에 스트림 물로 병을 채우고 그것을 덤프. 이 과정을 두 번 더 반복하여 병을 조절합니다. 전체 병을 네 번째로 채우고 캡을 씌웁니다.
참고: 1L 병을 재사용하는 경우 2분 동안 10%의 표백제로 헹구고, 그 다음에는 탈이온화된 물로 3회 헹구고, 70%에탄올로 한 번 헹구고, 마지막으로 설정으로 자동 적으로 자동화할 수 있습니다: 121.1°C에서 30분 노출 시간 및 15분 건조 시간. 오토클레이브 하는 동안, 병의 캡은 프로세스에서 압축 되는 병을 피하기 위해 매우 느슨한 해야 합니다. - 안정된 표면에 일단, 멸균 Luer 잠금 주사기를 사용하고 전체 볼륨을 그립니다. 그런 다음 주사기를 멸균 및 DNA/RNA가 없는 직경 1.7cm의 폴로테셔술폰 필터에 0.22 μm의 모공 크기로 연결하고 플런저를 끝까지 눌러 필터를 통해 전체 부피를 밀어 넣습니다. 병(1L)에서 수집된 총 부피가 필터를 통해 푸시될 때까지 이 과정을 반복한다.
참고: 주사기의 부피는 필터를 통해 밀어붙이는 총 물의 양이 추적되는 경우 가변적일 수 있다. 그러나 일반적으로 60mL이 바람직하다. 1 L은 이상적이지만, 일화적으로, 적어도 200mL의 부피는 DNA와 RNA의 추출을 위해 충분한 바이오매스 (mL당 ~20,000 세포를 가정)를 수집 할 가능성이 높습니다. - 약 20mL 상당의 공기를 주사기에 끌어서 필터를 통해 밀어필터에서 여분의 물을 제거합니다.
참고: 2.4단계가 수행되면 방부제의 손실을 방지하는 데 도움이 됩니다. - P1000 마이크로피펫을 사용하여 필터를 수평으로 유지하면서 필터의 더 큰 개구부(주사기에 부착된 곳)를 통해 방부제의 2mL를 추가합니다. 파이펫이 방부제가 필터에 들어가는지 확인하기 위해 피펫이 우울할 때 파이펫의 끝은 필터의 배럴 내에 있어야 합니다. 각 사용 후 팁을 변경합니다.
참고: 퇴적물 수집과 마찬가지로, 이 단계는 필요하지 않지만, 특히 RNA의 경우 나중에 핵산 수율을 높이는 데 강력히 권장됩니다. - 파라핀 필름 의 한 사각형을 벗겨 밀봉 필터의 각 개구부 / 끝 주위에 단단히 포장. 파라핀 필름 포장 필터를 멸균 샘플 백에 넣은 다음 수집 중에 전체 가방을 얼음 위에 놓습니다.
참고: 필터를 감싸는 데 사용되는 측면이 이전에 환경에 노출되지 않은 멸균되었는지 확인합니다. - 샘플링에서 돌아오면 필터를 -20°C에서 16S 또는 메타 전사학용 -70°C에 저장합니다.
3. 핵산 추출 및 정량화
- 샘플 전송을 시작하기 전에 10 % 표백제와 70 %의 에탄올로 작업 영역을 청소하십시오.
- 퇴적물(1.5단계에서)의 경우 일반적으로 ~0.25 g의 시료를 사용하십시오. 화염은 70%의 에탄올비커에 담그고 샘플 사이에 에탄올을 태우는 것으로 금속 공구를 살균합니다.
- 필터(2.6단계에서)의 경우 필터 용지를 멸균 튜브로 이동하여 추출합니다. 이렇게하려면 아래 단계를 따르십시오.
- 접이식 의 내부 부분이 외부 환경에 노출되지 않도록 알루미늄 호일을 접고 접힌 조각을 설정으로 자동화하여 멸균, DNA 및 RNA 자유 표면을 만듭니다: 121.1°C 및 5분 건조 시간.
- 70% 에탄올과 화염으로 바이스 그립을 살균합니다. 그런 다음 바이스 그립을 사용하여 멸균 표면의 필터 케이스를 열고 케이스에서 코어를 제거합니다.
- 멸균 메스를 사용하여 필터 용지를 코어에서 잘라내고 위쪽과 아래쪽을 잘라낸 다음 솔기를 따라 잘라냅니다. 멸균 핀셋을 사용하여 필터 용지를 접은 다음 필터를 메스를 사용하여 작은 조각으로 자른다.
- 추출을 위해 미세 원심 분리기 튜브에 필터 조각을 놓습니다. 필터 용지가 살균되지 않거나 핵산이 존재할 수 있는 표면과 접촉하지 않는지 확인하십시오.
- 이전에 설명된 바와 같이 DNA절연을 수행 13 또는 상업적으로 사용 가능한 컬럼 기반 키트를 사용하여 (재료의 표참조). 나열된 상용 키트의 단계는 아래에 간략하게 설명되어 있습니다.
- 비드 튜브로 옮기고 적어도 5 분 동안 고속으로 세포 파괴기로 복종하여 샘플 내의 세포를 lyse. 원심분리기및 상피체를 멸균 미세원심분리기 튜브로 이송합니다.
- 상체(1:1 볼륨)에 용해 버퍼를 추가하고 제공된 필터(노란색)로 옮김합니다. 필터를 원심분리합니다.
- 필터를 새로운 멸균 미세 원심분리기 튜브로 옮기십시오. 준비 버퍼(400 μL), 원심분리기를 추가하고 흐름을 폐기합니다.
- 세척 버퍼(700 μL), 원심분리기를 추가하고 흐름을 폐기합니다. 그런 다음 세척 버퍼 (400 μL), 원심 분리기를 추가하고 흐름을 다시 폐기합니다.
- 필터를 새로운 멸균 미세 원심분리기 튜브로 옮기십시오. DNase/RNase 의 50 μL을 가진 엘ute는 원심분리하기 전에 실온에서 5 분 동안 앉게하십시오.
- 큐베이션 기간 동안, 수집 튜브에 배치하고 HRC 준비 용액(600 μL)을 추가하여 III-HRC 필터를 준비하고 8,000 x g에서3분의 원심 분리 단계를 수행한다.
- 준비된 필터를 멸균 미세 원심분리기 튜브로 이동합니다. 3.4.5 단계에서 이 필터및 원심분리기로 3분 동안 16,000 x g로 동경의 DNA를 전달합니다. 통해 흐름은 추출 된 DNA를 포함.
- -20°C에서 퇴적물 및 필터 모두에 대한 DNA 추출물을 저장합니다.
참고: DNA 추출물은 안정적인 온도, 제한된 광 노출 및 유해한 오염물질(14)을가정하여 -20°C에서 약 8년 동안 보관할 수 있다. - 제조업체의 프로토콜에 따라 RNA 절연을 수행합니다. RNA 추출물을 -80°C에 저장합니다.
- 비드 튜브로 옮기고 적어도 5 분 동안 고속으로 세포 파괴기로 복종하여 샘플 내의 세포를 lyse. 원심분리기및 상피체를 멸균 미세원심분리기 튜브로 이송합니다.
- 상체(1:1 볼륨)에 리시스 버퍼를 추가하고 제공된 열(노란색)으로 옮김합니다. 열을 원심분리합니다.
- 95-100% 에탄올의 동일한 볼륨을 흐름에 추가하고 5번 위아래로 피펫팅하여 혼합합니다.
- 멸균 미세 원심 분리기 튜브에 IICG 컬럼(녹색)을 놓습니다. 혼합 솔루션을 컬럼 및 원심분리기로 전송합니다.
- 세척 버퍼(400 μL), 원심분리기를 추가하고 흐름을 폐기합니다.
- DNase I 5 μL과 75 μL의 DNA 소화 버퍼를 컬럼에 넣고 실온에서 15분 동안 배양합니다.
- 준비 버퍼(400 μL), 원심분리기를 추가하고 흐름을 폐기합니다.
- 세척 버퍼(700 μL), 원심분리기를 추가하고 흐름을 폐기합니다. 그런 다음 세척 버퍼 (400 μL), 원심 분리기를 추가하고 흐름을 다시 폐기합니다.
- 컬럼을 새로운 멸균 미세 원심분리기 튜브로 옮기습니다. DNase/RNase 의 50 μL을 가진 엘루테는 원심분리기 전에 5분 동안 앉게 하십시오.
- 그 인큐베이션 기간 동안, 수집 튜브에 배치하고 HRC 준비 솔루션 (600 μL)을 추가하여 III-HRC 필터를 준비하고 8,000 x g에서3 분의 원심 분리 단계를 수행합니다.
- 준비된 필터를 멸균 미세 원심분리기 튜브로 이동합니다. 3.6.9 단계에서 이 필터및 원심분리기로 3분 동안 16,000 x g로 동경 RNA를 전달한다. 관통하는 흐름에는 추출된 RNA가 포함되어 있습니다.
참고: RNA 추출물은15의분해를 시작하기 전에 1년 동안만 저장할 수 있습니다. DNA와 RNA 추출물은 반복된 동결 해동에 의해 저하됩니다. 일부 프로토콜은 동일한샘플(16,17)에서DNA와 RNA를 모두 추출할 수 있도록허용한다.
- 발광계 또는 분광계를 사용하여 추출된 DNA 및 RNA 샘플을 정량화한다. 예를 들어 형광계 DNA 농도 값을 표 1을 참조하십시오. 예를 들어 분광계 정량화 프로토콜은 참조18을참조하십시오. 재료표에 기재된 키트를 가진 퇴적DNA 농도 값은 일반적으로 1 내지 40 ng/μL범위, 필터 DNA 농도 값은 0.5 내지 10 ng/μL. 재료표에 나열된 키트를 가진 퇴적 RNA 농도 값은 일반적으로 약 1 내지 20 ng/μL 범위, 필터 RNA 농도 값이 낮아지는 경향이 있는 동안, 일반적으로 0.5에서 5 ng/μL에 이르기까지 다양합니다.
4. 16S rRNA 라이브러리 생성
- 10% 표백제와 70% 에탄올로 작업 영역을 청소하십시오. 작업 영역은 라미나르 유량 조건(라미나르 플로우 후드)을 생성할 수 있는 밀폐된 공간이어야 합니다.
- DNA 추출물(3.5단계에서)을 사용하고, 라미나르 유량 조건 하에서 16S rRNA19의 V4 초변 영역을 증폭시키는 지구 미생물군유전체 의 웹 사이트에 설명된 것과 같은 표준 PCR 프로토콜을 사용하여 16S rRNA 앰플리콘 염기서열분석용 샘플을 준비한다.
- 앞서 설명한 대로 2% 아가로즈 젤을 준비하고17을고화시키게 한다. PCR 제품의 7 μL과 DNase 프리 워터의 13 μL을 혼합합니다. 젤 로딩 염료를 1x의 최종 농도에 추가합니다. 아가로즈가 고화되면 이 PCR 제품을 2% 아가로즈 젤에 섞어 적재하십시오.
참고: 또는, 이러한 젤이 더 빨리 실행되고 미리 만들어지기 때문에 미리 발주된 젤을 대신 사용할 수 있습니다. - 60-90분 동안 90V에서 젤을 실행하여 지구 미생물군유전체의 프로토콜을 사용하여 16S rRNA V4 앰플리턴에 대한 성공적인 증폭으로 386의 대역 크기를 확인합니다.
5. DNA 16S rRNA 라이브러리 정화
- 적절한 크기의 멸균 미세 센심분리기 튜브에서 희미한 밴드를 산출한 시료에 대해 밝은 대역과 13 μL을 산출한 샘플용 PCR 제품의 풀 10 μL.
- 불소계 또는 분광계를 사용하여 결과 풀의 농도를 확인하고 이전과 같이 2 % 아가로즈 젤을 준비하십시오. 이상적으로, 수영장은 적어도 10 ng/μL의 농도가 있어야 하며, 대부분의 샘플은 약 25 ng/μL의 농도가 있어야 합니다.
- 농도 및 부피 허용, 2% 아가로즈 젤의 우물에 약 150-200 ng로드.
- 90볼트에서 60-90분 동안 젤을 실행합니다.
- 2% 아가로즈 젤을 실행하여 풀이 있는 라이브러리를 정화합니다.
- 겔에서 386 bp DNA 밴드를 소비하고 이전에 설명된 바와 같이 시판된 키트를 사용하여 풀라이브러리를정화한다. 10mM Tris-Cl(pH 8.5)의 30 μL로 정제된 DNA를 엘테. 젤을 절단하면 PCR 앰플리턴을 실험자 및 주변 지역에 확산시키기 때문에 DNA 또는 RNA 추출과는 다른 영역에서 이 단계를 수행하여 향후 오염을 방지합니다.
- 불소계 또는 분광계를 사용하여 정제 된 풀의 농도를 확인하십시오. 정화가 잘 되면, 그 농도는 순수하지 않은 수영장의 절반 이상이어야합니다. 일반적으로 최종 농도는 5~20ng/μL 범위여야 합니다.
- 다음 세대 시퀀싱을 위해 정제된 라이브러리를 보냅니다. 선적 컨테이너에 드라이 아이스를 포함시켜 운송 중에 차갑게 유지되도록 하십시오.
6. RNA 라이브러리 생성 및 정화
- 몇몇 상용 키트는 RNA 라이브러리를 만들기 위하여 이용될 수 있습니다. 어느 것이 든 사용되든 멸균 라미나르 흐름 환경에서 작업하는 동안 작성된 대로 제조업체의 프로토콜을 따릅니다. 재료 표의 키트에 대한 프로토콜의 매우 요약된 버전은21아래에 표시됩니다.
- 첫 번째 가닥 cDNA 합성 마스터 믹스(뉴클레아제없는 물의 8 μL 및 첫 번째 스트랜드 합성 Enyzme Mix의 2 μL)를 만들어 시료에 추가합니다. 프로토콜에 지정된 조건과 함께 샘플을 열순환기에 배치합니다.
- 얼음에 두 번째 가닥 cDNA 합성 마스터 믹스 (제 2 가닥 합성 반응 버퍼의 8 μL, 4 μL 두 번째 가닥 합성 효소 믹스, 뉴클레아제없는 물의 48 μL)를 만들어 샘플에 추가합니다. 1시간 동안 16°C로 설정된 열순환기에 놓습니다.
- 제공된 구슬(144 μL)을 추가하고 2개의 80% 에탄올 세세(200 μL)를 수행하여 반응을 정화합니다.
- 제공된 TE 버퍼(53 μL)를 엘ute하고 상체의 50 μL을 깨끗한 PCR 튜브로 옮기습니다. PCR 튜브를 얼음 위에 놓습니다.
- 얼음에 최종 준비 마스터 믹스 (최종 준비 반응 버퍼의 7 μL 및 최종 준비 효소 믹스의 3 μL)를 확인하고 PCR 튜브에 추가합니다. 프로토콜에 지정된 조건과 함께 PCR 튜브를 열순환기에 배치합니다.
- 희석된 어댑터(2.5 μL), 리딩 마스터 믹스(30 μL) 및 리기션 인핸서(1 μL) 솔루션을 얼음에 섞습니다. 혼합 솔루션을 샘플에 추가하고 20°C에서 15분 동안 열순환기를 배치합니다.
- 제공된 구슬(87 μL)을 추가하고 에탄올 세세스(200 μL)를 수행하고 용출을 수행하여 반응을 정화하되, TE의 17μL만 추가한다.
- 인덱스(10 μL)와 Q5 마스터 믹스(25 μL) 용액을 추가하고 프로토콜에 설명된 조건이 있는 열순환기에 배치합니다.
- 제공된 구슬(45 μL)을 추가하고 2개의 에탄올 세세이션(200 μL)을 추가수행하고 TE의 23μL을 가진 엘루트를 추가하여 반응을 정화한다. 깨끗한 PCR 튜브에 20 μL을 전송합니다.
- 생체 분석기, 불소계 또는 분광계를 사용하여 RNA의 검출 가능한 농도에 대한 라이브러리를 확인하십시오.
- 메타전사 라이브러리를 대략 적인 비율로 풀.
- 250~400bp 사이의 절제 조각을 제외하고, 16S 라이브러리 정화에 대해 동일한 프로토콜에 따라 라이브러리를 정화한다. 16S 라이브러리에는 증폭 된 영역을 나타내는 뚜렷한 밴드가 있었지만, 여기에 결과는 얼룩입니다.
- 정수된 도서관의 농도를 이전과 같이 확인합니다.
- 정화된 도서관에 드라이 아이스를 시퀀싱 시설로 배송합니다.
참고: 대안적으로, RNA 추출물은 도서관 준비 및 시퀀싱을 위해 대학 또는 민간 기업으로 보낼 수 있다.
7. 미생물 커뮤니티 분석
- 시퀀싱이 완료되면 샘플 데이터에 액세스합니다. 사용 가능한 컴퓨터에 다운로드합니다.
참고: 이상적으로는 장치에 최소 16기가바이트의 RAM이 있어야 합니다. (Qiime2의 경우) 컴퓨팅 요구 사항에 대한 토론은 https://forum.qiime2.org/t/recommended-specifications-to-run-qiime2/9808 참조하십시오. - 16S rRNA 데이터를 분석하기 위해 모들, QIIME2 및 R과 같은 소프트웨어를 사용합니다. 예를 들어 QIIME2 16S 분석 자습서를 보려면 여기 https://docs.qiime2.org/2020.11/tutorials/moving-pictures/ 참조하십시오.
- 형이상학 (RNA) 데이터를 위해, HUMAnN2와 ATLAS를 사용하여 샘플에 존재하는 유전자와 경로를 결정합니다.
참고: 다양성과 무작위 포리스트 분석의 절정을 이루는 메타전사 파이프라인이 보충 정보 파일에나와 있습니다. 모든 명령은 Mac 사용자를 위한 터미널과 같은 명령줄을 통해 실행됩니다.
결과
DNA 및 RNA 추출의 성공은 다양한 장비 및 프로토콜을 사용하여 평가될 수 있다. 일반적으로, 어느 것의 검출 가능한 농도는 추출이 성공했다는 결론을 내리기에 충분하다고 여겨진다. 다음 표 1을 검사하면 하나를 제외한 모든 추출이 성공적이라고 불립니다. 이 단계에서의 실패는 종종 추출 중에 초기 바이오매스, 열악한 샘플 보존 또는 사람의 오류로 인해 발생합니다. 필터의 경우 농도가 검출 이하인 경우에도 추출이 성공했을 수 있습니다. 이러한 추출물이 PCR(16S를 수행하는 경우) 또는 라이브러리 준비(메타전사학)를 수행한 후 검출 가능한 농도에 대한 대역을 산출하지 않으면 실제로 실패했을 가능성이 큽니다.
16S 프로토콜을 따르는 경우, PCR 증폭에 따라 밝은 밴드, 도 1에서우물 4 및 6에서 볼 수 있듯이, 성공을 나타내며, 상단 행의 다른 우물에서 볼 수 있듯이 대역의 부족은 실패를 나타냅니다. 더욱이, 부정적인 PCR 제어를 포함하는 젤 레인의 밝은 밴드는 또한 부정적인 제어에 영향을 미치는 오염이 샘플에 영향을 미치지 않았다고 가정하는 것이 위험할 것이기 때문에 실패를 나타낼 것이다.
16S 및 메타전사학 모두에 대해, 시퀀싱의 성공은 획득된 서열의 수를 보고 평가될 수있다(도 2). 16S 샘플은 최소 1,000개의 시퀀스를 가져야 하며, 최소 5,000개 이상의 시퀀스가 있어야한다(그림 2A). 마찬가지로, 형이상학 샘플은 최소 500,000 시퀀스를 가져야하며 적어도 2,000,000명이 이상적(그림2B)이어야한다. 그 최소보다 더 적은 순서를 가진 견본은 정확하게 그들의 세균성 지역 사회를 나타내지 않을 수 있기 때문에 분석에 이용해서는 안됩니다. 그러나 최소값과 이상사이에 떨어지는 샘플은 여전히 사용할 수 있지만 많은 샘플이 해당 범위에 속하는 경우 결과가 더 신중하게 해석되어야 합니다.
후속 다운스트림 분석의 성공여부는 예상 출력 파일을 받았는지 여부에 따라 간단히 결정할 수 있습니다. 어쨌든 QIIME2 및R(그림 3)과같은 프로그램은 파쇄에 기초한 세균 계 지역 사회 간의 잠재적 차이평가를 허용해야 한다. 도 3의 데이터는 16S 및 메타전사학 분석을 위한 13개의 다른 스트림에서 21개의 다른 사이트에서 퇴적물 견본을 수집하여 얻어졌습니다. 그 21 개의 사이트 중 12 곳은 파쇄 활동의 하류에 있었고 HF +로 분류되었으며, 그 중 9 곳은 파쇄 활동의 상류 또는 파쇄가 발생하지 않은 분수령에 있었습니다. 이러한 스트림은 HF-로 분류되었다. 파쇄 활동의 존재 외에도 스트림은 그렇지 않으면 비교할 수 있었습니다.
이러한 차이는 파쇄 상태에 따라 일관된 조성 변화의 형태를 취할 수 있습니다. 이 경우, HF+와 HF-샘플은 도 3A 및 도 3B의 경우와 같이 PCoA 플롯에서 서로 클러스터될 것으로 예상됩니다. 이러한 명백한 교대체가 단지 성임 방법의 아티팩트가 아니라는 것을 확인하려면 추가 통계 분석이 필요합니다. 예를 들어, 도 3A 및 도 3B가 파쇄 상태에 기초하여 입증된 중요한 클러스터링을 기반으로 하는 거리 매트릭스에 대한 PERMANOVA22 테스트는 플롯에서 관찰된 분리가 성임 의 유물 대신 샘플의 세균 계열 간의 차이와 일치한다는 것을 의미합니다. 중요한 PERMANOVA 또는 ANOSIM 결과는 HF+ 및 HF-견본 사이 일관된 다름의 강한 표시입니다, HF+ 견본이 파쇄에 의해 영향을 받았다는 것을 표시하는 동안, 높은 p-값은 견본이 영향을 받지 않았다는 것을 표시할 것입니다. 메타전사 데이터는 마찬가지로 동일한 방법을 사용하여 시각화하고 평가할 수 있습니다.
차동 특징 (미생물 또는 기능)을 검토하면 샘플이 영향을 받았다는 증거를 밝힐 수 있습니다. 차동 피처를 결정하는 한 가지 방법은 임의의 포리스트 모델을 만드는 것입니다. 임의 포리스트 모델을 사용하여 샘플의 파쇄 상태를 올바르게 분류할 수 있는 방법을 확인할 수 있습니다. 모델이 예상보다 더 나은 성능을 경우, 이는 파쇄 상태에 따라 차이점의 추가 증거가 될 것입니다. 또한 가장 중요한 예측 변수는 시료를 올바르게 차별화하는 데 가장 중요한 특징을나타냅니다(그림 3C). 그런 다음 이러한 기능은 파쇄 상태에 따라 일관되게 다른 값을 가졌을 것입니다. 이러한 차동 기능이 결정되면 문헌을 검토하여 이전에 파쇄와 연관되었는지 확인할 수 있습니다. 그러나, 대부분의 16S rRNA 조성 데이터를 사용 했기 때문에 차동 기능을 결정 하는 연구를 찾기 어려울 수 있습니다. 따라서, 차동 기능의 의미를 평가하기 위한, 1개의 가능한 방법은 이전에 파쇄 유체에 일반적으로 사용되는 살생물제에 대한 잠재적 저항과 연관되었거나 고도로 식염수 조건을 용인하는 데 도움이 될 수 있는지 확인하는 것입니다. 또한, 관심있는 분류의 기능 프로필을 검사하면 파쇄의 영향(그림 3D)의증거를 공개 할 수 있습니다. 예를 들어, 임의의 삼림 모델에 의해 분류가 차분으로 확인되면 HF+ 샘플의 항균 저항 프로파일은 HF 샘플의 프로파일과 비교될 수 있으며 크게 다르면 생물체를 함유한 파쇄 유체가 스트림에 들어갔다는 것을 시사할 수 있습니다.
| 샘플 ID | 농도(ng/μL) |
| 1 | 1.5 |
| 2 | 1.55 |
| 3 | 0.745 |
| 4 | 0.805 |
| 5 | 7.82 |
| 6 | 0.053 |
| 7 | 0.248 |
| 8 | 0.945 |
| 9 | 1.82 |
| 10 | 0.804 |
| 11 | 0.551 |
| 12 | 1.69 |
| 13 | 4.08 |
| 14 | Below_Detection |
| 15 | 7.87 |
| 16 | 0.346 |
| 17 | 2.64 |
| 18 | 1.15 |
| 19 | 0.951 |
표 1: 불소계 1x DS DNA 고감도 분석기준 예DNA 농도. 14를 제외한 이러한 모든 샘플에 대한 추출은 검출 가능한 양의 DNA를 가지고 있기 때문에 성공한 것으로 간주될 것입니다.
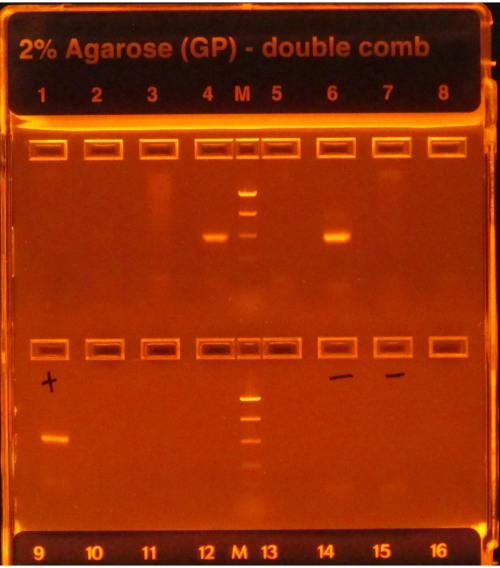
그림 1: PCR 제품이 있는 예예 e-gel. 젤은 자외선 아래에서 미리 염색되고 시각화되어 그 위에 존재하는 DNA가 빛을 발합니다. PCR은 둘 다 예상 크기의 하나의 밝은 밴드 (사다리에 따라)를 가지고 있기 때문에 첫 번째 행에서 우물 4 및 6의 샘플에 대해 일했습니다. 그들은 어떤 밴드를 생산하지 않았기 때문에, 다른 여섯 우물에서 샘플에 대한 PCR은 실패했습니다. 양수 컨트롤(첫 번째 음, 두 번째 행)에는 PCR이 제대로 수행되었음을 나타내는 밝은 밴드가 있었고 음수 컨트롤(Wells 6 및 7, 2열)에는 샘플이 오염되지 않았음을 나타내는 대역이 없었습니다. 음수가 샘플만큼 밝은 대역을 가지고 있다면, 샘플에 오염의 결과가 아닌 앰플리턴이 있다고 가정하는 것이 위험하기 때문에 PCR은 실패로 간주되었을 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
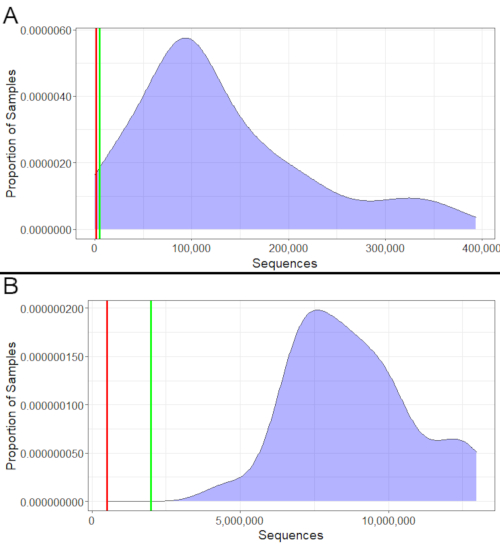
그림 2: 예 시퀀스 카운트. (A)16S 예제 시퀀스가 카운트. 거의 모든 16S 샘플은 1,000개 이상의 시퀀스를 가지고 있었습니다. 1,000개 미만의 시퀀스를 가진 극소수의 경우 세균 계류를 정확하게 나타내기 에는 시퀀스가 충분하지 않아 다운스트림 분석에서 제외되어야 합니다. 여러 시퀀스는 1,000에서 5,000 개의 시퀀스를 가지고 있었습니다. 이상적이지는 않지만 최소 값을 초과하기 때문에 여전히 사용할 수 있으며 대부분의 샘플도 5,000을 초과합니다. (B)메타전사학 예가 카운트. 모든 샘플은 최소(500,000)과 이상적인 최소(2,000,000) 시퀀스 수를 모두 초과했습니다. 따라서 시퀀싱은 모두 성공했으며 모두 다운스트림 해석에 사용될 수 있었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
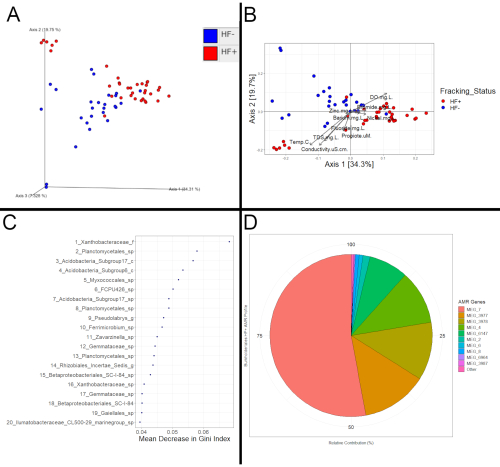
그림 3: 예제 분석. (A)QIIME2를 통해 생성및 시각화된 가중치 유니프락 거리 매트릭스로 계산된 좌표를 기반으로 하는 PCoA 플롯. (B)QIIME2로부터 내보낸 가중치 유니프락 거리 매트릭스로 계산된 좌표를 기반으로 하는 PCoA 플롯. 좌표는 Phyloseq 및 ggplot2 패키지를 사용하여 시각화되었으며, R. 메타데이터 벡터의 ggplot2 패키지는 비건 패키지를 사용하여 플롯에 장착되었습니다. 각 점은 샘플의 세균 커뮤니티를 나타내며, 더 유사한 커뮤니티 구성을 나타내는 가까운 지점을 나타냅니다. 이러한 16S 퇴적물 샘플에 대한 파쇄 상태에 따라 클러스터링이 관찰되었습니다(PERMANOVA, p=0.001). 더욱이, 벡터는 HF+ 샘플이 HF-샘플에 비해 상이한 세균성 지역 사회 조성에 대응하는 바륨, 브로마이드, 니켈 및 아연의 상부를 갖는 경향이 있음을 나타낸다. (C)세균성 풍부를 사용하여 샘플 중 파쇄 상태를 예측하는 데 사용될 수 있는 무작위 삼림 모델에 대한 최고의 예측 변수플롯. 임의포리스트 모델은 임의포리스트 패키지를 사용하여 R을 통해 만들어졌습니다. 상위 20개 예측변수는 시료를 분리하는 데 활용될 때 지니 지수의 평균 감소 의 형태로 불순물의 감소(HF+ 및 HF-시료의 측정)뿐만 아니라 표시됩니다. (D)형이상전사 데이터를 기반으로 한 버크홀더리알 프로파일의 항균 저항 프로파일을 보여주는 원형 차트. 시퀀스는 크라켄2에 먼저 추가하여 어떤 택시가 속해 있는지 를 결정했습니다. BLAST는 그 에 의해 그 불투과 말기 2.0 데이터베이스와 함께 사용되었다 어떤 항균 저항 유전자를 결정하기 위해 (의 형태로 "MEG_#") 적극적으로 표현되고 있었다. 버크홀더리알의 구성원에 의해 표현된 항균 저항 유전자는 그 때 그 분류 중 가장 널리 퍼진 것을 보기 위하여 추출되었습니다. 형이상학은 비용이 많이 들고 시간이 많이 소요되지만, 메타전사학은 16S 데이터로는 수행할 수 없는 기능 분석을 허용합니다. 특히, Kraken2는 HUMAnN2 대신이 예제 분석에 사용되었습니다. 크라켄2는 HUMAnN2보다 빠릅니다. 그러나, 그것은 단지 조성 물, 대신 조성 정보를 출력, 기여, 및 기능 (유전자) 및 HUMAnN2 같은 경로. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일: 예를 들어 메타전사 파이프라인. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 논문에 설명된 방법은 2014년과 2018년 사이에 우리 그룹이 발표한 여러연구를 통해 개발및 정제되었으며,곧 출판을 위한 논문을 제출할 3년 프로젝트에서 수생 커뮤니티에 대한 파쇄의 영향을 조사하기 위해 협력 프로젝트에 성공적으로 사용되었습니다. 이러한 메서드는 프로젝트의 나머지 기간 동안 계속 활용됩니다. 또한, 스트림 및 생태계에 파쇄가 미치는 영향을 조사하는 다른 현재 문헌은 샘플 수집, 처리 및 분석7,8,10,11에대한 유사한 방법을 설명합니다. 그러나, 그 논문 중 어느 것도 메타 전사 분석을 사용하지 않았으며,이 논문은 이러한 분석이 근처 스트림에 파쇄의 영향을 설명하는 방법을 가장 먼저 설명합니다. 또한, 샘플 수집을 위해 여기에 제시된 방법은 오염을 피하기 위해 취한 단계와 마찬가지로 보다 자세합니다.
프로토콜의 가장 중요한 단계 중 하나는 초기 샘플 수집 및 보존입니다. 현장 샘플링 및 수집에는 수집 중에 무균 또는 멸균 환경을 유지하는 것이 어려울 수 있기 때문에 특정 과제가 있습니다. 이 단계에서는 오염시 샘플을 방지하는 것이 중요합니다. 이를 위해 장갑을 착용해야 하며 멸균 용기와 도구만 시료와 접촉할 수 있어야 합니다. 또한 핵산 분해를 완화하기 위해 수집 후 즉시 얼음에 샘플을 배치해야합니다. 수집 시 상업용 핵산 방부제를 추가하면 핵산 수율을 높이고 수집 후 장기간 시료를 저장할 수 있습니다. 핵산 추출이 수행될 때마다 적절한 양의 시료를 사용하는 것이 중요하며, 추출에 사용되는 스핀 필터를 너무 많이 막을 수 있지만(그 프로토콜을 사용하는 프로토콜의 경우) 너무 적으면 수율이 낮아질 수 있습니다. 사용 되는 키트에 대 한 지침을 따라야 합니다.
필드 수집과 유사하게, 오염을 피하거나 최소화하는 것은 특히 최적 퇴적물 샘플(많은 양의 자갈 또는 바위를 함유한 샘플)이나 물 샘플과 같은 낮은 핵산 수율 샘플로 작업할 때 핵산 추출 및 샘플 준비 중에 중요합니다. 따라서 샘플 수집과 마찬가지로 오염을 줄이기 위해 이러한 모든 단계에서 장갑을 착용해야 합니다. 또한 실험실 절차 중에 사용되는 모든 작업 표면은 10 % 표백제 용액으로 닦아 70 % 에탄올 용액을 사용하여 사전에 멸균해야합니다. 파이펫팅 단계(3-6)의 경우 필터 팁을 사용하여 파이펫 자체로 인한 오염을 방지하고 비멸 상태 표면을 만질 때마다 팁이 변경되어야 합니다. 파이펫을 포함한 실험실 작업에 사용되는 모든 도구는 표백제 및 에탄올 용액으로 전후에 닦아야 합니다. 오염을 평가하기 위해, 추출 블랭크 및 네거티브 (멸균 액체)는 핵산 추출 및 PCR 반응의 모든 세트 동안 포함되어야한다. 추출 후 정량화하면 네거티브에서 검출 가능한 양의 DNA/RNA가 드러나면 충분한 샘플이 남아 있으면 추출이 반복될 수 있다. PCR에 대한 음수 샘플이 증폭을 표시하는 경우 소스를 결정하기 위해 문제 해결을 수행해야 하며 샘플을 다시 실행해야 합니다. 낮은 수준의 오염을 고려하려면 계산 분석 중에 오염 물질을 식별하고 제거할 수 있도록 추출 블랭크 및 PCR 네거티브를 시퀀스하는 것이 좋습니다. 반대로, PCR 증폭은 또한 다양한 원인으로 인해 실패할 수 있었습니다. 환경 샘플의 경우, PCR 반응의 억제는 종종 범인이며, 이는 Taq폴리머라제(23)를방해하는 다양한 물질때문일 수 있다. 억제가 의심되는 경우, PCR 등급 물(재료표참조)을 사용하여 DNA 추출물을 희석시킬 수 있다.
이 프로토콜에는 몇 가지 주목할 만한 제한 사항과 잠재적인 어려움이 있습니다. 샘플 수집은 물과 퇴적물 샘플 모두에 어려울 수 있습니다. 충분한 바이오매스를 얻으려면 이상적으로 1 L의 스트림 워터를 필터를 통해 밀어내야 합니다. 필터의 기공은 미생물을 포착하기 위해 작아야하지만 퇴적물을 함정에 빠뜨릴 수도 있습니다. 최근 강우로 인해 많은 퇴적물이 물에 있는 경우 필터가 막히면 필터를 통해 전체 부피를 밀어내기가 어려워질 수 있습니다. 퇴적물 컬렉션의 경우 수집 중에 퇴적물의 깊이를 추정하는 것이 어려울 수 있습니다. 더욱이, 모은 퇴적물이 주로 토양인지 확인하는 것이 중요하며, 자갈과 바위는 핵산 수율을 낮추고 미생물 공동체의 정확한 표현이 아닐 수 있기 때문에 주로 토양이다. 마지막으로, 특히 방부제를 사용하지 않는 경우, 샘플이 수집 후 얼음에 보관되는 것이 중요합니다.
이 프로토콜은 형이상술과 16S 실험실 프로토콜을 모두 다루지만 이 두 가지 방법은 프로세스와 제공하는 데이터 유형 모두에서 매우 다르다는 점을 강조해야 합니다. 16S rRNA 유전자는 박테리아와 고고학에서 매우 보존되는 일반적으로 표적화된 영역이며 샘플에서 세균 커뮤니티를 특성화하는 데 유용합니다. 표적적이고 구체적인 접근법이지만, 종 수준 해상도는 종종 달성할 수 없으며 새로 다진 종이나 균주를 특성화하는 것은 어렵습니다. 반대로, 메타전사학은 샘플 내에 존재하는 모든 활성 유전자와 미생물을 포착하는 광범위한 접근법입니다. 16S는 식별을 위한 데이터만 제공하는 반면, 형이상학은 발현 유전자 및 대사 경로와 같은 기능성 데이터를 제공할 수 있습니다. 둘 다 귀중하고 결합할 때, 어떤 박테리아가 존재하는지 그리고 어떤 유전자가 표현되고 있는지 밝힐 수 있습니다.
이 논문에서는 파쇄를 연구하는 맥락에서 16S rRNA 및 메타전사 분석 모두에 대한 현장 수집 및 샘플 처리 방법을 설명합니다. 또한 저바이오매스 샘플과 장기 저장을 위한 고품질 DNA/RNA에 대한 수집 방법을 자세히 설명합니다. 여기에 설명된 방법은 파쇄가 미생물 커뮤니티의 구조와 기능을 검토하여 주변 스트림에 미치는 영향을 배우기 위한 노력의 샘플 수집 및 처리에 대한 당사의 경험의 절정입니다. 미생물은 교란에 신속하게 반응하고, 결과적으로 미생물이 존재하고 그들이 표현하는 유전자는 생태계에 파쇄의 영향에 대한 정보를 제공 할 수 있습니다. 전반적으로 이러한 방법은 파쇄가 이러한 중요한 생태계에 미치는 영향에 대한 우리의 이해에 매우 유용할 수 있습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
저자는 이러한 방법의 개발을 주도 프로젝트에 대한 자금 원천을 인정하고 싶습니다, 그 소스와 함께: 하워드 휴즈 의료 연구소 (http://www.hhmi.org) 유치원 및 학부 과학 교육 프로그램을 통해, 뿐만 아니라 국립 과학 재단에 의해 (http://www.nsf.gov) NSF 상을 통해 DBI-1248096 및 CBET-1805549.
자료
| Name | Company | Catalog Number | Comments |
| 200 Proof Ethanol | Thermo Fisher Scientific | A4094 | 400 mL need to be added to Buffer PE (see Qiagen QIAQuck Gel Extraction kit protocol) and 96 mL needs to be added to the DNA/RNA Wash Buffer (see ZymoBIOMICS DNA/RNA Miniprep kit protocol). Additional ethanol is needed for the ZymoBIOMICS DNA/RNA Miniprep and NEBNext® Ultra™ II RNA Library Prep with Sample Purification Beads kits. |
| Agarose | Thermo Fisher Scientific | BP1356-100 | 100 g per bottle. 0.6 g of agarose would be needed to make one 2% 30 mL gel. |
| Disinfecting Bleach | Walmart (Clorox) | No catalog number | Use a 10% bleach solution for cleaning the work area before and after lab procedures |
| DNA gel loading dye | Thermo Fisher Scientific | R0611 | Each user-made (i.e. non-e-gel) should include loading dye with all of the samples in the ratio of 1 µL dye to 5 µL sample |
| DNA ladder | MilliporeSigma | D3937-1VL | A ladder should be run on every gel/e-gel |
| DNA/RNA Shield (2x) | Zymo Research | R1200-125 | 3 mL per sediment sample (50 mL conical) and 2 mL per water sample (filter) |
| Ethidium bromide | Thermo Fisher Scientific | BP1302-10 | Used for staining user-made e-gels |
| Forward Primer | Integrated DNA Technologies (IDT) | 51-01-19-06 | 0.5 µL per PCR reaction |
| Isopropanol | MilliporeSigma | 563935-1L | Generally less than 2 mL per library. Volume needed varies by mass of excised gel fragment (see Qiagen QIAQuick Gel Extraction kit protocol). |
| PCR-grade water | MilliporeSigma | 3315932001 | 13 µL per PCR reaction (assuming 1 µL of sample DNA template is used) |
| Platinum Hot Start PCR Master Mix (2x) | Thermo Fisher Scientific | 13000012 | 10 µL per PCR reaction |
| Reverse Primer | Integrated DNA Technologies (IDT) | 51-01-19-07 | 0.5 µL per PCR reaction |
| TBE Buffer (Tris-borate-EDTA) | Thermo Fisher Scientific | B52 | 1 L of 10x TBE buffer (30 mL of 1x TBE buffer would be needed to make one 30 mL gel) |
| 1 L bottle | Thermo Fisher Scientific | 02-893-4E | One needed per stream (the same bottle can be used for multiple streams if it is sterilized between uses) |
| 1.5 mL Microcentrifuge tubes | MilliporeSigma | BR780400-450EA | 5 microcentrifuge tubes are needed per DNA extraction and an additional 3 are needed to purify RNA (see ZymoBIOMICS DNA/RNA Miniprep kit protocol) |
| 2% Agarose e-gel | Thermo Fisher Scientific | G401002 | Each gel can run 10 samples (so 9 with a PCR negative and 8 if the extraction negative is run on the same gel) |
| 50 mL Conicals | CellTreat | 229421 | 1 50 mL conical needed per sediment samples |
| 500 mL Beaker | MilliporeSigma | Z740580 | Only 1 needed (for flame sterilization) |
| Aluminum foil | Walmart (Reynolds KITCHEN) | No number | Aluminum foil can be folded and autoclaved. The part not exposed to the environment can then be used as a sterile, DNA and RNA free surface for processing filters (one folded piece per filter to avoid cross-contamination) |
| Autoclave | Gettinge | LSS 130 | Only one needed |
| Centrifuge | MilliporeSigma | EP5404000138-1EA | Only 1 needed |
| Cooler | ULINE | S-22567 | Just about any cooler can be used. This one is listed due to being made of foam, making it lighter and thus easier to take along for field sampling. |
| Disruptor Genie | Bio-Rad | 3591456 | Only one needed |
| Electrophoresis chamber | Bio-Rad | 1664000EDU | Only 1 needed |
| Electrophoresis power supply | Bio-Rad | 1645050 | Only 1 needed |
| Freezer (-20 C) | K2 SCIENTIFIC | K204SDF | One needed to store DNA extracts |
| Freezer (-80 C) | K2 SCIENTIFIC | K205ULT | One needed to store RNA extracts |
| Gloves | Thermo Fisher Scientific | 19-020-352 | The catalog number is for Medium gloves. |
| Heat block | MilliporeSigma | Z741333-1EA | Only one needed |
| Lab burner | Sterlitech | 177200-00 | Only one needed |
| Laminar Flow Hood | AirClean Systems | AC624LFUV | Only 1 needed |
| Library purification kit | Qiagen | 28704 | One kit has enough for 50 reactions |
| Magnet Plate | Alpaqua | A001219 | Only one needed |
| Microcentrifuge | Thermo Fisher Scientific | 75004061 | Only one needed |
| Micropipette (1000 µL volume) | Pipette.com | L-1000 | Only 1 needed |
| Micropipette (2 µL volume) | Pipette.com | L-2 | Only 1 needed |
| Micropipette (20 µL volume) | Pipette.com | L-20 | Only 1 needed |
| Micropipette (200 µL volume) | Pipette.com | L-200R | Only 1 needed |
| NEBNext Ultra II RNA Library Prep with Sample Purification Beads | New England BioLabs Inc. | E7775S | One kit has enough reagents for 24 samples. |
| Parafilm | MilliporeSigma | P7793-1EA | 2 1" x 1" squares are needed per filter |
| PCR Tubes | Thermo Fisher Scientific | AM12230 | One tube needed per reaction |
| Pipette tips (for 1000 µL volume) | Pipette.com | LF-1000 | Pack of 576 tips |
| Pipette tips (for 20 µL volume) | Pipette.com | LF-20 | Pack of 960 tips |
| Pipette tips (for 200 µL volume) | Pipette.com | LF-250 | Pack of 960 tips |
| PowerWulf ZXR1+ computer cluster | PSSC Labs | No number | This is just an example of a supercomputer powerful enough to perform metatranscriptomics analysis in a timely manner. Only one needed. |
| Qubit fluorometer starter kit | Thermo Fisher Scientific | Q33239 | Comes with a Qubit 4 fluorometer, enough reagent for 100 DNA assays, and 500 Qubit tubes |
| Scoopula | Thermo Fisher Scientific | 14-357Q | Only one needed |
| Sterile blades | AD Surgical | A600-P10-0 | One needed per filter |
| Sterivex-GP Pressure Filter Unit | MilliporeSigma | SVGP01050 | 1 filter needed per water sample |
| Thermocycler | Bio-Rad | 1861096 | Only one needed |
| Vise-grip | Irwin | 2078500 | Only one needed (for cracking open the filters) |
| Vortex-Genie 2 | MilliporeSigma | Z258415-1EA | Only 1 needed |
| WHIRL-PAK bags | ULINE | S-22729 | 1 needed per filter |
| ZymoBIOMICS DNA/RNA Miniprep kit | Zymo Research | R2002 | One kit has enough reagents for 50 samples. |
참고문헌
- The process of unconventional natural gas production. US EPA Available from: https://www.epa.gov/uog/process-unconventional-natural-gas-production (2013)
- Brittingham, M. C., Maloney, K. O., Farag, A. M., Harper, D. D., Bowen, Z. H. Ecological risks of shale oil and gas development to wildlife, aquatic resources, and their habitats. Environmental Science & Technology. 48 (19), 11034-11047 (2014).
- McBroom, M., Thomas, T., Zhang, Y. Soil erosion and surface water quality impacts of natural gas development in East Texas, USA. Water. 4 (4), 944-958 (2012).
- Maloney, K. O., Weller, D. E. Anthropogenic disturbance, and streams: land use and land-use change affect stream ecosystems via multiple pathways. Freshwater Biology. 56 (3), 611-626 (2011).
- Meyer, J. L., et al. The contribution of headwater streams to biodiversity in river networks1. JAWRA Journal of the American Water Resources Association. 43 (1), 86-103 (2007).
- Alexander, R. B., Boyer, E. W., Smith, R. A., Schwarz, G. E., Moore, R. B. The role of headwater streams in downstream water quality. Journal of the American Water Resources Association. 43 (1), 41-59 (2007).
- Ulrich, N., et al. Response of aquatic bacterial communities to hydraulic fracturing in Northwestern Pennsylvania: A five-year study. Scientific Reports. 8 (1), 5683 (2018).
- Chen See, J. R., et al. Bacterial biomarkers of Marcellus shale activity in Pennsylvania. Frontiers in Microbiology. 9, 1697 (2018).
- Rausch, P., et al. Comparative analysis of amplicon and metagenomic sequencing methods reveals key features in the evolution of animal metaorganisms. Microbiome. 7 (1), 133 (2019).
- Louca, S., Doebeli, M., Parfrey, L. W. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem. Microbiome. 6 (1), 41 (2018).
- Trexler, R., et al. Assessing impacts of unconventional natural gas extraction on microbial communities in headwater stream ecosystems in Northwestern Pennsylvania. Frontiers in Microbiology. 5, 522 (2014).
- Mumford, A. C., et al. Shale gas development has limited effects on stream biology and geochemistry in a gradient-based, multiparameter study in Pennsylvania. Proceedings of the National Academy of Sciences. 117 (7), 3670-3677 (2020).
- JoVE Core Biology DNA Isolation. Journal of Visualized Experiments Available from: https://www.jove.com/cn/science-education/10814/dna-isolation (2020)
- Oxford Gene Technology DNA Storage and Quality. OGT Available from: https://www.ogt.com/resources/literature/403_dna_storage_and_quality (2011)
- ThermoFisher SCIENTIFIC Technical Bulletin #159: Working with RNA. Thermoscientific Available from: https://www.thermofisher.com/us/en/home/references/ambion-tech-support/nuclease-enzymes/general-articles/working-with-rna.html (2020)
- QIAGEN AllPrep DNA/RNA Mini Kit. Qiagen Available from: https://www.qiagen.com/us/products/discovery-and-translational-research/dna-rna-purification/multianalyte-and-virus/allprep-dnarna-mini-kit/#orderinginformation (2020)
- ZymoBIOMICS DNA/RNA Miniprep Kit. Zymo Research Available from: https://www.zymoresearch.com/products/zymobiomics-dna-rna-miniprep-kit (2020)
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- 16S Illumina amplicon protocol: Earth microbiome project. Earth microbiome project Available from: https://earthmicrobiome.org/protocols-and-standards/16s/ (2018)
- Gel Purification: Binding, washing and eluting a sample | Protocol. Journal of Visualized Experiments Available from: https://www.jove.com/v/5063/gel-purification (2020)
- New England Biolabs protocol for the use with NEBNext Poly(A) mRNA magnetic isolation module (E7490) and NEBNext Ultra II RNA library prep kit for Illumina (E7770, E7775). New England Biolabs Available from: https://www.neb.com/protocols/2017/03/04/protocol-for-use-with-purified-mrna-or-rrna-depleted-rna-and-nebnext-ultra-ii-rna-library-prep-ki (2020)
- Anderson, M. J. Permutational multivariate analysis of variance (PERMANOVA). Wiley StatsRef: Statistics Reference Online. , 1-15 (2017).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유