Method Article
使用微生物分子特征评估水力压裂对溪流的影响
摘要
在这里,我们提出了一个协议,通过分析水分和沉积物微生物群落来调查水力压裂对附近溪流的影响。
摘要
水力压裂 (HF) 通常称为"压裂",它使用高压水、沙子和化学物质的混合物压裂岩石,释放石油和天然气。这一进程彻底改变了美国能源工业,因为它提供了以前无法获得的资源,现在生产的天然气占美国天然气总量的三分之二。尽管压裂对美国经济产生了积极影响,但一些研究强调了压裂对环境的有害影响。特别令人关切的是压裂对水流的影响,由于压裂对整个流域的健康影响过大,因此尤为重要。这些溪流中的细菌可以用作流健康指标,因为在受干扰的溪流中存在的细菌及其丰度预计将与本来可比但不受干扰的溪流中的细菌不同。因此,本协议旨在使用细菌群落来确定溪流是否受到压裂的影响。为此,必须收集来自压裂附近的溪流(可能受到影响)和上游或不同压裂活动分水岭(未受影响)的沉积物和水样。然后,这些样本需要提取核酸、库准备和测序,以调查微生物群落的组成。随后可以采用相关分析和机器学习模型来识别哪些特征可以解释社区的变化,以及识别用于压裂影响的预测生物标志物。这些方法可以揭示地下水流中微生物群落的各种差异,基于压裂的接近程度,并作为今后调查压裂活动对环境影响的基础。
引言
水力压裂(HF)或"压裂"是一种天然气开采方法,随着对化石燃料的需求持续上升,这种开采方式变得越来越普遍。这项技术包括使用大功率钻井设备将水、沙子和化学物质混合注入富含甲烷的页岩矿床中,通常释放被困气体1。
由于这些非常规的采伐技术相对较新,因此必须调查这种做法对附近水道的影响。压裂活动要求清理大片土地,用于设备运输和井垫建设。每口井垫2必须清理约1.2至1.7公顷的土地,这可能会影响系统3的径流和水质。压裂液的确切化学成分缺乏透明度,包括使用什么杀菌剂。此外,压裂废水往往是高盐度2。此外,废水可能含有金属和天然放射性物质2。因此,压裂液因人为错误或设备故障而泄漏和溢出的可能性令人关注。
众所周知,溪流生态系统对周围景观的变化非常敏感,对于维持整个流域内的生物多样性5和适当的养分循环6非常重要。微生物是淡水溪流中最丰富的生物,因此对营养循环、生物降解和初级生产至关重要。微生物群落的组成和功能由于对扰动的敏感性而成为获取生态系统信息的绝佳工具,最近的研究表明,基于接近压裂活性的观察细菌组合发生了明显变化。例如,贝耶林基亚、伯克霍尔德里亚和甲基细菌被确定为在压裂附近的溪流中富集,而伪心电图、硝基和罗多细菌在不接近压裂7的溪流中富集。
下一代测序的16S核糖核糖核瘤RNA(rRNA)基因是一种负担得起的方法来确定细菌群落组成,这是更快和更便宜的全基因组测序方法9。分子生态学领域的一个常见做法是使用16S rRNA基因高度可变的V4区域进行测序分辨率,通常下降到具有广泛识别9的属水平,因为它是不可预知的环境样本的理想选择。这项技术已在已发表的研究中广泛应用,并已成功应用于识别压裂作业对水生环境的影响。然而,值得注意的是,细菌有不同的拷贝数的16S rRNA基因,这影响其检测到的丰度10。有几个工具来解释这一点,但他们的功效是值得怀疑的10。另一种在流行率上迅速增长并缺乏这种弱点的做法是元定性测序,其中对所有RNA进行测序,使研究人员能够识别活性细菌及其基因表达。
因此,与先前发表的研究7、8、11、12的方法不同,本协议还涵盖了用于研究微生物群落功能(元体学)的样本收集、保存、处理和分析。这里详细的步骤使研究人员能够看到压裂对微生物在其溪流中表达的基因和途径(包括抗微生物抗药性基因)产生了什么影响(如果有的话)。此外,还改进了样品收集的详细程度。虽然一些步骤和笔记在有经验的研究人员看来似乎是显而易见的,但对于那些刚刚开始研究的人来说,它们可能是无价的。
在此,我们描述了样品收集和处理的方法,以产生细菌遗传数据,作为根据我们的实验室多年的经验来研究压裂对附近溪流的影响的一种手段。这些数据可用于下游应用,以识别与压裂状态相对应的差异。
研究方案
1. 收集沉积物样本用于提取核酸
- 将无菌的 50 mL 圆锥管浸入溪水中。在样品采集过程中戴手套,以避免引入不必要的人类污染。执行此步骤从岸边或朝上,如果在水中。
- 当圆锥管被淹没时,取下盖子,并用它从1到3厘米的深度中挖出约3mL的沉积物到圆锥管中。
- 从水中取出锥形管并倾倒所有水,但覆盖沉积物样本(约 1 mL)的薄层除外。
- 使用 1000 μL 移液器和适当的移液器提示,在收集的样品中加入 3 mL 的 DNA/RNA 防腐剂(参见防腐剂规格 材料表 )。将移液器提示保存在无菌移液器尖端盒中,仅在使用前立即将其连接并丢弃。倒置封顶锥形管10次,以确保防腐剂和样品被彻底混合。
注步骤 1.4 是没有必要的,但如果以后要从沉积物中提取RNA,则强烈建议使用。 - 将样品放在冰上,以进行其余样品采集。从收集回来后,如果样品要用于 16S 分析 (DNA), 或 -70 °C,如果样品要用于元数据学分析 (RNA),则存放在 -20 °C 的冰柜中。
2. 用于提取核酸的过滤器集合
- 取下无菌 1 L 瓶盖。当面对上游或从岸边,填充瓶与流水到顶部,然后倾倒出来。重复此过程两次,以调节瓶子。第四次装满整瓶酒并盖上盖子。
注:如果重复使用1L瓶,可以用10%的漂白剂冲洗2分钟进行消毒,然后用去离子水冲洗3次,然后用70%乙醇冲洗一次,最后用设置自动清洁:30分钟暴露时间在121.1°C和15分钟干燥时间。在自动包装过程中,瓶子上的盖应非常松动,以避免瓶子在这个过程中被压缩。 - 一旦在稳定的表面上,使用无菌的Luer锁注射器,并绘制一个完整的体积。然后将注射器连接到无菌和脱氧核糖核酸/无RNA 1.7 厘米直径的聚醚硫酮过滤器,孔径为 0.22 μm,通过按下柱塞一路将整个体积推入过滤器。重复此过程,直到瓶子中收集的总体积 (1 L) 通过过滤器。
注:如果跟踪通过过滤器推入的总水量,注射器的体积可能是可变的。但是,一般来说,首选 60 mL。虽然1L是理想的,传闻,至少200ml的体积可能仍然收集足够的生物质(假设每mL20,000个细胞)提取DNA和RNA。 - 将大约 20 mL 的空气抽入注射器并推动其通过过滤器,从过滤器中取出多余的水。
注意:如果执行第 2.4 步,这将有助于防止防腐剂丢失。 - 使用 P1000 微管,通过通过过滤器较大的开口(将其连接到注射器上)将其放电,同时水平握住滤清器,加入 2 mL 的 DNA/RNA 防腐剂。当移液器处于抑郁状态时,移液器的尖端应位于过滤器的桶内,以确保防腐剂进入过滤器。每次使用后更改提示。
注:与沉积物收集一样,此步骤是没有必要的,但强烈建议以后增加核酸产量,尤其是RNA。 - 剥下一平方的石蜡薄膜,紧紧包裹在过滤器的每个开口/末端,以密封。将石蜡薄膜包裹的过滤器放入无菌样品袋中,然后在收集过程中将整个包装在冰上。
注意:确保用于包装过滤器的侧面是无菌的,即以前未暴露在环境中。 - 取样后,将过滤器存储在 -20 °C 的 16S 或 -70 °C 的元转录学。
3. 核酸提取和量化
- 在开始样品转移之前,用 10% 漂白剂和 70% 乙醇清洁工作区域。
- 对于沉积物(从第 1.5 步开始),通常使用 ±0.25 克样品。火焰通过将金属工具浸入 70% 乙醇的烧嘴中,并在样品之间燃烧乙醇来消毒。
- 对于过滤器(从第 2.6 步开始),将滤纸移入无菌管进行提取。要做到这一点,请按照下面的步骤进行操作。
- 通过折叠铝箔创建无菌、DNA 和 RNA 自由表面,使折叠的内部部分不暴露在外部环境中,并自动将折叠件与设置进行解密:121.1 °C 和 5 分钟干燥时间。
- 用 70% 乙醇和明火消毒小火。然后使用 vise 抓地力打破无菌表面上的滤芯外壳,从外壳中取出核心。
- 使用无菌手术刀将滤纸从核心切开,在顶部和底部切片,然后沿着接缝切开。使用无菌钳子折叠滤纸,然后使用手术刀将滤清器切成小块。
- 将过滤件放入微中心管中进行提取。确保滤纸不会接触到任何未消毒或可能存在核酸的表面,因为这会导致样品受到不必要的污染。
- 执行前述 13 描述的 DNA 隔离,或使用市售的基于列的工具包(参见 材料表)。下面简要介绍了列出的商业工具包的步骤。
- 将样本中的细胞转移到珠子管中,并使其高速受细胞干扰至少5分钟,从而对样本中的细胞进行取景。离心机并将超导管转移到无菌微中心管中。
- 将裂解缓冲器添加到超高纳特(1:1 体积),并转移到提供的过滤器(黄色)。离心过滤器。
- 将过滤器转移到新的无菌微中心管。添加制备缓冲器 (400 μL)、离心机,并丢弃流经。
- 加入洗涤缓冲器 (700 μL)、离心机,并丢弃流经。然后加入洗涤缓冲器 (400 μL),离心机,并再次丢弃流过。
- 将过滤器转移到新的无菌微中心管。Elute 配有 50 微升 DNase/RNase 免费水,在离心前在室温下坐 5 分钟。
- 在催化期间,准备 III-HRC 过滤器,将其放入收集管中,并在其中添加 HRC 预置解决方案 (600 μL),然后在 8,000 x g下进行 3 分钟的离心步骤。
- 将准备好的过滤器移到无菌微中心管上。将脱氧核糖核酸从第 3.4.5 步转移到此过滤器和离心机,速度为 16,000 x g,3 分钟。流经包含提取的DNA。
- 在 -20 °C 下存储沉积物和过滤器的 DNA 提取物。
注:DNA提取物可以在-20°C下储存约8年,假设温度稳定,光线暴露有限,且无有害污染物14。 - 根据制造商的协议执行 RNA 隔离。将RNA提取物储存在-80°C。
- 将样本中的细胞转移到珠子管中,并使其高速受细胞干扰至少五分钟,从而对样本中的细胞进行取景。离心机并将超导管转移到无菌微中心管中。
- 将裂解缓冲器添加到超高纳特(1:1 体积)并转移到所提供的列(黄色)。离心柱。
- 将等量的 95-100% 乙醇添加到流经中,通过上下管道混合五次。
- 将 IICG 柱(绿色)放在无菌微中心管上。将混合溶液转移到柱子和离心机。
- 加入洗涤缓冲器 (400 μL)、离心机,并丢弃流经。
- 将 5 μL 的 DNase I 和 75 μL 的 DNA 消化缓冲添加到柱中,并在室温下孵育 15 分钟。
- 添加预备缓冲器 (400 μL)、离心机,并丢弃流经。
- 加入洗涤缓冲器 (700 μL)、离心机,并丢弃流经。然后加入洗涤缓冲器 (400 μL),离心机,并再次丢弃流过。
- 将柱子转移到新的无菌微中心管。Elute 配有 50 微升的 DNase/RNase 免费水,让离心前坐 5 分钟。
- 在潜伏期,准备III-HRC过滤器,将其放入收集管中,并在其中添加HRC预制液(600微升),然后在8,000 x g下进行3分钟的离心步。
- 将准备好的过滤器移到无菌微中心管上。将 el2 RNA 从第 3.6.9 步转移到此过滤器和离心机,速度为 16,000 x g,3 分钟。流经包含提取的RNA。
注:RNA提取物只能储存一年,然后才开始降解15。DNA和RNA提取物都通过反复冷冻解冻而降解。有些协议允许从同一样本16,17中提取DNA和RNA。
- 使用荧光仪或光谱光度计对提取的DNA和RNA样本进行量化。例如,请参阅表 1的荧计 DNA 浓度值。例如光谱光度计量化协议,请参阅参考18。材料表中所列工具包的沉积物DNA浓度值一般在1至40 ng/μL之间,而过滤器DNA浓度值一般在0.5至10ng/μL之间。材料表中所列套件的沉积物RNA浓度值一般在1至20ng/μL之间,而过滤RNA浓度值往往较低, 通常范围从 0.5 到 5 ng/μL。
4. 16S rRNA 库创建
- 用 10% 漂白剂和 70% 乙醇清洁工作区域。工作区域应该是一个封闭的空间,能够产生层流条件(层流罩)。
- 使用 DNA 提取物(从第 3.5 步开始),使用标准 PCR 协议为 16S rRNA 放大器测序准备样本,例如地球微生物群网站上描述的在层压流条件下放大 16S rRNA19 的 V4 超变区域。
- 准备一个2%的玫瑰凝胶如前所述,让它凝固17。混合 7 μL 的 PCR 产品和 13 μL 的 DNase 免费水。将凝胶加载染料添加到最终浓度为 1 倍。一旦糖凝固,将此 PCR 产品混合在 2% 的玫瑰凝胶上。
注意:或者,可以使用预铸凝胶,因为这些凝胶运行得更快,并且是预制的。 - 使用地球微生物群协议,在 90 V 处运行凝胶 60-90 分钟,以检查带大小为 386,作为 16S rRNA V4 放大器的成功放大。
5. DNA 16S rRNA 库净化
- 池 10 μL 的 PCR 产品用于产生明亮频带的样品,13 μL 用于在适当大小的无菌微中心管中产生微弱频段的样品。
- 使用荧光仪或光谱光度计检查产生的池的浓度,并像以前一样准备 2% 的玫瑰凝胶。理想情况下,池的浓度应至少为 10 ng/μL,大多数样品的浓度应约为 25 ng/μL。
- 浓度和体积允许,在2%的玫瑰凝胶井中装载约150-200 ng。
- 在 90 伏特下运行凝胶 60-90 分钟。
- 通过运行 2% 的阿加罗斯凝胶来净化池中库。
- 从凝胶中取出386个基点的DNA带,并使用之前描述的20个商业可用的试剂盒净化池库。用 30 微升 10 m Tris-Cl (pH 8.5) 来提取纯化的 DNA。在与 DNA 或 RNA 提取不同的区域执行此步骤,以防止未来的污染,因为切割凝胶将把 PCR 放大器传播到实验者和周围区域。
- 使用荧光仪或光谱光度计检查净化池的浓度。如果净化顺利,其浓度应至少是未净化池的一半。一般来说,最终浓度应介于 5 到 20 ng/μL 之间。
- 发送纯化库进行下一代测序。确保运输过程中保持寒冷,在集装箱中包括干冰。
6. RNA 库创建和净化
- 可以使用几个商业工具包创建 RNA 库。无论使用哪种,在无菌层压流环境中工作时,请按照制造商的流程书写。 材料表 中套件协议的非常总结版本呈现在21以下。
- 制作第一股 cDNA 合成主组合(8 μL 无核糖水和第 2 μL 第一链合成 Enyzme 混合),并将其添加到样品中。将样品放在符合协议中指定条件的热循环器中。
- 使第二股cDNA合成主组合(8 μL的第二股合成反应缓冲器,4微升第二链合成酶混合,和48微升无核糖水),并添加到样品中。放置在温度循环器设置为 16 °C 一个小时。
- 通过添加提供的珠子 (144 μL) 和进行两次 80% 乙醇洗涤 (200 μL) 来净化反应。
- 配备提供的 TE 缓冲器 (53 μL), 并将 50 μL 的超高纳特转移到干净的 PCR 管中。将 PCR 管放在冰上。
- 在冰上制作端准备主组合(7 μL 的末端准备反应缓冲器和 3 μL 的端准备酶混合),并将其添加到 PCR 管中。将 PCR 管放入符合协议中指定条件的热循环器中。
- 将稀释适配器 (2.5 μL)、配体主混合 (30 μL) 和 Ligation 增强器 (1 μL) 解决方案混合在冰上。将混合溶液添加到样品中,并在 20 °C 下放在热循环器中 15 分钟。
- 通过添加提供的珠子 (87 μL) 和执行乙醇洗涤 (200 μL) 和洗涤像以前一样净化反应, 除了只添加 17 μL 的 TE.
- 添加指数 (10 μL) 和第 5 季度主混合 (25 μL) 解决方案,并将其放置在符合协议中描述条件的热循环器中。
- 通过添加所提供的珠子 (45 μL) 并添加两个乙醇洗涤 (200 μL) 和 23 μL 的 TE 来净化反应。将 20 μL 转移到干净的 PCR 管中。
- 使用生物分析仪、荧光仪或光谱光谱仪检查库,了解RNA的可检测浓度。
- 以大致等值的比例将元描述性库池入池中。
- 按照 16S 库净化的相同协议净化库,但 250 到 400 桶/日之间的切除片段除外。虽然 16S 库有一个代表放大区域的不同波段,但此处的结果是涂片。
- 检查纯化库的浓度。
- 将纯化后的库与干冰一起运送到测序设施。
注:或者,RNA提取物可以发送到大学或私人公司进行图书馆准备和测序。
7. 微生物群落分析
- 测序完成后,访问样本数据。将其下载到可用的计算机。
注:理想情况下,该设备应具有至少 16 GB 的 RAM。有关计算要求的讨论(对于 Qiime2),请参阅 https://forum.qiime2.org/t/recommended-specifications-to-run-qiime2/9808。 - 使用软件(如飞蛾、QIIME2 和 R)分析 16S rRNA 数据。请参阅此处 https://docs.qiime2.org/2020.11/tutorials/moving-pictures/ 示例 QIIME2 16S 分析教程。
- 对于甲基定性学 (RNA) 数据,请使用 HUMANN2 和 ATLAS 来确定样本中存在哪些基因和通路。
注:在 补充信息文件中提供了一个以多样性和随机森林分析为高潮的元数据学管道示例。所有命令都通过命令线运行,例如,Mac 用户的终端。
结果
DNA和RNA提取的成功率可以使用各种设备和协议来评估。一般来说,任何可检测到的浓度都足以得出提取成功的结论。然后检查 表 1, 除表 1 外,所有提取都将被称为成功。此步骤的失败通常是由于初始生物量低、样品保存不良或提取过程中的人为错误。在过滤器的情况下,即使浓度低于检测,萃取也可能成功。如果这些提取物没有产生PCR的带(如果做16S)或库准备后可检测到的浓度(元数据学),那么它们可能确实失败了。
如果遵循 16S 协议,则 PCR 放大后的明亮频段(如图 1 中的 4 号和 6 号井)表示成功,而上排其他油井中显示的频段不足则表示失败。此外,凝胶车道中含有负 PCR 控制的明亮带也表示故障,因为假设影响负控制的污染不会影响样品是有风险的。
对于 16S 和元定性学,测序的成功率可以通过查看获得的序列数(图 2)来评估。16S 样品应至少具有 1,000 个序列,其中至少 5,000 个序列是理想的(图 2A)。同样,元数据学样本至少应有50万个序列,其中至少200万个序列是理想的(图2B)。序列少于这些最低序列的样本不应用于分析,因为它们可能无法准确代表其细菌群落。但是,在最小值和理想值之间可以使用样本,但如果许多样本落在该范围内,则应更谨慎地解释结果。
后续下游分析的成功与否仅取决于是否获得了预期的输出文件。无论如何,项目,如QIIME2和R(图3),应允许评估细菌群落之间潜在的重大差异的基础上压裂。 图3 的数据是通过收集13个不同流的21个不同地点的沉积物样本,进行16S和元数据学分析获得的。在这21个地点中,有12个位于压裂活动下游,被列为HF+,其中9个地点位于压裂活动的上游或没有压裂的流域内;这些流被归类为 HF-。除了压裂活动的存在,溪流是可比的。
这些差异可以采取基于压裂状态的一致构图变化的形式。如果是这样的话,HF+和HF样本将有望在PCOA图中彼此分离,图3A和图3B就是如此。为了确认这些明显的转变不仅仅是协调方法的一个伪影,还需要进一步的统计分析。例如,PERMANOVA22对图 3A 和图 3B的距离基质进行测试,该测试基于基于压裂状态的显露显著聚类,这意味着在绘图中观察到的分离与样本细菌群落之间的差异一致,而不是协调的产件。 PERMANOVA 或 ANOSIM 的显著结果是,HF® 和 HF 样本之间一致差异的有力指示,这将表明 HF® 样本受到压裂的影响,而高 p 值则表明样本未受到影响。类似,可以用同样的方法对元数据进行可视化和评估。
检查微分特征(微生物或功能)可以揭示样品也受到影响的证据。确定微分特征的一种方法是创建随机森林模型。随机森林模型可用于查看样品的压裂状态如何正确分类。如果模型的表现比预期的要好,那将是取决于压裂状态的差异的额外证据。此外,最重要的预测器将揭示哪些特征对于正确区分样本(图3C)最为重要。然后,这些特征也会基于压裂状态产生一致不同的值。一旦确定这些差异特征,就可以审查这些文献,看看它们以前是否与压裂有关。然而,找到确定微分函数的研究可能具有挑战性,因为大多数研究只使用了 16S rRNA 组成数据。因此,为了评估微分函数的影响,一种可能的方法是看看它们以前是否与压裂液中常用的杀菌剂的潜在耐药性有关,或者它们是否有助于耐受高盐水条件。此外,检查利息分类的功能配置文件可以揭示压裂的影响的证据(图3D)。例如,如果分类被随机森林模型识别为差分,HF®样本中的抗菌电阻特征可以与HF样本中的特征进行比较,如果差异很大,则可能表明含有杀菌剂的压裂液进入流中。
| 样本 | 浓度 (ng/μL) |
| 1 | 1.5 |
| 2 | 1.55 |
| 3 | 0.745 |
| 4 | 0.805 |
| 5 | 7.82 |
| 6 | 0.053 |
| 7 | 0.248 |
| 8 | 0.945 |
| 9 | 1.82 |
| 10 | 0.804 |
| 11 | 0.551 |
| 12 | 1.69 |
| 13 | 4.08 |
| 14 | Below_Detection |
| 15 | 7.87 |
| 16 | 0.346 |
| 17 | 2.64 |
| 18 | 1.15 |
| 19 | 0.951 |
表1:基于荧光仪1xDSDNA高灵敏度检测的DNA浓度示例。 除14个样本外,所有这些样本的提取都被认为是成功的,因为有可检测到的DNA量。
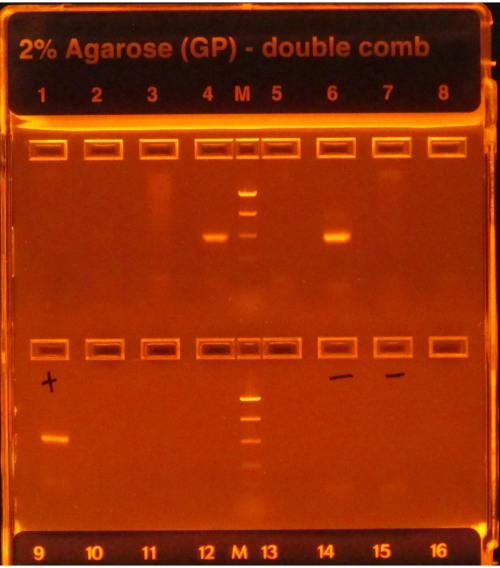
图1:PCR产品电子凝胶示例。 凝胶在紫外线下预先染色和可视化,导致其上存在的任何DNA发光。PCR 在第一排的 4 号和 6 号井中为样品工作,因为它们都有一个预期尺寸的明亮波段(基于梯子)。其他六口井中样品的PCR出现故障,因为它们没有产生任何波段。正控(第一井,第二排)具有明亮的带,表明PCR执行得当,负控(井6和7,第二排)没有任何带,表明样品没有受到污染。如果阴性有一个像样品一样亮的带子,PCR 将被视为失败,因为假设样品的安培不仅仅是污染的结果是有风险的。 请单击此处查看此图的较大版本。
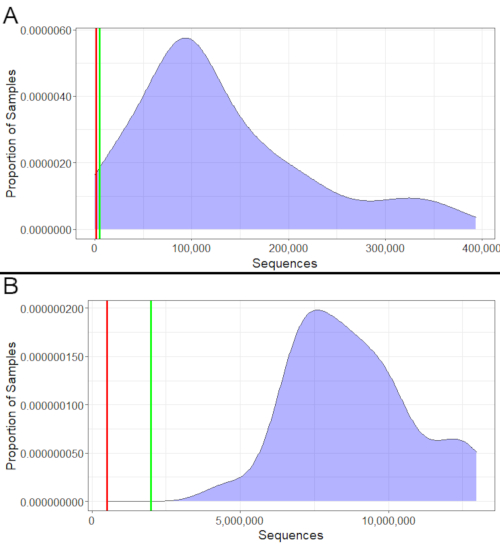
图2: 示例序列计数。 (A) 16s 示例序列计数。几乎所有的这16S样本都有超过1000个序列。极少数序列少于1,000个序列的人应该被排除在下游分析之外,因为他们没有足够的序列来准确代表他们的细菌群落。几个序列有1,000到5,000个序列;虽然不理想,他们仍然可以,因为他们超过最低值,大多数样品也超过理想的最低5000。(B) 甲基经学示例很重要。所有样本都超过了最低(500,000)和理想最小(2,000,000)序列数。因此,测序对于所有测序都是成功的,并且都可以用于下游分析。 请单击此处查看此图的较大版本。
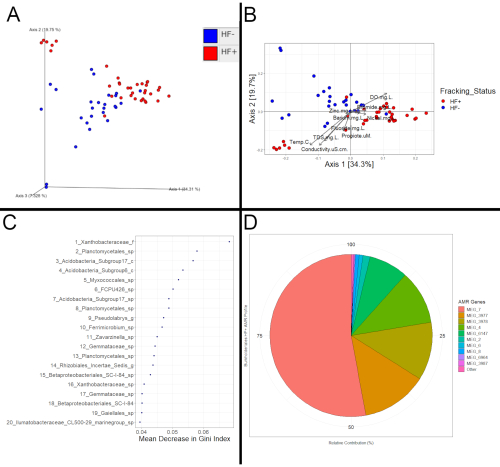
图3: 示例分析。 (A) PCoA 图基于通过 QIIME2 创建和可视化的加权 Unifrac 距离矩阵计算的坐标。(B) PCoA 图基于与 QIIME2 输出的加权 Unifrac 距离矩阵计算的坐标。坐标使用 Phyloseq 进行可视化,R. 元数据载体中的 ggplot2 包使用素食封装安装到图中。每个点表示样本的细菌群落,更近的点表示更相似的社区组成。根据这些 16S 沉积物样本的压裂状态进行聚类(PERMANOVA,p=0.001)。此外,病媒显示,HF®样品的氦、溴、镍和锌含量往往较高,与HF样本相比,这些样本与不同的细菌群落组成相对应。(C) 随机森林模型的最佳预测图,用于测试细菌丰度可用于预测样品中的压裂状态。随机森林模型是通过 R 使用随机森林包创建的。前 20 个预测因素以及由此产生的杂质减少(HF+ 和 HF 样本组合在一起的测量)在使用 Gini 指数的平均减少以单独样品的形式显示。(D) 饼图,显示基于元数据的伯克霍尔德里亚斯配置文件的抗菌电阻配置文件。序列首先与 Kraken2 注释,以确定它们属于哪个分类。然后,BLAST 与这些注释序列和 MEGARes 2.0 数据库一起使用,以确定哪些抗微生物药物耐药基因(以"MEG_#"的形式)正在积极表达。然后提取伯克利雅成员表达的抗微生物药物耐药基因,看看哪些基因在该分类中最为普遍。虽然更昂贵和更耗时,但元数据学确实允许进行功能分析,例如,16S 数据无法完成此分析。值得注意的是,Kraken2 用于此示例分析,而不是 HUMANN2。克拉肯2比胡曼2快:然而,它只输出组成信息,而不是组成,贡献和功能(基因)和路径,如HUMANN2做。 请单击此处查看此图的较大版本。
补充文件:甲基线学管道示例。 请点击这里下载此文件。
讨论
本文所描述的方法在2014年至2018年7、8、10年间由我们小组发表的几项研究过程中得到开发和完善,并已成功地应用于一个合作项目,以调查压裂对水生社区的影响,该项目为期三年,不久将提交论文出版。这些方法将继续在项目剩余时间内使用。此外,其他调查压裂对溪流和生态系统影响的现有文献描述了类似的样本收集、处理和分析方法,第7、8、10、11。然而,这些论文都没有使用元数据学分析,这使得本文首次描述了如何利用这些分析来阐明压裂对附近溪流的影响。此外,这里介绍的样品收集方法更为详细,为避免污染而采取的步骤也更加详细。
我们协议中最重要的步骤之一是初始样本收集和保存。现场采样和收集伴随着某些挑战,因为在采集过程中保持无菌或无菌环境可能很困难。在此步骤中,避免污染样品至关重要。为此,应佩戴手套,只允许无菌容器和工具接触样品。采集后还应立即将样品放在冰上,以减轻核酸降解。在采集时添加商业核酸防腐剂还可以增加核酸产量,并允许样品在采集后储存更长时间。每当进行核酸萃取时,使用适量的样品很重要,太多可以堵塞用于提取的自旋过滤器(用于使用它们的协议),但太少会导致产量低。请务必按照使用任何工具包的说明操作。
与现场采集类似,避免或尽量减少污染在核酸提取和样品制备过程中也很重要,尤其是在处理低核酸产量样品(如次优沉积物样品(含有大量砾石或岩石的样品)或水样时。因此,与样品收集一样,在所有这些步骤中应佩戴手套,以减少污染。此外,在实验室过程中使用的所有工作表面应事先用 10% 漂白液擦拭,然后使用 70% 乙醇溶液进行消毒。对于管道处理步骤 (3-6),应使用滤芯来避免由于移液器本身造成的污染,每次触摸非无菌表面时都会更改管道。所有用于实验室工作的工具,包括移液器,应在漂白剂和乙醇溶液前后擦除。为了评估污染,每组核酸提取和PCR反应中都应包括提取空白和底片(无菌液体)。如果提取后的量化显示底片中可检测到的 DNA/RNA 量,则如果还剩下足够的样本,可以重复提取。如果 PCR 的阴性样本显示放大,则应进行故障排除以确定源,然后重新运行样本。为了解释低污染水平,建议对提取空白和PCR底片进行测序,以便在计算分析期间,如有必要,可以识别和去除污染物。相反,PCR 放大也可能由于各种原因而失败。对于环境样品,抑制PCR反应往往是罪魁祸首,这可能是由于各种物质干扰Taq聚合酶23。如果怀疑抑制,PCR级水(见 材料表)可用于稀释DNA提取物。
本协议有一些明显的局限性和潜在的困难。样品收集对水和沉积物样本来说都是具有挑战性的。为了获得足够的生物质,理想情况下,1升的溪水需要通过过滤器推。过滤器的毛孔需要小,以捕获微生物,但也可以捕获沉积物。如果由于最近的降雨,水中有很多沉积物,过滤器可能会堵塞,因此很难将整个体积推入过滤器。对于沉积物收集,在收集过程中估计沉积物的深度可能具有挑战性。此外,必须确保收集的沉积物主要是土壤,因为鹅卵石和岩石将导致核酸产量降低,而且可能无法准确反映微生物群落。最后,收集后将样品保存在冰上也至关重要,尤其是在未使用防腐剂的情况下。
虽然此协议涵盖元数据学和 16S 实验室协议,但应强调这两种方法在过程和提供的数据类型上都大不相同。16S rRNA基因是一个常见的靶点区域,在细菌和古生物中保存高度,可用于在样本中描述细菌群落。虽然采用有针对性和具体的方法,但物种水平的分辨率往往无法实现,很难确定新分化的物种或菌株的特征。相反,甲基定性学是一种更广泛的方法,可以捕获样本中存在的所有活性基因和微生物。16S只提供识别数据,而代谢学可以提供功能数据,如表达基因和代谢通路。两者都是有价值的,当结合时,它们可以揭示哪些细菌存在,哪些基因在表达。
本文介绍了16S rRNA和元数据学分析在研究压裂背景下的现场采集和样品处理方法。此外,它还详细介绍了从低生物质样品中采集高质量DNA/RNA的方法,以及长期储存的方法。这里描述的方法是我们收集和处理样品经验的顶点,我们努力通过检查其微生物群落的结构和功能来了解压裂如何影响附近的溪流。微生物对扰动反应迅速,因此,微生物的存在及其表达的基因可以提供有关压裂对生态系统的影响的信息。总的来说,这些方法对于我们理解压裂如何影响这些重要的生态系统可能是无价的。
披露声明
作者没有什么可透露的。
致谢
作者要确认导致这些方法发展的项目的资助来源,这些来源是:霍华德·休斯医学院(http://www.hhmi.org)通过预科和本科科学教育方案,以及国家科学基金会(http://www.nsf.gov)通过NSF奖励DBI-1248096和CBET-1805549。
材料
| Name | Company | Catalog Number | Comments |
| 200 Proof Ethanol | Thermo Fisher Scientific | A4094 | 400 mL need to be added to Buffer PE (see Qiagen QIAQuck Gel Extraction kit protocol) and 96 mL needs to be added to the DNA/RNA Wash Buffer (see ZymoBIOMICS DNA/RNA Miniprep kit protocol). Additional ethanol is needed for the ZymoBIOMICS DNA/RNA Miniprep and NEBNext® Ultra™ II RNA Library Prep with Sample Purification Beads kits. |
| Agarose | Thermo Fisher Scientific | BP1356-100 | 100 g per bottle. 0.6 g of agarose would be needed to make one 2% 30 mL gel. |
| Disinfecting Bleach | Walmart (Clorox) | No catalog number | Use a 10% bleach solution for cleaning the work area before and after lab procedures |
| DNA gel loading dye | Thermo Fisher Scientific | R0611 | Each user-made (i.e. non-e-gel) should include loading dye with all of the samples in the ratio of 1 µL dye to 5 µL sample |
| DNA ladder | MilliporeSigma | D3937-1VL | A ladder should be run on every gel/e-gel |
| DNA/RNA Shield (2x) | Zymo Research | R1200-125 | 3 mL per sediment sample (50 mL conical) and 2 mL per water sample (filter) |
| Ethidium bromide | Thermo Fisher Scientific | BP1302-10 | Used for staining user-made e-gels |
| Forward Primer | Integrated DNA Technologies (IDT) | 51-01-19-06 | 0.5 µL per PCR reaction |
| Isopropanol | MilliporeSigma | 563935-1L | Generally less than 2 mL per library. Volume needed varies by mass of excised gel fragment (see Qiagen QIAQuick Gel Extraction kit protocol). |
| PCR-grade water | MilliporeSigma | 3315932001 | 13 µL per PCR reaction (assuming 1 µL of sample DNA template is used) |
| Platinum Hot Start PCR Master Mix (2x) | Thermo Fisher Scientific | 13000012 | 10 µL per PCR reaction |
| Reverse Primer | Integrated DNA Technologies (IDT) | 51-01-19-07 | 0.5 µL per PCR reaction |
| TBE Buffer (Tris-borate-EDTA) | Thermo Fisher Scientific | B52 | 1 L of 10x TBE buffer (30 mL of 1x TBE buffer would be needed to make one 30 mL gel) |
| 1 L bottle | Thermo Fisher Scientific | 02-893-4E | One needed per stream (the same bottle can be used for multiple streams if it is sterilized between uses) |
| 1.5 mL Microcentrifuge tubes | MilliporeSigma | BR780400-450EA | 5 microcentrifuge tubes are needed per DNA extraction and an additional 3 are needed to purify RNA (see ZymoBIOMICS DNA/RNA Miniprep kit protocol) |
| 2% Agarose e-gel | Thermo Fisher Scientific | G401002 | Each gel can run 10 samples (so 9 with a PCR negative and 8 if the extraction negative is run on the same gel) |
| 50 mL Conicals | CellTreat | 229421 | 1 50 mL conical needed per sediment samples |
| 500 mL Beaker | MilliporeSigma | Z740580 | Only 1 needed (for flame sterilization) |
| Aluminum foil | Walmart (Reynolds KITCHEN) | No number | Aluminum foil can be folded and autoclaved. The part not exposed to the environment can then be used as a sterile, DNA and RNA free surface for processing filters (one folded piece per filter to avoid cross-contamination) |
| Autoclave | Gettinge | LSS 130 | Only one needed |
| Centrifuge | MilliporeSigma | EP5404000138-1EA | Only 1 needed |
| Cooler | ULINE | S-22567 | Just about any cooler can be used. This one is listed due to being made of foam, making it lighter and thus easier to take along for field sampling. |
| Disruptor Genie | Bio-Rad | 3591456 | Only one needed |
| Electrophoresis chamber | Bio-Rad | 1664000EDU | Only 1 needed |
| Electrophoresis power supply | Bio-Rad | 1645050 | Only 1 needed |
| Freezer (-20 C) | K2 SCIENTIFIC | K204SDF | One needed to store DNA extracts |
| Freezer (-80 C) | K2 SCIENTIFIC | K205ULT | One needed to store RNA extracts |
| Gloves | Thermo Fisher Scientific | 19-020-352 | The catalog number is for Medium gloves. |
| Heat block | MilliporeSigma | Z741333-1EA | Only one needed |
| Lab burner | Sterlitech | 177200-00 | Only one needed |
| Laminar Flow Hood | AirClean Systems | AC624LFUV | Only 1 needed |
| Library purification kit | Qiagen | 28704 | One kit has enough for 50 reactions |
| Magnet Plate | Alpaqua | A001219 | Only one needed |
| Microcentrifuge | Thermo Fisher Scientific | 75004061 | Only one needed |
| Micropipette (1000 µL volume) | Pipette.com | L-1000 | Only 1 needed |
| Micropipette (2 µL volume) | Pipette.com | L-2 | Only 1 needed |
| Micropipette (20 µL volume) | Pipette.com | L-20 | Only 1 needed |
| Micropipette (200 µL volume) | Pipette.com | L-200R | Only 1 needed |
| NEBNext Ultra II RNA Library Prep with Sample Purification Beads | New England BioLabs Inc. | E7775S | One kit has enough reagents for 24 samples. |
| Parafilm | MilliporeSigma | P7793-1EA | 2 1" x 1" squares are needed per filter |
| PCR Tubes | Thermo Fisher Scientific | AM12230 | One tube needed per reaction |
| Pipette tips (for 1000 µL volume) | Pipette.com | LF-1000 | Pack of 576 tips |
| Pipette tips (for 20 µL volume) | Pipette.com | LF-20 | Pack of 960 tips |
| Pipette tips (for 200 µL volume) | Pipette.com | LF-250 | Pack of 960 tips |
| PowerWulf ZXR1+ computer cluster | PSSC Labs | No number | This is just an example of a supercomputer powerful enough to perform metatranscriptomics analysis in a timely manner. Only one needed. |
| Qubit fluorometer starter kit | Thermo Fisher Scientific | Q33239 | Comes with a Qubit 4 fluorometer, enough reagent for 100 DNA assays, and 500 Qubit tubes |
| Scoopula | Thermo Fisher Scientific | 14-357Q | Only one needed |
| Sterile blades | AD Surgical | A600-P10-0 | One needed per filter |
| Sterivex-GP Pressure Filter Unit | MilliporeSigma | SVGP01050 | 1 filter needed per water sample |
| Thermocycler | Bio-Rad | 1861096 | Only one needed |
| Vise-grip | Irwin | 2078500 | Only one needed (for cracking open the filters) |
| Vortex-Genie 2 | MilliporeSigma | Z258415-1EA | Only 1 needed |
| WHIRL-PAK bags | ULINE | S-22729 | 1 needed per filter |
| ZymoBIOMICS DNA/RNA Miniprep kit | Zymo Research | R2002 | One kit has enough reagents for 50 samples. |
参考文献
- The process of unconventional natural gas production. US EPA Available from: https://www.epa.gov/uog/process-unconventional-natural-gas-production (2013)
- Brittingham, M. C., Maloney, K. O., Farag, A. M., Harper, D. D., Bowen, Z. H. Ecological risks of shale oil and gas development to wildlife, aquatic resources, and their habitats. Environmental Science & Technology. 48 (19), 11034-11047 (2014).
- McBroom, M., Thomas, T., Zhang, Y. Soil erosion and surface water quality impacts of natural gas development in East Texas, USA. Water. 4 (4), 944-958 (2012).
- Maloney, K. O., Weller, D. E. Anthropogenic disturbance, and streams: land use and land-use change affect stream ecosystems via multiple pathways. Freshwater Biology. 56 (3), 611-626 (2011).
- Meyer, J. L., et al. The contribution of headwater streams to biodiversity in river networks1. JAWRA Journal of the American Water Resources Association. 43 (1), 86-103 (2007).
- Alexander, R. B., Boyer, E. W., Smith, R. A., Schwarz, G. E., Moore, R. B. The role of headwater streams in downstream water quality. Journal of the American Water Resources Association. 43 (1), 41-59 (2007).
- Ulrich, N., et al. Response of aquatic bacterial communities to hydraulic fracturing in Northwestern Pennsylvania: A five-year study. Scientific Reports. 8 (1), 5683 (2018).
- Chen See, J. R., et al. Bacterial biomarkers of Marcellus shale activity in Pennsylvania. Frontiers in Microbiology. 9, 1697 (2018).
- Rausch, P., et al. Comparative analysis of amplicon and metagenomic sequencing methods reveals key features in the evolution of animal metaorganisms. Microbiome. 7 (1), 133 (2019).
- Louca, S., Doebeli, M., Parfrey, L. W. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem. Microbiome. 6 (1), 41 (2018).
- Trexler, R., et al. Assessing impacts of unconventional natural gas extraction on microbial communities in headwater stream ecosystems in Northwestern Pennsylvania. Frontiers in Microbiology. 5, 522 (2014).
- Mumford, A. C., et al. Shale gas development has limited effects on stream biology and geochemistry in a gradient-based, multiparameter study in Pennsylvania. Proceedings of the National Academy of Sciences. 117 (7), 3670-3677 (2020).
- JoVE Core Biology DNA Isolation. Journal of Visualized Experiments Available from: https://www.jove.com/cn/science-education/10814/dna-isolation (2020)
- Oxford Gene Technology DNA Storage and Quality. OGT Available from: https://www.ogt.com/resources/literature/403_dna_storage_and_quality (2011)
- ThermoFisher SCIENTIFIC Technical Bulletin #159: Working with RNA. Thermoscientific Available from: https://www.thermofisher.com/us/en/home/references/ambion-tech-support/nuclease-enzymes/general-articles/working-with-rna.html (2020)
- QIAGEN AllPrep DNA/RNA Mini Kit. Qiagen Available from: https://www.qiagen.com/us/products/discovery-and-translational-research/dna-rna-purification/multianalyte-and-virus/allprep-dnarna-mini-kit/#orderinginformation (2020)
- ZymoBIOMICS DNA/RNA Miniprep Kit. Zymo Research Available from: https://www.zymoresearch.com/products/zymobiomics-dna-rna-miniprep-kit (2020)
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- 16S Illumina amplicon protocol: Earth microbiome project. Earth microbiome project Available from: https://earthmicrobiome.org/protocols-and-standards/16s/ (2018)
- Gel Purification: Binding, washing and eluting a sample | Protocol. Journal of Visualized Experiments Available from: https://www.jove.com/v/5063/gel-purification (2020)
- New England Biolabs protocol for the use with NEBNext Poly(A) mRNA magnetic isolation module (E7490) and NEBNext Ultra II RNA library prep kit for Illumina (E7770, E7775). New England Biolabs Available from: https://www.neb.com/protocols/2017/03/04/protocol-for-use-with-purified-mrna-or-rrna-depleted-rna-and-nebnext-ultra-ii-rna-library-prep-ki (2020)
- Anderson, M. J. Permutational multivariate analysis of variance (PERMANOVA). Wiley StatsRef: Statistics Reference Online. , 1-15 (2017).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。