Method Article
Оценка влияния гидроразрыва пласта на потоки с использованием микробных молекулярных сигнатур
В этой статье
Резюме
Здесь мы представляем протокол для исследования воздействия гидравлического разрыва пласта на близлежащие потоки путем анализа их водных и осадочных микробных сообществ.
Аннотация
Гидравлический разрыв пласта (HF), обычно называемый «фрекингом», использует смесь воды высокого давления, песка и химических веществ для разрушения горных пород, высвобождая нефть и газ. Этот процесс произвел революцию в энергетической отрасли США, поскольку он дает доступ к ресурсам, которые ранее были недоступны, и теперь производит две трети всего природного газа в Соединенных Штатах. Хотя фрекинг оказал положительное влияние на экономику США, несколько исследований выявили его пагубные экологические последствия. Особую озабоченность вызывает воздействие фрекинга на верховья ручьев, которое особенно важно из-за их непропорционально большого воздействия на здоровье всего водосборного бассейнов. Бактерии в этих потоках могут быть использованы в качестве индикаторов здоровья потока, поскольку ожидается, что бактерии, присутствующие и их обилие в нарушенном потоке, будут отличаться от бактерий в сопоставимом, но ненарушенном потоке. Таким образом, этот протокол направлен на использование бактериального сообщества для определения того, были ли потоки затронуты фрекингом. С этой целью необходимо собирать пробы отложений и воды из ручьев вблизи фрекинга (потенциально пострадавших) и вверх по течению или в другом водоразделе деятельности по фрекингу (без последствий). Затем эти образцы подвергают экстракции нуклеиновых кислот, подготовке библиотеки и секвенированию для изучения состава микробного сообщества. Корреляционный анализ и модели машинного обучения могут быть впоследствии использованы для определения того, какие признаки являются эксплантативными для вариаций в сообществе, а также для идентификации прогностических биомаркеров воздействия фрекинга. Эти методы могут выявить различные различия в микробных сообществах между верховьями ручьев, основанные на близости к фрекингу, и служить основой для будущих исследований воздействия деятельности по фрекингу на окружающую среду.
Введение
Гидравлический разрыв пласта (ВС), или «фрекинг», является методом добычи природного газа, который становится все более распространенным по мере роста спроса на ископаемое топливо. Этот метод заключается в использовании мощного бурового оборудования для закачки смеси воды, песка и химических веществ в богатые метаном сланцевые месторождения, обычно для высвобождения захваченных газов1.
Поскольку эти нетрадиционные методы сбора урожая являются относительно новыми, важно изучить влияние такой практики на близлежащие водные пути. Деятельность по гидроразрыву пласта требует расчистки больших участков земли для транспортировки оборудования и строительства колодезных площадок. Приблизительно 1,2-1,7 гектара земли должны быть расчищаемы для каждой площадкискважины 2,что потенциально влияет на сток и качество воды системы3. Отсутствует прозрачность, окружающая точный химический состав жидкости для фрекинга, в том числе то, какие биоциды используются. Кроме того, сточные воды фрекинга, как правило, имеют высокую засоленную2. Кроме того, сточные воды могут содержать металлы и природные радиоактивные вещества2. Поэтому вызывает беспокойство возможность утечек и разливов жидкости для гидроразрыва пласта из-за человеческой ошибки или неисправности оборудования.
Известно, что реточные экосистемы очень чувствительны к изменениям в окружающих ландшафтах4 и важны для поддержания биоразнообразия5 и надлежащего круговорота питательных веществ6 во всем водоразделе. Микробы являются наиболее распространенными организмами в пресноводных потоках и, таким образом, необходимы для круговорота питательных веществ, биодеградации и первичного производства. Состав и функции микробных сообществ служат отличными инструментами для получения информации об экосистеме из-за их чувствительности к возмущениям, и недавние исследования показали отчетливые сдвиги в наблюдаемых бактериальных сборках, основанных на близости к деятельности фрекинга7,8. Например, Beijerinckia, Burkholderiaи Methanobacterium были идентифицированы как обогащенные в ручьях вблизи фрекинга, в то время как Pseudonocardia, Nitrospiraи Rhodobacter были обогащены в потоках, не близких к фрекингу7.
Секвенирование следующего поколения гена рибосомальной РНК (рРНК) следующего поколения является доступным методом определения состава бактериального сообщества, который быстрее и дешевле, чем секвенирование всего генома приближается к9. Обычной практикой в области молекулярной экологии является использование высоко вариативной области V4 гена 16S рРНК для разрешения секвенирования, часто вплоть до уровня рода с широким спектром идентификации9,поскольку она идеально подходит для непредсказуемых образцов окружающей среды. Этот метод был широко внедрен в опубликованных исследованиях и был успешно использован для выявления воздействия операций фрекинга на воднуюсреду 7,8. Однако стоит отметить, что бактерии имеют различное число копий гена 16S рРНК, что влияет на их обнаруженное изобилие10. Есть несколько инструментов, чтобы объяснить это, но их эффективность сомнительна10. Другой практикой, которая быстро растет в распространенности и не имеет этой слабости, является метатранскриптомное секвенирование, при котором все РНК секвенируются, что позволяет исследователям идентифицировать как активные бактерии, так и экспрессию их генов.
Поэтому, в отличие от методов в ранее опубликованных исследованиях7,8,11,12,этот протокол также охватывает сбор образцов, сохранение, обработку и анализ для исследования функции микробного сообщества (метатранскриптомика). Шаги, подробно описанные здесь, позволяют исследователям увидеть, какое влияние, если таковое имеется, фрекинг оказал на гены и пути, экспрессируемые микробами в их потоках, включая гены устойчивости к противомикробным препаратам. Кроме того, повышается уровень детализации, представляемой для сбора проб. Хотя некоторые из шагов и заметок могут показаться очевидными для опытных исследователей, они могут быть бесценными для тех, кто только начинает исследования.
Здесь мы описываем методы сбора и обработки образцов для получения бактериальных генетических данных в качестве средства исследования влияния фрекинга на близлежащие потоки на основе многолетнего опыта наших лабораторий. Эти данные могут быть использованы в последующих приложениях для выявления различий, соответствующих статусу фрекинга.
протокол
1. Сбор проб отложений для экстракции нуклеиновых кислот
- Погружайте стерильную коническую трубку 50 мл в воду ручья. Надевайте перчатки во время сбора проб, чтобы избежать нежелательного загрязнения человеком. Выполните этот шаг либо с берега, либо лицом вверх по течению, если в воде.
- Пока коническая трубка погружена, снимите колпачок и используйте его, чтобы зачерпнуть примерно 3 мл осадка с глубины от 1 до 3 см в коническую трубку.
- Извлеките коническую трубку из воды и выбросьте всю воду, за исключением тонкого слоя, покрывающего образец осадка (примерно 1 мл).
- Используя пипетку 1000 мкл и соответствующие наконечники пипетки, добавьте 3 мл консерванта ДНК/РНК (см. Таблицу материалов для спецификаций консервантов) к собранному образцу. Держите наконечники пипеток в стерильном ящике для пипеток и прикрепляйте их только непосредственно перед использованием и выбрасывайте после использования. Переверьте колпашку конической трубки 10 раз, чтобы убедиться, что консервант и образец тщательно перемешаны.
Примечание Шаг 1.4 не является необходимым, но настоятельно рекомендуется, если РНК должна быть извлечена из отложений позже. - Поместите образцы на лед для остальной части сбора проб. По возвращении из сбора храните в морозильной камере при -20 °C, если образцы должны быть использованы для анализа 16S (ДНК), или -70 °C, если они должны использоваться для метатранскриптомического анализа (РНК).
2. Фильтр-коллектор для экстракции нуклеиновых кислот
- Снимите крышку стерильной бутылки объемом 1 л. Стоя лицом вверх по течению или от берега, наполните бутылку струйной водой наверх, а затем выбросьте ее. Повторите этот процесс еще два раза, чтобы кондиционировать бутылку. Заполните всю бутылку в четвертый раз и за крышкой.
ПРИМЕЧАНИЕ: При повторном ополаскивательном флаконе объемом 1 л ее можно стерилизовать 10% отбеливателем в течение 2 мин, затем трижды промыть деионизированной водой, а затем один раз 70% этанолом и, наконец, автоклавированием с настройками: 30-минутное время воздействия при 121,1 °C и 15-минутное время сушки. Во время автоклавирования крышка на бутылке должна быть очень свободной, чтобы избежать сжатия бутылки в процессе. - Оказавшись на устойчивой поверхности, используйте стерильный шприц Luer Lock и нарисуйте полный объем. Затем подключите шприц к стерильному и не имеющего ДНК/РНК полиэфирсульфонового фильтра диаметром 1,7 см с размером пор 0,22 мкм и протолкните весь объем через фильтр, нажав плунжер до самого низу. Повторяйте этот процесс до тех пор, пока общий объем, собранный во флаконе (1 л), не будет протолкнут через фильтр.
ПРИМЕЧАНИЕ: Объем шприца может быть переменным, если отслеживается общее количество воды, проталкиваемой через фильтр. Однако, как правило, 60 мл является предпочтительным. Хотя 1 л является идеальным, как ни странно, объем не менее 200 мл, вероятно, все равно будет собирать достаточно биомассы (при условии ~ 20 000 клеток на мл) для экстракции ДНК и РНК. - Удалите лишнюю воду из фильтра, втянув примерно 20 мл воздуха в шприц и протолкнув его через фильтр.
ПРИМЕЧАНИЕ: Это поможет предотвратить потерю консерванта, если выполняется шаг 2.4. - Используя микропипетку P1000, добавьте 2 мл консерванта ДНК / РНК, выведя его через большее отверстие фильтра (где он был прикреплен к шприцевой части), удерживая фильтр горизонтально. Наконечник пипетки должен находиться в бочке фильтра, когда пипетка нажата, чтобы обеспечить попадание консерванта в фильтр. Меняйте наконечник после каждого использования.
ПРИМЕЧАНИЕ: Как и в случае со сбором отложений, этот этап не является необходимым, но он настоятельно рекомендуется для увеличения выхода нуклеиновых кислот позже, особенно для РНК. - Снимите один квадрат парафиновой пленки и плотно оберните его вокруг каждого отверстия / конца фильтра для герметизации. Поместите обернутый парафиновой пленкой фильтр в стерильный мешок для образцов, а затем поместите весь пакет на лед во время сбора.
ПРИМЕЧАНИЕ: Убедитесь, что сторона, используемая для обертывания фильтра, стерильна, т.е. ранее не подвергалась воздействию окружающей среды. - После возврата после отбора проб храните фильтры при -20 °C для 16S или -70 °C для метакриптомики.
3. Экстракция и количественная оценка нуклеиновых кислот
- Очистите рабочую зону 10% отбеливателем и 70% этанолом перед началом переноса пробы.
- Для осадка (начиная со ступени 1.5), как правило, используют ~0,25 г образца. Пламя стерилизует металлический инструмент, погружая его в замок с 70% этанола и сжигая этанол между образцами.
- Для фильтров (из шага 2.6) переместите фильтровальную бумагу в стерильную трубку для экстракции. Для этого выполните следующие действия.
- Создают стерильную, ДНК и РНК свободную поверхность путем складывания алюминиевой фольги таким образом, чтобы внутренняя часть складки не подвергалась воздействию внешней среды и автоклавирования сложенного куска с настройками: 121,1 °C и 5 мин времени высыхания.
- Стерилизуйте тиски 70% этанолом и открытым пламенем. Затем используйте тиски-рукоятку, чтобы разорвать корпус фильтра на стерильной поверхности и удалить сердечник из корпуса.
- Используйте стерильный скальпель, чтобы отрезать фильтровальную бумагу от сердцевины, нарезав сверху и снизу, а затем вдоль шва. Сложите фильтровальную бумагу с помощью стерильного пинцете, а затем разрежьте фильтр на мелкие кусочки с помощью скальпеля.
- Поместите части фильтра в микроцентрифужную трубку для экстракции. Убедитесь, что фильтрованная бумага не вступает в контакт с какими-либо поверхностями, которые не стерилизованы или на которых может присутствовать нуклеиновая кислота, так как это приведет к нежелательному загрязнению образца.
- Выполняют выделение ДНК, как описановыше 13, или с помощью коммерчески доступного набора на основе столбцов (см. Таблицу материалов). Перечисленные шаги для коммерческого комплекта кратко описаны ниже.
- Различают клетки внутри образца, перенося его в шариковую трубку и подвергая его разрушителя клеток с высокой скоростью в течение не менее 5 минут. Центрифугировать и перенести супернатант в стерильную микроцентрифужную трубку.
- Добавьте буфер лизиса к супернатанту (объем 1:1) и перенесите на предоставленный фильтр (желтый). Центрифугировать фильтр.
- Перенесите фильтр в новую стерильную микроцентрифужную трубку. Добавьте буфер подготовки (400 мкл), центрифугу и отбросьте поток.
- Добавьте буфер промывки (700 мкл), центрифугу и отбросьте поток. Затем добавьте промывочный буфер (400 мкл), центрифугу и снова отбросьте поток.
- Перенесите фильтр в новую стерильную микроцентрифужную трубку. Элюировать 50 мкл воды без ДНКазы/РНКазы и дать постоять в течение 5 мин при комнатной температуре перед центрифугированием.
- В течение этого периода кубирования подготовьте фильтр III-HRC, поместив его в сборную трубку и добавив к ней раствор для подготовки HRC (600 мкл) с последующей этапом центрифугирования 3 мин при 8000 х г.
- Переместите подготовленный фильтр на стерильную микроцентрифужную трубку. Перенесите элюированную ДНК со ступени 3.4.5 на этот фильтр и центрифугу при 16 000 х г в течение 3 мин. Поток содержит извлеченную ДНК.
- Храните экстракты ДНК как для отложений, так и для фильтров при -20 °C.
ПРИМЕЧАНИЕ: Экстракты ДНК могут храниться около 8 лет при -20 °C при стабильной температуре, ограниченном освещении и отсутствии вредных загрязняющих веществ14. - Выполните изоляцию РНК в соответствии с протоколом производителя. Хранить экстракты РНК при -80 °C.
- Различают клетки в образце, перенося его в шариковую трубку и подвергая его разрушителя клеток на высокой скорости в течение не менее пяти минут. Центрифугировать и перенести супернатант в стерильную микроцентрифужную трубку.
- Добавьте буфер лизиса к надёжному (объем 1:1) и перенесите в предоставленный столбец (желтый). Центрифугировать колонну.
- Добавьте равный объем 95-100% этанола в поток и перемешайте, пипетируя вверх и вниз пять раз.
- Поместите колонку IICG (зеленую) на стерильную микроцентрифужную трубку. Переложите смешанный раствор на колонну и центрифугу.
- Добавьте буфер промывки (400 мкл), центрифугу и отбросьте поток.
- Добавьте в колонку 5 мкл ДНКазы I и 75 мкл буфера для пищеварения ДНК и инкубируют при комнатной температуре в течение 15 минут.
- Добавьте подготовительный буфер (400 мкл), центрифугу и отбросьте поток.
- Добавьте буфер промывки (700 мкл), центрифугу и отбросьте поток. Затем добавьте промывочный буфер (400 мкл), центрифугу и снова отбросьте поток.
- Перенесите колонну в новую стерильную микроцентрифужную трубку. Элюировать 50 мкл воды, свободной от ДНКазы/РНКазы, и дать постоять в течение 5 мин перед центрифугированием.
- В течение этого инкубационного периода подготовьте фильтр III-HRC, поместив его в сборную трубку и добавив к нему раствор для подготовки HRC (600 мкл) с последующей этапом центрифугирования 3 мин при 8000 х г.
- Переместите подготовленный фильтр на стерильную микроцентрифужную трубку. Перенос элюированной РНК со ступени 3.6.9 на этот фильтр и центрифугу при 16 000 х г в течение 3 мин. Поток через содержит извлеченную РНК.
ПРИМЕЧАНИЕ: Экстракты РНК могут храниться только в течение одного года, прежде чем они начнут разлагаться15. Как ДНК, так и экстракты РНК разлагаются при повторном замораживании-оттаивании. Некоторые протоколы позволяют извлекать как ДНК, так и РНК из одного и того же образца16,17.
- Количественно оцените извлеченные образцы ДНК и РНК с помощью флуорометра или спектрофотометра. См. таблицу 1, например, значения концентрации ДНК флюорометра. Пример протокола количественной оценки спектрофотометра см. в ссылке18. Значения концентрации ДНК осадка в наборе, указанном в Таблице материалов, обычно варьируются от 1 до 40 нг/мкл, в то время как значения концентрации ДНК фильтра, как правило, колеблются от 0,5 до 10 нг/мкл. Значения концентрации РНК в осадке с набором, перечисленным в Таблице материалов, обычно варьируются от 1 до 20 нг/мкл, в то время как значения концентрации фильтрующей РНК, как правило, ниже, обычно в диапазоне от 0,5 до 5 нг/мкл.
4. Создание библиотеки рРНК 16S
- Очистите рабочую зону 10% отбеливателем и 70% этанолом. Рабочая зона должна представлять собой закрытое пространство, способное создавать ламинаричные условия потока (ламинарной вытяжка).
- Используйте экстракты ДНК (из шага 3.5) и подготовьте образцы для секвенирования ампликона 16S рРНК со стандартным протоколом ПЦР, таким как тот, который описан на веб-сайте микробиома Земли, который усиливает гипервариабельную область V4 16S рРНК19 в условиях ламинарного потока.
- Приготовьте 2% агарозный гель, как описано ранее, и дайте ему затвердеть17. Смешайте 7 мкл продукта ПЦР и 13 мкл воды без ДНКазы. Добавьте гелеобразующий краситель до конечной концентрации 1x. Как только агароза затвердеет, загрузите эту смесь продуктов ПЦР на 2% агарозный гель.
ПРИМЕЧАНИЕ: В качестве альтернативы можно использовать сборный гель, так как эти гели работают быстрее и поставляются предварительно изготовленными. - Запустите гель при 90 В в течение 60-90 мин, чтобы проверить размер полосы 386 в качестве успешного усиления для ампликонов 16S рРНК V4, используя протокол микробиома Земли.
5. Очистка библиотеки рРНК ДНК 16S
- Пул 10 мкл продуктов ПЦР для образцов, которые дали яркие полосы и 13 мкл для образцов, которые дали слабые полосы в стерильной микроцентрифужной трубке соответствующего размера.
- Проверьте концентрацию полученного бассейна с помощью флуорометра или спектрофотометра и приготовьте 2% агарозный гель, как и раньше. В идеале бассейн должен иметь концентрацию не менее 10 нг/мкл, а большинство образцов должны иметь концентрацию около 25 нг/мкл.
- Концентрация и объем позволяют, нагрузка около 150-200 нг в скважину 2% агарозного геля.
- Запускают гель в течение 60-90 мин при 90 вольтах.
- Очистите объединенную библиотеку, испуская 2% агарозный гель.
- Высеките полосу ДНК 386 bp из геля и очистите объединенную библиотеку, используя коммерчески доступный набор, как описаноранее 20. Элюдирует очищенную ДНК 30 мкл 10 мМ Tris-Cl (рН 8,5). Выполните этот шаг в области, отличной от экстракции ДНК или РНК, чтобы предотвратить будущее загрязнение, так как разрезание геля приведет к распространению ампликонов ПЦР как на экспериментатора, так и на окружающую область.
- Проверьте концентрацию очищенного бассейна с помощью флуорометра или спектрофотометра. Если очищение прошло хорошо, его концентрация должна составлять не менее половины неочищенного бассейна. Как правило, конечная концентрация должна составлять от 5 до 20 нг/мкл.
- Отправьте очищенные библиотеки для секвенирования следующего поколения. Убедитесь, что они остаются холодными во время транспортировки, включив сухой лед в транспортный контейнер.
6. Создание и очистка библиотеки РНК
- Несколько коммерческих наборов могут быть использованы для создания библиотек РНК. Для любого из них используется, следуйте протоколу производителя, как написано при работе в стерильной ламинарной среде потока. Очень обобщенный вариант протокола для комплекта в Таблице материалов представлен ниже21.
- Сделайте мастер-микс синтеза кДНК первой нити (8 мкл воды без нуклеазы и 2 мкл первой цепи синтеза Enyzme Mix) и добавьте ее в образец. Поместите образец в термоциклер с условиями, указанными в протоколе.
- Сделайте на льду мастер-смесь синтеза кДНК второй цепи (8 мкл реакционного буфера синтеза второй цепи, 4 мкл смеси ферментов синтеза второй цепи и 48 мкл воды без нуклеазы) и добавьте ее в образец. Поместите в термоциклер, установленный при 16 °C, на один час.
- Очистите реакцию, добавив предоставленные шарики (144 мкл) и выполнив две 80% промывки этанола (200 мкл).
- Элюировать с помощью предоставленного TE-буфера (53 мкл) и переносить 50 мкл супернатанта в чистую ПЦР-трубку. Поместите трубку ПЦР на лед.
- Сделайте мастер-микс для конечной подготовки (7 мкл реакционного буфера конечной подготовки и 3 мкл смеси ферментов конечной подготовки) на льду и добавьте его в трубку ПЦР. Поместите ПЦР-трубку в термоциклер с условиями, указанными в протоколе.
- Смешайте на льду растворы разбавленный адаптер (2,5 мкл), Ligation Master Mix (30 мкл) и лигационный усилитель (1 мкл). Добавьте смешанные растворы к образцу и поместите в термоциклер на 15 мин при 20 °C.
- Очистите реакцию, добавив предоставленные шарики (87 мкл) и выполнив промывку этанола (200 мкл) и элюдируя, как и раньше, за исключением добавления только 17 мкл ТЭ.
- Добавьте индексы (10 мкл) и раствор Q5 Master Mix (25 мкл) и поместите в термоциклер с условиями, описанными в протоколе.
- Очищают реакцию путем добавления предоставленных шариков (45 мкл) и выполнения добавления двух промывок этанола (200 мкл) и элюда с 23 мкл ТЭ. Переложить 20 мкл в чистую ПЦР-трубку.
- Проверьте библиотеки на наличие обнаруживаемых концентраций РНК с помощью биоанализатора, флуорометра или спектрофотометра.
- Объединяем метатранскриптомные библиотеки в примерно эквимолярном соотношении.
- Очистите библиотеку по тому же протоколу для очистки библиотеки 16S, за исключением фрагментов акцизов между 250 и 400 bp. В то время как библиотека 16S имела четкую полосу, представляющую усиленную область, результатом здесь является мазок.
- Проверьте концентрацию очищенной библиотеки, как и раньше.
- Отправляйте очищенную библиотеку с сухим льдом на установку секвенирования.
ПРИМЕЧАНИЕ: В качестве альтернативы, экстракты РНК могут быть отправлены в университет или частную компанию для подготовки библиотеки и секвенирования.
7. Анализ микробного сообщества
- После завершения виртуализации получите доступ к образцу данных. Загрузите его на пригодный для использования компьютер.
ПРИМЕЧАНИЕ: В идеале устройство должно иметь не менее 16 гигабайт оперативной памяти. Обсуждение вычислительных требований (для Qiime2) см. в https://forum.qiime2.org/t/recommended-specifications-to-run-qiime2/9808. - Используйте программное обеспечение, такое как mothur, QIIME2 и R, для анализа данных 16S рРНК. Здесь https://docs.qiime2.org/2020.11/tutorials/moving-pictures/ приведен пример учебника по анализу QIIME2 16S.
- Для получения данных метатранскриптомики (РНК) используйте HUMAnN2 и ATLAS, чтобы определить, какие гены и пути присутствуют в образцах.
ПРИМЕЧАНИЕ: Пример конвейера метатранскриптомики, кульминацией которого является анализ разнообразия и случайных лесов, представлен в файле дополнительной информации. Все команды выполняются через командную строку, например, для пользователей терминала для Mac.
Результаты
Успех экстракции ДНК и РНК можно оценить с помощью различных средств и протоколов. Как правило, любая обнаруживаемая концентрация любого из них считается достаточной для вывода о том, что экстракция была успешной. Изучив таблицу 1, все извлечения, за исключением одного, будут названы успешными. Неудача на этом этапе часто связана с низкой начальной биомассой, плохой сохранностью образца или человеческой ошибкой во время экстракции. В случае фильтров экстракция может быть успешной, даже если концентрация ниже обнаружения. Если эти экстракты не дают полос для ПЦР (если делать 16S) или обнаруживаемую концентрацию после подготовки библиотеки (метатранскриптомика), то они, вероятно, действительно потерпели неудачу.
Если следовать протоколу 16S, яркие полосы после ПЦР-амплификации, как видно из скважин 4 и 6 на рисунке 1,указывают на успех, в то время как отсутствие полос, как видно в других скважинах в верхнем ряду, указывает на отказ. Кроме того, яркая полоса в гелевой полосе, содержащая отрицательный контроль ПЦР, также будет указывать на сбой, поскольку было бы рискованно предполагать, что загрязнение, влияющее на отрицательный контроль (ы), не повлияло на образцы.
Как для 16S, так и для метатранскриптомики успех секвенирования можно оценить, посмотрев на количество полученных последовательностей(рисунок 2). Образцы 16S должны иметь не менее 1000 последовательностей, причем не менее 5000 должны быть идеальными(рисунок 2A). Аналогичным образом, образцы метатранскриптомики должны иметь не менее 500 000 последовательностей, причем по крайней мере 2 000 000 должны быть идеальными(рисунок 2B). Образцы с меньшим количеством последовательностей, чем эти минимумы, не должны использоваться для анализов, поскольку они могут не точно представлять их бактериальное сообщество. Тем не менее, образцы, которые попадают между минимальным и идеальным, все еще могут использоваться, хотя результаты следует интерпретировать более осторожно, если многие образцы попадают в этот диапазон.
Успех последующего анализа может быть определен просто на основе того, были ли получены ожидаемые выходные файлы или нет. В любом случае, такие программы, как QIIME2 и R(рисунок 3),должны позволять оценивать потенциальные значимые различия между бактериальными сообществами на основе фрекинга. Данные для рисунка 3 были получены путем сбора образцов отложений с двадцати одного различных участков в тринадцати различных потоках для анализа 16S и метатранскриптомики. Из этих двадцати одного участка двенадцать из них находились ниже по течению от гидроразрыва пласта и классифицировались как HF+, а девять из них находились либо выше по течению от деятельности по фрекингу, либо в водосборном бассейне, где фрекинг не проводился; эти потоки были классифицированы как HF-. Помимо наличия гидроразрыва пласта, потоки были сопоставимы в остальном.
Эти различия могут принимать форму последовательных структурных изменений в зависимости от статуса фрекинга. Если бы это было так, то ожидается, что выборки HF+ и HF- будут группироваться отдельно друг от друга на графике PCoA, как это показано на рисунке 3A и рисунке 3B. Чтобы подтвердить, что эти кажущиеся сдвиги являются не просто артефактом метода рукоположения, необходим дальнейший статистический анализ. Например, тест PERMANOVA22 на матрице расстояний, на которых основаны рисунки 3A и 3B, выявил значительную кластеризацию, основанную на статусе фрекинга, что означает, что разделение, наблюдаемое на участке, согласуется с различиями между бактериальными сообществами образцов, а не с артефактом рукоположения. Значительный результат PERMANOVA или ANOSIM является убедительным свидетельством последовательных различий между HF+ и HF-образцами, что указывает на то, что на образцы HF+ повлиял фрекинг, в то время как высокое значение p указывает на то, что образцы не были затронуты. Метатранскриптомные данные также могут быть визуализированы и оценены с использованием тех же методов.
Изучение дифференциальных особенностей (микробов или функций) может выявить доказательства того, что образцы также были затронуты. Одним из методов определения дифференциальных признаков является создание случайной модели леса. Модель случайного леса может быть использована для того, чтобы увидеть, насколько хорошо можно правильно классифицировать состояние фрекинга образцов. Если модель работает лучше, чем ожидалось случайно, это будет дополнительным доказательством различий, зависящих от статуса фрекинга. Более того, наиболее важные предикторы выявят, какие признаки наиболее важны для правильной дифференциации выборок(рисунок 3C). Эти характеристики также имели бы постоянно разные значения в зависимости от статуса фрекинга. Как только эти дифференциальные особенности определены, литература может быть просмотрена, чтобы увидеть, были ли они ранее связаны с фрекингом. Тем не менее, может быть сложно найти исследования, которые определяли дифференциальные функции, так как большинство из них использовали только композиционные данные 16S рРНК. Поэтому для оценки последствий дифференциальных функций одним из возможных методов было бы увидеть, были ли они ранее связаны с потенциальной устойчивостью к биоцидам, обычно используемым в жидкости для фрекинга, или они могут помочь в переносе сильно соленых условий. Кроме того, изучение функционального профиля интересующего таксона может выявить доказательства воздействия фрекинга(рисунок 3D). Например, если таксон идентифицирован как дифференциальный с помощью случайной лесной модели, его профиль устойчивости к противомикробным препаратам в образцах HF+ может быть сопоставлен с его профилем в HF-пробах, и если они сильно различаются, это может свидетельствовать о том, что жидкость для фрекинга, содержащая биоциды, попала в поток.
| ПримерID | Концентрация (нг/мкл) |
| 1 | 1.5 |
| 2 | 1.55 |
| 3 | 0.745 |
| 4 | 0.805 |
| 5 | 7.82 |
| 6 | 0.053 |
| 7 | 0.248 |
| 8 | 0.945 |
| 9 | 1.82 |
| 10 | 0.804 |
| 11 | 0.551 |
| 12 | 1.69 |
| 13 | 4.08 |
| 14 | Below_Detection |
| 15 | 7.87 |
| 16 | 0.346 |
| 17 | 2.64 |
| 18 | 1.15 |
| 19 | 0.951 |
Таблица 1: Пример концентрации ДНК на основе анализа высокой чувствительности ДНК флуорометра 1x DS. Извлечение всех этих образцов, за исключением 14, будет считаться успешным из-за наличия обнаруживаемых количеств ДНК.
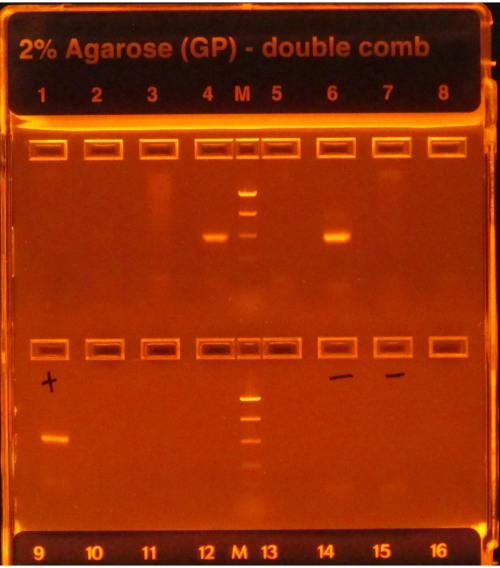
Рисунок 1:Пример электронного геля с продуктами ПЦР. Гель был предварительно окрашен и визуализирован под ультрафиолетовым светом, заставляя любую ДНК, присутствующую на нем, светиться. ПЦР работала для образцов в скважинах 4 и 6 в первом ряду, так как они оба имели одну единственную яркую полосу ожидаемого размера (исходя из лестницы). ПЦР для образцов в других шести скважинах не удалось, так как они не произвели никаких полос. Положительный контроль (первая скважина, второй ряд) имел яркую полосу, указывающую на то, что ПЦР была выполнена правильно, а отрицательные контрольные (скважины 6 и 7, второй ряд) не имели никаких полос, что указывает на то, что образцы не были загрязнены. Если бы у отрицательного была такая же яркая полоса, как у образцов, ПЦР считалась бы неудачной, поскольку было бы рискованно предполагать, что образцы имели ампликоны, которые не были просто результатом загрязнения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
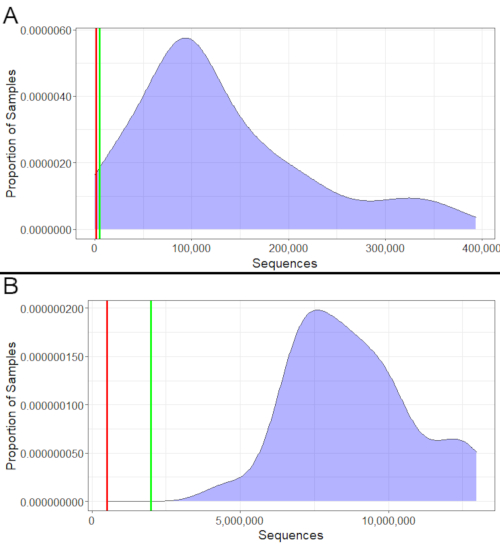
Рисунок 2:Количество примеров последовательностей. (A) 16S примеры последовательностей. Почти все эти образцы 16S имели более 1000 последовательностей. Очень немногие, у которых было менее 1000 последовательностей, должны быть исключены из последующих анализов, поскольку у них было недостаточно последовательностей для точного представления их бактериальных сообществ. Несколько последовательностей имели от 1000 до 5000 последовательностей; хотя они и не идеальны, они все равно будут пригодны для использования, поскольку они превышают минимум, и большинство образцов также превышают идеальный минимум в 5000. (B) Пример метатранскриптомики имеет значение. Все выборки превышали как минимальное (500 000), так и идеальное минимальное (2 000 000) количество последовательностей. Таким образом, последовательность была успешной для всех из них, и все они могли быть использованы в последующем анализе. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
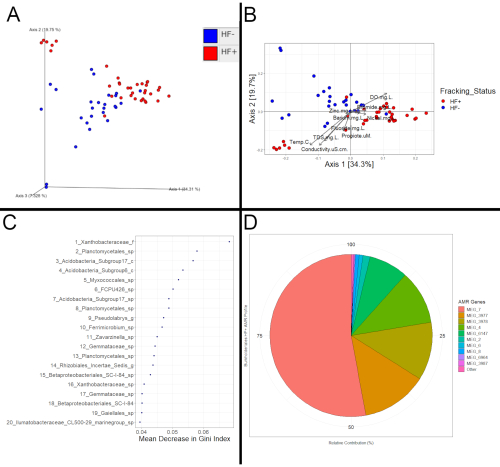
Рисунок 3:Пример анализа. (A)График PCoA на основе координат, рассчитанных с помощью взвешенной матрицы расстояний Unifrac, созданной и визуализированной с помощью QIIME2. (B) График PCoA на основе координат, рассчитанных с помощью взвешенной матрицы расстояний Unifrac, экспортированной из QIIME2. Координаты были визуализированы с использованием пакетов Phyloseq и ggplot2 в R. Векторы метаданных были подогнаны к графику с помощью пакета Vegan. Каждая точка представляет бактериальное сообщество образца, причем более близкие точки указывают на более похожие составы сообществ. Наблюдалось кластеризация на основе состояния фрекинга для этих проб отложений 16S (PERMANOVA, p=0,001). Кроме того, векторы показывают, что образцы HF+, как правило, имеют более высокие уровни бария, бромида, никеля и цинка, что соответствовало различному составу бактериального сообщества по сравнению с образцами HF-. (C)График наилучших предикторов для случайной модели леса, которая проверяла, где бактериальное изобилие может быть использовано для прогнозирования состояния фрекинга среди образцов. Модель случайного леса была создана с помощью R с помощью пакета randomForest. Показаны 20 лучших предикторов, а также результирующее уменьшение примесей (измерение количества HF+ и HF-образцов, сгруппированных вместе) в виде среднего снижения индекса Джини при их использовании для разделения образцов. (D) Круговая диаграмма, показывающая профиль устойчивости к противомикробным препаратам профиля Burkholderiales на основе метатранскриптомных данных. Последовательности были впервые аннотированы Kraken2, чтобы определить, к каким таксонам они принадлежат. Затем BLAST был использован с этими аннотированными последовательностями и базой данных MEGARes 2.0 для определения того, какие гены устойчивости к противомикробным препаратам (в форме «MEG_#») активно экспрессируются. Затем были извлечены гены устойчивости к противомикробным препаратам, экспрессируемые членами Burkholderiales, чтобы увидеть, какие из них наиболее распространены среди этого таксона. Несмотря на то, что метатранскриптомика является более дорогостоящей и трудоемкой, она позволяет проводить функциональный анализ, подобный этому, который не может быть выполнен с данными 16S. Примечательно, что для этого примера анализа был использован Kraken2 вместо HUMAnN2. Kraken2 быстрее, чем HUMAnN2; однако он выводит только композиционную информацию, а не состав, вклад, функции (гены) и пути, как это делает HUMAnN2. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный файл: пример конвейера метатранскриптомики. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Методы, описанные в этойстатье,были разработаны и усовершенствованы в ходе нескольких исследований, опубликованных нашей группой в период с 2014 по 2018 год7,8,10 и были успешно использованы в совместном проекте по исследованию воздействия фрекинга на водные сообщества в трехлетнем проекте, который вскоре представит документ для публикации. Эти методы будут по-прежнему использоваться в течение оставшейся части проекта. Кроме того, в другой современной литературе, исследующей влияние фрекинга на потоки и экосистемы, описываются аналогичные методы сбора, обработки и анализа проб7,8,10,11. Однако ни в одной из этих работ не использовался метатранскриптомный анализ, что делает эту статью первой, описывающей, как эти анализы могут быть использованы для выяснения влияния фрекинга на близлежащие потоки. Кроме того, методы, представленные здесь для сбора проб, являются более подробными, как и шаги, предпринятые во избежание загрязнения.
Одним из наиболее важных шагов нашего протокола является первоначальный сбор и сохранение образцов. Отбор проб и сбор в полевых условиях сопряжены с определенными проблемами, поскольку поддержание асептической или стерильной среды во время сбора может быть затруднено. На этом этапе жизненно важно избегать заражения образцов. Для этого следует надевать перчатки, а также допускать контакт с образцами только стерильные контейнеры и инструменты. Образцы также должны быть немедленно помещены на лед после сбора, чтобы смягчить деградацию нуклеиновых кислот. Добавление коммерческого консерванта нуклеиновых кислот при сборе также может увеличить выход нуклеиновых кислот и позволить хранить образцы в течение более длительных периодов времени после сбора. Всякий раз, когда выполняется экстракция нуклеиновых кислот, важно использовать соответствующее количество образца, слишком много может засорить спиновые фильтры, используемые для экстракции (для тех протоколов, которые их используют), но слишком мало может привести к низкому выходу. Обязательно следуйте инструкциям для любого используемого комплекта.
Подобно полевому сбору, предотвращение или минимизация загрязнения также важны во время экстракции нуклеиновых кислот и подготовки образцов, особенно при работе с образцами с низким выходом нуклеиновых кислот, такими как субоптимальные образцы отложений (образцы, содержащие большое количество гравия или горных пород) или образцы воды. Поэтому, как и при сборе проб, перчатки следует носить на всех этих этапах, чтобы уменьшить загрязнение. Кроме того, все рабочие поверхности, используемые во время лабораторных процедур, должны быть предварительно стерилизовано путем протирания 10% раствором отбеливателя, а затем 70% раствором этанола. Для этапов пипетки (3-6) следует использовать наконечники фильтров, чтобы избежать загрязнения из-за самой пипетки, причем наконечники меняются каждый раз, когда они касаются нестерильной поверхности. Все инструменты, используемые для лабораторных работ, включая пипетки, следует протирать до и после растворами отбеливателя и этанола. Для оценки загрязнения экстракционные заготовки и негативы (стерильная жидкость) должны быть включены в каждый набор экстракции нуклеиновых кислот и реакций ПЦР. Если количественная оценка после экстракции показывает обнаруживаемое количество ДНК / РНК в отрицательных результатах, экстракции могут быть повторены, если осталось достаточное количество образца. Если отрицательные образцы для ПЦР показывают амплификацию, следует выполнить поиск и устранение неисправностей для определения источника, а затем образцы должны быть повторно использованы. Для учета низких уровней загрязнения рекомендуется упорядочить экстракционные заготовки и отрицательные ПЦР-негативы таким образом, чтобы загрязняющие вещества могли быть идентифицированы и удалены, при необходимости, в ходе вычислительного анализа. И наоборот, амплификация ПЦР также может потерпеть неудачу из-за различных причин. Для образцов окружающей среды ингибирование реакции ПЦР часто является виновником, что может быть связано с различными веществами, мешающими Taq полимеразе23. При подозрении на ингибирование для разбавления экстрактов ДНК может быть использована вода класса ПЦР (см. Таблицу материалов).
Этот протокол имеет несколько заметных ограничений и потенциальных трудностей. Сбор проб может быть сложным как для проб воды, так и для проб отложений. Чтобы получить достаточное количество биомассы, в идеале 1 л речной воды должен быть протолкнут через фильтр. Поры фильтра должны быть небольшими, чтобы захватывать микробы, но также могут задерживать осадок. Если в воде много осадков из-за недавних осадков, фильтр может засориться, что затрудняет проталкивание всего объема через фильтр. Для сбора отложений может быть сложно оценить глубину осадка во время сбора. Кроме того, важно обеспечить, чтобы собранные отложения были преимущественно почвой, поскольку галька и горные породы приведут к снижению выхода нуклеиновых кислот и могут не быть точным представлением микробного сообщества. Наконец, также важно, чтобы образцы хранились на льду после сбора, особенно если консервант не используется.
Хотя этот протокол охватывает как метатранскриптомику, так и лабораторные протоколы 16S, следует подчеркнуть, что эти два метода очень различаются как по процессу, так и по типу данных, которые они предоставляют. Ген 16S рРНК является обычно целевой областью, высоко сохраненной в бактериях и археях и полезной для характеристики бактериального сообщества в образце. Несмотря на целенаправленный и специфический подход, разрешение на уровне видов часто недостижимо, и характеристика новых расходящихся видов или штаммов затруднена. Напротив, метатранскриптомика — это более широкий подход, который захватывает все активные гены и микробы, присутствующие в образце. В то время как 16S предоставляет только данные для идентификации, метатранскриптомика может предоставить функциональные данные, такие как экспрессированные гены и метаболические пути. Оба они ценны, и в сочетании они могут выявить, какие бактерии присутствуют и какие гены они экспрессируют.
В данной работе описываются методы полевого сбора и обработки проб как для 16S рРНК, так и для метатранскриптомного анализа в контексте изучения фрекинга. Кроме того, в нем подробно описываются методы сбора высококачественной ДНК / РНК из образцов с низким содержанием биомассы и для долгосрочного хранения. Методы, описанные здесь, являются кульминацией нашего опыта сбора и обработки образцов в наших усилиях по изучению того, как фрекинг влияет на близлежащие потоки путем изучения структуры и функции их микробных сообществ. Микробы быстро реагируют на нарушения, и, следовательно, какие микробы присутствуют и гены, которые они экспрессируют, могут предоставить информацию о влиянии фрекинга на экосистемы. В целом, эти методы могут быть бесценными в нашем понимании того, как фрекинг влияет на эти важные экосистемы.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Авторы хотели бы отметить источники финансирования проектов, которые привели к разработке этих методов, причем этими источниками являются: Медицинский институт Говарда Хьюза (http://www.hhmi.org) через Программу научного образования для подготовки к колледжу и бакалавриату, а также Национальный научный фонд (http://www.nsf.gov) через награды NSF DBI-1248096 и CBET-1805549.
Материалы
| Name | Company | Catalog Number | Comments |
| 200 Proof Ethanol | Thermo Fisher Scientific | A4094 | 400 mL need to be added to Buffer PE (see Qiagen QIAQuck Gel Extraction kit protocol) and 96 mL needs to be added to the DNA/RNA Wash Buffer (see ZymoBIOMICS DNA/RNA Miniprep kit protocol). Additional ethanol is needed for the ZymoBIOMICS DNA/RNA Miniprep and NEBNext® Ultra™ II RNA Library Prep with Sample Purification Beads kits. |
| Agarose | Thermo Fisher Scientific | BP1356-100 | 100 g per bottle. 0.6 g of agarose would be needed to make one 2% 30 mL gel. |
| Disinfecting Bleach | Walmart (Clorox) | No catalog number | Use a 10% bleach solution for cleaning the work area before and after lab procedures |
| DNA gel loading dye | Thermo Fisher Scientific | R0611 | Each user-made (i.e. non-e-gel) should include loading dye with all of the samples in the ratio of 1 µL dye to 5 µL sample |
| DNA ladder | MilliporeSigma | D3937-1VL | A ladder should be run on every gel/e-gel |
| DNA/RNA Shield (2x) | Zymo Research | R1200-125 | 3 mL per sediment sample (50 mL conical) and 2 mL per water sample (filter) |
| Ethidium bromide | Thermo Fisher Scientific | BP1302-10 | Used for staining user-made e-gels |
| Forward Primer | Integrated DNA Technologies (IDT) | 51-01-19-06 | 0.5 µL per PCR reaction |
| Isopropanol | MilliporeSigma | 563935-1L | Generally less than 2 mL per library. Volume needed varies by mass of excised gel fragment (see Qiagen QIAQuick Gel Extraction kit protocol). |
| PCR-grade water | MilliporeSigma | 3315932001 | 13 µL per PCR reaction (assuming 1 µL of sample DNA template is used) |
| Platinum Hot Start PCR Master Mix (2x) | Thermo Fisher Scientific | 13000012 | 10 µL per PCR reaction |
| Reverse Primer | Integrated DNA Technologies (IDT) | 51-01-19-07 | 0.5 µL per PCR reaction |
| TBE Buffer (Tris-borate-EDTA) | Thermo Fisher Scientific | B52 | 1 L of 10x TBE buffer (30 mL of 1x TBE buffer would be needed to make one 30 mL gel) |
| 1 L bottle | Thermo Fisher Scientific | 02-893-4E | One needed per stream (the same bottle can be used for multiple streams if it is sterilized between uses) |
| 1.5 mL Microcentrifuge tubes | MilliporeSigma | BR780400-450EA | 5 microcentrifuge tubes are needed per DNA extraction and an additional 3 are needed to purify RNA (see ZymoBIOMICS DNA/RNA Miniprep kit protocol) |
| 2% Agarose e-gel | Thermo Fisher Scientific | G401002 | Each gel can run 10 samples (so 9 with a PCR negative and 8 if the extraction negative is run on the same gel) |
| 50 mL Conicals | CellTreat | 229421 | 1 50 mL conical needed per sediment samples |
| 500 mL Beaker | MilliporeSigma | Z740580 | Only 1 needed (for flame sterilization) |
| Aluminum foil | Walmart (Reynolds KITCHEN) | No number | Aluminum foil can be folded and autoclaved. The part not exposed to the environment can then be used as a sterile, DNA and RNA free surface for processing filters (one folded piece per filter to avoid cross-contamination) |
| Autoclave | Gettinge | LSS 130 | Only one needed |
| Centrifuge | MilliporeSigma | EP5404000138-1EA | Only 1 needed |
| Cooler | ULINE | S-22567 | Just about any cooler can be used. This one is listed due to being made of foam, making it lighter and thus easier to take along for field sampling. |
| Disruptor Genie | Bio-Rad | 3591456 | Only one needed |
| Electrophoresis chamber | Bio-Rad | 1664000EDU | Only 1 needed |
| Electrophoresis power supply | Bio-Rad | 1645050 | Only 1 needed |
| Freezer (-20 C) | K2 SCIENTIFIC | K204SDF | One needed to store DNA extracts |
| Freezer (-80 C) | K2 SCIENTIFIC | K205ULT | One needed to store RNA extracts |
| Gloves | Thermo Fisher Scientific | 19-020-352 | The catalog number is for Medium gloves. |
| Heat block | MilliporeSigma | Z741333-1EA | Only one needed |
| Lab burner | Sterlitech | 177200-00 | Only one needed |
| Laminar Flow Hood | AirClean Systems | AC624LFUV | Only 1 needed |
| Library purification kit | Qiagen | 28704 | One kit has enough for 50 reactions |
| Magnet Plate | Alpaqua | A001219 | Only one needed |
| Microcentrifuge | Thermo Fisher Scientific | 75004061 | Only one needed |
| Micropipette (1000 µL volume) | Pipette.com | L-1000 | Only 1 needed |
| Micropipette (2 µL volume) | Pipette.com | L-2 | Only 1 needed |
| Micropipette (20 µL volume) | Pipette.com | L-20 | Only 1 needed |
| Micropipette (200 µL volume) | Pipette.com | L-200R | Only 1 needed |
| NEBNext Ultra II RNA Library Prep with Sample Purification Beads | New England BioLabs Inc. | E7775S | One kit has enough reagents for 24 samples. |
| Parafilm | MilliporeSigma | P7793-1EA | 2 1" x 1" squares are needed per filter |
| PCR Tubes | Thermo Fisher Scientific | AM12230 | One tube needed per reaction |
| Pipette tips (for 1000 µL volume) | Pipette.com | LF-1000 | Pack of 576 tips |
| Pipette tips (for 20 µL volume) | Pipette.com | LF-20 | Pack of 960 tips |
| Pipette tips (for 200 µL volume) | Pipette.com | LF-250 | Pack of 960 tips |
| PowerWulf ZXR1+ computer cluster | PSSC Labs | No number | This is just an example of a supercomputer powerful enough to perform metatranscriptomics analysis in a timely manner. Only one needed. |
| Qubit fluorometer starter kit | Thermo Fisher Scientific | Q33239 | Comes with a Qubit 4 fluorometer, enough reagent for 100 DNA assays, and 500 Qubit tubes |
| Scoopula | Thermo Fisher Scientific | 14-357Q | Only one needed |
| Sterile blades | AD Surgical | A600-P10-0 | One needed per filter |
| Sterivex-GP Pressure Filter Unit | MilliporeSigma | SVGP01050 | 1 filter needed per water sample |
| Thermocycler | Bio-Rad | 1861096 | Only one needed |
| Vise-grip | Irwin | 2078500 | Only one needed (for cracking open the filters) |
| Vortex-Genie 2 | MilliporeSigma | Z258415-1EA | Only 1 needed |
| WHIRL-PAK bags | ULINE | S-22729 | 1 needed per filter |
| ZymoBIOMICS DNA/RNA Miniprep kit | Zymo Research | R2002 | One kit has enough reagents for 50 samples. |
Ссылки
- The process of unconventional natural gas production. US EPA Available from: https://www.epa.gov/uog/process-unconventional-natural-gas-production (2013)
- Brittingham, M. C., Maloney, K. O., Farag, A. M., Harper, D. D., Bowen, Z. H. Ecological risks of shale oil and gas development to wildlife, aquatic resources, and their habitats. Environmental Science & Technology. 48 (19), 11034-11047 (2014).
- McBroom, M., Thomas, T., Zhang, Y. Soil erosion and surface water quality impacts of natural gas development in East Texas, USA. Water. 4 (4), 944-958 (2012).
- Maloney, K. O., Weller, D. E. Anthropogenic disturbance, and streams: land use and land-use change affect stream ecosystems via multiple pathways. Freshwater Biology. 56 (3), 611-626 (2011).
- Meyer, J. L., et al. The contribution of headwater streams to biodiversity in river networks1. JAWRA Journal of the American Water Resources Association. 43 (1), 86-103 (2007).
- Alexander, R. B., Boyer, E. W., Smith, R. A., Schwarz, G. E., Moore, R. B. The role of headwater streams in downstream water quality. Journal of the American Water Resources Association. 43 (1), 41-59 (2007).
- Ulrich, N., et al. Response of aquatic bacterial communities to hydraulic fracturing in Northwestern Pennsylvania: A five-year study. Scientific Reports. 8 (1), 5683 (2018).
- Chen See, J. R., et al. Bacterial biomarkers of Marcellus shale activity in Pennsylvania. Frontiers in Microbiology. 9, 1697 (2018).
- Rausch, P., et al. Comparative analysis of amplicon and metagenomic sequencing methods reveals key features in the evolution of animal metaorganisms. Microbiome. 7 (1), 133 (2019).
- Louca, S., Doebeli, M., Parfrey, L. W. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem. Microbiome. 6 (1), 41 (2018).
- Trexler, R., et al. Assessing impacts of unconventional natural gas extraction on microbial communities in headwater stream ecosystems in Northwestern Pennsylvania. Frontiers in Microbiology. 5, 522 (2014).
- Mumford, A. C., et al. Shale gas development has limited effects on stream biology and geochemistry in a gradient-based, multiparameter study in Pennsylvania. Proceedings of the National Academy of Sciences. 117 (7), 3670-3677 (2020).
- JoVE Core Biology DNA Isolation. Journal of Visualized Experiments Available from: https://www.jove.com/cn/science-education/10814/dna-isolation (2020)
- Oxford Gene Technology DNA Storage and Quality. OGT Available from: https://www.ogt.com/resources/literature/403_dna_storage_and_quality (2011)
- ThermoFisher SCIENTIFIC Technical Bulletin #159: Working with RNA. Thermoscientific Available from: https://www.thermofisher.com/us/en/home/references/ambion-tech-support/nuclease-enzymes/general-articles/working-with-rna.html (2020)
- QIAGEN AllPrep DNA/RNA Mini Kit. Qiagen Available from: https://www.qiagen.com/us/products/discovery-and-translational-research/dna-rna-purification/multianalyte-and-virus/allprep-dnarna-mini-kit/#orderinginformation (2020)
- ZymoBIOMICS DNA/RNA Miniprep Kit. Zymo Research Available from: https://www.zymoresearch.com/products/zymobiomics-dna-rna-miniprep-kit (2020)
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- 16S Illumina amplicon protocol: Earth microbiome project. Earth microbiome project Available from: https://earthmicrobiome.org/protocols-and-standards/16s/ (2018)
- Gel Purification: Binding, washing and eluting a sample | Protocol. Journal of Visualized Experiments Available from: https://www.jove.com/v/5063/gel-purification (2020)
- New England Biolabs protocol for the use with NEBNext Poly(A) mRNA magnetic isolation module (E7490) and NEBNext Ultra II RNA library prep kit for Illumina (E7770, E7775). New England Biolabs Available from: https://www.neb.com/protocols/2017/03/04/protocol-for-use-with-purified-mrna-or-rrna-depleted-rna-and-nebnext-ultra-ii-rna-library-prep-ki (2020)
- Anderson, M. J. Permutational multivariate analysis of variance (PERMANOVA). Wiley StatsRef: Statistics Reference Online. , 1-15 (2017).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены