A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
15 N CPMG التشتت الاسترخاء للتحقيق في ديناميات تشكيل البروتين على مقياس زمني μs-ms
In This Article
Summary
هنا، يتم توفير وصف مفصل للبروتوكول المنفذ في المختبر للحصول على وتحليل 15N ملامح التشتت الاسترخاء عن طريق التحليل الطيفي NMR الحل.
Abstract
تلعب الديناميكيات التوافقية للبروتين أدوارا أساسية في تنظيم الحفز الأنزيمي ، وربط الليغند ، والتستوستيرون ، والإشارات ، وهي عمليات بيولوجية مهمة. إن فهم كيفية تحكم التوازن بين البنية والديناميكيات للوظيفة البيولوجية هو حدود جديدة في البيولوجيا الهيكلية الحديثة وأشعل العديد من التطورات التقنية والمنهجية. من بين هذه, CPMG الاسترخاء حل التشتت أساليب NMR توفير فريدة من نوعها, معلومات الدقة الذرية على هيكل, الحركية, والديناميكا الحرارية من البروتين تشكيلية التوازنات التي تحدث على مقياس زمني μs-ms. هنا ، تقدم الدراسة بروتوكولات مفصلة للحصول على وتحليل تجربة تشتت الاسترخاء 15N. على سبيل المثال ، يظهر خط أنابيب لتحليل ديناميات μs-ms في المجال C-terminal من البكتيريا Enzyme I.
Introduction
كار بورسيل ميبوم جيل (CPMG) يتم استخدام تجارب تشتت الاسترخاء (RD) على قاعدة روتينية لتوصيف التوازنات التوافقية التي تحدث على مقياس الوقت μs-ms عن طريق التحليل الطيفي NMR الحل1،2،3،4،5. بالمقارنة مع الطرق الأخرى للتحقيق في الديناميات التوافقية ، فإن تقنيات CPMG سهلة التنفيذ نسبيا على مطياف NMR الحديث ، ولا تتطلب خطوات إعداد عينات متخصصة (أي التبلور أو تجميد العينة أو المحاذاة ، و / أو اقتران التكافؤ مع علامة فلورية أو مغناطيسية) ، وتوفر وصفا شاملا للتوازنات التركيبية العائدة للمعلومات الهيكلية والحركية والدينامية الحرارية حول عمليات التبادل. ولكي تقدم تجربة CPMG تقريرا عن التوازن التوافقي، يجب تطبيق شرطين: '1' يجب أن تكون للدورات المعدلة بالرنين المغناطيسي الملاحظة تحولات كيميائية مختلفة في الولايات التي تخضع لتبادل تشكيلي (حالات صغرى) و'2' يجب أن يحدث التبادل في مقياس زمني يتراوح بين ~ 50 ميكروس إلى ~ 10 مللي ثانية. في ظل هذه الظروف، ومعدل الاسترخاء العرضي الملاحظ 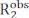 () هو مجموع R2 الجوهرية (R2 تقاس في غياب ديناميات ميكروس مللي ثانية،
() هو مجموع R2 الجوهرية (R2 تقاس في غياب ديناميات ميكروس مللي ثانية، 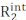 ) ومساهمة الصرف إلى الاسترخاء العرضي (Rex). يمكن إخمادمساهمة R ex إلى R2obs تدريجيا عن طريق تقليل التباعد بين النبضات 180 درجة التي تشكل كتلة CPMG من تسلسل النبض ، ويمكن تصميم منحنيات RD الناتجة باستخدام نظرية Bloch-McConnell للحصول على الفرق في التحول الكيميائي بين الدول الصغيرة ، والسكان الكسور في كل ولاية صغيرة ، ومعدلات التبادل بين الدول الصغيرة (الشكل 1)1،2،3.
) ومساهمة الصرف إلى الاسترخاء العرضي (Rex). يمكن إخمادمساهمة R ex إلى R2obs تدريجيا عن طريق تقليل التباعد بين النبضات 180 درجة التي تشكل كتلة CPMG من تسلسل النبض ، ويمكن تصميم منحنيات RD الناتجة باستخدام نظرية Bloch-McConnell للحصول على الفرق في التحول الكيميائي بين الدول الصغيرة ، والسكان الكسور في كل ولاية صغيرة ، ومعدلات التبادل بين الدول الصغيرة (الشكل 1)1،2،3.
وقد تم الإبلاغ عن عدة تسلسلات نبض مختلفة وبروتوكولات التحليل في الأدبيات ل15تجارب N CPMG. هنا، يتم وصف البروتوكول المنفذ في المختبر. وعلى وجه الخصوص، سيتم إدخال الخطوات الحاسمة لإعداد عينة NMR، وإعداد واكتساب تجارب NMR، ومعالجة وتحليل بيانات NMR(الشكل 2). لتسهيل نقل البروتوكول إلى مختبرات أخرى، يتم توفير برنامج النبض، ومخطوطات المعالجة والتحليل، وإحدى مجموعات البيانات على سبيل المثال كملفات تكميلية وهي متاحة للتنزيل في (https://group.chem.iastate.edu/Venditti/downloads.html). يتضمن تسلسل النبض المقدم دورة مرحلية من أربع خطوات في كتلة CPMG لقمع القطع الأثرية المعتمدة على الإزاحة6 ويتم ترميزها للحصول على العديد من التجارب المتشابكة. هذه التجارب interleaved لها فترة استرخاء متطابقة ولكن أعداد مختلفة من نبضات إعادة التركيز من أجل تحقيق مختلف الحقول CPMG7. من المهم أيضا ملاحظة أن برنامج النبض الموصوف يقيس 15N R2 من مكون TROSY لإشارة NMR8. عموما، تم تطبيق البروتوكول بنجاح لتوصيف التبادل التوافقي في البروتينات متوسطة وكبيرة الحجم4،5،9،10. بالنسبة للأنظمة الأصغر (<20 كيلودا)، ينصح باستخدام تسلسل النبض الذبذبي الأحادي الترونو نووي (HSQC)11و12.
Protocol
1. إعداد عينة NMR
- التعبير عن وتنقية 2H،15N-labled عينة من البروتين من الفائدة.
ملاحظة: في حين يمكن استخدام عينة بروتين 15N المسمى للحصول على تجربة CPMG RD، perdeuteration (حيثما أمكن) يزيد بشكل كبير من جودة البيانات التي تم الحصول عليها. بروتوكولات لإنتاج البروتينات perdeuterated متوفرة في الأدب13. - المخزن المؤقت تبادل عينة البروتين المنقى في مخزن مؤقت NMR degassed.
- نقل عينة NMR إلى أنبوب NMR.
ملاحظة: يجب تحسين تركيز عينة NMR بعناية من أجل زيادة نسبة الإشارة إلى الضوضاء وتقليل حدوث التفاعلات بين البروتين والبروتين. بشكل عام، يتراوح نطاق التركيز النموذجي لتجارب CPMG بين 0.1 و1.0 مللي متر.
2. أول مرة إعداد تجربة NMR
- تحميل وفك الملفات التكميلية.
- نسخ bits.vv الملفات و trosy_15N_cpmg.vv (الموجود في المجلد المسمى برنامج نبض) في دليل برنامج النبض (<باث>/إكسب/ستان/nmr/lists/pp/user).
ملاحظة: تأكد من المسار إلى bits.vv المسرودة في السطر الأول من الملف trosy_15N_cpmg.vv الصحيح. - فتح برنامج الاستحواذ ونسخ تجربة HSQC 1H-15N تشغيل سابقا في تجربة جديدة باستخدام الأمر edc.
- باستخدام pulprogالأمر ، تحميل برنامج نبض trosy_15N_cpmg.vv في التجربة التي تم إنشاؤها حديثا.
- إعداد تجربة CPMG باستخدام التعليمات المتوفرة في نهاية ملف برنامج النبض (trosy_15N_cpmg.vv).
3. الإعداد الروتيني لتجربة NMR
- إدخال العينة في المغناطيس وتنفيذ جميع الخطوات الأساسية للحصول على أي تجربة NMR: قفل وshim العينة؛ تطابق وضبط 1H و 15N القنوات.
- تعيين p1 و p7 لمدة 1H و 15N الثابت 90° البقول, على التوالي.
ملاحظة: للحصول على أفضل النتائج، من المهم معايرة نبض 15N 90 درجة بعناية فائقة. عادة ما يتم إنجاز المعايرة باستخدام عينة 100 mM من 15ن المسمى اليوريا في DMSO كما هو موضح في دليل المطياف. بالإضافة إلى ذلك، من الممكن التحقق من المعايرة مباشرة على عينة NMR العاملة عن طريق الحصولعلى الزيادة الأولى من طيفH-15N HSQC الذي يتم فيه تحويل نبض 15N 90° من الكتلة INEPT الأولى إلى نبض 180 درجة. إذا كانت المعايرة صحيحة، يجب الحصول على قيمة خالية. - في نافذة الامتلاك، قم بتعيين العرض الوسطي الطيفي لأبعاد 1H و 15N.
ملاحظة: مركز الطيف 1H في وتيرة إشارة المياه لقمع المياه الأمثل. - تعيين تأخير الاسترخاء (d30) يساوي 0.7 T2, حيث T2 هو المتوقع 15N وقت الاسترخاء العرضي للبروتين الخاص بك.
ملاحظة: يمكن تحسين قيمة d30 تجريبيا للحصول على أفضل النتائج. - استخدم vclist الأمر لإنشاء قائمة أرقام عدد صحيح المطابقة إلى المعلمة n في الشكل 1A والشكل 1B. تأكد من أن كل إدخال في القائمة يتوافق مع حقل CPMG مختلف (νcpmg) وفقا ل νcpmg = 4 × n / d30. تأكد من أن الرقم الأول في القائمة هو 0 (وهذا يتوافق مع التجربة المرجعية التي يتم تخطي كتلة CPMG وd30 = 0 s) وعدم استخدام أرقام أكبر من 1000 × d30 / 4. أرقام أكبر من هذه العتبة يؤدي إلى νcpmg > كيلوهرتز 1 وقد تلحق الضرر التحقيق.
- تعيين l8 إلى عدد الإدخالات في vclist، l3 إلى عدد من النقاط الحقيقية للبعد غير المباشر (عادة ما يتم تعيين نطاق من 64-200 ل L3)، و 1 td يساوي l8 × l3 × 2.
ملاحظة: تنفيذ الخطوات 3.7-3.11 لتحسين منع المياه. - تعيين كسب المتلقي (rg) إلى 1; فتح ملف برنامج نبض (edcpul) ، انتقل إلى السطر 91 ، وإزالة الفاصلة المنقوطة السابقة goto 999، وحفظ الملف.
- باستخدام الأمر gs ضبط المعلمات spdb0 (أو spdw0) من أجل تقليل كثافة إشارة FID.
- إعادة إدخال الفاصلة المنقوطة في السطر 91 من ملف برنامج النبض وحفظ الملف.
- كرر الخطوات 3.7-3.9 لتحسين spdb11 (سطر 168 من ملف برنامج النبض) ، spdb2 (سطر 179 من ملف برنامج النبض) ، وpldb2 (سطر 184 من ملف برنامج النبض).
- تشغيل rga الأمر لتحسين كسب المتلقي.
- تعيين عدد عمليات المسح(ns)إلى مضاعف 8.
- تشغيل الأمر zg لبدء التجربة.
4. معالجة وتحليل بيانات NMR
- نسخ المجلد المسمى عملية (ملفات تكميلية) في الدليل الذي يحتوي على ملف ser.
- استخدم الملفات sep_fid.com ft2D.com لمعالجة بيانات NMR.
ملاحظة: يتم توفير إرشادات حول كيفية تحرير البرامج النصية المعالجة ضمن sep_fid.com وملفات ft2D.com. - افتح الملفات ucsf المضمنة في الدليل CPMG_Sparky_files في Sparky.
ملاحظة: يتم إنشاء ملفات ucsf بواسطة البرامج النصية المعالجة. هناك ملف ucsf واحد لكل إدخال في vclist. يحتوي الملف الأول ucsf (test_1.ucsf) على التجربة المرجعية. ملفات ucsf اللاحقة (test_2.ucsf، test_3.ucsf,... test_n.ucsf) يتم ترتيبها من أدنى إلى أكبر قيمة من νcpmg. - اختر قمم عبور NMR على طيف NMR المرجعي (test_1.ucsf).
- حدد جميع القمم المتقاطعة التي تم انتقاؤها باستخدام الأمر Sparky pa.
- تشغيل الأمر سباركي rh. يفتح هذا الأمر إطار حوار. حدد ارتفاعات الخيار في نفس الموضع في كل طيف. انقر على الإعداد والتحقق من جميع الأطياف NMR. انقر على تحديث وحفظ ملف الإخراج في دليل العمل. يحتوي ملف الإخراج على مصفوفة شدة الإشارة على جميع أطياف NMR المفتوحة.
ملاحظة: وقد وصفت بروتوكولات أكثر دقة التي تستخدم خط شكل المناسب لاستعادة كثافة من التجارب الزائفة 3D متشابكة في الأدب14. تتوفر برامج متاحة مجانا لتركيب خط الخطوط في https://pint-nmr.github.io/PINT/ (PINT) https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA) وداخل NMRPipe (وحدة nLinLS) و SPARKY (وحدة تكنولوجيا المعلومات). - لكل حقل CPMG، تحويل كثافة إشارة إلى معدلات R2 باستخدام
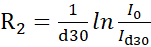 الصيغة، حيث أنا0 وأنا d30 هي كثافة المرجع (vc = 0) واسترخاء (vc > 0) أطياف NMR، على التوالي.
الصيغة، حيث أنا0 وأنا d30 هي كثافة المرجع (vc = 0) واسترخاء (vc > 0) أطياف NMR، على التوالي.
ملاحظة: في الملفات التكميلية، يتم توفير قالب ملف جدول البيانات (R2_calc.xls) المستخدمة لتحويل كثافة الإشارة إلى معدلات R2 وتصور ملفات تعريف RD. - قراءة مستوى الضوضاء في الطيف المرجعي باستخدام St الأمر سباركي ونشر الخطأ على معدلات R2.
ملاحظة: في الملفات التكميلية، يتم توفير قالب ملف جدول البيانات (R2_calc.xls) المستخدمة لنشر الخطأ على معدلات R2 المقاسة.
5. تركيب منحنيات RD
- نسخ المجلد المسمى RD_fitting(ملفات تكميلية)في دليل العمل.
- لكل ذروة NMR المعينة، تقدير Rالسابقين عن طريق حساب
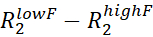 .
. 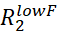
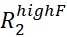
- بصريا فحص منحنيات RD مع Rالسابقين أكبر من مرتين الخطأ المقدر وتجاهل جميع منحنيات RD التي هي صاخبة جدا لتكون على غرار بدقة.
ملاحظة: يمكن نشر الضوضاء على Rex من الأخطاء على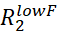 و
و 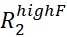 .
. - قم بإعداد ملف إدخال للبرنامج النصي المناسب باستخدام كافة منحنيات RD مع Rex أكبر من مرتين الخطأ المقدر.
ملاحظة: يتم توفير إرشادات مفصلة حول إعداد ملفات الإدخال ضمن البرامج النصية المناسب. يتم توفير ملفات الإدخال المثال كملفات تكميلية. عادة، يتم قياس بيانات RD في حقلين ثابتين مختلفين وتركيبها في وقت واحد. في هذا البروتوكول، مطلوب ملفين إدخال مختلفين(ملفات تكميلية). - احتواء بيانات RD باستخدام البرامج النصية المتوفرة في ملفات تكميلية.
ملاحظة: يتم توفير برنامجين نصيين مختلفين لتنفيذ نوبة عمومية من منحنيات RD المستندة إلى المخلفات أو على التوالي. كلا البرنامجين النصيين تناسب منحنيات RD باستخدام نموذج تبادل موقعين ومعادلة كارفر ريتشاردز. يتم توفير تعليمات أكثر تفصيلا ضمن البرامج النصية المناسب. حزم برامج إضافية مثل Chemex (https://github.com/gbouvignies/ChemEx) وCATIA (https://www.ucl.ac.uk/hansen-lab/catia/) متاحة لتنفيذ البيانات المناسبة باستخدام معادلات بلوك ماكونيل. - اختبار موثوقية المعلمات المجهزة تقدير انخفاض خي2 كدالة فب وكالسابقين.
ملاحظة: يتم توفير خي 2 مخفضة في ملف الإخراج. فب و كالسابقين يمكن تقييدها لقيم محددة باستخدام الحدود الدنيا والعليا في إجراءات المناسب. في نصوصنا، الحدود الدنيا والعليا لpb هي lb(2) وub(2)، على التوالي. الحدود السفلى والعليا ل كالسابقين هي lb(3) وub(3) ، على التوالي. - تقدير الخطأ في المعلمات المجهزة. يمكن القيام بذلك عن طريق تعيين قيمة MC_fac في البرنامج النصي إلى 1 وتكرار المناسب عدة مرات (عادة >20 يكرر). يتم تقدير الخطأ في كل معلمة على أنه الانحراف المعياري للتوزيع.
ملاحظة: إعداد MC_fac إلى 1 يولد مجموعة بيانات اصطناعية يتم فيها إضافة خطأ موزع غاوسي (محسوب استنادا إلى الخطأ التجريبي) إلى البيانات التجريبية.
النتائج
البروتوكول الموصوف هنا يؤدي إلى الحصول على ملامح RD لكل ذروة في الطيف 1H-15N TROSY(الشكل 3A). من ملامح RD المكتسبة، فمن الممكن لتقدير مساهمة الصرف إلى الاسترخاء العرضي N 15من كل مجموعة أميد العمود الفقري(الشكل 3A،3B). من خلال رسمR السابقين
Discussion
تصف هذه المخطوطة البروتوكول المنفذ في المختبر للحصول على 15N RD من البيانات المتعلقة بالبروتينات وتحليلها. وعلى وجه الخصوص، يتم تغطية الخطوات الحاسمة لإعداد عينة NMR، وقياس بيانات NMR، وتحليل ملامح RD. وفيما يلي بعض الجوانب الهامة المتعلقة باقتناء وتحليل تجارب RD. ومع ذلك ، للحصول على وصف ?...
Disclosures
وقد قرأ جميع المؤلفين المخطوطة ووافقوا عليها. نحن لا نعلن عن أي تضارب في المصالح.
Acknowledgements
وقد تم دعم هذا العمل بأموال من NIGMS R35GM133488 ومن صندوق روي ج. كارفر الخيري إلى V.V.
Materials
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
References
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved