Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
15 μs-ms Zaman Ölçeğinde Protein Konformasyonsal Dinamiklerinin Araştırılması için N CPMG Gevşeme Dağılımı
Bu Makalede
Özet
Burada 15 Ngevşeme dispersiyon profillerinin çözelti NMR spektroskopisi ile edinilmesi ve analizi için laboratuvarda uygulanan protokolün detaylı bir açıklaması sağlanmaktadır.
Özet
Protein konformasyonsal dinamikleri, önemli biyolojik süreçler olan enzimatik kataliz, ligand bağlama, allosteri ve sinyalizasyonun düzenlenmesinde temel roller oynar. Yapı ve dinamikler arasındaki dengenin biyolojik işlevi nasıl yönettiğini anlamak, modern yapısal biyolojide yeni bir sınırdır ve çeşitli teknik ve metodolojik gelişmelerin fitilini ateşlemiştir. Bunlar arasında, CPMG gevşeme dispersiyon çözümü NMR yöntemleri, μs-ms zaman ölçeğinde meydana gelen protein konformasyonsal dengenin yapısı, kinetiği ve termodinamiği hakkında benzersiz, atomik çözünürlüklü bilgiler sağlar. Burada, çalışma 15 Ngevşeme dispersiyon deneyinin edinilmesi ve analizi için ayrıntılı protokoller sunmaktadır. Örnek olarak, bakteri Enzim I'in C-terminal etki alanındaki μs-ms dinamiklerinin analizi için boru hattı gösterilmiştir.
Giriş
Carr-Purcell Meiboom-Gill (CPMG) gevşeme dispersiyon (RD) deneyleri, μs-ms zaman ölçeğinde meydana gelen konformasyonel dengeyi NMR spektroskopisi1, 2,3,4,5çözeltisi ile karakterize etmek için rutin bir tabanda kullanılır. Konformasyonel dinamiklerin araştırılması için diğer yöntemlerle karşılaştırıldığında, CPMG tekniklerinin modern NMR spektrometrelerinde uygulanması nispeten kolaydır, özel numune hazırlama adımları (örneğin, kristalizasyon, numune dondurma veya hizalama ve/veya floresan veya paramanyetik etiketli kovalent konjugasyon) gerektirmez ve değişim süreçlerinde yapısal, kinetik ve termodinamik bilgileri döndüren konformasyonel dengenin kapsamlı bir şekilde nitelendirilmesini sağlar. Bir CPMG deneyinin konformasyonsal bir dengeyi rapor edebilmesi için iki koşul geçerli olmalıdır: (i) gözlemlenen NMR dönüşleri, konformasyonsal değişime uğrayan durumlarda farklı kimyasal kaymalara sahip olmalıdır (mikro durumlar) ve (ii) değişimin ~50 μs ile ~10 ms arasında değişen bir zaman ölçeğinde gerçekleşmesi gerekir. Bu koşullar altında, gözlemlenen enine gevşeme oranı ( 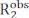 ) iç R 2 'nin(μs-ms dinamiklerinin yokluğunda ölçülen R2)
) iç R 2 'nin(μs-ms dinamiklerinin yokluğunda ölçülen R2) 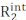 ve enine gevşemeye değişim katkısının (Rex)toplamıdır. R2obs'a Rex katkısı, darbe dizisinin CPMG bloğunu oluşturan 180 ° darbeler arasındaki aralığı azaltarak kademeli olarak söndürülebilir, ve ortaya çıkan RD eğrileri, mikro devletler arasındaki kimyasal kayma farkını, her mikro devletteki kesirli popülasyonu ve mikro devletler arasındaki değişim oranlarını elde etmek için Bloch-McConnell teorisi kullanılarak modellenebilir (Şekil 1)1,2,3.
ve enine gevşemeye değişim katkısının (Rex)toplamıdır. R2obs'a Rex katkısı, darbe dizisinin CPMG bloğunu oluşturan 180 ° darbeler arasındaki aralığı azaltarak kademeli olarak söndürülebilir, ve ortaya çıkan RD eğrileri, mikro devletler arasındaki kimyasal kayma farkını, her mikro devletteki kesirli popülasyonu ve mikro devletler arasındaki değişim oranlarını elde etmek için Bloch-McConnell teorisi kullanılarak modellenebilir (Şekil 1)1,2,3.
Literatürde 15 NCPMG deneyi için birkaç farklı nabız dizisi ve analiz protokolü bildirilmiştir. Burada laboratuvarda uygulanan protokol açıklanmıştır. Özellikle, NMR örneğinin hazırlanması, NMR deneylerinin kurulması ve alınması ve NMR verilerinin işlenmesi ve analizi için önemli adımlar tanıtılacaktır (Şekil 2). Protokolün diğer laboratuvarlara aktarılmasını kolaylaştırmak için, darbe programı, işleme ve analiz komut dosyaları ve bir örnek veri kümesi Ek Dosyalar olarak sağlanır ve (https://group.chem.iastate.edu/Venditti/downloads.html. Sağlanan darbe dizisi, ofset bağımlı yapıtların6'nın bastırılması için CPMG bloğunda dört adımlı bir faz döngüsü içerir ve birkaç aralanmış deneyin alınması için kodlanmıştır. Bu aralanmış deneyler aynı gevşeme süresine sahiptir, ancak farklı CPMG alanları elde etmek için farklı sayıda yeniden odaklama darbeleri7. Açıklanan darbe programının NMR sinyali8'inTROSY bileşeninin 15N R 2'yi ölçtüklerine dikkat etmek de önemlidir. Genel olarak, orta ve büyük boyutlu proteinler 4 ,5,9,10'dakonformasyonsal değişimin karakterizasyonu için protokolbaşarıylauygulanmıştır. Daha küçük sistemler (<20 kDa) için Heteronuclear Tek Kuantum Tutarlılığı (HSQC) tabanlı darbe dizisi11,12 kullanılması önerilir.
Protokol
1. NMR örneğinin hazırlanması
- İlgi proteininin 2H,15N-labled örneğini eksprese edin ve arındırın.
NOT: CPMG RD deneyinin alınması için 15N etiketli protein örneği kullanılabilirken, perdeuterasyon (mümkünse) elde edilen verilerin kalitesini önemli ölçüde artırır. Perdeuterated proteinlerin üretimine ilişkin protokoller literatürde mevcuttur13. - Tampon, saflaştırılmış protein örneğini gazsız bir NMR tamponuna değiştirir.
- NMR örneğini NMR tüpüne aktarın.
NOT: Sinyal-gürültü oranını en üst düzeye çıkarmak ve protein-protein etkileşimlerinin oluşumunu en aza indirmek için NMR örneğinin konsantrasyonunun dikkatlice optimize edilmesi gerekir. Genel olarak, CPMG deneyleri için tipik konsantrasyon aralığı 0.1-1.0 mM'dir.
2. NMR deneyinin ilk kurulumu
- Ek Dosyaları indirin ve fermini çözün.
- Bits.vv ve trosy_15N_cpmg.vv dosyalarını (pulseprogram adlı klasörde bulunur) darbe programı dizinine (
- Edinme yazılımını açın ve daha önce çalıştırılan 1H-15 NHSQC denemesini edckomutunu kullanarak yeni bir denemeye kopyalayın.
- Komut pulprogkullanarak, darbe programı trosy_15N_cpmg.vv yeni oluşturulan deney içine yükleyin.
- Nabız programı dosyasının (trosy_15N_cpmg.vv) sonunda sağlanan yönergeleri kullanarak CPMG denemesini ayarlayın.
3. NMR deneyinin rutin kurulumu
- Numuneyi mıknatısa tanıtın ve herhangi bir NMR deneyinin alınması için tüm temel adımları uygulayın: numuneyi kilitleyin ve dolgunlanın; 1H ve 15 Nkanallarını eşleştirin ve ayarlayın.
- p1 ve p7'yi sırasıyla 1H ve 15 Nsert 90° darbe süresine ayarlayın.
NOT: Optimum sonuçlar için 15 N90° darbeyi büyük bir dikkatle kalibre etmek önemlidir. Kalibrasyon genellikle spektrometre kılavuzunda açıklandığı gibi DMSO'daki 15N etiketli ürenin 100 mM'lik bir örneği kullanılarak gerçekleştirilir. Ek olarak, ilk INEPT bloğunun 15N 90° darbesinin 180 ° darbeye geçirildiği 1 H-15N HSQC spektrumunun ilk artımını alarak kalibrasyonu doğrudan çalışan NMR örneğinde iki kez kontrol etmek mümkündür. Kalibrasyon doğruysa, bir null elde edilmelidir. - Edinme penceresinde, 1 H ve 15 Nboyutları için merkez ve spektral genişliği ayarlayın.
NOT: Optimum su bastırma için 1H spektrumunu su sinyalinin frekansı cinsinden ortalayın. - Gevşeme gecikmesini (d30) 0,7 T2'ye eşit olarak ayarlayın , burada T2 proteininizin beklenen 15N enine gevşeme süresidir.
NOT: D30 değeri, en iyi sonuçları elde etmek için ampirik olarak optimize edilebilir. - Şekil 1A ve Şekil 1B'deki n parametresine karşılık gelen tamsayı numaralarının listesini oluşturmak için komut vclist'inikullanın. Listedeki her girişin νcpmg= 4 x n / d30'a göre farklı bir CPMG alanına (νcpmg) karşılık geldiğine emin olun. Listedeki ilk sayının 0 olduğundan emin olun (bu, CPMG bloğunun atladığı başvuru deneyine karşılık gelir ve d30 = 0 s) ve 1.000 x d30 / 4'ten büyük sayılar kullanmamaya dikkat edin. Bu eşikten daha büyük sayılar νcpmg > 1 kHz ile sonuçlanır ve proba zarar verebilir.
- l8'i vclist'teki girdi sayısına, l3'ü dolaylı boyut için gerçek nokta sayısına (genellikle l3 için 64-200 aralığı ayarlanır) ve l8 x l3 x 2'ye eşit 1 td olarak ayarlayın.
NOT: Su bastırmayı optimize etmek için 3.7-3.11 adımlarını uygulayın. - Alıcı kazancını (rg) 1 olarak ayarlayın; pulse program dosyasını (edcpul) açın, satır 91'e gidin, goto 999'danönceki noktalı virgülü çıkarın ve dosyayı kaydedin.
- Gs komutunu kullanarak, FID sinyalinin yoğunluğunu en aza indirmek için spdb0 (veya spdw0) parametrelerini ayarlayın.
- Darbe programı dosyasının 91.
- SPDB11 (pulse program dosyasının satır 168), spdb2 (pulse program dosyasının satır 179) ve pldb2 (pulse program dosyasının satır 184) optimize etmek için 3.7-3.9 adımlarını tekrarlayın.
- Alıcı kazancını en iyi duruma getirmek için rga komutunu çalıştırın.
- Tarama sayısını (ns) 8'in katına ayarlayın.
- Denemeyi başlatmak için zg komutunu çalıştırın.
4. NMR verilerinin işlenmesi ve analizi
- İşlem ( EkDosyalar) adlı klasörü, ser dosyasını içeren dizine kopyalayın.
- NMR verilerini işlemek için sep_fid.com ve ft2D.com dosyaları kullanın.
NOT: İşleme komut dosyalarının nasıl düzenleneceği ile ilgili talimat sep_fid.com ve ft2D.com dosyalarında sağlanır. - Sparky'deki dizin CPMG_Sparky_files bulunan ucsf dosyalarını açın.
NOT: Ucsf dosyaları işleme komut dosyaları tarafından oluşturulur. Vclist her girdi için bir ucsf dosyası vardır. İlk ucsf dosyası (test_1.ucsf) başvuru deneyini içerir. Sonraki ucsf dosyaları (test_2.ucsf, test_3.ucsf,... test_n.ucsf) en düşükten en büyük νcpmgdeğerine kadar sıralanır. - Referans NMR spektrumunda (test_1.ucsf) NMR çapraz zirvelerini seçin.
- Sparky komut pa'sını kullanarak tüm seçili çapraz tepeleri seçin.
- Sparky komut rh komutunu çalıştırın. Bu komut bir iletişim penceresi açar. Her spektrumda aynı konumdaki seçenek yükseklikleriniseçin. Kur'a tıklayın ve tüm NMR spektrumlarını kontrol edin. Güncelleştir'i tıklatın ve çıktı dosyasını çalışma dizinine kaydedin. Çıkış dosyası, tüm açılan NMR spektrumları üzerindeki sinyal yoğunluklarının matrisini içerir.
NOT: Aralanmış sahte 3D deneylerden kaynaklanan yoğunlukları kurtarmak için çizgi şekli uydurması kullanan daha doğru protokoller literatürde açıklanmıştır14. Hat şekli montajı için serbestçe kullanılabilen yazılım https://pint-nmr.github.io/PINT/ (PINT), https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA) ve NMRPipe (nLinLS modülü) ve SPARKY (it modülü) içinde mevcuttur. - Her CPMG alanı için, I0 ve I
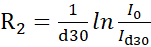 d30 sırasıyla referansın (vc = 0) ve rahat (vc > 0) NMR spektrumunun yoğunluğu olduğu formülü kullanarak sinyal yoğunluklarını R 2 oranlarına dönüştürün.
d30 sırasıyla referansın (vc = 0) ve rahat (vc > 0) NMR spektrumunun yoğunluğu olduğu formülü kullanarak sinyal yoğunluklarını R 2 oranlarına dönüştürün.
NOT: Ek Dosyalar'da, sinyal yoğunluklarını R2 oranlarına dönüştürmek ve RD profillerini görselleştirmek için kullanılan elektronik tablo dosyasının (R2_calc.xls) bir şablonu sağlanır. - Sparky komutunu kullanarak referans spektrumundaki gürültü düzeyini okuyun ve hatayı R2 hızlarında yayın.
NOT: Ek Dosyalar'da, ölçülen R2 oranlarında hatayı yaymak için kullanılan elektronik tablo dosyasının (R2_calc.xls) bir şablonu sağlanır.
5. RD eğrilerini sığdırma
- RD_fitting (Ek Dosyalar) adlı klasörü çalışma dizinine kopyalayın.
- Atanan her NMR zirvesi için, Rex'i hesaplayarak tahmin
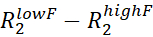 edin ve
edin ve 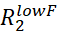
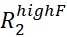 vclist'teki en düşük ve en yüksek νcpmg'de ölçülen R2 oranlarıdır.
vclist'teki en düşük ve en yüksek νcpmg'de ölçülen R2 oranlarıdır.
- Rd eğrilerini tahmini hatanın iki katından daha büyük Rex ile görsel olarak inceleyin ve doğru şekilde modellenemeyecek kadar gürültülü olan tüm RD eğrilerini atın.
NOT: Rex üzerindeki gürültü, üzerindeki ve üzerindeki hatalardan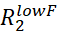
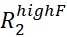 yayılabilir.
yayılabilir. - Rex tahmini hatanın iki katından daha büyük olan tüm RD eğrilerini kullanarak uydurma komut dosyası için bir giriş dosyası hazırlayın.
NOT: Giriş dosyalarının hazırlanmasına ilişkin ayrıntılı talimat, uygun komut dosyaları içinde sağlanır. Örnek giriş dosyaları Ek Dosyalarolarak sağlanır. Genellikle, RD verileri iki farklı statik alanda ölçülür ve aynı anda takılır. Bu protokolde, iki farklı giriş dosyası gereklidir (Ek Dosyalar). - Ek Dosyalar 'da sağlanan komut dosyalarını kullanarak RD verilerini sığdırın.
NOT: RD eğrilerinin artık tabanlı veya genel bir sığdırmasını gerçekleştirmek için sırasıyla iki farklı komut dosyası sağlanır. Her iki komut dosyası da iki siteli değişim modeli ve Carver-Richards denklemi kullanarak RD eğrilerine uyar. Uygun komut dosyaları içinde daha ayrıntılı talimatlar sağlanır. Block-McConnell denklemlerini kullanarak veri uydurmayı gerçekleştirmek için Chemex (https://github.com/gbouvignies/ChemEx) ve CATIA (https://www.ucl.ac.uk/hansen-lab/catia/) gibi ek yazılım paketleri mevcuttur. - Azaltılmış χ 2'yi pb ve k ex işlevi olarak tahmin eden takılı parametrelerin güvenilirliğini testedin.
NOT: Azaltılmış χ2 çıktı dosyasında sağlanır. pb ve kex, montaj prosedürlerinde alt ve üst sınırlar kullanılarak belirli değerlere göre kısıtlanabilir. Komut dosyalarımızda, pb için alt ve üst sınırlar sırasıyla lb(2) ve ub(2'dir. kex için alt ve üst sınırlar sırasıyla lb(3) ve ub(3)'dir. - Takılan parametrelerdeki hatayı tahmin edin. Bu, komut dosyasındaki MC_fac değerini 1 olarak ayarlayarak ve sığdırma işlemi birden çok kez yinelenerek yapılabilir (genellikle >20 yineler). Her parametredeki hata, dağılımın standart sapması olarak tahmin edilir.
NOT: MC_fac 1 olarak ayarlamak, gauss dağıtılmış hatasının (deneysel hataya göre hesaplanır) deneysel verilere eklenmediği sentetik bir veri kümesi oluşturur.
Sonuçlar
Burada açıklanan protokol, 1 H- 15 NTROSY spektrumundaki her tepe için RD profillerinin alınmasıyla sonuçlanır (Şekil 3A). Edinilen RD profillerinden, her omurga amid grubunun 15 Nenine gevşemesine değişim katkısını tahmin etmek mümkündür (Şekil 3A,3B). Rex'i incelenen proteinin 3D yapısı üzerine çizerek, μs-ms zaman ölçeğinde konformasyonsal değişime uğrayan yapısal bölg...
Tartışmalar
Bu makalede, proteinler üzerinde 15 NRD verinin eldei ve analizi için laboratuvarda uygulanan protokol açıklanmaktadır. Özellikle, NMR örneğinin hazırlanması, NMR verilerinin ölçümü ve RD profillerinin analizi için önemli adımlar ele alınmıştır. Aşağıda RD deneylerinin edinimi ve analizi ile ilgili bazı önemli hususlar tartışılmıştır. Bununla birlikte, deney ve veri analizinin daha derinlemesine tanımlanması için, orijinal literatürün dikkatli bir şekilde incelenmesi şidd...
Açıklamalar
Tüm yazarlar makaleyi okudu ve onayladı. Çıkar çatışması ilan ediyoruz.
Teşekkürler
Bu çalışma NIGMS R35GM133488 ve Roy J. Carver Charitable Trust'tan V.V.'ye fonlarla desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
Referanslar
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır