Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
15. N CPMG Dispersión De Relajación Para La Investigación De La Dinámica Conformacional De Proteínas En La Escala De Tiempo μs-ms
En este artículo
Resumen
Aquí, se proporciona una descripción detallada del protocolo implementado en el laboratorio para la adquisición y análisis de perfiles de dispersión de relajación de 15N por solución de espectroscopia de RMN.
Resumen
La dinámica conformacional de proteínas juega un papel fundamental en la regulación de la catálisis enzimática, la unión de ligandos, el alosterismo y la señalización, que son procesos biológicos importantes. Comprender cómo el equilibrio entre la estructura y la dinámica gobierna la función biológica es una nueva frontera en la biología estructural moderna y ha encendido varios desarrollos técnicos y metodológicos. Entre estos, los métodos de RMN de la solución de dispersión de relajación de CPMG proporcionan información única de resolución atómica sobre la estructura, cinética y termodinámica de los equilibrios conformacionales de proteínas que ocurren en la escala de tiempo de μs-ms. Aquí, el estudio presenta protocolos detallados para la adquisición y el análisis de un experimento de dispersión de relajación de 15N. Como ejemplo, se muestra la tubería para el análisis de la dinámica μs-ms en el dominio C-terminal de la enzima I.
Introducción
Carr-Purcell Meiboom-Gill (CPMG) experimentos de dispersión de relajación (RD) se utilizan sobre una base de rutina para caracterizar los equilibrios conformacionales que ocurren en la escala de tiempo de μs-ms por solución espectroscopia de RMN1,2,3,4,5. En comparación con otros métodos para la investigación de la dinámica conformacional, las técnicas de CPMG son relativamente fáciles de implementar en los espectrómetros de RMN modernos, no requieren pasos especializados de preparación de muestras (es decir, cristalización, congelación o alineación de muestras, y / o conjugación covalente con una etiqueta fluorescente o paramagnética), y proporcionan una caracterización completa de los equilibrios conformacionales que devuelven información estructural, cinética y termodinámica sobre los procesos de intercambio. Para que un experimento de CPMG informe sobre un equilibrio conformacional, se deben aplicar dos condiciones: (i) los espines de RMN observados deben poseer diferentes cambios químicos en los estados sometidos a intercambio conformacional (microestados) y (ii) el intercambio tiene que ocurrir en una escala de tiempo que va de ~50 μs a ~10 ms. En estas condiciones, la tasa de relajación transversal observada ( 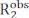 ) es la suma delR2 intrínseco (elR2 medido en ausencia de dinámica de μs-ms,
) es la suma delR2 intrínseco (elR2 medido en ausencia de dinámica de μs-ms, 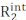 ) y la contribución de intercambio a la relajación transversal (Rex). La contribución de Rex aR2obs se puede apagar progresivamente reduciendo el espaciamiento entre los pulsos de 180° que constituyen el bloque CPMG de la secuencia de pulsos, y las curvas de RD resultantes se pueden modelar utilizando la teoría de Bloch-McConnell para obtener la diferencia de desplazamiento químico entre microestados, la población fraccionaria de cada microestado y las tasas de intercambio entre microestados(Figura 1),1,2,3.
) y la contribución de intercambio a la relajación transversal (Rex). La contribución de Rex aR2obs se puede apagar progresivamente reduciendo el espaciamiento entre los pulsos de 180° que constituyen el bloque CPMG de la secuencia de pulsos, y las curvas de RD resultantes se pueden modelar utilizando la teoría de Bloch-McConnell para obtener la diferencia de desplazamiento químico entre microestados, la población fraccionaria de cada microestado y las tasas de intercambio entre microestados(Figura 1),1,2,3.
Varias diversas secuencias del pulso y protocolos del análisis se han divulgado en la literatura para 15experimentos de N CPMG. Adjunto, el protocolo puesto en ejecución en el laboratorio se describe. En particular, se introducirán los pasos cruciales para la preparación de la muestra de RMN, la configuración y adquisición de los experimentos de RMN, y el procesamiento y análisis de los datos de RMN (Figura 2). Para facilitar la transferencia del protocolo a otros laboratorios, el programa de pulsos, los scripts de procesamiento y análisis y un conjunto de datos de ejemplo se proporcionan como archivos suplementarios y están disponibles para su descarga en (https://group.chem.iastate.edu/Venditti/downloads.html). La secuencia de pulsos proporcionada incorpora un ciclo de fase de cuatro pasos en el bloque CPMG para la supresión de artefactos dependientes de desplazamiento6 y está codificada para la adquisición de varios experimentos intercalados. Estos experimentos intercalados tienen un período de relajación idéntico pero diferentes números de pulsos de reenfoque para lograr diferentes camposcpmg 7. También es importante notar que el programa de impulsos descrito mide los 15N R2 de la componente TROSY de la señal de RMN8. En general, el protocolo se ha aplicado con éxito para la caracterización del intercambio conformacional en proteínas de tamaño mediano y grande4,5,9,10. Para sistemas más pequeños (<20 kDa), se recomienda el uso de una secuencia de pulsosheteronuclearbasada en coherencia cuántica única (HSQC)11, 12.
Protocolo
1. Preparación de la muestra de RMN
- Expresar y purificar una muestra de 2H,15N-labled de la proteína de interés.
NOTA: Mientras que una muestra de proteína 15con etiqueta N se puede utilizar para la adquisición del experimento CPMG RD, la perdeuteración (cuando sea posible) aumenta dramáticamente la calidad de los datos obtenidos. Los protocolos para la producción de proteínas perdeuteradas están disponibles en la literatura13. - Intercambio del almacenador intermediario la muestra purificada de la proteína en un almacenador intermediario desgastado del RMN.
- Transfiera la muestra de RMN al tubo de RMN.
NOTA: La concentración de la muestra de RMN debe optimizarse cuidadosamente para maximizar la relación señal-ruido y minimizar la aparición de interacciones proteína-proteína. En general, el rango de concentración típico para los experimentos de CPMG es de 0.1-1.0 mM.
2. Primera puesta a día del experimento de RMN
- Descargue y descomprima los archivos suplementarios.
- Copie los archivos bits.vv y trosy_15N_cpmg.vv (ubicados en la carpeta denominada pulseprogram) en el directorio del programa pulse (
- Abra el software de adquisición y copie un experimento HSQC previamenteejecutado de 1 H-15N en un nuevo experimento utilizando el comando edc.
- Usando el comando pulprog, cargue el programa de pulsos trosy_15N_cpmg.vv en el experimento recién creado.
- Configure el experimento CPMG siguiendo las instrucciones proporcionadas al final del archivo de programa de pulsos (trosy_15N_cpmg.vv).
3. Configuración rutinaria del experimento de RMN
- Introducir la muestra en el imán y realizar todos los pasos básicos para la adquisición de cualquier experimento de RMN: bloquear y caldo la muestra; coincide y sintoniza los canales 1H y 15N.
- Fije p1 y p7 a la duración de los pulsos duros de 90° de 1H y 15N, respectivamente.
NOTA: Para obtener resultados óptimos, es importante calibrar el pulso de 15N 90° con mucho cuidado. La calibración se logra generalmente usando una muestra de 100 milímetros de urea N-etiquetada 15en DMSO según lo descrito en el manual del espectrómetro. Además, es posible volver a comprobar la calibración directamente en la muestra de RMN de trabajo adquiriendo el primer incremento de unespectro HSQC de 1 H-15N en el que el pulso de 15N 90° del primer bloque INEPT se cambia a un pulso de 180°. Si la calibración es correcta, se debe obtener un nulo. - En la ventana de adquisición, establezca el centro y el ancho espectral para las dimensiones 1H y 15N.
NOTA: Centre el espectro de 1H en la frecuencia de la señal de agua para una supresión óptima del agua. - Establezca el retardo de relajación (d30) igual a 0.7T2,dondeT2 es el tiempo de relajación transversal esperado de 15N de su proteína.
Nota: El valor de d30 se puede optimizar empíricamente para obtener los mejores resultados. - Utilice el comando vclist para crear una lista de números enteros correspondientes al parámetro n en la Figura 1A y la Figura 1B. Asegúrese de que cada entrada de la lista corresponde a un campo CPMG diferente (νcpmg) según νcpmg = 4 x n / d30. Asegúrese de que el primer número de la lista es 0 (esto corresponde al experimento de referencia para el que se omite el bloque CPMG y d30 = 0 s) y no utilice números mayores que 1.000 x d30 / 4. Los números mayores que este umbral dan como resultado νcpmg > 1 kHz y podrían dañar la sonda.
- Fije l8 al número de entradas en el vclist, l3 al número de puntos verdaderos para la dimensión indirecta (generalmente un rango de 64-200 se fija para l3), y 1 td igual a l8 x l3 x 2.
NOTA: Realice los pasos 3.7-3.11 para optimizar la supresión de agua. - Establezca la ganancia del receptor (rg) en 1; abra el archivo de programa de pulso (edcpul), vaya a la línea 91, quite el punto y coma que precede a goto 999y guarde el archivo.
- Usando el comando gs ajuste los parámetros spdb0 (o spdw0) para minimizar la intensidad de la señal FID.
- Vuelva a introducir el punto y coma en la línea 91 del archivo de programa de pulsos y guarde el archivo.
- Repita los pasos 3.7-3.9 para optimizar spdb11 (línea 168 del archivo de programa de pulsos), spdb2 (línea 179 del archivo de programa de pulsos) y pldb2 (línea 184 del archivo de programa de pulsos).
- Ejecute el comando rga para optimizar la ganancia del receptor.
- Establezca el número de exploraciones (ns) en un múltiplo de 8.
- Ejecute el comando zg para iniciar el experimento.
4. Tratamiento y análisis de los datos de RMN
- Copie la carpeta denominada Proceso (Archivos suplementarios)en el directorio que contiene el archivo ser.
- Utilice los archivos sep_fid.com y ft2D.com para procesar los datos de RMN.
Nota : instrucción en cuanto a cómo editar las secuencias de comandos de procesamiento se proporciona dentro de los archivos de sep_fid.com y ft2D.com. - Abra los archivos ucsf contenidos en el directorio CPMG_Sparky_files en Sparky.
Nota : los archivos ucsf se crean mediante las secuencias de comandos de procesamiento. Hay un archivo ucsf para cada entrada en el vclist. El primer archivo ucsf (test_1.ucsf) contiene el experimento de referencia. Los archivos ucsf posteriores (test_2.ucsf, test_3.ucsf,... test_n.ucsf) se ordenan del valor más bajo al más grande de νcpmg. - Elija los picos cruzados de RMN en el espectro de RMN de referencia (test_1.ucsf).
- Seleccione todos los picos cruzados seleccionados utilizando el comando Sparky pa.
- Ejecute el comando Sparky rh. Este comando abre una ventana de diálogo. Seleccione las alturas de opción en la misma posición en cada espectro. Haga clic en la configuración y marque todos los espectros nmr. Haga clic en Actualizar y guarde el archivo de salida en el directorio de trabajo. El archivo de salida contiene la matriz de las intensidades de la señal sobre todos los espectros abiertos del RMN.
NOTA: En la literatura14se han descrito protocolos más precisos que utilizan el ajuste de forma de línea para recuperar intensidades de experimentos pseudo-3D intercalados. El software disponible gratuitamente para el ajuste de forma de línea está disponible en https://pint-nmr.github.io/PINT/ (PINT), https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA) y dentro de NMRPipe (módulo nLinLS) y SPARKY (módulo it). - Para cada campo CPMG, convierta las intensidades de la señal a velocidadesR2 utilizando la
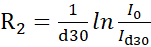 fórmula, donde I0 e Id30 son la intensidad de los espectros de RMN de referencia (vc = 0) y relajados (vc > 0), respectivamente.
fórmula, donde I0 e Id30 son la intensidad de los espectros de RMN de referencia (vc = 0) y relajados (vc > 0), respectivamente.
Nota: En los archivos suplementarios,se proporciona una plantilla del archivo de hoja de cálculo (R2_calc.xls) utilizado para convertir las intensidades de señal a velocidadesR2 y para visualizar los perfiles de RD. - Lea el nivel de ruido en el espectro de referencia utilizando el comando sparky st y propague el error en las velocidadesR2.
Nota : en los archivos suplementarios,se proporciona una plantilla del archivo de hoja de cálculo (R2_calc.xls) utilizado para propagar el error en las tasas de R2 medidas.
5. Ajuste de curvas RD
- Copie la carpeta denominada RD_fitting (Archivos suplementarios) en el directorio de trabajo.
- Para cada pico de RMN asignado, estime Rex calculando el
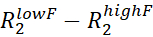 . y son las
. y son las 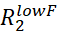
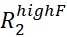 tasas R2 medidas en el νcpmg más bajo y más alto en el vclist.
tasas R2 medidas en el νcpmg más bajo y más alto en el vclist.
- Inspeccione visualmente las curvas RD con Rex mayor que el doble del error estimado y descarte todas las curvas RD que son demasiado ruidosas para ser modeladas con precisión.
NOTA: El ruido en Rex se puede propagar a partir de los errores en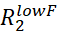 y
y 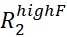 .
. - Prepare un fichero de entrada para el script de conexión utilizando todas las curvas RD con Rex mayor que el doble del error estimado.
NOTA: Las instrucciones detalladas sobre la preparación de los archivos de entrada se proporcionan dentro de los scripts de ajuste. Los archivos de entrada de ejemplo se proporcionan como archivos suplementarios. Comúnmente, los datos de RD se miden en dos campos estáticos diferentes y se ajustan simultáneamente. En este protocolo, se requieren dos archivos de entrada diferentes (Archivos suplementarios). - Ajuste los datos de ESCRITORIO REMOTO mediante los scripts proporcionados en Archivos suplementarios.
NOTA: Se proporcionan dos secuencias de comandos diferentes para realizar un ajuste basado en residuos o global de las curvas rd, respectivamente. Ambos scripts se ajustan a las curvas de RD utilizando un modelo de intercambio de dos sitios y la ecuación de Carver-Richards. Se proporcionan instrucciones más detalladas dentro de los guiones de conexión. Paquetes de software adicionales como Chemex (https://github.com/gbouvignies/ChemEx) y CATIA (https://www.ucl.ac.uk/hansen-lab/catia/) están disponibles para llevar a cabo el ajuste de datos utilizando las ecuaciones de Block-McConnell. - Probar la fiabilidad de los parámetros ajustados estimando la reducción de χ2 en función de pb y kex.
Nota: El χ reducido2 se proporciona en el archivo de salida. pb y kex pueden restringirse a valores específicos utilizando límites inferiores y superiores en los procedimientos de ajuste. En nuestros scripts, los límites inferior y superior para pb son lb(2) y ub(2), respectivamente. Los límites inferior y superior para kex son lb(3) y ub(3), respectivamente. - Estime el error en los parámetros ajustados. Esto se puede hacer estableciendo el valor de MC_fac en el script en 1 y repitiendo el ajuste varias veces (normalmente >20 repeticiones). El error en cada parámetro se estima como la desviación estándar de la distribución.
Nota : establecer MC_fac en 1 genera un conjunto de datos sintético en el que se agrega un error distribuido gaussiano (calculado en función del error experimental) a los datos experimentales.
Resultados
El protocolo aquí descrito da como resultado la adquisición de perfiles de DR para cada pico en elespectro TROSY de 1H-15N (Figura 3A). A partir de los perfiles de DR adquiridos, es posible estimar la contribución de intercambio a la relajación transversal de 15N de cada grupo amida de la columna vertebral (Figura 3A,3B). Trazando el Rex sobre la estructura 3D de la proteína bajo investigación, e...
Discusión
Este manuscrito describe el protocolo implementado en el laboratorio para la adquisición y análisis de 15N RD de datos sobre proteínas. En particular, se cubren los pasos cruciales para la preparación de la muestra de RMN, la medición de los datos de RMN y el análisis de los perfiles de DR. A continuación se discuten algunos aspectos importantes con respecto a la adquisición y el análisis de experimentos de RD. Sin embargo, para una descripción más profunda del experimento y el análisis de los dato...
Divulgaciones
Todos los autores han leído y aprobado el manuscrito. Declaramos que no hay conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por fondos de NIGMS R35GM133488 y del Roy J. Carver Charitable Trust a V.V.
Materiales
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
Referencias
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados