Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
15 N CPMG Relaxation Dispersion zur Untersuchung der Proteinkonformationsdynamik auf der μs-ms Zeitskala
In diesem Artikel
Zusammenfassung
Hier wird eine detaillierte Beschreibung des im Labor implementierten Protokolls zur Erfassung und Analyse von 15 NRelaxationsdispersionsprofilen mittels Lösungs-NMR-Spektroskopie bereitgestellt.
Zusammenfassung
Die Proteinkonformationsdynamik spielt eine grundlegende Rolle bei der Regulierung der enzymatischen Katalyse, Ligandenbindung, Allostersion und Signalisierung, die wichtige biologische Prozesse sind. Zu verstehen, wie das Gleichgewicht zwischen Struktur und Dynamik die biologische Funktion steuert, ist eine neue Grenze in der modernen Strukturbiologie und hat mehrere technische und methodische Entwicklungen ausgelöst. Unter diesen liefern CPMG-Relaxationsdispersionslösungs-NMR-Methoden einzigartige, atomar aufgelöste Informationen über die Struktur, Kinetik und Thermodynamik von Proteinkonformationsgleichgewichten, die auf der μs-ms-Zeitskala auftreten. Hier präsentiert die Studie detaillierte Protokolle zur Erfassung und Analyse eines 15 NRelaxationsdispersionsexperiments. Als Beispiel wird die Pipeline zur Analyse der μs-ms-Dynamik in der C-terminalen Domäne des Bakteriums Enzym I gezeigt.
Einleitung
Carr-Purcell Meiboom-Gill (CPMG) Relaxation Dispersion (RD) Experimente werden auf einer Routinebasis verwendet, um Konformationsgleichgewichte auf der μs-ms-Zeitskala durch Lösungs-NMR-Spektroskopie1,2,3,4,5zu charakterisieren. Im Vergleich zu anderen Methoden zur Untersuchung der Konformationsdynamik sind CPMG-Techniken auf modernen NMR-Spektrometern relativ einfach zu implementieren, erfordern keine speziellen Probenvorbereitungsschritte (z. B. Kristallisation, Probengefrieren oder -ausrichtung und/ oder kovalente Konjugation mit einem fluoreszierenden oder paramagnetischen Tag) und bieten eine umfassende Charakterisierung von Konformationsgleichgewichten, die strukturelle, kinetische und thermodynamische Informationen über Austauschprozesse liefern. Damit ein CPMG-Experiment über ein Konformationsgleichgewicht berichten kann, müssen zwei Bedingungen gelten: (i) Die beobachteten NMR-Spins müssen unterschiedliche chemische Verschiebungen in den Zuständen aufweisen, die einem Konformationsaustausch unterzogen werden (Mikrozustände) und (ii) der Austausch muss auf einer Zeitskala von ~ 50 μs bis ~ 10 ms stattfinden. Unter diesen Bedingungen ist die beobachtete Querrelaxationsrate ( 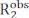 ) die Summe des intrinsischenR2 (das in Abwesenheit von μs-ms-Dynamik gemesseneR2)
) die Summe des intrinsischenR2 (das in Abwesenheit von μs-ms-Dynamik gemesseneR2) 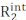 und des Austauschbeitrags zur Querrelaxation (Rex). Der Rex-Beitrag zuR2-Obs kann schrittweise abgeschreckt werden, indem der Abstand zwischen den 180°-Pulsen, die den CPMG-Block der Pulssequenz ausmachen, verringert wird, und die resultierenden RD-Kurven können mit der Bloch-McConnell-Theorie modelliert werden, um die chemische Verschiebungsdifferenz zwischen Mikrozuständen, die fraktionierte Population jedes Mikrostaates und die Austauschraten zwischen Mikrozuständen zu erhalten (Abbildung 1)1,2,3.
und des Austauschbeitrags zur Querrelaxation (Rex). Der Rex-Beitrag zuR2-Obs kann schrittweise abgeschreckt werden, indem der Abstand zwischen den 180°-Pulsen, die den CPMG-Block der Pulssequenz ausmachen, verringert wird, und die resultierenden RD-Kurven können mit der Bloch-McConnell-Theorie modelliert werden, um die chemische Verschiebungsdifferenz zwischen Mikrozuständen, die fraktionierte Population jedes Mikrostaates und die Austauschraten zwischen Mikrozuständen zu erhalten (Abbildung 1)1,2,3.
Für 15N-CPMG-Experimente wurden in der Literatur mehrere verschiedene Pulssequenzen und Analyseprotokolle berichtet. Hierin wird das im Labor implementierte Protokoll beschrieben. Insbesondere werden die entscheidenden Schritte zur Vorbereitung der NMR-Probe, zum Aufbau und zur Erfassung der NMR-Experimente sowie zur Verarbeitung und Analyse der NMR-Daten vorgestellt (Abbildung 2). Um die Übertragung des Protokolls auf andere Labore zu erleichtern, werden das Pulsprogramm, Verarbeitungs- und Analyseskripte sowie ein Beispieldatensatz als Supplemental Files zur Verfügung gestellt und stehen unter (https://group.chem.iastate.edu/Venditti/downloads.html) zum Download zur Verfügung. Die bereitgestellte Pulssequenz beinhaltet einen vierstufigen Phasenzyklus im CPMG-Block zur Unterdrückung von offsetabhängigen Artefakten6 und ist für die Erfassung mehrerer verschachtelter Experimente codiert. Diese verschachtelten Experimente haben eine identische Relaxationsperiode, aber eine unterschiedliche Anzahl von Refokussierpulsen, um verschiedene CPMG-Felder zu erreichen7. Wichtig zu beachten ist auch, dass das beschriebene Pulsprogramm die 15N R2 der TROSY-Komponente des NMR-Signals8 misst. Insgesamt wurde das Protokoll erfolgreich zur Charakterisierung des Konformationsaustausches in mittelgroßen und großformatigen Proteinen4,5,9,10angewendet. Für kleinere Systeme (<20 kDa) empfiehlt sich die Verwendung einer auf Heteronukleärer Single Quantum Coherence (HSQC) basierenden Pulssequenz11,12.
Protokoll
1. Vorbereitung der NMR-Probe
- Exprimieren und reinigen Sie eine 2H,15N-labierte Probe des interessierenden Proteins.
HINWEIS: Während eine 15N-markierte Proteinprobe für die Erfassung des CPMG RD-Experiments verwendet werden kann, erhöht die Perdeuteration (wo möglich) die Qualität der erhaltenen Daten dramatisch. Protokolle zur Herstellung von perdeuterierten Proteinen sind in der Literatur13verfügbar. - Puffer tauschen die gereinigte Proteinprobe in einen entgasten NMR-Puffer aus.
- Die NMR-Probe wird in das NMR-Röhrchen überführen.
HINWEIS: Die Konzentration der NMR-Probe muss sorgfältig optimiert werden, um das Signal-Rausch-Verhältnis zu maximieren und das Auftreten von Protein-Protein-Interaktionen zu minimieren. Im Allgemeinen liegt der typische Konzentrationsbereich für CPMG-Experimente bei 0,1-1,0 mM.
2. Erstmaliger Aufbau des NMR-Experiments
- Laden Sie die Ergänzenden Dateien herunter und entpacken Sie sie.
- Kopieren Sie die Dateien bits.vv und trosy_15N_cpmg.vv (im Ordner pulseprogram) in das Pulse-Programmverzeichnis (
- Öffnen Sie die Erfassungssoftware und kopieren Sie ein zuvor ausgeführtes 1H-15N HSQC-Experiment mit dem Befehl edcin ein neues Experiment.
- Laden Sie mit dem Befehl pulprogdas Impulsprogramm trosy_15N_cpmg.vv in das neu erstellte Experiment.
- Richten Sie das CPMG-Experiment mit den Anweisungen am Ende der Pulse-Programmdatei (trosy_15N_cpmg.vv) ein.
3. Routinemäßiger Aufbau des NMR-Experiments
- Führen Sie die Probe in den Magneten ein und führen Sie alle grundlegenden Schritte für die Erfassung eines NMR-Experiments durch: Sperren und Verschließen der Probe; Passen Sie die Kanäle 1H und 15N an und stimmen Sie sie ab.
- Stellen Sie p1 und p7 auf die Dauer der 1H bzw. 15N harten 90° Impulse ein.
HINWEIS: Für optimale Ergebnisse ist es wichtig, den 15 N90° Puls mit großer Sorgfalt zu kalibrieren. Die Kalibrierung erfolgt in der Regel mit einer 100 mM-Probe von 15N-markiertem Harnstoff in DMSO, wie im Spektrometerhandbuch beschrieben. Darüber hinaus ist es möglich, die Kalibrierung direkt an der arbeitenden NMR-Probe zu überprüfen, indem das erste Inkrement eines 1 H-15NHSQC-Spektrums entnommen wird, in dem der 15N 90°-Impuls des ersten INEPT-Blocks auf einen 180°-Impuls umgeschaltet wird. Wenn die Kalibrierung korrekt ist, sollte ein Nullwert erreicht werden. - Legen Sie im Erfassungsfenster die Mittlere und Spektralbreite für die Abmessungen 1H und 15N fest.
HINWEIS: Zentrieren Sie das 1H-Spektrum auf die Frequenz des Wassersignals für eine optimale Wasserunterdrückung. - Stellen Sie die Entspannungsverzögerung (d30) auf 0,7T2ein, wobeiT2 die erwartete 15 NQuerrelaxationszeit Ihres Proteins ist.
HINWEIS: Der Wert von d30 kann empirisch optimiert werden, um die besten Ergebnisse zu erzielen. - Verwenden Sie den Befehl vclist, um eine Liste ganzzahliger Zahlen zu erstellen, die dem Parameter n in Abbildung 1A und Abbildung 1Bentsprechen. Stellen Sie sicher, dass jeder Eintrag in der Liste einem anderen CPMG-Feld (νcpmg) gemäß νcpmg = 4 x n / d30 entspricht. Stellen Sie sicher, dass die erste Zahl in der Liste 0 ist (dies entspricht dem Referenzexperiment, für das der CPMG-Block übersprungen wird und d30 = 0 s) und keine Zahlen größer als 1.000 x d30 / 4 zu verwenden. Zahlen, die diesen Schwellenwert überschreiten, führen zu νcpmg > 1 kHz und können die Sonde beschädigen.
- Setzen Sie l8 auf die Anzahl der Einträge in der vclist, l3 auf die Anzahl der reellen Punkte für die indirekte Dimension (normalerweise wird für l3 ein Bereich von 64-200 festgelegt) und 1 td gleich l8 x l3 x 2.
HINWEIS: Führen Sie die Schritte 3.7-3.11 aus, um die Wasserunterdrückung zu optimieren. - Stellen Sie die Empfängerverstärkung (rg) auf 1 ein; Öffnen Sie die Pulse-Programmdatei (edcpul), wechseln Sie zu Zeile 91, entfernen Sie das Semikolon vor Goto 999,und speichern Sie die Datei.
- Mit dem Befehl gs passen Sie die Parameter spdb0 (oder spdw0) an, um die Intensität des FID-Signals zu minimieren.
- Setzen Sie das Semikolon in Zeile 91 der Impulsprogrammdatei wieder ein und speichern Sie die Datei.
- Wiederholen Sie die Schritte 3.7-3.9, um spdb11 (Zeile 168 der Pulse-Programmdatei), spdb2 (Zeile 179 der Pulse-Programmdatei) und pldb2 (Zeile 184 der Pulse-Programmdatei) zu optimieren.
- Führen Sie den Befehl rga aus, um die Empfängerverstärkung zu optimieren.
- Legen Sie die Anzahl der Scans (ns) auf ein Vielfaches von 8 fest.
- Führen Sie den Befehl zg aus, um das Experiment zu starten.
4. Verarbeitung und Analyse der NMR-Daten
- Kopieren Sie den Ordner Process (Supplemental Files) in das Verzeichnis, das die ser-Datei enthält.
- Verwenden Sie die Dateien sep_fid.com und ft2D.com, um die NMR-Daten zu verarbeiten.
HINWEIS: Anweisungen zum Bearbeiten der Verarbeitungsskripte finden Sie in den sep_fid.com- und ft2D.com Dateien. - Öffnen Sie die ucsf-Dateien, die im Verzeichnis CPMG_Sparky_files in Sparky enthalten sind.
HINWEIS: Die ucsf-Dateien werden von den Verarbeitungsskripten erstellt. Für jeden Eintrag in der vclist gibt es eine ucsf-Datei. Die erste ucsf-Datei (test_1.ucsf) enthält das Referenzexperiment. Die nachfolgenden ucsf-Dateien (test_2.ucsf, test_3.ucsf,... test_n.ucsf) sind vom niedrigsten bis zum größten Wert von νcpmggeordnet. - Wählen Sie die NMR-Kreuzpeaks im Referenz-NMR-Spektrum (test_1.ucsf).
- Wählen Sie alle ausgewählten Kreuzspitzen mit dem Sparky-Befehl paaus.
- Führen Sie den Sparky-Befehl rhaus. Mit diesem Befehl wird ein Dialogfenster geöffnet. Wählen Sie die Optionshöhen an der gleichen Position in jedem Spektrumaus. Klicken Sie auf Setup und überprüfen Sie alle NMR-Spektren. Klicken Sie auf Aktualisieren und speichern Sie die Ausgabedatei im Arbeitsverzeichnis. Die Ausgabedatei enthält die Matrix der Signaldicken über alle geöffneten NMR-Spektren.
HINWEIS: Genauere Protokolle, die linienförmige Anpassung zur Wiederherstellung von Intensitäten aus verschachtelten Pseudo-3D-Experimenten verwenden, wurden in der Literatur14beschrieben. Frei verfügbare Software für den Lineshape Fitting ist an https://pint-nmr.github.io/PINT/ (PINT), https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA) sowie in NMRPipe (nLinLS-Modul) und SPARKY (it-Modul) verfügbar. - Konvertieren Sie für jedes CPMG-Feld die Signalintensitäten inR2-Raten mit der Formel
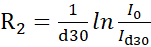 , wobei I0 und Id30 die Intensität der Referenz- (vc = 0) bzw. der entspannten (vc > 0) NMR-Spektren sind.
, wobei I0 und Id30 die Intensität der Referenz- (vc = 0) bzw. der entspannten (vc > 0) NMR-Spektren sind.
HINWEIS: In den Zusatzdateienwird eine Vorlage der Tabellenkalkulationsdatei (R2_calc.xls) bereitgestellt, die verwendet wird, um die Signalraten inR2-Raten umzuwandeln und die RD-Profile zu visualisieren. - Lesen Sie den Rauschpegel im Referenzspektrum mit dem Sparky-Befehl st und verbreiten Sie den Fehler auf den R2-Raten.
HINWEIS: In den Zusatzdateienwird eine Vorlage der Tabellenkalkulationsdatei (R2_calc.xls) bereitgestellt, die verwendet wird, um den Fehler auf den gemessenen R2-Raten zu verbreiten.
5. RD-Kurven anpassen
- Kopieren Sie den Ordner RD_fitting (Supplemental Files) in das Arbeitsverzeichnis.
- Schätzen Sie für jeden zugewiesenen NMR-Peak Rex durch Berechnung der
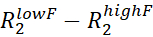 . und sind die
. und sind die 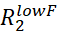
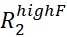 R2-Raten, die am niedrigsten und höchsten νcpmg in der vclist gemessenwerden.
R2-Raten, die am niedrigsten und höchsten νcpmg in der vclist gemessenwerden.
- Untersuchen Sie die RD-Kurven visuell mit Rex, die größer als das Zweifache des geschätzten Fehlers sind, und verwerfen Sie alle RD-Kurven, die zu laut sind, um genau modelliert zu werden.
HINWEIS: Rauschen auf Rex kann von den Fehlern auf und übertragen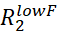
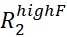 werden.
werden. - Bereiten Sie eine Eingabedatei für das Formstückskript vor, indem Sie alle RD-Kurven mit Rex verwenden, die größer als das Zweifache des geschätzten Fehlers sind.
HINWEIS: Detaillierte Anweisungen zur Vorbereitung der Eingabedateien finden Sie in den Anpassungsskripten. Beispieleingabedateien werden als Ergänzende Dateienbereitgestellt. Üblicherweise werden RD-Daten an zwei verschiedenen statischen Feldern gemessen und gleichzeitig angepasst. In diesem Protokoll sind zwei verschiedene Eingabedateien erforderlich (Supplemental Files). - Passen Sie die RD-Daten mithilfe der skripts unter Ergänzende Dateien bereitgestellten Skripts an.
HINWEIS: Es werden zwei verschiedene Skripts bereitgestellt, um eine rückstandsbasierte bzw. globale Anpassung der RD-Kurven durchzuführen. Beide Skripte passen die RD-Kurven unter Verwendung eines Zwei-Standort-Austauschmodells und der Carver-Richards-Gleichung an. Detailliertere Anweisungen finden Sie in den Anpassungsskripten. Zusätzliche Softwarepakete wie Chemex (https://github.com/gbouvignies/ChemEx) und CATIA (https://www.ucl.ac.uk/hansen-lab/catia/ ) stehen zur Verfügung, um die Datenanpassung unter Verwendung der Block-McConnell-Gleichungen durchzuführen. - Prüfung der Zuverlässigkeit der angepassten Parameter zur Schätzung des reduzierten χ2 als Funktion von pb und kex.
HINWEIS: Das reduzierte χ2 ist in der Ausgabedatei enthalten. pb und kex können mit unteren und oberen Grenzen in den Anpassungsverfahren auf bestimmte Werte beschränkt werden. In unseren Skripten sind die untere und obere Grenze für pb lb(2) bzw. ub(2). Die untere und obere Grenze für kex sind lb(3) bzw. ub(3). - Schätzen Sie den Fehler bei den angepassten Parametern. Dies kann geschehen, indem Sie den Wert von MC_fac im Skript auf 1 setzen und das Formstück mehrmals wiederholen (in der Regel >20 Wiederholungen). Der Fehler bei jedem Parameter wird als Standardabweichung der Verteilung geschätzt.
HINWEIS: Wenn Sie MC_fac auf 1 setzen, wird ein synthetischer Datensatz generiert, in dem den experimentellen Daten ein Gaußscher verteilter Fehler (berechnet auf der Grundlage des experimentellen Fehlers) hinzugefügt wird.
Ergebnisse
Das hier beschriebene Protokoll führt zur Erfassung von RD-Profilen für jeden Peak im 1H-15N TROSY-Spektrum (Abbildung 3A). Aus den gewonnenen RD-Profilen ist es möglich, den Austauschbeitrag zur 15N Querrelaxation jeder Backbone-Amid-Gruppe abzuschätzen (Abbildung 3A,3B). Durch die Darstellung des Rex auf der 3D-Struktur des untersuchten Proteins ist es möglich, die Strukturregionen, die sich ein...
Diskussion
Dieses Manuskript beschreibt das im Labor implementierte Protokoll zur Erfassung und Analyse von 15N RD-Daten über Proteine. Insbesondere werden die entscheidenden Schritte zur Vorbereitung der NMR-Probe, zur Messung der NMR-Daten und zur Analyse der RD-Profile behandelt. Im Folgenden werden einige wichtige Aspekte bezüglich der Erfassung und Analyse von RD-Experimenten diskutiert. Für eine eingehendere Beschreibung des Experiments und die Datenanalyse wird jedoch ein sorgfältiges Studium der Originalliter...
Offenlegungen
Alle Autoren haben das Manuskript gelesen und genehmigt. Wir erklären keine Interessenkonflikte.
Danksagungen
Diese Arbeit wurde durch Mittel von NIGMS R35GM133488 und vom Roy J. Carver Charitable Trust an V.V. unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
Referenzen
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten