A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
15 (15) N פיזור הרפיה CPMG לחקירת דינמיקה קונפורמטיבית חלבון על ציר הזמן μs-ms
In This Article
Summary
כאן, תיאור מפורט של הפרוטוקול מיושם במעבדה לרכישה וניתוח של 15N פרופילי פיזור הרפיה על ידי פתרון ספקטרוסקופיה NMR מסופק.
Abstract
דינמיקה קונפורמציה של חלבונים ממלאת תפקידי יסוד בוויסות קטליזה אנזימטית, כריכת ליגנד, אלוסטריה ואיתות, שהם תהליכים ביולוגיים חשובים. הבנת האופן שבו האיזון בין מבנה לדינמיקה שולט בתפקוד הביולוגי היא גבול חדש בביולוגיה המבנית המודרנית והציתה מספר התפתחויות טכניות ומתודולוגיות. בין אלה, פתרון פיזור הרפיה CPMG שיטות NMR לספק ייחודי, מידע ברזולוציה אטומית על המבנה, קינטיקה, תרמודינמיקה של שיווי משקל קונפורמי חלבון המתרחשים על ציר הזמן μs-ms. כאן, המחקר מציג פרוטוקולים מפורטים לרכישה וניתוח של ניסוי פיזור הרפיה 15N. כדוגמה, הצינור לניתוח הדינמיקה μs-ms בתחום C-מסוף של אנזים חיידקים אני מוצג.
Introduction
Carr-פרסל Meiboom-Gill (CPMG) הרפיה פיזור (RD) ניסויים משמשים על בסיס שגרתי לאפיון שיווי משקל קונפורמי המתרחשים על ציר הזמן μs-ms על ידי פתרון ספקטרוסקופיה NMR1,2,3,4,5. בהשוואה לשיטות אחרות לחקירת דינמיקה קונפורמטיבית, טכניקות CPMG קלות יחסית ליישום בספקטרומטרים מודרניים של NMR, אינן דורשות שלבי הכנה מיוחדים לדוגמה (כלומר, התגבשות, הקפאה או יישור מדגם, ו/או הטיה קוולנטית עם תג פלואורסצנטי או פרמגנטי), ומספקות אפיון מקיף של שיווי משקל קונפורמי המחזיר מידע מבני, קינטי ותרמודינמי על תהליכי החלפה. על מנת שניסוי CPMG ידווח על שיווי משקל קונפורמי, יש להחיל שני תנאים: (i) ספינים NMR שנצפו חייבים להחזיק משמרות כימיות שונות במדינות העוברות חילופי קונפורמציה (microstates) ו- (ii) ההחלפה צריכה להתרחש בסולם זמן הנע בין ~ 50 מיקרוס ל ~ 10 אלפיות השנייה. בתנאים אלה, שיעור ההרפיה הרוחבי שנצפה ( 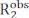 ) הוא סכום R2 המהותי (R2 נמדד בהעדר דינמיקה μs-ms),
) הוא סכום R2 המהותי (R2 נמדד בהעדר דינמיקה μs-ms), 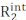 ואת תרומת ההחלפה הרפיה רוחבית (Rex). ניתן להרוותבהדרגה את תרומת Rex לאובס R2על-ידי הפחתת הריווח בין פולסים של 180° המהווים את בלוק ה- CPMG של רצף הדופק, ואת עקומות המו"פ הנובעות מכך ניתן לעצב באמצעות תיאוריית בלוך-מקונל כדי להשיג את ההבדל הכימי בין מיקרוסטאטים, את אוכלוסיית השברים של כל מיקרוסטאט ואת שיעורי ההחלפה בין מיקרוסטטים (איור1).
ואת תרומת ההחלפה הרפיה רוחבית (Rex). ניתן להרוותבהדרגה את תרומת Rex לאובס R2על-ידי הפחתת הריווח בין פולסים של 180° המהווים את בלוק ה- CPMG של רצף הדופק, ואת עקומות המו"פ הנובעות מכך ניתן לעצב באמצעות תיאוריית בלוך-מקונל כדי להשיג את ההבדל הכימי בין מיקרוסטאטים, את אוכלוסיית השברים של כל מיקרוסטאט ואת שיעורי ההחלפה בין מיקרוסטטים (איור1).
מספר רצפי דופק שונים ופרוטוקולי ניתוח דווחו בספרות עבור 15ניסויי N CPMG. להלן מתואר הפרוטוקול המיושם במעבדה. בפרט, יוכנסו השלבים המכריעים להכנת מדגם NMR, הקמה ורכישה של ניסויי NMR, ועיבוד וניתוח של נתוני NMR (איור 2). כדי להקל על העברת הפרוטוקול למעבדות אחרות, תוכנית הדופק, סקריפטים עיבוד וניתוח, ו ערכת נתונים אחת לדוגמה מסופקים כקבצים משלימים זמינים להורדה ב (https://group.chem.iastate.edu/Venditti/downloads.html). רצף הדופק שסופק משלב מחזור פאזה בן ארבעה שלבים בבלוק CPMG לדיכוי חפצים תלויי אופסט6 והוא מקודד לרכישת מספר ניסויים שזורים. ניסויים אלה interleaved יש תקופת הרפיה זהה אבל מספר שונה של פולסים מיקוד מחדש על מנת להשיג שדות CPMG שונים7. חשוב גם לשים לב כי תוכנית הדופק המתוארת מודדת את 15N R2 של רכיב TROSY של אות NMR8. בסך הכל, הפרוטוקול הוחל בהצלחה על אפיון חילופי קונפורמציה בחלבונים בינוניים וגדולים4,5,9,10. עבור מערכות קטנות יותר (<20 kDa), מומלץ להשתמש בקוהרנטיות קוונטית אחת הטרונוקלארית (HSQC) המבוססת עלפולסים 11,12.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. הכנת מדגם NMR
- לבטא ולטהר 2H,15N-labled מדגם של החלבון של עניין.
הערה: בעוד שמדגם חלבון בעל תווית N 15יכול לשמש לרכישת ניסוי CPMG RD, התייבשות (במידת האפשר) מגדילה באופן דרמטי את איכות הנתונים המתקבלים. פרוטוקולים לייצור חלבונים מיובשים זמינים בספרות13. - חוצץ להחליף את דגימת החלבון המטוהר לתוך מאגר NMR degassed.
- העבר את דגימת NMR לתוך צינור NMR.
הערה: יש למטב בזהירות את הריכוז של דגימת NMR על מנת למקסם את יחס האות לרעש ולמזער את התרחשותן של אינטראקציות חלבון-חלבון. באופן כללי, טווח הריכוז האופייני לניסויי CPMG הוא 0.1-1.0 מ"מ.
2. הגדרה ראשונה של ניסוי NMR
- הורד ובטל את ההסתעפות של הקבצים המשלימים.
- העתק את הקבצים bits.vv ו- trosy_15N_cpmg.vv (הממוקמים בתיקיה בשם pulseprogram) לספריית תוכניות הדופק (<פת>/exp/stan/nmr/רשימות/עמ'/משתמש).
הערה: ודא שהנתיב ל- bits.vv המפורט בשורה הראשונה של הקובץ trosy_15N_cpmg.vv נכון. - פתח תוכנת רכישה והעתק ניסוי H-15N HSQC שנערך בעבר לניסוי חדש באמצעות הפקודה edc.
- באמצעות pulprogהפקודה , לטעון את תוכנית הדופק trosy_15N_cpmg.vv לתוך הניסוי החדש שנוצר.
- הגדר את ניסוי CPMG באמצעות ההוראות שסופקו בסוף קובץ תוכנית הדופק (trosy_15N_cpmg.vv).
3. הקמה שגרתית של ניסוי NMR
- הציגו את המדגם במגנט ובצעו את כל השלבים הבסיסיים לרכישת כל ניסוי NMR: נעלו ושימו את הדגימה; להתאים ולכוונן את 1H ו 15N ערוצים.
- הגדר p1 ו p7 למשך של 1H ו 15N קשה 90° פולסים, בהתאמה.
הערה: לקבלת תוצאות מיטביות, חשוב לכייל את הדופק של 15N 90° בזהירות רבה. כיול מושגת בדרך כלל באמצעות מדגם 100 מ"מ של 15אוריאה N-labeled ב DMSO כמתואר במדריך ספקטרומטר. בנוסף, ניתן לבדוק שוב את הכיול ישירות על דגימת NMR העובדת על ידי רכישת ההפרש הקבוע הראשון של ספקטרום H-15N HSQC שבו הדופק של 15N 90° של בלוק INEPT הראשון עובר לפולס של 180°. אם הכיול נכון, יש להשיג Null. - בחלון הרכישה, הגדר את הרוחב המרכזי והספקטרלי עבור הממדים 1H ו- 15N.
הערה: מרכז את הספקטרום 1H בתדר של אות המים לדיכוי מים אופטימלי. - הגדר את השהיית הרפיה (d30) שווה 0.7 T2, שבו T2 הוא צפוי 15N זמן הרפיה רוחבי של החלבון שלך.
הערה: ניתן למטב את הערך של d30 באופן אמפירי כדי להשיג את התוצאות הטובות ביותר. - השתמש ברשימת vclist של הפקודות כדי ליצור רשימה של מספרים שלמים התואמים לפרמטר n באיור 1A ובאיור 1B. ודא שכל ערך ברשימה מתאים לשדה CPMG אחר (νcpmg)בהתאם ל- νcpmg = 4 x n / d30. ודא שהמספר הראשון ברשימה הוא 0 (פעולה זו תואמת לניסוי ההפניה שעבורו מדלגים על בלוק CPMG ו- d30 = 0 s) ולא להשתמש במספרים הגדולים מ- 1,000 x d30 / 4. מספרים הגדולים מסף זה גורמים ל- νcpmg > 1 kHz ועלולים לגרום נזק לבדיקה.
- הגדר את l8 למספר הערכים ברשימת ה- vclist, l3 למספר הנקודות הממשיות עבור הממד העקיף (בדרך כלל טווח של 64-200 מוגדר עבור l3) ו- 1 td שווה ל- l8 x l3 x 2.
הערה: בצע את השלבים 3.7-3.11 כדי למטב את דיכוי המים. - הגדר את רווח המקלט (rg) ל- 1; פתח את קובץ תוכנית הדופק (edcpul), עבור לקו 91, הסר את הנקודה-פסיק שלפני goto 999ושמור את הקובץ.
- באמצעות הפקודה gs להתאים את הפרמטרים spdb0 (או spdw0) על מנת למזער את עוצמת אות FID.
- הצג מחדש את הנקודה-פסיק בשורה 91 של קובץ תוכנית הדופק ושמור את הקובץ.
- חזור על השלבים 3.7-3.9 כדי לייעל spdb11 (שורה 168 של קובץ תוכנית הדופק), spdb2 (שורה 179 של קובץ תוכנית הדופק), ו pldb2 (שורה 184 של קובץ תוכנית הדופק).
- הפעל את הפקודה rga כדי למטב את רווח המקלט.
- הגדר את מספר הסריקות (ns)לכפולה של 8.
- הפעל את הפקודה zg כדי להתחיל בניסוי.
4. עיבוד וניתוח של נתוני NMR
- העתק את התיקיה בשם Process (קבצים משלימים) לספריה המכילה את קובץ ser.
- השתמש בקבצים sep_fid.com ft2D.com כדי לעבד את נתוני NMR.
הערה: הוראה כיצד לערוך את קבצי ה- Script לעיבוד מסופקת בתוך קבצי sep_fid.com והקבצים ft2D.com. - פתח את קבצי ucsf הכלולים בספריה CPMG_Sparky_files בספרקי.
הערה: קבצי ucsf נוצרים על-ידי קבצי ה- Script של העיבוד. יש קובץ ucsf אחד עבור כל ערך ברשימת ה- vclist. קובץ ה- ucsf הראשון (test_1.ucsf) מכיל את ניסוי הייחוס. קבצי ucsf הבאים (test_2.ucsf, test_3.ucsf,... test_n.ucsf) מסודרים מהנמוך ביותר לערך הגדול ביותר של νcpmg. - בחר את הפסגות הצולבות של NMR בספקטרום NMR הייחוס (test_1.ucsf).
- בחר את כל הפסגות הצולבות שנבחרו באמצעות הרשותלפקודה Sparky .
- הפעל את הפקודהספרקי rh . פקודה זו פותחת חלון דו-שיח. בחר את גובה האפשרויות באותו מיקום בכל ספקטרום. לחץ על ההתקנה ובדוק את כל ספקטרום NMR. לחץ על עדכן ושמור את קובץ הפלט בספריית העבודה. קובץ הפלט מכיל את המטריצה של עוצמות האות מעל כל ספקטרום NMR שנפתח.
הערה: פרוטוקולים מדויקים יותר המשתמשים התאמת lineshape לשחזור עוצמות מניסויים פסאודו-3D שזורים תוארו בספרות14. תוכנה זמינה בחופשיות להתאמת lineshape זמינה https://pint-nmr.github.io/PINT/ (PINT), https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA), ובתוך NMRPipe (מודול nLinLS) ו SPARKY (מודול זה). - עבור כל שדה CPMG, המר את עוצמת האותות לקצבי R2 באמצעות הנוסחה
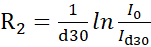 , כאשר I0 ו- Id30 הם עוצמת ההפניה (vc = 0) וספקטרום NMR רגוע (vc > 0), בהתאמה.
, כאשר I0 ו- Id30 הם עוצמת ההפניה (vc = 0) וספקטרום NMR רגוע (vc > 0), בהתאמה.
הערה: בקבצים המשלימים, מסופקת תבנית של קובץ הגיליון האלקטרוני (R2_calc.xls) המשמש להמרת עוצמת האותות לתעריפי R2 ולהמחיש את פרופילי המו"פ. - קרא את רמת הרעש בספקטרום ההפניות באמצעות הפקודה Sparky st והפץ את השגיאה בשיעורי R2.
הערה: בקבצים המשלימים, מסופקת תבנית של קובץ הגיליון האלקטרוני (R2_calc.xls) המשמש להפצת השגיאה בשיעורי R2 הנמדדים.
5. התאמת עקומות RD
- העתק את התיקיה בשם RD_fitting (קבצים משלימים) לספריית העבודה.
- עבור כל שיא NMR שהוקצה, להעריך Rex על ידי חישוב
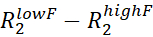 .
. 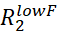
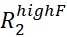
- בדוק באופן חזותי את עקומות המו"פ עם Rex גדול מפי שניים מהשגיאה המשוערת והשליך את כל עקומות המו"פ הרועשים מכדי שניתן יהיה לדגמן במדויק.
הערה: רעש על Rex ניתן להפיץ מן השגיאות על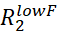 ו
ו 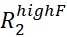 .
. - הכן קובץ קלט עבור קובץ ה- Script המתאים באמצעות כל עקומות המו"פ עם Rex הגדול מפי שניים מהשגיאה המשוערת.
הערה: הוראה מפורטת על הכנת קבצי הקלט מסופקים בתוך הסקריפטים המתאימים. קבצי קלט לדוגמה מסופקים כקבצים משלימים. בדרך כלל, נתוני המו"פ נמדדים בשני שדות סטטיים שונים ומותאמים בו-זמנית. בפרוטוקול זה נדרשים שני קבצי קלט שונים (קבצים משלימים). - התאם את נתוני ה- RD באמצעות קבצי ה- Script הכלולים בקבצים משלימים.
הערה: שני סקריפטים שונים מסופקים לביצוע התאמה מבוססת שאריות או התאמה גלובלית של עקומות המו"פ, בהתאמה. שני התסריטים מתאימים לעקומות ה-RD באמצעות מודל חילופי שני אתרים ומשוואת קארבר-ריצ'רדס. הוראות מפורטות יותר ניתנות בתוך הסקריפטים המתאימים. חבילות תוכנה נוספות כגון Chemex (https://github.com/gbouvignies/ChemEx) ו- CATIA (https://www.ucl.ac.uk/hansen-lab/catia/) זמינות לביצוע התאמת נתונים באמצעות משוואות בלוק-מקונל. - בדוק את המהימנות של הפרמטרים המותאמים להערכת χהמופחת 2 כפונקציה של pb ו- kex.
הערה: χהמופחת 2 מסופק בקובץ הפלט. pb ו- kex ניתן לרסן לערכים ספציפיים באמצעות הגבולות התחתונים והעליון בהליכי ההתאמה. בתסריטים שלנו, הגבולות התחתונים והעליון עבור pb הם lb(2) ו- ub(2), בהתאמה. הגבולות התחתונים והעליון עבור kex הם lb(3) ו- ub(3), בהתאמה. - הערכת השגיאה בפרמטרים המותאמים. ניתן לעשות זאת על ידי הגדרת הערך של MC_fac בקובץ ה- Script ל- 1 וחזרה על ההתאמה מספר פעמים (בדרך כלל >20 חוזר). השגיאה בכל פרמטר מוערכת כסטיית התקן של ההתפלגות.
הערה: הגדרת MC_fac ל- 1 יוצרת ערכת נתונים סינתטית שבה שגיאה מבוזרת של גאוסיאני (המחושבת בהתבסס על השגיאה הניסיונית) מתווספת לנתונים הניסיוניים.
Access restricted. Please log in or start a trial to view this content.
תוצאות
הפרוטוקול המתואר כאן מביא לרכישת פרופילי RD עבור כל שיא בספקטרום 1H-15N TROSY (איור 3A). מפרופילי המו"פ הנרכשים ניתן להעריך את תרומת ההחלפה לרגיעה רוחבית של 15N של כל קבוצת אמידה(איור 3A, 3B). על ידי התוויית Rex על המבנה 3D של החלבון תחת חקי?...
Access restricted. Please log in or start a trial to view this content.
Discussion
כתב יד זה מתאר את הפרוטוקול המיושם במעבדה לרכישה וניתוח של 15נתוני N RD על חלבונים. בפרט, השלבים הקריטיים להכנת מדגם NMR, מדידת נתוני NMR וניתוח פרופילי המו"פ מכוסים. להלן כמה היבטים חשובים לגבי רכישה וניתוח של ניסויי מו"פ נדונים. עם זאת, לתיאור מעמיק יותר של הניסוי וניתוח הנתונים, מומלץ מאו?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
כל הסופרים קראו ואישרו את כתב היד. אנחנו לא מצהירים על ניגודי אינטרסים.
Acknowledgements
עבודה זו נתמכה על ידי כספים מ NIGMS R35GM133488 ומ קרן הצדקה רוי ג'יי קרבר לV.V.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
References
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960(2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. 3rd A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. 3rd A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G. 3rd, Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747(2019).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved