É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
15 N CPMG Relaxation Dispersão para a Investigação da Dinâmica Conformacional de Proteínas na escala de tempo μs-ms
Neste Artigo
Resumo
Aqui, é fornecida uma descrição detalhada do protocolo implementado no laboratório para aquisição e análise de perfis de dispersão de relaxamento de 15N por espectroscopia de solução NMR.
Resumo
A dinâmica conformacional proteica desempenha papéis fundamentais na regulação da catálise enzimática, ligação de ligantes, aosteria e sinalização, que são importantes processos biológicos. Entender como o equilíbrio entre estrutura e dinâmica rege a função biológica é uma nova fronteira na biologia estrutural moderna e tem inflamado diversos desenvolvimentos técnicos e metodológicos. Entre estes, os métodos NMR da solução de relaxamento cpmg fornecem informações únicas de resolução atômica sobre a estrutura, cinética e termodinâmica do equilíbrio conformacional proteico que ocorre na escala de tempo μs-ms. Aqui, o estudo apresenta protocolos detalhados para aquisição e análise de um experimento de dispersão de relaxamento de 15N. Como exemplo, o pipeline para a análise da dinâmica μs-ms no domínio terminal C da bactéria Enzima I é mostrado.
Introdução
Os experimentos de dispersão de relaxamento (RD) Carr-Purcell Meiboom-Gill (CPMG) são usados em uma base de rotina para caracterizar o equilíbrio conformacional que ocorre na escala de tempo μs-ms por solução espectroscopia NMR1,2,3,4,5. Em comparação com outros métodos de investigação da dinâmica conformacional, as técnicas de CPMG são relativamente fáceis de implementar em espectrômetros de RN modernos, não exigem etapas especializadas de preparação de amostras (ou seja, cristalização, congelamento ou alinhamento de amostras e/ou conjugação codinâmica com uma etiqueta fluorescente ou paramagnética), e fornecem uma caracterização abrangente do equilíbrio conformacional retornando informações estruturais, cinéticas e termodinâmicas em processos de troca. Para que um experimento cpmg informe sobre um equilíbrio conformacional, duas condições devem ser aplicadas: (i) as rodadas de RMN observadas devem possuir diferentes mudanças químicas nos estados submetidos à troca conformacional (microestados) e (ii) a troca deve ocorrer em uma escala de tempo que varia de ~50 μs a ~10 ms. Nessas condições, a taxa de relaxamento transversal observada 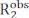 é a soma do R2 intrínseco (o R2 medido na ausência de dinâmica μs-ms)
é a soma do R2 intrínseco (o R2 medido na ausência de dinâmica μs-ms) 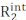 e a contribuição cambial para o relaxamento transversal (Rex). A contribuiçãoR ex para R2obs pode ser progressivamente saciada reduzindo o espaçamento entre os pulsos de 180° que constituem o bloco CPMG da sequência de pulso, e as curvas RD resultantes podem ser modeladas usando a teoria Bloch-McConnell para obter a diferença de mudança química entre os microestados, a população fracionária de cada microestado, e as taxas de troca entre microestados (Figura 1)1,2,3.
e a contribuição cambial para o relaxamento transversal (Rex). A contribuiçãoR ex para R2obs pode ser progressivamente saciada reduzindo o espaçamento entre os pulsos de 180° que constituem o bloco CPMG da sequência de pulso, e as curvas RD resultantes podem ser modeladas usando a teoria Bloch-McConnell para obter a diferença de mudança química entre os microestados, a população fracionária de cada microestado, e as taxas de troca entre microestados (Figura 1)1,2,3.
Várias sequências de pulso diferentes e protocolos de análise foram relatados na literatura para experimentos de 15N CPMG. Aqui, o protocolo implementado no laboratório é descrito. Em particular, serão introduzidas as etapas cruciais para a elaboração da amostra de RMN, a configuração e aquisição dos experimentos de RMN e o processamento e análise dos dados da RMN(Figura 2). Para facilitar a transferência do protocolo para outros laboratórios, o programa de pulso, scripts de processamento e análise e um exemplo de conjunto de dados são fornecidos como Arquivos Suplementares e estão disponíveis para download em (https://group.chem.iastate.edu/Venditti/downloads.html). A sequência de pulso fornecida incorpora um ciclo de fase de quatro etapas no bloco CPMG para supressão de artefatos dependentes de deslocamento6 e é codificada para aquisição de vários experimentos intercalados. Esses experimentos intercalados têm um período de relaxamento idêntico, mas números diferentes de pulsos de refoco, a fim de alcançar diferentes campos CPMG7. Também é importante notar que o programa de pulso descrito mede o 15N R2 do componente TROSY do sinal NMR8. No geral, o protocolo foi aplicado com sucesso para a caracterização da troca conformacional em proteínas de médio e grande porte4,5,9,10. Para sistemas menores (<20 kDa), o uso de uma sequência de pulso baseada em Coerência Quântica Única Heteronuclear (HSQC)é aconselhável.
Protocolo
1. Preparação da amostra de RMN
- Expresse e purifique uma amostra de 2H,15N-labled da proteína de interesse.
NOTA: Enquanto uma amostra de proteína de 15N pode ser usada para aquisição do experimento CPMG RD, a perdateração (quando possível) aumenta drasticamente a qualidade dos dados obtidos. Protocolos para a produção de proteínas perdidos estão disponíveis na literatura13. - Tampão troque a amostra de proteína purificada em um tampão de RMR desgaseado.
- Transfira a amostra de RMN para o tubo NMR.
NOTA: A concentração da amostra de RMN precisa ser cuidadosamente otimizada para maximizar a relação sinal-ruído e minimizar a ocorrência de interações proteína-proteína. Em geral, a faixa de concentração típica para experimentos cpmg é de 0,1-1,0 mM.
2. Primeira configuração do experimento NMR
- Baixe e descompacte os Arquivos Suplementares.
- Copie os arquivos bits.vv e trosy_15N_cpmg.vv (localizados na pasta denominada pulseprogram) no diretório do programa de pulso (
- Abra o software de aquisição e copie um experimentoH-15N HSQC anteriormente executadoem um novo experimento usando o edcde comando .
- Usando o pulprog decomando, carregue o programa de pulso trosy_15N_cpmg.vv no experimento recém-criado.
- Configure o experimento CPMG usando as instruções fornecidas no final do arquivo do programa de pulso (trosy_15N_cpmg.vv).
3. Configuração de rotina do experimento NMR
- Introduza a amostra no ímã e execute todas as etapas básicas para aquisição de qualquer experimento NMR: bloquear e shim a amostra; combinar e sintonizar os canais 1H e 15N.
- Defina p1 e p7 até a duração dos pulsos de 1H e 15N duros de 90°, respectivamente.
NOTA: Para obter ótimos resultados, é importante calibrar o pulso de 15N 90° com muito cuidado. A calibração é geralmente realizada usando uma amostra de 100 mM de 15ureias rotuladas em N em DMSO, conforme descrito no manual do espectrômetro. Além disso, é possível verificar novamente a calibração diretamente na amostra NMR de trabalho adquirindo o primeiro incremento de umespectroH-15N HSQC no qual o pulso de 15N 90° do primeiro bloco INEPT é alterado para um pulso de 180°. Se a calibração estiver correta, deve ser obtido um nulo. - Na janela de aquisição, defina a largura central e espectral para as dimensões de 1H e 15N.
NOTA: Centralizar o espectro de 1H na frequência do sinal de água para a supressão ideal da água. - Defina o atraso de relaxamento (d30) igual a 0,7 T2, onde T2 é o tempo de relaxamento transversal esperado de 15N de sua proteína.
NOTA: O valor do d30 pode ser otimizado empiricamente para obter os melhores resultados. - Use o vclist de comando para criar uma lista de números inteiros correspondentes ao parâmetro n na Figura 1A e Figura 1B. Certifique-se de que cada entrada na lista corresponde a um campo CPMG diferente (νcpmg) de acordo com νcpmg = 4 x n / d30. Certifique-se de que o primeiro número da lista é 0 (isso corresponde ao experimento de referência para o qual o bloco CPMG é ignorado e d30 = 0 s) e não usar números maiores que 1.000 x d30 / 4. Números maiores do que esse limiteresultam em > de 1 kHz e podem danificar a sonda.
- Defina l8 para o número de entradas no vclist, l3 para o número de pontos reais para a dimensão indireta (geralmente uma faixa de 64-200 é definida para l3), e 1 td igual a l8 x l3 x 2.
NOTA: Realize as etapas 3.7-3.11 para otimizar a supressão da água. - Definir o ganho do receptor (rg) para 1; abrir o arquivo do programa de pulso (edcpul), ir para a linha 91, remover o ponto e vírgula anterior ao goto 999e salvar o arquivo.
- Utilizando os gs de comando ajuste os parâmetros spdb0 (ou spdw0) a fim de minimizar a intensidade do sinal FID.
- Reintroduza o ponto e vírgula na linha 91 do arquivo do programa de pulso e salve o arquivo.
- Repita as etapas 3.7-3.9 para otimizar o spdb11 (linha 168 do arquivo do programa de pulso), spdb2 (linha 179 do arquivo do programa de pulso) e pldb2 (linha 184 do arquivo do programa de pulso).
- Execute a rga de comando para otimizar o ganho do receptor.
- Defina o número de scans(ns) para um múltiplo de 8.
- Execute o comando zg para iniciar o experimento.
4. Processamento e análise dos dados de RMN
- Copie a pasta chamada Process (Arquivos Suplementares) no diretório que contém o arquivo ser.
- Use os arquivos sep_fid.com e ft2D.com para processar os dados NMR.
NOTA: Instruções sobre como editar os scripts de processamento são fornecidas dentro dos arquivos sep_fid.com e ft2D.com. - Abra os arquivos da UCSF contidos no diretório CPMG_Sparky_files em Sparky.
NOTA: Os arquivos ucsf são criados pelos scripts de processamento. Há um arquivo ucsf para cada entrada no vclist. O primeiro arquivo ucsf (test_1.ucsf) contém o experimento de referência. Os arquivos subsequentes da UCSF (test_2.ucsf, test_3.ucsf,... test_n.ucsf) são encomendados do menor ao maior valor deν cpmg. - Escolha os picos cruzados NMR no espectro NMR de referência (test_1.ucsf).
- Selecione todos os picos cruzados escolhidos usando o comando Sparky pa.
- Executar o comando Sparky rh. Este comando abre uma janela de diálogo. Selecione as alturas deopção na mesma posição em cada espectro . Clique em Configuração e verifique todos os espectros NMR. Clique em Atualizar e salve o arquivo de saída no diretório de trabalho. O arquivo de saída contém a matriz das intensidades de sinal sobre todos os espectros NMR abertos.
NOTA: Protocolos mais precisos que usam o ajuste de linha para recuperar intensidades de experimentos pseudo-3D intercalados foram descritos na literatura14. O software disponível gratuitamente para montagem de linha está disponível em https://pint-nmr.github.io/PINT/ (PINT), https://www.ucl.ac.uk/hansen-lab/fuda/ (FUDA) e dentro do NMRPipe (módulo nLinLS) e SPARKY (módulo de ti). - Para cada campo CPMG, converta as intensidades de sinal para taxas de R2 utilizando a
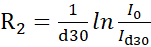 fórmula, onde I0 e Id30 são a intensidade da referência (vc = 0) e relaxada (vc > 0) espectros NMR, respectivamente.
fórmula, onde I0 e Id30 são a intensidade da referência (vc = 0) e relaxada (vc > 0) espectros NMR, respectivamente.
NOTA: Nos Arquivos Suplementares,é fornecido um modelo do arquivo de planilha (R2_calc.xls) usado para converter as intensidades de sinal em taxas de R2 e visualizar os perfis RD. - Leia o nível de ruído no espectro de referência usando o comando Sparky st e propigue o erro nas taxas R2.
NOTA: Nos Arquivos Suplementares,é fornecido um modelo do arquivo de planilha (R2_calc.xls) usado para propagar o erro nas taxas de R2 medidos.
5. Montagem de curvas RD
- Copie a pasta nomeada RD_fitting(Arquivos Suplementares)no diretório de trabalho.
- Para cada pico de RMR atribuído, estimar Rex calculando as
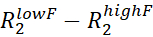 taxas R
taxas R 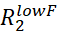
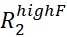 2 medidas nas taxas mais baixas e mais altas do vclist.
2 medidas nas taxas mais baixas e mais altas do vclist.
- Inspecione visualmente as curvas RD com Rex maior que duas vezes o erro estimado e descarte todas as curvas RD que são muito barulhentos para serem modeladas com precisão.
NOTA: O ruído noex R pode ser propagado a partir dos erros ligados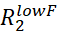 e
e 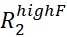 .
. - Prepare um arquivo de entrada para o script de montagem usando todas as curvas RD comR ex maior que duas vezes o erro estimado.
NOTA: Instruções detalhadas sobre a preparação dos arquivos de entrada são fornecidas dentro dos scripts de montagem. Os arquivos de entrada de exemplo são fornecidos como Arquivos Suplementares. Comumente, os dados RD são medidos em dois campos estáticos diferentes e instalados simultaneamente. Neste protocolo, são necessários dois arquivos de entrada diferentes(Arquivos Suplementares). - Encaixe os dados RD usando os scripts fornecidos em Arquivos Suplementares.
NOTA: Dois scripts diferentes são fornecidos para executar um ajuste global baseado em resíduos ou um ajuste global das curvas RD, respectivamente. Ambos os scripts se encaixam nas curvas RD usando um modelo de troca de dois sites e a equação carver-richards. Instruções mais detalhadas são fornecidas dentro dos scripts de montagem. Pacotes de software adicionais como Chemex (https://github.com/gbouvignies/ChemEx) e CATIA (https://www.ucl.ac.uk/hansen-lab/catia/ ) estão disponíveis para realizar a montagem de dados usando as equações Block-McConnell. - Teste a confiabilidade dos parâmetros instalados estimando o χ2 reduzido em função de pb e kex.
NOTA: O χ2 reduzido é fornecido no arquivo de saída. pb e kex podem ser contidos a valores específicos usando limites inferiores e superiores nos procedimentos de montagem. Em nossos scripts, os limites inferiores e superiores para pb são lb(2) e ub(2), respectivamente. Os limites inferior e superior para kex são lb(3) e ub(3), respectivamente. - Estime o erro nos parâmetros instalados. Isso pode ser feito definindo o valor de MC_fac no script para 1 e repetindo o encaixe várias vezes (normalmente >20 repetições). O erro em cada parâmetro é estimado como o desvio padrão da distribuição.
NOTA: A configuração MC_fac para 1 gera um conjunto de dados sintético no qual um erro distribuído gaussiano (calculado com base no erro experimental) é adicionado aos dados experimentais.
Resultados
O protocolo descrito aqui resulta na aquisição de perfis RD para cada pico no espectro 1H-15N TROSY(Figura 3A). A partir dos perfis de RD adquiridos, é possível estimar a contribuição cambial para o relaxamento transversal de 15N de cada grupo backbone amide(Figura 3A,3B). Ao traçar oR ex na estrutura 3D da proteína sob investigação, é possível identificar as regiões estruturais submetidas...
Discussão
Este manuscrito descreve o protocolo implementado em laboratório para aquisição e análise de dados de 15N RD sobre proteínas. Em particular, são abordadas as etapas cruciais para a preparação da amostra de RMN, medição dos dados de RMN e análise dos perfis rd. Abaixo alguns aspectos importantes sobre a aquisição e análise de experimentos de RD são discutidos. No entanto, para uma descrição mais aprofundada do experimento e análise de dados, o estudo cuidadoso da literatura original é altamen...
Divulgações
Todos os autores leram e aprovaram o manuscrito. Não declaramos conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado por fundos da NIGMS R35GM133488 e do Roy J. Carver Charitable Trust para V.V.
Materiais
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
Referências
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados