JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
15 Μs-ms 타임스케일에 단백질 형성 역학의 조사를 위한 N CPMG 이완 분산
요약
여기서, 솔루션 NMR 분광법에 의한 15N이완 분산 프로파일의 수집 및 분석을 위해 실험실에서 구현된 프로토콜에 대한 자세한 설명이 제공된다.
초록
단백질 형성 역학은 중요한 생물학적 과정인 효소 촉매, 리간드 결합, 알로스터리 및 시그널링의 조절에 근본적인 역할을 합니다. 구조와 역학 사이의 균형이 생물학적 기능을 어떻게 지배하는지 이해하는 것은 현대 구조 생물학의 새로운 개척지이며 몇 가지 기술적 및 방법론적 발전에 불을 붙였습니다. 이들 중, CPMG 이완 분산 용NMR 방법은 μs-ms 타임스케일에서 발생하는 단백질 형태 평형의 구조, 운동학 및 열역학에 대한 고유하고 원자분해능적인 정보를 제공한다. 여기서, 연구 결과는 15N 이완 분산 실험의 취득 그리고 분석을 위한 상세한 프로토콜을 제시합니다. 예를 들어, 박테리아 효소의 C-말단 도메인에서 μs-ms 역학의 분석을 위한 파이프라인이 나타난다.
서문
Carr-Purcell Meiboom-Gill (CPMG) 이완 분산 (RD) 실험은 용액 NMR분광법 1,2,3,4,5에의해 μs-ms 시간 척도에서 발생하는 형태 평형을 특성화하기 위해 일상적인 베이스에 사용된다. 균역학을 조사하는 다른 방법에 비해 CPMG 기술은 현대 NMR 분광계에서 비교적 구현하기 쉽고, 특수 시료 준비 단계(즉, 결정화, 샘플 동결 또는 정렬 및/또는 형광 또는 파라마그네틱 태그와 함께 동월 결합)을 필요로 하지 않으며, 구조적, 역학적 재형성, 역학적 재형성, 역학적 재해석, 동역학적 재보행의 포괄적인 특성화를 제공한다. CPMG 실험이 형태 평형에 대해 보고하기 위해서는, 두 가지 조건이 적용되어야 한다: (i) 관찰된 NMR 스핀은 형태 교환(microstates)을 겪고 있는 상태에서 다른 화학적 변화를 가져야 하며 (ii) 교환은 ~50 μs에서 ~10ms에 이르는 시간 척도로 이루어져야 한다. 이러한 조건하에서, 관찰된 횡방향 이완속도()는 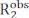 본질R2(μs-ms 역학의 부재하에서 측정된R2)의
본질R2(μs-ms 역학의 부재하에서 측정된R2)의 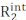 합이며, 횡방향 이완(Rex)에대한 교환 기여도이다. R2obs에 대한 R전 기여도는 펄스 서열의 CPMG 블록을 구성하는 180° 펄스 사이의 간격을 감소시킴으로써 점진적으로 담금질될 수 있으며, 그 결과 RD 곡선은 블로흐-맥코넬 이론을 이용하여 마이크로스테이트 간의 화학적 변화 차이, 각 마이크로스테이트의 분수 인구, 및 소행성 간 교환속도(도1)중 의 화학적 변화 차이를 얻을 수있다.
합이며, 횡방향 이완(Rex)에대한 교환 기여도이다. R2obs에 대한 R전 기여도는 펄스 서열의 CPMG 블록을 구성하는 180° 펄스 사이의 간격을 감소시킴으로써 점진적으로 담금질될 수 있으며, 그 결과 RD 곡선은 블로흐-맥코넬 이론을 이용하여 마이크로스테이트 간의 화학적 변화 차이, 각 마이크로스테이트의 분수 인구, 및 소행성 간 교환속도(도1)중 의 화학적 변화 차이를 얻을 수있다.
15NCPMG 실험에 대한 문헌에서 여러 가지 상이한 펄스 서열 및 분석 프로토콜이 보고되었다. 본원에서, 실험실에서 구현된 프로토콜은 기술된다. 특히 NMR 샘플의 제조, NMR 실험의 설정 및 획득, NMR 데이터의 처리 및 분석을 위한 중요한 단계가 도입될것이다(그림 2). 프로토콜을 다른 실험실로 쉽게 전송하기 위해 펄스 프로그램, 처리 및 분석 스크립트 및 하나의 예제 데이터 집합은 보충 파일로 제공되며(https://group.chem.iastate.edu/Venditti/downloads.html)에서 다운로드할 수 있습니다. 제공된 펄스 서열은 오프셋 종속 아티팩트6의 억제를 위해 CPMG 블록에 4단계 위상 주기를 통합하고 여러 인터리브 된 실험을 수집하기 위해 코딩됩니다. 이러한 인터리브 된 실험은 동일한 휴식 기간을 가지고 있지만 다른 CPMG 필드7을달성하기 위해 펄스를 다시 초점을 맞추는 다른 수. 또한 기술된 펄스 프로그램이 NMR 신호8의TROSY 성분의 15N R2를 측정한다는 것을 통지하는 것도 중요하다. 전반적으로, 프로토콜은 중형 및 대형단백질4,5,9,10에서형성 교류의 특성화를 성공적으로 적용하였다. 소형 시스템(<20 kDa)의 경우, 이종핵 단일 양자 일관성(HSQC)-기반 펄스 서열11,12의 사용은 바람직하다.
프로토콜
1. NMR 샘플 의 준비
- 관심 있는 단백질의 2H,15N-labled 샘플을 표현하고 정화합니다.
참고: 15개의N 라벨 단백질 샘플이 CPMG RD 실험의 획득에 사용될 수 있지만, 감단(가능한 경우)은 얻어진 데이터의 품질을 극적으로 증가시킵니다. 정해성 단백질의 생산을 위한 프로토콜은문헌(13)에서유효하다. - 완충제는 정제된 단백질 샘플을 탈가스 NMR 버퍼로 교환합니다.
- NMR 샘플을 NMR 튜브로 전송합니다.
참고: NMR 샘플의 농도는 신호 대 잡음 비율을 최대화하고 단백질-단백질 상호 작용의 발생을 최소화하기 위해 신중하게 최적화되어야 합니다. 일반적으로 CPMG 실험의 전형적인 농도 범위는 0.1-1.0 mM이다.
2. NMR 실험의 첫 번째 설정
- 추가 파일을 다운로드하고 압축 해제합니다.
- 파일 bits.vv 및 trosy_15N_cpmg.vv(펄스 프로그램라는 폴더에 위치)를 펄스 프로그램 디렉토리(
- 오픈 획득 소프트웨어를 복사하고 명령 edc를 사용하여 이전 실행 1H-15N HSQC 실험을 새로운 실험으로 복사합니다.
- 명령 펄프로그를사용하여 펄스 프로그램 trosy_15N_cpmg.vv를 새로 만든 실험에 로드합니다.
- 펄스 프로그램 파일(trosy_15N_cpmg.vv)의 끝에 제공된 지침을 사용하여 CPMG 실험을 설정합니다.
3. NMR 실험의 일상적인 설정
- 자석에 샘플을 소개하고 NMR 실험을 획득하기위한 모든 기본 단계를 수행 : 잠그고 샘플을 심; 일치하고 1H 및 15N 채널을 조정합니다.
- p1 및 p7을 각각 1H및 15N 하드 90° 펄스의 지속 시간으로 설정합니다.
참고: 최적의 결과를 얻으려면 15N 90° 펄스를 세심한 주의를 기울여 보정하는 것이 중요합니다. 교정은 일반적으로 분광계 매뉴얼에 설명된 바와 같이 DMSO에서 15N 라벨 우레아의 100mMM 샘플을 사용하여 수행됩니다. 또한, 첫 번째 INEPT 블록의 15N 90° 펄스가 180° 펄스로 전환되는 1H-15 N HSQC 스펙트럼의 첫 번째 증분을 획득하여 작업NMR 샘플에서 직접 교정을 다시 확인할 수 있다. 교정이 정확하면 null을 얻어야 합니다. - 획득 창에서 1H및 15N 치수의 중심 및 스펙트럼 너비를 설정합니다.
참고: 최적의 물 억제를 위해 물 신호의 주파수에서 1H스펙트럼을 중심으로 합니다. - T2가 단백질의 예상 15N횡방향 이완 시간인 0.7 T2와동일한 이완 지연(d30)을 설정합니다.
참고: d30의 값을 경험적으로 최적화하여 최상의 결과를 얻을 수 있습니다. - 명령 vclist를 사용하여 도 1A 및 도 1B에서매개 변수 n에 해당하는 정수 번호 목록을 만듭니다. 목록의 각 항목이다른 CPMG 필드(θcpmg)에해당하는지 확인하여 cpmg = 4 x n/d30에 따라 다릅니다. 목록의 첫 번째 번호가 0인지 확인하십시오 (이것은 CPMG 블록을 건너 뛰고 d30 = 0 s를 건너 뛰는 참조 실험에 해당하며 1,000 x d30 / 4보다 큰 숫자를 사용하지 않아야합니다. 이 임계값보다 큰 숫자는 1kHz>cpmg을 생성하고 프로브를 손상시킬 수 있습니다.
- l8을 vclist의 항목 수로 설정하고 l3은 간접 차원의 실제 포인트 수(일반적으로 64-200 의 범위가 l3로 설정됨) 및 l8 x l3 x 2와 동일한 1 td를 설정합니다.
참고: 3.7-3.11 단계를 수행하여 물 억제를 최적화합니다. - 수신기 게인(rg)을 1로 설정합니다. 펄스 프로그램 파일(edcpul)을 열고, 91줄로 이동하고, goto 999앞에 있는 세미콜론을 제거하고, 파일을 저장한다.
- 명령 GS를 사용하여 FID 신호의 강도를 최소화하기 위해 매개 변수 spdb0(또는 spdw0)을 조정합니다.
- 펄스 프로그램 파일의 91줄에서 세미콜론을 다시 도입하고 파일을 저장합니다.
- 스Pdb11(펄스 프로그램 파일의 라인 168), spdb2(펄스 프로그램 파일의 라인 179) 및 pldb2(펄스 프로그램 파일의 라인 184)를 최적화하기 위해 3.7-3.9 단계를 반복한다.
- 명령 rga를 실행하여 수신기 게인을 최적화합니다.
- 스캔횟수(ns)를8의 배수로 설정합니다.
- 명령을 실행하여 실험을 시작합니다.
4. NMR 데이터의 처리 및 분석
- 프로세스(보충 파일)라는폴더를 ser 파일이 포함된 디렉토리에 복사합니다.
- 파일을 sep_fid.com ft2D.com 사용하여 NMR 데이터를 처리합니다.
참고: 처리 스크립트를 편집하는 방법에 대한 지침은 sep_fid.com 및 ft2D.com 파일 내에 제공됩니다. - 스파키의 디렉토리 CPMG_Sparky_files 포함된 ucsf 파일을 엽니다.
참고: ucsf 파일은 처리 스크립트에 의해 만들어집니다. vclist의 각 항목에 대해 하나의 ucsf 파일이 있습니다. 첫 번째 ucsf 파일(test_1.ucsf)에는 참조 실험이 포함되어 있습니다. 후속 ucsf 파일 (test_2.ucsf, test_3.ucsf,... test_n.ucsf)는 가장 낮은 값에서 가장 큰 값으로주문됩니다. - 참조 NMR 스펙트럼(test_1.ucsf)에서 NMR 교차 피크를 선택합니다.
- 스파키 명령 pa를사용하여 선택한 모든 교차 피크를 선택합니다.
- 스파키 커맨드 rh를실행합니다. 이 명령은 대화 창을 엽니다. 각 스펙트럼에서 동일한 위치에서옵션 높이를 선택합니다. 설정을 클릭하고 모든 NMR 스펙트럼을 확인합니다. 업데이트를 클릭하고 작업 디렉터리에서 출력 파일을 저장합니다. 출력 파일에는 열린 모든 NMR 스펙트럼에 대한 신호 강도의 행렬이 포함되어 있습니다.
참고: 인터리브 된 의사 3D 실험에서 강도를 복구하기 위해 라인 모양 피팅을 사용하는 보다 정확한 프로토콜이 문헌14에설명되었습니다. 라인 셰이프 피팅에 사용할 수 있는 소프트웨어는 https://pint-nmr.github.io/PINT/(PINT), https://www.ucl.ac.uk/hansen-lab/fuda/(FUDA), NMRPipe(nLinLS 모듈) 및 SPARKY(it 모듈) 내에서 사용할 수 있습니다. - 각 CPMG 필드에 대해,
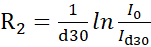 I0과 Id30이 참조(vc = 0) 및 이완(vc > 0) NMR 스펙트럼의 강도인 수식을 사용하여 신호 강도를 R 2 비율로 변환한다.
I0과 Id30이 참조(vc = 0) 및 이완(vc > 0) NMR 스펙트럼의 강도인 수식을 사용하여 신호 강도를 R 2 비율로 변환한다.
참고: 보충 파일에서신호 강도를 R2 비율로 변환하고 RD 프로파일을 시각화하는 데 사용되는 스프레드시트 파일(R2_calc.xls)의 템플릿이 제공됩니다. - 스파키 명령 st를 사용하여 참조 스펙트럼의 노이즈 레벨을 읽고 R2 속도에 오류를 전파합니다.
참고: 보충 파일에서측정된 R2 요금에 오류를 전파하는 데 사용되는 스프레드시트 파일(R2_calc.xls)의 템플릿이 제공됩니다.
5. RD 곡선 피팅
- RD_fitting(보충 파일)이라는폴더를 작업 디렉토리에 복사합니다.
- 할당된 각 NMR 피크에 대해 Rex를 계산하여
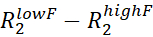
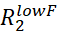 추정하고
추정하고 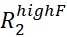 vclist에서 가장 낮고 가장 높은cpmg에서 측정된 R2 요율입니다.
vclist에서 가장 낮고 가장 높은cpmg에서 측정된 R2 요율입니다.
- 예상 오차의 두 배보다 큰Rex로 RD 곡선을 시각적으로 검사하고 너무 시끄러운 모든 RD 커브를 정확하게 모델링할 수 있습니다.
참고: Rex의 노이즈는 오류에서 전파할 수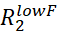
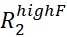 있으며.
있으며. - 예상 오차의 두 배보다 큰 Rex가 있는 모든 RD 커브를 사용하여 피팅 스크립트에 대한 입력 파일을 준비합니다.
참고: 입력 파일 준비에 대한 자세한 지침은 피팅 스크립트 내에 제공됩니다. 예제 입력 파일은 보충 파일로제공됩니다. 일반적으로 RD 데이터는 두 개의 서로 다른 정적 필드에서 측정되고 동시에 장착됩니다. 이 프로토콜에서는 두 개의 서로 다른 입력 파일이필요합니다(추가 파일). - 보충 파일에제공된 스크립트를 사용하여 RD 데이터에 적합합니다.
참고: 각각 RD 커브의 잔류물 기반 또는 전역 맞춤을 수행하기 위해 두 개의 서로 다른 스크립트가 제공됩니다. 두 스크립트 모두 2사이트 교환 모델과 카버-리차즈 방정식을 사용하여 RD 곡선에 적합합니다. 피팅 스크립트 내에서 보다 자세한 지침이 제공됩니다. Chemex(https://github.com/gbouvignies/ChemEx) 및 CATIA(https://www.ucl.ac.uk/hansen-lab/catia/)와 같은 추가 소프트웨어 패키지를 사용하여 블록-맥코넬 방정식을 사용하여 데이터 피팅을 수행할 수 있습니다. - pb 및 k 전의 함수로서 감소된 2를 추정하는 장착 된 매개 변수의 신뢰성을테스트합니다.
참고: 감소된 θ2가 출력 파일에 제공됩니다. pb 및 kex는 피팅 절차에서 하부 및 상한을 사용하여 특정 값에 구속될 수 있습니다. 스크립트에서 pb의 하부 및 상한은 각각 lb(2) 및 ub(2)입니다. kex의 하부 및 상한은 각각 lb(3) 및 ub(3)입니다. - 장착된 매개 변수의 오류를 추정합니다. 스크립트의 MC_fac 값을 1로 설정하고 피팅을 여러 번 반복하여 수행할 수 있습니다(일반적으로 >20 반복). 각 매개 변수의 오류는 분포의 표준 편차로 추정됩니다.
참고: MC_fac 1로 설정하면 가우시안 분산 오차(실험 오류에 따라 계산)가 실험 데이터에 추가되는 합성 데이터 집합이 생성됩니다.
결과
여기서 설명된 프로토콜은 1H-15N TROSY스펙트럼(그림 3A)에서각 피크에 대한 RD 프로파일을 획득하는 결과를 낳는다. 인수된 RD 프로파일로부터, 각 백본 아마이드 그룹의 15N횡방향 이완에 대한 교환 기여도를 추정할 수있다(도 3A,3B). 조사 중인 단백질의 3D 구조에 Rex를 플로팅함으로써, μs-ms 시간
토론
이 원고는 단백질에 대한 15NRD 데이터를 수집 및 분석을 위해 실험실에서 구현된 프로토콜을 설명합니다. 특히 NMR 샘플 의 준비, NMR 데이터 측정 및 RD 프로파일 분석을 위한 중요한 단계가 다룹니다. Rd 실험의 인수 및 분석에 관한 몇 가지 중요한 측면에 대해 논의됩니다. 그러나, 실험 및 데이터 분석에 대한 보다 심층적인 설명을 위해, 본문문헌을 주의 깊게 공부하는 것이
공개
모든 저자는 원고를 읽고 승인했습니다. 우리는 이해 상충을 선언하지 않습니다.
감사의 말
이 작품은 NIGMS R35GM133488의 자금과 로이 J. 카버 자선 신탁에서 V.V.V.에 이르기까지 자금으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Cryoprobe | Bruker | 5mm TCI 800 H-C/N-D cryoprobe | Improve sensitivity |
| Deuterium Oxide | Sigma Aldrich | 756822-1 | >99.8% pure, utilised in preparing NMR samples and deuterated cultures |
| Hand driven centrifuge | United Scientific supply | CENTFG1 | Used to remove any air bubbles or residual liquid stuck on the walls of NMR tube. |
| High Field NMR spectrometer | Bruker | Bruker Avance II 600, Bruker Avance 800 | Acquisition of the NMR data |
| MATLAB | MathWorks | https://www.mathworks.com/products/get-matlab.html | Modeling of the NMR data |
| NMR pasteur Pipette | Corning Incorporation | 7095D-NMR | Pyrex glass pastuer pipette to transfer liquid sample in NMR tube |
| NMR tube | Willmad Precision | 535-PP-7 | 5mm thin wall 7'' cylinderical glass tube |
| NMRPipe | Institute of Biosciences and Biotechnology research | https://www.ibbr.umd.edu/nmrpipe/install.html | NMR data processing |
| SPARKY | University of California, San Francisco | https://www.cgl.ucsf.edu/home/sparky/ | Analysis of the NMR data |
| Tospin 3.2 (or newer) | Bruker | https://www.bruker.com/protected/en/services/software-downloads/nmr/pc/pc-topspin.html | Acquisition Software |
참고문헌
- Anthis, N. J., Clore, G. M. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48 (1), 35-116 (2015).
- Lisi, G. P., Loria, J. P. Solution NMR spectroscopy for the study of enzyme allostery. Chemical Reviews. 116 (11), 6323-6369 (2016).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Venditti, V., Clore, G. M. Conformational selection and substrate binding regulate the monomer/dimer equilibrium of the C-terminal domain of Escherichia coli enzyme I. Journal of Biological Chemistry. 287 (32), 26989-26998 (2012).
- Venditti, V., et al. Large interdomain rearrangement triggered by suppression of micro- to millisecond dynamics in bacterial Enzyme I. Nature Communications. 6, 5960 (2015).
- Yip, G. N., Zuiderweg, E. R. A phase cycle scheme that significantly suppresses offset-dependent artifacts in the R2-CPMG 15N relaxation experiment. Journal of Magnetic Resonance. 171 (1), 25-36 (2004).
- Mulder, F. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (micros-ms) time scale dynamics in protein side chains by (15)N relaxation dispersion NMR spectroscopy: application to Asn and Gln residues in a cavity mutant of T4 lysozyme. Journal of the American Chemical Society. 123 (5), 967-975 (2001).
- Loria, J. P., Rance, M., Palmer, A. G. A TROSY CPMG sequence for characterizing chemical exchange in large proteins. Journal of Biomolecular NMR. 15 (2), 151-155 (1999).
- Dotas, R. R., et al. Hybrid thermophilic/mesophilic enzymes reveal a role for conformational disorder in regulation of bacterial Enzyme I. Journal of Molecular Biology. 432 (16), 4481-4498 (2020).
- Purslow, J. A., et al. Active site breathing of human Alkbh5 revealed by solution NMR and accelerated molecular dynamics. Biophysical Journal. 115, 1895-1905 (2018).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr−Purcell−Meiboom−Gill sequence for characterizing chemical exchange by NMR Spectroscopy. Journal of the American Chemical Society. 121 (10), 2331-2332 (1999).
- Hansen, D. F., Vallurupalli, P., Kay, L. E. An improved 15N relaxation dispersion experiment for the measurement of millisecond time-scale dynamics in proteins. Journal of Physical Chemistry B. 112 (19), 5898-5904 (2008).
- Tugarinov, V., Kanelis, V., Kay, L. E. Isotope labeling strategies for the study of high-molecular-weight proteins by solution NMR spectroscopy. Nature Protocols. 1 (2), 749-754 (2006).
- Niklasson, M., et al. Comprehensive analysis of NMR data using advanced line shape fitting. Journal of Biomolecular NMR. 69, 93-99 (2017).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods in Enzymology. 339, 204-238 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. Journal of the American Chemical Society. 123, 11341-11352 (2001).
- Carver, J. P., Richards, R. E. A general two-site solution for the chemical exchange produced dependence of T2 upon the Carr-Purcell pulse separation. Journal of Magnetic Resonance. 6 (1), 89-105 (1972).
- Egner, T. K., et al. Surface Contrast' NMR Reveals Non-innocent Role of Support in Pd/CeO2 Catalyzed Phenol Hydrogenation. ChemCatChem. 12 (6), 4160-4166 (2020).
- Egner, T. K., Naik, P., Nelson, N. C., Slowing, I. I., Venditti, V. Mechanistic Insight into Nanoparticle Surface Adsorption by Solution NMR Spectroscopy in an Aqueous Gel. Angewandte Chemie (International Edition in English). 56, 9802-9806 (2017).
- Tugarinov, V., Libich, D. S., Meyer, V., Roche, J., Clore, G. M. The energetics of a three-state protein folding system probed by high-pressure relaxation dispersion NMR spectroscopy. Angewandte Chemie (International Edition in English). 54, 11157-11161 (2015).
- Korzhnev, D. M., Kloiber, K., Kanelis, V., Tugarinov, V., Kay, L. E. Probing slow dynamics in high molecular weight proteins by methyl-TROSY NMR spectroscopy: application to a 723-residue enzyme. Journal of the American Chemical Society. 126 (12), 3964-3973 (2004).
- Mayzel, M., Ahlner, A., Lundstrom, P., Orekhov, V. Y. Measurement of protein backbone (13)CO and (15)N relaxation dispersion at high resolution. Journal of Biomolecular NMR. 69, 1-12 (2017).
- Pritchard, R. B., Hansen, D. F. Characterising side chains in large proteins by protonless (13)C-detected NMR spectroscopy. Nature Communications. 10, 1747 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유