Method Article
عزل النوى من كلية الفأر البالغة لتسلسل الحمض النووي الريبي أحادي النواة
* These authors contributed equally
In This Article
Summary
هنا ، نقدم بروتوكولا لعزل النوى عالية الجودة من كلى الفئران المجمدة التي تعمل على تحسين تمثيل أنواع خلايا الكلى النخاعية وتجنب آثار التعبير الجيني من تفكك الأنسجة الأنزيمية.
Abstract
تنظم الكليتان العمليات الحيوية المتنوعة مثل الماء والكهارل والاتزان الداخلي الحمضي القاعدي. يتم تنفيذ الوظائف الفسيولوجية للكلية من خلال أنواع متعددة من الخلايا مرتبة في بنية معقدة عبر المحور القشري النخاعي للعضو. أدت التطورات الحديثة في علم النسخ أحادي الخلية إلى تسريع فهم التعبير الجيني الخاص بنوع الخلية في فسيولوجيا الكلى والمرض. ومع ذلك ، فإن بروتوكولات تفكك الأنسجة القائمة على الإنزيم ، والتي تستخدم بشكل متكرر لتسلسل الحمض النووي الريبي أحادي الخلية (scRNA-seq) ، تتطلب في الغالب أنسجة جديدة (غير مؤرشفة) ، وتقدم استجابات إجهاد النسخ ، وتفضل اختيار أنواع الخلايا الوفيرة من قشرة الكلى مما يؤدي إلى نقص تمثيل خلايا النخاع.
هنا ، نقدم بروتوكولا يتجنب هذه المشاكل. يعتمد البروتوكول على عزل النوى عند 4 درجات مئوية من أنسجة الكلى المجمدة. يتم عزل النوى من قطعة مركزية من كلية الفأر تتكون من القشرة والنخاع الخارجي والنخاع الداخلي. هذا يقلل من التمثيل المفرط للخلايا القشرية النموذجية لعينات الكلى الكاملة لصالح الخلايا النخاعية بحيث تمثل البيانات المحور القشري النخاعي بأكمله بوفرة كافية. البروتوكول بسيط وسريع وقابل للتكيف ويوفر خطوة نحو توحيد النسخ أحادي النوى في أبحاث الكلى.
Introduction
تعرض الكلى بنية أنسجة معقدة للغاية. وهي تتكون من أجزاء متميزة وظيفيا وتشريحيا على طول محور قشري ووظائف بيولوجية وسيطة ، مثل تنظيم حجم السائل خارج الخلية ، أو توازن الكهارل ، أو التوازن الحمضيالقاعدي 1.
مكنت التطورات في علم النسخ أحادي الخلية من التوصيف المتعمق للأنسجة المعقدة وتسريع فهم التعبير الجيني الخاص بنوع الجزء والخلية في فسيولوجيا الكلى والتطور والمرض2،3،4.
ومع ذلك ، فإن بروتوكولات التفكك القائمة على الإنزيم والتي يتم استخدامها بشكل متكرر ل scRNA-seq تظهر العديد من العيوب والقيود. اعتمادا على البروتوكول ، فإنها تولد استجابات الإجهاد النسخي وتحيز تفكك الأنسجة نحو أنواع الخلايا القشرية الأسهل في الانفصال 5,6. على الرغم من أن البروتوكولات التي تستخدم البروتياز النشط على البارد للكلى الجنينية قادرة على التخفيف من التغيرات النسخية المرتبطة بالإجهاد ، إلا أنها تفشل في التغلب على تحيز التفكك تجاه الخلايا القشرية وقد لا تكون قابلة للتكيف بسهولة مع أنواع مختلفة من أنسجة الكلى المريضة7. بالإضافة إلى ذلك ، لا تتوافق مناهج الخلية الواحدة بسهولة مع عينات الأنسجة المجمدة ، مما يحد من تطبيقها في الغالب على الأنسجة الطازجة غير المؤرشفة ، مما يجعل جمع الأنسجة عاملا مقيدا6.
يمكن لتسلسل الحمض النووي الريبي أحادي النوى (snRNA-seq) التحايل على هذه القيود 8,9. هنا ، نقدم بروتوكولا لعزل النوى من شريحة مركزية من أنسجة كلى الفئران البالغة المجمدة (الشكل 1)10. بروتوكولنا بسيط ويوفر نهجا موحدا للحصول على مكتبات تسلسل الحمض النووي الريبي مع تمثيل متوازن لأنواع خلايا الكلى المتنوعة للنماذج التجريبية التي لا تنطوي على تغييرات قوية في الأنسجة الإقليمية. في الحالة الأخيرة ، يمكن أيضا إجراء بروتوكولنا بالكلى الكاملة.
Protocol
أجريت جميع التجارب على الحيوانات وفقا لقانون رعاية الحيوان (TierSchG) ولائحة التجارب لرعاية الحيوان (TierSchVersV) وتم تفويضها من قبل السلطات المحلية ومسؤولي رعاية الحيوان في مؤسستنا (MDC).
1. إعداد الأنسجة
- قم بإعداد صفيحة من 6 آبار تحتوي على 2 مل من 1x محلول ملحي مخزن بالفوسفات (PBS) لكل بئر لكل كلية سيتم الحصول عليها. تحضير لوحة 6 آبار تحتوي على 2 مل من محلول تثبيت الحمض النووي الريبي لكل بئر وكلية. قبل تبريد كلا اللوحين على الجليد.
- القتل الرحيم لذكر C57BL / 6 يبلغ من العمر 3 إلى 6 أشهر. ضع الماوس على صينية تشريح ، وقم بتثبيت الأطراف وتعقيم البطن بنسبة 70٪ من الإيثانول.
- افتح البطن حتى القفص الصدري باستخدام ملقط ومقص. ارفع الأمعاء والأعضاء الأخرى إلى الجانب وقم بإزالة الكلى عن طريق قطع الحالب والشريان الكلوي والوريد بعناية بالمقص. اغسل الكلى في برنامج تلفزيوني معد مسبقا وبارد 1x وقم بإزالة اللفافة الكلوية وأي دهون متبقية من الكلى حتى تتم إزالة جميع الأنسجة البيضاء (الشكل 2 أ).
- ضع الكلى على صفيحة تشريح باردة واستخدم مشرطا حادا أو شفرة حلاقة للحصول على شريحة متوسطة من 1-2 مم. تأكد من أن قطعة الأنسجة تحتوي على محور القشرية النخاعي بالكامل (الشكل 2 ب). استخدم مقص التشريح المجهري والملقط لتقليم القشرة بعناية من جوانب القطعة المركزية. داخل قطعة الأنسجة المشرحة ، يجب أن تكون الأجزاء الثلاثة القشرة والنخاع الخارجي والنخاع الداخلي مرئية بوضوح (الشكل 2C).
ملاحظة: يجب ألا يتجاوز سمك الشريحة 2 مم أو وزن 20 مجم لضمان كميات عازلة كافية لتحلل الأنسجة الفعال ومن أجل تقليل الحمض النووي الريبي في الخلفية المحيطة في مكتبات cDNA. يهدر الحمض النووي الريبي المحيط قدرة التسلسل ، لأنه لا يرتبط بنواة واحدة. - نقل قطعة الكلى إلى محلول تثبيت الحمض النووي الريبي المعد مسبقا واحتضانها لمدة 24 ساعة عند 4 درجات مئوية لتجنب تدهور الحمض النووي الريبي. بعد 24 ساعة ، قم بإزالة محلول تثبيت الحمض النووي الريبي وتخزين الأنسجة في -80 درجة مئوية حتى الاستخدام مرة أخرى. قم بإزالة المحلول الزائد بعناية باستخدام المناديل الورقية.
2. عزل النوى
- خطوات التنظيف والتحضير
- نظف أسطح الطاولة والماصات باستخدام 70٪ من الإيثانول ومحلول إزالة التلوث RNase.
- قم بتنظيف أنبوب مطحنة الأنسجة ذو القاع المستدير سعة 2 مل ومطابقة المدقة A و B بمحلول إزالة التلوث RNase ، متبوعا بنسبة 70٪ من الإيثانول والماء الخالي من RNase (1 أنبوب مطحنة ومجموعة مدقة لكل عينة). دعها تجف تماما.
- قم بتبريد جهاز الطرد المركزي مسبقا إلى 4 درجات مئوية.
- قم بتسمية وتبريد ثلاثة أنابيب تجميع سعة 15 مل ، وأنبوب تجميع سعة 1.5 مل ، وأنبوب تجميع لفرز الخلايا المنشط بالفلورة (FACS) سعة 5 مل ، وأنبوب مطحنة جافة لكل عينة على الجليد.
- إعداد العازلة
- قم بتسخين محلول مرق مركب ريبونوكليوسيد وفاناديل إلى 65 درجة مئوية حتى يعاد تشكيله إلى محلول شفاف أخضر-أسود وفقا لتعليمات الشركة المصنعة. 11
- تحضير 1x PBS يحتوي على 4٪ ألبومين مصل بقري (BSA) كما هو موضح في الجدول 1 أ. بالإضافة إلى ذلك ، قم بإعداد 1x PBS مع 0.04٪ BSA (الجدول 1B). قم بتصفية كلا المحلولين باستخدام مرشح حقنة غشائي خال من المواد الخافضة للتوتر السطحي (SFCA) 0.2 ميكرومتر واحتفظ به على الثلج حتى استخدامه مرة أخرى.
- تحضير المخزن المؤقت لتحلل النوى 1 (NLB1 ، الجدول 1C). أضف 4 مل من المخزن المؤقت لتحلل EZ لمحلول تحلل النوى 2 (NLB2 ، الجدول 1D) و 2 مل من 0.04٪ BSA / PBS للمخزن المؤقت لتعليق النوى (NSB ، الجدول 1E) إلى أنابيب 15 مل. أضف محلول مثبط RNase إلى NLB2 و NSB مباشرة قبل الاستخدام كما هو موضح أدناه في البروتوكول. الحفاظ على الجليد حتى استخدام مرة أخرى.
- تحضير محلول تحلل EZ مع 10٪ سكروز (محلول تدرج السكروز ، الجدول 1F). امزج جيدا وقم بتصفية المخزن المؤقت في أنبوب جديد سعة 15 مل باستخدام مرشح حقنة غشائي SFCA 0.2 ميكرومتر. الحفاظ على الجليد حتى استخدام مرة أخرى.
- تجانس الأنسجة وتحلل الخلايا
ملاحظة: من أجل تقليل تدهور الحمض النووي الريبي ، يتم تنفيذ جميع الخطوات على الجليد. يجب تبريد أنبوب المطحنة وطبق بتري وجميع المخازن المؤقتة. تتم جميع خطوات إعادة التعليق عن طريق سحب تعليق النوى بعناية. لا تقم بدوامة العينة لتجنب قوى القص وتلف النوى.- خذ قطعة الكلى المجمدة وانقلها إلى طبق بتري من البوليسترين 60 مم على ثلج يحتوي على 1 مل من NLB1.
- فرم الأنسجة جيدا باستخدام شفرة حلاقة أو مشرط (الشكل 3 أ).
- اقطع طرف طرف ماصة سعة 1 مل وانقل المنديل المفروم والمخزن المؤقت إلى أنبوب المطحنة. تأكد من نقل جميع قطع المناديل. اغسل طبق بتري 5-10 مرات باستخدام المخزن المؤقت ، إذا لزم الأمر.
- تجانس التعليق على الجليد عن طريق تحريك المدقة A ببطء ، 25x لأعلى ولأسفل في أنبوب المطحنة. تجنب فقاعات الهواء الناتجة عن الحركة السريعة (الشكل 3 ب).
- مرر التجانس من خلال مصفاة 100 ميكرومتر في أنبوب تجميع 15 مل مبرد مسبقا واغسل الفلتر ب 1 مل آخر من NLB1.
- اغسل أنبوب المطحنة بمحلول تحلل نوى EZ البارد وتخلص من المخزن المؤقت.
- انقل المجانس مرة أخرى إلى أنبوب المطحنة وقم بتجانس التعليق على الجليد عن طريق تحريك المدقة B ببطء ، 15x لأعلى ولأسفل في أنبوب المطحنة. تجنب فقاعات الهواء الناتجة عن الحركة السريعة (الشكل 3C).
- انقل التجانس إلى أنبوب تجميع سعة 15 مل مبرد مسبقا. اغسل أنبوب المطحنة ب 2 مل أخرى من NLB1 وتأكد من نقل جميع شظايا الأنسجة إلى أنبوب التجميع. احتضان التجانس (الحجم الكلي 4 مل) لمدة 5 دقائق على الجليد لتحلل الخلايا.
- تنقية النوى
- مرر التجانس من خلال مصفاة 40 ميكرومتر في أنبوب تجميع 15 مل مبرد مسبقا. قم بتدوير أنبوب التجميع لمدة 5 دقائق عند 500 × جم عند 4 درجات مئوية في جهاز طرد مركزي مع دوار دلو متأرجح. في غضون ذلك ، أضف محلول مثبط RNase إلى NLB2 (الجدول 1D).
- قم بإزالة المادة الطافية دون إزعاج الحبيبات. أعد تعليق الحبيبات بعناية في 4 مل من NLB2.
- قم بتغطية التعليق بعناية بوسادة سعة 1 مل من المخزن المؤقت المتدرج للسكروز. جهاز طرد مركزي عند 500 × جم لمدة 5 دقائق عند 4 درجات مئوية في جهاز طرد مركزي مع دوار دلو متأرجح. في غضون ذلك ، أضف محلول مثبط RNase إلى NSB (الجدول 1E).
- بعد الطرد المركزي ، قم بإزالة أنبوب التجميع برفق من جهاز الطرد المركزي واحرص على عدم إزعاج الطبقتين عند التعامل مع أنبوب التجميع. يمكن رؤية حطام الخلية بين الطبقتين. قم بإزالة المادة الطافية بعناية بدءا من الحطام. قم بإزالة المادة الطافية المتبقية دون إزعاج حبيبات النوى وأعد تعليق الحبيبات بعناية في 1 مل من NSB.
ملاحظة: يعتمد حجم إعادة التعليق على كمية الأنسجة المستخدمة للعزل وحجم الحبيبات المكتسبة بعد خطوة الطرد المركزي الأخيرة. قد يلزم تكييف الحجم مع العدد المتوقع للنوى. - مرر المجانسة من خلال مصفاة 20 ميكرومتر في أنبوب تجميع FACS المبرد مسبقا سعة 5 مل.
3. فرز النوى
- أضف 20 ميكرولتر من 4 ′ ، 6-دياميدينو -2-فينيليندول (DAPI) لكل مل من NSB إلى تركيز نهائي قدره 2 ميكرومتر إلى التجانس في أنبوب تجميع FACS واخلطه بعناية. احتضان لمدة 5 دقائق على الجليد.
- قم بإعداد أنبوب التجميع المبرد مسبقا سعة 1.5 مل مع 20 ميكرولتر من 4٪ BSA / 1x PBS وأضف محلول مثبط RNase 0.5 ميكرولتر إلى تركيز نهائي قدره 1 وحدة / ميكرولتر. انتقل فورا إلى الفرز.
- فرز النوى باستخدام فارز الخلية.
- امزج معلق النوى لفترة وجيزة قبل إدخال أنبوب تجميع FACS في آلة الفرز.
- قم بتعيين البوابة الأولى P1 بناء على التشتت الأمامي (FSC) والتشتت الجانبي (SSC) لاستبعاد الحطام والركام (الشكل 4A).
- لاستبعاد النوى والمضاعفات الفارغة أو التالفة ، قم بتعيين بوابة لاحقة بناء على منطقة DAPI مقابل ارتفاع DAPI (DAPI-A مقابل DAPI-H) (الشكل 4B).
- قم بفرز النوى المفردة في أنبوب تجميع سعة 1.5 مل يحتوي على 4٪ BSA / 1x PBS مع محلول مثبط RNase 1 U / μL محضر في 3.2.
4. مراقبة الجودة
- قم بقياس تركيز النوى النهائي تحت مجهر مضان أو في غرفة عد آلية في عددين مستقلين على الأقل وتقييم جودة التعليق (الشكل 5).
ملاحظة: التركيزات المثلى هي بين 700 - 1200 نواة / ميكرولتر. قد تكون تركيزات الخلايا المنخفضة مثل 700 نواة / ميكرولتر مفضلة لأن مكتبات cDNA الناتجة تحتوي على RNA خلفية أقل محيطا (نسخ غير مرتبطة بنوى فردية). - احسب الحجم المطلوب لتعليق النوى من أجل الاسترداد المطلوب للنوى المفردة المتتابعة. من أجل تجنب تراكم النوى وتدهور الحمض النووي الريبي ، انتقل فورا إلى إعداد المكتبة.
النتائج
لتحديد أداء بروتوكولنا ، استخدمنا 10x Genomics Chromium Single Cell 3 'Gene Expression Kit v3.1 لإعداد المكتبة وقمنا بتحليل بيانات snRNA-seq باستخدام حزمة Seurat12,13.
يوضح الشكل 6 النتائج من مكتبة snRNA-seq التمثيلية. لتقييم جودة نواتنا ، قمنا برسم عدد الجينات مقابل عدد النسخ (المحددة بواسطة المعرفات الجزيئية الفريدة (UMIs)) الملونة بجزء من قراءات الميتوكوندريا (الشكل 6 أ). تظهر النوى ذات النوعية الجيدة عموما أعدادا أعلى من القراءات ، وتربط بين UMI وأرقام الجينات ، وكسور قراءة الميتوكوندريا المنخفضة.
للتحليل اللاحق ، تم استبعاد النوى التي تحتوي على أقل من 500 أو أكثر من 4000 جين محسوب ، أو أكثر من 5٪ من الحمض النووي الريبي للميتوكوندريا (ن = 828). تم تضمين الجينات المعبر عنها فقط في ثلاث نوى على الأقل. اكتشفنا حوالي 20000 جين في المجموع في النوى ال 6000 المتبقية مع 1600 جين متوسط و 2800 UMIs متوسطة لكل نواة (الشكل 6B).
استند التجميع إلى جينات متغيرة للغاية. حددنا ما مجموعه 18 مجموعة. تم شرح هويات الخلايا بناء على جينات علامة معروفة (غير معروضة). تم تلخيص المجموعات الفرعية لنوع واحد من الخلايا في مجموعة واحدة مما أدى إلى ما مجموعه 11 نوعا متميزا من الخلايا: الخلايا الدموية ، النبيبات القريبة (PT) ، الأطراف الرقيقة (tL) ، الطرف الصاعد السميك (TAL) ، النبيبات الملتوية البعيدة (DCT) ، الأنابيب المتصلة (CNT) ، تجميع القناة الرئيسية والخلايا المقحمة (CD-PC ، A-IC ، B-IC) ، ظهارة النخاع العميق للحوض (DMEP) والبطانة. تم تصور أنماط التعبير الجيني للعلامات المخصبة بالكتلة في مخطط نقطي (الشكل 6C) ومجموعات من نوع الخلية في مخطط تضمين عشوائي عشوائي موزع على t (t-SNE) (الشكل 6D).
لتقييم توزيعات نوع الخلية في عينتنا ، تم حساب النسبة المئوية لكل نوع خلية (الشكل 6E) واستخدامها لتحديد نسبة PT إلى TAL. يقع PT بشكل رئيسي في قشرة الكلى وغالبا ما يتم تمثيله بشكل مفرط في مجموعات بيانات الخلية المفردة للكلى حيث يسهل فصل خلايا PT ووفيرة للغاية في عينات الكلى الكاملة. من ناحية أخرى ، يمتد TAL عبر النخاع الخارجيبأكمله 14. وبالتالي ، فإن نسبة كسور PT و TAL تمثل مقياسا جيدا لإثراء أنواع الخلايا النخاعية في مجموعة بيانات خلية واحدة في الكلى. بشكل عام ، تراوحت نسبة PT / TAL في مجموعات بيانات الكلى الكاملة أحادية الخلية من 8 (بيانات غير منشورة من أنسجة الكلى الكاملة المعالجة بالبروتياز البارد) إلى 45 للأنسجة المنفصلة إنزيميا10،14،15. في مجموعة بيانات snRNA-seq المعروضة هنا ، تمكنا من الوصول إلى نسبة PT / TAL تبلغ 2. توضح هذه النتيجة أن إزالة القشرة الزائدة أثناء تشريح الأنسجة جنبا إلى جنب مع snRNA-seq يؤدي إلى تحسين تمثيل نوع خلايا الكلى بشكل لافت للنظر.
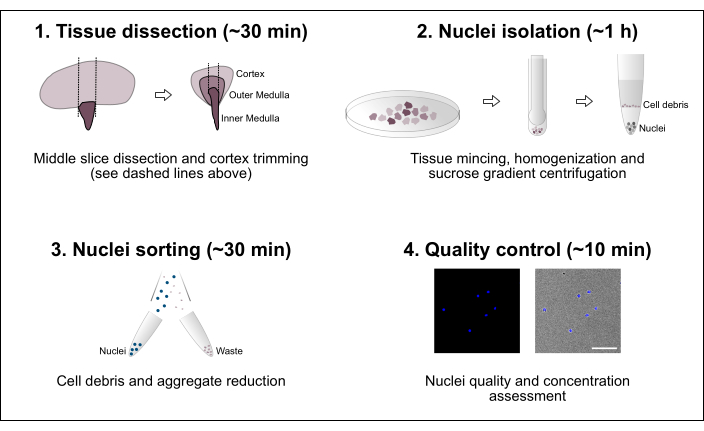
الشكل 1: نظرة عامة تخطيطية على سير العمل. يتكون البروتوكول من أربع خطوات رئيسية تشمل تشريح الأنسجة يليها عزل النوى وفرز النوى وتقييم النقاء والتركيز النهائي. شريط المقياس = 100 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الشكل.
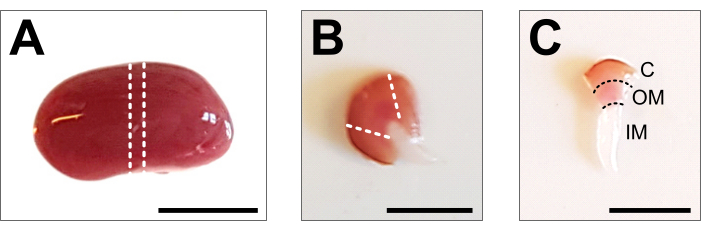
الشكل 2: تشريح الكلى وإعداد الأنسجة. أ: صورة تمثيلية للكلية الكاملة المقطعة. تشير الخطوط المنقطة إلى التخفيضات المطلوبة للحصول على شريحة متوسطة من 1-2 مم مع تمثيل لجميع أنواع الخلايا الكلوية. (ب) صورة تمثيلية للشريحة الوسطى التي تم الحصول عليها. تشير الخطوط المنقطة إلى تخفيضات تقليم القشرة من الجانب. (ج) صورة تمثيلية لقطعة الكلى المركزية مع قشرة مشذبة. القشرة (C) والنخاع الخارجي (OM) والنخاع الداخلي (IM) مرئية بوضوح. شريط المقياس = 500 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3: تجانس الأنسجة وتنقية النوى. أ: صورة تمثيلية توضح أنسجة الكلى المفرومة بدرجة كافية. شريط المقياس = 500 ميكرومتر. (ب) التجانس بعد خطوة التجانس الأولى (25 ضربة مع أنبوب مطحنة مدقة A ، 2 مل). (ج) التجانس بعد خطوة التجانس الثانية (15 ضربة مع المدقة B ، أنبوب مطحنة 2 مل). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
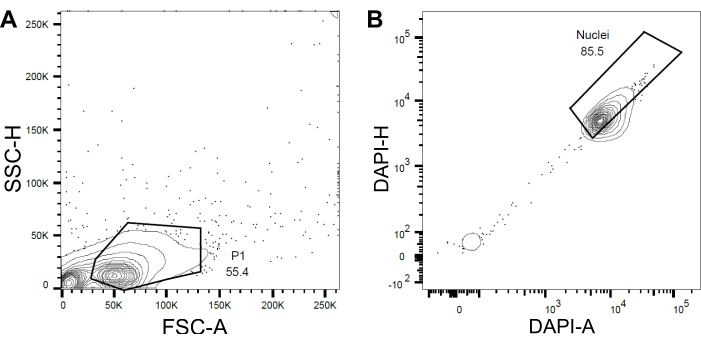
الشكل 4: استراتيجية البوابات لفرز النوى. (أ ) تم تعيين بوابة أولى P1 على أساس التشتت الأمامي (FSC) مقابل التشتت الجانبي (SSC) لاستبعاد الحطام والركام. (ب ) بوابة لاحقة تعتمد على منطقة دابي (DAPI-A) مقابل ارتفاع دابي (DAPI-H) تستبعد النوى والمضاعفات الفارغة أو التالفة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
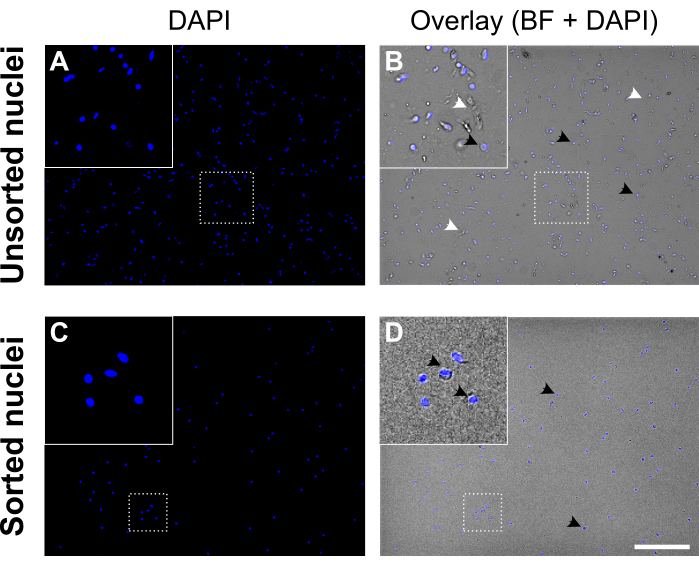
الشكل 5: معلق النوى قبل فرز النوى وبعده. (أ، ج) نوى ملطخة ب DAPI (زرقاء). (ب، د) تراكب قناة DAPI وبرايتفيلد (BF). قبل الفرز (اللوحة العلوية) ، يحتوي تعليق النوى على حطام الخلايا والركام (المسمى برؤوس أسهم بيضاء). بعد الفرز (اللوحة السفلية) ، يبدو تعليق النوى أكثر نظافة. يتم تمييز أمثلة النوى الملطخة ب DAPI برؤوس أسهم سوداء. تظهر النوى ذات النوعية الجيدة مستديرة وناعمة مع غشاء سليم ومنفصلة جيدا ، في حين أن النوى ذات النوعية الرديئة تظهر مجعدة وتظهر فقدان الغشاء النووي. شريط المقياس = 250 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الشكل.
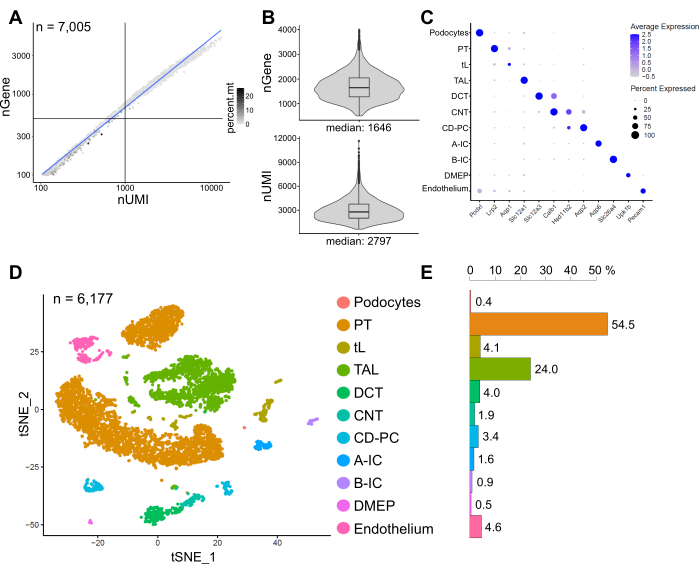
الشكل 6: مراقبة الجودة وتحليل مجموعة بيانات snRNA-seq التمثيلية. (أ) عدد الجينات (nGene) المرسومة مقابل عدد المعرفات الجزيئية الفريدة (nUMI) الملونة بجزء قراءات الميتوكوندريا (percent.mt). تتوافق النوى منخفضة الجودة مع الربع السفلي الأيسر من المؤامرة (n = 828) وتم استبعادها من التحليل اللاحق. (ب) توزيع ومتوسط nGene و nUMI المكتشف لكل نواة في مجموعة بيانات snRNA-seq ، والتي تمثل 6177 نواة (> 500 جين). تم تسلسل المكتبات إلى عمق متوسط ~ 8200 قراءة معينة لكل نواة. (ج) مخطط نقطي يوضح أنماط التعبير الجيني للعلامات العنقودية المخصبة (المحور السيني) لأنواع الخلايا الفردية (المحور ص). يتوافق حجم النقطة مع نسبة الخلايا التي تعبر عن الجين المشار إليه. اللون يتوافق مع متوسط التعبير. (د) مخطط تضمين الجار العشوائي الموزع على شكل حرف T (t-SNE) لأنواع الخلايا المحددة. (ه) توزيع نوع الخلية في مجموعة بيانات snRNA-seq. PT ، النبيب القريب. tL ، طرف رفيع. TAL ، طرف صاعد سميك ، DCT ، نبيب ملتوي بعيد ؛ CNT ، ربط النبيب ؛ CD-PC ، جمع الخلايا الرئيسية للقناة ؛ A-IC ، الخلايا المقحمة من النوع A ؛ B-IC ، الخلايا المقحمة من النوع B ؛ DMEP ، ظهارة النخاع العميق للحوض. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
| الكاشف | التركيز النهائي | الحجم (مل) | ||
| (أ) 4٪ BSA / PBS | ||||
| محلول ملحي مخزن بالفوسفات (PBS) مع 10٪ ألبومين بقري | 4% | 2 | ||
| PBS (محلول ملحي مخزن بالفوسفات) 1X بدون كالسيوم أو مغنيسيوم | - | 3 | ||
| (ب) 0.04٪ BSA / PBS | ||||
| 4٪ BSA / برنامج تلفزيوني | 0.04 % | 0.5 | ||
| PBS (محلول ملحي مخزن بالفوسفات) 1X بدون كالسيوم أو مغنيسيوم | - | 49.5 | ||
| (ج) المخزن المؤقت لتحلل النوى 1 (NLB1) | ||||
| عازلة تحلل EZ النووية | - | 4 | ||
| مثبط RiboLock RNase (40 وحدة / ميكرولتر) | 1 وحدة / ميكرولتر | 0.1 | ||
| مركب ريبونوكليوسيد فاناديل (200 مللي مول) | 10 مللي متر | 0.2 | ||
| (د) المخزن المؤقت لتحلل النوى 2 (NLB2) | ||||
| عازلة تحلل EZ النووية | - | 4 | ||
| مثبط RiboLock RNase (40 وحدة / ميكرولتر) | 1 وحدة / ميكرولتر | 0.1 | ||
| (ه) العازلة المعلقة للنوى (NSB) | ||||
| 0.04٪ BSA / PBS | - | 2 | ||
| مثبط RiboLock RNase (40 وحدة / ميكرولتر) | 1 وحدة / ميكرولتر | 0.05 | ||
| (F) عازلة تدرج السكروز (10٪ سكروز) | ||||
| الوزن 1 غرام من السكروز | ||||
| يذوب في 6 مل من العازلة النووية EZ Lysis | ||||
| املأ ما يصل إلى 10 مل بمحلول تحلل EZ النووي | ||||
| قم بالتصفية من خلال مرشح حقنة 0.2 ميكرومتر في أنبوب جديد | ||||
الجدول 1: وصفات الحل: (أ) تحضير 4٪ BSA / 1x PBS. قم بالتصفية باستخدام مرشح حقنة غشاء SFCA 0.2 ميكرومتر واحفظه على الثلج حتى الاستخدام. (ب) إعداد 0.04٪ BSA / 1 × PBS. قم بالتصفية باستخدام مرشح حقنة غشاء SFCA 0.2 ميكرومتر واحفظه على الثلج حتى الاستخدام. (ج) تحضير محلول تحلل النوى 1 (NLB1). يتم توفير الأحجام المشار إليها لكل عينة. الحفاظ على الجليد حتى الاستخدام. (د) تحضير محلول تحلل النوى 2 (NLB2). يتم توفير الأحجام المشار إليها لكل عينة. أضف مثبط RiboLock RNase إلى NLB2 مباشرة قبل الاستخدام كما هو مذكور في البروتوكول. الحفاظ على الجليد حتى الاستخدام. (ه) تحضير العازل المعلق للنوى (NSB). يتم توفير الأحجام المشار إليها لكل عينة. أضف مثبط RiboLock RNase إلى NSB مباشرة قبل الاستخدام كما هو مذكور في البروتوكول. الحفاظ على الجليد حتى الاستخدام. (و) تحضير عازل متدرج للسكروز. قم بالتصفية باستخدام مرشح حقنة غشاء SFCA 0.2 ميكرومتر واحفظه على الثلج حتى الاستخدام.
Discussion
تعمل النسخ أحادية الخلية على تعزيز فهم التعبير الجيني الخاص بنوع الخلية في فسيولوجيا الكلى والمرض. هنا ، قدمنا طريقة بسيطة وقابلة للتكرار لعزل النوى المفردة عالية الجودة من أنسجة كلى الفئران المجمدة ل snRNA-seq بطريقة موحدة.
بالنسبة إلى snRNA-seq ، من الأهمية بمكان استخدام نوى عالية الجودة كمدخلات لتوليد المكتبة وتجنب تدهور الحمض النووي الريبي أثناء معالجة الأنسجة. لذلك ، فإن حضانة قطع الأنسجة في محلول تثبيت الحمض النووي الريبي مباشرة بعد التشريح أمر ضروري لحماية واستقرار الحمض النووي الريبي الخلوي ويسمح بتخزين العينات عند - 80 درجة مئوية إلى أجل غير مسمى. عند تطبيق هذا البروتوكول على الأنسجة المجمدة دون معالجة محلول تثبيت الحمض النووي الريبي ، مثل المواد الأرشيفية ، يلزم إجراء اختبار تجريبي ، ويجب تقييم جودة الحمض النووي الريبي حيث لاحظنا خسارة كبيرة في سلامة الحمض النووي الريبي في الأنسجة المجمدة المفاجئة دون حضانة مسبقة في محلول تثبيت الحمض النووي الريبي.
بشكل عام ، تعد المعالجة المناسبة للعينات أمرا بالغ الأهمية لتحقيق أقصى قدر من استعادة النوى المفردة السليمة. يجب تنفيذ جميع خطوات إعادة التعليق عن طريق السحب بعناية لتجنب إجهاد القص والأضرار المادية. يجب أن تحتوي المخازن المؤقتة لإعادة تعليق النوى النهائية وفرز النوى على BSA لتجنب فقدان النوى وتجميعها.
تم تحسين أحجام المخزن المؤقت في هذا البروتوكول لعينات الأنسجة الصغيرة جدا (~ 15 ملغ). من الأهمية بمكان ضمان تحلل الخلايا بالكامل والغسيل الكافي لتوليد معلقات عالية الجودة. ستؤدي كتل الأنسجة الكبيرة أو عينات الكلى الكاملة إلى تركيزات مفرطة من النوى تؤدي إلى التكتل والتجميع ، ووفرة عالية من الحمض النووي الريبي المحيط ، وجودة تعليق رديئة بشكل عام. إذا تمت معالجة عينات أكبر أو أنسجة أخرى ، يوصى بشدة بإجراء عمليات تجريبية لتحديد أحجام المخزن المؤقت المثلى للحد الأدنى من مستويات الحمض النووي الريبي المحيطة. يجب فحص جودة وتركيزات النوى والحمض النووي الريبي بعناية لأن التحميل الزائد يؤدي إلى ضعف الأداء بشكل عام.
بالإضافة إلى ذلك ، فإن كميات كبيرة من حطام الخلايا ، مما يتسبب في مستويات عالية من الحمض النووي الريبي المحيط غير المرتبط بنواة واحدة يؤثر سلبا على نتائج التسلسل. إن توضيح تعليق النوى عن طريق الطرد المركزي من خلال وسادة السكروز يخفف من هذه المشكلة إلى حد ما ، ولكنه يمكن أن يؤدي أيضا إلى التحيز في تمثيل نوع الخلية عن طريق الاختيار المضاد مقابل النوى الكثيفة والصغيرة الموجودة ، على سبيل المثال ، في الخلايا المناعية16. إذا كان هذا مصدر قلق ، فيجب حذف تدرج السكروز. على النقيض من ذلك ، وجدنا أن قياس التدفق الخلوي القائم على تلطيخ DAPI كان أمرا بالغ الأهمية لتقليل كمية حطام الخلايا من أجل إنتاج تعليق أحادي النواة عالي الجودة.
يتمتع عزل النوى المفردة بمزايا كبيرة عند مقارنته بنهج الخليةالواحدة 8. إنه متوافق مع الأنسجة المجمدة بشكل صحيح ، مما يجعل جمع الأنسجة أكثر مرونة ، ويتحايل على الحاجة إلى تفكك الأنسجة القائم على الإنزيم ، والذي يمكن أن يؤدي إلى استجابات الإجهاد النسخي 6,17. علاوة على ذلك ، فإنه يتغلب على تحيز التفكك الذي يفضل اختيار أنواع الخلايا القابلة للانفصال بسهولة في القشرة الكلوية ، مما قد يؤدي إلى نقص تمثيل أنواع الخلايا النخاعية في بعض الأساليب القائمة على الإنزيم5،6،10.
إن استخدام قطعة الكلى المركزية بدلا من أنسجة الكلى الكاملة يوفر الموارد ويصحح التمثيل المفرط لأنواع الخلايا الوفيرة كما هو موضح سابقا10. ومع ذلك ، اعتمادا على نموذج الماوس أو النمط الظاهري الذي تم فحصه ، قد يكون من المفيد استخدام عينات الكلى الكاملة بدلا من شريحة متوسطة واحدة. قد تكون عينات الكلى الكاملة أكثر تمثيلا لنسب الخلايا الحقيقية ، أو التغيرات التي تحدث في الكلية بأكملها ، في حين أثبتت الشريحة الوسطى المشذبة أنها مفيدة للأنماط الظاهرية النخاعية أو عندما تكون مادة العينة محدودة. لذلك ، فإن هذا القرار خاص جدا بالمستخدم ويجب النظر فيه بعناية.
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
نشكر منصة علم الجينوم العلمية في مركز ماكس ديلبروك للطب الجزيئي في جمعية هيلمهولتز ، برلين على الدعم الفني.
تم دعم JL و KMSO من قبل مجموعة التدريب البحثي GRK 2318 التابعة لمؤسسة الأبحاث الألمانية (DFG) ووحدة الأبحاث FOR 2841. تم دعم KMSO من خلال منحة البحوث التعاونية 1365. تم دعم AB بتمويل من جائزة جوتفريد فيلهلم لايبنتز من DFG الممنوحة إلى NR.
Materials
| Name | Company | Catalog Number | Comments |
| Cell sorter | - | - | For fluorescence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. |
| Centrifuge 5810 R | Eppendorf | 5811000015 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | Needs to contain DAPI light cube to count nuclei. Alternatively, nuclei can be counted manually under fluorescence microscope. |
| 4′,6-Diamidino-2-phenyl-indol-dihydrochlorid (DAPI) | Biotrend | 40043/b | Stock solution prepared with a concentration of 100 µM. Used for nuclei staining in a final concentration of 2 µM. |
| D(+)-Sucrose ≥99.9%, ultrapure DNAse-, RNAse-free | VWR | 0335-500G | |
| DNA LoBind Microcentrifuge Tubes (1.5 mL) | Eppendorf | 22431021 | |
| Ethanol, 70 % | - | - | |
| FACS tubes | pluriSelect | 43-10100-46 | |
| KIMBLE Dounce tissue grinder set 2 mL complete | Sigma-Aldrich | D8938-1SET | |
| Minisart Syringe Filters 0.2 µm | Sartorius | 16534-GUK | |
| Nuclease-free Water | Invitrogen | AM9937 | |
| Nuclei EZ Prep Nuclei Isolation Kit | Sigma-Aldrich | NUC-101 | Nuclei EZ Lysis Buffer (Product No. N3408) needed for buffer preparation. |
| PBS (Phosphate-Buffered Saline) 1X without calcium or magnesium | Corning | 21-040-CV | |
| Petri dishes, polystyrene 60 mm | Sigma-Aldrich | P5481 | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin | Sigma-Aldrich | SRE0036 | |
| pluriStrainer Mini 100 µm | pluriSelect | 43-10100-46 | |
| pluriStrainer Mini 20 µm | pluriSelect | 43-10020-40 | |
| pluriStrainer Mini 40 µm | pluriSelect | 43-10040-40 | |
| Polystyrene Centrifuge Tube (15 mL) | Falcon | 352099 | |
| Razor blades | - | - | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher | EO0384 | |
| Ribonucleoside-vanadyl complex | New England Biolabs | S1402S | Follow manufacturer's instructions (https://international.neb.com/products/s1402-ribonucleoside-vanadyl-complex#Product%20Information). Upon use the 200 mM stock solution is reconstituted to a green-black clear solution by incubating at 65 °C. |
| RNAlater Stabilization Solution | Invitrogen | AM7020 | |
| RNase AWAY | Fisher Scientific | 11952385 |
References
- Thomas, R. S. Kidney modeling and systems physiology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1 (2), 172-190 (2009).
- Potter, S. S. Single-cell RNA sequencing for the study of development, physiology and disease. Nature Reviews Nephrology. 14 (8), 479-492 (2018).
- Park, J., Liu, C. L., Kim, J., Susztak, K. Understanding the kidney one cell at a time. Kidney International. 96 (4), 862-870 (2019).
- Clark, A. R., Greka, A. The power of one: advances in single-cell genomics in the kidney. Nature Reviews Nephrology. 16 (2), 73-74 (2020).
- Lake, B. B., et al. A single-nucleus RNA-sequencing pipeline to decipher the molecular anatomy and pathophysiology of human kidneys. Nature Communications. 10 (1), 2832(2019).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 30 (1), 23-32 (2019).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144 (19), 3625-3632 (2017).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Muto, Y., et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nature Communications. 12 (1), 2190(2021).
- Hinze, C., et al. Kidney single-cell transcriptomes predict spatial corticomedullary gene expression and tissue osmolality gradients. Journal of the American Society of Nephrology. 32 (2), 291(2021).
- Berger, S. L. Isolation of cytoplasmic RNA: ribonucleoside-vanadyl complexes. Methods in Enzymology. 152, 227-234 (1987).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411-420 (2018).
- Stuart, T., et al. Comprehensive Integration of single-cell data. Cell. 177 (7), 1888-1902 (2019).
- Park, J., et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease. Science. 360 (6390), 758-763 (2018).
- Kirita, Y., Wu, H., Uchimura, K., Wilson, P. C., Humphreys, B. D. Cell profiling of mouse acute kidney injury reveals conserved cellular responses to injury. Proceedings of the National Academy of Sciences of the United States of America. 117 (27), 15874-15883 (2020).
- Schneeberger, S., et al. The neuroinflammatory interleukin-12 signaling pathway drives Alzheimer's disease-like pathology by perturbing oligodendrocyte survival and neuronal homeostasis. bioRxiv. , 441313(2021).
- Nguyen, Q. H., Pervolarakis, N., Nee, K., Kessenbrock, K. experimental considerations for single-cell RNA sequencing approaches. Frontiers in Cell and Developmental Biology. 6, 108(2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved