Method Article
Isolement de noyaux à partir de reins de souris adultes pour le séquençage de l’ARN mononucléaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous présentons un protocole pour isoler des noyaux de haute qualité à partir de reins de souris congelés qui améliorent la représentation des types de cellules rénales médullaires et évitent les artefacts d’expression génique de la dissociation des tissus enzymatiques.
Résumé
Les reins régulent divers processus biologiques tels que l’eau, l’électrolyte et l’homéostasie acido-basique. Les fonctions physiologiques du rein sont exécutées par plusieurs types de cellules disposées dans une architecture complexe à travers l’axe corticomédullaire de l’organe. Les progrès récents de la transcriptomique unicellulaire ont accéléré la compréhension de l’expression génique spécifique au type cellulaire dans la physiologie et les maladies rénales. Cependant, les protocoles de dissociation tissulaire à base d’enzymes, qui sont fréquemment utilisés pour le séquençage de l’ARN unicellulaire (scRNA-seq), nécessitent principalement des tissus frais (non archivés), introduisent des réponses de stress transcriptionnel et favorisent la sélection de types de cellules abondantes du cortex rénal, ce qui entraîne une sous-représentation des cellules de la moelle.
Ici, nous présentons un protocole qui évite ces problèmes. Le protocole est basé sur l’isolement des noyaux à 4 °C à partir de tissus rénaux congelés. Les noyaux sont isolés d’une pièce centrale du rein de souris composée du cortex, de la moelle externe et de la moelle interne. Cela réduit la surreprésentation des cellules corticales typiques des échantillons de reins entiers au profit des cellules médullaires, de sorte que les données représenteront l’ensemble de l’axe corticomédullaire en abondance suffisante. Le protocole est simple, rapide et adaptable et constitue une étape vers la normalisation de la transcriptomique à noyau unique dans la recherche rénale.
Introduction
Les reins présentent une architecture tissulaire très complexe. Ils sont constitués de segments fonctionnellement et anatomiquement distincts le long d’un axe corticomédullaire et interviennent dans les fonctions biologiques, telles que la régulation du volume de liquide extracellulaire, l’équilibre électrolytique ou l’homéostasie acido-basique1.
Les progrès de la transcriptomique unicellulaire ont permis la caractérisation approfondie de tissus complexes et accéléré la compréhension de l’expression génique spécifique au segment et au type cellulaire dans la physiologie, le développement et la maladie rénales 2,3,4.
Cependant, les protocoles de dissociation enzymatique fréquemment utilisés pour le séquençage de l’ARNc présentent plusieurs inconvénients et contraintes. Selon le protocole, ils génèrent des réponses de stress transcriptionnel et un biais de dissociation tissulaire vers des types de cellules corticales plus faciles à dissocier 5,6. Bien que les protocoles utilisant des protéases actives à froid pour les reins embryonnaires soient en mesure d’atténuer les altérations transcriptionnelles liées au stress, ils ne parviennent pas à surmonter le biais de dissociation vers les cellules corticales et pourraient ne pas être facilement adaptables à différents types de tissus rénaux malades7. De plus, les approches unicellulaires ne sont pas facilement compatibles avec les échantillons de tissus congelés, ce qui limite leur application principalement aux tissus frais non archivés, ce qui fait de la collecte de tissus un facteurde restriction 6.
Le séquençage de l’ARN mononucléiforme (snRNA-seq) permet de contourner ces limitations 8,9. Nous présentons ici un protocole d’isolement des noyaux à partir d’une tranche centrale de tissu rénal de souris adulte congelé (Figure 1)10. Notre protocole est simple et fournit une approche standardisée pour obtenir des bibliothèques de séquençage d’ARN avec une représentation équilibrée de divers types de cellules rénales pour des modèles expérimentaux qui n’impliquent pas de forts changements tissulaires régionaux. Dans ce dernier cas, notre protocole peut également être effectué avec des reins entiers.
Protocole
Toutes les expérimentations sur les animaux ont été menées conformément à la loi sur le bien-être des animaux (TierSchG) et au règlement sur le bien-être des animaux expérimentaux (TierSchVersV) et ont été autorisées par les autorités locales et les responsables du bien-être animal de notre institution (MDC).
1. Préparation des tissus
- Préparer une plaque de 6 puits contenant 2 mL de 1x solution saline tamponnée au phosphate (PBS) par puits pour chaque rein qui sera obtenu. Préparer une plaque de 6 puits contenant 2 mL de solution de stabilisation de l’ARN par puits et par rein. Pré-refroidir les deux assiettes sur de la glace.
- Euthanasier une souris C57BL/6 mâle âgée de 3 à 6 mois. Placez la souris sur un plateau de dissection, épinglez les extrémités et stérilisez l’abdomen avec de l’éthanol à 70%.
- Ouvrez l’abdomen jusqu’à la cage thoracique à l’aide de forceps et de ciseaux. Soulevez l’intestin et les autres organes sur le côté et retirez les reins en coupant soigneusement l’uretère, l’artère rénale et la veine avec un ciseau. Lavez le rein dans le 1x PBS glacé préalablement préparé et retirez le fascia rénal et toute graisse restante du rein jusqu’à ce que tous les tissus blancs soient enlevés (Figure 2A).
- Placez le rein sur une plaque de dissection à froid et utilisez un scalpel tranchant ou une lame de rasoir pour obtenir une tranche centrale de 1-2 mm. Assurez-vous que le morceau de tissu contient tout l’axe corticomédullaire (figure 2B). Utilisez des ciseaux de microdissection et des pinces pour couper soigneusement le cortex des côtés de la pièce centrale. Dans la pièce de tissu disséquée, les trois segments du cortex, de la moelle externe et de la moelle interne doivent être clairement visibles (Figure 2C).
REMARQUE : La tranche ne doit pas dépasser une épaisseur de 2 mm ou un poids de 20 mg pour assurer des quantités tampons suffisantes pour une lyse tissulaire efficace et pour minimiser l’ARN de fond ambiant dans les banques d’ADNc. L’ARN ambiant gaspille la capacité de séquence, car il n’est pas associé à des noyaux uniques. - Transférer le morceau de rein dans la solution de stabilisation de l’ARN préalablement préparée et incuber pendant 24 h à 4 °C pour éviter la dégradation de l’ARN. Après 24 h, retirer la solution de stabilisation de l’ARN et conserver le tissu à -80 °C jusqu’à nouvelle utilisation. Retirez délicatement l’excès de solution avec du papier de soie.
2. Isolement des noyaux
- Étapes de nettoyage et de préparation
- Nettoyez les paillasses et les pipettes avec une solution de décontamination à 70 % d’éthanol et de RNase.
- Nettoyez un tube de broyeur de tissu à fond rond de 2 ml et le pilon A et B assorti avec une solution de décontamination RNase, suivi d’une solution de décontamination à base d’éthanol à 70 % et d’eau sans RNase (1 tube de broyeur et jeu de pilon par échantillon). Laissez-le sécher complètement.
- Pré-refroidir la centrifugeuse à 4 °C.
- Étiqueter et prérefroidir trois tubes de collecte de 15 mL, un tube de collecte de 1,5 mL, un tube de prélèvement de cellules activées par fluorescence (FACS) de 5 mL et un tube de broyage sec pour chaque échantillon sur glace.
- Préparation du tampon
- Réchauffer la solution mère du complexe ribonucléoside-vanadyle à 65 °C jusqu’à ce qu’elle soit reconstituée en une solution transparente vert-noir conformément aux instructions du fabricant. 11
- Préparer 1x PBS contenant 4 % d’albumine sérique bovine (BSA) comme décrit dans le tableau 1A. De plus, préparez 1x PBS avec 0,04% BSA (tableau 1B). Filtrer les deux solutions à l’aide d’un filtre à seringue à membrane en acétate de cellulose sans surfactant (SFCA) de 0,2 μm et conserver sur la glace jusqu’à nouvelle utilisation.
- Préparer le tampon de lyse des noyaux 1 (NLB1, tableau 1C). Ajouter 4 mL de tampon de lyse EZ pour le tampon de lyse des noyaux 2 (NLB2, tableau 1D) et 2 mL de BSA / PBS à 0,04 % pour le tampon de suspension de noyaux (NSB, tableau 1E) à des tubes de 15 mL. Ajouter la solution d’inhibiteur de la RNase au NLB2 et au NSB directement avant utilisation, comme indiqué ci-dessous dans le protocole. Garder sur la glace jusqu’à nouvel usage.
- Préparer le tampon de lyse EZ avec 10 % de saccharose (tampon de gradient de saccharose, tableau 1F). Bien mélanger et filtrer le tampon dans un nouveau tube de 15 ml à l’aide d’un filtre à seringue à membrane SFCA de 0,2 μm. Garder sur la glace jusqu’à nouvel usage.
- Homogénéisation tissulaire et lyse cellulaire
NOTE: Afin de minimiser la dégradation de l’ARN, toutes les étapes sont effectuées sur la glace. Le tube du broyeur, la boîte de Petri et tous les tampons doivent être pré-refroidis. Toutes les étapes de remise en suspension sont effectuées en pipetant soigneusement la suspension de noyaux. Ne pas vortex l’échantillon pour éviter les forces de cisaillement et les dommages aux noyaux.- Prenez le morceau de rein congelé et transférez-le dans une boîte de Petri en polystyrène de 60 mm sur de la glace contenant 1 mL de NLB1.
- Hachez soigneusement le tissu à l’aide d’une lame de rasoir ou d’un scalpel (figure 3A).
- Coupez l’extrémité d’une embout de pipette de 1 mL et transférez le tissu haché et le tampon dans le tube du broyeur. Assurez-vous de transférer tous les morceaux de tissu. Lavez la boîte de Petri 5 à 10 fois avec le tampon, si nécessaire.
- Homogénéiser la suspension sur la glace en déplaçant lentement le pilon A, 25x de haut en bas dans le tube du broyeur. Évitez les bulles d’air causées par des mouvements rapides (figure 3B).
- Passez l’homogénat dans une crépine de 100 μm dans un tube de collecte prérefroidi de 15 mL et lavez le filtre avec un autre 1 mL de NLB1.
- Lavez le tube du broyeur avec un tampon de lyse froid pour les noyaux EZ et jetez le tampon.
- Transférer l’homogénat dans le tube du broyeur et homogénéiser la suspension sur la glace en déplaçant lentement le pilon B, 15x de haut en bas dans le tube du broyeur. Évitez les bulles d’air causées par des mouvements rapides (figure 3C).
- Transférer l’homogénat dans un tube de prélèvement prérefroidi de 15 mL. Lavez le tube du broyeur avec 2 mL de NLB1 et assurez-vous de transférer tous les fragments de tissu dans le tube de prélèvement. Incuber l’homogénat (volume total de 4 mL) pendant 5 min sur de la glace pour lyser les cellules.
- Purification des noyaux
- Faire passer l’homogénat dans une crépine de 40 μm dans un tube de prélèvement prérefroidi de 15 mL. Faire tourner le tube collecteur pendant 5 min à 500 x g à 4 °C dans une centrifugeuse à l’aide d’un rotor à godet oscillant. En attendant, ajouter une solution d’inhibiteur de la RNase à NLB2 (Tableau 1D).
- Retirer le surnageant sans déranger la pastille. Remettez délicatement la pastille en suspension dans 4 mL de NLB2.
- Sous-tendre soigneusement la suspension avec un coussin de 1 ml de tampon dégradé de saccharose. Centrifuger à 500 x g pendant 5 min à 4 °C dans une centrifugeuse à rotor à godet oscillant. Entre-temps, ajouter une solution d’inhibiteur de la RNase au NSB (tableau 1E).
- Après la centrifugation, retirez délicatement le tube de collecte de la centrifugeuse et veillez à ne pas déranger les deux couches lors de la manipulation du tube de collecte. Les débris cellulaires sont visibles entre les deux couches. Retirez soigneusement le surnageant en commençant par les débris. Retirer le surnageant restant sans perturber la pastille de noyaux et remettre délicatement la pastille en suspension dans 1 mL de NSB.
NOTE: Le volume de remise en suspension dépend de la quantité de tissu utilisée pour l’isolement et de la taille des pastilles obtenues après la dernière étape de centrifugation. Il peut être nécessaire d’adapter le volume au nombre attendu de noyaux. - Faire passer l’homogénat dans une crépine de 20 μm dans le tube de collecte FACS prérefroidi de 5 mL.
3. Tri des noyaux
- Ajouter 20 μL de 4′,6-diamidino-2-phénylindole (DAPI) par mL de NSB à une concentration finale de 2 μM à l’homogénat dans le tube de collecte FACS et mélanger soigneusement. Incuber pendant 5 min sur la glace.
- Préparer le tube de collecte prérefroidi de 1,5 mL avec 20 μL de BSA à 4 % /1x PBS et ajouter la solution inhibitrice de la RNase à 0,5 μL jusqu’à une concentration finale de 1 U/μL. Procéder immédiatement au tri.
- Triez les noyaux à l’aide d’un trieur de cellules.
- Mélanger brièvement la suspension de noyaux avant d’insérer le tube de collecte FACS dans le trieur.
- Définir une première porte P1 basée sur la diffusion vers l’avant (FSC) et la diffusion latérale (SSC) pour exclure les débris et les agrégats (figure 4A).
- Pour exclure les noyaux et les multiplets vides ou endommagés, définissez une porte suivante en fonction de la zone DAPI par rapport à la hauteur DAPI (DAPI-A vs DAPI-H) (Figure 4B).
- Trier les noyaux simples dans le tube de collecte de 1,5 mL contenant 4% de BSA / 1x PBS avec 1 solution d’inhibiteur de RNase U/μL préparée en 3.2.
4. Contrôle de la qualité
- Mesurer la concentration finale des noyaux au microscope à fluorescence ou dans une chambre de comptage automatisée en au moins deux comptages indépendants et évaluer la qualité de la suspension (figure 5).
REMARQUE : Les concentrations optimales se situent entre 700 et 1 200 noyaux/μL. Des concentrations cellulaires plus faibles, telles que 700 noyaux/μL, peuvent être préférables, car les banques d’ADNc résultantes contenaient moins d’ARN de fond ambiant (transcrits non associés à des noyaux individuels). - Calculer le volume requis de noyaux en suspension pour la récupération souhaitée de noyaux simples séquencés. Afin d’éviter l’agrégation des noyaux et la dégradation de l’ARN, procéder immédiatement à la préparation de la bibliothèque.
Résultats
Pour déterminer la performance de notre protocole, nous avons utilisé le 10x Genomics Chromium Single Cell 3' Gene Expression Kit v3.1 pour la préparation de la bibliothèque et analysé les données de snRNA-seq avec le paquet Seurat12,13.
La figure 6 montre les résultats d’une bibliothèque représentative de snRNA-seq. Pour évaluer la qualité de nos noyaux, nous avons tracé le nombre de gènes par rapport au nombre de transcrits (définis par des identifiants moléculaires uniques (UMI)) colorés par la fraction de lectures mitochondriales (Figure 6A). Les noyaux de bonne qualité présentent généralement un nombre plus élevé de lectures, corrélant le nombre d’UMI et de gènes, et de faibles fractions de lecture mitochondriales.
Pour l’analyse ultérieure, les noyaux avec moins de 500 ou plus de 4000 gènes comptés, ou plus de 5% d’ARN mitochondrial ont été exclus (n = 828). Seuls les gènes exprimés dans un minimum de trois noyaux ont été inclus. Nous avons détecté environ 20 000 gènes au total dans les 6 000 noyaux restants avec 1 600 gènes médians et 2 800 UMI médians par noyau (Figure 6B).
Le regroupement était basé sur des gènes très variables. Nous avons identifié un total de 18 grappes. Les identités cellulaires ont été annotées en fonction de gènes marqueurs connus (non représentés). Les sous-grappes d’un type cellulaire ont été résumées en une seule grappe, ce qui donne un total de 11 types de cellules distinctes : podocytes, tubule proximal (PT), membre mince (tL), membre ascendant épais (TAL), tubule convoluté distal (DCT), tubule de connexion (CNT), principal du canal collecteur et cellules intercalées (CD-PC, A-IC, B-IC), épithélium médullaire profond du bassin (DMEP) et endothélium. Les profils d’expression génique des marqueurs enrichis en grappes ont été visualisés dans un diagramme à points (figure 6C) et les groupes de types cellulaires dans un diagramme d’incorporation de voisins stochastiques distribués en t (t-SNE) (figure 6D).
Pour évaluer les distributions des types cellulaires dans notre échantillon, le pourcentage de chaque type de cellule a été calculé (figure 6E) et utilisé pour déterminer le rapport du PT au TAL. Le PT est principalement situé dans le cortex rénal et souvent surreprésenté dans les ensembles de données sur les cellules uniques du rein, car les cellules du PT sont faciles à dissocier et très abondantes dans les échantillons de reins entiers. Le TAL, quant à lui, s’étend sur toute la moelle extérieure14. Ainsi, le rapport des fractions PT et TAL représente une bonne mesure de l’enrichissement des types de cellules médullaires dans un ensemble de données unicellulaires rénales. En général, le rapport PT/TAL dans les ensembles de données sur les reins entiers unicellulaires variait de 8 (données non publiées provenant de tissus rénaux entiers traités à la protéase froide) à 45 pour les tissus dissociés enzymatiquement10,14,15. Dans l’ensemble de données snRNA-seq présenté ici, nous avons pu atteindre un rapport PT/TAL de 2. Ce résultat illustre que l’élimination de l’excès de cortex pendant la dissection tissulaire combinée à un séquençage de l’ARNn entraîne une amélioration frappante de la représentation du type de cellules rénales.
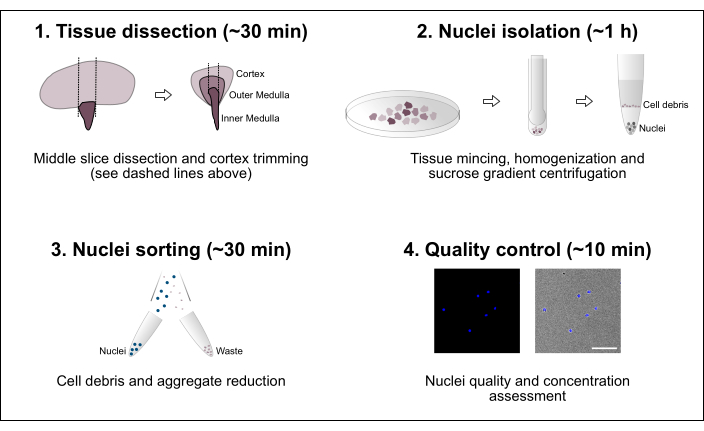
Figure 1 : Vue d’ensemble schématique du flux de travail. Le protocole comprend quatre étapes principales qui comprennent la dissection tissulaire suivie de l’isolement des noyaux, du tri des noyaux et d’une évaluation finale de la pureté et de la concentration. Barre d’échelle = 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
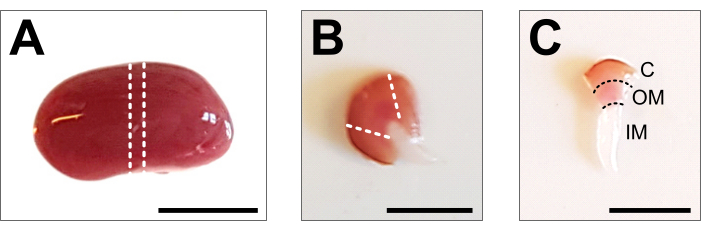
Figure 2 : Dissection rénale et préparation tissulaire. (A) Image représentative d’un rein entier disséqué. Les lignes pointillées indiquent les coupes nécessaires pour obtenir une tranche médiane de 1-2 mm avec une représentation de tous les types de cellules rénales. (B) Image représentative de la tranche du milieu obtenue. Les lignes pointillées indiquent les coupes pour la coupe du cortex sur le côté. (C) Image représentative d’un morceau de rein central avec cortex coupé. Le cortex (C), la moelle externe (OM) et la moelle interne (IM) sont clairement visibles. Barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Homogénéisation tissulaire et purification des noyaux. (A) Image représentative montrant du tissu rénal suffisamment haché. Barre d’échelle = 500 μm. (B) Homogéner après la première étape d’homogénéisation (25 coups avec le pilon A, tube de meulage de 2 mL). (C) Homogéner après deuxième étape d’homogénéisation (15 coups avec le pilon B, tube de meulage de 2 mL). Veuillez cliquer ici pour voir une version agrandie de cette figure.
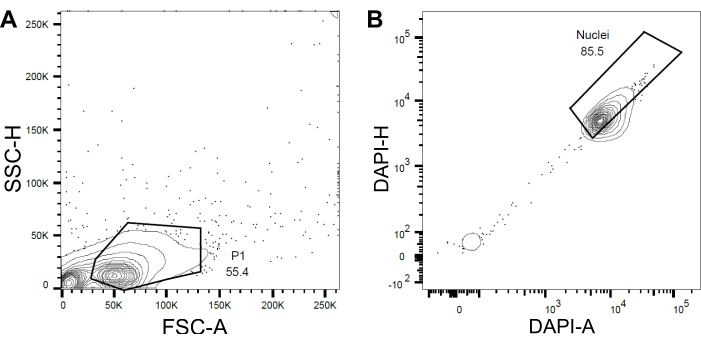
Figure 4 : Stratégie de contrôle pour le tri des noyaux. (A) Une première porte P1 a été définie sur la base de la diffusion vers l’avant (FSC) par rapport à la diffusion latérale (SSC) pour exclure les débris et les agrégats. (B) Une porte ultérieure basée sur la zone DAPI (DAPI-A) par rapport à la hauteur DAPI (DAPI-H) excluait les noyaux et les multiplets vides ou endommagés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
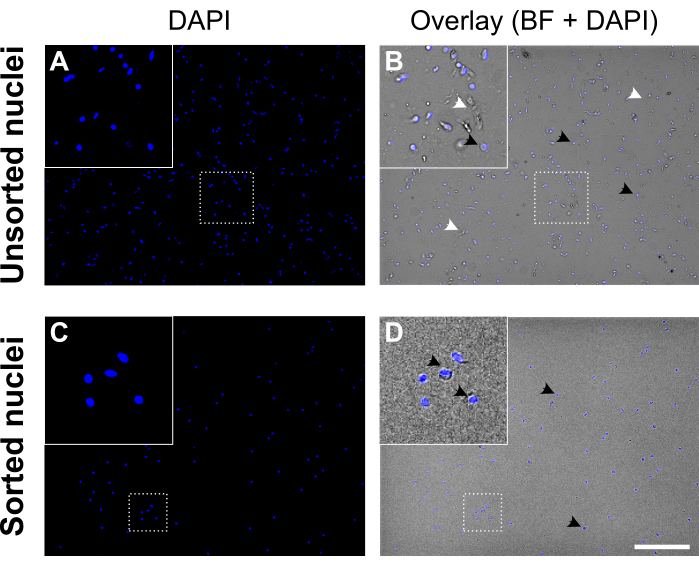
Figure 5 : Suspension de noyaux avant et après tri des noyaux. (A, C) Noyaux colorés DAPI (bleu). (B, D) Superposition de DAPI et de canal en fond clair (BF). Avant le tri (panneau supérieur), la suspension de noyaux contient des débris cellulaires et des agrégats (marqués avec des pointes de flèches blanches). Après tri (panneau inférieur), la suspension des noyaux semble beaucoup plus propre. Des exemples de noyaux colorés DAPI sont étiquetés avec des pointes de flèches noires. Les noyaux de bonne qualité apparaissent ronds et lisses avec une membrane intacte et sont bien séparés, tandis que les noyaux de mauvaise qualité apparaissent ridés et présentent une perte de la membrane nucléaire. Barre d’échelle = 250 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
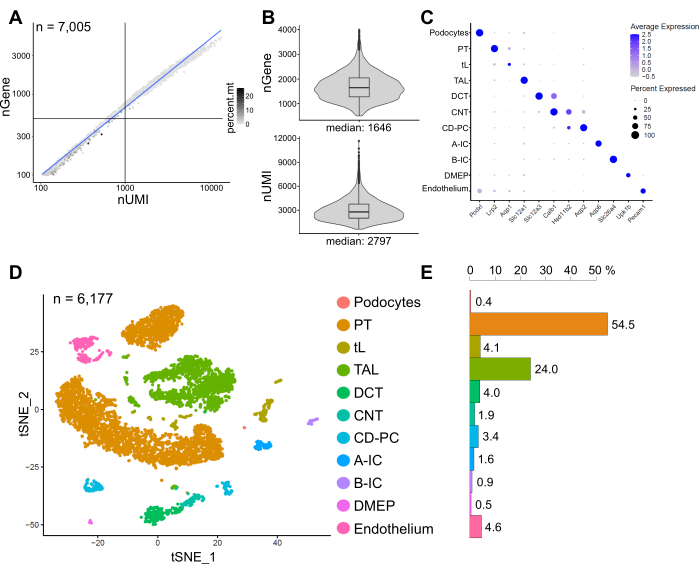
Figure 6 : Contrôle de la qualité et analyse d’un ensemble de données représentatif de l’ARNn-seq. (A) Nombre de gènes (nGene) tracé en fonction du nombre d’identificateurs moléculaires uniques (nUMI) colorés par la fraction de lectures mitochondriales (percent.mt). Les noyaux de faible qualité correspondent au quadrant inférieur gauche du graphique (n = 828) et ont été exclus de l’analyse ultérieure. (B) Distribution et médiane du gène nGene et du nUMI détectés par noyau dans l’ensemble de données snRNA-seq, représentant 6 177 noyaux (> 500 gènes). Les bibliothèques ont été séquencées à une profondeur médiane de ~ 8 200 lectures mappées par noyau. (C) Diagramme à points montrant les profils d’expression génique des marqueurs enrichis en grappes (axe des x) pour les types de cellules individuels (axe des y). La taille du point correspond à la proportion de cellules exprimant le gène indiqué. La couleur correspond à l’expression moyenne. (D) Tracé d’incorporation stochastique de voisins distribués en T (t-SNE) de types cellulaires identifiés. (E) Distribution des types cellulaires dans l’ensemble de données snRNA-seq. PT, tubule proximal; tL, membre mince; TAL, membre ascendant épais, DCT, tubule convolué distal; CNT, tubule de raccordement; CD-PC, cellules principales de collecte des conduits; A-IC, cellules intercalées de type A; B-IC, cellules intercalées de type B; DMEP, épithélium médullaire profond du bassin. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Réactif | Concentration finale | Volume (mL) | ||
| a) 4 % BSA / PBS | ||||
| Solution saline tamponnée au phosphate (PBS) contenant 10 % d’albumine bovine | 4% | 2 | ||
| PBS (solution saline tamponnée au phosphate) 1X sans calcium ni magnésium | - | 3 | ||
| b) 0,04 % BSA / PBS | ||||
| 4 % BSA / PBS | 0.04 % | 0.5 | ||
| PBS (solution saline tamponnée au phosphate) 1X sans calcium ni magnésium | - | 49.5 | ||
| (C) Tampon de lyse des noyaux 1 (NLB1) | ||||
| Tampon de lyse nucléaire EZ | - | 4 | ||
| Inhibiteur de la RNase RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| Complexe ribonucléoside-vanadyle (200 mM) | 10 mM | 0.2 | ||
| d) Tampon de lyse des noyaux 2 (NLB2) | ||||
| Tampon de lyse nucléaire EZ | - | 4 | ||
| Inhibiteur de la RNase RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| e) Tampon de suspension de noyaux (NSB) | ||||
| 0,04 % BSA / PBS | - | 2 | ||
| Inhibiteur de la RNase RiboLock (40 U/μL) | 1 U/μL | 0.05 | ||
| f) Tampon de gradient de saccharose (10 % saccharose) | ||||
| Poids 1 g de saccharose | ||||
| Dissoudre dans 6 ml de tampon de lyse EZ nucléaire | ||||
| Remplir jusqu’à 10 mL avec le tampon de lyse EZ nucléaire | ||||
| Filtrer à travers un filtre à seringue de 0,2 μm dans un tube neuf | ||||
Tableau 1 : Recettes de solution : (A) Préparation de 4 % de BSA/1x PBS. Filtrer à l’aide d’un filtre à seringue à membrane SFCA de 0,2 μm et conserver sur la glace jusqu’à utilisation. (B) Préparation de 0,04% BSA/1 x PBS. Filtrer à l’aide d’un filtre à seringue à membrane SFCA de 0,2 μm et conserver sur la glace jusqu’à utilisation. c) Préparation du tampon de lyse des noyaux 1 (NLB1). Les volumes indiqués sont fournis par échantillon. Conserver sur la glace jusqu’à utilisation. d) Préparation du tampon de lyse des noyaux 2 (NLB2). Les volumes indiqués sont fournis par échantillon. Ajouter l’inhibiteur de la RNase RiboLock à NLB2 directement avant utilisation, comme indiqué dans le protocole. Conserver sur la glace jusqu’à utilisation. e) Préparation du tampon de suspension de noyaux (NSB). Les volumes indiqués sont fournis par échantillon. Ajouter l’inhibiteur de la RNase RiboLock au NSB directement avant utilisation, comme indiqué dans le protocole. Conserver sur la glace jusqu’à utilisation. f) Préparation du tampon de gradient de saccharose. Filtrer à l’aide d’un filtre à seringue à membrane SFCA de 0,2 μm et conserver sur la glace jusqu’à utilisation.
Discussion
La transcriptomique unicellulaire fait progresser la compréhension de l’expression génique spécifique au type cellulaire dans la physiologie et la maladie rénales. Ici, nous avons fourni une méthode simple et reproductible pour isoler des noyaux uniques de haute qualité à partir de tissus rénaux de souris congelés pour le séquençage de l’ARNn de manière standardisée.
Pour le snRNA-seq, il est essentiel d’utiliser des noyaux de haute qualité comme entrée pour la génération de bibliothèques et d’éviter la dégradation de l’ARN pendant le traitement des tissus. Par conséquent, l’incubation de morceaux de tissu dans une solution de stabilisation de l’ARN immédiatement après la dissection est essentielle pour protéger et stabiliser l’ARN cellulaire et permet de stocker les échantillons à - 80 °C indéfiniment. Lors de l’application de ce protocole à des tissus congelés sans traitement en solution de stabilisation de l’ARN, tels que du matériel d’archives, un essai est nécessaire et la qualité de l’ARN doit être évaluée car nous avons observé une perte significative de l’intégrité de l’ARN dans les tissus congelés sans incubation préalable dans une solution de stabilisation de l’ARN.
En général, une manipulation appropriée des échantillons est cruciale pour maximiser la récupération des noyaux uniques intacts. Toutes les étapes de remise en suspension doivent être effectuées par pipetage avec précaution pour éviter les contraintes de cisaillement et les dommages physiques. Les tampons pour la remise en suspension finale des noyaux et le tri des noyaux doivent contenir de l’ABS pour éviter la perte et l’agrégation des noyaux.
Les volumes tampons dans ce protocole sont optimisés pour de très petits échantillons de tissus (~15 mg). Il est essentiel d’assurer une lyse cellulaire complète et un lavage suffisant pour générer des suspensions de haute qualité. Des blocs de tissus plus grands ou des échantillons de reins entiers entraîneront des concentrations excessives de noyaux qui entraîneront l’agglutination et l’agrégation, une abondance élevée d’ARN ambiant et une mauvaise qualité globale de la suspension. Si des échantillons plus grands ou d’autres tissus sont traités, il est fortement recommandé d’effectuer des essais pour déterminer les volumes tampons optimaux pour des niveaux minimaux d’ARN ambiant. La qualité et les concentrations des noyaux et de l’ARN doivent être examinées avec soin, car une surcharge entraîne une performance globalement médiocre.
En outre, de grandes quantités de débris cellulaires, provoquant des niveaux élevés d’ARN ambiant non associés à des noyaux uniques, influencent négativement les résultats du séquençage. La clarification de la suspension des noyaux par centrifugation à travers un coussin de saccharose atténue ce problème dans une certaine mesure, mais elle peut également conduire à un biais dans la représentation du type cellulaire en contre-sélectionnant contre les petits noyaux denses présents, par exemple, dans les cellules immunitaires16. Si cela est préoccupant, le gradient de saccharose doit être omis. En revanche, nous avons constaté que la cytométrie en flux basée sur la coloration DAPI était essentielle pour réduire la quantité de débris cellulaires afin de produire une suspension mononucléiforme de haute qualité.
L’isolement de noyaux uniques présente des avantages considérables par rapport aux approches unicellulaires8. Il est compatible avec les tissus correctement congelés, ce qui rend la collecte de tissus plus flexible et contourne le besoin de dissociation tissulaire à base d’enzymes, qui peut introduire des réponses de stress transcriptionnel 6,17. De plus, il surmonte le biais de dissociation qui favorise la sélection de types cellulaires facilement dissociables du cortex rénal, ce qui peut conduire à une sous-représentation des types de cellules médullaires dans certaines approches enzymatiques 5,6,10.
L’utilisation d’un morceau de rein central au lieu d’un tissu rénal entier permet d’économiser davantage de ressources et de corriger la surreprésentation des types de cellules abondantes comme décrit précédemment10. Cependant, selon le modèle murin ou le phénotype étudié, il peut être avantageux d’utiliser des échantillons de reins entiers au lieu d’une seule tranche du milieu. Les échantillons de rein entier peuvent être plus représentatifs des proportions cellulaires réelles ou des changements qui se produisent dans le rein entier, alors qu’une tranche du milieu parée s’est avérée avantageuse pour les phénotypes médullaires ou lorsque le matériel de l’échantillon était limité. Cette décision est donc très spécifique à l’utilisateur et doit être examinée avec soin.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions la plateforme de génomique scientifique du Centre Max Delbrück de médecine moléculaire de l’Association Helmholtz, à Berlin, pour son soutien technique.
JL et KMSO ont été soutenus par le groupe de formation à la recherche GRK 2318 de la Fondation allemande pour la recherche (DFG) et par l’unité de recherche FOR 2841. KMSO a été soutenu par la subvention de recherche collaborative 1365. AB a été soutenu par le financement du prix Gottfried Wilhelm Leibniz de la DFG décerné à NR.
matériels
| Name | Company | Catalog Number | Comments |
| Cell sorter | - | - | For fluorescence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. |
| Centrifuge 5810 R | Eppendorf | 5811000015 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | Needs to contain DAPI light cube to count nuclei. Alternatively, nuclei can be counted manually under fluorescence microscope. |
| 4′,6-Diamidino-2-phenyl-indol-dihydrochlorid (DAPI) | Biotrend | 40043/b | Stock solution prepared with a concentration of 100 µM. Used for nuclei staining in a final concentration of 2 µM. |
| D(+)-Sucrose ≥99.9%, ultrapure DNAse-, RNAse-free | VWR | 0335-500G | |
| DNA LoBind Microcentrifuge Tubes (1.5 mL) | Eppendorf | 22431021 | |
| Ethanol, 70 % | - | - | |
| FACS tubes | pluriSelect | 43-10100-46 | |
| KIMBLE Dounce tissue grinder set 2 mL complete | Sigma-Aldrich | D8938-1SET | |
| Minisart Syringe Filters 0.2 µm | Sartorius | 16534-GUK | |
| Nuclease-free Water | Invitrogen | AM9937 | |
| Nuclei EZ Prep Nuclei Isolation Kit | Sigma-Aldrich | NUC-101 | Nuclei EZ Lysis Buffer (Product No. N3408) needed for buffer preparation. |
| PBS (Phosphate-Buffered Saline) 1X without calcium or magnesium | Corning | 21-040-CV | |
| Petri dishes, polystyrene 60 mm | Sigma-Aldrich | P5481 | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin | Sigma-Aldrich | SRE0036 | |
| pluriStrainer Mini 100 µm | pluriSelect | 43-10100-46 | |
| pluriStrainer Mini 20 µm | pluriSelect | 43-10020-40 | |
| pluriStrainer Mini 40 µm | pluriSelect | 43-10040-40 | |
| Polystyrene Centrifuge Tube (15 mL) | Falcon | 352099 | |
| Razor blades | - | - | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher | EO0384 | |
| Ribonucleoside-vanadyl complex | New England Biolabs | S1402S | Follow manufacturer's instructions (https://international.neb.com/products/s1402-ribonucleoside-vanadyl-complex#Product%20Information). Upon use the 200 mM stock solution is reconstituted to a green-black clear solution by incubating at 65 °C. |
| RNAlater Stabilization Solution | Invitrogen | AM7020 | |
| RNase AWAY | Fisher Scientific | 11952385 |
Références
- Thomas, R. S. Kidney modeling and systems physiology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1 (2), 172-190 (2009).
- Potter, S. S. Single-cell RNA sequencing for the study of development, physiology and disease. Nature Reviews Nephrology. 14 (8), 479-492 (2018).
- Park, J., Liu, C. L., Kim, J., Susztak, K. Understanding the kidney one cell at a time. Kidney International. 96 (4), 862-870 (2019).
- Clark, A. R., Greka, A. The power of one: advances in single-cell genomics in the kidney. Nature Reviews Nephrology. 16 (2), 73-74 (2020).
- Lake, B. B., et al. A single-nucleus RNA-sequencing pipeline to decipher the molecular anatomy and pathophysiology of human kidneys. Nature Communications. 10 (1), 2832(2019).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 30 (1), 23-32 (2019).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144 (19), 3625-3632 (2017).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Muto, Y., et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nature Communications. 12 (1), 2190(2021).
- Hinze, C., et al. Kidney single-cell transcriptomes predict spatial corticomedullary gene expression and tissue osmolality gradients. Journal of the American Society of Nephrology. 32 (2), 291(2021).
- Berger, S. L. Isolation of cytoplasmic RNA: ribonucleoside-vanadyl complexes. Methods in Enzymology. 152, 227-234 (1987).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411-420 (2018).
- Stuart, T., et al. Comprehensive Integration of single-cell data. Cell. 177 (7), 1888-1902 (2019).
- Park, J., et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease. Science. 360 (6390), 758-763 (2018).
- Kirita, Y., Wu, H., Uchimura, K., Wilson, P. C., Humphreys, B. D. Cell profiling of mouse acute kidney injury reveals conserved cellular responses to injury. Proceedings of the National Academy of Sciences of the United States of America. 117 (27), 15874-15883 (2020).
- Schneeberger, S., et al. The neuroinflammatory interleukin-12 signaling pathway drives Alzheimer's disease-like pathology by perturbing oligodendrocyte survival and neuronal homeostasis. bioRxiv. , 441313(2021).
- Nguyen, Q. H., Pervolarakis, N., Nee, K., Kessenbrock, K. experimental considerations for single-cell RNA sequencing approaches. Frontiers in Cell and Developmental Biology. 6, 108(2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon