Method Article
Isolamento de Núcleos do Rim de Rato Adulto para Sequenciamento de RNA de Núcleo Único
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para isolar núcleos de alta qualidade de rins de camundongos congelados que melhoram a representação de tipos de células renais medulares e evitam os artefatos de expressão gênica da dissociação do tecido enzimático.
Resumo
Os rins regulam diversos processos biológicos, como água, eletrólito e homeostase ácido-base. As funções fisiológicas do rim são executadas por vários tipos de células dispostas em uma arquitetura complexa em todo o eixo corticomedular do órgão. Avanços recentes na transcriptômica unicelular aceleraram a compreensão da expressão gênica específica do tipo celular na fisiologia renal e na doença. No entanto, os protocolos de dissociação tecidual baseados em enzimas, que são frequentemente utilizados para sequenciamento de RNA de célula única (scRNA-seq), requerem principalmente tecido fresco (não arquivado), introduzem respostas de estresse transcricional e favorecem a seleção de tipos celulares abundantes do córtex renal, resultando em uma sub-representação das células da medula.
Aqui, apresentamos um protocolo que evita esses problemas. O protocolo baseia-se no isolamento de núcleos a 4 °C do tecido renal congelado. Os núcleos são isolados de uma peça central do rim do rato composta pelo córtex, medula externa e medula interna. Isso reduz a super-representação de células corticais típicas de amostras de rim inteiro para o benefício de células medulares, de modo que os dados representarão todo o eixo corticomedular em abundância suficiente. O protocolo é simples, rápido e adaptável e fornece um passo em direção à padronização da transcriptômica de núcleos únicos na pesquisa renal.
Introdução
Os rins exibem uma arquitetura de tecido altamente complexa. Consistem em segmentos funcional e anatomicamente distintos ao longo de um eixo corticomedular e mediam funções biológicas, como regulação do volume de líquido extracelular, equilíbrio eletrolítico ou homeostase ácido-base1.
Os avanços na transcriptômica unicelular permitiram a caracterização aprofundada de tecidos complexos e aceleraram a compreensão da expressão gênica específica do segmento e do tipo celular na fisiologia, desenvolvimento e doença renal 2,3,4.
No entanto, os protocolos de dissociação baseados em enzimas que são frequentemente utilizados para scRNA-seq exibem várias desvantagens e restrições. Dependendo do protocolo, geram respostas transcricionais ao estresse e viés de dissociação tecidual em direção a tipos de células corticais mais fáceis de dissociar 5,6. Embora os protocolos que utilizam proteases ativas a frio para rins embrionários sejam capazes de mitigar as alterações transcricionais relacionadas ao estresse, eles não conseguem superar o viés de dissociação em relação às células corticais e podem não ser facilmente adaptáveis a diferentes tipos de tecidos renais doentes7. Além disso, as abordagens unicelulares não são facilmente compatíveis com amostras de tecido congelado, limitando sua aplicação principalmente a tecidos frescos e não arquivados, tornando a coleta de tecidos um fator restritivo6.
O sequenciamento de RNA de núcleos únicos (snRNA-seq) pode contornar essas limitações 8,9. Aqui, apresentamos um protocolo para isolamento de núcleos de um corte central de tecido renal adulto de camundongo congelado (Figura 1)10. Nosso protocolo é simples e fornece uma abordagem padronizada para obter bibliotecas de sequenciamento de RNA com uma representação equilibrada de diversos tipos de células renais para modelos experimentais que não envolvem fortes alterações teciduais regionais. Neste último caso, nosso protocolo também pode ser realizado com rins inteiros.
Protocolo
Todos os experimentos em animais foram conduzidos de acordo com a Lei de Bem-Estar Animal (TierSchG) e o Regulamento de Bem-Estar Animal Experimental Animal (TierSchVersV) e foram autorizados pelas autoridades locais e pelos Oficiais de Bem-Estar Animal de nossa instituição (MDC).
1. Preparação de tecidos
- Prepare uma placa de 6 poços contendo 2 mL de 1x solução salina tamponada com fosfato (PBS) por poço para cada rim que será obtido. Prepare uma placa de 6 poços contendo 2 mL de solução de estabilização de RNA por poço e rim. Pré-resfrie ambas as placas no gelo.
- Eutanasiar um rato macho C57BL/6 de 3 a 6 meses de idade. Coloque o rato em uma bandeja de dissecação, prenda as extremidades e esterilize o abdômen com etanol a 70%.
- Abra o abdômen até a caixa torácica usando fórceps e tesoura. Levante o intestino e outros órgãos para o lado e remova os rins cortando cuidadosamente o ureter, a artéria renal e a veia com uma tesoura. Lave o rim no PBS 1x gelado previamente preparado e remova a fáscia renal e qualquer gordura restante do rim até que todo o tecido branco seja removido (Figura 2A).
- Coloque o rim em uma placa de dissecação fria e use um bisturi afiado ou lâmina de barbear para obter uma fatia média de 1-2 mm. Certifique-se de que a peça de tecido contenha todo o eixo corticomedular (Figura 2B). Use tesouras e pinças de microdissecação para aparar cuidadosamente o córtex dos lados da peça central. Dentro da peça de tecido dissecada, os três segmentos córtex, medula externa e medula interna devem ser claramente visíveis (Figura 2C).
NOTA: A fatia não deve exceder uma espessura de 2 mm ou um peso de 20 mg para garantir quantidades tampão suficientes para uma lise tecidual eficaz e para minimizar o ARN de fundo ambiente nas bibliotecas de cDNA. O RNA ambiente desperdiça a capacidade de sequência, pois não está associado a núcleos únicos. - Transfira a peça renal para a solução de estabilização de RNA previamente preparada e incube por 24 h a 4 °C para evitar a degradação do RNA. Após 24 h, retirar a solução estabilizadora de ARN e conservar o tecido a -80 °C até nova utilização. Remova cuidadosamente o excesso de solução com papel de seda.
2. Isolamento dos núcleos
- Etapas de limpeza e preparação
- Limpe bancadas e pipetas com etanol a 70% e solução de descontaminação de RNase.
- Limpe um tubo moedor de tecido de fundo redondo de 2 mL e compatibilhe combinando pilão A e B com solução de descontaminação de RNase, seguido de etanol a 70% e água livre de RNase (1 tubo de moedor e pilão por amostra). Deixe secar completamente.
- Pré-arrefecer a centrífuga a 4 °C.
- Rotule e pré-resfrie três tubos de coleta de 15 mL, um tubo de coleta de 1,5 mL, um tubo de coleta de 5 mL de classificação celular ativada por fluorescência (FACS) e um tubo de moedor seco para cada amostra em gelo.
- Preparação do buffer
- Aquecer a solução-mãe do complexo ribonucleosídeo-vanadilo a 65 °C até ser reconstituída numa solução límpida verde-preta de acordo com as instruções do fabricante. 11
- Preparar 1x PBS contendo 4% de albumina sérica bovina (BSA), conforme descrito na Tabela 1A. Além disso, preparar 1x PBS com 0,04% de BSA (Tabela 1B). Filtrar ambas as soluções utilizando um filtro de seringa de membrana de acetato de celulose (SFCA) isento de surfactante de 0,2 μm e manter no gelo até nova utilização.
- Prepare o tampão de lise de núcleos 1 (NLB1, Tabela 1C). Adicionar 4 mL de tampão de lise EZ para Nuclei Lysis Buffer 2 (NLB2, Tabela 1D) e 2 mL de 0,04% BSA/PBS para o Nuclei Suspension Buffer (NSB, Tabela 1E) a tubos de 15 mL. Adicione a solução inibidora de RNase ao NLB2 e NSB diretamente antes de usar, conforme indicado abaixo no protocolo. Mantenha no gelo até novo uso.
- Prepare o tampão de lise EZ com sacarose a 10% (Sucrose Gradient Buffer, Tabela 1F). Misture bem e filtre o tampão para um tubo fresco de 15 mL usando um filtro de seringa de membrana SFCA de 0,2 μm. Mantenha no gelo até novo uso.
- Homogeneização tecidual e lise celular
NOTA: A fim de minimizar a degradação do RNA, todas as etapas são realizadas no gelo. O tubo do moedor, a placa de Petri e todos os tampões precisam ser pré-resfriados. Todas as etapas de ressuspensão são feitas pipetando cuidadosamente a suspensão dos núcleos. Não vortex a amostra para evitar forças de cisalhamento e danos aos núcleos.- Pegue a peça de rim congelada e transfira-a para uma placa de Petri de poliestireno de 60 mm em gelo contendo 1 mL de NLB1.
- Pique bem o tecido usando uma lâmina de barbear ou bisturi (Figura 3A).
- Corte a ponta de uma ponta de pipeta de 1 mL e transfira o tecido picado e o tampão para o tubo do moedor. Certifique-se de transferir todas as peças de tecido. Lave a placa de Petri 5-10 vezes com o tampão, se necessário.
- Homogeneize a suspensão no gelo movendo lentamente o pilão A, 25x para cima e para baixo no tubo do moedor. Evite bolhas de ar causadas por movimentos rápidos (Figura 3B).
- Passar o homogeneizado através de um filtro de 100 μm em um tubo de coleta de 15 mL pré-resfriado e lavar o filtro com mais 1 mL de NLB1.
- Lave o tubo do moedor com o tampão de lise de núcleos EZ frio e descarte o tampão.
- Transfira o homogeneizado de volta para o tubo do moedor e homogeneize a suspensão no gelo movendo lentamente o pilão B, 15x para cima e para baixo no tubo do moedor. Evite bolhas de ar causadas por movimentos rápidos (Figura 3C).
- Transfira o homogeneizado para um tubo de coleta de 15 mL pré-resfriado. Lave o tubo moedor com mais 2 mL de NLB1 e certifique-se de transferir todos os fragmentos de tecido para o tubo de coleta. Incubar o homogeneizado (volume total de 4 mL) por 5 min no gelo para lisar as células.
- Purificação de núcleos
- Passar o homogeneizado através de um filtro de 40 μm para um tubo de recolha de 15 ml pré-arfriado. Gire o tubo de recolha durante 5 min a 500 x g a 4 °C numa centrífuga com um rotor de balde. Entretanto, adicione a solução inibidora da RNase ao NLB2 (Tabela 1D).
- Remova o sobrenadante sem perturbar o pellet. Ressuscite cuidadosamente o pellet em 4 mL de NLB2.
- Cuidadosamente submersa na suspensão com uma almofada de 1 ml de tampão gradiente de sacarose. Centrífuga a 500 x g durante 5 min a 4 °C numa centrífuga com rotor de balde. Entretanto, adicione a solução inibidora da RNase ao NSB (Tabela 1E).
- Após a centrifugação, remova suavemente o tubo de coleta da centrífuga e tenha cuidado para não perturbar as duas camadas ao manusear o tubo de coleta. Os detritos celulares são visíveis entre as duas camadas. Remova o sobrenadante cuidadosamente começando com os detritos. Remova o sobrenadante restante sem perturbar o pellet dos núcleos e ressuspenda cuidadosamente o pellet em 1 mL de NSB.
NOTA: O volume de ressuspensão depende da quantidade de tecido utilizado para o isolamento e do tamanho do pellet ganho após a última etapa de centrifugação. O volume pode precisar ser adaptado ao número esperado de núcleos. - Passar o homogeneizado através de um filtro de 20 μm para o tubo de recolha de FACS de 5 ml pré-arfriado.
3. Classificação de núcleos
- Adicionar 20 μL de 4′,6-diamidino-2-fenilindol (DAPI) por ml de NSB a uma concentração final de 2 μM ao homogeneizado no tubo de recolha de FACS e misturar cuidadosamente. Incubar por 5 min no gelo.
- Preparar o tubo de recolha de 1,5 ml pré-arrefecido com 20 μL de BSA a 4% /1x PBS e adicionar 0,5 μL de solução inibidora de RNase a uma concentração final de 1 U/μL. Proceder imediatamente à classificação.
- Classifique os núcleos usando um classificador de células.
- Misture brevemente a suspensão dos núcleos antes de inserir o tubo de recolha FACS no classificador.
- Defina uma primeira porta P1 com base no espalhamento para frente (FSC) e dispersão lateral (SSC) para excluir detritos e agregados (Figura 4A).
- Para excluir núcleos e múltiplos vazios ou danificados, defina uma porta subsequente com base na área DAPI VERSUS DAPI-Height (DAPI-A vs DAPI-H) (Figura 4B).
- Classificar núcleos únicos no tubo de coleta de 1,5 mL contendo BSA a 4% /1x PBS com solução inibidora de RNase 1 U/μL preparada em 3.2.
4. Controle de qualidade
- Medir a concentração final de núcleos sob um microscópio de fluorescência ou em uma câmara de contagem automatizada em pelo menos duas contagens independentes e avaliar a qualidade da suspensão (Figura 5).
NOTA: As concentrações ótimas estão entre 700 - 1.200 núcleos/μL. Concentrações celulares mais baixas, como 700 núcleos/μL, podem ser preferíveis, pois as bibliotecas de cDNA resultantes continham menos RNA de fundo ambiente (transcritos não associados a núcleos individuais). - Calcular o volume necessário de suspensão de núcleos para a recuperação desejada de núcleos únicos sequenciados. A fim de evitar a agregação de núcleos e a degradação do RNA, proceda imediatamente à preparação da biblioteca.
Resultados
Para determinar o desempenho do nosso protocolo, utilizamos o 10x Genomics Chromium Single Cell 3' Gene Expression Kit v3.1 para a preparação da biblioteca e analisamos os dados de snRNA-seq com o pacote Seurat12,13.
A Figura 6 mostra os resultados de uma biblioteca snRNA-seq representativa. Para avaliar a qualidade de nossos núcleos, plotamos o número de genes contra o número de transcritos (definidos por identificadores moleculares únicos (UMIs)) coloridos pela fração de leituras mitocondriais (Figura 6A). Núcleos de boa qualidade geralmente apresentam maior número de leituras, correlacionando UMI e números de genes, e baixas frações de leitura mitocondrial.
Para a análise subsequente, foram excluídos núcleos com menos de 500 ou mais de 4000 genes contados, ou mais de 5% de RNA mitocondrial (n = 828). Apenas genes expressos em um mínimo de três núcleos foram incluídos. Detectamos cerca de 20.000 genes no total nos 6.000 núcleos restantes, com 1.600 genes medianos e 2.800 UMIs medianas por núcleo (Figura 6B).
O agrupamento foi baseado em genes altamente variáveis. Identificamos um total de 18 clusters. As identidades celulares foram anotadas com base em genes marcadores conhecidos (não mostrados). Subaglomerados de um tipo de célula foram resumidos em um aglomerado, resultando em um total de 11 tipos celulares distintos: podócitos, túbulo proximal (TP), membro fino (tL), membro ascendente espesso (TAL), túbulo contorcido distal (DCT), túbulo de conexão (CNT), ducto coletor principal e células intercaladas (CD-PC, A-IC, B-IC), epitélio medular profundo da pelve (DMEP) e endotélio. Os padrões de expressão gênica de marcadores enriquecidos com clusters foram visualizados em um gráfico de pontos (Figura 6C) e os clusters de tipo celular em um gráfico de incorporação de vizinhos estocásticos distribuídos em t (t-SNE) (Figura 6D).
Para avaliar as distribuições do tipo celular em nossa amostra, a porcentagem de cada tipo de célula foi calculada (Figura 6E) e utilizada para determinar a razão entre TP e TAL. O TP está localizado principalmente no córtex renal e frequentemente super-representado em conjuntos de dados de célula única renal, pois as células do PT são fáceis de dissociar e altamente abundantes em amostras renais inteiras. O TAL, por outro lado, estende-se por toda a medula externa14. Assim, a razão das frações PT e TAL representa uma boa medida para o enriquecimento dos tipos de células medulares em um conjunto de dados de célula única renal. Em geral, a relação PT/TAL em conjuntos de dados de rim inteiro unicelular variou de 8 (dados não publicados de tecido renal inteiro tratado com protease a frio) a 45 para tecido dissociado enzimaticamente10,14,15. No conjunto de dados snRNA-seq aqui apresentado foi possível atingir uma relação PT/TAL de 2. Este resultado ilustra que a remoção do excesso de córtex durante a dissecção tecidual combinada com snRNA-seq resulta em uma representação do tipo de célula renal notavelmente melhorada.
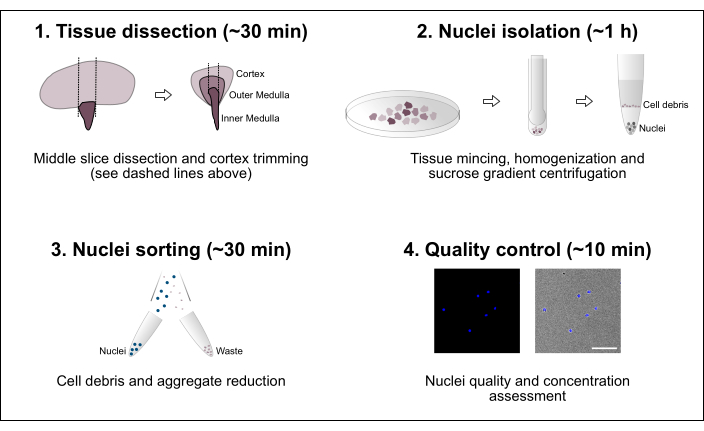
Figura 1: Visão geral esquemática do fluxo de trabalho. O protocolo consiste em quatro etapas principais que incluem dissecção tecidual seguida de isolamento de núcleos, classificação de núcleos e uma avaliação final de pureza e concentração. Barra de escala = 100 μm. Clique aqui para ver uma versão maior desta figura.
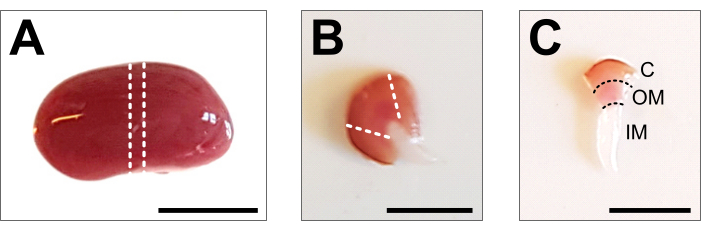
Figura 2: Dissecção renal e preparo tecidual . (A) Imagem representativa de todo o rim dissecado. As linhas pontilhadas indicam os cortes necessários para obter uma fatia média de 1-2 mm com uma representação de todos os tipos de células renais. (B) Imagem representativa da fatia média obtida. As linhas pontilhadas indicam os cortes para o corte do córtex do lado. (C) Imagem representativa da peça renal central com córtex aparado. O córtex (C), a medula externa (MO) e a medula interna (IM) são claramente visíveis. Barra de escala = 500 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Homogeneização tecidual e purificação de núcleos . (A) Imagem representativa mostrando tecido renal suficientemente picado. Barra de escala = 500 μm. (B) Homogeneizado após a primeira etapa de homogeneização (25 braçadas com pilão A, tubo moedor de 2 mL). (C) Homogeneizado após segunda etapa de homogeneização (15 braçadas com pilão B, tubo moedor de 2 mL). Por favor, clique aqui para ver uma versão maior desta figura.
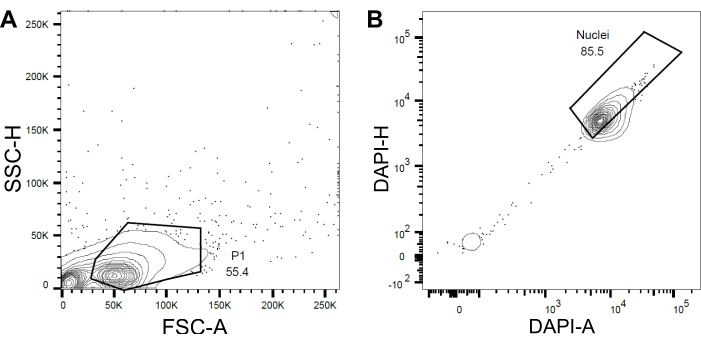
Figura 4: Estratégia de bloqueio para classificação de núcleos. (A ) Uma primeira porta P1 foi definida com base na dispersão dianteira (FSC) vs dispersão lateral (SSC) para excluir detritos e agregados. (B ) Uma porta subsequente baseada em DAPI-Área (DAPI-A) vs DAPI-Altura (DAPI-H) excluiu núcleos e múltiplos vazios ou danificados. Por favor, clique aqui para ver uma versão maior desta figura.
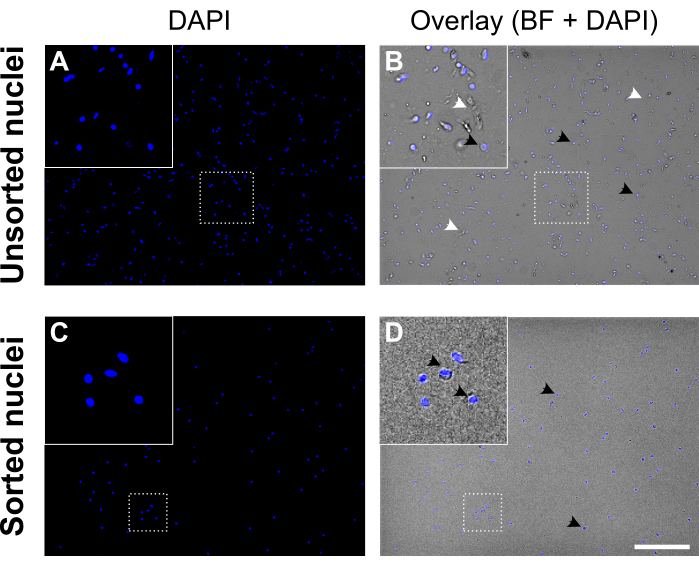
Figura 5: Suspensão dos núcleos antes e depois da classificação dos núcleos. (A, C) Núcleos corados com DAPI (azul). (B, D) Sobreposição de DAPI e canal de campo brilhante (BF). Antes da classificação (painel superior), a suspensão de núcleos contém detritos celulares e agregados (rotulados com pontas de seta brancas). Após a classificação (painel inferior) a suspensão dos núcleos parece muito mais limpa. Exemplos de núcleos corados com DAPI são rotulados com pontas de seta pretas. Núcleos de boa qualidade aparecem redondos e lisos com uma membrana intacta e são bem separados, enquanto núcleos de baixa qualidade parecem enrugados e apresentam perda da membrana nuclear. Barra de escala = 250 μm. Clique aqui para ver uma versão maior desta figura.
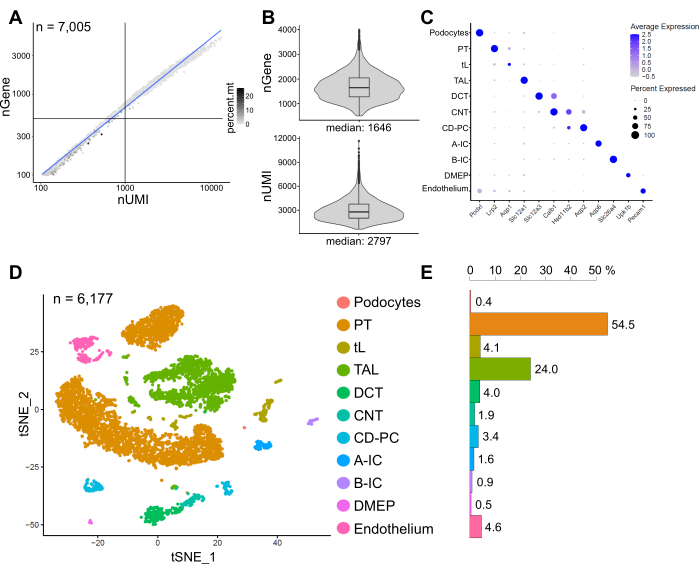
Figura 6: Controle de qualidade e análise de um conjunto de dados snRNA-seq representativo. (A) Número de genes (nGene) plotados contra o número de identificadores moleculares únicos (nUMI) coloridos pela fração de leituras mitocondriais (percent.mt). Núcleos de baixa qualidade correspondem ao quadrante inferior esquerdo da parcela (n = 828) e foram excluídos da análise subsequente. (B) Distribuição e mediana de nGene e nUMI detectados por núcleo no conjunto de dados snRNA-seq, representando 6.177 núcleos (> 500 genes). As bibliotecas foram sequenciadas a uma profundidade mediana de ~ 8.200 leituras mapeadas por núcleo. (C) Gráfico de pontos mostrando padrões de expressão gênica de marcadores enriquecidos com aglomerados (eixo x) para tipos de células individuais (eixo y). O tamanho do ponto corresponde à proporção de células que expressam o gene indicado. A cor corresponde à expressão média. (D) Gráfico de incorporação de vizinhos estocásticos distribuídos por T (t-SNE) de tipos de células identificados. (E) Distribuição do tipo de célula no conjunto de dados snRNA-seq. TP, túbulo proximal; tL, membro fino; TAL, membro ascendente espesso, DCT, túbulo contorcido distal; CNT, túbulo de conexão; CD-PC, células principais do ducto coletor; A-IC, células intercaladas do tipo A; B-IC, células intercaladas do tipo B; DMEP, epitélio medular profundo da pelve. Por favor, clique aqui para ver uma versão maior desta figura.
| Reagente | Concentração final | Volume (mL) | ||
| (A) 4 % BSA / PBS | ||||
| Salina tamponada com fosfato (PBS) com 10% de albumina bovina | 4% | 2 | ||
| PBS (Phosphate-Buffered Saline) 1X sem cálcio ou magnésio | - | 3 | ||
| (B) 0,04 % BSA / PBS | ||||
| 4 % BSA / PBS | 0.04 % | 0.5 | ||
| PBS (Phosphate-Buffered Saline) 1X sem cálcio ou magnésio | - | 49.5 | ||
| (C) Núcleo Lysis Buffer 1 (NLB1) | ||||
| Tampão de lise EZ nuclear | - | 4 | ||
| Inibidor da RNase RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| Complexo ribonucleosídeo-vanadilo (200 mM) | 10 mM | 0.2 | ||
| (D) Núcleo Lysis Buffer 2 (NLB2) | ||||
| Tampão de lise EZ nuclear | - | 4 | ||
| Inibidor da RNase RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| (E) Buffer de Suspensão de Núcleos (NSB) | ||||
| 0,04 % BSA / PBS | - | 2 | ||
| Inibidor da RNase RiboLock (40 U/μL) | 1 U/μL | 0.05 | ||
| F) Tampão gradiente de sacarose (10 % de sacarose) | ||||
| Peso 1 g de sacarose | ||||
| Dissolver em 6 ml de tampão nuclear de lise EZ | ||||
| Encha até 10 mL com Nuclear EZ Lysis Buffer | ||||
| Filtrar através de um filtro de seringa de 0,2 μm para um tubo fresco | ||||
Tabela 1: Receitas da solução: (A) Preparação de 4% BSA/1x PBS. Filtrar utilizando um filtro de seringa de membrana SFCA de 0,2 μm e manter no gelo até à sua utilização. (B) Preparação de 0,04% BSA/1 x PBS. Filtrar utilizando um filtro de seringa de membrana SFCA de 0,2 μm e manter no gelo até à sua utilização. (C) Preparação do Núcleo Lysis Buffer 1 (NLB1). Os volumes indicados são fornecidos por amostra. Mantenha no gelo até o uso. (D) Preparação do Buffer de Lise de Núcleos 2 (NLB2). Os volumes indicados são fornecidos por amostra. Adicione o Inibidor de RNase RiboLock ao NLB2 diretamente antes de usar, conforme observado no protocolo. Mantenha no gelo até o uso. (E) Preparação do Buffer de Suspensão de Núcleos (NSB). Os volumes indicados são fornecidos por amostra. Adicione o Inibidor de RNase RiboLock ao NSB diretamente antes do uso, conforme observado no protocolo. Mantenha no gelo até o uso. (F) Preparação de tampão gradiente de sacarose. Filtrar utilizando um filtro de seringa de membrana SFCA de 0,2 μm e manter no gelo até à sua utilização.
Discussão
A transcriptômica unicelular avança na compreensão da expressão gênica específica do tipo celular na fisiologia renal e na doença. Aqui, fornecemos um método simples e reprodutível para isolar núcleos únicos de alta qualidade do tecido renal de camundongo congelado para snRNA-seq de maneira padronizada.
Para o snRNA-seq, é fundamental usar núcleos de alta qualidade como entrada para a geração de bibliotecas e evitar a degradação do RNA durante o processamento tecidual. Portanto, a incubação de pedaços de tecido em solução de estabilização de RNA imediatamente após a dissecção é essencial para proteger e estabilizar o RNA celular e permite armazenar amostras a - 80 °C indefinidamente. Ao aplicar este protocolo a tecidos congelados sem tratamento com solução de estabilização de RNA, como material de arquivo, é necessário um ensaio clínico e a qualidade do RNA precisa ser avaliada, pois observamos uma perda significativa da integridade do RNA no tecido congelado sem incubação prévia na solução de estabilização de RNA.
Em geral, o manuseio adequado da amostra é crucial para maximizar a recuperação de núcleos únicos intactos. Todas as etapas de ressuspensão devem ser realizadas por pipetagem cuidadosa para evitar tensão de cisalhamento e danos físicos. Os tampões para a ressuspensão final dos núcleos e a classificação dos núcleos devem conter QUE para evitar a perda e agregação dos núcleos.
Os volumes tampão neste protocolo são otimizados para amostras de tecido muito pequenas (~15 mg). É fundamental garantir a lise celular completa e lavagem suficiente para gerar suspensões de alta qualidade. Blocos de tecido maiores ou amostras de rim inteiro resultarão em concentrações excessivas de núcleos que levam à aglomeração e agregação, alta abundância de RNA ambiente e má qualidade geral da suspensão. Se amostras maiores ou outros tecidos forem processados, é altamente recomendável realizar ensaios para determinar volumes de tampão ideais para níveis mínimos de RNA ambiente. A qualidade e as concentrações de núcleos e RNA precisam ser examinadas com cuidado, pois a sobrecarga resulta em um desempenho geral ruim.
Além disso, grandes quantidades de detritos celulares, causando altos níveis de RNA ambiente não associados a núcleos únicos, influenciam negativamente os resultados do sequenciamento. O esclarecimento da suspensão dos núcleos por centrifugação através de uma almofada de sacarose atenua esse problema até certo ponto, mas também pode levar a um viés na representação do tipo celular, contra-selecionando contra núcleos densos e pequenos presentes, por exemplo, em células imunes16. Se isso for preocupante, o gradiente de sacarose deve ser omitido. Por outro lado, descobrimos que a citometria de fluxo baseada na coloração DAPI foi fundamental para reduzir a quantidade de detritos celulares, a fim de produzir uma suspensão de núcleos únicos de alta qualidade.
O isolamento de núcleos únicos tem vantagens consideráveis quando comparado às abordagens unicelulares8. É compatível com tecido adequadamente congelado, tornando a coleção tecidual mais flexível, e contorna a necessidade de dissociação tecidual baseada em enzimas, o que pode introduzir respostas transcricionais ao estresse 6,17. Além disso, supera o viés de dissociação que favorece a seleção de tipos celulares facilmente dissociáveis do córtex renal, o que pode levar a uma sub-representação dos tipos de células medulares em algumas abordagens enzimáticas 5,6,10.
O uso de uma peça renal central em vez de tecido renal inteiro economiza ainda mais recursos e corrige a super-representação de tipos de células abundantes, conforme descrito anteriormente10. No entanto, dependendo do modelo de camundongo ou fenótipo investigado, pode ser benéfico usar amostras de rim inteiras em vez de uma única fatia média. Amostras de rim inteiro podem ser mais representativas de proporções de células verdadeiras, ou alterações que ocorrem em todo o rim, enquanto um corte médio aparado mostrou-se vantajoso para fenótipos medulares ou quando o material da amostra foi limitado. Essa decisão, portanto, é altamente específica do usuário e deve ser considerada com cuidado.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos à Plataforma de Genômica Científica do Centro Max Delbrück de Medicina Molecular da Associação Helmholtz, em Berlim, pelo apoio técnico.
JL e KMSO foram apoiados pelo Grupo de Formação em Investigação GRK 2318 da Fundação Alemã de Investigação (DFG) e pela Unidade de Investigação FOR 2841. A KMSO foi apoiada pelo Collaborative Research Grant 1365. A AB foi apoiada pelo financiamento do Prêmio Gottfried Wilhelm Leibniz da DFG concedido à NR.
Materiais
| Name | Company | Catalog Number | Comments |
| Cell sorter | - | - | For fluorescence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. |
| Centrifuge 5810 R | Eppendorf | 5811000015 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | Needs to contain DAPI light cube to count nuclei. Alternatively, nuclei can be counted manually under fluorescence microscope. |
| 4′,6-Diamidino-2-phenyl-indol-dihydrochlorid (DAPI) | Biotrend | 40043/b | Stock solution prepared with a concentration of 100 µM. Used for nuclei staining in a final concentration of 2 µM. |
| D(+)-Sucrose ≥99.9%, ultrapure DNAse-, RNAse-free | VWR | 0335-500G | |
| DNA LoBind Microcentrifuge Tubes (1.5 mL) | Eppendorf | 22431021 | |
| Ethanol, 70 % | - | - | |
| FACS tubes | pluriSelect | 43-10100-46 | |
| KIMBLE Dounce tissue grinder set 2 mL complete | Sigma-Aldrich | D8938-1SET | |
| Minisart Syringe Filters 0.2 µm | Sartorius | 16534-GUK | |
| Nuclease-free Water | Invitrogen | AM9937 | |
| Nuclei EZ Prep Nuclei Isolation Kit | Sigma-Aldrich | NUC-101 | Nuclei EZ Lysis Buffer (Product No. N3408) needed for buffer preparation. |
| PBS (Phosphate-Buffered Saline) 1X without calcium or magnesium | Corning | 21-040-CV | |
| Petri dishes, polystyrene 60 mm | Sigma-Aldrich | P5481 | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin | Sigma-Aldrich | SRE0036 | |
| pluriStrainer Mini 100 µm | pluriSelect | 43-10100-46 | |
| pluriStrainer Mini 20 µm | pluriSelect | 43-10020-40 | |
| pluriStrainer Mini 40 µm | pluriSelect | 43-10040-40 | |
| Polystyrene Centrifuge Tube (15 mL) | Falcon | 352099 | |
| Razor blades | - | - | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher | EO0384 | |
| Ribonucleoside-vanadyl complex | New England Biolabs | S1402S | Follow manufacturer's instructions (https://international.neb.com/products/s1402-ribonucleoside-vanadyl-complex#Product%20Information). Upon use the 200 mM stock solution is reconstituted to a green-black clear solution by incubating at 65 °C. |
| RNAlater Stabilization Solution | Invitrogen | AM7020 | |
| RNase AWAY | Fisher Scientific | 11952385 |
Referências
- Thomas, R. S. Kidney modeling and systems physiology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1 (2), 172-190 (2009).
- Potter, S. S. Single-cell RNA sequencing for the study of development, physiology and disease. Nature Reviews Nephrology. 14 (8), 479-492 (2018).
- Park, J., Liu, C. L., Kim, J., Susztak, K. Understanding the kidney one cell at a time. Kidney International. 96 (4), 862-870 (2019).
- Clark, A. R., Greka, A. The power of one: advances in single-cell genomics in the kidney. Nature Reviews Nephrology. 16 (2), 73-74 (2020).
- Lake, B. B., et al. A single-nucleus RNA-sequencing pipeline to decipher the molecular anatomy and pathophysiology of human kidneys. Nature Communications. 10 (1), 2832(2019).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 30 (1), 23-32 (2019).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144 (19), 3625-3632 (2017).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Muto, Y., et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nature Communications. 12 (1), 2190(2021).
- Hinze, C., et al. Kidney single-cell transcriptomes predict spatial corticomedullary gene expression and tissue osmolality gradients. Journal of the American Society of Nephrology. 32 (2), 291(2021).
- Berger, S. L. Isolation of cytoplasmic RNA: ribonucleoside-vanadyl complexes. Methods in Enzymology. 152, 227-234 (1987).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411-420 (2018).
- Stuart, T., et al. Comprehensive Integration of single-cell data. Cell. 177 (7), 1888-1902 (2019).
- Park, J., et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease. Science. 360 (6390), 758-763 (2018).
- Kirita, Y., Wu, H., Uchimura, K., Wilson, P. C., Humphreys, B. D. Cell profiling of mouse acute kidney injury reveals conserved cellular responses to injury. Proceedings of the National Academy of Sciences of the United States of America. 117 (27), 15874-15883 (2020).
- Schneeberger, S., et al. The neuroinflammatory interleukin-12 signaling pathway drives Alzheimer's disease-like pathology by perturbing oligodendrocyte survival and neuronal homeostasis. bioRxiv. , 441313(2021).
- Nguyen, Q. H., Pervolarakis, N., Nee, K., Kessenbrock, K. experimental considerations for single-cell RNA sequencing approaches. Frontiers in Cell and Developmental Biology. 6, 108(2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados