A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تقييم الركيزة في كل مكان بواسطة E3 Ubiquitin-ligase في تحلل خلايا الثدييات
In This Article
Summary
نحن نقدم بروتوكولا مفصلا لفحص الانتشار في كل مكان لركيزة معينة و E3 ubiquitin-ligase في خلايا الثدييات. تم استخدام خطوط الخلايا HEK293T للإفراط في التعبير عن البروتين ، وتم تنقية الركيزة متعددة التواجدات في كل مكان من تحلل الخلايا عن طريق الترسيب المناعي ، وتم حلها في SDS-PAGE. تم استخدام النشاف المناعي لتصور هذا التعديل بعد الترجمة.
Abstract
في كل مكان هو تعديل ما بعد الترجمة الذي يحدث في الخلايا حقيقية النواة التي تعتبر حاسمة لتنظيم العديد من المسارات البيولوجية، بما في ذلك بقاء الخلايا، وانتشار، والتمايز. إنها عملية عكسية تتكون من ارتباط تساهمي للأوبيكويتين بالركيزة من خلال تفاعل متتالي لثلاثة إنزيمات مختلفة على الأقل ، تتكون من E1 (إنزيم تنشيط Ubiquitin) ، E2 (إنزيم يوبيكويتين المقترن) ، و E3 (إنزيم Ubiquitin-ligase). يلعب مجمع E3 دورا مهما في التعرف على الركيزة والانتشار في كل مكان. هنا ، يتم وصف بروتوكول لتقييم انتشار الركيزة في خلايا الثدييات باستخدام النقل المشترك العابر للبلازميد الذي يشفر الركيزة المحددة ، و E3 ubiquitin ligase ، و ubiquitin المعلمة. قبل التحلل ، يتم التعامل مع الخلايا المنقولة باستخدام مثبط البروتيازوم MG132 (كاربوبنزوكسي-لو-لو-ليوسينال) لتجنب تدهور البروتيسومال الركيزة. علاوة على ذلك ، يتم تقديم مستخلص الخلايا إلى الترسيب المناعي على نطاق صغير (IP) لتنقية الركيزة متعددة الكل كل مكان للكشف عنها لاحقا عن طريق النشاف الغربي (WB) باستخدام أجسام مضادة محددة لعلامة ubiquitin. وبالتالي ، يتم وصف بروتوكول متسق وغير معقد لفحص الانتشار في كل مكان في خلايا الثدييات لمساعدة العلماء في معالجة انتشار ركائز محددة وغازات E3 في كل مكان.
Introduction
تعد التعديلات اللاحقة للترجمة (PTMs) آلية مهمة فيما يتعلق بتنظيم البروتين ، وهو أمر ضروري لتوازن الخلايا. انتشار البروتين في كل مكان هو تعديل ديناميكي ومعقد يخلق مجموعة متنوعة من الإشارات المختلفة مما يؤدي إلى العديد من النتائج الخلوية في الكائنات حقيقية النواة. في كل مكان هو عملية عكسية تتكون من ربط بروتين يوبيكويتين يحتوي على 76 من الأحماض الأمينية بالركيزة ، ويحدث في سلسلة إنزيمية تتكون من ثلاثة تفاعلات متميزة1. تتميز الخطوة الأولى بتنشيط يوبيكويتين ، والذي يعتمد على التحلل المائي ATP لتشكيل يوبيكويتين عالي الطاقة مرتبط بالثيوستر بين ubiquitin C-terminus وبقايا السيستين الموجودة في الموقع النشط لإنزيم E1. في وقت لاحق ، يتم نقل يوبيكويتين إلى إنزيم E2 الذي يشكل مركبا محبوبا بالثيوستر مع يوبيكوتين. بعد ذلك ، يتم ربط ubiquitin تساهميا بالركيزة بواسطة E2 ، أو في كثير من الأحيان ، بواسطة إنزيم E3 ، الذي يتعرف على الركيزة 2,3 ويتفاعل معها. في بعض الأحيان ، تكون إنزيمات E4 (عوامل استطالة سلسلة Ubiquitin) ضرورية لتعزيز تجميع سلسلة multiubiquitin3.
يحتوي Ubiquitin على سبعة بقايا ليسين (K6 و K11 و K27 و K29 و K33 و K48 و K63) ، مما يسمح بتكوين سلاسل polyubiquitin التي تولد روابط متميزة لإنتاج هياكل ثلاثية الأبعاد مختلفة سيتم التعرف عليها بواسطة العديد من البروتينات المستجيبة 4,5. وبالتالي ، فإن نوع سلسلة polyubiquitin التي تم إدخالها في الركيزة ضروري لتحديد مصير الخلية 6,7,8. علاوة على ذلك ، يمكن أيضا نشر الركيزة في كل مكان من خلال بقاياها الطرفية N التي تسمى N-degrons. الغازات المتساوية في كل مكان E3 المحددة هي المسؤولة عن التعرف على N-degron ، مما يسمح بتعدد الوعل في كل مكان لبقايا الليسينالقريبة 9.
في الوقت الحاضر ، هناك أكثر من 40 ركيزة مختلفة خاصة ب SCF مميزة. من بين هؤلاء ، يمكن العثور على المنظمين الرئيسيين للعديد من المسارات البيولوجية ، بما في ذلك تمايز الخلايا وتطورها بالإضافة إلى بقاء الخلايا وموتها ،10،11،12،13. وبالتالي ، فإن تحديد ركائز محددة لكل E3 ubiquitin-ligase أمر ضروري لتصميم خريطة شاملة لمختلف الأحداث البيولوجية. على الرغم من أن تحديد الركائز الحقيقية يمثل تحديا كيميائيا حيويا ، إلا أن استخدام الأساليب القائمة على الكيمياء الحيوية مناسب جدا لتقييم خصوصية السلسلة والتمييز بين أحادية ومتعددة في كل مكان14. تصف هذه الدراسة بروتوكولا كاملا لفحص الانتشار في كل مكان باستخدام خط خلية الثدييات HEK293T الذي يبالغ في التعبير عن الركيزة UXT-V2 (التي يتم التعبير عنها في كل مكان مثل المرافقة الشبيهة بالبريفولدين isoform 2) مع مركب E3 ubiquitin-ligase SCF (Fbxo7). UXT-V2 هو عامل مساعد أساسي لإشارات NF-κB ، وبمجرد هدم هذا البروتين في الخلايا ، فإنه يمنع تنشيط NF-κB الناجم عن TNF α11. وبالتالي ، للكشف عن UXT-V2 متعدد الانتشار ، يتم استخدام مثبط البروتيازوم MG132 لأنه لديه القدرة على منع النشاط البروتيني للوحدة الفرعية 26S من مجمع البروتيازوم15. علاوة على ذلك ، يتم تقديم مستخلص الخلايا إلى IP صغير النطاق لتنقية الركيزة ، باستخدام جسم مضاد محدد مثبت إلى راتنج الأغاروز للكشف عنه لاحقا من قبل البنك الدولي باستخدام أجسام مضادة مختارة. هذا البروتوكول مفيد جدا للتحقق من صحة انتشار الركيزة في البيئة الخلوية ، ويمكن أيضا تكييفه مع أنواع مختلفة من خلايا الثدييات وغيرها من مجمعات E3 ubiquitin-ligase. ومع ذلك ، من الضروري التحقق من صحة الركيزة التي تم اختبارها من خلال فحص الانتشار في المختبر أيضا ، لأن كلا البروتوكولين يكملان بعضهما البعض فيما يتعلق بتحديد الركائز الحقيقية.
Protocol
ملاحظة: يتم تمثيل نظرة عامة على بروتوكول فحص الانتشار في كل مكان في خلايا الثدييات في الشكل 1.
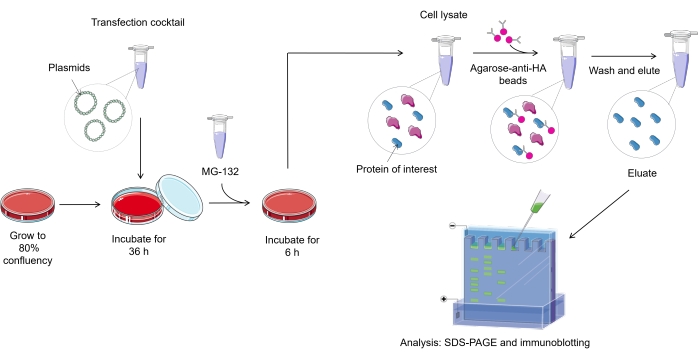
الشكل 1. نظرة عامة على إجراء فحص الانتشار في كل مكان. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
1. زراعة الخلايا
- قم بزراعة خط الخلايا HEK293T في طبق استزراع معالج بتقنية TC مقاس 100 مم إلى التقاء بنسبة 80٪ -90٪ في وسائط النمو (الجلوكوز العالي من Dulbecco's Modified Eagle's Medium (DMEM) مع 10٪ من مصل البقر الجنيني (FBS) والبنسلين (100 وحدة) والستربتومايسين (100 ميكروغرام) والجلوتامين L (0.292 مجم / مل)). احتضان الثقافة عند 37 درجة مئوية في حاضنة زراعة الخلايا الرطبة بنسبة 5٪ CO2.
- لتمرير الخلايا ، استنشق الوسائط من طبق الثقافة باستخدام ماصة مصلية. اغسل الخلايا مرة واحدة ب 1 مل من محلول ملحي معقم 1x مخزن بالفوسفات (PBS 1x).
- افصل الخلايا عن طريق إضافة 1 مل من محلول التربسين / EDTA (حمض الإيثيلين ديامين رباعي الأسيتيك). احتضن الطبق على حرارة 37 درجة مئوية لمدة 5 دقائق. أعد تعليق الخلايا باستخدام ماصة مصلية في 2 مل من وسائط النمو.
- انقل نظام التعليق الخلوي إلى أنبوب جديد ونظيف سعة 15 مل وأجهزة طرد مركزي بسرعة 500 × جم لمدة 5 دقائق في درجة حرارة الغرفة (RT). قم بإزالة السوبرناتانت عن طريق سكبه بعناية. أعد تعليق حبيبات الخلية بلطف في 3 مل من وسائط النمو عن طريق السحب لأعلى ولأسفل للحصول على تعليق خلوي متجانس.
- انقل 1 مل من تعليق الخلية إلى طبق استزراع معالج بتقنية TC مقاس 100 مم يحتوي على 9 مل من وسائط النمو.
ملاحظة: إذا كانت الخلايا في ظروف جيدة ، فإن كل طبق استزراع من HEK293T ، حيث يكون الالتقاء 80٪ -90٪ ، يمكن أن يولد ثلاثة أطباق استزراع مع التقاء 80٪ بعد يومين من المرور.
2. نقل الخلايا
ملاحظة: لا ينصح بنقل مزرعة الخلايا إذا كان الالتقاء الذي تم الوصول إليه أقل من 80٪.
- قبل النقل ، قم بالتصديق على ما إذا كانت الخلايا خالية من التلوث وعند التقاء مناسب لعمليات النقل العابرة.
- لكل عينة نقل ، قم بإعداد مجمعات الحمض النووي - البولي إيثيلينيمين (PEI 1 ميكروغرام / ميكرولتر عند درجة الحموضة 7.2) على النحو التالي:
- تمييع 3 ميكروغرام من كل بلازميد في 100 ميكرولتر من opti-MEM I خفضت وسط المصل دون مكملات ومزجها بلطف عن طريق سحب المحلول لأعلى ولأسفل.
ملاحظة: هنا ، تم نقل الخلايا مع 4 ميكروغرام من كل بلازميد: ناقلات فارغة (pcDNA3) أو هياكل FLAG-Fbxo7 و UXT-V2-HA ، مع أو بدون 6xHis-myc-ubiquitin. كان إجمالي محتوى الحمض النووي 12 ميكروغرام. - قم بإذابة جزيرة الأمير إدوارد في RT وأضفها إلى المحلول بعد نسبة 3 ميكرولتر من جزيرة الأمير إدوارد لكل 1 ميكروغرام من الحمض النووي. تجانس الحل عن طريق السحب لأعلى ولأسفل. ثم احتضنها لمدة 15 دقيقة في RT للسماح بتكوين مجمعات DNA-PEI.
ملاحظة: تختلف النسبة المثلى لحجم جزيرة الأمير إدوارد لكل كمية من الحمض النووي وفقا لخط الخلية المحدد.
- تمييع 3 ميكروغرام من كل بلازميد في 100 ميكرولتر من opti-MEM I خفضت وسط المصل دون مكملات ومزجها بلطف عن طريق سحب المحلول لأعلى ولأسفل.
- أضف الحجم الإجمالي لمجمعات DNA-PEI إلى كل طبق يحتوي على مزرعة الخلايا واخلطها بلطف عن طريق هز الطبق ذهابا وإيابا. احتضان الخلايا عند 37 درجة مئوية في حاضنة زراعة الخلايا الرطبة عند 5٪ CO2.
- استبدل وسط النمو بعد 5 ساعات ، لأن التعرض المطول لجزيرة الأمير إدوارد يمكن أن يكون ساما لخلايا HEK293T. احتضان الخلايا عند 37 درجة مئوية في حاضنة زراعة الخلايا الرطبة عند 5٪ CO2 لمدة 36 ساعة.
3. تحلل الخلايا والترسيب المناعي
- بعد فترة الحضانة و 6 ساعات قبل تحلل الخلايا ، عالج الخلايا المنقولة ب 10 ميكرومتر من مثبط البروتيازوم MG-132. مرة أخرى ، احتضن الخلايا عند 37 درجة مئوية في حاضنة زراعة الخلايا الرطبة بنسبة 5٪ CO2.
- استنشق الوسائط من كل طبق استزراع باستخدام ماصة مصلية واغسلها مرة واحدة باستخدام 1 مل من 1x PBS. افصل الخلايا بإضافة 1 مل من التربسين واحتضان الطبق عند 37 درجة مئوية لمدة 5 دقائق. أعد تعليق الخلايا في 1 مل من وسائط النمو.
- انقل تعليق الخلية إلى أنبوب 15 مل جديد ونظيف وقم بطرده مركزيا عند 500 × g لمدة 5 دقائق في RT.
- قم بإزالة السوبرناتانت عن طريق سكبه بعناية. أعد تعليق حبيبات الخلية بلطف في 200 ميكرولتر من المخزن المؤقت لتحلل NP-40 البارد (50 mM Tris-HCl pH 7.2 و 225 mM KCl و 1٪ NP-40) ، مع كوكتيل مثبطات البروتياز والفوسفاتيز (10 mM NaF و 1 mM Na3VO4) ، وانقل المحلول إلى أنبوب دقيق نظيف 1.5 مل.
- احتضن الخلية محللة لمدة 30 دقيقة على الجليد. بعد الحضانة ، قم بالطرد المركزي للخلية محللة عند 16900 × g لمدة 20 دقيقة عند 4 درجات مئوية.
- وفي الوقت نفسه ، قم بموازنة حبات الأغاروز المضادة للحمض الهيدروجيني مع المخزن المؤقت للتحلل NP-40 البارد على الجليد. استخدم 15 ميكرولتر من حبات الأغاروز المضادة للحمض الوراثي لكل عينة. اغسل الخرز ب 200 ميكرولتر من المخزن المؤقت لتحلل NP-40 عن طريق نبضه في أنبوب جهاز طرد مركزي دقيق عند 3000 × g لمدة 1 دقيقة عند 4 درجات مئوية. مع ماصة ، استنشق وتخلص من supernatant بعناية فائقة ؛ كرر هذه العملية ثلاث مرات. بعد ذلك ، حافظ على توازن الخرز على الثلج حتى الاستخدام.
- بعد الطرد المركزي للخلية ، قم باستعادة supernatant. حدد كمية محتوى البروتين في المحلول الكلي باستخدام طريقة برادفورد16.
- تأكد من أن كل عينة تخضع لهطول الأمطار المناعية تقدم كمية متساوية من البروتين. احتضن الحجم اللازم من تحلل الخلايا مع حبات الأغاروز المتوازنة المضادة ل HA لمدة 4 ساعات ، مع الدوران بلطف في حاضنة دوارة عند 4 درجات مئوية ، مما يسمح ل UXT-V2-HA بالارتباط بحبات الأغاروز المضادة للحمض النووي.
- اجمع حبات الأغاروز المضادة للحمض الوراثي عن طريق نبضها في أنبوب جهاز طرد مركزي دقيق عند 3000 × جم لمدة 1 دقيقة عند 4 درجات مئوية. استنشق بعناية وتخلص من supernatant. اغسل الخرز ثلاث مرات باستخدام المخزن المؤقت لتحلل الخلايا NP-40 البارد ومرتين باستخدام المخزن المؤقت FLAG / HA البارد (10 mM Hepes pH 7.9 و 15 mM MgCl2 و 225 mM KCl و 0.1٪ NP-40).
- بعد الغسيل النهائي ، قم بإزالة جميع المواد الفائقة بعناية باستخدام ماصة وقم بالتخلص من البروتين متعدد الكل مع ببتيد HA (300 ميكروغرام / مل) المخفف على المخزن المؤقت FLAG / HA. احتضن حبات الأغاروز المضادة ل HA مع ببتيد HA لمدة 1 ساعة عند 4 درجات مئوية في منصة اهتزاز هزازة.
- قم بتدوير الخرز عند 3000 × جم لمدة 2 دقيقة عند 4 درجات مئوية وقم بماصة السوبر نات التي تحتوي على البروتينات متعددة الكل في كل مكان بعناية. إذا لزم الأمر ، قم بتخزين اللوات في أنبوب دقيق جديد ونظيف عند -20 درجة مئوية.
- حل الإيلوات وتحلل الخلية في 10٪ SDS-PAGE (كبريتات الصوديوم دوديسيل بولي أكريلاميد هلام الكهربائي الكهربائي )17 والنشاف المناعي.
- في هذه الدراسة ، تم إجراء نقل رطب WB. لهذا النوع من النقل ، ضع الجل في شطيرة نقل تتكون من ورق ترشيح - هلام - غشاء - ورق ترشيح ، وقم بتبطينه بمنصات ، واضغط عليه معا بواسطة شبكة دعم. ضع هذا النظام عموديا في خزان مملوء بمخزن مؤقت للنقل وبين أقطاب أسلاك الفولاذ المقاوم للصدأ / البلاتين. يحدث النقل لمدة 90 دقيقة عند 150 فولت في مخزن النقل الرطب (الجلايسين 192 مللي متر ، قاعدة التريس 25 ملليمتر ، 0.025٪ SDS ، 20٪ ميثانول).
ملاحظة: نظرا لأن مستخلصات الخلايا قد تم تحديدها كميا (الخطوة 3.7)، قم بتشغيل SDS-PAGE بكمية متساوية من البروتين لكل عينة. أيضا، لحل التوضيح، قم بتشغيل وحدات تخزين متساوية لكل عينة. - التحقيق في غشاء اللطخة المناعية باستخدام أجسام مضادة مختارة 11. تأكد من اكتشاف إشارة مسحة في اللطاخة من الركيزة متعددة الكل في كل مكان التي تم سحبها في عملية IP باستخدام الأجسام المضادة المضادة للميك في العينات التي تحتوي على ليغاز E3 من النوع البري والركيزة و myc-ub. في محللات الخلية (المدخلات) ، تأكد من اكتشاف الإشارة من الركيزة المختارة ، وبروتين Fbxo7 ، والبروتينات الموجودة في كل مكان ، وبروتين التدبير المنزلي (على سبيل المثال ، GAPDH و β-actin) لضمان نفس الكمية من البروتينات في كل حارة.
ملاحظة: تم إعداد التخفيف لكل جسم مضاد مستخدم وفقا لتعليمات الشركة المصنعة.
النتائج
UXT (نسخة يتم التعبير عنها في كل مكان) هو بروتين يشبه ما قبل الفولدين يشكل مجمعات قابلة للطي للبروتين يتم التعبير عنها في كل مكان في أنسجة الفئران والبشر مثل القلب والدماغ والعضلات الهيكلية والمشيمة والبنكرياس والكلى والكبد18. تم وصف شكلين متساويين للربط من UXT ، يطلق عليهما UXT-V1 و...
Discussion
يعد التواجد في كل مكان تعديلا أساسيا بعد الترجمة ينظم مستويات العديد من البروتينات ويلعب دورا حاسما في العديد من مسارات الإشارات والعمليات البيولوجية ، مما يضمن بيئة صحية داخل الخلايا. يعد نظام يوبيكوتين بروتيازوم (UPS) أحد المحاور الرئيسية للبحوث الصيدلانية الحديثة ، مما يوفر إمكانية تثب...
Disclosures
ويعلن صاحبا البلاغ أنه لا يوجد تضارب في المصالح.
Acknowledgements
F.R.T مدعوم بمنحة FAPESP رقم 2020/15771-6 و CNPq Universal 405836/2018-0. يتم دعم P.M.S.P و V.S بواسطة CAPES. تم دعم C.R.S.T.B.C بمنحة FAPESP رقم 2019/23466-1. نشكر ساندرا ر. س. ماروياما (FAPESP 2016/20258-0) على الدعم المادي.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
References
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved